摘要
背景
非寄主抗性(NHR)为全球粮食安全提供了一种引人注目的长期植物保护策略,但对NHR的遗传基础仍知之甚少。用于许多疾病,包括小麦茎锈病[致病有机体]小麦锈菌(Pg)], NHR在很大程度上是未被探索的,因为开发一种遗传上可控制的系统,其中抗性隔离的固有挑战。目前的研究转向病原体的替代宿主,小檗(小檗属植物Spp .),以克服这一挑战。
结果
在本研究中,一个种间映射种群来源于之间的杂交Pg防小檗属植物thunbergii(英国电信),Pg易受影响b .寻常的是为了调查Pg-NHR由英国电信。为了便于QTL分析和后续的性状解剖,构建了两个亲本种的第一个遗传连锁图,并建立了染色体尺度的参考基因组英国电信组装(PacBio + Hi-C)。QTL分析结果显示,在其短臂上鉴定了一个13 cM的区域(约5.1 Mbp,横跨13个物理contigs)英国电信3号染色体。基因差异表达分析结合两个亲本种之间的序列变异分析,在QTL区域内确定了几个候选基因的优先级,其中一些属于先前涉及抗病的基因家族。
结论
基础遗传和基因组资源开发小檗属植物spp.使QTL的识别和注释与Pg-NHR。虽然还需要后续的验证和精细映射研究,但本研究证明了解剖的可行性,并为解剖奠定了基础Pg-NHR是农业中最具破坏性的病原体之一。
背景
茎锈病,由真菌病原体引起小麦锈菌(Pg),数千年来一直是小麦及相关小谷物最具破坏性的疾病之一[1,2,3.].在二十世纪中叶,通过协同开发抗性小麦品种和去除小麦锈病,实现了对该病的有效控制Pg的替代宿主,小檗(小檗属植物寻常的L.),源自主要小麦产地[3.,4].然而,在过去20年里,新的强毒茎锈病品种的出现使一些长期使用的抗性基因失效[5,6].例如,当小麦茎锈蚀赛跑Ug991998年在东非首次发现,据估计,世界上80%以上的小麦种质资源对其广泛应用的抗性基因具有前所未有的毒性Sr31[7].的快速分布和持续演化Ug99种族家族,结合最近欧洲茎锈病爆发[8],强调需要寻找新的耐药性来源[9].传统上,这种新的资源几乎完全是从多样化的资源中寻找的小麦属植物下。尽管小麦改良的可转译性可能不那么直接,甚至可能无法实现,但一种补充的方法可能会超越这一基因库,寻找对复合物的非宿主抗性(NHR)的潜在机制Pg致病源
NHR是一种抗性形式,其中潜在宿主物种的所有个体对潜在病原体的所有个体(例如种族)表现出免疫[10].作为最常见的抗病形式和具有内在持久性的抗病形式,NHR为实现对许多植物病原体的广谱持久保护提供了令人信服的策略,包括小麦茎锈病的致病有机体[11,12].潜在的遗传机制Pg-NHR在很大程度上仍是未知的,特别是与研究相对充分的种族特异性和定量、种族非特异性宿主抗性机制相比。然而,在过去的十年中,人们通过各种模型和非模型植物来了解NHR对锈病病原体的影响。许多植物种类,包括拟南芥,Brachypodium distachyon、大米、大麦和豇豆[13,14,15,16,17,18],已被用来研究NHRp . striiformisf . sp。tritici小麦条锈病的病原。与之相反,NHR对小麦茎锈病病原菌有抑制作用Pg迄今为止只在水稻中进行了研究[13],区别于中间体的研究Pg在大麦和b . distachyon[19,20.].
作为唯一对所有已知锈病免疫的全球重要小谷物,水稻(选用的逻辑潜在来源Pg-NHR基因。遗传研究Pg-NHR在水稻中是困难的,然而,正是因为根据定义,非寄主群体无法隔离产生抗性。虽然有些进展有限Pg在水稻中已发现感染,因此提高了解剖的可能性Pg-NHR在该系统中,感染过程几乎没有变化,需要繁琐的微观研究来表征,最终无法完成[13].作为大米的替代品小檗属植物-Pg系统是近年来提出的一种易于处理的病理系统Pg-NHR [21].大量物种在高度多样化小檗属植物或小檗属易受感染Pg感染(如欧洲小檗b .寻常的L., 20世纪小麦种植区大规模消灭的目标)[22,23].然而,其他的则被认为是非东道主。日本伏牛花b . thunbergii直流。,for example, is considered a non-host ofPg1)经过美国农业部谷物疾病实验室近一个世纪的广泛测试,答案是否定的Pg曾在该物种中观察到感染[24,25,26,27,28,29,30.,31,32,332)没有Pg已观察到感染b . thunbergii在自然条件下,尽管物种在景观中猖獗繁殖。因为这种寄主和非寄主物种之间的杂交已知发生在自然界中(例如。B。×ottawensis沙伊德)[34],种间小檗杂交种群体的研究为研究小檗的遗传基础提供了一种潜在的手段Pg-NHR。
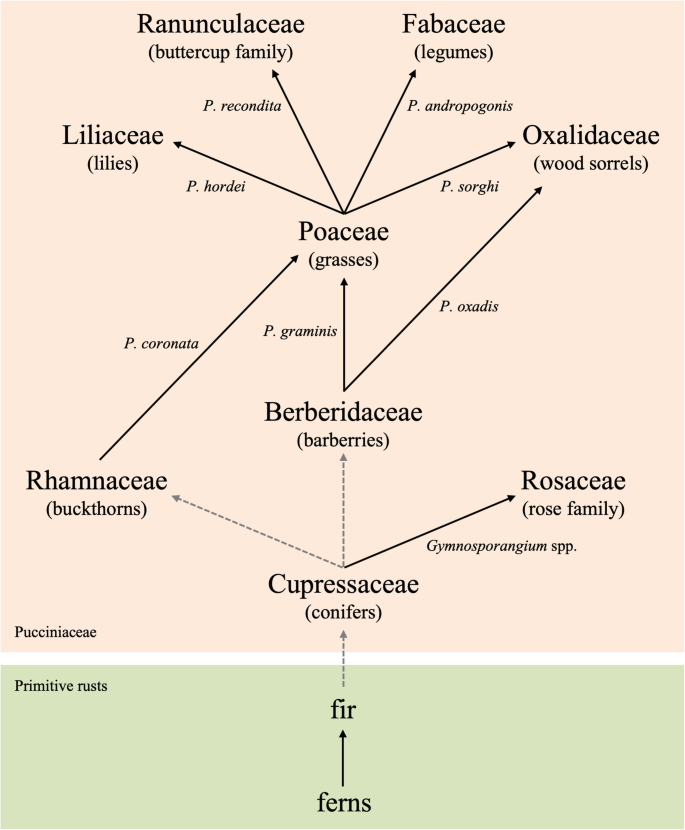
由于其他原因,barberries也是一个引人注目的模型。不像水稻,水稻和Pg在美国,barberries被认为是第一批被锈病寄生的eudicots之一(图。1).事实上,多项证据支持这样的观点,即倒钩莓可能在锈菌的进化中发挥了重要作用。首先,小檗属植物植物宿主锈病的多样性广泛,包括许多大环,异种种柄锈菌(如。Pg, P. striiformis, P. montanensis, P. brachypodii, P. pigmea, P. koeleriae,p . arrhenatheri)、一些自体锈病(例如:Cumminsiella属,属于蒲草科;Edythea属,属于蛇草科;而且Pucciniosira锈斑属(puciniosiraceae),甚至还有一些变质锈斑(例如锈斑)。Acedidium而且Uredospp)。其次,各种大环锈菌的端孢子之间仅存在微小的形态差异[35],表明这些病原体有单一的进化起源。第三,最近的古植物学发现b . wuyunensis从中国东北5500万至6500万年前的沉积物层中发现的证据表明,这种植物是最早的被子植物群之一[36].
具体到草锈病,已知有八种柄锈菌在小檗上完成有性(无性)阶段,在禾本科禾本科植物上完成无性(有性和无性)阶段。这种关系,结合这两个植物科的相对年龄,表明柄锈菌spp.可能在其宿主扩展到草之前寄生在小檗科。今天,属柄锈菌由2000多个物种组成;在这个多样化的属中,宿主跳跃而不是共同物种形成被认为是物种形成的主要手段[37].作为最近的例子,宿主从禾本科跳转到毛茛科可能产生p . recondita复合体及其排列的种,很可能是由百合科进化而来的p . hordei和其排列的物种,并可能产生跳转到草科p . sorghi和排列的物种。因为倒钩莓和铁锈之间的关系很可能在这种物种形成之前就存在了。1),探讨一些当代小檗的NHR机制具有重要意义。
在本研究中,种间B。×ottawensis建立了定位群体,以研究假定基础基因的遗传Pg -NHR的b . thunbergii.为了支持这项工作,研究人员开发了必要的遗传和基因组资源,包括两个亲本种的遗传连锁图(b . thunbergii而且b .寻常的)和染色体尺度的参考基因组b . thunbergii.本研究不仅为研究的开展奠定了基础资源小檗属植物-Pg还展示了它们在初步解剖中的用途Pg-NHR,长期希望有助于洞察对茎锈病病原体持久抗性的可能新机制。
结果
变异检测和连锁图构建
为两个亲本系(b .寻常的继承“马车山”和b . thunbergii' BtUCONN1 ')及其182个种间B。×ottawensisF1生成总共60gb的数据[~ 4.01亿个150-bp成对端(PE)读取]。经过高质量的解析和多路复用,GBS-SNP-CROP管道平均每个基因型保留了300万个高质量的reads [38](附加文件1).使用来自两个亲本的高质量读取,生成了一个由87089个质心(即一致的GBS片段)组成的模拟参考(MR),总长度约为15.4 Mbp。
共检测到15411个多态性标记,其中14043个SNPs(平均深度D单核苷酸多态性= 41.5)和1368个索引(Dindels= 36.4),通过将总体的所有高质量reads映射到mr来识别。表中提供了通过一系列过滤器筛选这些标记以获得用于链接映射构建的最终标记集的详细说明1.利用双向伪测试交叉映射策略,为每个亲本种构建单独的遗传连锁图[39].剔除个体F后1> 30%缺失数据的子代,保留161个和162个b . thunbergii而且b .寻常的分别构建链接映射。的b . thunbergii地图共使用1757个标记(分别来自标记集1和2的1497和260个;见表1),以及b .寻常的地图共使用706个标记(分别来自标记集3和4的600和106)构建。对于这两个亲本种,其余标记合并成14个不同的连锁群,与报道的染色体数一致小檗属植物spp.(附加文件2:图S1)。
两种遗传连锁图的汇总统计见表2.的b . thunbergii地图由598个重组箱(即映射位点)组成,总长度为1474厘米。在14个连锁群(LG14)中,每个连锁群的箱数从23个(LG14)到60个(LG2)不等,相邻箱间的平均距离为2.6 cM。相比之下,b .寻常的地图由347个箱子组成,总长1714厘米。在这14个LG14中,每个容器的数量从13 (LG14)到37 (LG2)不等,相邻容器之间的平均距离为5.5 cM。附加文件中提供了标记名称、等位基因和遗传位置(cM),以及映射种群中所有成员中重组事件的彩色编码可视化3.(b . thunbergii)及附加文件4(b .寻常的).
疾病表现型
确定疾病的反应Pg,家长和所有F1用越冬胚芽萌发的胚芽孢子喷射出的担子孢子接种子代Pg在自然感染上发现大被.后代分离成四个明确的表型类,从抗性到易感(图。2、表3.).用于构建连锁图谱的153个子代成功获得了疾病表型。其中25例表现出明显的耐药反应b . thunbergii父(图。2C)和61表现出明显的敏感反应,类似于b .寻常的父(图。2f).在其余67个品系中,38个表现出中等抗性(图2)。2d), 29例表现为中度易感性(图。2e)。
两个作图群体父母及其F1后代。一个抗性反应b . thunbergii登记' BtUCONN1 ',未显示任何视觉症状;b敏感反应b .寻常的登上“马车山”,上叶表面有浓密的皮草,下叶表面有多产、发育良好的毛虫草;c抵抗反应(4分制中1分)B。×ottawensis后代WH15-039,斑点稀疏;d中度耐药反应(2分)B。×ottawensis子代WH15-063,显示明显坏死病变和部分脓毒症形成;e中度敏感反应(得分3)B。×ottawensis子代‘WH15-128’,显示发育良好的脓肿和脓肿,以及稀疏的坏死病灶;而且f易感反应(4分)B。×ottawensis子代‘WH15-149’,呈发育良好的脓肿和脓肿,无明显坏死。所有照片均于接种后14天拍摄
QTL分析
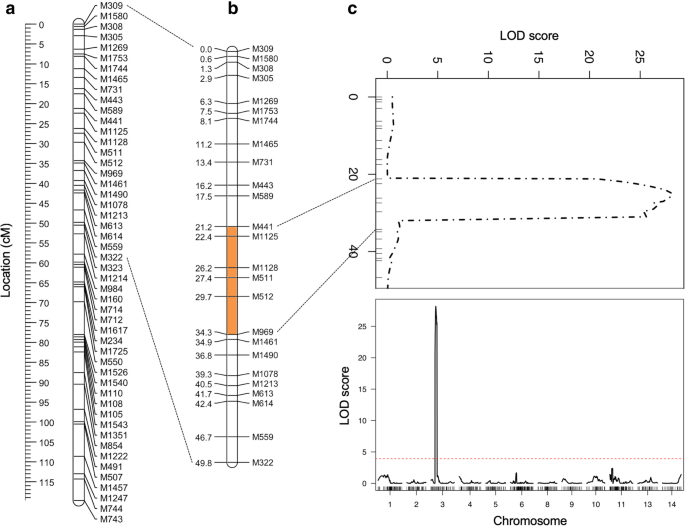
映射与之关联的区域Pg-NHR在b . thunbergii,利用上述父本和茎锈病4点反应型的连锁图谱进行了复合区间映射(CIM)分析。基于排列分析得出的LOD阈值为3.9,CIM分析发现了一个显著QTL(峰值LOD值为28.2),位于离短臂端粒25 cM处b . thunbergii染色体3(图;3.).这个13cm QTL区域的侧翼标记,以下简称为QPgr-3S,通过对F1在QTL标记峰值两侧有重组事件的个体M1128.远端侧翼标记M441由Pg-抗性个体WH15-192,以及近端侧翼标记M969由Pg-抗性个体WH15-101(附加文件3.).未检测到显著QTLb .寻常的地图.
建立一个参考基因组b . thunbergii简历。“小鬼”
从115个PacBio单分子实时(SMRT)细胞(RS II上的P6-C4化学)中生成了大约129 Gb的序列数据,平均读取长度为10,409 bp, N50为15,021 bp(额外文件)2:表S1)。狗头人的单倍体基因组大小,一种广泛的绿叶b . thunbergii根据k-mer分析估计为1.37 Gbp,根据流式细胞仪估计为1.72 Gb(数据未显示),这两个值与先前发表的结果相结合b . thunbergii单倍体基因组大小(1C)为1.51 Gb [40].FALCON-Unzip管道[41]的结果为1.36 Gb,由4671个主contig组成,contig长度N50为0.67 Mbp4).它们对应的7144个相相单倍体的总长度为0.88 Gb,约占主contig空间的64%。进一步以嵌合体断裂和神秘单倍体鉴定的形式(见材料和方法)进行筛选,最终得到1.23磅的装配,由2698个主contigs组成,contig长度N50为0.76 Mbp4).最终组装中的haplotig数量增加到8790,总长度为0.99 Gb(>主contig空间的80%)。
基因组完整性和污染分析显示,最终的基因组组装质量可接受,具有BUSCO核心植物基因集的80.9%的完整表达,只有15.1%的BUSCO基因缺失。83.0%的BtUCONN1 GBS片段、80.71%的PacBio preads和92.2%的RNA-seq数据(以适当的配对方式)与最终组装对齐。经FALCON-Unzip组装后,119个主contigs与植物cpDNA和mtDNA序列具有显著的序列相似性;但由于密集的单倍体清洗和整理,这个数字在最终组装中下降到只有一个主contig。
基于染色体构象捕获分析(Hi-C)获得的三维接近信息,最终组装中的主要contigs被引导到染色体水平的支架(伪分子)[42].在2698个初代contigs中,97%(2611个contigs, 1.20 Gbp)成功组装成14个伪分子,代表14条染色体b . thunbergii,如Hi-C热图所示2:图S2)。其余3%(156个contigs, 33.5 Mbp)被指定为无支架contigs。组成的14个伪分子的详细摘要统计b . thunbergii简历。“狗头人”参考程序集可以在附加文件中找到2:表S2。
将遗传连锁映射到物理装配上并分配染色体数目
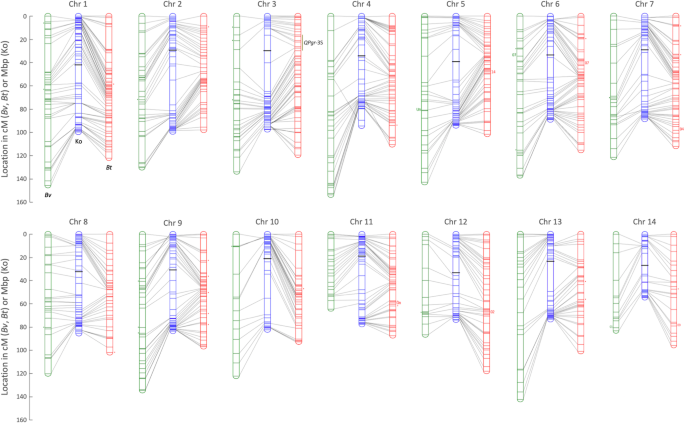
使用BLASTn和MR质心作为查询,在最终Hi-C组装中映射的GBS标记的位置被用于将两个亲本物种的遗传连锁图锚定到狗头人物理图上。如图所示。4,在两个物种之间观察到非常高的同步性,狗头人物理地图的共线性为95.1和92.9%b . thunbergii而且b .寻常的分别为连杆映射。在两个连锁图谱中,都有一小部分位点的物理位置b . thunbergii5.1%的b .寻常的)是模棱两可的,因为它们不能被分配到物理集会中的唯一位置。另一个小百分比的位点(0.93%b . thunbergii1.12%的b .寻常的)在不同的染色体上表现出明显的BLAST命中,如图中的点所示。4.大约着丝粒的位置可以从Hi-C热图中直观地推断出来2:图S2)。
BtUCONN1(红色)和Wagon Hill(绿色)基因图谱的同步性可视化。小檗属植物寻常的(Bv,绿色)及b . thunbergii(英国电信(红色)的基因图谱(单位:cM)被固定在b . thunbergii简历。“狗头人”参考组件(Ko,蓝色;(单位:Mbp)通过GBS质心。BLAST超出预期链接组的7个GBS标记由小数字(01-14)表示,表示它们关联的链接组。BLAST到无支架contigs的四个GBS标记用“Un”表示。连锁图旁边的小点表示在整个基因组中有多个不明确的定位位点。狗头人物理图上粗体的水平黑条表示着着丝点的大致位置,基于Hi-C热图。QTL区位置QPgr-3S标记在3号染色体的连锁图谱旁b . thunbergii父母“BtUCONN1”
为了将染色体编号分配给连锁群,狗头人物理组装中的伪分子按从长到短进行了排序。在狗头人装配中最长的伪分子(99.76 Mbp)上锚定的连接基团(LG)被指定为LG1;第二长的伪分子为LG2 (99.56 Mbp);依此类推到LG14 (54.72 Mbp)(见附加文件)2:表S2)。因为观察到的连锁群数量与该物种的预期染色体数量完全一致[40], LG1被简单地重新分配为1号染色体,以此类推。
转录组的组装
通过对10个不同组织的库进行测序,获得了59.6 Gb的数据,包括约1.98亿个150 bp PE readsb . thunbergii参考加入'狗头人',包括接种后不同时间点取样的未成熟叶片组织Pg(附加文件2:表S3)。使用Trinity管道[43]和最终的狗头人装配作为指导,组装了189.3 Mbp转录组,包含122,872个假定转录本和55,186个cDNA序列(完整的orf)(见表5用于汇总统计)。通过TransRate评估转录组组装的质量和完整性[44]和BUSCO分析[45].迄今为止,TransRate评分为0.22,超过了NCBI TSA中已发表的从头组装转录组的50% [44].相比之下,狗头人转录组的TransRate评分为0.40,表明其相对质量。完整性统计也可以接受,事实表明,在BUSCO组的1440个核心植物基因中,有1286个(89.3%)在转录组中被表达,其中651个(45.2%)为单拷贝,635个(44.1%)为重复。
候选基因的鉴定
13厘米QPgr-3S区域在物理装配中对应5.35 Mbp区域,共包含20个contigs(长度N50 = 389.7 kbp)。为了完善QTL区域内的组装,这20个contigs使用canu进行局部重新组装[46],最终得到13个contig,总长度减少了5.10 Mbp, contig长度N50增加了508.5 kbp。使用RepeatMasker [47的5.6% (~ 373 kbp)Qpgr-3S区域被掩盖为重复元素使用答:芥作为模型。共发现219个逆转录因子,其中ltr 178个(79Ty1/Copia和99年吉普赛/DIRS1)和41是LINEs (L1/CIN4).另一个大约9 kbp的序列被发现对应于DNA转座子。简单序列重复区域总长度为130 kbp,共发现32个小rna。
的功能注释QPgr-3S结果鉴定出576个高信度(HC)基因。其中,450个基于参考转录组(循证)注释,126个基于基因预测模型(从头算)注释。以帮助确定一个候选基因的名单,可能与Pg-NHR并优先用于正在进行的研究,HC基因列表与其他两项分析的结果进行了交叉引用:差异基因表达(DGE)和存在/缺失分析(见材料和方法)。时间过程DGE分析鉴定出5个基因(TR27614、TR9306、TR20791、TR5393和TR12856)在基因表达上存在差异Pg接种(附加文件)2:图S3和图S4)。基因TR27614和TR9306表现出类似的从接种后48h开始逐渐下调的模式。基因TR20791在前48 hpi表达上调,72 hpi表达下调。相比之下,TR5339和TR12856基因在72 hpi后开始出现下调,然后逐渐回升到原来的水平。存在/缺失分析确定了两个基因存在于b . thunbergii参考,但似乎是完全缺失(MA26)或缺失整个外显子(MA262)b .寻常的(附加文件2:图S5)。可能缺席的证据b .寻常的由于MA026的高覆盖率,MA026的影响特别强b .寻常的在基因的直接附近读取(附加文件2:图S5)。
结合QTL分析的连锁证据,时间过程DGE和存在/缺失分析的结果将上述7个基因提升到与QTL相关的候选基因的地位Pg-NHR。因此,这些候选对象被选中进行详细的功能注释;在UNIPROT和Phytozome数据库中发现了其中3个(TR20791、TR27614和TR12856)的同源序列。具体而言,基因TR20791与休眠相关的生长素抑制蛋白家族有关;TR27614与锌指dna结合蛋白具有较高的序列相似性;TR12856属于谷氨酰胺合成酶(谷氨酸-氨连接酶活性)蛋白家族(附加文件5).其他四个候选基因在任何用于功能注释的公共数据库中都没有命中,因此可能是小檗属植物-特异性基因,或者至少是以前在其他物种中没有特征的新基因。随着新一代测序在基因组研究中的应用已成为常规,在模式物种和非模式物种中都发现了大量完全新颖的转录本(例如,参见[48,49,50,51])。
讨论
遗传和基因组资源开发
植物遗传连锁图谱构建中常用的定位群体包括分离F2系,回交群体,双单倍体,重组自交系。然而,在自交不亲和的多年生植物物种中,尤其是像杨梅这样世代时间较长的植物,这种典型的定位种群即使不是不可能,也是很难产生的。为了克服这些挑战,Grattapaglia和Sederoff(1994)首先提出了所谓的“伪测试杂交”策略,并成功应用于构建森林树木的遗传连锁图谱[39].根据这一策略,绘制了全兄妹F1后代是由两个不相关的高度杂合(即非近交)个体杂交而产生的。配子重组可以在这样的种群中跟踪,因为战略选择的标记集服从典型测试杂交中发现的分离模式。这种方法已广泛应用于其他方法不适合用的植物种类[52,53,54].
在这项研究中,使用伪测试交叉策略,为两者开发了遗传连锁图b . thunbergii而且b .寻常的从单一种间F1人口的映射。由于对所使用的一组从头开始的GBS标记应用了严格的质量过滤器,几乎100%的标记都成功地放置在两个物种的连锁图中。虽然流式细胞术分析表明两个亲本之间的基因组大小相当(b . thunbergii: 1.72英镑;b .寻常的: 1.69 Gbp), BtUCONN1的总长度(b . thunbergii)的连锁图谱比Wagon Hill (b .寻常的)地图(1474厘米vs. 1714厘米)。这种与物理基因组大小的预期差异的不一致可能是由于可用的标记明显较少b .寻常的与可用的地图进行比较b . thunbergii(706 vs. 1757).低标记密度往往导致遗传距离膨胀[55],因此,预计额外的标记将减少的总长度b .寻常的连锁图。可用于标记的数量显著降低b .寻常的可能是由于该物种在从欧洲殖民引入北美期间被认为存在严重的遗传瓶颈,因此其多样性水平相对较低的结果[21].
在本研究中开发的两个连锁图是毛茛科植物目中任何物种的第一个。标记在两个物种的14条染色体上的相对均匀分布使得初始QTL分析具有可接受的分辨率,大约87%和65%的标记间距离小于5 cMb . thunbergii而且b .寻常的,分别。此外,在两个独立的地图之间观察到的强同向性是它们可靠性的有力证据(图。4).
作为遗传资源的补充,如绘制种群和连锁图,高质量的参考基因组可以作为解剖qtl、识别潜在候选基因和促进其详细描述的宝贵资源。在这项研究中,当代测序和脚手架技术被用于开发一个高度连续的从头参考基因组b . thunbergii.利用PacBio SMRT测序和染色体构象捕获数据,得到1.2 Gb的单倍体组装体b . thunbergii简历。“狗头人”被成功组装成14个染色体尺度的伪分子。与链接图一样,这是第一个涉及小檗科和毛茛目成员的链接图。考虑到之前对barberries分子资源的缺乏,本研究中组装的参考基因组体现了最近技术的力量,即使在非模型系统中也能取得快速进展,并为具有中等大小基因组的高度杂合子植物物种的重新组装建立了基准。
综上所述,建立了包括种间基因分型定位群体、两个亲本种的连锁图谱、染色体尺度参考基因组和多组织转录组在内的基础遗传和基因组资源小檗属植物作为一个可行的研究模式进行研究Pg-NHR。此外,这些资源有望促进相关工作,包括全球锈病监测工作和观赏园艺育种。
QPgr-3 s以及候选基因的鉴定Pg-NHR
本研究的长期目标是确定调控的候选基因Pg-NHR在b . thunbergii.作为朝着这一方向迈出的第一步,这里开发的遗传和基因组资源能够在短臂上鉴定一个具有较大影响的单个QTL (LOD > 28)b . thunbergii染色体3(图;3.).这个13cm的QTL区域被命名为Qpgr-3S,被发现跨越13个物理contigs,共包含576个高置信基因。其中,7个被列为后续研究相对优先的候选基因,其中3个与公共数据库中的基因表现出同源性,包括休眠相关生长素抑制蛋白(TR20791)、锌离子结合蛋白(TR27614)和谷氨酰胺合成酶蛋白(TR12856)。
目前的抗病模型表明,植物的免疫反应可以大致分为两大类,即由病原体相关分子模式触发的入侵前防御(pamp触发的免疫)和由病原体效应物触发的入侵后防御(效应物触发的免疫)[56,57],两者都已被证明涉及广泛的防御相关蛋白质。在本研究中鉴定的7个候选基因中的3个与文献中涉及抗病的基因家族具有同源性。例如,已知生长素是水杨酸的调节剂,水杨酸是诱导植物系统性获得性抗性所必需的植物激素[58];锌指转录因子参与了一种影响锈病芽管分化的基因的调控[59];已知谷氨酰胺合成酶蛋白通过氨基酸代谢在植物防御病原体中发挥关键作用[60].
两者的识别QPgr-3S区域和一组高优先级候选基因证明了本研究中开发的遗传和基因组资源在探测潜在基因方面的实用性Pg-NHR由b . thunbergii.然而,这样的结果只是确定控制基因的第一步Pg-NHR;除了测试候选基因假设外,还需要进一步的工作来验证和解剖QTL区域。
的可能继承模式Pg-NHR
从育种提高小麦茎秆锈病抗性的实际角度出发,提出了小麦茎秆锈病防治的核心问题Pg-NHR涉及基础基因的性质和遗传模式。正如先前在天然种间小檗杂交种群中观察到的[21), F1种间杂交表现出一系列反应Pg从完全抵抗到完全易感,有各种中间形式。这一系列反应在F1为本研究开发的映射种群(图;2c-f和表3.).如果有人假设Pg为在b . thunbergii是否由单个基因控制,减数分裂时的独立分类必然导致纯合子Pg易受影响b . thunbergii后代。然而,到目前为止,没有加入b . thunbergii尽管进行了广泛的调查(见背景),但仍表现出这种易感性;因此,一个单一的基因控制Pg为在b . thunbergii是不可能的。在其他研究中也发现了多基因NHR,包括水稻NHR对小麦茎锈病的作用,大麦NHR对白粉病、燕麦茎锈病和其他非适应性锈病的作用[19,61,62].
如果真的QPgr-3S地域起着一定的作用Pg-NHR,数据表明其潜在基因对抗性是必要的,但不是充分的。换句话说,这项研究最多提供了对更大的基因网络调控的第一次洞察Pg-NHR在b . thunbergii.的确,鉴于在非寄主亲本物种中缺乏分离b . thunbergii, F1杂交表明某些关键基因(s)的可能存在,根据定义固定在b . thunbergii上游的GenepoolQPgr-3S.因为它们内部的固定状态b . thunbergii,这样的基因(s)不能映射到F1人口;但如果是隐性的,他们的单剂量是F1会使易感性Pg,从而可以检测背景抗性基因(例如。QPgr-3S).十有八九,QPgr-3S关键区域不是赋予吗Pg-NHR,而是一个地区的贡献Pg阻力。F1子代和/或回交b . thunbergii是否有必要检验这一假设并确定那些关键的调控基因Pg-NHR在b . thunbergii目前的研究表明,这项工作是可行的。
结论
在这篇文章中,我们报告了公共可用的基础遗传和基因组资源的新发展小檗属植物-Pg病理系统,包括两个的第一个基因图谱小檗属植物物种(b . thunbergii而且b .寻常的),一个染色体尺度的参考基因组b . thunbergii,以及相关的转录组,以促进遗传机制的表征Pg-NHR。未来的工作应集中在已确定QTL的验证、进一步表征和解剖,包括测试候选基因假设。除此之外,现在小檗属植物-Pg病理系统已被证明是一种可行的手段,探索机制Pg-NHR在b . thunbergii在美国,未来的工作还必须努力解决这种对小麦抗性的潜在可翻译性这一重要问题。这种可译性当然不是给定的,特别是考虑到感染的孢子是不同的小檗属植物(basidi孢子)和草(uredinio孢子)宿主。但是,由于所讨论的两个生命阶段属于同一致病菌,并且因为小檗属植物可能是该生物在其宿主扩展到草类之前的祖先宿主(见背景),可能存在的机制Pg-NHR在b . thunbergii为小麦抗性育种提供了新的思路。通过这项研究,为最终回答这个问题奠定了基础。
方法
绘制人口发展图
一个B。×ottawensis由182个F1个体是由种间杂交而来的b . thunbergii添加' BtUCONN1 '(花粉亲本)和b .寻常的继承' Wagon Hill '(女性父母)。BtUCONN1是茎锈病病原菌的非寄主,是一种小灌木(0.5-2.5 m高),全叶长1.3-3.8 cm,花序长1-2 cm,很少有伞形花序,但大部分是单花。相比之下,马车山易受茎锈病的影响,是一种相对较高的灌木(约3米高),具有2-5厘米长的倒卵形至倒卵形长圆形叶片,边缘具有高度锯齿(> 50锯齿),有5-8厘米长的垂状总状花序,花为亮黄色。花粉亲本BtUCONN1是一种野生植物,保存在康涅狄格大学研究农场的小梅收藏中(N41°47′40.63″,W072°13′39.61″),母本Wagon Hill是一种野生植物,生长在新罕布什尔州达勒姆的大海湾口海岸线(N43°07′30.64″,W70°52′17.95″)。
为了进行种间杂交,使用前面描述的正戊烷方法从BtUCONN1的成熟花中收获花粉[63]并保存在4°C,直到马车山的花达到繁殖成熟。雌性花朵的阉割和人工授粉是在所谓的气球阶段进行的,此时花瓣开始在顶部略微分开,在打开之前形成充气气球的外观。为了在播种前打破休眠,成功杂交的种子在培养皿中4°C的湿沙中分层,为期3个月。两个亲本的繁殖剪枝与F1在直径11.5厘米的塑料盆中绘制人口分布图;6.5厘米高)填充PRO-MIX HP生长介质在麦克法兰温室设施在新罕布什尔大学。
为了验证假定的F1根据现有的GBS数据,设计了基于pcr的种特异性标记[21].一个通用引物对被设计用来扩增在两个亲本之间表现出长度多态性的短基因组序列。具体来说,引物(F: 5 ' -CCTGATTGGGGCTCATTATC-3 ';R: 5 ' -AGTGAGGAATTCCGAGCTGA-3 ')在Wagon Hill中扩增了一个208 bp的片段,但在BtUCONN1中只扩增了一个195 bp的片段,因为存在一个13 bp的indel(见附加文件)6:文本S1)。PCR反应总体积为20 μl(每个引物0.25 mM,每个dNTP 100 μM, 0.75 U Taq DNA聚合酶,10倍标准Taq缓冲液,100 ng模板DNA),循环条件为:94℃下5 min;32个周期,94°C 30 s, 52°C 30 s, 68°C 15 s;在68°C下5分钟。扩增产物在3% TBE/EtBr琼脂糖凝胶上分离60分钟,在75 V下,用紫外透光成像。F1如果来自两个亲本种的两个条带都被检测到,则假定的杂交个体的状态被认为是有效的2:图S6)。
基因分型和变异检测
182株经鉴定的F1采用改良的CTAB法从约100 mg冻干叶片组织中提取个体和双亲[64].在制备GBS文库之前,使用Zymo Research的Genomic DNA Clean & Concentrator™对分离的DNA进行纯化-10列(目录# D4011),遵循制造商的协议。用双酶(PstI-MspI)波兰等人描述的GBS协议[65]并在哈伯德基因组研究中心的Illumina HiSeq 2500上通过150 bp对端(PE)读取进行测序。
使用CASAVA 1.8.3生成原始FASTQ文件,并使用无参考生物信息学管道GBS-SNP-CROP进行分析[38,66].利用来自两名家长的高质量体育阅读构建模拟参考(MR);通过对来自亲本和所有F的高质量PE reads进行比对,确定了假定的变异,包括snp和indels1子代到MR,遵循管道的二倍体物种推荐参数。本分析中使用的GBS-SNP-CROP命令行的完整细节,包括所有指定的管道参数,在附加文件中提供6:文本S2。
遗传连锁图谱构建
表中总结了用于获得链接映射构建的最终标记集的过滤器序列1.简而言之,如果一个标记符合以下任何一个标准,它就被剔除了:1)种群中超过30%的个体没有得分;2)双亲均为杂合;3)未在群体中分离(即该标记的所有子代均为杂合);4)主候补等位基因深度的平均比值显著偏离预期的1:1;和/或5)其分离比明显偏离1:1的预期比例,根据其标记类别。作为最终筛选,去除>缺失数据30%的基因型。
采用R包ONEMAP v2.0-4进行连锁分析[67],并根据双向伪测试交叉映射策略为两个亲本构建单独的连锁映射[30.].BtUCONN1链接映射使用标记集1和2构建,而Wagon Hill映射使用标记集3和4构建(见表1).对于每张地图,首先对所有标记对进行两点测试,使用最小LOD评分为4,最大重组分数为0.25,将标记分组为连锁组(lg)。接下来,使用ONEMAP中的“try”算法对每个LG中的标记进行排序。
识别GBS数据中常见的潜在基因分型错误[68],地图被手动检查是否存在单例(明显的双交叉)[69],它们被替换为缺失的值。如果发现多个标记映射到同一个遗传库,则选择一组标记的共识来表示链接库,进行最终的映射迭代,直到没有由' ripple '生成的替代顺序。seq的函数。使用Kosambi映射函数计算最终地图距离[70],而表意文字则使用Mapchart 2.0生成[71].
茎锈病表型
为了确定疾病反应,父母和所有F1将定位群体中的个体接种从萌发的微孢子中喷射出来的担子孢子Pg发现特莉娅就自然感染了大被,如上文所述[21].花粉亲本BtUCONN1表现出明显的典型的非寄主反应b . thunbergii.相比之下,母本马车希尔表现出明显的易受影响的反应b .寻常的叶的背面可见发育良好的成熟的被毛。典型反应的图像的父母和个人在F1映射总体如图所示。2.详情见表3.,针对在该人群中观察到的特定隔离特征,开发了一个4分制量表。该量表的级别是基于以下症状:1)斑点的程度;2)坏死灶的存在及程度;3)脓虫、绦虫的存在和密度。接种茎锈病14 d后,对所有植株进行茎锈病反应评分。
QTL分析
QTL分析Pg利用R包R/qtl v1.39-5的亲本和母本遗传连锁图进行抗性分析[72].哈雷-诺特回归[73],基于复合区间映射法(CIM);如果一个QTL的LOD评分峰值超过排列分析确定的阈值(1000个排列,5%显著性水平),则认为该QTL显著。
参考基因组组装
因为它不仅与Pg-NHR研究,也用于观赏育种,b . thunbergiicv“狗头人”(Kobold)是观赏工业中常见的一种商业绿叶品种,被选中进行全基因组测序。狗头人是一种杂合二倍体(2n = 2x = 28),是茎锈病的非寄主[33].狗头人的剪枝从康涅狄格大学的小檗收藏中获得,在标准的小檗条件下生根,并在UNH的麦克法兰温室中保存[21].为了测序,从4 ~ 6株无性系植株中采集了~ 2g的新鲜嫩叶,并在液氮中快速冷冻。采用改良的CTAB程序提取基因组DNA [74]并通过荧光测定(Qubit,赛默飞世尔科学,沃尔瑟姆,美国)和琼脂糖凝胶电泳与lambda DNA标准定量。使用20kb的BluePippin试剂盒(PacBio)进行单分子实时(SMRT)文库制备;115个SMRT细胞在加州大学戴维斯基因组中心的PacBio RS II系统上使用P6-C4化学进行测序。所有数据收集为6h测序视频。
FALCON和FALCON- unzip工具包(FALCON- integration v1.8.2) [41]用于全基因组组装和分期。FALCON被描述为一个分层基因组组装过程(Hierarchical Genome Assembly Process)管道,它通过以下基本步骤从长PacBio reads生成基因组组装:1)通过对齐子reads来纠正原始读取错误;2)预装配经过纠错的长读数;3)预装读块重叠检测;4)重叠滤波;5)重叠图构造;6)基于图的contig构造。在这个初始组装之后,FALCON-Unzip用于高度杂合子的物种,根据结构变异和相关snp(即单倍型块)的模式来解析不同的haploes(即解压缩基因组)。这个解压缩过程产生了一组所谓的主contigs(主组装)和一组相关的haplotigs(主contigs的阶段变体,在高杂合度区域)。本研究中使用的FALCON配置文件的完整细节在附加文件中提供6:文本S3。最后,' GenomicConsensus ' PacBio包中的Arrow算法(https://github.com/PacificBiosciences/GenomicConsensus)用于打磨相控的主contigs及其相关的haplotigs。基因组大小的估计使用校正PacBio reads的k-mer分析[75]以及碘化丙啶流式细胞术分析使用Pisum一L. Citrad (2C = 9.09 pg)作为内部标准(BD Accuri™C6流式细胞仪)[76].
使用Purge Haplotigs管道完成了对组件的进一步抛光和管理[77].某些基因组区域的高杂合度会导致单倍体作为不同的原始contigs的错误分配[77].为了识别此类错误并正确地将同源contigs分配到haplotig池,清除Haplotigs管道首先使用BEDTools执行读深度分析[78]将异常低或高覆盖率的contigs标记为潜在的嵌合体,然后执行BLAST [79]对整个组合进行比对,以确定推定的主contigs彼此之间表现出高度同源性。在此过程中,生成对齐点图,并手动筛选这些点图以破坏可能的嵌合体,将最终的主contigs集定义为参考序列,并将剩余的syntenic contigs分配为haplotigs。清除Haplotigs过程的完整细节在附加文件中提供6:文本S4。
基因组组装质量和hi-C支架的评估
使用QUAST评估最终策划装配的质量[80],利用BUSCO v3中1440个核心植物基因集评估组装完整性[45].为了识别和清除污染物contigs,最后的组装被发送到以下可能的污染物数据库:被子植物的质粒DNA (cpDNA和mtDNA),人类基因组(GRCh38.p7),和DNA大肠杆菌基因组(CP017100.1),以及16S和18S rrna。rRNA数据库是使用SILVA项目创建的[81],其他基因则是通过从Genbank中取样而产生的。为了进一步评估完整性,PacBio纠错读取(preads)、为转录组组装生成的RNA-seq数据(见下文)以及为连锁映射生成的BtUCONN1亲本的GBS数据也使用BLASR与最终组装进行了比对[82], gmap [83],及BWA [84),分别。
为了将主要contigs线性排序并定向为染色体尺度的伪分子,使用Phase Genomics的Proximo™染色体构象捕获(Hi-C)技术进行了近距离引导组装[42].由Phase Genomics (Seattle, WA, USA)进行组织处理、染色质分离、文库制备、测序和Hi-C分析。最后,利用JuiceBox,利用BtUCONN1遗传连锁图手动策划Hi-C组装[85],在出现歧义的情况下,提供独立的信息来指导一组锚点contig的排序。
基因连接的锚定映射到物理装配
采用正交标记建立亲本间的遗传连锁图谱;因此,这两张地图没有共同的标记,阻碍了对这两个物种之间同步性的直接评估。然而,物理组件提供了一种可以比较两个映射的潜在“公共语言”,前提是链接映射中的标记可以唯一地位于物理组件中(即锚定在物理组件上)。为了做到这一点,BLASTn [86]在MR质心(查询)和策展程序集(主题)之间执行。仅使用那些在参考基因组中显示独特位置的质心,使用Matplotlib绘图库的Pacth函数生成同步性图(https://matplotlib.org/index.html).采用上述锚定方法对检测到的信号进行投影Pg-NHR QTL区域的物理图谱,从而允许洞察其潜在的物理序列。
转录组的组装
对于转录组组装,10种不同的组织,包括不同时间点后的未成熟叶片组织Pg接种,从无性繁殖的植物b . thunbergii简历。“狗头人”(附加文件2:表S3)。新鲜的组织在液氮中快速冷冻,然后用研钵和杵磨成细粉。根据制造商的协议,使用Zymo Research RNA Clean & Concentrator™试剂盒(目录# R1015)分离总RNA。RNAseq文库使用Illumina TruSeq®RNA文库准备试剂盒制备,并在UNH哈伯德基因组研究中心的Illumina HiSeq 2500上通过150 bp对端(PE)读取进行测序。
使用BFC v1.0软件对casava处理的原始序列进行错误校正[87],按照Oyster River Protocol For Transcriptome Assembly的建议[88].使用Trimmomatic v.0.33处理纠正错误的读取以删除Illumina适配器,并轻轻修剪以删除低质量的读取(Phred≤5)[89].来自这10个组织的所有后处理reads被汇总,转录组使用Trinity (reference-guided de novo assembly)进行组装[34].使用TransRate [44],利用BUSCO v3中1440个核心植物基因集评价其完整性[45].除了提供基本的汇总统计和质量指标外,TransRate还提供了基于一套映射指标的转录组邻近的总体评分;BUSCO基于期望的单拷贝正交向量的表示来评估装配内容。
候选基因的鉴定
为了促进候选基因的鉴定,可以解释所检测的QTL区域与Pg响应时,跨越QTL区域的物理contigs使用canu进行局部重组[46].然后使用RepeatMasker对重新组装的QTL区域进行结构注释[47并使用Maker管道进行功能注释[90],使用ab-initio和基于转录组的分析。基于Maker’s Annotation Edit Distance质量度量(AED < 0.7)和基因长度在500 - 10,000 bp之间不重叠的要求,定义QTL区域内支持良好的基因集,以下称为高置信度(HC)基因。
我们采取了多种方法的组合,以减少HC的全部基因,只保留那些更可能导致HC的基因Pg-NHR。进行了差异基因表达(DGE)分析实验,以确定其表达水平在胁迫下可检测到变化的基因Pg.从无性系繁殖的未成熟叶片中取样3个生物重复b . thunbergii简历。“狗头人”植物在四个不同的时间点:接种前(T0)和接种后48、72和144小时(T48、T72和T144)。总RNA按照上述方法提取、测序和处理。利用Kallisto定量转录本丰度[91],时间过程分析采用Sleuth [92].用于成绩单丰度和时间过程分析的参数的完整细节在附加文件中提供6:文本S5。
为了补充上述DGE分析,~ 4.28亿PE 100 bp霰弹枪从b .寻常的将母株Wagon Hill(即>30倍重测序深度)与QTL区进行比对,以鉴定HC中没有明显同源的基因b .寻常的.高优先级候选基因的最终列表由QTL区域中差异表达的HC基因组成Pg接种或至少有一个完整的CDS序列缺失b .寻常的(附加文件2:图S5)。假定的蛋白质功能和基因本体(GO)术语被分配到候选基因,使用Phytozome v.12.1 [93]及UniProtKB [94)数据库。
数据和材料的可用性
所有原始序列数据和最终集合(基因组和转录组)都可通过NCBI数据库获得。经过解析的高质量GBS数据生成的两个亲本系和182 F1子代可通过NCBI短读档案,SRA ID的附加文件提供1.RNAseq数据从十b . thunbergii简历。用于DGE和转录组组装的“狗头人”组织连接到NCBI BioProject PRJNA478022;组装的转录组本身可在TSA ID GGRA00000000下获得。与狗头人基因组组装相关的数据,包括FALCON-Unzip主contigs和单倍体,最终Hi-C引导的染色体水平组装,以及所有未支架的contigs,均保存在NCBI中,生物项目登录号为PRJNA478403。狗头人基因组也可通过UNH WebApollo基因组浏览器进行可视化和注释http://genome.sr.unh.edu/jbrowse.
缩写
- CIM:
-
复合区间映射
- DGE:
-
差异基因表达
- GBS:
-
Genotyping-by-sequencing
- HC的基因:
-
高自信基因
- 现病史:
-
接种后小时
- 格林:
-
连锁群
- 先生:
-
模拟参考
- NHR:
-
不耐
- PAMP时:
-
病原体相关的分子模式
- 体育:
-
成对的结束
- Pg:
-
小麦锈菌
- SMRT:
-
单分子实时
参考文献
- 1.
Leonard KJ, Szabo LJ。茎锈病引起的小谷物和草小麦锈菌.植物病理学杂志2005;6:99-111。
- 2.
陈建平,李志强,陈志强,等。小麦茎锈病真菌新品种的出现和传播:对粮食安全和遗传控制前景的持续威胁。植物病理学。2015;105:872 - 84。
- 3.
张志刚,张志刚,张志刚,等。茎秆锈病会摧毁世界小麦作物吗?Adv Agron, 2008; 98:271-309。
- 4.
彼得森警局,莱纳德·KJ,鲁尔夫斯美联社,萨顿·TB。根除小檗对山茱萸种群变化的影响小麦锈菌在明尼苏达州。植物科学学报,2005;39(2):339 - 339。
- 5.
金勇,Szabo LJ, Pretorius ZA, Singh RP, Ward R, Fetch T Jr.抗性基因毒力检测Sr24TTKS的小麦锈菌f . sp。tritici.植物科学学报,2008;42(2):344 - 344。
- 6.
黄志强,王志强,王志强,等。小麦茎锈病抗性基因毒力检测Sr31在小麦锈菌.f。Sp。tritici在乌干达。植物科学2000;84:203。
- 7.
张志刚,张志刚,张志刚,等。的出现Ug99茎锈病真菌是世界小麦生产的一大威胁。植物病理学杂志,2011;49:465-81。
- 8.
Lewis CM, persons A, Bebber DP, Kigathi RN, Maintz J, Findlay K,等。小麦茎锈病在英国重新出现的可能性。社区生物学2018;1:13。
- 9.
李国强,李国强,李国强,等。小麦茎秆锈病抗性研究进展。植物学报,2008;11:187-92。
- 10.
李卡U, Fuchs R, Kuhns C, Petutschnig E,李卡V.对白粉病的非寄主抗性拟南芥。中国生物医学工程学报(英文版);2010;
- 11.
对非宿主抗性过程的新见解。植物学报,2003;6:351-7。
- 12.
迈索尔k,柳崇明。非宿主抗性:我们知道多少?植物科学进展2004;9:97-104。
- 13.
Ayliffe M, Devilla R, Mago R, White R, Talbot M, Pryor A,等。水稻对锈病病原的非寄主抗性。植物与微生物相互作用。2011;24:1143-55。
- 14.
程燕,张辉,姚娟,王旭,徐娟,韩强,等。蚕豆对小麦条锈病的非寄主抗性特征。BMC植物生物学。2012;12:96。
- 15.
程燕,张辉,姚杰,韩强,王霞,黄玲,等。植物非寄主抗性的细胞学和分子特征拟南芥预防小麦条锈病。植物化学学报。2013;62:11-8。
- 16.
安涛,蔡勇,赵松,周杰,宋波,布克斯华,等。Brachypodium distachyonT-DNA插入系:研究小麦条锈病非寄主抗性的模型病理系统。科学代表2016;6:25510。
- 17.
道森AM,弗格森JN,加德纳M,格林P,哈伯德A,莫斯库MJ。隔离和精细映射Rps6:大麦抗小麦条锈病的中间寄主基因。理论应用,2016;129:831-43。
- 18.
李凯,Hegarty J,张超,万安,吴娟,Guedira GB,等。大麦位点精细定位Rps6抗小麦条锈病。理论应用,2016;129:845-59。
- 19.
德拉卡托斯PM, Ayliffe M, Khatkar MS, Fetch T Jr, Singh D, Park RF。吸前抵抗遗传小麦锈菌f . sp。avenae在大麦中(大麦芽l .)。植物与微生物相互作用。2014;27:1253-62。
- 20.
菲格罗亚M,议员S,加文DF, Pfender WF。感染Brachypodium distachyon的形式和特殊情况小麦锈菌:早期感染事件和宿主-病原体不相容。PLoS One, 2013。https://doi.org/10.1371/journal.pone.0056857.
- 21.
杨晓明,杨晓明,杨晓明,杨晓明。一种种间杂交小檗对茎锈病病原菌非寄主抗性的遗传分析小麦锈菌.中国生物医学工程学报,2018;
- 22.
Jin Y.角色小檗属植物在产生新种属时作为替代宿主小麦锈菌而且p . striiformis.Euphytica。2011;179:105-8。
- 23.
赵娟,王磊,王震,陈鑫,张宏,姚洁,等。十八人的鉴定小檗属植物作为替代宿主的种柄锈菌striiformisf . sp。tritici中国自然侵染小檗病原菌的毒力变异。植物病理学。2013;103:927-34。
- 24.
皮特森PD。“小檗还是面包”:20世纪初美国根除普通小檗的公众运动。可以从:https://www.apsnet.org/edcenter/apsnetfeatures/Pages/Barberry.aspx
- 25.
Levine MN, Cotter RU。易感性和耐药性小檗属植物和有关属小麦锈菌:美国农业部;1932.报告没有。: 300
- 26.
美联社鲁尔夫斯,布什内尔WR。疾病、分布、流行病学与控制。正确的做法:谷物锈病。奥兰多:学术;1985.7 - 9页。
- 27.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.ars.usda.gov/ARSUserFiles/50620500/Barberry/fedreg67-36.pdf.2019年5月28日访问。
- 28.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.ars.usda.gov/ARSUserFiles/50620500/Barberry/fedreg71-23.pdf.2019年5月28日访问。
- 29.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.ars.usda.gov/ARSUserFiles/50620500/Barberry/2007%20fedreg72-112.pdf.2019年5月28日访问。
- 30.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.ars.usda.gov/ARSUserFiles/50620500/Barberry/2010%20fedreg75-100.pdf.2019年5月28日访问。
- 31.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.ars.usda.gov/ARSUserFiles/50620500/Barberry/2010%20fedreg75-173.pdf.2019年5月28日访问。
- 32.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.govinfo.gov/content/pkg/FR-2013-05-13/pdf/2013-11318.pdf.2019年5月28日访问。
- 33.
农业部。黑茎锈病;防锈品种的鉴定要求和添加。https://www.govinfo.gov/content/pkg/FR-2017-09-05/pdf/2017-18712.pdf.2019年5月28日访问。
- 34.
黑尔(Hale IL),康诺利(Connolly BA),巴陶拉(Bartaula r)。小檗属植物×ottawensis(小檗科),产于新罕布什尔州和罗德岛。北美杜鹃花。2015;117:384-7。
- 35.
Arthur JC,康明斯GB公司。美国和加拿大的锈蚀手册。拉斐特:普渡研究基金会;1934.
- 36.
李永林,kvaekek Z, Ferguson DK,王永峰,李春生,杨杰,等。化石记录小檗属植物(小檗科),以及该属的演化和植物地理的解释。古机器人。2010;160:10-31。
- 37.
贝根豪泽,李志强,李志强,李志强。对锈病病原的非寄主抗性是连续的延续。植物科学进展(英文版);
- 38.
Melo AT, Bartaula R, Hale I. GBS-SNP-CROP:利用可变长度、配对端测序数据进行SNP发现和植物种质鉴定的参考可选管道。BMC Bioinform, 2016。https://doi.org/10.1186/s12859-016-0879-y.
- 39.
葛兰,王晓明桉树茅而且桉树urophylla使用伪测试交叉:映射策略和RAPD标记。遗传学。1994;137:1121-37。
- 40.
朗萨维尔TJ,兰尼TG。倍性水平和基因组大小小檗属植物l .,十大功劳纳特。种,杂交种和栽培品种。HortScience。2010; 45:1029-33。
- 41.
陈春生,彭丽娟,陈志强,陈志强,等。分阶段二倍体基因组组装单分子实时测序。Nat Methods. 2016;13:1050。
- 42.
张志刚,张志刚,张志刚,张志刚。基于染色质相互作用的新基因组组装的染色体尺度支架。中国生物工程学报,2013;
- 43.
李志强,李志强,李志强,等。利用三位一体平台从RNA-seq中重建转录本序列,用于参考生成和分析。Nat protocol . 2013;8:1494。
- 44.
Smith-Unna R, Boursnell C, Patro R, Hibberd JM, Kelly S. TransRate:从头转录组组装的无参考质量评估。基因组决议2016;26:1134-44。
- 45.
Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:评估基因组组装和单拷贝orthologs注释完整性。生物信息学。2015;31:3210-2。
- 46.
Koren S, Walenz BP, Berlin K, Miller JR, Bergman NH, Phillippy AM。Canu:通过自适应k-mer加权和重复分离可伸缩和精确的长读取组装。基因组决议2017;27:722-36。
- 47.
陈宁。利用重复掩码技术识别基因组序列中的重复元素。生物信息学。2004;5:4-10。
- 48.
Srivastava R, Budak G, Dash S, Lachke SA, Janga SC.发育中的晶状体转录组分析揭示了大量新的转录本和广泛的剪接改变。科学通报2017;7:11572。
- 49.
怪人T, Militello G, Müller R, John D, Dimmeler S, Uchida S. RNA-seq数据中新转录本的鉴定和表征。生物信息学报。2015;17:678-85。
- 50.
王玲,曹超,马强,曾强,王华,程忠,等。大豆多分生组织的RNA-seq分析:新的和替代转录本,进化和功能意义。BMC植物生物学。2014;14:169。
- 51.
Aghamirzaie D, Batra D, Heath LS, Schneider A, greene R, Collakova E.在发育中的大豆胚胎中,转录组范围的功能特征揭示了差异表达转录本之间的新关系。BMC基因组学。2015;16:928。
- 52.
何东,刘勇,蔡敏,潘宏,张强。紫薇首个遗传连锁图谱(l . agerstroemia)基于扩增片段长度多态性和简单序列重复序列标记。植物育种,2014;133:138-44。
- 53.
Myles S, Mahanil S, Harriman J, Gardner KM, Franklin JL, Reisch BI,等。利用SNP微阵列强度值在葡萄中的遗传定位。Mol Breed. 2015;35:88。
- 54.
Myburg AA, Griffin AR, Sederoff RR, Whetten RW。比较遗传连锁图桉树茅,蓝桉以及基于双伪回交映射方法的F1混合算法。理论应用,2003;107:1028-42。
- 55.
Di Pierro EA, Gianfranceschi L, Di Guardo M, Koehorst-van Putten HJ, Kruisselbrink JW, Longhi S,等。一个高密度的,多亲本的苹果SNP遗传图谱验证了一种新的异交物种映射方法。Hortic Res. 2016;3:16057。
- 56.
Chisholm ST, Coaker G, Day B, Staskawicz BJ。宿主-微生物的相互作用:形成植物免疫反应的进化。细胞。2006;124:803-14。
- 57.
琼斯JD, Dangl JL。植物免疫系统。大自然。2006;444:323-9。
- 58.
德莱尼TP, Uknes S, Vernooij B, Friedrich L, Weymann K, Negrotto D,等。水杨酸在植物抗病中的核心作用。科学。1994;266:1247-50。
- 59.
李的,李H-Y, Seo E,李J,金正日sb,哦,等。目前对植物非寄主抗性的认识。植物与微生物相互作用,2017;30:5-15。
- 60.
杨晓明,杨晓明,杨晓明,Höfte杨晓明。谷氨酸代谢在植物病害防治中的作用。植物与微生物相互作用,2013;26:475-85。
- 61.
Romero CC, Vermeulen JP, Vels A, Himmelbach A, Mascher M, Niks RE.大麦白粉病抗性图谱揭示了一个大效应非寄主抗性QTL。理论应用,2018;131:1031-45。
- 62.
杨晓明,王晓明,王晓明,等。大麦对不同外源锈病真菌的天然非宿主免疫是由特异性不同且重叠的抗性基因组控制的。植物与微生物相互作用,2006;19:1270-9。
- 63.
A.常红杨梅的育种(小檗属植物spp)。伍迪·奥纳姆,1992。https://doi.org/10.17660/ActaHortic.1992.320.11.
- 64.
柯南道尔JJ。一种用于少量新鲜叶片组织的快速DNA分离程序。植物化学通报1987;19:11-5。
- 65.
Poland JA, Brown PJ, Sorrells ME, Jannink J-L。利用一种新的双酶基因测序方法开发大麦和小麦的高密度遗传图谱。PLoS One, 2012。https://doi.org/10.1371/journal.pone.0032253.
- 66.
Melo AT, Hale I.扩展了功能,提高了准确性,并增强了速度新创基因分型测序管道GBS-SNP-CROP。生物信息学》2018。https://doi.org/10.1093/bioinformatics/bty873.
- 67.
玛格丽多·格拉,苏扎AP,加西亚AAF。OneMap:异交物种遗传图谱软件。Hereditas。2007;144:78-9。
- 68.
杨晓东,李志强,李志强,陈志强,陈志强。基于gbs的抗寒猕猴桃种群的反褶积分析(猕猴桃spp)种质。PLoS One, 2017。https://doi.org/10.1371/journal.pone.0170580.
- 69.
范·奥斯,斯坦姆·P,维瑟·RG,范·艾克·HJ。SMOOTH:从高密度遗传连锁数据中成功去除基因分型错误的统计方法。《应用理论》2005;112:187-94。
- 70.
Kosambi DD.从重组值估计映射距离。安·哈姆·热内1943年。https://doi.org/10.1111/j.1469-1809.1943.tb02321.x.
- 71.
MapChart:用于链接映射和qtl的图形化表示的软件。王志强,2002;29(1):77 - 8。
- 72.
吴海平,吴海平,吴海平,吴海平。qtl: qtl实验的分析工具。R软件包版本。2010。https://rdrr.io/cran/qtl/
- 73.
Haley CS, Knott SA。利用侧翼标记在系杂交中定位数量性状位点的一种简单回归方法。遗传。1992;69:315-24。
- 74.
Stoffel K, van Leeuwen H, Kozik A, Caldwell D, Ashrafi H,崔x,等。开发和应用650万特征Affymetrix Genechip®,用于大规模并行发现生菜的单位置多态性(摘要以spp)。BMC基因组学。2012;13:185。
- 75.
王晓明,王晓明。基因组组装中k-mer大小的自动选择。生物信息学。2013;30:31-7。
- 76.
王志强,李志强,李志强,等。流式细胞仪检测植物细胞核DNA含量的研究。Nat protocol . 2007;2:2233。
- 77.
罗奇MJ,施密特SA, Borneman AR.清除单倍体:第三代二倍体基因组组装的等位基因contig重分配。BMC bioinfo . 2018;19:460。
- 78.
昆兰AR,霍尔IM。BEDTools:用于比较基因组特征的一套灵活的实用程序。生物信息学。2010;26:841-2。
- 79.
卡马乔C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K,等。BLAST+:架构和应用程序。BMC bioinfo . 2009;10:421。
- 80.
郭晓明,王晓明,王晓明,王晓明,等。qast:基因组组装质量评估工具。生物信息学。2013;29:1072-5。
- 81.
Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P,等。SILVA核糖体RNA基因数据库项目:改进的数据处理和基于网络的工具。核酸研究,2012;41:D590-6。
- 82.
Chaisson MJ, Tesler G.利用基本局部比对逐次细化(BLASR)绘制单分子测序reads:应用与理论。BMC Bioinform, 2012。https://doi.org/10.1186/1471-2105-13-238.
- 83.
吴道明,渡边CK。GMAP:用于mRNA和EST序列的基因组定位和比对程序。生物信息学。2005;21:1859 - 75。
- 84.
李华,杜斌。基于Burrows-Wheeler变换的快速精确短读对齐。生物信息学。2009; 25:1754-60。
- 85.
杜琴科,沙米姆MS,巴特拉S,杜兰德NC,穆索NT,莫斯托法R,等。Juicebox组装工具模块可以使用染色体长度的支架进行哺乳动物基因组的从头组装,价格低于1000美元。bioRxiv。2018.https://doi.org/10.1101/254797
- 86.
张军,张震,张志强,等。gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸研究,1997;25:3389-402。
- 87.
Li H. BFC:纠正Illumina测序错误。生物信息学。2015;31:2885-7。
- 88.
为非模式生物转录组的从头组装和评估建立基于证据的最佳实践。bioRxiv。2016.https://doi.org/10.1101/035642
- 89.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活修剪器。生物信息学。2014;30:2114-20。
- 90.
Cantarel BL, Korf I, Robb SM, Parra G, Ross E, Moore B,等。MAKER:为新兴模式生物基因组设计的易于使用的注释管道。基因组决议2008;18:188-96。
- 91.
Bray NL, Pimentel H, Melsted P, Pachter L.近最优概率rna序列定量。生物工程学报。2016;34:525。
- 92.
Pimentel H, Bray NL, Puente S, Melsted P, Pachter L. RNA-seq定量不确定性的差异分析。2017;14:687。
- 93.
郭德华,陈志强,张志强,等。Phytozome:绿色植物基因组学的比较平台。核酸决议2011。https://doi.org/10.1093/nar/gkr944.
- 94.
UniProt:通用蛋白质知识库。核酸决议2016。https://doi.org/10.1093/nar/gkw1099.
确认
我们感谢L. Hydock对温室的支持;M. BtUCONN1花粉提供品牌及小檗授粉繁殖指导;T. Davis的批评性反馈;M.辅助流式细胞术的护盾;以及M. Macmanes在参考和转录组组装方面的指导。我们也要感谢处理编辑和匿名审稿人对这篇手稿早期版本的批判性反馈。
资金
这项工作得到了比尔和梅林达·盖茨基金会的支持[OPPGD1389, OPP1133199];以及美国农业部NIFA Hatch多州项目NE009 [NH00611-R]。部分资金由新罕布什尔州农业实验站提供(科学贡献号8203)。
作者信息
从属关系
贡献
IH对研究进行了概念化。IH和RB设计实验并协调各方面的工作。RB、AM和IH进行数据分析。SK参与了参考基因组的组装和评价。RB、AM、YJ、IH起草稿件;所有作者都修改了手稿并批准了最终形式。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
SK是开发单分子测序技术的太平洋生物科学公司的全职员工。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
附加文件
附加文件1:
测序详情b . thunbergii加入“BtUCONN1”,b .寻常的继承' Wagon Hill ',种间F1 (b×ottawensis)本研究中使用的映射种群。(xlsx20 kb)
附加文件2:
补充数字和表格。图S1。遗传连锁图b . thunbergii' BtUCONN1 '和b .寻常的“马车山”。图S2。支架主contigs的Hi-C热图b . thunbergii简历。“小鬼”。图S3.高优先候选基因的维恩图有待进一步研究。图S4。通过DGE分析发现的5个候选基因的时间过程表达图。图S5。逐基覆盖图b .寻常的通过存在-缺失分析鉴定出的两个候选基因为“Wagon Hill”。图S6.标记的凝胶图像用于验证F1人口的映射。表S1。获得的原始PacBio数据的摘要b . thunbergii简历。“小鬼”。表S2。总结统计了14种伪分子b . thunbergii简历。“狗头人”参考汇编。表S3。详细的十种组织库b . thunbergii简历。“狗头人”用于转录组组装。(PDF 5620 kb)
附加文件3:
的联动图b . thunbergii' BtUCONN1 '和相关信息。(xlsx412kb)
附加文件4:
的联动图b .寻常的附加“马车山”和相关信息。(xlsx251kb)
附加文件5:
7个候选基因的MAKER特征和详细功能注释。(xlsx13kb)
附加文件6:
补充文本。文本S1。用于验证F1组成B. × .的个体的状况ottawensis人口的映射。文本S2。本研究中使用的GBS-SNP-CROP命令行详细记录。文本S3。本研究中使用的FALCON总成参数的完整细节。文本S4。用于清除单plotigs的脚本的完整细节。文本S5。用于量化转录本的参数和时间过程分析的侦探R代码的完整细节。(PDF 128 kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
巴陶拉,R.,甜瓜,A.T.O,金安,S.。et al。种间小檗杂交种对茎锈病病原的非寄主抗性图谱。BMC植物生物学19日,319(2019)。https://doi.org/10.1186/s12870-019-1893-9
收到了:
接受:
发表:
关键字
- 小麦
- 茎锈病
- 伏牛花
- 不耐
- 持久的阻力
- 参考基因组