摘要
背景
盐度作为主要的非生物胁迫之一,严重威胁着包括水稻在内的世界主要粮食作物的生长和肥力。为了深入了解耐盐基因型响应盐胁迫的分子机制,我们提出了一种综合meta分析方法来寻找耐盐的关键基因。在此基础上,利用微阵列和RNA-seq数据进行全基因组meta分析,鉴定了耐盐水稻基因型的差异表达基因(DEGs)。然后通过meta-QTL分析和文献综述确认了DEGs。
结果
在46个Meta-QTL位置中检测到总共3449只DEG,其中分别在根,芽,幼苗和叶组织中观察到1286,86,1729和348°。Moreover, functional annotation of DEGs located in the meta-QTLs suggested some involved biological processes (e.g., ion transport, regulation of transcription, cell wall organization and modification as well as response to stress) and molecular function terms (e.g., transporter activity, transcription factor activity and oxidoreductase activity). Remarkably, 23 potential candidate genes were detected inSaltol和覆盖产量组分和离子稳态性状原始qtl的热点区域;其中,有许多未被报道的盐度响应基因。检测到果胶酯酶、过氧化物酶、转录调节剂、高亲和力钾转运蛋白、细胞壁组织、蛋白丝氨酸/苏氨酸磷酸酶和CBS结构域蛋白等候选基因。
结论
结果表明,耐盐基因型在盐胁迫的感知和信号传递、转录调节、离子稳态和活性氧清除等方面发挥了一定的作用。
背景
目前,水稻是世界上除小麦和玉米之外最重要的粮食作物,为全球超过35亿人提供了主要的热量来源[1.,2.].然而,水稻在苗期和繁殖期都被列为对盐分非常敏感的作物,而土壤盐分过剩是亚洲和欧洲一些河流三角洲最普遍的非生物胁迫之一[3.,4.].苗期的盐胁迫会导致水稻植株生长停滞或死亡,从而显著降低产量[5.,6.];因此,提高幼苗阶段的盐度耐受性将有效地改善水稻中的环境适应和产量维持。有必要了解盐度胁迫耐受性的机制,因为增加了人口,有限的耕地和气候变化,这可以为我们提供更好的视角,了解如何管理对高产大米的不断增加的需求[2.,7.].耐盐性在遗传和生理上都是一个复杂的性状[8.].米饭,作为一种研究的模型生物,特别是促进盐度应力反应的浓度7.].许多qtl最终在水稻育种计划中被发现[9,10.,11.,12.,13.,14.,15.,16.,包括1号染色体上的一个主要位点,即Saltol,参与Pokkali和SKC1衍生的Na/K稳态(OsHKT1;源自Nona Bokra [17.].分离已识别的与耐盐性相关的qtl可能对改善全球农业和粮食安全非常有益,但这也是一项具有挑战性的任务[18.].虽然已经发现了许多qtl,但目前对水稻耐盐相关基因网络的研究还很有限。基于测序方法的微阵列和基因表达谱等技术加速了对与环境应激反应相关的遗传机制的全面理解[19.,20.].近年来,高通量测序技术的快速发展和价格的下降,使得RNA测序在各种物种中的广泛应用[21.].因此,通过提到的技术在对比样本中鉴定了许多差异表达的基因(DEGS)。研究人员最近使用了DEG和QTL的集成为识别潜在候选基因的自信方法[22.].目前,大量多样的基因组数据已经公开;随后,大量可访问数据的组合可以提高结果的一致性和通用性。结合从独立但相关的研究中获得的结果被称为“meta分析(MA)”;因此,研究人员可以通过增加MA的统计功率来获得关于差异基因表达的更精确的估计[23.,24.].由于qtl在不同遗传背景和环境中的相互冲突,已鉴定的qtl的渐渗育种受到限制[25.];而meta QTL分析提供了一个利用不同遗传背景的不同作图群体的QTL数据来检测QTL准确位置的机会[26.].若干研究已经确定了具有各种特征的准确的Meta-QTL,用于在水稻和其他作物植物中开采候选基因[26.,27.,28.,29.,30.].然而,本研究采用综合荟萃分析的方法,发现了几个有前景的耐盐基因,其中一些重要的基因/基因家族被列出并在稍后进行讨论,以支持它们在水稻中的候选基因。利用以往研究中获得的所有数据,对水稻耐盐相关候选基因进行鉴定,然后利用meta分析对候选基因进行确认。本研究结果为水稻耐盐基因和途径的研究提供了有价值的信息。
结果
盐度耐受性相关的Meta-QTL
在本研究中使用简单的序列重复(SSR)标记,共收集与32个特征相关的265个QTLS(表S1,S2),从中筛选出126个和139个qtl,在正常和盐度条件下进行进一步分析(表2)S3).大部分qtl属于耐盐评分(STS)(27个)、地上部钾浓度(KS)(26个)、地上部钠浓度(NS)(21个)、叶绿素含量(CHL)(19个)和地上部干重(DSW)性状(19个)(图s1)。而少数qtl属于不育小穗数(NSS) [20.[死去的幼苗速率(DSR),叶钾浓度(KLV),减少幼苗高度(RSH)和叶面积(RLA)特征的还原(图3)。在染色体1(37 QTL)和2(36 QTL)上观察到最多的QTLS,然后是染色体7(29 QTL),而染色体8(12 QTL)和11(12 QTL)具有最低数量的QTL(图.s2)。由原始QTL描述的表型方差不同于0.7〜33.25%,标记的置信区间(CI)不同于0.99至84.36厘米(表S3)在整合所有收集到的QTL后,在水稻12条染色体上鉴定出46个meta QTL(图。1.).基于最低赤池信息标准(AIC)值的meta- qtl的CI为95%。值得注意的是,Chr7: M-QTL2、Chr2: M-QTL2和Chr1: M-QTL2上的第2个元qtl的初始qtl数量最高,分别为17、16和12个,覆盖的CI相对较窄,分别为4.78、1.82和2.84 cMS4).这些元qtl支持重要性状;例如,茎部钠钾浓度(NKS)、可育小穗数(NFS)、根长(RTL)和叶绿素含量(表S4).CHR12:M-QTL4,CHR 9:M-QTL3和CHR3:M-QTL2具有最高的表型变异百分比(R2.),可以被认为是涉及的特征的主要有效QTL(表S4).共检测到46个meta-QTL位点上的9366个基因,其中Chr8: M-QTL2包含的基因数量最多(868个);而Chr12: M-QTL2所含基因最少,为14个S4).此外,与分配假定功能的未注释基因的约73%相比,具有功能特征的注释基因的比例(27%)实际上是有限的。值得注意的是,在Chr1: M-QTL2上鉴定了81个基因,这些基因位于Saltol地区。
与盐耐盐相关的特征的Meta-QTL位置(表S1)在12米的12染色体上。染色体左侧的垂直线显示每个QTL的置信区间。标记名称和位置(共识地图上的CM)显示在左侧。颜色表示与耐盐性相关的特征的Meta-QTL位置
大米盐度耐受基因型中的表达分析分析
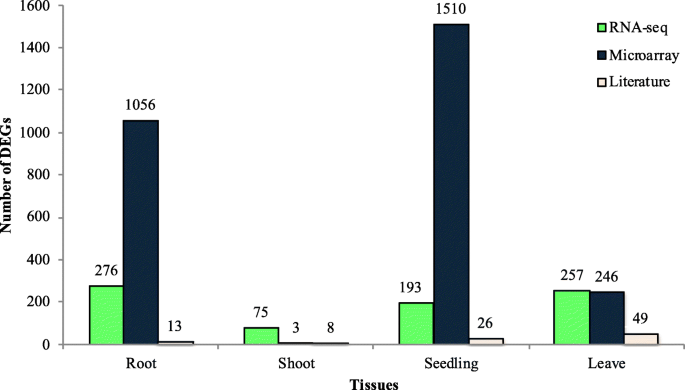
在耐盐基因型中,与对照相比,在盐胁迫条件下鉴定了DEGs。FL478的根中共检测到1714个耐盐基因型的deg,其中927和787在盐条件下上调和下调[31.].组合来自多个RNA-SEQ数据集的DEGS,将DEG分为根,芽,幼苗和叶子,以更深入地了解盐度耐受水稻基因型中的盐响应基因。共有3030,396,703和723℃分别以根,芽,幼苗和叶片鉴定出来(图3)。此外,下载了来自九个独立实验的原始微阵列数据(表S5),并进行统一分析。微阵列元分析显示有11694个DEGs,其中根、茎、苗、叶中分别有4121、13、6247和1199个DEGs单独表达(图s4)。在耐盐基因型中,仅有4763和5862个基因表达上调和下调。
来自两种meta分析方法的deg的整合
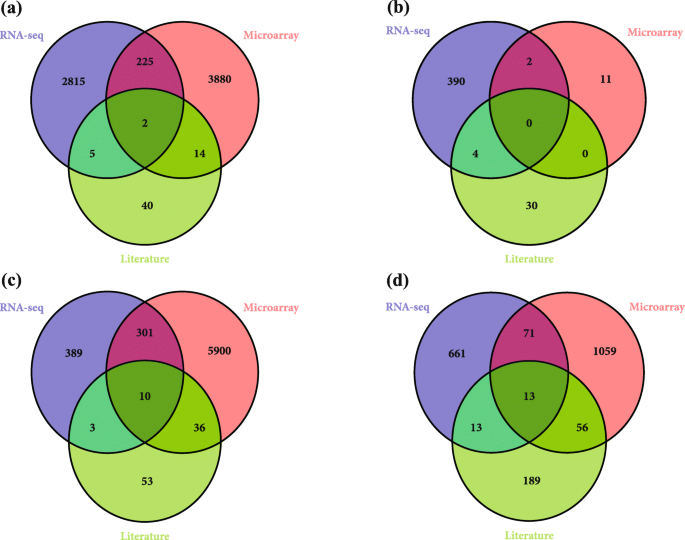
组合RNA-SEQ和微阵列间分析中的鉴定的DEG以确认所得结果的一致性。在除去所有重复基因后,在四个组织中检测到重叠的次数列表。
比较转录组分析表明,RNA-Seq和microarray在根、茎、苗和叶组织中分别检测到227、2 311和84个deg(图2)。2.).通过RNA-Seq和微阵列荟萃分析共鉴定出4255和10980个deg,而此前文献仅报道了156个deg(图)。2.).
检测meta-QTL位置上的deg
在根、茎、苗和叶的meta-QTL位置上分别有1345、86、1729和552个DEGs(图2)。3.).在元QTL位置确定的DEG中,通过RNA-Seq和微阵列荟萃分析分别确定了664和2359个DEG,而文献中之前仅报告了位于元QTL位置的82个DEG(图。3.).
位于meta-QTL位置的DEGs的功能注释
通过基因本体论富集分析确定位于meta-QTL位置的DEGs的生物学作用。在生物过程中,碳水化合物代谢过程、细胞过程调控、转录调控、应激反应和氮化合物代谢过程调控为优势项(BP)(图s5)。此外,转录调控、无机阴离子转运、阴离子转运、离子转运以及基因表达调控、细胞壁组织调控和修饰调控等BP术语显著富集(图s5)。最显著的过代表分子功能(MF)项为核苷酸结合、ATP结合、阴离子跨膜转运蛋白活性、无机阴离子跨膜转运蛋白活性、转录因子活性和氧化还原酶活性(图s5)。在细胞成分(CC)本体论中,最显著的富集项是膜固有项和膜固有项(Fig.S5)。
在meta-QTL位置上挖掘潜在的候选基因
通过对常见基因的meta-QTL区域的探索,在根中发现了60个潜在的候选基因(表1)S6),其中只有4个基因之前被报道与盐度反应有关。在ch1中发现了编码果胶酯酶的LOC_Os01g20980.1Saltol地区(表S6).在Chr1: M-QTL2中也发现了控制KLV、NS、NKS、KS和RN性状的离子稳态相关qtlS4).总的来说,将鉴定的潜在候选基因分类为根组织中的几个术语,例如转录因子(例如,TIFY,肝,霍克斯,怀疑和my家庭),信号(例如,OsWAK125,果胶酯酶,osmkk1.,奇特15)、运输(例如,oshkt1.以及一些编码跨膜转运和阴离子转运的基因)和一些其他功能(如:NUDIX.家族,编码天冬氨酸蛋白酶的基因)(表S6).
ch2、3和8的亚区中有4个基因被鉴定为芽中潜在的候选基因,如文献所述;例如,TIP2–1(loc_os02g44080.1)在chr2:m-qtl4中(表S6).Chr2: M-QTL4与7个控制RTL和其他相关性状(如S、KS、NKS、SIS和NS)的初始qtl整合(表2)S4).此外,在Chr3: M-QTL1和Chr8: M-QTL2中分别鉴定出两个转录因子(LOC_Os03g08310.1和LOC_Os08g15050.1)为可能的候选基因(表1)S6),分别支持根长和光合作用相关性状(表S4).值得注意的是,loc_os03g08310.1(编码)TIFY11A)被鉴定为根和茎中常见的候选基因(表S6).
我们的结果表明了幼苗中的98个潜在候选基因,其中包括尚未报告的M-QTL中的84次DEG。然而,文献中已经考虑了14个基因(表S6).这些潜在候选基因的功能分类进一步表明它们与转录调节有关(例如,AP2,怀疑,霍克斯,克家族)、信号传导(如:CIPK24,GDSL),在盐胁迫下,有一些具有激酶、磷酸酶、转运蛋白等功能的基因(表1)S6).值得注意的是,LOC_Os01g20830.1(编码转运蛋白)和LOC_Os01g21144.1(功能未知)在Saltolch1上的区域:M-QTL2S6).同时,在热点区域也有一些潜在的候选基因;例如,WRKY70(LOC_Os05g39720.1)在Chr5: M-QTL4和PP2C(loc_os06g483001) in Chr6: M-QTL4 (R2.= 10.31%)(表S4,S6).此外,一些基因被鉴定为CHR2:M-QTL1,CHR8:M-QTL1,CHR10:M-QTL3和CHR11:M-QTL1中的潜在候选基因;这些元区域综合了光合作用,稻草干重,产量组分(例如QGW,DF和NFS)和RTL特征的重要性(表格S4,S6).
在叶片中共鉴定出28个潜在候选基因,其中文献中发现14个。LOC_Os01g22249.1(编码过氧化物酶)位于Saltolch1: M-QTL2区被鉴定为另一个领先候选基因。值得注意的是,oshkt1.(LOC_Os06g48810.1)和PP2C(loc_os06g483001)位于Chr6: M-QTL4的热点区域(表1)S4,S6).
结果表明,20个基因位于含有产量构成和离子稳态性状的原始qtl的热点区域,这些基因可能是有希望的候选基因(图2)。4., 桌子1.).有希望的基因与以下功能有关:果胶酯酶、过氧化物酶、转录调节、高亲和力钾转运体、蛋白丝氨酸/苏氨酸磷酸酶、细胞壁组织和一个含CBS结构域的基因,其中有2个基因Saltol地区(表1.).
qRT-PCR方法验证差异基因表达
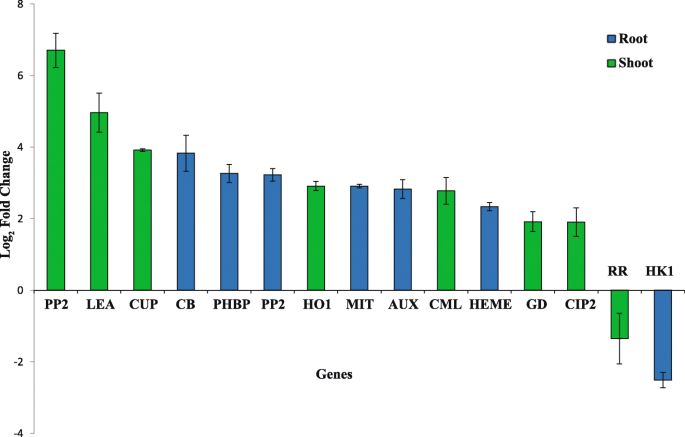
为了进一步验证候选基因,我们在FL478中选择15个基因作为耐盐基因型进行qRT-PCR(图4)。5.)qRT PCR结果证实了荟萃分析的结果(图S6)。
讨论
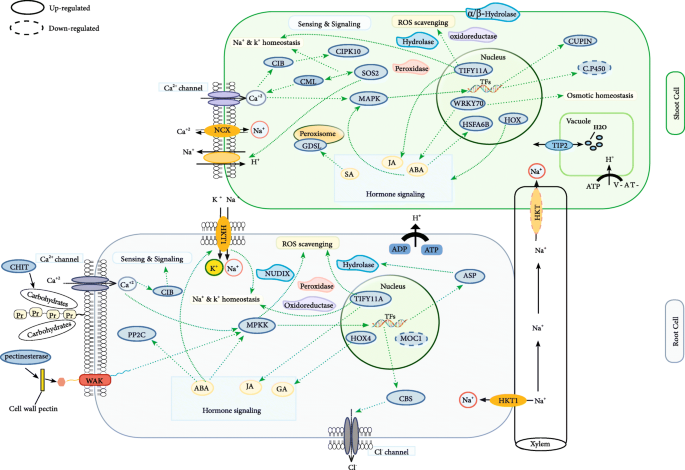
大米受到幼苗和生殖阶段的盐度应力的高度影响。高盐度浓度导致离子紊乱,脱水,渗透胁迫和氧化损伤。因此,重要的是识别最准确的QTL和所涉及的候选基因。在本文中,在耐受基因型中提供了位于Meta-QTL区域上的潜在候选基因和盐度应力条件中的差异表达的基因(图。6.).
传感和信号
通过复杂的多组分信号传导途径激活植物抵抗包括盐度的非生物应力的耐受性,以返回细胞稳态并促进存活率[32.].植物细胞壁是生物和非生物刺激感知的第一层,细胞壁重塑提供了应激反应的一般机制[33.].这是一些编码细胞膜和细胞壁组织完整组成部分的基因在热点区域。OsWAK125在Chr12: M-QTL1中发现,并在根中表达上调(表S6,无花果。6.),属于壁相关激酶家族,主要被研究为细胞壁“传感器”的潜在候选[34.,35.].壁相关激酶(WAKs)牢固地结合细胞壁的果胶网络,突出细胞膜,并将其连接到细胞质,在细胞质中丝氨酸/苏氨酸(Ser/Thr)激酶域负责进一步的信号传导[34.,35.].细胞壁相关的干旱和盐度响应基因(以果胶酯酶为代表)也被发现Saltol区域在根中上调S6,无花果。6.).在盐和干旱胁迫下,大豆、小麦和番茄等多种作物耐受性品种的果胶重塑酶水平高于敏感基因型[33.].在热点区域的幼苗阶段,几种Ser / Thr磷酸酶基因在叶片中差异表达(表S6).丝氨酸/苏氨酸磷酸酶在马铃薯、小麦、水稻等多种作物的适应性胁迫响应和信号通路调控中发挥重要作用[36.,37.,38.,39.,40].
osmkk1.Ch6中:M-QTL12和OsCHIT15在CH110:也检测到M-QTL3,在根中上调,并在水稻中介导盐度信号(表S6,无花果。6.)[41.].植物几丁质酶在应对非生物胁迫中起着重要作用;也有报道称几丁质酶水解碳水化合物链表明其可能在信号传导或渗透调节功能中发挥作用[34.].此外,位于meta-QTL区域的deg中有7个参与信号通路的水解酶编码基因(表1)S6),其中Chr5:M-QTL2和Chr6:M-QTL1中的两种gdsl样脂肪酶/酰基水解酶在盐胁迫下表达上调(表1)S6,无花果。6.).此外,oscipk24.(SOS2)在Chr6:M-QTL3和oscipk10ch3:M-QTL2在幼苗中上调(表2)S6,无花果。4.).CIPK (CBL- interaction Protein kinase)通路已成为调节水稻耐盐性的主要信号通路[42.,43.].一般的信号转导途径始于信号感知,接着是第二信使的产生)。,inositol phosphates and Reactive Oxygen Species (ROS)) and the transcription factors controlling the specific sets of stress-regulated genes [44.].
转录调控
转录因子对任何表型的出现都很重要,因为它们能够调节所有相关基因的表达[32.].HSFA6B(位于Chr1:M-QTL3,在幼苗中上调)作为ABA信号下游的正调控因子,直接与脱水反应元件结合(DREB)启动子结合,并增加其表达(表1)S6,无花果。6.)脱水反应元件结合蛋白2A(DREB2A)的上调可以激活不同植物物种中与胁迫耐受性相关的各种基因[45.].也有报道说,过度表达OSTIfI11(位于Chr3:M-QTL1,在茎和根中上调)通过茉莉酸(Jasmonic Acid, JA)信号和钾稳态调节提高了对盐胁迫的耐受性(表1)S6,无花果。6.)[46.].有OsHOX22和OsHOX24来自同源盒家族的Chr3:M-QTL1和Chr4:M-QTL3在幼苗中均表达上调(表1)S6,无花果。6.).OsHOX24是耐盐基因型(FL478)中在150 mM NaCl下上调最多的基因;而在盐敏感基因型(IR29)中则高度下调[31.].此外,角色OsHOX24已被发现通过微调水稻中应激响应基因的表达来调节非生物应激反应[47.].此外,有OsWRKY70在Chr5:M-QTL4中表达,并在幼苗中上调S6,无花果。6.).据说OsWRKY70作为通过SA-和ABA介导的信号传导的气孔闭合的负调节剂,在植物耐受渗透胁迫中起重要作用[48.].此外,GRAS(位于Chr5:M-QTL4,在根中下调)蛋白属于植物特异性转录因子家族,参与许多植物过程,包括植物的生长发育和非生物胁迫响应(表)S6,无花果。6.)[49.,50].据报道MOC1.编码GRAS家族的一个核转录因子。MOC1.充当侧分支或增加分蘖数的正调节因子[51].
ROS抑制
提高植物对有害环境条件(包括高盐浓度)适应能力的关键机制之一是调节有毒ROS水平[33.,52].Nudix水解酶在CHR4:M-QTL3中发现,并在根中上调(表S6,无花果。6.),一般清除过量的有毒代谢物或控制代谢途径中中间体的可及性[53].此外,ch1:M-QTL2中有一个属于抗氧化系统的过氧化物酶编码基因在叶片中上调(表)S6,无花果。6.)转基因拟南芥植物表达胞浆过氧化物酶基因,据报道表现出较高的耐盐性[20.].此外,存在属于含有蛋白质中的α/β折叠家族结构域的水解酶编码基因在CHR3中的蛋白质:M-QT13在幼苗中上调(表S6,无花果。6.).有报道称,与野生型相比,过表达一个编码α/β水解酶折叠酶的基因可显著提高甘薯的耐盐性,这是因为保护了膜的完整性,提高了甘薯清除ROS的能力[54].
离子稳态
在盐度应力下的离子助焊剂的调节是细胞在低水平下保持毒性离子浓度并收集必需离子的必要条件。盐度压力上调追踪族的追踪性基因如Na+和K+运输器和漂湿的na+/小时+换热器(55].在meta-QTL位点上观察到几种转运体,其中:HKT1在Chr6中发现:M-QTL4;在叶中下调,在根中上调(热点区,表S6,无花果。6.).高亲和力K+被称为Na的转运体+/ K+共同运输机减少了NA的运输+并积极调节水稻和拟南芥的耐盐性[56].在CHR1:M-QTL3和CHR3:M-QTL2中鉴定了用信号肽结构域编码真空蛋白的两个基因(表S6);在幼苗中上调。在Chr12中编码NCX的基因为M-QTL4;在幼苗中上调(表S6,无花果。6.),在CA中发挥着重要作用2+信号和离子稳态。钠/钙交换剂使用钠+通过质膜挤出细胞内Ca的电化学梯度2+[57,58].
其他耐盐相关的潜在候选基因
共发现23个未知的潜在候选基因,其中5个基因序列中含有CBS或cupin结构域。例如,一个含有CBS结构域的基因位于根中上调的Chr2:M-QTL1。以往的报道表明,它通过影响氯离子通道在耐盐和抗氧化应激方面发挥作用(Kushwaha et al. 2009)。也有报道说,过度表达OscBSX4.提高烟草转基因品系对盐和氧化胁迫的耐受性[59].
此外,在不同的M-QTL位点上发现了4个具有cupin结构域的基因(见表1)S6),而在幼苗中则上调。根据以往的报道,cupin结构域可能在盐胁迫下水稻种子的萌发中发挥作用,因为在与种子休眠、种子储备利用和种子萌发相关的qtl位置附近发现了具有cupin结构域的蛋白质[60].
结论
为了探究耐盐基因型响应盐胁迫的分子机制,我们采用综合方法鉴定了水稻耐盐相关候选基因。结果表明,耐盐基因型利用更有效的机制应对盐胁迫(图。6.)特别是在1)中的感测和盐胁迫的信号;编码细胞壁组织的几个基因,在差异基因型中差异表达的热点区域观察到甲基酯酶,甲基磷酸酶,Chitina酶,CIPKWERE。2)转录的调节;在Meta-QTL地区和DEGS中,发现了几种不同家庭的盐度敏感转录因子(TFS),包括细胞,MyB,HSF,Hox,Wrky,AP2和GRAS系列,这些都被证明可以发挥必要角色在水稻中的盐度耐受性。3)离子和渗透性稳态;一些运输司机也是如此有希望的候选基因等HKT1(NA / K运输机),NCX(钠/钙交换剂)和TIP2-1(水通道蛋白)。4) ROS清除;位于meta-QTL位置的DEGs中有许多与解毒相关的重要基因,如水解酶、氧化还原酶和过氧化物酶。对这些有潜力的候选基因的进一步研究,可以为通过基因工程或分子育种提高基因型的耐盐性提供有益的信息。
方法
荟萃分析的主要
准备QTL数据
收集了所有报道的与水稻耐盐性相关的qtl(2009 - 2018年),包括之前发表的15项研究中确定的qtl [9,10.,11.,12.,13.,14.,16.,61,62,63,64,65,66,67,68].提供了QTL资料,包括亲本、定位群体类型和数量以及每个性状的QTL数量。此外,对每个QTL的侧翼分子标记、置信区间(CI)、QTL位置、概率对数(LOD)评分和表型方差解释比例(PVE或R2)进行了评价。本研究使用的qtl来源于不同群体类型(包括:F2、回交株系(BC3F4)、重组自交系(RILs))以及不同组织在幼苗和生殖发育阶段的大小(从87到285株)(见表)S1).
共识图谱和QTL定位
使用BioMercator Software识别共识QTL区域[69].国际水稻微卫星倡议(IRMI)的地图可在以下网站获得https://archive.grachene.org.(IRMI_2003)作为Meta-QTL分析的参考图。在将QTL投影到共识图上之前,使用以下公式计算初始QTL的95% CI:
- (我)
F2行:\(CI= frac{530}{N\times {R}^2} \)
- (2)
对于双单倍体(DH)系:\(ci = \ frac {287} {n \ times {r} ^ 2} \)
- (iii)
对于RIL:\(ci = \ frac {163} {n \ times {r} ^ 2} \)
N表示人口规模和R2.是相关QTL解释的表型变异的百分比。初始QTL的标记间隔之间的缩放规则用于共识染色体地图上的QTL位置。
QTLS的Meta分析
使用BioMercator V4.2工具中的默认参数集进行Meta分析。利用软件计算了1、2、3和n个QTL。采用赤池信息准则(AIC)筛选各染色体上的QTL模型[70].根据AIC值,具有最低AIC值的QTL模型被认为是一个重要的模型。
RNA序列
RNA-SEQ数据是从我们以前的两项对比基因型的研究中获得的栽培稻在盐度胁迫下[31.].简单地说,用150 mM NaCl处理FL478(耐盐)和IR29(盐敏感)幼苗,并在盐胁迫开始24小时后采集根系样品。此外,还收集了正常样本(在相同条件下但未经过盐度处理)作为对照样本[31.].用纯化的RNA构建cDNA文库;随后使用IlluminaHiSeq™2500测序仪对合格库进行测序。转录组原始数据包括FL478的对照(SRR7944745和SRR7944784)和盐处理样品(SRR7944792和SRR7944793), IR29的对照样品(SRR7945188和SRR7945229)和盐处理样品(SRR7945230和SRR7945234)在NCBI数据库的SRA (Sequence Read Achieve)中可查到。数据集的质量是使用FastQC工具进行的[71].利用TopHat技术,利用水稻参考基因组序列IRGSP 1.0 (ftp://ftp.ensemblgenomes.org/pub/植物) [71].原始测序reads然后通过Cufflinks和Cuffmerge元汇编工具组装[71].最后,通过Cuffdiff效用识别出具有log2 fold change≥1(上调基因)和≤(−1)(下调基因)的degQ -临界值≤0.05。
根据组织对基因表达数据进行meta分析
RNA-seq Meta-dataset
到本分析完成时,可获得的盐胁迫下水稻植株转录组数据集来自美国国家生物技术中心(NCBI)数据库(见表)S7).基因− 1. ≥ log2倍变化≥ 1和有效Q值(FDR ≤ 5%)被认为是来自这些RNA-seq数据集的DEG,并被分为四个组织(即地上部、根、幼苗和叶)。
微阵列元分析
盐胁迫下水稻的表达数据来源于NCBI的基因表达综合库(GEO) [71,72].从affymetrix平台Rice Genome Array (affymetrix或Agilent微阵列平台)下载了9组GEO数据集(表1)S5).对每一组表达式数据分别进行预处理。R程序中的LIMMA包用于分析Agilent微阵列数据[73],而Affy软件包在R程序中处理affymetrix平台。每个源的原始数据通过分位数归一化和鲁棒多阵列平均背景校正进行预处理。然后,从程序标准设置中删除低强度和非信息性的探针;然后,将探针转换为o它们的相关基因组位置。采用RMA对后续MA的值进行标准化。然后,使用LIMMA软件包计算每个治疗组与其对照组之间的差异。将数据拟合到线性模型后,使用简单的经验贝叶斯模型修正标准误差。对于每个基因中的每个对比,计算差异表达的缓和t统计量和对数几率− 1. ≥ log2倍变化≥ 1及Q -4个组织中DEGs的临界值均≤0.05。
融合了大部分基因表达和对DEG的文献引用
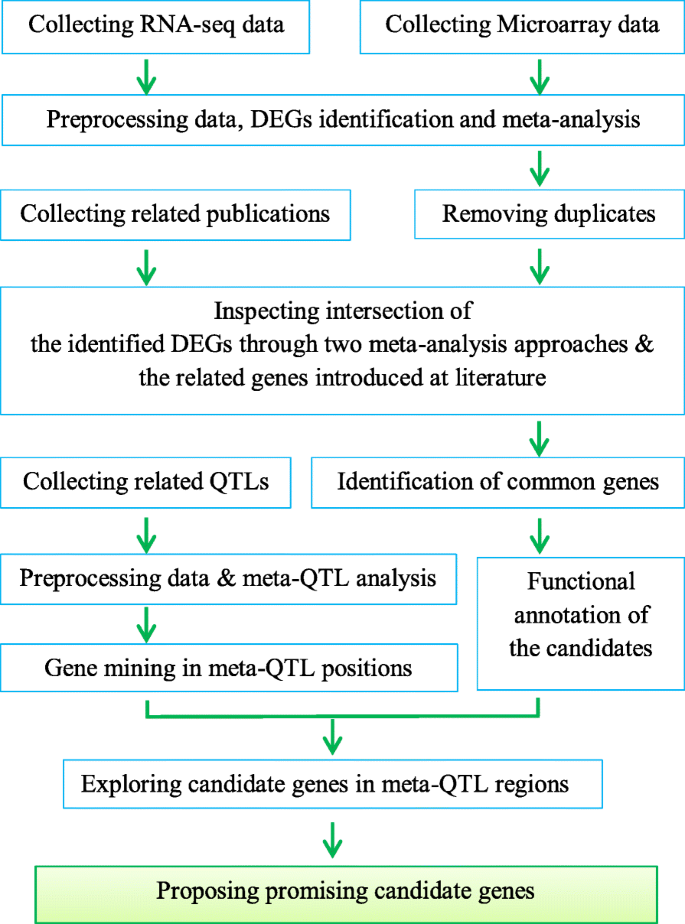
本研究提出了一个新的数据处理管道,整合不同的数据类型来识别水稻耐盐相关的候选基因(图。4.).一方面,在微阵列和RNA-SEQ技术中响应水稻中的盐度胁迫而集成了DEG。另一方面,NCBI(NCBI;www.ncbi.nlm.nih.gov)检索文献以鉴定已发表的关于水稻耐盐基因的报道。本研究回顾了111篇论文,收集了所有已报道的水稻耐盐相关基因。所有已鉴定的基因分为四个组织(包括地上部、根系、幼苗和叶)(表1)S8).用维恩图(使用R包)比较不同方法(包括RNA-seq、微阵列和文献综述)检测到的每个组织的重叠基因,并检测常见基因。最后,在耐盐相关的meta qtl区域中寻找与meta qtl位置一致的DEGs。为了鉴定meta-QTL区域内的deg,利用已鉴定的mqtl的侧边标记检测每个meta-QTL的物理间隔。然后,根据水稻基因组组装IRGSP 1.0找到位于meta- qtl区域的基因。
功能辅助和途径分析
使用Agrigo公共网络工具进行DEG的浓缩分析[74]。使用Fisher精确检验(Q值)将过度代表的GO术语分为三大类,包括“生物过程”、“分子功能”和“细胞成分”< 0.05),并通过错误发现率(FDR)方法在P< 0.05。
耐盐相关候选基因的meta-QTL定位
在本文中称为至少两种方法(包括RNA-SEQ,微阵列,微阵列,文献综述)的基因被称为常见基因。寻求盐度公差相关的Meta-QTLS区域以找到常见的基因。潜在的候选基因。位于覆盖原始QTL的热点区域的潜在候选基因被认为是产生组分和离子稳态性状性状的原始QTL,作为承诺的候选基因(图。4.).
植物生长与盐胁迫处理
耐盐水稻FL478种子(栽培稻L.)基因型由国际水稻研究所(IRRI)提供。如前所述进行种子灭菌和萌发,以及植物生长条件[31.].盐胁迫开始24小时后,采集21日龄幼苗的根和茎样品,用150mm NaCl处理,立即置于液氮中,置于−80℃,直到RNA提取。
RNA提取和cDNA文库合成
通过100mg芽和根组织的RNEAY植物试剂盒(QIAGEN)进行总RNA提取。使用NanoDrop ND-1000®分光光度计和琼脂糖凝胶电泳检查RNA样品的完整性和质量。使用与制造商的指示一致的IscriptTM cDNA合成试剂盒(生物缺陷)进行cDNA文库合成。
利用qRT-PCR方法验证耐盐相关候选基因
从候选基因列表中随机选取15个基因在每个组织中(表1)S6)通过定量实时PCR(RT-QPCR)进行验证。每个基因的特异性引物对(表S9对于列表底漆)由Oligo 7.0(National Bioscience Inc.,USA)设计。具有三个独立生物重复的QRT-PCR是由LightCycler®96实时PCR系统(Roche Life Scients,德国)和Sybr预混物完成的,而不是基于制造商的协议。将水稻(OS03G0836000)的肌动蛋白基因作为合适的内部对照基因。从三个生物重复的提名基因的转录物水平计算为2-ΔΔct[75].
缩写
- 度:
-
差异表达基因
- QTL:
-
数量性状位点
- 嘛:
-
荟萃分析
- ROS:
-
反应性氧气
- KLV:
-
K+在叶营养
- NS:
-
拍摄钠浓度
- NKS:
-
茎部钠、钾浓度之比
- KS:
-
拍摄钾浓度
- RN:
-
根Na+浓度
- 姐姐:
-
盐损伤分数
- QGW:
-
1000粒重(克)
- DF:
-
天开花
- NFS:
-
可育小穗数
- BP:
-
生物过程
- 走:
-
基因本体论
- MF:
-
分子功能
- CC:
-
蜂窝组件
- m-qtl:
-
Meta-QTL
- 置信区间:
-
置信区间
- LOD:
-
比值比的对数
- AIC:
-
Akaike信息标准
参考文献
- 1。
张辉,等。我国马铃薯主粮研究与产业发展进展。农业工程学报。2017;16(12):2924-32。
- 2。
邢颖,张强。水稻产量的遗传和分子基础研究。植物学报2010;61:421-42。
- 3.
黄涛,等。水稻耐盐性的提高:挑战与机遇。农学。2016;6(4):54。
- 4.
Frouin J等人。粳稻温和盐度应激的耐受性:基因组 - 宽协会映射研究突出了钙信号传导和代谢基因。Plos一个。2018; 13(1):E0190964。
- 5.
曾吕,香农MC,莱克斯SM。盐度应力的时间影响水稻生长和产量组分。农业水管理。2001; 48(3):191-206。
- 6.
赵旭,等。两种水稻基因型苗期耐盐性的比较代谢谱。《公共科学图书馆•综合》。2014;9 (9):e108020。
- 7.
Ray DK等人。气候变化解释了全球农作物产量变化的三分之一。Nat Commun。2015;6:5989。
- 8.
Joseph B,Jini D,Sujatha S.不同水稻植物非生物盐胁迫反应特异性的生物学和生理学观点.亚洲农业科学杂志,2010;2(3):99-105。
- 9.
水稻渐渗群体耐盐性状和耐盐预选育系qtl的鉴定。植物分子生物学报告。2018:1-15。
- 10.
Mohammadi R,等。水稻(Oryza sativa L.)生殖期盐胁迫下与产量和产量构成相关的数量性状位点定位。基因杂志。2013;92(3):433-43。
- 11.
王胜,等。结合RNA测序和QTL定位,对水稻苗期耐盐相关候选基因进行了鉴定。植物学报2017;8:1427。
- 12.
耐盐水稻地方品种‘Pokkali’渗入系苗期耐盐qtl的鉴定与验证。《公共科学图书馆•综合》。2017;12 (4):e0175361。
- 13。
Kim D-M等。水稻品种间渐渗系群体耐盐性qtl的定位。作物科学与生物技术。2009;12(3):121。
- 14。
梁j -l等。在盐或碱性应激下在水稻幼苗阶段的盐或碱性耐受鉴定QTL。Euphytica。2015; 201(3):441-52。
- 15.
潘伟迪等。结合QTL定位和转录组分析,鉴定水稻耐盐基因的功能多态性。中国生物医学工程学报。2010;28(2):121-36。
- 16.
Sabouri H等人。QTLS与幼米幼苗盐耐盐相关的生理性状的映射。BIOL植物。2009; 53(4):657-62。
- 17.
Rahman MA等。通过分子和生理特性探索水稻耐盐性的新遗传来源。安机器人。2016;117(6):1083 - 97。
- 18.
任Z-H等人。用于耐盐性的水稻定量性状基因座编码钠转运蛋白。NAT Genet。2005; 37(10):1141。
- 19.
Barnes M等人。染色型和Illumina基因表达分析平台的实验比较与交叉验证。核酸RES。2005; 33(18):5914-23。
- 20。
Lu Z,Liu D,Liu S.两种水稻细胞质抗坏血酸过氧化物酶差异提高转基因拟南芥的耐盐性。植物细胞代表2007;26(10):1909–17.
- 21.
加尔伯M,等。转录组注释和使用RNA-SEQ定量的计算方法。NAT方法。2011; 8(6):469。
- 22.
徐慧敏,等。甘蓝型油菜荚果的RNA-Seq转录组分析及脂质相关候选基因的鉴定。BMC基因组学。2015;16(1):858。
- 23.
ramasamy a等人。对基因表达微阵列数据集进行荟萃分析的关键问题。Plos med。2008; 5(9):E184。
- 24.
Tseng GC, Ghosh D, Feingold E.微阵列meta分析的综合文献综述和统计考虑。核酸学报2012;40(9):3785-99。
- 25.
价格啊。信不信由你,qtl是准确的!植物科学进展。2006;11(5):213-6。
- 26.
水稻苗期耐盐性相关数量性状的荟萃分析(oryza sativa l.).植物。2019;8(2):33。
- 27.
Swamy BM,Sarla N. Meta分析的产量QTL源自稻米际跨越米米米的互联网和候选基因。植物mol biol报告。2011; 29(3):663-80。
- 28.
吴y等。与水稻穗相关性状的定量特性鉴定与荟萃分析。Mol Gen Genomics。2016; 291(5):1927-40。
- 29.
等。水稻根系遗传结构:来自干旱QTL数据库的meta分析。大米。2009;2(2):115。
- 30.
张H等人。玉米低磷耐受的荟萃分析及候选基因开采。J Intent Plant Biol。2014; 56(3):262-70。
- 31.
等。解剖水稻耐盐的分子机制:对比基因型的比较转录分析。大米。2019;12(1):13。
- 32.
陈志强等。水稻耐盐性研究进展。Theor Appl Genet. 2019:1-20。
- 33.
等。禾本科与非生物胁迫:干旱和盐胁迫的重点,最近的见解和展望《前线工厂科学》2017;8:1214。
- 34.
植物细胞壁蛋白在盐胁迫反应中的作用。Sci World J. 2014;2014。
- 35.
wall -associated kinase WAK1与细胞壁果胶以钙诱导构象相互作用。植物生理学报。2005;46(2):268-78。
- 36。
País SM等。马铃薯(Solanum tuberosum)和番茄(Solanum lycopersicum)蛋白磷酸酶2A型催化亚基的特性及其在胁迫反应中的参与足底。2009;230(1):这边是。
- 37。
País SM, Téllez-Iñón MT, Capiati DA。2A型丝氨酸/苏氨酸蛋白磷酸酶及其在胁迫信号中的作用。植物学报。2009;4(11):1013-5。
- 38。
余荣民,等。水稻(Oryza sativa L.)蛋白磷酸酶2A催化亚基基因第二亚家族的结构、进化和表达。足底。2005;222(5):757 - 68。
- 39。
余荣民,等。两个编码蛋白磷酸酶2A催化亚基的基因在水稻中有差异表达。植物学报。2003;51(3):295-311。
- 40。
徐c,等。小麦(Triticum aestivum)蛋白磷酸酶2a催化亚单位基因在烟草中提供增强的抗旱耐受性。Ann Bot。2007; 99(3):439-50。
- 41。
植物中MAP激酶网络的信号转导。生物化学学报。2006;452(1):55-68。
- 42。
刘文志,等。油菜钙调磷酸酶b样蛋白CBL4与cbl相互作用蛋白激酶CIPK24相互作用,调控植物的耐盐性。生物化学学报。2015;467(3):467 - 71。
- 43。
Abdula se等。BRCIPK1基因的过度表达通过增加水稻中的脯氨酸生物合成来增强非生物胁迫耐受性。植物mol biol报告。2016; 34(2):501-11。
- 44。
朱建坤。寒冷、干旱和盐胁迫下的细胞信号。植物学报。2002;14(增刊1):S165-83。
- 45.
黄B等人。Eruca Vesicaria Subsp Sativa中脱水响应元结合蛋白2A基因的克隆与表征。genet mol res。2016; 15:23-9。
- 46.
叶华,等。水稻胁迫和激素反应中TIFY家族基因的鉴定和表达谱分析acta botanica sinica(云南植物学报). 2009;40(3):457 - 461。
- 47.
Bhattacharjee A,Sharma R,Jain M. OSHOX24的过表达赋予转基因水稻中非生物胁迫的增强易感性通过调节应力响应基因表达。前植物SCI。2017; 8:628。
- 48.
李军,等。防御相关转录因子WRKY70和WRKY54通过调节拟南芥的气孔大小来调节其对渗透胁迫的耐受性。新植醇。2013;200(2):457 - 72。
- 49.
Gremplet J等人。葡萄牙葡萄牙Gras基因家族的结构和功能分析表明GRAS蛋白在控制发育和应激反应中的作用。前植物SCI。2016; 7:353。
- 50.
李鹏,等。BrLAS是一种GRAS转录因子甘蓝型油菜,参与转基因拟南芥的抗旱性。植物学报2018;9:1792。
- 51.
Sakamoto T,Matsuoka M.水稻产量基因的识别和利用.Curr Opin Plant Biol.2008;11(2):209-14。
- 52.
侯赛因MS, Dietz K-J。盐胁迫下氧化还原调节机制、活性氧和氧化还原稳态的调节。植物学报2016;7:548。
- 53.
等。通过敲除Nudix水解酶19 (AtNUDX19)基因,拟南芥(Arabidopsis thaliana)叶片和根系中nadph循环系统在砷胁迫下的激活得以加速。植物生理学杂志。2016;192:81-9。
- 54.
刘丹,等。一个新的α/β水解酶基因IbMas增强甘薯耐盐性。《公共科学图书馆•综合》。2014;9 (12):e115128。
- 55.
朱镕基j - k。盐胁迫下离子稳态的调节。植物学报。2003;6(5):441-5。
- 56.
王锐,等。水稻高亲和钾转运体1;1参与耐盐性,受myb型转录因子调控。植物杂志。2015;168(3):1076 - 90。
- 57.
廖军,等。钠/钙交换器胞外离子交换和结合位点闭塞的机制。中国生物医学工程学报。2016;23(6):590。
- 58.
王志强,王志强,王志强,等。钠钙交换(NCX)蛋白中离子转运和变构调控的结构特征。杂志。2016;7。
- 59.
等。水稻CBS结构域蛋白的过表达提高了转基因烟草的耐盐性、氧化性和重金属性。生物科技摩尔》。2012;52(3):205 - 16。
- 60.
徐娥,等。蛋白质组学分析揭示了盐胁迫下水稻种子吸胀过程中涉及的蛋白质。前沿植物科学。2017;7:2006。
- 61.
pram VRR等。利用耐盐地方品种博克纳渐渗系对水稻苗期耐盐性的遗传分析。J在这里。2017;108(6):658 - 70。
- 62.
王志,等。不同NaCl胁迫下水稻根系和地上部Na+和K+含量的QTL分析(oryza sativa l.).《公共科学图书馆•综合》。2012;7 (12):e51202。
- 63.
潘伟迪等。结合QTL定位和转录组分析,鉴定水稻耐盐基因的功能多态性。中国生物医学工程学报。2010;28(2):121-36。
- 64。
等。水稻耐盐盐盐醇数量性状位点的鉴定。大米。2010;3(2):148。
- 65。
田丽,等。水稻苗期耐盐性相关数量性状位点的鉴定。中国基因工程杂志。2011;38(12):593-601。
- 66。
王志,等。不同盐胁迫下水稻幼苗耐盐性主要互作、上位性互作和QTL×环境互作qtl的鉴定中国科学(d辑:地球科学)2012;
- 67。
郑华,等。基于连锁关联分析的NaCl胁迫下粳稻地上部和根系Na+和K+浓度QTL分析Euphytica。2015;201(1):109 - 21所示。
- 68。
程L等人。用两套互易血液渗入耐盐背景效果鉴定耐盐性QTLS。基因组。2011; 55(1):45-55。
- 69.
生物erbc V3:遗传图谱编制和数量性状位点元分析算法的升级。生物信息学,2012,28(15):2082 - 3。
- 70。
数量性状位点:一项荟萃分析。遗传学。2000;155(1):463 - 73。
- 71.
Trapnell C,等。TopHat和袖扣RNA-seq实验的差异基因和转录表达分析。Nat Protoc.2012;7(3):562。
- 72.
Barrett T等。NCBI GEO:功能基因组学数据集更新档案。核酸研究。2012;41(D1):D991-5。
- 73.
史密斯星期。Limma:用于微阵列数据的线性模型,在生物信息学和计算生物学解决方案中使用R和Bioconductor:施普林格;2005.p . 397 - 420。
- 74.
Tian T et al. agriGO v2。0:用于农业社区的GO分析工具包,2017年更新。核酸Res. 2017;45(W1): W122-9。
- 75.
Livak KJ, Schmittgen TD。使用实时定量PCR和2−ΔΔCT方法分析相关基因表达数据。方法。2001;25(4):402 - 8。
致谢
作者感谢伊朗农业生物技术研究所(ABRII)的支持,以及穆罕默德·杰达里先生帮助创作的艺术品。
资金
本研究得到了伊朗伊斯兰共和国生物技术发展委员会的财务支持,授予:950618(和伊朗国家科学基金会(INSF授予NO:98014939)。
作者信息
隶属关系
贡献
RMM进行了实验,并起草了手稿。Z-SS构思了项目,监督和协调了研究,并修改了手稿。RMM分析RNA- seq数据,RMM和PD进行meta-QTL分析,RMM和S-MM分析Microarray数据。NBJ和MRG检查了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意
Springer Nature在公布的地图和机构附属机构的管辖权主张方面保持中立。
补充信息
附加文件1:表S1。
用于水稻耐盐性状meta-QTL分析的QTL定位研究列表表S2。对与耐盐性状相关的qtl进行了总结。表S3。meta分析中包含的与耐盐性相关的qtl。表S4。稻米荟萃分析鉴定的32种性状的共识QTL。表S5。选择用于盐胁迫下水稻meta分析的原始微阵列数据集。表S6。meta- qtl区域可能的候选基因列表(meta位置列上的星号表示位于热点位置的有潜力的基因)。表S7。本研究使用了可公开访问的RNA序列数据集列表。表S8。在文献综述的基础上,将已报道的水稻耐盐相关基因列表分为4个组织(包括茎、根、苗和叶)。表的年代9.用于qRT-PCR分析的引物列表。图S1。与每个耐盐相关性状相关的原始QTL数量(表S2中提供了性状及其缩写)。图。S2。水稻各染色体中与耐盐性相关的qtl数量。图S3。通过RNA-seq荟萃分析在四种组织(包括茎、根、苗和叶)中发现的差异表达基因(DEGs)的数量。图S4。在四种组织(包括茎、根、苗和叶)中,通过微阵列元分析鉴定出差异表达基因(DEGs)的数量。图。S5。将meta-QTL定位的DEGs进行GO项划分,主要分为细胞成分、分子功能和生物过程三大类。图。S6。FL478候选基因的qRT-PCR熔体曲线。
权利和权限
开放访问本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图像或其他第三方材料都包含在本文的知识共享许可中,除非在该材料的信用额度中另有说明。如果资料不包括在文章的知识共享许可协议中,并且你的预期用途没有被法律规定允许或超过允许用途,你将需要直接从版权所有者获得许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有用入数据的信用额度。
关于这篇文章
引用这篇文章
Mirdar Mansuri, R., Shobbar, ZS。巴巴多斯耶洛达尔et al。水稻耐盐候选基因的整合荟萃分析方法。BMC植物BIOL.20,452(2020)。https://doi.org/10.1186/s12870-020-02679-8
收到了:
认可的:
发表:
关键字
- 元分析
- RNA-SEQ.
- 微阵列
- 法
- 盐度的压力
- 栽培稻