图3
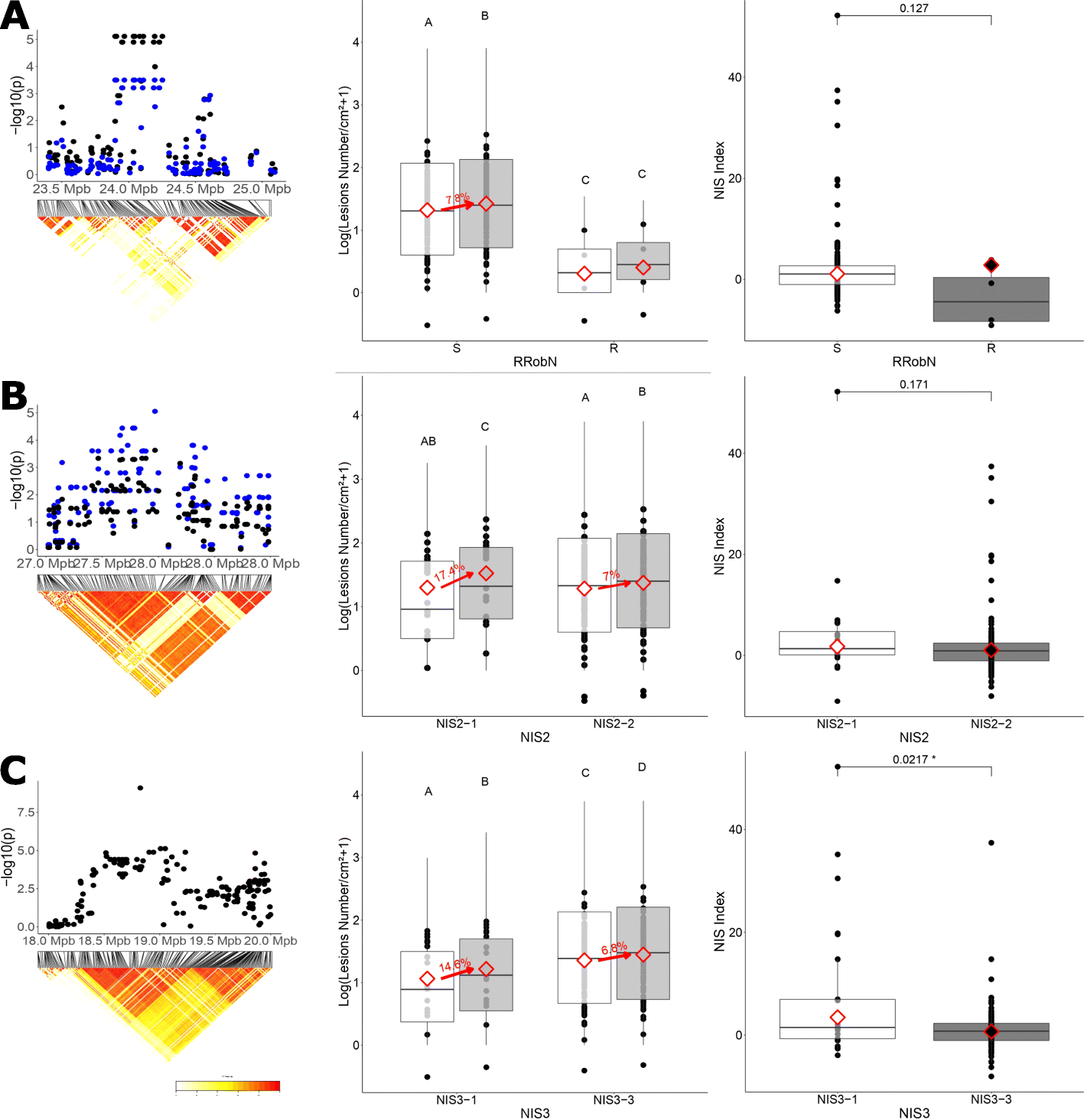
温带粳稻中控制NIS的3个位点与稻瘟菌的遗传和表型关联。通过对CD203分离株的疾病水平进行GWAS分析,鉴定出3个基因座: RRobN1位点(23,9 Mpb−24,2Mpb,位于第6染色体;一个),NIS2位点(第5染色体27.55-27.96 Mb;b),NIS3位点(第11染色体上18,4mpb - 19,2mpb;c)。对于每个位点,左边的数字表示QTL在各自染色体上的物理位置。热图显示了由R2每个snp之间的计算。建立了包含显著单核苷酸多态性的整个LD区段的QTL。对于A和B,这些点表示snpp- GWAS的爆炸敏感性值。黑点代表p-值表示在N0条件下得到的值,蓝色表示N1条件。对于C,这些点表示在GWAS中鉴定的NIS指数表型的snp的p值。中间的图显示了在两种氮条件下,每个QTL的每个单倍型对CD203菌株的敏感性。每个黑点对应一个基因型稻瘟病病斑数的最小平均数。白色中,N0对应低氮条件,灰色中N1对应高氮条件。红钻石对应于每一处理的平均lmean。红色箭头表示氮条件显著影响时敏感性增加的值。统计组是通过与Tukey调整的两两比较确定的,Tukey调整是基于试验和基因型作为协变量的模型。右边的数字代表每个QTL单倍型的NIS指数。虽然该指数仅用于识别NIS3,我们也展示了它RRobN1和NIS2进行比较。每个点对应每个基因型的NIS指数。红钻石对应于每个单倍型的Lsmeans。P的Wilcoxon检验得到的值一个和b,和一个学生测试后的对数变换c
