摘要
背景
豇豆(豇豆属unguiculata)是干旱半干旱地区的重要作物,是研究耐旱性的良好模型。MicroRNAs (miRNAs)在植物胁迫响应中发挥着重要作用,但在豇豆中尚未发现与干旱相关的miRNAs。此外,miRNAs如何在不同基因型豇豆中发挥不同的耐旱能力还不清楚。
结果
我们从两种豇豆基因型(CB46,干旱敏感型和IT93K503-1,耐旱型)中提取了深测序小RNA,它们生长在充足的水分和干旱胁迫条件下。我们将小RNA reads映射到豇豆基因组序列,并鉴定出属于89个家族的157个miRNA基因。44个干旱相关miRNAs中,30个在干旱条件下上调,14个下调。尽管miRNA在两种基因型中的表达基本一致,但我们发现有9种miRNA在其中一种基因型中主要或独家表达,有11种miRNA仅在一种基因型中受干旱调控,而在另一种基因型中不受干旱调控。
结论
这些结果表明,mirna可能在豇豆抗旱性中发挥重要作用,并可能是决定不同基因型豇豆抗旱性水平的关键因素。
背景
干旱是导致作物减产或完全损失的主要非生物因素之一。由于世界上许多地区的农业用水越来越有限,因此研究耐旱的自然机制是了解干旱胁迫反应的生物学基础和选择具有更好耐旱性的植物的重要策略[1,2].豇豆(豇豆属unguiculata(l)Walp。]在非洲、亚洲、中美洲和南美洲的半干旱和干旱热带地区,豇豆是一种重要的经济作物,在这些地区,豇豆被用作人类的食物和牲畜的营养饲料[3.,4].作为豆科植物,豇豆属于同一部落(Phaseoleae),如普通豆和黄豆。与这些近亲和大多数其他作物相比,豇豆能够很好地适应这些地区,因为它能够在贫瘠的土壤中固定氮,并且具有更强的耐旱性。4,5].因此,豇豆是研究其耐旱性遗传基础的优良体系。
人们已努力鉴定与豇豆干旱胁迫反应有关的遗传元素。例如,通过cdna的克隆和表征,已有十几种基因被证明与干旱胁迫反应有关[6- - - - - -12].此外,还在豇豆中定位了10个与幼苗耐旱性相关的QTL [13].然而,目前还不清楚干旱相关豇豆基因或位点的表达是如何被调控的,以及小rna是如何参与调控的。
MicroRNAs (miRNAs)是20-24 nt单链RNA分子,由折叠成茎环结构的RNA前体加工而成[14].MiRNAs在转录后水平调控靶mrna的基因表达,这是通过近乎完美的碱基互补来识别的。在mirna -靶标识别后,通常通过mRNA切割或翻译抑制对靶标进行负调控[15].功能分析表明,miRNAs参与植物的多种发育过程[16].此外,miRNAs在植物抵抗各种非生物和生物胁迫方面发挥着关键作用[17- - - - - -19].特别是,已经有几种方法用于研究与植物抗旱能力有关的mirna。在一项关于应激反应mirna的开创性研究中,Sunkar和Zhu [20.]利用小RNA克隆技术鉴定出26种新型miRNAs,其中miR393、miR397b和miR402脱水上调,miR389a下调。另一个miRNA家族miR169被发现在干旱胁迫下通过aba依赖的途径下调。miR169的抑制导致其靶基因NFYA5的高表达,进而增强植物的抗旱性[21].通过微阵列杂交或小RNA深度测序的全局miRNA表达谱实验,发现了更多在干旱条件下上调或下调的miRNA [22- - - - - -25].
尽管在包括豆科植物在内的许多植物物种中已经发现了大量的miRNAsMedicago truncatula[26,27],大豆[28]、和豆[29],在miRBase注册表中仅报道了两个豇豆的序列。最近在豇豆中预测了47个潜在miRNA,属于13个miRNA家族[30.].在另一项研究中,鉴定出了属于16个家族的18个保守mirna [31].两项研究都使用同源搜索方法来鉴定豇豆在其他植物中保守的mirna。在这项研究中,我们使用Illumina深度测序技术生成小RNA reads,并利用这些reads鉴定豇豆中的mirna,特别是豇豆特异性mirna和与耐旱性相关的mirna。据我们所知,这是第一个通过直接克隆豇豆小RNA鉴定出miRNAs的报道。
尽管豇豆品种具有抗旱性,但它们的抗旱性却有显著差异[32- - - - - -34].对干旱敏感性不同的植物基因型的研究和比较是发现自然耐旱机制的一种有前途的途径[35].为了深入了解mirna在耐旱性中的作用,我们使用了两种具有代表性的豇豆基因型:加州黑眼46号(CB46)和IT93K503-1。对干旱敏感的CB46是美国种植最广泛的黑眼睛型品种,由加州大学戴维斯分校开发[36].IT93K503-1是由尼日利亚伊巴丹国际热带农业研究所(IITA)开发的耐旱育种品系。我们在水分充足和干旱胁迫条件下培养了这两种基因型,并使用营养阶段的叶子构建了四个小的RNA文库。通过从这些文库中读取小RNA,我们鉴定出157个候选miRNAs。文库间mirna表达谱的比较表明,部分mirna在不同基因型中表达水平不同,这可能是导致其抗旱性水平不同的关键因素。
结果
豇豆中mirna的鉴定
为了研究miRNAs在耐旱性中的作用,我们在温室中种植豇豆(CB46和IT93K503-1),在充足的水分和干旱胁迫条件下。对30日龄植株进行干旱胁迫处理。经过10至15天的中度干旱胁迫(ψw= -1.5 MPa),两种基因型的耐旱性存在明显差异。在IT93K503-1植株继续较好生长的同时,CB46植株表现出严重的干旱胁迫症状,如叶片褪绿(图)1).
我们构建了4个小RNA文库(2基因型× 2种生长条件),从每个文库中平均获得690万条(范围:6.5 - 730万条)干净的小RNA(深度测序数据已存入NCBI/GEO数据库,登录号为GSE26402)。每个图书馆的平均独立阅读数是430万(范围:390 - 460万)。使用材料和方法部分中描述的程序和标准,我们将独特的小RNA reads映射到豇豆EST组装、BAC末端序列和甲基化过滤序列、dbGSS中的GSS序列和豇豆基因组组装草案,并分别预测了14、78、6和125个miRNA前体。然后将这四组假定的miRNA前体相互比较以去除冗余,我们获得了157个候选miRNA基因(详细信息请参阅附加文件)1).根据成熟miRNA序列的相似性,将这些miRNA基因聚为89个家族。而miRBase中有27个家族(93个miRNAs)与其他植物的miRNAs相匹配(release 16) [37], 62个家族(64个miRNAs)似乎是豇豆特异性的。使用豇豆EST组装,我们还鉴定了112个(71%)miRNAs的假定靶蛋白编码基因。
miRNAs的基因型特异性表达
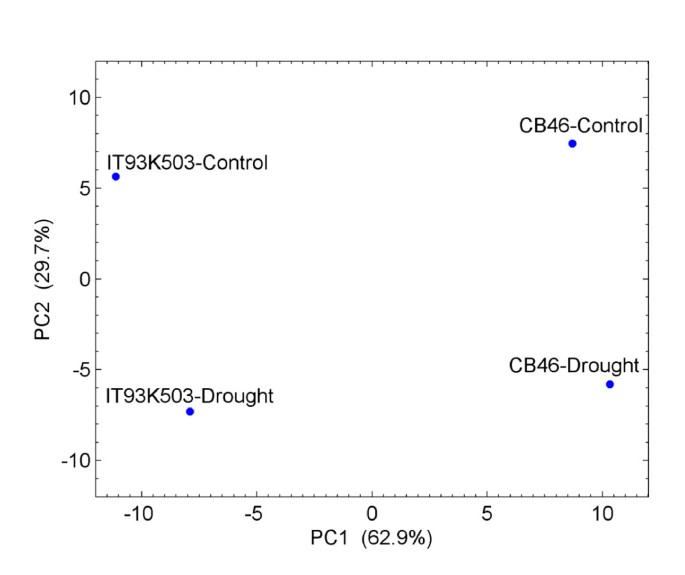
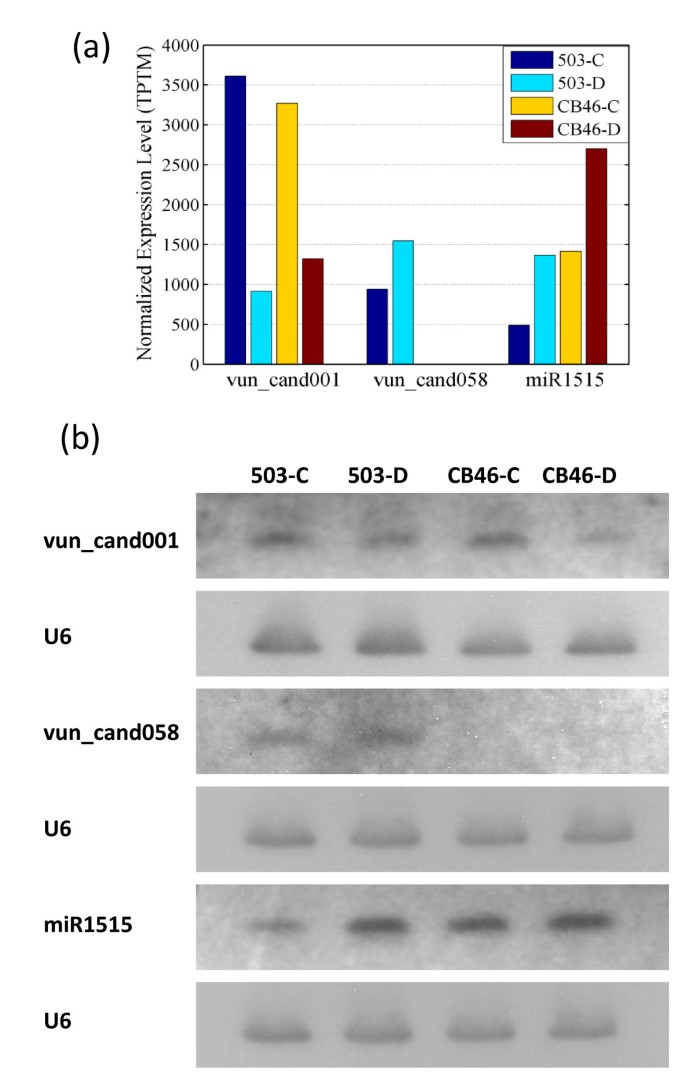
由于小RNA文库的测序深度很高,成熟mirna的数量可以用来评估它们在不同基因型和生长条件下的相对表达水平。我们首先将主成分分析(PCA)应用于四个文库中包含至少50个TPTM表达的91个独特成熟mirna的log2归一化计数(TPTM,每1000万个转录本)。如图所示2,前两个组成部分占数据集变化的93%以上,其中第一个组成部分占63%。第一个成分(PC1)将一个基因型的两个样本从另一个基因型的两个样本中分离出来,表明基因型是决定miRNA表达水平的主要因素。事实上,9个miRNAs占PC1变异的75%,它们表现出明确的基因型特异性表达(表2)1,关于预测发夹结构和小RNA读取前体的映射,请参阅附加文件2而且3.).两种miRNAs (vun_cand014和vun_cand054)主要在CB46中表达,其他7种miRNAs仅或主要在IT93K503-1植物中表达。northern blot检测证实了IT93K503-1特异性miRNA vun_cand058的表达模式3 b).
由于将小RNA测序结果映射到豇豆序列中进行miRNA预测需要完全匹配,因此成熟miRNAs中的基因型间单核苷酸多态性(SNPs)可能导致miRNAs的基因型特异性表达。为了解决这种可能性,我们将从每个文库中读取的干净小RNA重新映射到表中9个miRNAs的前体1,允许最多一次不匹配。这些成熟mirna的归一化计数基本没有变化(数据未显示)。因此,这些mirna的基因型特异性表达是真实的,而不是reads映射过程的产物。
Drought-associated microrna
为了鉴定干旱相关的mirna,我们使用Audic和Claverie开发的统计方法,检测了干旱胁迫下各基因型和相应对照样品中mirna的差异表达。38].我们使用以下标准来识别干旱相关的miRNAs:(1)在两个比较中至少有一个调整后的p值小于0.01;(2)四个库中有一个库的归一化计数(TPTM)至少为100;(3)在2个基因型中,干旱库与对照库归一化计数的log2比值大于1或小于-1。对于差异表达分析,我们只考虑独特的成熟miRNA,因为它们是miRNA的活性形式,在某些情况下,相同的成熟miRNA可以由两个或多个同源miRNA基因产生。我们发现了44种与干旱相关的独特成熟miRNAs,它们属于28个家族(附加文件)4).所有44个miRNA在两种基因型中的变化方向均具有统计学意义,表明IT93K503-1和CB46的miRNA基因表达对干旱胁迫的总体响应相似。44个miRNAs中有30个在干旱胁迫条件下上调,14个在一个或两个基因型中下调。
在44个干旱相关的miRNAs中,22个miRNAs(17个家族)在干旱条件下的表达与对照组相比至少发生了两倍的变化(附加文件)4).在之前的研究中发现其中一些miRNA家族与干旱胁迫有关,包括miR156和miR166 [39], miR159 [40], miR167 [24], miR169 [22], miR171 [25,41], miR395 [40], miR396 [24,39],以及miR482 [29].大多数预测目标编码转录因子(附加文件4).其他miRNA家族miR162、miR164、miR319、miR403、miR828和4个豇豆特异性miRNA (vun_cand001、vun_cand010、vun_cand041和vun_cand057)首次被发现与干旱胁迫相关(vun_cand001的北方杂交证实如图所示3 b).
我们还发现,仅在IT93K503-1中,有12个mirna显示出至少2倍的变化2), CB46中仅有10个(表3.).尽管统计测试表明,其中一些mirna(如miR1515,如图所示,通过northern blot验证3 b)在干旱胁迫下均上调或下调,且无2倍变化,11个mirna仅在一种基因型中被明显调控。而miR160a、miR160b、miR171e、vun_cand015、vun_cand033和vun_cand048仅在IT93K503-1植株中被干旱胁迫显著调控,miR171b、miR171d、miR2111b、miR390b和miR393仅在CB46植株中被调控。
讨论
通过miRNAs与其目标mrna之间的序列特异性相互作用调节基因表达,为植物对环境刺激的响应提供了一种精确的、可遗传的机制[18].由于水资源的限制,干旱是限制许多作物地理分布和产量的主要压力。因此,人们对干旱耐受性的遗传元素和机制进行了广泛的研究,包括干旱相关miRNAs的发现。豇豆是半干旱、干旱地区重要的抗旱作物,为研究其抗旱性提供了良好的体系。在这里,我们对两种豇豆基因型的小RNA文库进行了深度测序,并鉴定出157个miRNAs。通过比较干旱胁迫样品与对照样品中mirna的表达水平,我们还发现30个mirna在干旱条件下表达上调,14个mirna表达下调。这份与干旱相关的miRNA列表包括已知与其他植物物种的干旱相关的miRNA家族,这表明它们参与了保守的干旱响应途径。一些miRNA家族,包括一些豇豆特异性miRNA,首次被发现与干旱有关,这表明它们可能参与了谱系或物种特异性的应激反应途径和功能。
我们预测了44个干旱相关miRNAs中的32个的靶基因。预测的目标mrna编码多种功能的蛋白质,其中大多数是转录因子(附加文件)4).对于大多数保守的mirna,预期它们的靶标也是保守的。例如,我们的研究结果表明,miR156在豇豆干旱胁迫下表达上调。MiR156在拟南芥、玉米、水稻和小麦中响应非生物胁迫并靶向SPB转录因子[24,39,41- - - - - -43].该miRNA也参与了营养阶段变化的发育调控[42],表明发育的重新编程是植物应对干旱胁迫的关键一步。另一个miRNA miR169在两种豇豆基因型中均下调。在拟南芥中,干旱胁迫导致miR169下调,其靶点核因子Y转录因子NFYA5被诱导[21].MiR169很可能在豇豆中以类似的方式通过诱导NFYA5同源物的表达来增强耐旱性。
本研究所研究的豇豆基因型具有不同的耐旱能力。由于这两种基因型在基因组成上高度相似,它们的表型变异如耐旱性很可能是由调控过程的变化引起的,而不是蛋白质的变化[44].由于地理来源不同,这两种基因型都能适应其自然栖息地的特定环境条件。本构差异可能与植物代谢、能量资源利用、生物量动员、自由基系统结构、叶蜡沉积、气孔膜稳定性或密度等特征有关。我们发现9个miRNAs主要或仅在一种基因型中表达,与治疗无关。另一方面,有11个miRNAs在干旱胁迫下在一个基因型中表达差异,而在另一个基因型中没有。miRNA表达的改变有望引起两种基因型之间靶基因表达的改变。
在具有基因型特异性调控的miRNAs中,miR160a和miR160b在耐旱品种中响应干旱时上调,而在敏感品种中则没有2).它们的假定目标是生长素反应因子(ARFs)家族的成员。ARFs是生长素介导的生理和形态机制调节的关键元素,可能有助于胁迫适应[45].此外,miR160对ARF10的负调控被证明在种子萌发过程中至关重要拟南芥通过生长素和aba依赖通路之间的相互作用[46].另一方面,miR2111家族的两个成员在干旱敏感品种中表达上调,而在耐旱品种中没有3.).他们的假定目标是含有Kelch重复的F-box蛋白,该蛋白属于一个大家族,已知其成员参与了对生物和非生物应激的响应[47].此外,含有Kelch重复序列的F-box蛋白已被发现在豇豆的近亲鹰嘴豆中对干旱有反应[48].这表明基因型对miRNAs的特异性调控可能是部分豇豆基因型比其他基因型具有更强的耐旱性的原因。
在本研究中发现的新的候选miRNA中,有10个受干旱胁迫调控,其中5个被预测为靶基因。例如,vun_cand030因干旱而下调,并可能以锌指蛋白为目标。锌指蛋白已知参与发育和应激反应的多种功能[49].此外,vun_cand015在耐旱品种中被上调,可能是针对碱性螺旋-环-螺旋(bHLH)转录因子。这些蛋白质在应对非生物应激方面发挥作用,如缺铁[50]、冰冻和盐胁迫[51].这表明这些新的miRNAs可能确实是豇豆干旱反应的一个组成部分。
对于许多mirna,预测的靶点不止一个。一个miRNA具有多个靶标的可能性是常见的。为了确认这些预测的靶点,我们需要通过实验方法,如RACE和降解体分析,对miRNA识别位点mRNA靶点的裂解进行详细分析[52- - - - - -54].一旦我们验证了干旱相关miRNAs的靶点,我们就能更好地将miRNAs及其靶点的表达变化与豇豆的耐旱性差异联系起来。
由于我们没有完整的豇豆基因组序列,一些miRNA基因没有被识别出来,即使它们在我们的小RNA文库中有显著的表达。为了找出遗漏了多少个miRNA家族,我们绘制了独特的小RNA读取图,在miRBase中种植miRNA前体,允许最多2个错配。虽然我们没有遗漏大量的mirna,但我们确实发现miR2118、miR2911和miR529在我们的文库中有显著的表达(附加文件)5).后两者也是由干旱胁迫引起的。MiR529被鉴定为水稻干旱相关miRNA [25].然而,与豇豆中发现的模式相反,水稻在干旱胁迫下表达下调。目前还不清楚这是由不同的采样时间或组织引起的,还是由物种特有的应激反应机制引起的。
像蛋白质编码基因一样,许多miRNA家族拥有多个miRNA基因,来自同一家族的miRNA基因可能具有相同或相似但不同的成熟miRNA序列。在进化过程中,同源miRNA基因可能在功能上发生分歧。在我们在豇豆中鉴定的mirna中,来自miR166和miR167家族的成员显示了功能多样化的明确证据。当一个成员miRNA基因(miR166a, miR167b)被干旱胁迫诱导时,来自同一家族的另一个miRNA (miR166b, miR167a)显著下调(附加文件)4).
结论
利用深度测序技术,我们在豇豆中鉴定出157个mirna,其中44个与干旱相关。通过比较不同基因型和生长条件下成熟miRNA的数量,我们发现9个miRNA几乎只在一种基因型中表达,11个miRNA在一种基因型中受干旱胁迫调控,而在另一种基因型中不受干旱胁迫调控。我们的研究表明,小rna深度测序是一种低成本的miRNA发现和表达分析方法。与同源搜索方法相比,深度测序允许物种特异性mirna的检测和数字表达分析。我们的研究结果表明,即使在同一物种的两种基因型之间,一些miRNAs的表达模式也可能非常不同。进一步鉴定干旱相关miRNAs的靶点将有助于了解豇豆对干旱的反应和耐受性的细节。
方法
植物材料
CB46和IT93K503-1植物于2009年春季在加州大学河滨校区的温室中种植。白天温度为35°C,夜间温度为18°C,没有人为控制白昼长度。四颗种子在2加仑的罐子里发芽,罐子里装满了加州大学河滨分校(UC Riverside)的蒸汽消毒土壤混合物UCMIX-3,播种两周后,每罐稀释到两株。在完全随机区组设计中,每个处理设置三个重复锅。在植株生长30 d时,即营养后期,在胁迫盆上采用不浇水的亏缺灌溉处理,对照盆按土壤容量日水量灌溉。使用压力室监测第三叶水势(Cornallis, PMS仪器,美国)[55]作为压力水平的指标。对三个重复的新鲜叶片(从顶端开始的第二个叶片)进行取样,并在液氮中冷冻,这些叶片来自对照植物(水分充足,ψw= -0.5 MPa)和中等应力植物(ψw= -1.5 MPa)用于RNA提取。
小RNA文库构建及测序
按照制造商说明书,用TRIzol试剂(Invitrogen)提取总RNA。采用Sunkar和Zhu所使用的方法从豇豆叶中构建小RNA文库。20.]稍加修改[56].简单地说,对于每个处理/基因型组,从三个重复中汇集等量的总RNA,以产生约700 μg的RNA。聚合的总RNA在15%变性聚丙烯酰胺凝胶中溶解,提取20-30 nt小RNA片段并洗脱。使用T4 RNA连接酶将一个预腺苷化适配器(连接子1,IDT)连接到小RNA的3'端。然后将结扎产物进行凝胶纯化,随后将其连接到5'端RNA接头上。产物经结扎纯化后作为模板进行RT-PCR。经过合成和纯化,PCR产物用Illumina Genome Analyzer进行定量和测序。
microrna的识别
只有通过Illumina质量控制并包含清晰的适配器序列的小RNA reads被认为是用于进一步处理的良好reads。剪去适配器序列后,将18nt及以上的干净小RNA序列组合成唯一的序列。删除与已知植物重复序列、rrna、tRNAs、snrna和snoRNAs匹配的Reads。利用SOAP2将独特的小RNA reads映射到四个基因组序列资源[57]:豇豆EST组件可在HarvEST:豇豆[58) (http://harvest.ucr.edu,版本1.17,18,745个序列,但我们排除了那些似乎是蛋白质编码基因),260,642豇豆基因空间随机散弹枪序列的组合[59]和30,527个BAC末端序列(从m . c . c。罗,加州大学戴维斯分校,http://phymap.ucdavis.edu:8080/cowpea), 54,123条豇豆基因组调查序列(GSS)来自GenBank的dbGSShttp://www.ncbi.nlm.nih.gov/dbGSS/(296,868 contigs,总长度约186 MB)http://www.harvest-blast.org).完全匹配是必须的。
我们使用更新的植物miRNAs注释标准[60]并建立了一个内部的miRNA预测管道。具有至少10个副本冗余的唯一读取被用作锚定序列。一端锚定在距离地图位置10 bp处,每个锚定序列覆盖100 - 300 bp的DNA片段采样,步长为20 bp。利用UNAFold预测各节段的二级结构[61].然后,我们检查了这些结构,只有符合以下标准的结构才被认为是真正的miRNA候选者:(1)自由能低于或等于-35 kcal/mol;(2)假定miRNA与miRNA*的错配数为4或更少;(3)茎区不对称凸起数不大于1个,且每个不对称凸起大小不大于2个;(4)链偏倚-小RNA读取映射到发夹DNA片段的正链占所有映射读取的至少80%;(5)精确切割-映射到miRNA和miRNA*区域的reads(定义为5'和3'端上的miRNA或miRNA*加上2nt)占到前体映射的所有reads的至少75%。如果从同一区域预测到两个或两个以上的候选发夹,我们将这些发夹进行比较,并选择一个假定成熟miRNA表达最高、自由能最低或长度最短的发夹。
为了将mirna分类为家族,所有预测成熟的mirna都使用FASTA包(3.5版)中的研究35程序与自身进行比较[62].使用单链接算法,成熟的mirna最多有两个错配被包含在相同的簇中。然后将成熟的miRNAs与miRBase中的成熟miRNAs进行比较(Release 16) [37使用search35。如果豇豆miRNA集群中的一个成员在miRBase中有一个匹配(允许最多两个不匹配),则已知miRNA的家族号被分配给该集群,否则该集群被注释为一个新的家族。
miRNA靶点预测
以成熟miRNA序列作为查询,利用miRanda搜索豇豆EST组合的潜在靶点[63].从miRanda输出中提取mirna和潜在靶标之间的比对,并使用位置依赖的错配惩罚系统进行评分[64- - - - - -66].简单地说,miRNA-靶标双工被分为两个区域:一个核心区域,包括miRNA 5'端起2-13个位置,一个一般区域,包含其他位置。在一般区域,不匹配或单核苷酸凸起或缺口的惩罚得分为1,G:U对的惩罚得分为0.5。核心区域的分数翻了一番。如果miRNA与靶标的排列符合以下两个条件,则认为匹配为阳性:(1)惩罚分为4分或以下;(2)凸起和缺口总数小于2个。
主成分分析
每个成熟miRNA的计数首先归一化为转录本千万分之一(TPTM),根据四个文库中每个文库中干净的小RNA读取总数。选择组合表达至少50个TPTM的mirna进行主成分分析(PCA)。我们使用miRNA归一化计数的log2值构建表达式矩阵,并使用MATLAB (MathWorks Inc., Natick, MA)中的princomp函数进行PCA。
mirna差异表达的统计学检验
由于小RNA的深度测序提供了原始小RNA池中成熟mirna的随机采样,因此mirna的计数可以用泊松分布来建模。我们采用了一种既定的方法[38,67],计算干旱胁迫样本与对照样本之间miRNAs差异表达的p值。第一步是用公式计算条件概率:
在哪里N1是控制库中干净读取的总数,N2是受干旱影响的图书馆中干净的阅读总数,x为对照文库中成熟miRNA的数量,和y为干旱胁迫文库中相同成熟miRNA的数量。然后计算差分表达式的双尾p值为p= 2问,其中q为累计概率:
由于x↔y对称的p(y | x),如果问大于0.5时,p值可计算为p= 2 * (1 -问).采用Bonferroni方法调整p值进行多次比较。
Northern blot分析
~40 μg总RNA在15%变性聚丙烯酰胺凝胶中分解,转移到中性尼龙膜(Hybond NX)上。RNA通过化学交联转移并固定在膜上[68],然后杂交到与成熟miRNA序列互补的探针,在38°C下过夜。杂交后,用洗涤液(2X SSC, 0.1% SDS)在38°C下洗涤两次,每次5分钟,并将其暴露在x射线胶片上以显示信号。在三个重复样品中验证了Northern blot检测结果。
参考文献
- 1.
Tuberosa R, Salvi S:基于基因组学的提高作物耐旱性的方法。植物科学进展,2006,11:405-412。10.1016 / j.tplants.2006.06.003。
- 2.
Ashraf M:诱导植物的耐旱性:最新进展。生物技术,2010,28:169-183。10.1016 / j.biotechadv.2009.11.005。
- 3.
Singh BB, Ajeigbe HA, Tarawali SA, Fernandez-Rivera S, Abubakar M:提高豇豆作为食物和饲料的生产和利用。农学通报,2003,34(3):379 - 379。10.1016 / s0378 - 4290(03) 00148 - 5。
- 4.
Ehlers JD, Hall AE:豇豆(豇豆属unguiculataL Walp)。农学通报,1997,33(4):457 - 457。10.1016 / s0378 - 4290(97) 00031 - 2。
- 5.
Sanginga N, Lyasse O, Singh BB:西非衍生热带草原低磷土壤中豇豆品种的磷利用效率和氮平衡。土壤学报,2000,29(3):357 - 357。10.1023 /: 1004785720047。
- 6.
Iuchi S, yamaguchi ishinozaki K, Urao T, Terao T, Shinozaki K:高耐旱豇豆新型干旱诱导基因:cdna克隆及相关基因表达分析。植物生理学报,1996,37:1073-1082。
- 7.
Iuchi S, yamaguchi ishinozaki K, Urao T, Shinozaki K:高耐旱豇豆新型干旱诱导基因的两个cdna的鉴定。植物学报,1996,29(4):344 - 344。10.1007 / BF02344557。
- 8.
Iuchi S, Kobayashi M, Yamaguchi-Shinozaki K, Shinozaki K:耐干旱豇豆水分胁迫下脱落酸生物合成中9-顺式环氧类胡萝卜素双加氧酶的胁迫诱导基因。中国农业科学,2000,29(4):457 - 457。10.1104 / pp.123.2.553。
- 9.
Diop NN, Kidric M, guardiin A, Gareil M, d'Arcy-Lameta A, Thi ATP, zuly - fodil Y:干旱胁迫诱导豇豆多胱氨酸抑素(豇豆属unguiculata(l)Walp)。树叶。中国农业科学,2004,27(4):344 - 344。10.1016 / j.febslet.2004.10.014。
- 10.
El-Maarouf H, d'Arcy-Lameta A, Gareil M, zuly - fodil Y, phami - thi AT:豇豆叶片两个pi - plc编码cdna的克隆及干旱条件下的表达。植物化学学报,2001,29(3):344 - 344。10.1016 / s0981 - 9428(00) 01226 - 2。
- 11.
D'Arcy-Lameta A, Ferrari-Iliou R, Contour-Ansel D, phami - thi AT, Zuily-Fodil Y:豇豆叶片水分亏缺反应的4个抗坏血酸过氧化物酶cdna的分离与鉴定。王志强,2006,30(1):1 - 4。
- 12.
Contour-Ansel D, Torres-Franklin ML, De Carvalho MHC, D'Arcy-Lameta A:豇豆叶片谷胱甘肽还原酶的克隆、表达及在渐次干旱胁迫、干燥和脱落酸处理下的酶活性王志强,2006,30(1):1 - 7。10.1093 / aob / mcl217。
- 13.
穆切罗·W, Ehlers JD, Close TJ, Roberts PA:干旱胁迫诱导豇豆早衰老和成熟的QTL定位[豇豆属unguiculata(l)Walp。]。应用物理学报,2009,29(3):349 - 349。10.1007 / s00122 - 008 - 0944 - 7。
- 14.
巴特尔DP: MicroRNAs:基因组学,生物发生,机制和功能。电子学报,2004,34(1):1- 7。10.1016 / s0092 - 8674(04) 00045 - 5。
- 15.
Brodersen P, Sakvarelidze-Achard L, Bruun-Rasmussen M, Dunoyer P, Yamamoto YY, Sieburth L, Voinnet O:植物mirna和sirna的广泛翻译抑制。科学通报,2008,32(3):344 - 344。10.1126 / science.1159151。
- 16.
Jover-Gil S, Candela H, Ponce MR:植物microRNAs及其发育。中国生物医学工程学报,2005,29(3):344 - 344。10.1387 / ijdb.052015sj。
- 17.
小rna在非生物胁迫中的作用。中国农业科学,2007,27(4):457 - 457。10.1016 / j.febslet.2007.04.007。
- 18.
孙佳,朱娟,朱建军:小rna在植物非生物胁迫响应和营养剥夺中的作用。植物科学进展,2007,12:301-309。10.1016 / j.tplants.2007.05.001。
- 19.
Navarro L, Dunoyer P, Jay F, Arnold B, Dharmasiri N, Estelle M, Voinnet O, Jones JDG:植物miRNA通过抑制生长素信号通路促进抗菌抗性。科学通报,2006,31(4):439 -439。10.1126 / science.1126088。
- 20.
孙卡罗,朱建军:拟南芥中新型的胁迫调控microrna和其他小rna。中国生物医学工程学报,2004,16:2001-2019。10.1105 / tpc.104.022830。
- 21.
李文霞,Oono Y,朱建辉,何晓军,吴建明,Iida K,陆晓阳,崔雪平,金海林,朱建科:拟南芥NFYA5转录因子通过转录和转录后调控促进抗旱性。植物科学学报,2008,30(4):344 - 344。10.1105 / tpc.108.059444。
- 22.
赵婷婷,梁仁强,葛丽龙,李伟,肖海生,林海霞,阮kc,金玉霞:水稻干旱诱导microrna的鉴定。生物化学学报,2007,34(3):359 - 359。10.1016 / j.bbrc.2007.01.022。
- 23.
孙嘉荣,周晓峰,郑勇,张文祥,朱建军:利用高通量测序鉴定水稻新mirna和候选mirna。中国生物医学工程学报,2008,8:25-10.1186/1471-2229-8-25。
- 24.
刘海辉,田霞,李玉娟,吴昌昌,郑成昌:基于微阵列的应力调控microRNAs分析拟南芥.RNA。2008, 14: 836-843。10.1261 / rna.895308。
- 25.
周玲,刘颖,刘震,孔东,段明,罗丽:干旱响应microrna的全基因组鉴定与分析栽培稻.应用物理学报,2010,29(4):457 - 457。10.1093 / jxb / erq237。
- 26.
Szittya G, Moxon S, Santos DM, Jing R, Fevereiro MP, Moulton V, Dalmay T:高通量测序Medicago truncatula短rna鉴定出8个新的miRNA家族。中国生物医学工程学报,2008,9:593-10.1186/1471-2164-9-593。
- 27.
lelandaiss - briere C, Naya L, Sallet E, Calenge F, Frugier F, Hartmann C, Gouzy J, Crespi M:全基因组Medicago truncatula小RNA分析揭示了新的microRNAs和异构体在根和结节中有不同的调控。植物科学学报,2009,29(4):379 - 379。10.1105 / tpc.109.068130。
- 28.
苏巴曼尼亚·S,付毅,孙卡尔·R,朱建科,于奥:大豆根系中新型和结节调控的microrna。中国生物医学工程学报,2008,29(4):344 - 344。
- 29.
Arenas-Huertero C, Perez B, Rabanal F, Blanco-Melo D, De la Rosa C, Estrada-Navarrete G, Sanchez F, Covarrubias AA, Reyes JL:豆科植物中保守和新颖的mirna菜豆是对压力的反应。中国生物医学工程学报,2009,29(4):389 - 397。10.1007 / s11103 - 009 - 9480 - 3。
- 30.
陆永智,杨晓阳:新型microRNAs及其靶分子的计算鉴定豇豆属unguiculata.compp Funct Genomics 2010, pii: 128297。
- 31.
Paul S, Kundu A, Pal A:保守microRNAs的鉴定和验证及其在植物根中的差异表达豇豆属unguiculata在盐胁迫下生长。植物科学学报,2011,29(3):344 - 344。10.1007 / s11240 - 010 - 9857 - 7。
- 32.
Hall AE, Cisse N, Thiaw S, Elawad HOA, Ehlers JD, Ismail AM, Fery RL, Roberts PA, Kitch LW, Murdock LL, Boukar O, Phillips RD, McWatters KH:大豆/豇豆CRSP对豇豆品种和种质的开发。农学通报,2003,29(4):344 - 344。10.1016 / s0378 - 4290(03) 00033 - 9。
- 33.
A馆:豇豆抗旱抗热育种。王志强,2004,21:447-454。10.1016 / j.eja.2004.07.005。
- 34.
马晓明,王晓明,王晓明,等:不同基因型豇豆苗期干旱诱导表型及干旱响应基因分析。作物科学,2008,48:541-552。10.2135 / cropsci2007.07.0397。
- 35.
郑洁,付洁,勾明,怀娟,刘勇,简明,黄强,郭霞,董志,王宏,王刚:干旱胁迫下两个玉米自交系的全基因组转录组分析。中国生物医学工程学报,2010,27(3):344 - 344。10.1007 / s11103 - 009 - 9579 - 6。
- 36.
Helms D, Panella L, Buddenhagen IW, Tucker CL, Gepts PL:加州黑眼46豇豆注册。作物科学,1991,31:1703-1703。
- 37.
griffith - jones S, Saini HK, van Dongen S, Enright AJ: miRBase: microRNA基因组学的工具。中国生物医学工程学报,2008,36:D154-D158。10.1093 / nar / gkn221。
- 38.
Audic S, Claverie JM:数字基因表达谱的意义。基因组学报,1997,7:986-995。
- 39.
Kantar M, Lucas SJ, Budak H: miRNA的表达模式小麦属植物dicoccoides对冲击干旱胁迫的反应。植物学报,2011,29(4):344 - 344。10.1007 / s00425 - 010 - 1309 - 4。
- 40.
孙国强,张斌,孙国强:盐胁迫和干旱胁迫诱导烟草microRNA基因异常表达。Mol生物技术,2011年2月26日,Epub。
- 41.
沈娟,谢凯,熊玲:水稻microrna在非生物胁迫响应中的重要作用。中国生物医学工程学报,2010,27(3):344 - 344。10.1007 / s00438 - 010 - 0581 - 0。
- 42.
吴刚,李志刚,李志刚:植物茎部发育的时间调控拟南芥miR156和它的目标SPL3。发展,2006,133:3539-3547。10.1242 / dev.02521。
- 43.
丁东,张丽玲,王辉,刘志军,张志新,郑丽丽:玉米根系mirna对盐胁迫响应的差异表达。学报学报,2009,103:29-38。
- 44.
King MC, Wilson AC:人类和黑猩猩进化的两个层面。科学通报,1995,18(3):344 - 344。10.1126 / science.1090005。
- 45.
植物生长素反应因子的研究。植物学报,2007,10:453-460。10.1016 / j.pbi.2007.08.014。
- 46.
刘PP, Montgomery TA, Fahlgren N, Kasschau KD, Nonogaki H, Carrington JC: microRNA160抑制生长素反应因子10对种子萌发和萌发后阶段的影响。植物学报,2007,29(3):344 - 344。10.1111 / j.1365 - 313 x.2007.03218.x。
- 47.
闫玉生,陈晓霞,杨凯,孙志祥,付亚平,张玉明,方仁祥:水稻B-box蛋白基因过表达降低非生物胁迫耐受性,促进根系生长。植物化学,2011,4:190-197。10.1093 / mp / ssq066。
- 48.
贾勇,顾辉,王霞,陈强,史松,张娟,马玲,张宏,马宏:鹰嘴豆F-box家族基因carF-box1的克隆与分子特性分析(中投arietinuml .)。Mol Biol代表2011,Epub领先,2011年6月12日。
- 49.
杨洋O:植物抗旱和耐盐性:胁迫调节转录因子及其在细胞转录网络中的意义。植物学报,2011,30:1383-1391。10.1007 / s00299 - 011 - 1068 - 0。
- 50.
Long TA, Tsukagoshi H, Busch W, Lahner B, Salt DE, Benfey PN: bHLH转录因子POPEYE调控拟南芥根系缺铁反应中国生物医学工程学报,2010,29(3):344 - 344。10.1105 / tpc.110.074096。
- 51.
李峰,郭珊珊,赵艳,陈东,崇科,徐勇:转基因拟南芥中含同肽重复的bHLH蛋白基因OrbHLH001的过表达及其抗冻耐盐性。植物学报,2010,29:977-986。10.1007 / s00299 - 010 - 0883 - z。
- 52.
German MA, Pillay M, Jeong DH, Hetawal A, Luo SJ, Janardhanan P, Kannan V, Rymarquis LA, Nobuta K, German R, De Paoli E, Lu C, Schroth G, Meyers BC, Green PJ:通过并行分析microRNA-target RNA对的全局识别。生物技术学报,2008,26:941-946。10.1038 / nbt1417。
- 53.
Addo-Quaye C, Eshoo TW, Bartel DP, Axtell MJ:拟南芥内源性siRNA和miRNA靶点的测序动物学报,2008,18:758-762。10.1016 / j.cub.2008.04.042。
- 54.
吴玲,张qq,周海燕,倪瑞芳,吴晓霞,齐玉娟:水稻MicroRNA效应复合物及靶标。植物学报,2009,21:3421-3435。10.1105 / tpc.109.070938。
- 55.
Scholander PF, Bradstreet ED, Hemmingsen EA, Hammel HT:维管植物中的液压力-负静水压力可以在植物中测量。科学通报,1996,18(3):339-346。10.1126 / science.148.3668.339。
- 56.
Ghildiyal M, Seitz H, Horwich MD, Li CJ, Du TT, Lee S, Xu J, Kittler ELW, Zapp ML, wzp, Zamore PD:来自转座子和mrna的内源性sirna果蝇体细胞。科学通报,2008,32(3):357 - 357。10.1126 / science.1157396。
- 57.
李锐,于超,李勇,林文伟,姚明生,王娟:SOAP2:一种改进的短读对齐超快工具。生物信息学,2009,25:1966-1967。10.1093 /生物信息学/ btp336。
- 58.
Close TJ,沃纳梅克S,卢斯ML,里昂M:收获。方法Mol生物学,2007,406:161-177。
- 59.
Timko MP, Rushton PJ, Laudeman TW, Bokowiec MT, Chipumuro E,张F, Town CD, Chen X:豇豆基因丰富空间的测序与分析。中国生物医学工程学报,2008,29(3):344 - 344。
- 60.
Meyers BC, Axtell MJ, Bartel B, Bartel DP, Baulcombe D, Bowman JL, Cao X, Carrington JC, Chen X, Green PJ, griffths - jones S, Jacobsen SE, Mallory AC, Martienssen RA, Poethig RS, Qi Y, Vaucheret H, Voinnet O, Watanabe Y, Weigel D, Zhu JK:植物microrna注释的标准。植物学报,2008,20:3186-3190。10.1105 / tpc.108.064311。
- 61.
Markham NR, Zuker M: UNAFold:核酸折叠和杂交软件。在生物信息学,卷二结构,功能和应用。编辑:Keith JM托托瓦,新泽西州:Humana出版社;2008:3-31。
- 62.
Pearson WR:使用FASTA3程序包进行灵活的序列相似性搜索。方法Mol biology, 2000, 132: 185-219。
- 63.
Enright AJ, John B, Gaul U, Tuschl T, Sander C, Marks DS: MicroRNA靶在果蝇.中国生物工程学报,2004,5:1。
- 64.
谢志新,谢志新,李志刚,李志刚:植物反式作用siRNA生物发生过程中microrna定向的相位调控。光子学报,2005,29(3):344 - 344。10.1016 / j.cell.2005.04.004。
- 65.
Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ: miRBase: microRNA序列,靶标和基因命名法。中国生物医学工程学报,2006,34:D140-D144。10.1093 / nar / gkj112。
- 66.
Fahlgren N, Howell MD, Kasschau KD, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA, Law TF, Grant SR, Dangl JL, Carrington JC:拟南芥microrna的高通量测序:MIRNA基因频繁出生和死亡的证据。公共科学学报,2007,2:e219-10.1371/journal.pone.0000219。
- 67.
Gonzalez-Ballester D, Casero D, Cokus S, Pellegrini M, Merchant SS, Grossman AR:硫剥夺的RNA-Seq分析衣藻《细胞》揭示了对细胞生存至关重要的驯化方面。中国生物医学工程学报,2010,30(4):344 - 344。10.1105 / tpc.109.071167。
- 68.
Pall GS, Hamilton AJ:改进的北方印迹法增强小RNA检测。自然科学进展。2008,3:1077-1084。10.1038 / nprot.2008.67。
鸣谢及资助
这项工作得到了加州大学河滨分校初始补充基金和美国农业部哈奇基金(CA-R-BPS-7754H)对RL的支持,UCR农业实验站基金对TJC的支持,NIH资助R01GM070795和R01GM059138给J-KZ, UC- mexus和conacyt -墨西哥奖学金给BEB-F。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
BEB-F, J-KZ, TJC和RL构思了这项研究。BEB-F, ZW, NND, JDE和PAR进行了实验。BEB-F、LG、J-KZ和RL分析了数据,LG贡献了新的分析工具,RL、BEB-F、TJC和J-KZ撰写了论文。所有作者都阅读并批准了最终的手稿。
Blanca E Barrera-Figueroa, Lei Gao对这项工作做出了同样的贡献。
电子辅助材料
12870 _2011_911_moesm1_esm.xls
附加文件1:在豇豆中鉴定出的MiRNAs。预测的豇豆miRNAs及其靶标的详细信息。(xl204kb)
12870 _2011_911_moesm2_esm.pdf
附加文件2:预测9种基因型特异性miRNAs的发夹结构。9种基因型特异性miRNAs的预测结构,其中成熟的miRNAs用绿色标记。(pdf 151kb)
12870 _2011_911_moesm3_esm.pdf
附加文件3:小RNA的映射从四个文库读取到九个基因型特异性miRNAs的前体。每个图显示了前驱体序列,预测的发夹结构,以及每个独特的读取如何映射到前驱体。(pdf 11mb)
12870 _2011_911_moesm4_esm.xls
附加文件4:豇豆中的干旱相关miRNAs。干旱相关miRNAs及其靶标的详细信息。(xls 84 kb)
12870 _2011_911_moesm5_esm.xls
附加文件5:在豇豆中表达的其他保守miRNAs。在两种生长条件下,两种豇豆基因型中三个保守的mirna及其表达量。(xl26kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
巴雷拉-菲格罗亚,b.e.,高林,迪奥普,N.N.et al。两种豇豆基因型干旱相关microrna的鉴定与比较分析。BMC植物生物学11,127(2011)。https://doi.org/10.1186/1471-2229-11-127
收到了:
接受:
发表:
关键字
- 干旱胁迫
- 耐旱
- microrna的家庭
- microrna基因
- 成熟的microrna