- 研究文章
- 开放获取
- 发表:
新创雄性花和雌雄同体花的测序和转录组比较分析为雄花雌雄同体花的形成调控提供了新的思路Taihangia rupestris
BMC植物生物学体积17、物品编号:54(2017)
摘要
背景
Taihangia rupestris雄花,雄花雌雄同体的植物,在同一个体内同时有雄花和雌雄同体的花。然而,雄花和雌雄同体花的建立和发育在雄花和雌雄同体花中Taihangia由于有限的遗传和序列信息,仍然知之甚少。探讨其调控的潜在分子机制Taihangia花的形成,我们用过新创RNA测序比较雄性花和雌雄同体花发育早期和后期的转录组谱。
结果
构建雄性花芽、雌雄同体花芽、雄花和雌雄同体花4个cDNA文库,并利用Illumina RNA-Seq方法进行测序。总共获得84,596,426个符合条件的Illumina reads,然后组装成59,064个unigenes,其中24,753个unigenes被注释到NCBI非冗余蛋白数据库中。此外,共有12,214个、7,153个和8,115个单基因分别归属于53个基因本体(GO)功能群、25个同源类群(COG)类别和126个京都基因与基因组百科全书(KEGG)途径。通过对样品间单基因丰度的两两比较,鉴定出1668个差异表达基因(deg),其中包括176个转录因子(TFs)。在雌雄同体花蕾发育早期,共表达了263个上调基因和436个下调基因;在雌雄同体花蕾发育后期,共表达了844个上调基因和314个下调基因。GO和KEGG富集分析表明,大量的deg与广泛的功能有关,包括细胞周期、表观遗传过程、花发育和不饱和脂肪酸途径的生物合成。最后,采用实时定量PCR对本研究中鉴定的deg进行验证。
结论
在本研究中,转录组数据的这种罕见的雄激素Taihangia都是首次被报道。比较转录组分析显示,雌雄同体花在发育早期和后期的基因表达谱存在显著差异。的转录组数据Taihangia有助于进一步认识雄花雌雄同株植物花形成和单性花形成的分子调控机制。
背景
开花植物的花结构具有显著的多样性,这在很大程度上促进了被子植物性系统的多样性。1]。雄花雌雄同体是一种罕见的性系统,在这种系统中,一个个体同时拥有雄蕊和完美的花朵。在植物界,已在近4000种植物中发现它,占开花植物的1.7% [2]。雌雄同体作为雌雄同体向雌雄同体的过渡阶段,在不同的植物类群中独立进化了多次[3.]。鉴于植物性系统在进化中的重要地位,单性花决定的发育和遗传机制近年来引起了人们的广泛关注[4- - - - - -6]。
Taihangia rupestris玉和李(蔷薇科),一种濒临灭绝的多年生草本植物,属于杉木科,仅在中国太行山南部的悬崖面上零星分布[j]。7]。不像这个部落的其他属,比如新和水杨梅属植物,Taihangia在同一个体中总是产生两性花和单性雄花(附加文件)1:图S1),形成雌雄同体的性系统[8]。Taihangia单性雄花在开始时是两性的,在随后的发育阶段通过阻止雌蕊发育而变成单性的。在花原基形成过程中,温度对单性花或两性花的形成起着至关重要的作用。在低温条件下,两性花的雌蕊容易发生败育,导致单性雄花的比例很高[9]。几个与花器官发育有关的MADS-box基因Taihangia,包括B-, C-,D-和e类基因,在先前的研究中已被发现[10- - - - - -12]。在这些已鉴定的MADS基因中,B类MADS-box基因TrPI在花瓣和雄蕊中表达,而E类基因TrSEP3强烈地表达在三个内旋中[10,11]。对于C类基因TrAG当第一次观察到雄蕊原基时,它最初在雄蕊和心皮形成的分生组织域表达。在心皮发育的后期,TrAG主要在胚珠、花柱发育和柱头中表达[12]。尽管取得了上述进展,但植物花器官发育和花形成调控的潜在分子机制Taihangia由于这种非模式植物物种的遗传和序列信息有限,人们对其了解甚少。
目前,下一代测序(NGS)技术为在没有参考基因组的情况下,在非模式植物的全基因组水平上高效、全面地分析基因表达提供了机会[13]。强大的NGS技术已被广泛应用于一些植物物种雄花和雌花的大规模基因表达谱比较转录组研究,为鉴定参与性别分化和花发育的功能基因提供了大量的基因组数据[14- - - - - -16]。然而,由于雌雄同株植物单性雄花的发生频率较低,在一定程度上对单性雄花的规格和发育的认识仍然有限。到目前为止,对雌雄同株植物进行的转录组研究很少[6,17,18]。
探讨雄花雌雄同株花形成调控的可能分子机制Taihangia,我们表演了新创利用Illumina平台对雄花和雌雄同体花发育早期和晚期的转录组测序。基于测序文库中单基因丰度的差异,我们分别鉴定了雌雄同体花在发育早期和后期的差异表达基因(DEGs)。此外,我们还进行了GO和KEGG富集分析,以阐明DEGs的主要功能,并确定参与花形成调控的候选基因,以进行进一步的功能分析。本研究将为今后的基因发现提供基因组资源,揭示雌雄同体植物单性花形成和性别分化的潜在分子机制Taihangia。
方法
植物材料和RNA提取
植株材料于2014年4月移栽于中国河南一豆水(35°28′N, 113°23′e)和竹玉峰(35°27′N, 113°22′e),在河南理工大学大棚种植。2015年早春,采集雄花和雌雄同体花发育早期和后期,包括雄性花芽(EM)、雌雄同体花芽(EH)、雄花(LM)和雌雄同体花(LH)(图2)。1)。如图所示。1将花的发育分为幼芽(A期,< 0.5 cm)、伸长花蕾(B期,0.5-1 cm, C期,>1 cm)、花前(D期)、花期(E期,花瓣部分张开的花)和完全张开的花(F期)6个阶段。在本研究中,早期发育的花样品是指包括幼芽和伸长花蕾(A-C期)的花蕾。发育后期的花样品包括花前花(D期)、花期花(E期)和花瓣完全开放的成熟花(F期)。每个发育阶段(A-F期)分别采集6个不同个体的雄花和雌雄同体花样品,立即在液氮中冷冻,保存在- 80°C中提取RNA。
根据制造商手册,使用RNeasy Mini Kit (Qiagen, Germany)从100 mg均质植物材料中分离总RNA。采用1.5%琼脂糖凝胶电泳初步评价RNA质量,然后用紫外可见分光光度计(UV-2550,日本)定量。根据A260/A280比值为1.9 ~ 2.1,A260/A230比值为2.0 ~ 2.5选取RNA样本进行进一步分析。从每个早期发育阶段(A-C阶段)的雄花芽中提取等量的RNA混合成一个EM样品,而从每个发育后期(A-C阶段)的雄花中提取等量的RNA组合成一个LM样品D- f)。同样,EH和LH样品也采用上述方法获得。共制备3个生物重复的12个RNA样本,进行cDNA合成和qPCR分析。然后,通过结合相应生物三副本中相同数量的总RNA,获得EM、EH、LM和LH的特异性RNA池。分别使用Nanodrop 2000仪器(Thermo Scientific)和Chip RNA 7500 Series II生物分析仪(Agilent)进一步分析混合RNA的质量和数量。采用260/280比值(1.9 ~ 2.1)、260/230比值(2.0 ~ 2.5)和RIN (RNA完整性数)大于8.0的RNA样品进行cDNA文库构建和Illumina测序。
文库建设及RNA-Seq
4个文库的构建和RNA-Seq由biomker生物技术公司(北京,中国)完成。从每个样品(EM, EH, LM和LH)中提取5微克总RNA构建测序文库。按照制造商的建议,使用TruSeq RNA样品准备试剂盒(Illumina, San Diego, CA)生成RNA测序文库。简单地说,使用poly- t oligo连接的磁珠(Illumina, San Diego, CA, USA)从总RNA中纯化含有poly (A)的mRNA分子。然后,将mRNA裂解成短片段,作为cDNA合成的模板。利用SuperScript II、缓冲液、dNTPs、RNaseH、DNA聚合酶I和随机六聚体引物(Illumina)合成双链cDNA。之后,将“A”尾添加到修复后的cDNA片段的3 '端,并将Illumina的配对端适配器连接到cDNA末端。用PCR扩增结扎反应产物。PCR产物经磁珠(Illumina)纯化,并溶解于EB溶液中生成测序文库。每个测序文库的数量和质量通过Agilent 2100生物分析仪进行检测。 Finally, the four libraries were sequenced separately on Illumina HiSeq™ 2500 platform.
序列组装和基因注释
测序后,首先通过内部perl脚本处理对端原始读取[19,20.]。在这一步中,通过去除适配器污染的reads、低质量序列(模棱两可的“N”碱基大于5%的reads)和大于10% Q < 20碱基的reads来获得干净的reads。同时计算洁净数据的Q20、Q30、gc含量。所有下游分析均基于高质量的干净数据。新创转录组的组装由Trinity软件完成(2014-07-17发布)[13],参数默认,没有引用顺序。简而言之,尺蠖程序在第一步就将reads组装到contigs中。通过Chrysalis程序将最小重叠的contigs聚集成连接的组件,然后通过Butterfly程序构建转录本[14]。最后,根据核苷酸序列的同一性对转录本进行进一步聚类,将聚类单元中最长的转录本视为单基因,剔除冗余序列。为了检测不同阶段雄性花和雌雄同体花的转录谱,每个文库的clean reads最初分别组装。为了在样本中获得统一的转录组参考,将来自四个文库的所有干净读取数据汇集在一起并进行分析新创组装以产生组装评估,基因注释和表达分析的单一基因。原始测序数据存储在NCBI Short Read Archive (SRA)数据库(http://www.ncbi.nlm.nih.gov/sra/),登记号为SRP081195。
确定…的假定功能Taihangia我们使用BLASTx搜索(e值≤10)进行功能注释−5)比对一系列蛋白质数据库:国家生物技术信息中心non-redundant (Nr)、SwissProt、Pfam、Clusters of Orthologous Groups (COG)、Gene Ontology (GO)和京都基因与基因组百科全书(KEGG)数据库。使用Blast2GO软件包比较并确定单基因GO注释[21]。最后,使用WEGO软件对所有标注的基因进行GO功能分类[22]。
转录因子预测
中表示的tfTaihangia使用blastX在植物转录因子数据库PlantTFDB 4.0中搜索所有组装的单基因,截止e值为1 e−5[23]。为了便于转录因子预测,进一步筛选鉴定出的tf,相似度≥0.6,覆盖率≥0.4。
差异表达基因(DEGs)分析
为了计算每个单基因的基因表达量,RSEM [24用]来量化定位到组装转录组上的reads数量,并从定位结果中获得每个基因的reads计数。我们使用FPKM [25(每千个碱基的片段数,每百万次测序)算法来标准化每个文库中的基因表达丰度。通过对4个文库的两两比较,利用DEGseq (1.18.0) R软件包根据不同文库中每个基因的读取计数鉴定出deg [26]。此外,错误发现率(FDR)控制方法[27的阈值P显著性检验中的-value。差异表达基因的显著性定义为错误发现率< 0.01,绝对值为log2-比值≥2。用GO和KEGG分析方法对deg进行分类。使用Cytoscape插件BiNGO对差异表达基因(DEGs)进行氧化石墨烯富集分析,并使用校正的超几何测试P-value < 0.05 [28]。利用超几何分布模型的KOBAS对deg进行KEGG通路富集分析[29]。铀浓缩活动p-值采用Benjamin和Hochberg方法进行调整。
实时荧光定量PCR (qpcr)对测序数据的验证
随机选择16个deg,采用qRT-PCR检测表达谱,确认转录组数据。使用PrimeScript RT reagent Kit with gDNA Eraser (Perfect Real Time) (Takara, Japan)根据说明书,从用于文库构建的相同RNA样品合成第一链cDNA。采用SsoFast EvaGreen Supermix RT-PCR试剂盒(Bio-Rad Laboratories)和MiniOpticon real-time Detection System (Bio-Rad)进行实时荧光定量PCR (qRT-PCR)。PCR混合物由10 μL EvaGreen Supermix, 2.0 μL 1:10稀释的cDNA,每个引物0.5 μL (10 mM), 7.0 μL水组成,最终体积为20 μL。反应在以下循环条件下孵育:在50℃下孵育2 min,在95℃下孵育2 min,在95℃下孵育15 s,在58℃下孵育15 s,在72℃下孵育30s,最后在72℃下孵育2 min。最后一个循环结束后,在60℃到95℃之间进行单循环,间隔5 s进行熔化曲线分析,以验证反应的特异性。通过对第一链cdna模板的连续稀释进行实时PCR来计算引物组的效率[30.]。使用geNorm软件[31], EF1α和UBQ被确定为跨发育阶段稳定表达的内参基因。各基因的相对表达量采用标准的E——ΔΔCq方法(32],用内标基因EF1α和UBQ归一化。按照Bustin等提出的MIQE指南进行表达定量和数据分析。[33]。每个定量PCR实验进行3个独立的生物重复和3个技术重复。各靶基因的相对表达量采用单因素方差分析。对数据进行统计分析P采用SPSS 16.0统计分析,差异有统计学意义。
结果
Illumina测序和新创组装
获得雄花雌雄同株的完整花转录组Taihangia以覆盖发育早期和晚期的雌雄同体花为材料,构建了4个cDNA文库(EM、EH、LM和LH)。在Illumina Hiseq 2500平台上对四个构建的文库进行配对端测序,共产生90,806,187个原始reads。经过对低质量区域、适配器和测序标签的过滤,我们获得了84,596,426条合格的Illumina reads(93.16%),其中EM、EH、LM和LH文库的clean reads分别为21,136,736条(93.36%)、20,963,888条(92.81%)、21,096,120条(93.16%)和21,399,682条(93.32%)1)。总体而言,该文库产生了21.31G碱基对的干净数据,Q30碱基占91.43%(测序错误率< 0.1%的序列百分比),表明该通量测序足够准确,可以进行进一步分析。每个库的干净读取是分开的新创分别得到42,630个(EM)、41,419个(EH)、38,764个(LM)和39,857个(LH) unigenes,平均长度为855 ~ 905 bp。之后,我们汇集了来自四个独立库的所有高质量读取来执行新创统一参考转录组的组装。利用Trinity程序,共组装了59064个长度大于200 bp的单基因,其中N50长度为1594 bp,平均长度为840 bp,最大转录物长度为11665 bp。的长度分布Taihangiaunigenes显示在附加文件中2:图S2。
评价评价…的质量新创没有参考基因组的组装,一种常用的方法是计算可以映射回单一基因集的reads的百分比。基于这个指标,我们发现超过84%的干净读取被映射回Taihangia每个库的unigenes(附加文件3.:表1)。除计算机质量评估外,还进行了标准PCR和Sanger测序,以评估其质量和准确性Taihangiaunigenes组装。在设计的18对引物中,16个单基因的17对引物可以稳定生成预期的PCR产物。用Sanger测序仪对17个PCR产物进行测序,并与已组装的单基因进行比较。比对分析表明,Sanger测序序列与组装的单基因吻合良好,平均序列同源性为98.7%,证实了转录组中组装的单基因的准确性(附加文件)4表2)。总之,我们的结果支持高质量的新创装配在当前的测序数据。
同源基因的功能标注与分类
所有组装的59,064个单一基因进行BlastX比较,设置截止e值为10−5,对比包括Nr、Pfam、Swiss-Prot、GO、KEGG和COG等公共数据库。结果,共有25,231个单基因序列显示出与公开数据库中已知蛋白质的显著相似性(表1)2)。根据Nr数据库的注释结果,e值分布分析显示,63.02%的匹配序列具有强同源性,且e值< 1 e−50,而36.98%的匹配序列在1e ~ 1e之间−5和1 e−50。相似性分布分析显示,58.29%的序列相似性大于75%,22.36%的序列相似性在60% ~ 75%之间。在物种分布方面,Nr数据库查询结果显示占68.03%t . rupestris带注释的序列与的序列匹配草莓属vesca,其次是李属却已(6.43%),碧桃(6.41%),马吕斯有明显(4.46%)和Pyrus x bretschneideri(3.30%)。的同源性搜索的特点Taihangia在附加文件中显示了与NR数据库的差异5:图S3。
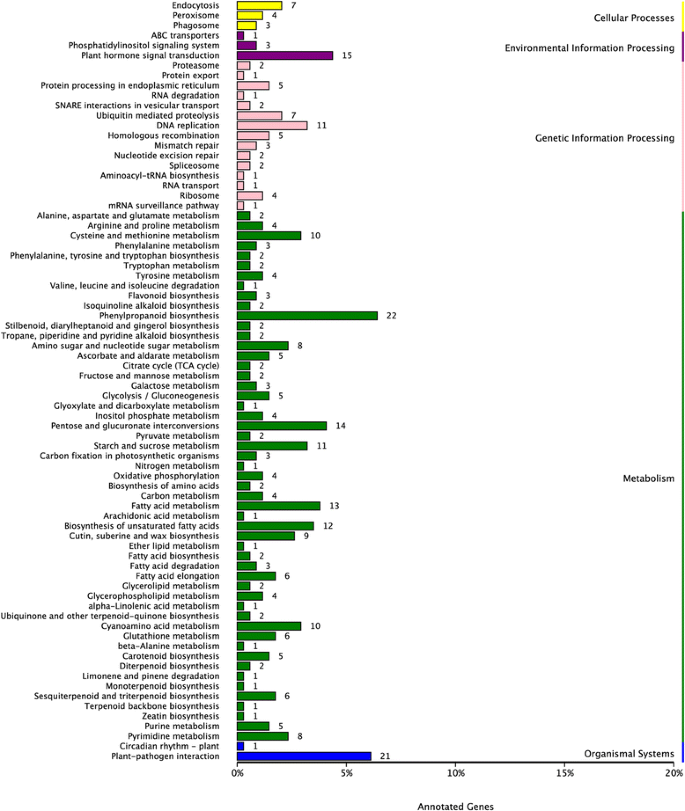
基于对Nr数据库的BLASTx结果,我们添加了GO术语,获得了这些组装的unique的GO功能注释和分类。在有Nr注释的24753个unique中,至少有一个GO项的共有12214个unique被分配到生物过程(24571个)、细胞成分(10195个)和分子功能(18239个)类别。这三个主要类别进一步划分为53个功能组,包括20个生物过程,17个细胞成分和16个分子功能。在分子功能类别中,“催化活性”是最具代表性的基团,其次是“结合”基团。在生物过程类别中,“代谢过程”和“细胞过程”是最具代表性的群体。细胞成分以“细胞”组和“细胞部分”组为主。氧化石墨烯分析表明,大量鉴定的独特基因与多种生物过程和分子功能有关Taihangia植物的组织。对标注的序列进行进一步的COG数据库搜索,以进行功能预测和分类。结果将7153个unigenes分为25个COG分类,其中“仅用于一般功能预测”的unigenes最多(2025个),其次是“复制、重组和修复”(1186个)、“转录”(1060个)、“信号转导机制”(904个)和“翻译后修饰、蛋白质转换、伴侣”(724个)。含有3个单基因的“核结构”类别是最小的一类。为了更好地了解unigenes的生物学功能,在KEGG数据库中检索了注释的序列。在25,231个注释的unigenes中,8,115个具有显著匹配,并被分配到126个KEGG通路。排名前五的途径分别是“核糖体”(ko03010)、“内质网蛋白质加工”(ko04141)、“碳代谢”(ko01200)、“氨基酸生物合成”(ko01230)和“剪接体”(ko03040)。功能GO, COG和KEGG通路注释和unigenes分类显示为附加文件6。综上所述,这些注释和分类分析将为今后的基因发现和功能基因组研究提供有价值的信息。
转录因子的鉴定
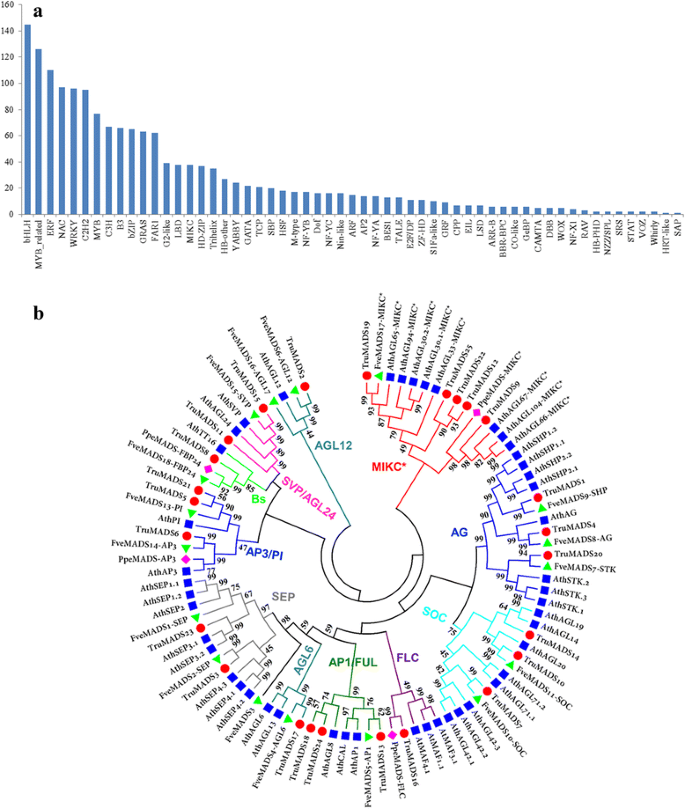
在本研究中,我们确定了1,661个TFs,占1.95%Taihangia经植物转录因子数据库PlantTFDB 4.0分类,共有57个TF家族。2、附加文件7)。在检测到的TF基因家族中,bHLH(145)是最丰富的TF家族,其次是MYB_related(126)、ERF(110)、NAC(97)、WRKY(96)和C2H2(95)。值得注意的是,共发现55个MADS-box基因,其中包括38个mikc型和17个m型tfTaihangia基于转录组数据。
研究的系统发育关系Taihangia基于预测的MADS-box蛋白全长序列的多序列比对,构建了邻链(Neighbor-Joining, NJ)系统发育树,并将其划分到已建立的亚家族中f . vesca,p . persica,拟南芥(无花果。2 b)。系统发育分析显示,25个MADS-box tf被分类为10个MIKC,其中包括8个前人报道的MADS-box基因c和一个MIKC*亚家族(附加文件8表S3)。此外,ABCDE模型中描述的aptala3 (AP3)、PISTILLATA (PI)、AGAMOUS (AG)、SHATERPROOF (SHP)、SEPALLATA (SEP)等MADS-box TFs也被包括在内。TruMADS13、TruMADS18和TruMADS24属于A类AP1/FUL亚家族,包括AP1和FUL进化支。TruMADS13被划分为AP1支系,TruMADS18和TruMADS24被划分为FUL支系。在功能b类(AP3/PI)组中,两个基因(TruMADS5和TruMADS21)属于PI支系,另一个基因(TruMADS6)属于TM6支系。三个MADS-box tf, TruMADS1, TruMADS4和TruMADS20被分配到AG家族,功能上被归类为C/D类组。TruMADS4属于c类AG,而TruMADS1和TruMADS20与c类AG最同源D-类官能团分别为SHP和STK。在E功能组中,两个MADS-box tf, TruMADS3和TruMADS23被分配到SEP亚家族。总体上,系统发育分析表明Taihangia在每个假定的官能团中都含有MADS蛋白。
雄性花和雌雄同体花的转录组谱
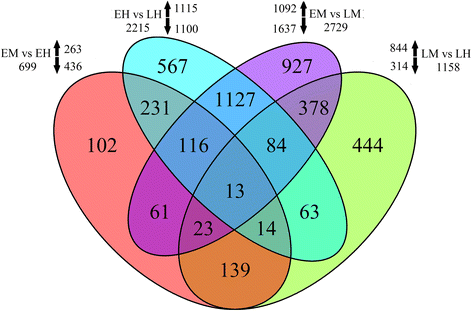
通过对四个文库(EM、EH、LM和LH)的两两比较,研究了雄性和雌雄同体在不同发育阶段基因表达谱的差异。共有36520个unigenes(53.26%)在所有文库中共享,而在单个文库中可检测到的unigenes约为15.51%(10639个)9:图S4)。在雌雄同体中,EH和LH分别有2176个和2475个单基因表达。同时,我们检测到3125个特异于EM的独特基因和2863个特异于LM的独特基因。共有12829个基因(18.71%)具有性别特异性,其中仅在雄花(EM + LM)和雌雄同体花(EH + LH)中分别检测到6304个和6525个。此外,14.06%的组装unique(9641个)仅在花蕾(EM + EH)中检测到,而9.77%的组装unique(6702个)仅在成熟花(LM + LH)中检测到,而在花蕾样品中没有检测到。综上所述,我们的研究结果表明,雄性或雌雄同体花中特异性表达的单基因可能是与雄花雌雄同体花形成调控有关的潜在候选基因Taihangia。
差异表达基因分析
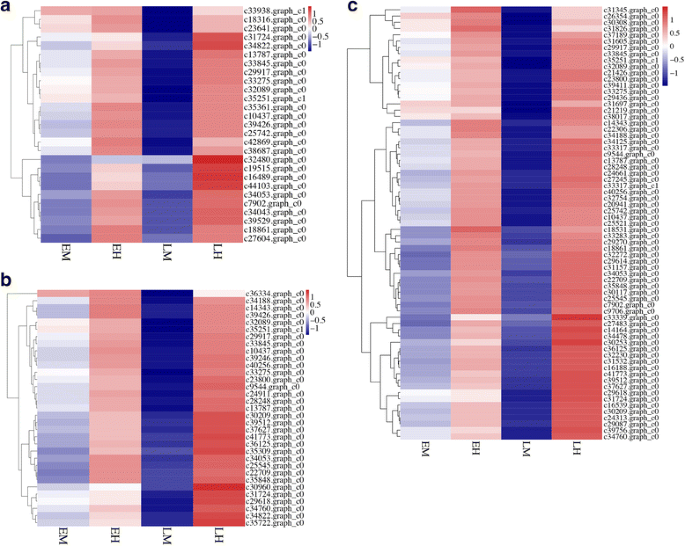
基于EM与EH、LM与LH、EM与LM、EH与LH的四项比较,我们共鉴定出4289个差异表达基因(DEGs)。其中,性别类型(EM + LM vs EH + LH)之间检测到1668个deg,发育阶段(EM + EH vs EH + LH)之间检测到3604个deg,表明大多数deg发生在不同发育阶段(图2)。3.)。为了鉴定参与花形成调控的相关基因,我们进一步分析了雄性和雌雄同体之间的1,668个基因。在发育早期,我们发现了263个在EH中表达的上调基因和436个在EH中表达的下调基因。另一方面,在发育后期LH中检测到844个上调基因和314个下调基因。有趣的是,180个基因在发育早期和后期都有差异表达,其中162个为雌雄同体偏异基因,18个为雄性偏异基因,这表明从花芽到成熟花的基因表达模式在不同发育阶段保持不变。
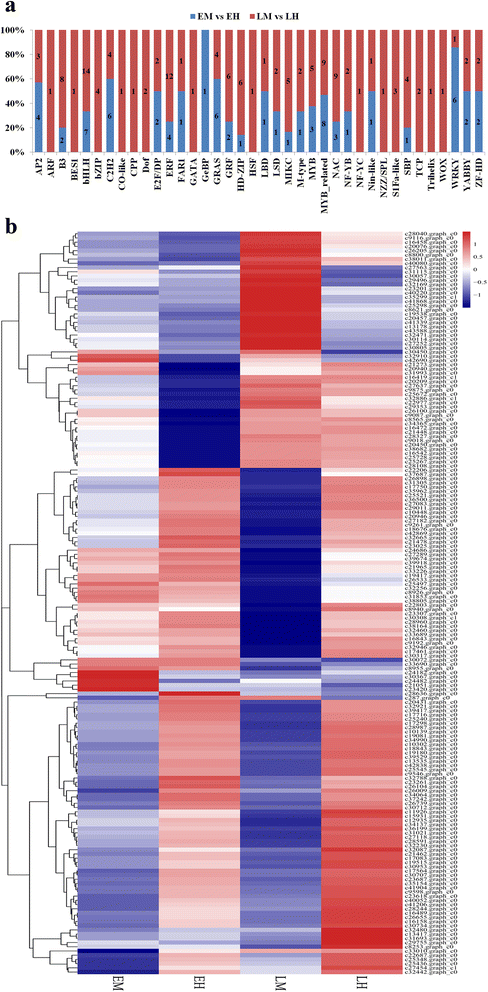
在本研究鉴定的1661个TF基因中,有176个TF基因在雌雄同体花中差异表达。这些TF基因被PlantTFDB 4.0分类为38个TF家族,其中bHLH家族(17个)代表了最多的差异表达TF,其次是ERF(15个)、MYB相关(14个)、WRKY(14个)和NAC(12个)。在花发育过程中,TF基因在雄花和雌雄同体花中表现出不同的表达模式(图2)。4),提示tf在发育早期和晚期可能有不同的作用。在发育早期,来自24个TF家族的67个TF基因在EM和EH的比较中存在差异表达。bHLH、MYB相关、ZF-HD、NAC、ERF等16个TF家族的33个TF基因显著上调,其余34个TF基因属于18个TF家族(如;GRF、MYB、C2H2和AP2)在EM中下调。在发育后期,通过LM和LH的比较,鉴定出135个推测的TF基因分为37个TF家族。其中,有105个TF基因在LH中表达显著上调。这些TF基因可能是雄花雌雄同体系统中雄花和雌雄同体花形成和发育的重要调控因子。由deg衍生的tf表达谱如图所示。4 b。
综上所述,雌雄同体花之间的DEG数量随着花的发育而显著增加,并且在EM与EH和LM与LH比较中观察到明显不同的DEG分布。考虑到Taihangia单性花和两性花发生在花发育早期,EM和EH之间的deg可能与性别分化有关,而LM和LH之间的deg可能与花器官发育和生殖有关。
氧化石墨烯与DEGs的途径分析
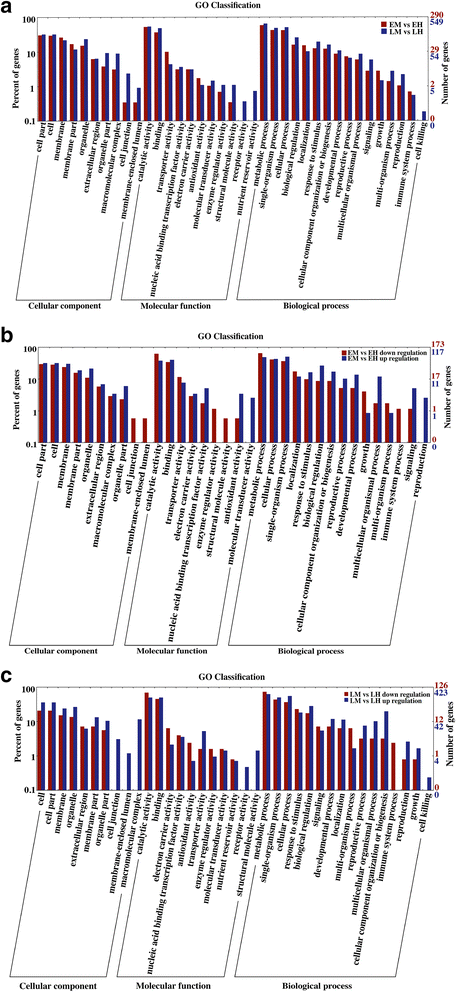
利用WEGO网站对雄性和雌雄同体样本之间的deg进行GO功能分类。在性别类型(EM + LM vs EH + LH)之间确定的1668个deg中,740个deg被分类为37个GO亚项。在细胞成分分类中,细胞(GO: 0005623)、细胞部分(GO: 0044464)、膜(GO: 0016020)和细胞器(GO: 0043226)是最丰富的亚术语。催化活性(GO: 0003824)和结合亚项(GO: 0005488)在分子功能分类中占主导地位。对于生物过程类别,高百分比的deg被分配给细胞过程(GO: 0009987),代谢过程(GO: 0044237)和单生物过程(GO: 0044699)子术语。5)。
为了更好地了解植物发育过程中花形成的调控机制Taihangia,我们重点分析了氧化石墨烯在EM与EH、LM与LH比较中上调和下调deg的功能分类。此外,使用BinGO程序中的超几何测试进行了GO富集分析p值≤0.05 [28]。在发育早期,290个deg被划分为34个GO亚项。EM中的173个上调基因和117个下调基因分别被分类为32和29个GO亚项(图2)。5 b)。在EM中,“花粉管发育”(GO: 0048868)、“花粉管生长调控”(GO: 0080092)、“水解酶活性,作用于糖基键”(GO: 0016798)中发现了氧化石墨烯术语的过度表达,表明这些基因参与了雄蕊发育。对于EH中上调的DEGs,大多数被过度描述的GO术语与细胞周期、发育过程和代谢过程有关,如“大分子代谢过程的调控”(GO: 0060255)、“细胞周期的调控”(GO: 0051726)、“解剖结构的发育”(GO: 0048856)、“膜的整体组成部分”(GO: 0016021)和“转录调控,dna模板化”(GO: 0006355)。在发育后期,在LM和LH的比较中发现了549个deg的38个GO亚项。126个上调基因和423个下调基因分别被分类为32和37个GO亚项(图2)。5度)。在LM中,氧化石墨烯术语的过度表达主要体现在“氧化还原酶活性”(GO: 0016491),而在LH中,氧化石墨烯术语的过度表达主要包括“细胞周期过程”(GO: 0022402)、“DNA甲基化”(GO: 0006306)、“花发育”(GO: 0009908)和“DNA结合”(GO: 0003677)。特别是,大量在早期发育阶段过多代表的GO术语,如“细胞周期调控”、“转录调控、dna模板化”和“发育过程调控”(GO: 0050793),在发育后期也得到了丰富。氧化石墨烯富集分析结果见附加文件10。
为了进一步研究这些deg的生化途径,我们将1,668个deg映射到KEGG数据库中的术语。其中219个基因具有KO ID,可分为78条通路,其中DEG数最多的是“苯丙类生物合成”(22条)、“植物与病原体相互作用”(21条)、“植物激素信号转导”(15条)、“戊糖与葡萄糖酸盐相互转化”(14条)、“脂肪酸代谢”(13条)和“不饱和脂肪酸代谢”(12条)6、附加文件11)。此外,我们对KEGG途径进行了富集分析,将DEGs划分为9个途径,如不饱和脂肪酸的生物合成、苯丙类生物合成以及戊糖和葡萄糖酸盐的相互转化(校正)p值≤0.05)。特别是两条途径“不饱和脂肪酸代谢”(12)和“DNA复制”(11)分别被高度代表,这与GO富集分析一致。
GO和KEGG通路分析表明,两性之间的deg具有广泛的功能,参与了许多生物学过程,如细胞增殖、染色质修饰和花的发育,这表明单性花和两性花的形成可能受到许多参与生物学过程的基因的控制。此外,所鉴定的GO亚项和KEGG通路可以为雌雄同株植物性别分化的分子机制提供进一步的信息Taihangia。
参与调控的基因taihangia雄花和雌雄同体花形成
花的发育
基于DEG分析和雄性花和雌雄同体花的基因表达谱,我们确定了一系列参与花发育的DEG(图2)。7一个)。其中,与花器官发育相关的MADS-box、WUSCHEL (WUS)、YABBY1 (YAB1)、lrr蛋白激酶BAM3等基因在雌雄同体花中表达上调。特别地,在AG、b姐妹(b)和MIKC*亚家族中发现了四个差异表达的MADS-box基因。其中,在雌雄同体花发育后期,TruMADS4 (AG)和TruMADS8 (FBP24)均上调表达。TruMADS20 (STK),属于D在整个花发育过程中,雌雄同体的表达量明显高于雄花。我们的研究结果表明,这些MADS-box基因在LH中表达较好,可能与雌雄同体花的雌蕊发育有关。相反,属于MIKC*亚家族的TruMADS22在EM的早期发育阶段被上调,这表明MADS-box TF可能参与了雄蕊发育。
表观遗传过程
表观遗传机制,包括DNA甲基化、组蛋白修饰、染色质构象和RNA干扰,在不改变DNA序列的情况下调节基因表达发挥重要作用。在本研究中,我们发现了一些参与DNA甲基化和组蛋白修饰的degTaihangia发育后期花(图2)7 b)。参与DNA甲基化过程的基因,包括三种DNA甲基化酶CMT3、MET和DRM2,在雌雄同体花中表现出差异表达。另一方面,组蛋白修饰相关基因,如组蛋白赖氨酸n -甲基转移酶ATXR6、ASHR3和SUVR4,在发育后期LM与LH的比较中表达差异。雌雄同体花发育早期表观遗传相关基因表达量差异不显著,而发育后期LH表达量均显著上调。值得注意的是,参与RNA沉默和RNA定向DNA甲基化(RdDM)途径的AGO 16基因的一个同源基因在LH发育后期表现出上调的表达模式。我们的发现表明,表观遗传过程可能广泛发生在雌雄同体花中,特别是在发育后期。
细胞周期过程
基于转录组数据,我们发现了大量参与细胞周期过程的deg,如DNA复制、细胞分裂和细胞周期调控,这些deg在雌雄同体花发育后期的表达显著增加(图2)。7 c)。许多deg,包括有丝分裂纺锤体检查点蛋白mad2样,atp依赖性DNA解旋酶q样,DNA拓扑异构酶3- α样和有丝分裂检查点丝氨酸/苏氨酸蛋白激酶BUB1 β样被分配到DNA复制功能组。同时,我们发现大多数属于细胞分裂功能组的deg在发育后期在LM中下调,包括65-kDa微管相关蛋白、G2/有丝分裂特异性细胞周期蛋白-1样、凝缩蛋白复合物亚基2样、DNA复制许可因子mcm2样和DNA复制许可因子MCM3。同样,参与细胞周期组的几个deg在雄性单性花中也表达下调,如微管相关蛋白RP/EB家族成员1C、DNA交联修复蛋白SNM1-like、FIZZY-RELATED 3-like和DNA交联修复蛋白SNM1-like。特别是,许多deg,包括G2/有丝分裂特异性cyclin-1样,fizzy -相关蛋白3样,有丝分裂纺锤体检查点蛋白mad2样,有丝分裂检查点丝氨酸/苏氨酸蛋白激酶BUB1 β样,cyclin- a1 -1样和cyclin- d3 -1样,在EH和LH的发育阶段均表现出显著的上调表达。总之,我们的结果表明,在雄性单性花中,细胞周期过程可能受到严重干扰。
不饱和脂肪酸的生物合成
不饱和脂肪酸被认为是必不可少的膜成分,在许多细胞事件中起着关键作用。不饱和脂肪酸的合成是一个复杂的过程,需要一系列酶的参与,如棕榈酰单半乳糖二酰基甘油Δ7去饱和酶(FAD5)和酰基-[酰基-载体蛋白]去饱和酶6 (ACP6)。在本研究中,几个DEGs编码了几个参与不饱和脂肪酸生物合成途径的关键酶,包括6个FAD5、3个超长链3-氧酰基辅酶a还原酶1样基因(ACPR)和1个ACP6基因。在雄花中被发现显著下调,表明不饱和脂肪酸的生物合成可能在雄花中受到限制。
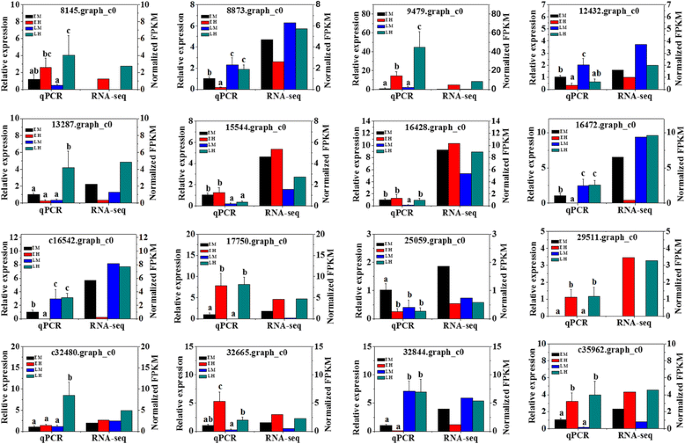
使用RT-qPCR验证unique基因和基因表达谱
为了证实RNA-seq数据鉴定的基因表达模式,我们用定量PCR检测了随机选择的16个deg的转录物水平。我们对这16个基因进行qRT-PCR,每组(EM、EH、LM和LH)进行3个生物重复(n = 12)。本研究选用的deg、id和引物对的详细信息见extra文件12表S4。结果发现,所选的16个deg均成功扩增出了预期大小的单条带,并且RT-qPCR检测到的基因表达模式与RNA-Seq数据一致(图2)8)。因此,RT-qPCR实验证实,从组装的转录组中获得的deg是准确的,从RNA-Seq数据中获得的基因表达谱是可靠的。
讨论
Taihangia转录组的概要文件
作为雌雄同株植物,Taihangia在统一的遗传背景下,为研究雌雄分化和建立单性花提供了良好的模式系统。在这项研究中,我们进行了新创利用Illumina RNA-Seq方法进行转录组测序,比较雄性花和雌雄同体花的转录谱。据我们所知,这是首次报道这种罕见的雌雄同株悬崖植物的转录组数据。从4个花测序文库中获得约84,596,426个合格的Illumina reads (Q30 > 91%),然后组装成59,064个unigenes。其中,25,231条(42.33%)已在公共数据库中被注释,33,833条(57.67%)未被功能鉴定。鉴于基因组和转录组学信息是有限的Taihangia,与独特花型相关的谱系特异性基因可能不包含在功能注释中,这意味着从我们的转录组数据中发现了潜在的新基因。通过对雄性和雌雄同体转录组数据的比较分析,我们发现了大量参与各种生物过程的候选deg,这些过程可能与雄性和雌雄同体花形成的调控有关。转录组数据为理解性别分化的特定生物学过程提供了重要资源,并促进了未来雄性雌雄同株研究中的基因发现Taihangia。
与花器官身份相关的MADS-box基因可能对花器官的形成至关重要
MADS-box转录因子最初被鉴定为花同源基因,是著名的“ABCDE”模型的主要组成部分,该模型描述了它们在决定花器官身份方面的作用[34]。基于Taihangia转录组数据,我们鉴定了25个MADS-box tf,包括ABCDE模型中描述的每个假定功能组中的几个MADS蛋白。根据“ABCDE”模型[35], A类、B类和e类MADS-box基因在男性和雌雄同体性别之间的表达水平相似,而C类和D- MADS基因在雌雄同体花中表达上调。在本研究中,大部分MADS-box TFs包括A-、B-和e类基因在雌雄同体花中没有差异表达。不出所料,两个MADS-box tf, TruAG (c级)和TruSTK (D-类),在雌雄同体花中表达上调。然而,值得注意的是,TruAG在EH中没有出现表达上调,而在LH中却出现了表达上调,这表明其表达模式似乎遵循雄花雌蕊发育受阻的规律。许多研究发现,在一些通过器官流产实现雌雄同体的雌雄异株和雌雄同株物种中,MADS盒子基因不参与性别决定[36,37]。在Taihangia在单性雄花中,雌蕊的起始和停止过程,性别决定的分子机制一定是作用于器官同一性的下游。因此,在雌雄同体花中更多表达的几个MADS-box tf的转录谱可能是雌雄同体花性别分化的结果而不是原因Taihangia, MADS-box基因与雌雄同体花的雌蕊发育有关。
表观遗传相关基因可能与雌雄同体花的生殖有关
表观遗传过程,如DNA甲基化、组蛋白修饰和小RNA,在花发育过程中基因表达的调控中发挥重要作用[qh]38]。作为参与DNA甲基化过程的关键酶,DNA甲基转移酶和组蛋白H3K9甲基转移酶对DNA甲基化的建立和维持至关重要[39]。在我们的研究中,三种DNA甲基转移酶(MET1, DRM2和CTM3)和三种组蛋白H3K9甲基转移酶(ASHR3, ATXR6和SUVH4)在LH中表现出较好的表达,表明它们可能在Taihangia发育后期的生殖此外,还鉴定了Argonaute 16 (AGO16)、基因沉默抑制因子3 (SGS 3, c26995.graph_c0)、RNA依赖性RNA聚合酶3 (RDR3, c36802.graph_c0)、Dicer同源3a样基因(DCL3, c35101 .graph_c0)和RPD1 (c38350.graph_c0)等可能参与RNA诱导沉默和RNA定向DNA甲基化(RdDM)途径的基因Taihangia转录组数据。特别是在发育后期,AGO 16在LH中的表达量比LM高约3倍。作为关键调控因子,AGO蛋白和DCL3以及RDR对表观遗传沉默至关重要[40]。基因表达分析显示,DCL、RDR、RPD三个基因在LH中的表达量比LM高2倍以上,表明广泛的小RNA相关过程可能发生在Taihangia发育阶段的雌雄同体花。目前,越来越多的证据支持表观遗传过程在植物生殖发育调控中发挥重要作用[41- - - - - -44]。例如,Galla等人。[41[j]报道,小RNA生成相关基因,包括DCL3、AGO、SGS3和RDR,参与植物生殖,如胚珠中细胞的身份和命运贯叶连翘。这些表观遗传相关基因的表达上调Taihangia雌雄同体花,特别是发育后期的雌雄同体花,可能表明表观遗传调控在植物生殖过程中发挥了重要作用,如雌性配子体的形成。表观遗传过程的作用t . rupestris目前尚不清楚,需要进一步研究相关基因在表观遗传过程中的作用。
细胞周期紊乱可能是雄花雌蕊败育的原因之一
在花发育过程中,雌蕊或雄蕊发育停滞是雌雄同株植物的正常现象[45- - - - - -47]。一些生物过程,如程序性细胞死亡(PCD)和细胞分裂的停止,对单性花的形成至关重要。48]。基于TaihangiaRNA-seq数据显示,大量参与细胞周期、DNA复制和细胞分裂的deg在雄花发育过程中显著下调,表明这些生物学过程在抑制雌蕊发育中发挥了重要作用Taihangia雄花。对于许多植物物种来说,PCD在植物形态发生的许多方面都是必不可少的,它是单性花中生殖器官发育停滞的一个常见过程[45,49,50]。然而,我们没有发现PCD参与雌蕊流产Taihangia基于转录组数据的雄花。据报道,不育性器官的发育停滞和随后的花的雌雄同体可能是由于细胞分裂的停止造成的[46]。同样,参与细胞分裂的相关基因表达下调可能导致细胞增殖受到限制,这可能与雌蕊发育受阻密切相关Taihangia单性雄花。
作为主要调控基因,细胞周期蛋白在控制细胞周期进程中发挥关键作用,包括DNA复制、G2/M转化和有丝分裂[qh]51]。本研究发现,与细胞周期调控相关的几个周期蛋白基因(CYCA1、CYCA2、CYCA3和CYCD3)在单性雄花中一致下调。特别是,cyclin-A1-1-like和cyclin-D3-1-like在EM和LM中跨发育阶段的表达水平显著降低,表明细胞周期调节与单性花的建立之间存在潜在的机制联系Taihangia。在花发育过程中,cyclin- a1和cyclin-D3被认为是参与调节花原基信息和分生组织活性的核心调控基因[52]。例如,cyclin-D3可以通过控制细胞分裂和细胞分化的平衡,在茎尖分生组织中定义不同的发育区域,作为侧枝器官之间的发育边界[51]。在Taihangia单性雄花中,cyca1和CYCD3表达下调可能导致雌蕊原基细胞周期紊乱,进而决定雌蕊发育受阻。这些结果表明,参与细胞周期进程的核心调控基因可能参与了雌蕊败育Taihangia由细胞周期调节的单性花。
冷反应基因可能影响单性花的形成
众所周知,单性花的形成受温度、光照强度、光周期和营养条件等环境因素的影响,并通过特定的基因表达[53- - - - - -55]。对于雄全同株的Taihangia,花原基起始时的温度是决定性别类型形态发生的关键,单性雄花的产生归因于低温下雌蕊败育[9]。为了应对不利的冷胁迫,植物发展出多种防御机制,通过调节分子、生理和发育过程获得耐寒性[j]。56]。例如,增加细胞膜的不饱和脂肪酸含量可以促进血浆和叶绿体膜的稳定,从而提高对冷胁迫的抵抗力[57]。作为参与不饱和脂肪酸生物合成的关键酶,FAD7、ACPR和acp6在EM中显著下调,表明多不饱和脂肪酸的生物合成可能受到了限制Taihangia雄花。在低温条件下,高等植物可以诱导许多冷调控基因,如LEA家族基因[54]。例如,LEA蛋白被认为在冷应力下通过与膜表面或水分子相互作用在膜稳定中起作用[58]。转录组数据显示,LEA Dc3、LEA B19.3、抗坏血酸氧化酶和过氧化物酶等抗寒基因在雄花中显著下调。这些结果与Shi等人最近对日本杏雄花和雌雄同体花的比较转录组分析的研究结果一致。[qh]6],认为雄花抗寒基因表达的减少可能与低温下雌蕊败育有关。综上所述,这些结果表明,在雄性雌雄同株中,早期冷响应基因表达的下调可能影响单性雄花的形成Taihangia。
候选TFs在雄花和雌雄同体花形成中的重要调控作用
在植物中,TF基因在植物生长发育的许多方面发挥着重要作用[59]。在本研究中,我们发现了大量的TF基因在雌雄同体花中表现出显著的表达差异。这些tf被认为参与了雄性花和雌雄同体花形成的调控Taihangia。结果表明,NAC、WRKY、bHLH、GRAS和ERF在雄花中的表达均有所增加,表明雄花可能比雌雄同体花受到的伤害更严重。两性花中大部分TFs的上调与花器官发育有关,包括WOX、YAB、AP2和C2H2。例如,已知在花分生组织中维持干细胞活性的多个WOX TF基因WUSCHEL (WUS) [60]。诱导花后,WUS可诱导AG的表达并促进AG在发育花中的上调[61]。在我们的研究中,在雌雄同体花的早期发育阶段没有观察到WUS的一个同源物的高表达。同样,TruAG)EM和EH的表达没有差异,表明可能存在不同的调控机制Taihangia花的发育。YABBY基因家族参与侧面器官的正面-背面极性,并参与多种过程,如花模式,器官生长和分生组织的维持[j]。62,63]。本研究发现的YABBY1同源基因在EH和LH中均出现上调,表明YABBY TFs与两性花的形成密切相关。有趣的是,在两性花中检测到5个编码生长调节因子(GRF)的基因,并显示出一致的表达增加。GRFs是植物特异性转录因子,通过细胞周期调控和包括花形成在内的核心发育过程在细胞增殖控制中发挥重要作用[64,65]。最近,Liang等报道了GRF表达水平的显著降低导致雌蕊异常,并提出了GRF在控制心皮数和雌蕊发育中的重要作用[66]。我们的结果与拟南芥证实了雄花细胞增殖受到干扰导致雌蕊败育。
结论
新创雄性雌雄同株的转录组测序Taihangia本研究采用Illumina配对端测序技术。总共从雄性和雌雄同体花中获得了59,064个独特序列,覆盖了发育的早期和晚期,并在公共数据库中注释了25,231个独特序列。重点研究雄花雌雄同株花形成的调控机制Taihangia在雄性花和雌雄同体花之间鉴定出1668个deg。GO和通路分析表明,这些deg参与了广泛的生物学过程,如花的发育和繁殖、细胞周期、表观遗传过程和转录调控,并为未来的功能分析提供了许多可能与性别分化相关的候选基因。的转录组数据Taihangia有助于加深对雌雄同株植物性别分化和单性花形成的分子机制的认识,并为雌雄同株植物花形成的调控提供理论依据。
缩写
- 齿轮:
-
同源基团簇
- 度:
-
差异表达基因
- FPKM:
-
每千个碱基的片段每一百万个片段映射的外显子
- 走:
-
基因本体论
- KEGG:
-
京都基因与基因组百科全书
- 存在:
-
定量逆转录聚合酶链反应
- RdDM:
-
RNA直接DNA甲基化
- RNA-Seq:
-
核糖核酸测序
- Taihangia rupestris:
-
Taihangia
- TF:
-
转录因子
参考文献
- 1.
了解植物生殖多样性。中国生物医学工程学报(英文版);2010;35(5):391 - 391。
- 2.
李建军,李建军。雌雄同体发育的研究进展chaerophyllum bulbosum(apiaceae-apioideae)。中国生物医学工程学报。2013;12(2):444 - 444。
- 3.
[张建平。植物性多样性的进化。]医学学报。2002;3:274-84。
- 4.
李建军,李建军,李建军,等。雄性雄性不育研究进展大戟属植物(大戟科)。安博。2002;89:571-7。
- 5.
Miller JS, Diggle PK.雌雄同株果实大小与性表达的相关进化茄属植物部分acanthophora和lasiocarpa(茄科)。[J] .中国生物医学工程学报。2007;24(4):591 - 591。
- 6.
石涛,高震,王磊,张震,庄伟,孙慧,钟伟。利用全基因组转录分析鉴定日本杏雌蕊败育相关差异基因。PLoS ONE。2012; 7: e47810。
- 7.
于涛,李超。Taihangia中国蔷薇科一新属。物理学报,1980;18:469-72。
- 8.
于涛,李晨。属的系统地位taihangia蔷薇科。物理学报,1983;21:29 - 35。
- 9.
陆伟。性器官发育taihangia rupestris两性花两性器官发育对温度的要求不同。医学学报。1996;38:174-9。
- 10.
王勇,田慧,杜鑫,卢森,卢伟,崇科,孟哲。一种推定的植物E类基因的分离与鉴定taihangia rupestris。植物学报,2009;39(4):444 - 444。
- 11.
吕松,范勇,刘磊,刘松,张伟,孟哲。TrPI异位表达[j]taihangia rupestris(蔷薇科)PI同源,引起拟南芥营养结构的改变。植物生理学报,2010;37(2):444 - 444。
- 12.
卢松,杜鑫,卢伟,钟凯,孟哲。太行菊(蔷薇科)两个AGAMOUS like MADS - box基因在C类和D类花同源功能进化中的独立轨迹。进化发展。2007;9:92-104。
- 13.
Grabherr M, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q.基于RNA-Seq数据的全基因组转录组组装。生物工程学报。2011;29:644-52。
- 14.
Ramos M, Coito JL, Silva HG, Cunha J, Costa MMR, Rocheta M.野生葡萄花发育和性别规格。基因工程学报,2014;15:10 . 95。
- 15.
李建军,李建军,李建军,李建军。芦笋雌雄异株基因表达的研究[j]。芦笋officinalis)。新植物学报。2015;07:883 - 92。
- 16.
陈J,郑Y,秦L,王Y,陈L, Y,范Z,陆microrna的识别和目标通过高通量测序和degradome分析男性和女性芦笋officinalis。植物学报,2016;16:1-19。
- 17.
宋勇,马凯,齐丹,陈强,田静,张丹。利用转录组、激素和DNA甲基化分析揭示雌雄异株植物两性二形花发育Populus tomentosa。中国生物医学工程学报。2013;33(3):559 - 559。
- 18.
Alagna F, Cirilli M, Galla G, Carbone F, Daddiego L, Facella P, Lopez L, Colao C, Mariotti R, Cultrera NGM。橄榄花发育的转录分析及调控事件齐墩果欧洲公司l .)。PLoS ONE。2016; 11: e0152943。
- 19.
苗艳,朱卓,郭强,朱艳,杨霞,孙艳。差异表达基因转录组分析为郁金香匍匐茎形成提供了新的视角。植物科学,2016;7:409。
- 20.
张勇,李伟,窦勇,张军,姜刚,苗磊,韩刚,刘勇,李宏,张震。花茶红黄果实RNA-Seq转录本定量分析差异基因PLoS ONE。2015; 10。
- 21.
陈建军,陈建军,陈建军,李建军,等。基于Blast2GO的功能基因组学研究进展。生物信息学。2005;21:3674-6。
- 22.
张你们J,方L,郑H, Y,陈J,张Z,李,李R, Bolund L,王J . WEGO: web工具绘制注释。中国生物医学工程学报,2006;33(4):893 - 897。
- 23.
金杰,田芳,杨D[3]孟彦强,孔磊,罗军,高刚。植物转录因子与调控相互作用的研究进展。核酸学报,2017;45:D1040-D1045。
- 24.
李波,杜威,CN。RSEM:从有或没有参考基因组的RNA-Seq数据中准确定量转录本。生物医学工程学报,2011;12:323。
- 25.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, Van Baren MJ, Salzberg SL, Wold BJ, Pachter L. RNA-Seq转录本装配和定量揭示细胞分化过程中无注释转录本和同型异构体转换。生物工程学报。2010;28:511 - 511。
- 26.
Anders S, Huber W.序列计数数据的差异表达分析。中国生物医学工程学报。2010;11(1):1 - 12。
- 27.
benjamin Y, Yekutieli D.依赖关系下多重测试中错误发现率的控制。统计学家。2001;29:1165-88。
- 28.
Maere S, Heymans K, Kuiper M. BiNGO:一个细胞景观插件来评估生物网络中基因本体类别的过度表示。生物信息学。2005;21:3448-9。
- 29.
毛翔,蔡涛,Olyarchuk JG,魏林。基于KEGG同源词的基因组自动标注与途径识别。生物信息学。2005;21:3787 - 93。
- 30.
Ginzinger DG。采用实时定量PCR进行基因定量。血液学杂志。2002;30:503-12。
- 31.
Vandesompele J, De Preter K, Pattyn F, Poppe B, Speleman F.多内控基因几何平均实时定量RT-PCR数据的精确归一化。基因组生物学。2002;3:1-12。
- 32.
Pfaffl兆瓦。实时RT-PCR相对定量的新数学模型。中国生物医学工程学报,2001;29(1):45。
- 33.
Bustin SA, Benes V, Garson JA, Hellemans J, Huggett JF, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL.实时荧光定量PCR实验的最小信息发布指南。临床化学。2009;55:611-22。
- 34.
王晓明,王晓明,王晓明,等。植物mads结构域因子的发育与进化多样性分析。发展。2012;139:3081 - 98。
- 35.
花器官同一性的发展:来自MADS的故事。植物学报,2001;4(4):775 - 785。
- 36.
Kater MM, Franken J, Carney KJ, Colombo L, agency GC。雌雄同株黄瓜的性别决定仅限于特定的花轮。植物学报。2001;13:481 - 493。
- 37.
男孩和女孩出来玩:雌雄异株植物的分子生物学。[j] .中华医学会杂志。2000;86:211-21。
- 38.
杨慧,常飞,游超,崔健,朱刚,王磊,郑勇,齐军,马辉。花发育过程中全基因组DNA甲基化模式及其与基因结构和表达的复杂关联拟南芥。植物科学学报,2015;31(1):391 - 391。
- 39.
雅各布森SE。建立、维持和修改植物和动物的DNA甲基化模式。中国农业科学,2010;11(1):1 - 4。
- 40.
张建平,李建平,李建平,等。小rna在植物繁殖过程中的多重作用。植物学报,2011;14(4):588 - 593。
- 41.
王晓明,王晓明,王晓明,等。基因序列的研究进展贯叶连翘L.花转录组鉴定与植物生殖感官相关的潜在基因。BMC Genomics. 2015;16:254。
- 42.
宋强,关旭,陈志军。异源四倍体棉花胚珠和纤维发育过程中小rna和DNA甲基化的动态作用。11.科学通报,2015;
- 43.
Garciaaguilar M, Michaud C, Leblanc O, Grimanelli D.玉米生殖器官DNA甲基化途径失活导致无融合样表型。植物学报。2010;22:3249-67。
- 44.
杨玲,吴艳,于敏,毛波,赵波,王杰。水稻雌性不育胚珠全基因组转录组分析揭示其败育机制。足底。2016;244:1011-28。
- 45.
李建军,李建军,李建军,等。植物生殖器官发育阻滞的研究进展葡萄ssp。silvestri。性学通报,2003;15(3):291 - 300。
- 46.
Daher A, Adam H, Chabrillange N, Collin M, Mohamed NA, Tregear J, Aberlencbertossi F.两性花芽向单性花过渡的细胞周期停滞特征凤凰dactylifera。生物医学工程学报。2010;26(6):591 - 591。
- 47.
Matsunaga S, Uchida W, Kawano S.雌雄异株植物沉默花发育过程中的性别特异性细胞分裂。植物学报。2004;45(5):795 - 802。
- 48.
Diggle PK, Stilio VSD, Gschwend AR, Golenberg EM, Moore RC, Russell JRW, Sinclair JP。多种发育过程是被子植物性别分化的基础。科学进展。2011;27:368-76。
- 49.
Gunawardena啊。植物细胞程序性死亡和组织重塑。[J] .中国生物医学工程学报,2008;29(1):445 - 451。
- 50.
细胞死亡和细胞保护基因决定了玉米雌蕊的命运。发展。1999;126:435-41。
- 51.
王刚,孔辉,孙艳,张欣,张伟,Altman N, Depamphilis CW,马慧。拟南芥细胞周期蛋白家族的全基因组分析及植物细胞周期蛋白样蛋白的系统发育比较分析。植物生理学报。2004;35(5):1084 - 1099。
- 52.
Gaudin V, Lunness P, Fobert PR, Towers MI, Rioukhamlichi C, Murray JAH, Coen E, Doonan JH。的表达式D-cyclin基因在金鱼鱼的顶端分生组织中定义了不同的发育区域,并受cycloidea基因的局部调控。植物生理学报。2000;22(2):1137 - 48。
- 53.
Reale L, Sgromo C, Ederli L, Pasqualini S, Orlandi F, Fornaciari M, Ferranti F, Romano B.橄榄花雌雄同体和雄蕊花形态细胞学发育和淀粉积累齐墩果欧洲公司l .)。性学通报,2009;22:109-19。
- 54.
Zinn KE, Tuncozdemir M, Harper JF。温度胁迫与植物有性生殖:揭示最薄弱环节。中国生物医学工程学报,2010;31(1):591 - 591。
- 55.
Sobral R, Silva HG, moraiscecilio L, Costa MMR。探究单性花发育背后的分子调控。植物科学,2016;7:160。
- 56.
刘建勇,洪森,赵松,吴军,李松,朴超。短茎远端粒冷响应C-repeat结合因子的分子和功能研究。植物学报,2014;14:15。
- 57.
Upchurch RG。植物对逆境反应中的脂肪酸不饱和、动员和调控。生物工程学报。2008;30:967-77。
- 58.
王晓明,王晓明,王晓明,王晓明,等。小麦叶绿体低温调控后胚发育的分子特征及功能分析。植物生理学报。2002;29(1):1 - 8。
- 59.
Schwechheimer C, Zourelidou M, Bevan MW。植物转录因子研究。植物生理学报,2003;49(1):127。
- 60.
Laux T, Mayer KF, Berger J, Jurgens G.拟南芥茎部和花分生组织完整性需要WUSCHEL基因。发展。1996;122:87 - 96。
- 61.
Lenhard M, Bohnert A, Jurgens G, Laux T.拟南芥花分生组织中WUSCHEL和AGAMOUS互作对干细胞维持终止的影响。细胞。2001;105:805-14。
- 62.
鲍曼杰。YABBY基因家族与细胞背面命运。植物学报,2000;3:17-22。
- 63.
Lugassi N, Nakayama N, Bochnik R, Zik M.一个新的丝状花等位基因揭示了花序与花分生组织和花形态发生关系的新见解。植物学报,2010;10(1):1 - 13。
- 64.
Omidbakhshfard MA, prost S, Fujikura U, Muellerroeber B.生长调节因子(GRFs):一个转录因子家族在植物生物学中的重要作用。植物学报,2015;8:998-1010。
- 65.
李斌,高健,李顺,李勇,朴健,金建辉。拟南芥grf -相互作用因子基因家族在决定器官大小和多种发育特性方面发挥重叠功能。植物生理学报。2009;51(1):655 - 658。
- 66.
梁刚,何红,李艳,王峰,于东。microRNA396介导拟南芥雌蕊发育的分子机制。植物生理学报,2014;24(4):559 - 564。
致谢
作者感谢刘森博士和张云星先生的样品采集。为了采集植物样本,爬上悬崖面既困难又危险。感谢毛宇翔博士对本文的改稿和语言修改提出的宝贵意见。
资金
国家自然科学基金(No. 31370434、31170354、31100182)和河南理工大学科技创新基金(B2008-25、T2013-2)资助。
数据和材料的可用性
原始测序数据存储在NCBI Short Read Archive (SRA)数据库(http://www.ncbi.nlm.nih.gov/sra/),登记号为SRP081195。其他支持数据作为附加文件包含。
作者的贡献
WL和YWZ构思和设计实验,ZD进行差异表达分析,QW和YDZ收集样本并进行实验,WL和LZ分析数据并撰写论文,LZ、HG和TC部分修改稿件。所有作者都阅读、审阅并批准了最终稿件。
相互竞争的利益
作者宣称他们没有竞争利益。
发表同意书
不适用。
伦理批准并同意参与
不适用。
作者信息
从属关系
相应的作者
附加文件
附加文件1:图S1。
雄花和雌雄同体的花产生在同一个体内Taihangia在悬崖上。(PDF 671kb)
附加文件2:图S2。
单基因序列的长度分布。(TIF 2980kb)
附加文件3:表S1。
对reads与单基因库比对的总结。(DOCX 14kb)
附加文件4:表S2。
使用Sanger测序验证组装的单基因。(docx17kb)
附加文件5图S3。
的同源性搜索的特点Taihangia与NR数据库进行比对。(PDF 136kb)
附加文件6:
功能GO、COG和KEGG通路的注释和分类Taihangiaunigenes。(XLSX 3235kb)
附加文件7:
针对PlnTFDB数据库的转录因子鉴定。(xlsx226kb)
附加文件8:表S3。
TaihangiaMADS-box基因名称和属性。(docx17kb)
附加文件9:图S4。
EM, EH, LM和LH样品中特定和共享的单基因数量的维恩图。(TIF 1967 kb)
附加文件10:
在EM与EH、LM与LH中,deg的GO术语分别具有过度代表性。(XLSX 74kb)
附加文件11:
雌雄同体花间deg的途径分类及富集分析。(xlsx25kb)
附加文件12:表S4。
选择用于RT-qPCR的deg, id和引物对。(docx18kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
李伟,张磊,丁志。et al。新创雄性花和雌雄同体花的测序和转录组比较分析为雄花雌雄同体花的形成调控提供了新的思路Taihangia rupestris。BMC Plant Biol17日,54(2017)。https://doi.org/10.1186/s12870-017-0990-x
收到了:
接受:
发表:
关键字
- 男花
- 两性的花
- 花的形成
- 性别分化
- Taihangia
- 转录组