摘要
背景
从头由短干扰RNA触发DNA甲基化被称为RNA指导的DNA甲基化(RdDM)。转录基因沉默(TGS)通过RdDM可以使用病毒载体来诱导。我们先前已经引起RdDM在35S启动子的绿色荧光蛋白(GFP) - 表达烟草benthamiana利用黄瓜花叶病毒载体的16c线。GFP荧光表型在第一个自交(S1)的不同程度的恢复从TGS上绿色荧光蛋白表达式。在自交的第二代(S2植物),表型再次分离。解释什么产生红色和橙色的类型可以回答表观遗传学中一个非常重要的问题:如何在TGS的稳定性RdDM诱导后的维护?
结果
在亚硫酸氢盐测序分析中,我们发现S基因中红色和橙色类型的启动子总体超甲基化模式存在显著差异1植物,但s的差异很小2植物。因此,我们假设某些特定胞嘧啶残基的甲基化可能是决定这两种表型的重要因素。为了找到区分稳定、稳健的TGS和不稳定、不完全遗传的TGS的因素,我们分析了甲基胞嘧啶残基对TGS的直接影响。由于尚未证实已知序列元件上少数特定胞嘧啶残基的DNA甲基化确实可以确定TGS的健壮性,我们新开发了一种方法,可以直接评估特定甲基化对启动子活性的影响。在本实验中,我们发现特定的胞嘧啶甲基化对TGS的影响在正链和负链之间是不同的。
结论
我们在病毒诱导的TGS植物后代中发现了两种不同的表型,稳定的和不稳定的TGS。我们的亚硫酸氢盐测序分析表明,35S启动子中某些特定胞嘧啶残基的甲基化作用决定了tgs是稳定的还是不稳定的。利用开发的方法,我们推断DNA甲基化的异质性在正链和负链之间可以差异地决定TGS。
背景
DNA甲基化是植物基因组中高度保守的表观遗传标志,控制基因表达。转基因和内源性基因的转录基因沉默(TGS)调控基因表达的研究已经广泛[1那2那3.那4.].在植物中,DNA甲基化发生在三个序列上下文:CG,CHG和CHH(其中,H是A,C或T)。的CG甲基化是通过METHYLTRANSFERASE1(MET1)维持,而CHG和CHH甲基化是由植物特异性CHROMOMETHYLASE3(CMT3)和CHROMOMETHYLASE 2(CMT2)分别维持,[1那2那3.那4.那5.].此外,新的DNA甲基转移酶,结构域重排甲基转移酶2 (DRM2)参与DNA甲基化的维持和启动[6.].
在植物中,DRM2需要从头DNA甲基化,它是由短干扰RNA(siRNA)触发6.].的siRNA引导DRM2对基因组DNA靶序列,并通过RNA指导的DNA甲基化引导从头DNA甲基化(RdDM)7.那8.那9.那10那11那12那13那14那15那16那17那18],其中两个植物特异性RNA聚合酶,聚合酶IV和Pol v播放重要作用。24个核苷酸(nt)siRNA被生成并由波尔IV,RNA依赖性RNA聚合酶2(RDR2)和DICER样3(DCL3)扩增。然后将24-nt的siRNA被装载到的Argonaute(AGO)用于随后的DRM2介导的DNA甲基化。AGO4最终与极化电压亚基,核RNA聚合酶E1(NRPE1)相互作用并与DRM2复杂起始DNA甲基化。近日,RdDM已经分为两个途径,规范和非规范的。在经典途径,酶反应(POL IV-RDR2-DCL3)的级联产生24-NT的siRNA在音乐会,而几个附加的小RNA似乎通过非经典途径参与RdDM [19].
假设RdDM通过的siRNA从一个启动子序列引发,有两个选项,人为地诱导特异性的DNA甲基化:转基因诱导的或病毒诱导的。第一种方法涉及其中双链RNA(dsRNA的)针对靶启动子序列中产生,例如转基因植物,创建由反向重复构建体[20.那21.那22.],而第二个涉及病毒诱导的转录基因沉默(VITG),这相当方便,因为我们不必产生转基因植物[23.].然而,存在这样的转基因诱导的siRNA的两种方法生成即使在后代植物但病毒驱动的siRNA被不可检测搬入到下一代无病毒感染之间的大的差异。
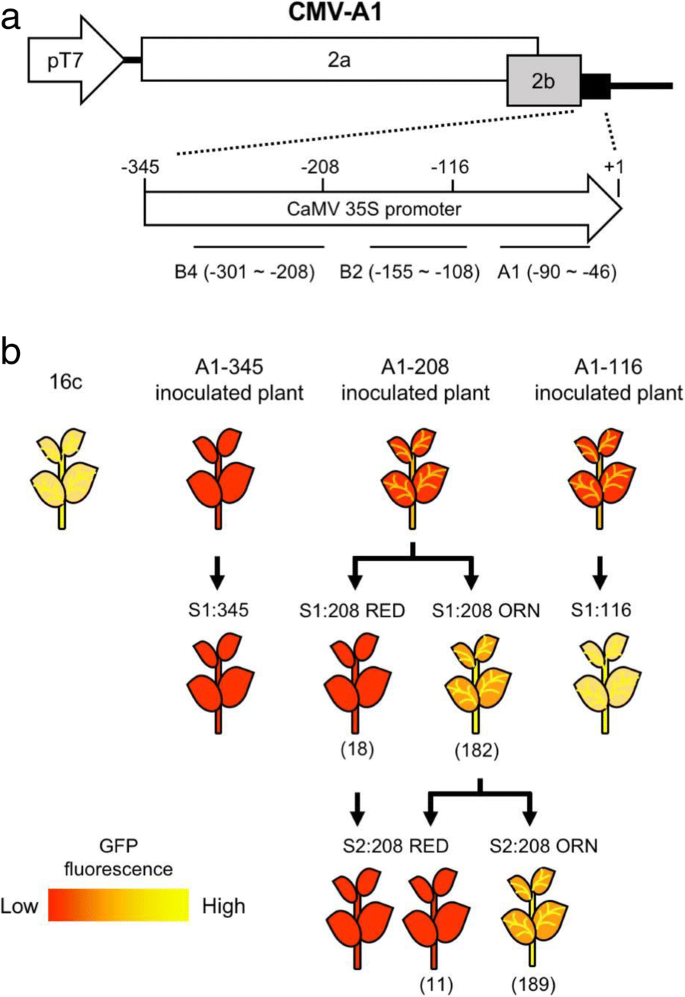
病毒相当数量已经为VITGS被开发出来,例如,苹果潜伏球形病毒(ALSV)24.那25.那26.],烟草响尾蛇病毒[27.那28.]、大麦条纹花叶病毒[29.]和马铃薯病毒X [30.那31.].我们以前已经开发了黄瓜花叶病毒(CMV)为基础的载体,CMV- a1用于PTGS和TGS [32.那33.那34.那35.那36.那37.].使用CMV-A1,我们可以成功地诱导针对一些内源性基因和转基因的VITGS [33.那38.].当我们接种表达绿色荧光蛋白(GFP)的转基因烟草benthamiana(16C)与含35S启动子序列的各种尺寸的CMV-A1的构建体,我们发现绿色荧光蛋白由于RdDM,表达水平下调,病毒整合序列的大小对DNA甲基化很重要[39.].
CAMV 35s启动子是一种强大的启动子,即它已广泛用于植物中的基因表达。已经鉴定了影响启动子活动的几个序列结构域。激活序列因子1和2(1和2(分别位于亚域A1和B1中,已被发现作为转录因子结合位点[40那41.那42.那43.那44.那45.].子域B2和B4也已报道是启动子活性重要[46.].然而,尚未证实这些序列元件上的特定DNA甲基化可以控制启动子的活性。
在我们观察到16C植物中对35s启动子的VITG,我们注意到了无病毒的s1当暴露于UV光时,后代植物包含两种表型:由于来自组合的叶绿素自发荧光和GFP荧光而导致的叶绿素的自发荧光引起的红颜色(红色)。大部分的s2S1ORN植株为ORN植株,但偶尔出现红色植株。35S启动子序列对应的病毒载体来源的sinas将不会在子代植物中产生,因为子代不再被重组病毒感染。对于DNA甲基化维持和启动的未知机制,我们提出了以下问题:红色和ORN植物的区别是什么?如何控制稳定和不稳定的TGS ?我们对两种表型中35S启动子DNA整体甲基化状态的初步分析表明,RED和ORN植株之间并没有太大差异,这表明目标序列上特定胞嘧啶残基的甲基化可能是TGS遗传的重要因素。为了回答上述问题,我们在这里详细分析了vitgs诱导的16c植物的DNA甲基化。
结果
VITGS的差动感应取决于靶序列的大小
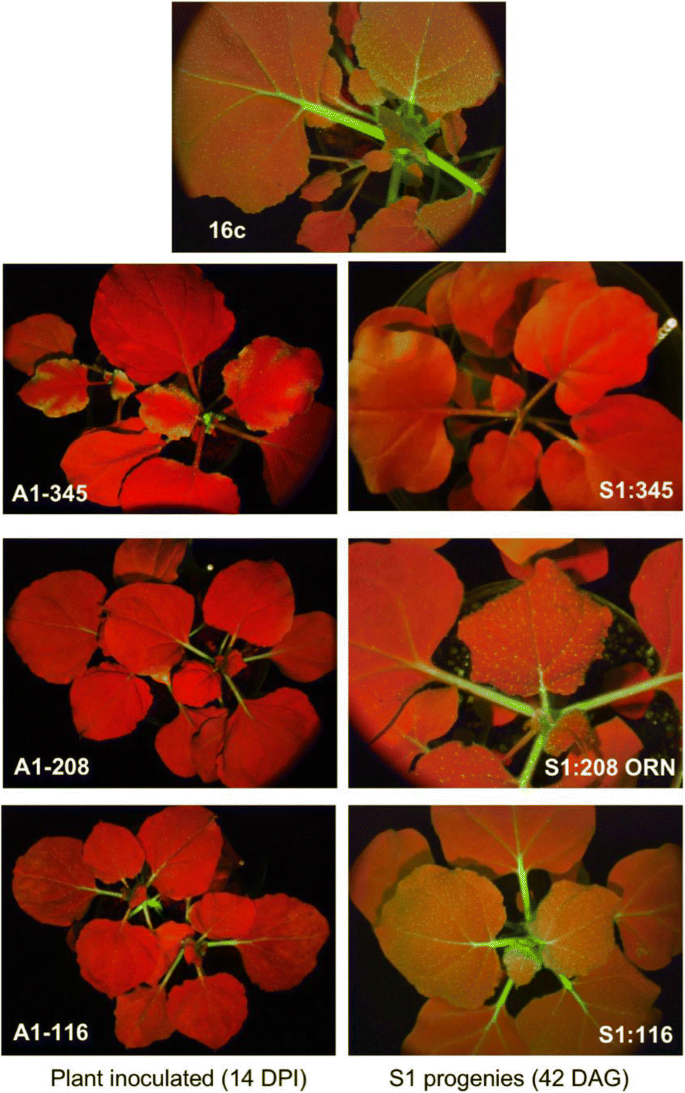
我们之前在gfp表达中诱导35S启动子的TGS底栖动物CMV-A1载体介导的植物(16c)1一种)。在这项研究中,我们进一步从机械的角度来看将这个VITGS。取决于集成到病毒载体的35S启动子序列的大小,发现不同程度的TGS诱导。当对应于几乎整个的35S启动子的345-nt的片段克隆,该病毒感染的植物开发高效VITGS,这是很好保持在第一自体受精(S1)后代植物(S1:345),该发荧光在UV光下(图红色。1乐队2).因为GFP表达大大降低了红色必定是由于叶绿素的自发荧光。在另一方面,116-NT片段可以诱导TGS在接种后的植株,在微弱的绿色荧光仍然留在叶脉,但未能维持TGS在S1后代的植物(S1:116);S1:116植株的GFP荧光恢复到16c植株的水平,叶片呈现黄色表型(fig .;1乐队2).对于208-nt片段,我们在a1 - 208接种植株中观察到VITGS诱导,但植株叶片叶柄中出现一定的GFP荧光。的年代1从A1-208-接种的植物的子代植物分成两种表型:赤型,其中丢失的GFP荧光(指定S1:208 RED)和橙色类型,这是S1之间的中间:345和16c(指定S1:208 ORN)(图.1乐队2).ORN植物似乎保留绿色荧光,这掩盖了红色自发荧光一定水平。在S的无1接种a1 - 208植株的后代植株的GFP荧光完全恢复。
CMV载体的构建和病毒感染植物及其后代的表型说明。一种CMV-A1vector的图构建。的recombinant CMV-A1 vectors contained the CaMV 35S promoter segments of three different sizes (positions relative to the transcription start site − 345 to + 1, − 208 to + 1 and − 116 to + 1). A1, B2 and B4 are sequence domains that affect promoter activity of the CaMV 35S sequences.B.病毒感染植物及其后代的示意图。烟草benthamiana用含有CaMV 35S启动子片段(A1-345、A1-208或A1-116)的CMV-A1载体接种16c植株,然后用含有CaMV 35S启动子片段(A1-345、A1-208或A1-116)的CMV-A1载体接种16c植株1)第二(S)2)获得自交后代。括号内表示表型分离。红色是由于TGS失去了GFP荧光而导致的叶绿素自身荧光的外观,黄色则是叶绿素自身荧光和GFP荧光的组合表达
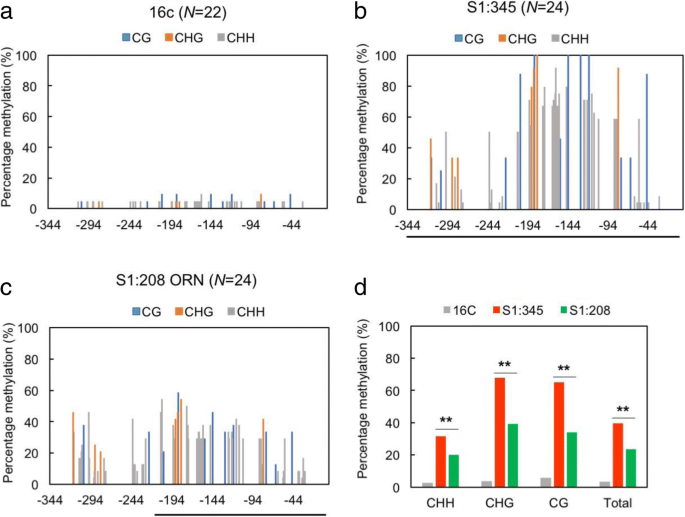
由于VITGS基本上通过RDDM操作,因此我们在S1:345,S1:208和16C植物中研究了35s启动子上的甲基化状态。我们的亚硫酸氢盐测序分析显示,S1:345的整个35s启动子的甲基化水平显着高于S1:208 ORN,16C中的35s启动子几乎没有甲基化(图。3.).
VITGS植物第二代自交后代35S启动子甲基化分析
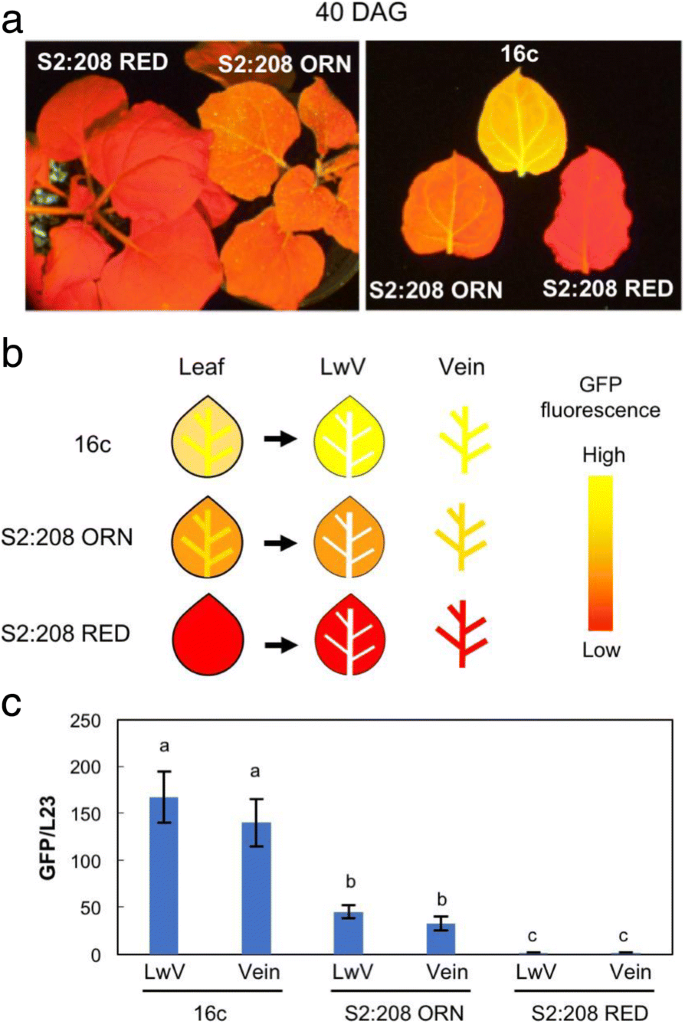
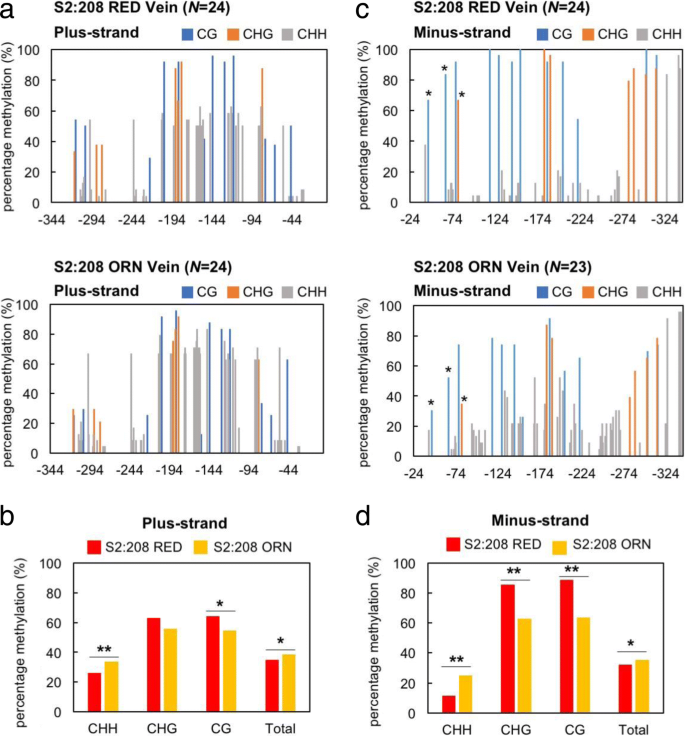
当我们生产的小号2通过S1:208 ORN植株的第二次自花受精,GFP荧光表型再次分化为RED和ORN植株:S2:208 RED和S2:208 ORN (fig . 20, fig . 20)。1乐队4.a).当S1:208 RED植株自交时,我们在S中只观察到RED表型植株2后代。这些结果提出的问题:是否稳定,继承TGS取决于甲基化水平?什么是稳定的TGS门槛甲基化水平?然而,因为它往往是那几个甲基化的胞嘧啶残基能抑制启动子活性的情况下,稳定的TGS可能不一定仅由甲基化差异解释。为了找到更多的线索,我们仔细研究了中间TGS在S2:208个后代。
在年代2后代,我们发现,在叶脉(静脉)发荧光比其他部分更亮黄色(叶无静脉,LWV)(图4.a和b)。我们分离了静脉和LwV,并比较了绿色荧光蛋白通过qRT-PCR检测这些组织的表达水平(图。4.C)。当我们从GFP荧光强度预计,绿色荧光蛋白在S2:208 ORN中表达约为16c的1/3,而在S2:208 RED中几乎未检测到。另一方面,不像我们的期望绿色荧光蛋白在静脉和LwV之间差异不大。
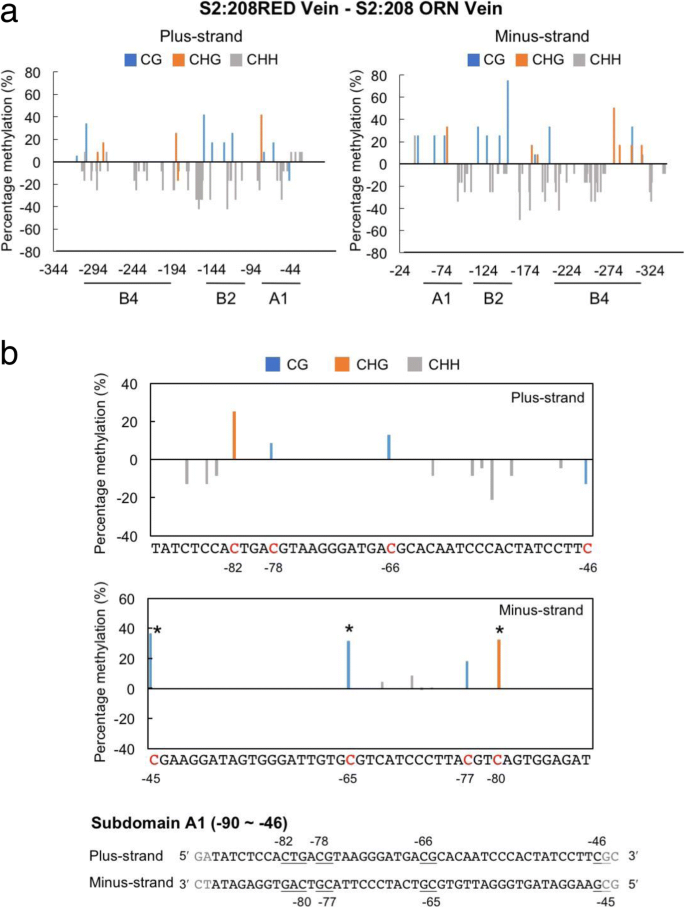
接下来,我们使用亚硫酸氢盐测序分析,以寻找S2:208 RED和ORN之间35S启动子甲基化模式的差异。如附加文件所示1:图S1,16C植物中的35s启动子中少量甲基化,在静脉和LWV组织之间的甲基化状态下没有观察到大的差异。在S2:208红色和S2:208 ORN中,35s启动子的正面甲基化的总体DNA甲基化如图2所示。5.静脉,并在附加文件2图S2为LwV组织。在s2: 208orn中,静脉组织的总甲基化水平较高(图2)。5.b)中,虽然在LWV组织中总的甲基化是在S2更高:208 RED(附加文件2:图开通)。另一方面,我们注意到不对称甲基化(CHH甲基化)显著降低,但对称CG甲基化在S2:208 RED静脉高于S2:208 ORN静脉(图2)。5.b).由于一条链上的不对称甲基化不会在其互补链的相应位置发生,也不会被后代遗传,我们进一步分析了互补负链上的甲基化状态。35S启动子负链的DNA甲基化如图所示。5.c代表静脉,在附加文件中2:图S2c为LwV。当对负链甲基化状态进行统计分析时,在Vein和LwV组织中,S2:208 RED的对称(CG和CHG)甲基化显著高于S2:208 ORN(图2)。5.d和附加文件2:图S2d)。此外,与S2:208 ORN相比,S2:208 RED的CHH甲基化整体显著降低。因此,特定位点的对称甲基化和CHH甲基化的降低可能与VITGS子代植物稳定、遗传的TGS相关。为了更容易发现RED和ORN之间的甲基化频率的差异,我们从S2:208 RED的值中减去S2:208 ORN的值(图2)。6.和额外的文件3.:图S3)。在A1亚结构域,参与对称甲基化(−45、−65、−77和−80)的胞嘧啶残基的甲基化水平在S2:208 RED中显著高于S2:208 ORN(图2)。6.b和附加文件3.:图S3b)。在S2:208 RED中,特定胞嘧啶位置(−82)上的对称甲基化倾向于高于S2:208 ORN,尽管统计上不支持。这些结果表明,对于稳定的TGS,负链的甲基化比正链的甲基化更重要。
S2:208 RED和S2:208 ORN静脉组织35S启动子胞嘧啶甲基化频率。一种S2:208 RED和S2:208 ORN之间35S启动子正义甲基化状态的比较。B.结果汇总一种以显示CHH、CHG、CG和总甲基化的差异。C比较S2:208 RED与ORN的35S启动子负序甲基化情况。星号表示RED和ORN之间的甲基化频率有显著差异的胞嘧啶残基,见图。6.湾D.结果汇总C以显示CHH、CHG、CG和总甲基化的差异。N是用于亚硫酸氢盐测序的克隆数。这个X-轴表示相对于转录起始位点(+ 1)的位置。(中的星号B.) 和 (D.)通过双尾Fisher精确检验表明甲基化频率具有统计学意义(*P.< 0.05, ** p < 0.01)
静脉组织中特定胞嘧啶甲基化在S2:208 RED和S2:208 ORN之间的差异。一种差异甲基化率在整体加功能和S2 35S启动子的负链:208 RED和S2:208 ORN。为了更容易找到甲基化率在每个链的任何差异,价值观S2的:208 RED静脉(图5.(a、c,上图)从S2:208 ORN静脉(图2)中减去。5.a和c,下面的图)。B.特写的子域A1在一种.的X-轴是A1子结构域的核苷酸序列。A1子结构域正负链的核苷酸序列如下图所示。甲基化的CG和CHG位点下划线。(中的星号B.)通过双尾Fisher精确检验(* .)表明,S2:208 RED与S2:208 ORN之间有统计学意义P. < 0.05)
在与VITGS实验的同时,我们正在创造转基因拟南芥筛选显示TGS的植物绿色荧光蛋白35S启动子下的基因;我们获得了含有35S启动子直接重复序列的转基因株系绿色荧光蛋白基因。在后代世代(T1T5.),我们注意到两种类型的GFP荧光:一种在早期表现出GFP荧光,然后发展了转录后基因沉默(PTGS)绿色荧光蛋白表达并最终在晚期诱导Tgs(PTGS-TGS植物);另一个已经在小幼苗(TGS植物)中具有完美的TGS,并且TGS通过下一代稳定地继承。从亚硫酸氢盐测序的结果分析这些植物中35s启动子序列的甲基化状态,我们发现在TGS植物中,亚域A1中的位置-46,-66,-78和-82处的Cg或Chg甲基化率高比在PTGS-TGS工厂中(附加文件4.:图S4)。此外,与PTGS-TGS植物相比,TGS植物的CHH甲基化整体水平大大降低。因此,这些观察结果与VITGS的结果非常吻合。
包含亚结构域A1的105 bp序列负链中的几个胞嘧啶残基的CG甲基化导致启动子活性显著降低
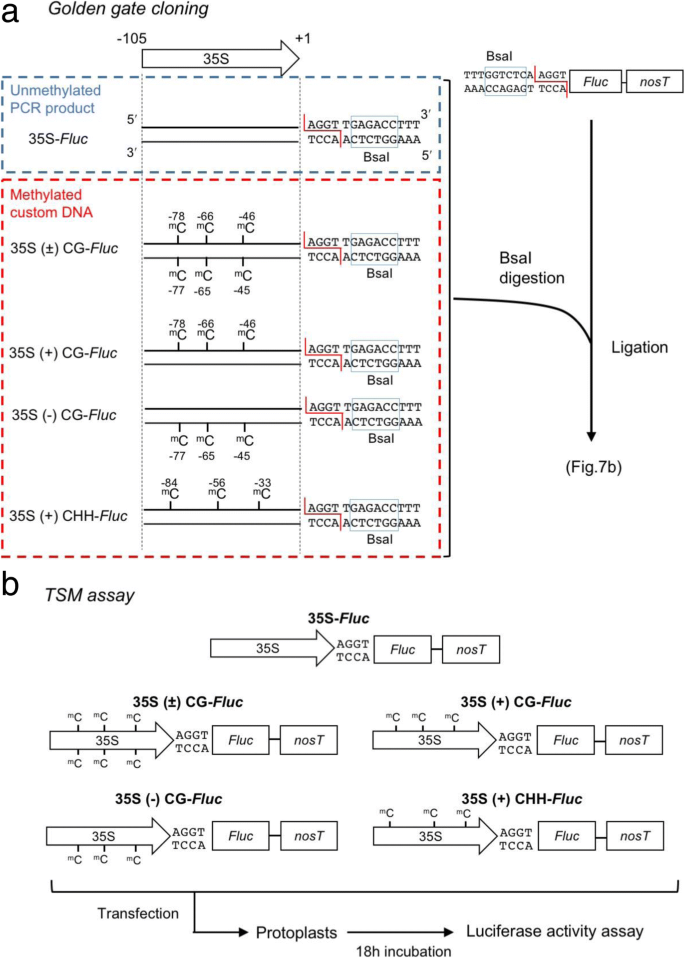
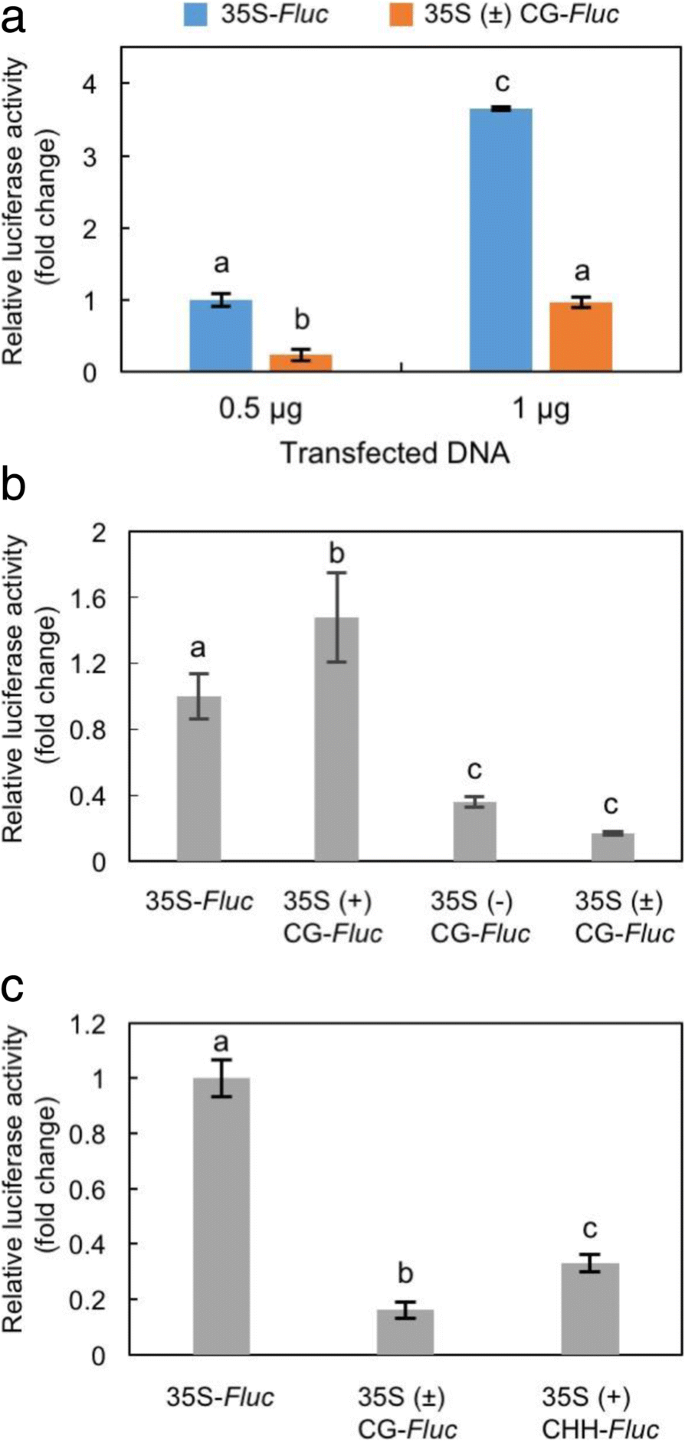
要验证的特定CG甲基化确实可以危及35S启动子下的基因表达,我们开发了一种新方法,其中,我们使用通过在金门杂交与甲基化的胞嘧啶残基的两个化学合成的寡核苷酸进行的105-bp的35S启动子序列克隆策略(图7.一种)。为了评价合成启动子的诱导下游基因表达的能力,我们把它连接到报告基因萤火虫荧光素酶(Fluc的),和Fluc的活性在该连接产物转染后的原生质体测定(图7.b)如图所示。8.A,当在胞嘧啶位置 - 45,46,65,66,77和-78处甲基化两者甲基化时,血管活性大大减少了。然而,当两种或减去股线时甲基化,我们发现在正链上的甲基化勉强抑制了琥珀瓣活动,有时甚至增加它。相比之下,减去股线的胞嘧啶甲基化大大减少了对照的1/3,35s-Fluc(无花果。8.b).有趣的是,当我们引入两股甲基化DNA时,Fluc至少被抑制到与负链相似的水平,或者似乎在降低启动子活性方面更有效,尽管差异没有统计学意义(图。8.b)。
在A1亚结构域的一些胞嘧啶残基上的CHH甲基化也有助于降低启动子活性
因为在CHH环境下,只有三个胞嘧啶残基(−33、−56和−84)在35S启动子的A域(−90 ~ + 1)被高度甲基化(图。5.一个额外的文件2图S2a),我们整合了这三个CHH位置的甲基胞嘧啶残基;两个残基位于子域A1,一个残基位于子域mp(范围从−45到+ 1)。如图所示。8.C,所引入的甲基化CHH减少荧光素酶活性下降到控制虽然所测试的特定CG甲基化两次更有效的(控制的1/6)的1/3。
讨论
不稳定TGS表型从VITGS-16C植物的后代
在我们对VITGS引起的35S启动子RdDM的分析,我们发现反对的TGS模式的离散表型绿色荧光蛋白35S启动子下游的基因。TGS似乎取决于插入病毒载体的35S启动子序列的大小。接种A1–345的16c植株中的TGS保持在S中1后代,而我们在S观察到的两种表型(RED和ORN)1接种后A1-208后代植株。红,叶绿素自发荧光,表示强烈绿色荧光蛋白而ORN似乎是介于16c和红色植物之间的中间TGS1来自植物的后代接种A1-116,大部分S1植物失去了TGS绿色荧光蛋白在接种病毒的植物中观察到的基因。基于这些观察,我们认为通过分析这些表型中的35S启动子,我们可能能够揭示一些重要的因素,控制rddm介导的TGS的稳健性。
然后,我们详细分析了S2:208植物,以获得关于为下一代固定稳定TGS的线索。如图所示。4.结果表明,S2:208 ORN植株茎和叶脉(Vein)的GFP荧光明显强于无脉叶区(LwV)。的绿色荧光蛋白S2:208 ORN植株的mRNA水平显著高于S2:208 RED植株。虽然我们最初期望绿色荧光蛋白Vein的水平会高于LwV,绿色荧光蛋白叶脉组织和叶脉组织的表达差异不大,可能是由于叶脉组织的叶绿素含量低于叶脉组织,且叶绿素自身荧光对GFP荧光的干扰较小。
胞嘧啶甲基的效果在35S启动子中一个TSM测定对下游基因的表达
在这里,我们成功地开发的测定法,使我们可以分析在下游基因表达的启动子序列特异性胞嘧啶甲基的效果。此外,我们甚至可以判别DNA链义当特定的甲基化被引入到靶启动子。在这里,我们把这种方法链的感测特定的甲基化(三条S甲基化,TSM)分析(图7.).此前,对TGS重要的启动子序列主要是通过点突变和亚结构域缺失策略确定的[40那41.那42.那43.那44.那45.那46.].然而,如果没有引入实际甲基化胞嘧啶具体残留,我们永远不能断定预测网站的甲基化是对下游基因表达的影响真正负责任。因为我们可以直接与一个TSM测定的目标启动子操纵胞嘧啶甲基化,不同的启动子序列可被分析以阐明特定胞嘧啶甲基化和启动子的活性之间的联系。我们的TSM测定的结果表明,在35S启动子的TGS水平可能取决于胞嘧啶甲基化和加锁存和对应于目标结构域序列负链之间的异质性的差异调节。在位置特别是胞嘧啶甲基贴近1element (− 83 to − 63) is very important for stable, inherited TGS, which agrees well with the previously predicted cytosine residues for the promoter activity [40那43.那45.].更具体地说,我们发现35S启动子的TGS在负链上的胞嘧啶残基(−78、−66和−46)被甲基化时有效地发生。另一方面,令人惊讶的是,正链上相应位点的甲基化对TGS的影响很小,但当两条链都在特定的胞嘧啶残基上甲基化时,TGS达到最大值。我们的TSM检测也表明,A1亚域的CHH甲基化显著降低了启动子的活性。然而,考虑到CHH甲基化在下一代中并没有维持,RED表型中稳定的遗传TGS将主要由一些特定的对称甲基化驱动。之前没有报道讨论过DNA甲基化在正负DNA链中及之间的异质性对TGS的影响。尽管我们一直假设在RdDM模型中,两条链会同时和均匀地甲基化[12,我们更应该考虑到这两条链在自然界中不一定是一致的甲基化。我们对35S启动子的观察是否可以普遍应用于其他启动子,应单独使用类似的检测方法进行检测。虽然在本研究中,我们只关注35S启动子中A1、B2和B4亚域的甲基化也被证明是启动子活性的关键[46.].利用TSM分析方法对35S启动子中其他元素的胞嘧啶甲基化进行评价是值得的。
遗传DNA甲基化中对称和不对称甲基化之间的联系
在我们的亚硫酸氢盐测序分析中,我们注意到一个有趣的现象,它总是与许多重复中的稳定遗传TGS相关。在S2:208中的红色植物中,我们发现在正链和负链中不对称(CHH)甲基化的总体水平显著降低,而对称甲基化的水平显著降低(CG和CHG)相反增加(图。5.和额外的文件2:图S2)。大部分的s1由a1 - 116接种植株产生的植株失去了TGS绿色荧光蛋白基因。下一代中的这种不稳定的Tgs可以与对称甲基化的降低和靶序列中的不对称甲基化的增加相关。对于TGS诱导的后代植物中的35S启动子的胞嘧啶甲基化(S.1到S4.),对ALSV载体的研究也显示了类似的观察结果[24.]为了了解甲基化状态在TGS诱导中的作用,我们可以通过TSM分析等方法测试35S启动子中的不对称甲基化水平如何影响TGS。尽管目前我们不知道不对称甲基化减少的确切原因,但可以想象生殖过程中的某些因素可能会影响RdDM与遗传性甲基化之间的关联。综合考虑这些结果,我们推断在VITGS子代植物中观察到的红色和ORN植物的产生可能取决于某些特定胞嘧啶位点上对称甲基化的积累水平以及子代植物中不对称甲基化的减少。
结论
我们诱导了TGS绿色荧光蛋白35S启动子下的基因通过RdDM通过一个含有不同大小的启动子序列的病毒载体。在自交的第一代中,稳定的TGS似乎与启动子上的高水平甲基化相关。然而,我们在第二代中观察到不稳定的TGS;年代2后代被分离为RED和ORN。我们的亚硫酸氢盐测序分析表明,在A1亚结构域的一些胞嘧啶残基上的特异性甲基化和不对称甲基化频率的降低可能是通过RdDM有效诱导TGS的重要因素。为了探究甲基化在特定胞嘧啶残基上的重要性,我们开发了一种方法,通过这种方法,我们可以以链特异性的方式分析甲基化胞嘧啶残基对TGS的直接影响。在本实验中,我们发现在正负感启动子序列上的胞嘧啶甲基化可以差异驱动启动子下游的基因表达。因此,我们推断,强大的TGS可能是由DNA甲基化在正链和负链之间的特定位点的异质性决定的。
方法
植物材料
烟草benthamiana从David Baulcombe博士获得第16C行。在24℃下使用16-H光(8-H暗)生长植物。
接种病毒和GFP观察
通过PCR扩增来自35S启动子的片段并插入CMV-A1载体中。底栖动物如Otagaki等人所述,16c系植物用CMV的体外转录本接种[36.].接种15 d后从感染组织中分离总RNA。使用2b-5up和R2-2814-R2引物对病毒cdna进行pcr扩增(附加文件)5.:表S1)和TaKaRa One Step RNA PCR Kit AMV (TaKaRa)试剂盒根据供应商的说明。为了证实病毒在复制过程中没有发生严重的突变,对扩增后的病毒cdna进行了直接测序:测序色谱图见附加文件6.:图S5。用带GFP滤光片(OP-42313)的Keyence显微镜VB-7000检测GFP荧光。
DNA甲基化测定
根据供应商的说明,使用Illusta DNA提取试剂盒PhytoPure(GE Healthcare)提取DNA。根据供应商的协议,使用EZ DNA甲基化闪电试剂盒(Zymo Research)对DNA进行亚硫酸氢盐处理。为了扩增目标序列,引物对35S-346F-bisuT/35S + 1A bisuA用于正链,引物对35S(−)-5-BS/35S(−)-3-BS用于负链(附加文件5.:表S1)。引物最初由程序MethPrimer (http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi).放大整个(~ 345 (bp)35S启动子的核心序列,我们对设计的引物进行修饰,使其含有尽可能少的胞嘧啶残基,并合成简并引物 94个周期 摄氏30度 s、 五十五 摄氏30度 s和72 摄氏30度 s、 然后使用Dyna Express TA PCR克隆试剂盒(Bio Dynamics Laboratory)将PCR产物克隆到pTAC1载体中。对于基于克隆的亚硫酸氢盐测序,我们使用22-24个克隆,测序数据使用MEGA版本6进行比对[47.].这些图形是在Microsoft Excel中创建的(附加文件7.:表S2)。
定量RT-PCR分析
按照供应商的说明书使用TRIzol试剂(Invitrogen)分离总RNA。对于S2子代的DNA和RNA提取,我们分离了I类和II类静脉(Vein) [48.]和叶组织在没有那些静脉(LWV)。Extracted RNA was treated by DNase I recombinant RNase-free (Roche Applied Science), and then cDNA was synthesized from 0.2 μg of RNA using the PrimeScript RT reagent Kit (TaKaRa) according to the supplier’s instruction. qRT-PCR analysis was carried out by the Applied Biosystems StepOnePlus Real-time PCR system using the PowerUp SYBR Green Master Mix (Applied Biosystems). The PCR cycling conditions were 40 cycles of 95 °C for 15 s, 55 °C for 30 s and 72 °C for 30 s. The绿色荧光蛋白使用比较CT方法计算表达水平并通过L23基因的表达归一化。使用引物对MgFP-5-160 / MGFP3-160进行靶序列进行扩增绿色荧光蛋白用于内控的基因和引物对Nb-L23–5-110/Nb-L23–3-110。
金门克隆和TSM测定
本研究中使用的所有寡核苷酸均在附加文件中列出5.S1:表。通过PCR从pBI221 (Clontech)中扩增出未甲基化的35S启动子片段。利用引物对35S-GGC-5/35S-GGC-3扩增目标序列。采用引物对LUC-GGC-5/LUC-GGC-3进行PCR扩增。自定义合成的在三个CG位点(−78,−66和−46)和三个CHH位点(−33,−56和−84)有特定甲基化的寡核苷酸(oligo DNA)由自定义DNA合成服务(北海道系统科学)制备。寡聚dna (100 μM)在寡聚退火缓冲液(10 mM Tris-HCl, 1 mM EDTA, 100 mM NaCl)中退火,95℃孵育4分钟后逐渐冷却。为了使DNA片段中的任何一条链都被甲基化,35S-CG-DNA (+), 35S-CG-DNA(-)和35S-CHH-DNA(+)被未甲基化的35s -3- 120pm或35s -5- 120pm的寡核苷酸退火,然后通过PCR扩增。PCR循环条件为94℃30 s、55℃30 s、72℃30 s共40个循环。所有四种DNA结构首先被BsaI消化,然后连接到萤火虫荧光素酶报告基因(Fluc)基本基本上被描述为Boldler等人的金门克隆。[49.].然后我们分析了原生质体中启动子的活性。我们称这种方法为链感特异性甲基化(triple S甲基化,TSM)分析。
原生质体转染和荧光素酶检测
原生质体由叶子制备底栖动物如Shimura等人所述[50].制备的flc -融合35S启动子片段(0.5 μg或1 μg)与pE-Rluc (0.45 μg)在聚乙二醇存在下直接转染原生质体。孵育18小时后,使用双荧光素酶报告基因分析系统(Promega)和荧光仪(Wallac 1420 ARVO MX)测定荧光素酶活性(Fluc和Rluc),如Shimura等人之前所描述的[50].
缩写
- 前:
-
Argonaute.
- ALSV:
-
苹果潜伏球形病毒
- CMT2:
-
CHROMOMETHYLASE2
- CMT3:
-
CHROMOMETHYLASE3
- CMV:
-
黄瓜花叶病毒
- DCL3:
-
DICER-LIKE 3
- DRM2:
-
域重新安排METHYLTRANSFERASE2
- 极:
-
双链RNA
- GFP:
-
绿色荧光蛋白
- MET1:
-
METHYLTRANSFERASE1
- NRPE1:
-
核RNA聚合酶E1
- PTGS:
-
转录后基因沉默
- RdDM:
-
RNA指导的DNA甲基化
- RDR2:
-
依赖RNA的RNA聚合酶2
- siRNA的:
-
短干扰RNA
- TGS:
-
转录后基因沉默
- VITGS:
-
病毒诱导的转录基因沉默
参考
- 1.
Baulcombe D.植物中的RNA沉默。自然。2004; 431:356-63。
- 2。
DNA甲基化与表观遗传学。植物学报2004;55:41-68。
- 3。
陈世伟,亨德森国际,雅各布森东南。基因组园艺:拟南芥DNA甲基化。纳特·杰内牧师。2005;6:351–60.
- 4.
Lucy AP,郭海生,李文霞,丁世文。定位于细胞核的植物病毒蛋白对转录后基因沉默的抑制。EMBO j . 2000; 19:1672 - 80。
- 5.
张Y,哈里斯CJ,刘谦,刘W,澳星我,大Y,小L,冯L,陈X,谢Y,陈X,詹L,冯S,李JJ,王H,翟Ĵ,雅各布森SE.大规模比较表观基因组学揭示了拟南芥非CG甲基化调控层次。国家科学院院刊美A. 2018; 115:E1069-74。
- 6.
曹X,雅各布森SE。拟南芥DRM甲基转移酶在从头DNA甲基化和基因沉默的作用。Curr Biol。2002; 12:1138年至1144年。
- 7.
曹X, Aufsatz W, Zilberman D, Mette MF, Huang MS, Matzke M, Jacobsen SE。DRM和CMT3甲基转移酶在rna导向DNA甲基化中的作用。咕咕叫医学杂志。2003;13:2212-7。
- 8.
Haag JR, Pikaard CS。多亚基RNA聚合酶IV和V:植物基因沉默的非编码RNA的提供者。细胞生物学杂志。2011;12:483-92。
- 9。
法JA,雅各布森SE。建立,维持和修饰植物和动物的DNA甲基化模式。NAT Rev Genet。2010; 11:204-20。
- 10
Matzke M, Aufsatz W, Kanno T, Daxinger L, Papp I, Mette MF。rna介导的转录基因沉默的遗传分析。生物化学学报。2004;37(4):449 - 454。
- 11
Matzke男,菅T,Daxinger L,胡特尔B,Matzke AJ。RNA介导的染色质基于沉默植物。CurrOgin Cell Biol。2009年; 21:367-76。
- 12.
Matzke MA, Kanno T, Matzke AJ。rna导向的DNA甲基化:开花植物复杂表观遗传途径的进化。植物学报2015;66:243-67。
- 13。
Mette MF, Aufsatz W, van der Winden J, Matzke M, Matzke AJM。双链RNA引发的转录沉默和启动子甲基化。EMBO j . 2000; 19:5194 - 201。
- 14。
Pikaard CS,Haag Jr,Pontes OM,Blevins T,Cockinl R. POL IV和POL V依赖性RNA定向DNA甲基化的转录叉模型。冷泉Harb Symp量子Biol。2012年; 77:205-12。
- 15.
Pontes O, Li CF, Costa Nunes P, Haag J, Ream T, Vitins A, Jacobsen SE, Pikaard CS。拟南芥染色质修饰核siRNA途径涉及核仁RNA加工中心。细胞。2006;126:79 - 92。
- 16.
植物的DNA甲基化:与小rna和组蛋白修饰的关系以及转座子失活的功能。acta botanica yunnanica(云南植物研究). 2012;53:766-84。
- 17.
Sijen T,Vijn I,Rebocho A,面包车Blokland R,罗洛夫斯D.转录和转录后基因沉默机制上是相关的。Curr Biol。2001; 11:436-40。
- 18.
张H,朱JK。RNA指导的DNA甲基化。CurrOp植物BIOL。2011; 14:142-7。
- 19.
Cuerda Gil D,Slotkin RK.非规范RNA指导的DNA甲基化。Nat植物。2016;2:16163。
- 20.
邓S,Chua NH。倒置重复RNA靶向FT内肾区促进拟南芥中的FT抑制。植物细胞生理。2015; 56:1667-78。
- 21.
Kinoshita Y,Saze H,Kinoshita T,Miura A,Soppe WJ,Koornneef M,Kakutani T.控制小鼠FWA基因沉默拟南芥与正弦相关的直接重复。植物j . 2007; 49:38-45。
- 22
三木d,岛本K.从头通过siRNA诱导的DNA甲基化靶向内源性转录序列是基因特异性和OsMet1无关的水稻。植物杂志2008; 56:539-49。
- 23
伯奇 - 史密斯TM,安德森JC,马丁GB,迪内希 - 库马尔SP。病毒诱导的基因沉默的应用和优势在植物基因功能研究。植物杂志2004; 39:734-46。
- 24。
关键词:苹果病毒,基因沉默,启动子序列,甲基化Microbiol前面。2014;5:595。
- 25。
陈志强,陈志强,陈志强,等。苹果、梨和日本梨病毒诱导基因沉默的研究进展苹果潜伏球形病毒向量。工厂方法。2011;15。
- 26。
在大豆种子山岸N,吉川N.病毒诱导的基因沉默和大豆植物与出现阶段苹果潜伏球形病毒载体。植物分子生物学。2009;71:15–24。
- 27。
徐H,徐L,杨p,曹y,唐y,他g,元s,明j.烟草慢性病毒诱导的植物去饱和酶(pds)和mg-chelatase h亚基(chlh)基因沉默茄属植物pseudocapsicumL. J.同行2018; 6:e4424。
- 28.
Jones L, Ratcliff F, Baulcombe D.在植物中,RNA导向的转录基因沉默可以独立于RNA触发而遗传,并且需要Met1来维持。咕咕叫医学杂志。2001;11:747-57。
- 29.
Holzberg S, Brosio P, Gross C, Pogue GP。大麦条纹花叶病毒-诱导的单子叶植物的基因沉默。植物j . 2002; 30:315-27。
- 30.
Faivre-横行O,Gilroy的EM,Hrubikova K,海因I,Millam S,Loake GJ,桦木P,泰勒男,Lacomme C.马铃薯病毒X.-诱导的马铃薯叶片和块茎基因沉默。植物杂志。2004;134:1308-16。
- 31.
琼斯L,汉密尔顿A,Voinnet O,托马斯C,莫尔A,Baulcombe D. RNA-DNA相互作用和DNA甲基化在转录后基因沉默。植物细胞。1999; 11:2291-301。
- 32.
Goto K, Kobori T, Kosaka Y, Natsuaki T, Masuta C.基于小rna结合能力的黄瓜花叶病毒沉默抑制基因2b的特性研究。植物生理学报。2007;48:1050-60。
- 33.
关键词:病毒,表观遗传修饰,植物,表型植物j . 2011; 65:156 - 68。
- 34
Nagamatsu A,Masuta C,Matsuura H,Kitamura K,Abe J,Kanazawa A.通过病毒诱导的基因沉默下调大豆类黄酮3′-羟化酶基因表达,揭示了与青春期色素沉着相关的阈值mRNA水平的存在。《植物生理学》2009;166:32–9。
- 35
Nagamatsu A, Masuta C, Senda M, Matsuura H, Kasai A, Hong JS。病毒诱导基因沉默介导大豆类黄酮合成相关基因功能分析生物技术学报2007;5:778-90。
- 36。
Otagaki S,Arai M,Takahashi A,Goto K,Hong JS,Masuta C.使用一种新的基因沉默技术快速诱导转录和转录后基因沉默黄瓜花叶病毒向量。生物科技植物》。2006;23:259 - 65。
- 37。
田崎K,寺田H,Masuta C,山岸M.病毒诱导的基因沉默(VIGS)的百合leichtlinii使用黄瓜花叶病毒向量。生物科技植物》。2016;33:373 - 81。
- 38。
金泽A,稻叶Ĵ,笠井男,志村H,Masuta C. RNA介导的由病毒载体靶向的内源基因的外遗传修饰:一种有效的基因沉默系统产生不携带转基因,但具有改变的性状的植物。植物西格Behav。2011; 8:1090-3。
- 39。
启动子RNA片段的大小和位置对病毒诱导的DNA甲基化和转录基因沉默的影响。表观遗传学。2011;6:681 - 91。
- 40.
Benfey PN, Chua NH。花椰菜花叶病毒35S启动子:植物转录的组合调控。科学。1990;250:959 - 66。
- 41.
任磊,蔡宁华。CaMV 35S增强子域的组合与协同特性。EMBO j . 1990; 9:1685 - 96。
- 42.
林E,Benfey PN,吉尔马丁下午,方RX,蔡NH。位点特异性突变改变在转基因植物中体外因子结合和变化的启动子的表达模式。国家科学院院刊美A. 1989; 86:7890-4。
- 43.
林娥,蔡新。ASF-2:结合花椰菜花叶病毒35S启动子和驾驶室启动子中保守的GATA基元的因子。植物细胞。1989;1:1147-56。
- 44.
方瑞x,张志强,蔡恩华。花椰菜花叶病毒35S启动子在转基因植物中最大表达的多个顺式调控元件植物细胞。1989;1:141-50。
- 45.
Kanazawa A, O ' Dell M, Hellens RP。核因子与CaMV 35S启动子as-1元件的结合在体外受胞嘧啶甲基化的影响。植物医学杂志。2007;9:435-41。
- 46
Bhullar S, Datta S, Advani S, Chakravarthy S, Gautam T, Pental D, Burma PK.花椰菜花叶病毒35S启动子功能分析:B5, B4和B2亚域在启动子活性中的作用。生物技术学报2007;5:696-708。
- 47
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物学杂志。2013;30:2725-9。
- 48
Mekuria T,Bamunusinghe D,Payton M,Verchot-Lubicz J.Ploem卸载马铃薯病毒X运动蛋白受病毒和宿主因子的调节。Mol植物微生物相互作用。2008; 8:1106-17。
- 49。
一罐一步精确克隆高产能的方法。《公共科学图书馆•综合》。2008;11:e3647。
- 50.
志村H,暮Y,后藤K,RNA沉默,并且病毒抑制的能力的Masuta C.度取决于在原生质体系统中的细胞种类。Ĵ根植物病理学。2008; 74:326-30。
作者信息
从属关系
贡献
CM和TM设计并协调了这项研究。HS指导原生质体实验。TI、SS和RI设计并进行了转基因实验拟南芥植物。WM进行了亚硫酸氢盐测序分析。稿件主要由WM和CM撰写。所有作者都对结果分析、手稿修订和最终批准发表做出了贡献。
相应的作者
伦理宣言
伦理批准和同意参加
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
图S1。在16C植物的35S启动子的正链甲基化状态。在静脉和LWV组织中的35S启动子的正链的甲基化频率,通过亚硫酸氢盐测序分析。N是用于测序的克隆的数量。的X-轴是相对于核苷酸距离转录起始位点的位置。(TIFF 2598 kb)
额外的文件2:
图S2。S2:208 RED和S2:208 ORN的LwV组织中35S启动子胞嘧啶甲基化频率。S2:208 RED和S2:208 ORN 35S启动子正位甲基化状态比较b Summary of the results in a to show CHH, CHG, CG and total methylationc S2:208 RED与ORN 35S启动子负位甲基化状态比较。星号表示RED和ORN之间的甲基化频率有显著差异的胞嘧啶残基,详见附加文件3.:图S3b。d总结c的结果显示CHH, CHG, CG和总甲基化的差异。N是用于亚硫酸氢盐测序的克隆数。这个X-轴表示相对于转录起始位点(+1)的位置。b和d中的星号表示双尾Fisher精确检验的甲基化频率具有统计学意义(*P.<0.05,**P.<0.01)。(TIFF 6706 KB)
额外的文件3:
图S3。S2:208 RED和S2:208 ORN LwV组织特异性胞嘧啶甲基化的差异。在S2:208 RED和S2:208 ORN中,35S启动子整体正链和负链的甲基化频率存在差异。为了更容易发现每条链中甲基化频率的任何差异,S2:208 RED LwV的值(附加文件2:图S2a和c,上图)从S2:208 ORN LwV(附加文件)中减去2:图S2a和c,下图)。b a中子域A1的特写X-轴是A1子结构域的核苷酸序列。A1子结构域正负链的核苷酸序列如下图所示。甲基化的CG和CHG位点下划线。B中的星号表示S2:208红色和S2:208 ORN之间的统计显着性通过双尾Fisher的确切测试(*P.< 0.05). (TIFF 7061 kb)
额外的文件4:
图S4。转基因植物35S启动子正链的甲基化状态a, b gfp沉默谱线甲基化频率。行2-6-1-7-6 (a)和2-6-1-7-2 (b)是T5.源于原始品系2-6-1-7的后代品系,该品系包含35S启动子的直接重复序列,随后是绿色荧光蛋白基因序列。绿色荧光蛋白2-6-1-7和2-6-1-7 -6的表达起初因PTGS (after -transcriptional gene silencing, PTGS)而降低,后来又因TGS而降低绿色荧光蛋白2-6-1-7-2的表达被TGS稳定抑制。12 ~ 15个克隆用于亚硫酸氢盐测序。c从b的值中减去a的值,表示2与6-1-7-2和2-6-1-7-6之间的甲基化频率差异。−82、−78、−66和−46表示位于A1亚域的特定胞嘧啶残基。的X-轴表示相对于转录起始位点(+ 1)的位置。子域A1,B2和B4的位置也指示。(TIFF 4447 kb)
额外的文件5:
表S1。本研究中使用的引物列表。(XLSX 10 KB)
附加文件6:
图S5。35S在来自受感染组织中分离的载体的病毒基因组中的启动子序列。A-C序列的插入件的色谱图,其包含345-,208-和116-BP部分(A至C)中的每一个。分离出15天的总RNA,直接测序RT-PCR扩增的片段。我们证实,集成到病毒载体中的原始序列在感染组织中没有变化。(TIFF 7535 KB)
权利和权限
开放访问本文采用知识共享署名4.0国际许可的条款分发(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
松永,W.,志村,H.,白川,S.。等等。35S驱动转基因的转录沉默取决于启动子在正负意义链上特定胞嘧啶上的甲基化异质性。BMC植物BIOL.19,24(2019)。https://doi.org/10.1186/s12870-019-1628-y
收到了:
公认:
发表:
关键词
- CaMV 35 s启动子
- 烟草benthamiana
- RNA指导的DNA甲基化
- VITGS