抽象的
背景
石龙科属于桃金娘目,属于archichlamydae的一部分。该科有31个属,包括大约620种草本植物、灌木和乔木。在这31个属中,5个大属各有35个或更多的种。他们是Lythrum.,35;Rotala, 45;Nesaea, 50;Lagerstroemia,56;和萼距花属植物,275种。
结果
我们报告了六种新序列的叶绿体(CP)基因组(Duabanga开大花的,Trapa•,Salicaria,Lawsonia inermis,Woodfordia后和Rotala rotundifolia),并与其他16种棘球蚴基因组进行比较。22种石藓科植物的cp基因组长度为152,049 bp ~ 160769 bp。每一种石藓科植物的cp基因组包含112个基因,包括78个蛋白质编码基因、4个核糖体rna和30个转移rna。此外,在6个分类中检测到211-332个简单重复序列(SSRs),在4个分类中检测到7-27个长重复序列。我们选择了十个不同的热点(ndhF, matK, ycf1, rpl22, rpl32, trnK-rps16, trnR-atpA, rpl32- trnl, trnH-psbA和trnG-trnR)在22种含量的物种中是潜在的分子标记。我们构建了来自42家Myrtales植物的系统发育树,用8个GerAniales植物作为群体。通过使用66蛋白编码基因构建的最大可能性(mL),最大判例(MP)和贝叶斯推理(BI)树,有效地区分了Myrtales物种之间的关系。一般来说,22种含量聚集成一个疏枝,这是姐妹患者到三种癌症种类。与Melastomataceae和Myrtaceae相比,Lytheraceae和Onagroceae在Myrtales后面有分化。
结论
该研究提供了十种潜在的分子标记作为候选DNA条形码,并在Myrtales内促进CP基因组资源进行进一步研究。
背景
Lythaceae属于Myrtales的命令,并以Genus命名Lythrum.[1].开花家族由五个亚属,Lythroideae,Punicoideae,Sonneratioideae,Duabangoideae和Trapoideae组成,具有31个属。亚家族旁观肌女性以前是Punicaceae的家庭,并且亚家族的Trapoideae以前是Trapaceae。属萼距花属植物,Lagerstroemia,Nesaea,Rotala,Lythrum.代表了Lythraceae中最大的类群。Lythraceae种类分布在世界各地,大多数分布在热带地区,一些分布在温带气候地区[2,3.,4,5,6,7].
大多数含量的睫状症物种是草药,而灌木或树木不太常见[8].Lythraceae与其他植物科的不同之处在于它们的花瓣,花瓣是在花蕾内皱折的,它们的种子有多层的外壳[2,3.].许多物种出现在水生或半水生生境中,例如滴点,Rotala,森和Trapa..家庭中的一些物种具有高经济价值,如石榴作为果树,Trapa•可食用的食物,海莫米娅myrtifolia作为一个重要的药用植物[9),Lawsonia inermis作为一种天然染料。总体而言,睫毛物种具有高经济和观赏价值,并广泛用于园艺[10.,11.].
过去对睫毛的研究集中在形态学上[2,12.],palynology [13.,14.]和解剖[15.].然而,这些研究没有区分睫毛内的内部关系。最近,为了深化我们对塞科西物种之间关系的理解,使用现代分支方法来初步估计睫毛物种内的系统发育[16.].基于rbcl.基因组数据,的PSAA-YCF3.cp基因组中的间隔体和它的核糖体DNA序列初步推断[17.].这两个非编码区域提高了物种间的分辨能力rbcl.分岔图(17.].然而,由于使用某些DNA片段,这些研究会导致结论不完全。完整的CP基因组将为睫毛内的关系重建提供更好的解决方案,并探讨其在Myrtales内的系统发育位置。
叶绿体是陆地植物必需的细胞器[18.],并且大多是遗传物品[19.].cp基因组通常由一个双链DNA分子组成,大多数cp基因组长度为120-220 kb,编码基因为120-140 [20.,21.].CP基因组通常具有四个部分:大单拷贝(LSC)区域,小单拷贝(SSC)区域,以及倒置重复区域(IRA和IRB)的两个副本。因为CP基因组比核和线粒体基因组更加保守,但长度较短,所以已经使用了一些CP基因组序列来区分物种和传导系统发育研究[22.,23.,24.,25.].由于完整的cp基因组序列提供了更好的数据来区分边缘类群,特别是物种水平以下的类群,近年来报道的cp基因组越来越多。
在这项研究中,我们报告了六种新测序的睫毛菌CP基因组,并将它们与睫毛内的其他16种物种的比较,包括九种已发表的CP基因组(P. GANATUM.,H. Myrtifolia.,Lagerstroemia fauriei.,Lagerstroemia多花植物,Lagerstroemia guilinensis,lagerstroemia indica.,lagerstroemia speciosa.,lagerstroemia subcostata.和Lagerstroemia媒介物)从Genbank和七个未发表的七LagerstroemiaCP基因组(Lagerstroemia excelsa,Lagerstroemia limii,Lagerstroemia摘要,Lagerstroemia siamica,lagerstroemia tomentosa,Lagerstroemia venusta和lagerstroemia calyculata.).我们的目标如下:(1)检测22种溶素物种的CP基因组之间的差异;(2)选择10个高度可变地区,以充当候选条形码,用于鉴定含有睫毛的相关物种;(3)重建系统发育关系以验证睫毛内的分支关系,探讨其在Myrtales中的地位。
结果
叶绿体基因组结构和含量
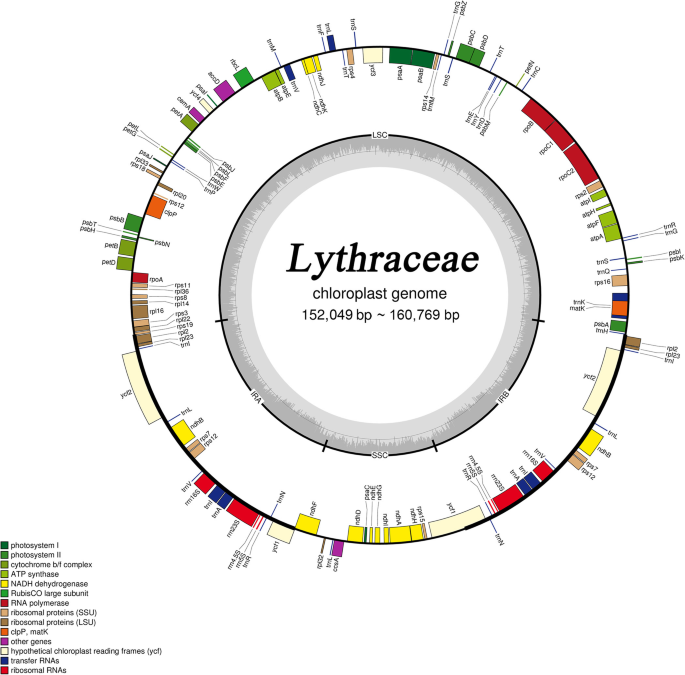
22种溶素物种的完整CP基因组长度为152,049 bp(l . subcostata)至160,769 bp(L. Villosa.)(表1).所有cp基因组都具有典型的四联体结构,包括由两个IR区分开的LSC和SSC区(图)。1).LSC范围为83,817 bp (l . guilinensis)至89,569 bp (W. Fruticosa.)占总长度的55.10-56.90%。SSC区域在16,501bp之间变化(D. Grandiflora.)和33,301 bp(L. Speciosa.)占总长度的10.60-21.80%。IR区范围从17,541 bp(L. Floribunda.)至26,906 bp (L. Villosa.),占总长度的11.50-17.00%。
22种石藓科植物的cp基因组共检测到112个独特基因,包括78个编码基因、30个tRNA基因和4个rRNA基因(见表)2).在22种睫状症物种中,蛋白质编码外显子的长度范围为73,401 bp(L. indica.)至81,047 bp (H. Myrtifolia.), rRNA范围为9022 bp (T. Natans.)至9068bp (L.fauriei),trna从2741 bp(l . guilinensis)至2913bp (L. Excelsa.),代际区域范围从44,031 bp(l . guilinensis)至51,367 bp (L. Villosa.),内含子区范围为14,786 bp (L. Calyculata.)至18099 bp (L. Villosa.).分别占总长度的37.00-38.00%、3.00-6.00%、1.80-1.90%、28.90-32.40%和9.70-11.30%3.).
在112个不同的基因中,共17个基因含有内含子。三个基因(RPS12.和YCF3.)包含两个内含子,类似于Melastomataceae cp基因组[26.].14个基因包含1个内含子,包括8个编码基因(RPS16,rpoC1,atpF,PETB.,佩德,ndhB,ndhA,rpl16.)和6个tRNA基因(trnk-uuu.,trnL-UAA,trnV-UAC,trnI-GAU,trna-ugc.,trnG-UCC).包含内含子的17个基因中,1个基因分布在SSC区域,3个基因分布在IR区域,13个基因分布在LSC区域(附加文件)1:表S1)。
密码子使用
共使用79个编码基因估算密码子使用频率。编码为25,068 (L. indica.)至27,111 (l . guilinensis)密码子。终止密码子是UGA,UAG和UAA。对于22种,GCU编码的丙氨酸具有最高的RSCU值,并且UAC编码的酪氨酸在约0.45的最低点。在22种含量中的大部分中,AAA编码的赖氨酸发生了最多的出现次数,在1000多个中。该结果也在CP基因组中报告H. Myrtifolia.,Aquilaria sinensis,epipremnum aureum和罂粟花rhoeas[9,27.,28.,29.].RSCU结果(表4,附加文件2表S2)显示,A或T在密码子第三位的核苷酸频率高于G或C。陆生物种的第三密码子位置往往倾向于A/T而非C/G, IR区域A/T的丰富度可能是主要原因[30.,31.].
22种石蒜科植物的基因组比较分析
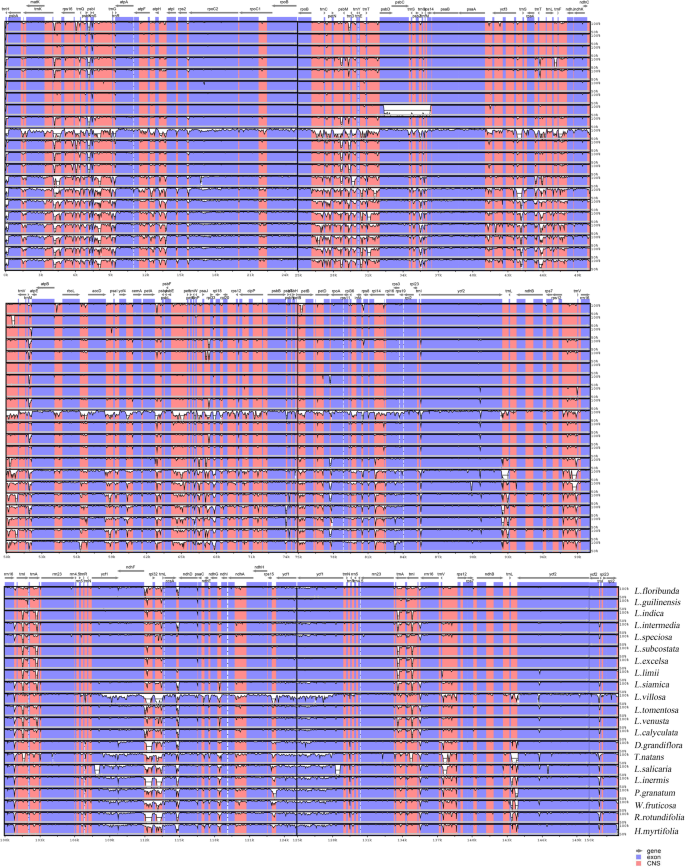
对L. Excelsa.作为参考,对22种石蒜科植物的cp基因组序列进行了MVISTA分析。将22个cp基因组进行配对比较后,我们发现序列之间的相似性相当高。从无花果。2,这很明显,14Lagerstroemia种从其他八种Lythraceae中分离出来。14个国家之间的分歧Lagerstroemia物种很低。LSC和SSC区变异程度大于IR区,非编码区分化程度大于编码区。一些区域包含更多的变异,例如ndhF,ndhH,mat,YCF2.,rpl22,accD,RPOB.,rbcl.,psbK在编码基因中和PSBM-TRND.,trnI-trnA,ndhf-rp132,RP132-TRNL.,ndhd-psac.,atpA-atpF,trnI-GAU内含子, trnK-rps16,trnh-psba.基因间区域(图。2).此前,这些地区也测量了类似的差异水平[32.,33.].
与22个cp基因组的LSC和SSC区域相比,IR区域在基因序列和数量上最为保守。然而,IR、LSC和SSC区域之间的联系也存在很大的差异。在比较的基因组中未检测到转位和易位。红外放大和收缩是造成这22个cp基因组大小差异的主要原因。
分析了22种含量物种的基因中存在进化率的显着差异。总体而言,大多数基因的平均Ka / ks小于0.5(92.21%)。17基因显示至少一个物种高于1的Ka / Ks。在17个基因中,七种基因(rbcl.,psbJ,rpl2.,rpl20.,rpl23,ccsA和YCF4.)呈现至少15种的高速率。结果表明,七种基因可能在阳性选择下。与光合作用相关的七个基因(psbN,psbI,psaC,atpH,佩德,psbD和psbM)显示最低的进化速度(平均Ka / ks = 0至0.0057),并在大多数物种中显示出均匀的速率。KA / KSpsbN,psbI,psaC和atpH为0,因为没有非同义替换(附加文件3.:表S3)。
为了检测特定系统发育分支可能的进化速率加速,我们分析了Ka/Ks变量最多的三个基因,即rpl23(核糖体的大亚基),rbcl.(rubisco的大亚基)和YCF4.(功能未知的基因)。自Ka/Ks比较14Lagerstroemia物种几乎是0,我们比较了KA / KSrpl23,rbcl.和YCF4.相比之下14Lagerstroemia物种和剩余的八种含杆菌物种。为了rpl23基因,KA / KS除了与...比较外,KA / Ks范围为0.891至1.8077D. Grandiflora..之间没有非同义替代Lagerstroemia物种和D. Grandiflora.此外L. Excelsa..从系统发育树中可以看出D. Grandiflora.和14Lagerstroemia物种比其他七种含杆菌物种更接近。为了rbcl.基因的Ka/Ks范围为0.1119 ~ 0.3849,这可能是由于Ks值较低(0.0046 ~ 0.0177)。为了YCF4.基因除了与之比较W. Fruticosa.(2.4259-2.8340),比例Lagerstroemia种与其他7种的变化范围为0.0305 ~ 0.8758。结果表明rpl23基因进化速度比rbcl.和YCF4..用于三种疏水板的Ka / ksL. Intermis.-r . rotundifolia由于KS是无效的.KA / KSYCF4.和rbcl.疏浚P. GANATUM.-W. Fruticosa.0.04和2.205,对rpl23无效(附加文件3.:表S3)。
基因组大小差异在22个睫毛CP基因组中
22种含杆菌物种,l . subcostata是最短(152,049 bp),和L. Villosa.是最长(160,769 bp)。除了L. Villosa.,CP基因组的长度Lagerstroemia物种在152,049 bp和152,519bp之间变化,而另一个属的Lythraceae的CP基因组在155,555 bp到159,380 bp(表1).一般来说,13的CP基因组Lagerstroemia种明显小于其他Lythraceae种。的cp基因组的较长长度L. Villosa.与6个新测序的Lythraceae物种相似,而不是与Lagerstroemia物种。代亚基地区(IGS)的长度在13个中的44,031bp至46,156 bp之间。Lagerstroemia结果表明,该基因组长度为45,923 bp ~ 51,357 bp,与全基因组长度一致4).与其他被子植物一样,IGS长度的差异在很大程度上决定了基因组大小的变化。22种植物叶绿体基因组中GC含量百分比为36.41 ~ 37.72%,平均为37.34%。的平均GC含量Lagerstroemia种占37.56%,高于其他属(36.88%)。
倒置重复的收缩和扩张(IRS)
22种石蒜属植物的基因组结构(包括基因数量和序列)高度保守。然而,IRA和IRB的边界发生了结构性变化(图。3.).虽然IR区域比其他区域更保守,但IR边界的扩大和收缩在基因组大小中起着主要作用[34.,35.,36.].
IRS的大小不同于24,421 BP(T. Natans.26,907 bp(L. Villosa.).在IRA-LSC的22个物种的边界内,18个物种的边界位于RPS19编码基因,导致RPS19在IRB地区的假膜。IRA-LSC边界L. Villosa.位于左侧RPS19编码基因与IRA-LSC的边界D. Grandiflora.,W. Fruticosa.和H. Myrtifolia.位于RPS19编码基因。之间的距离RPS19和IRA-LSC边界为3 ~ 279 bp。
除了14Lagerstroemia物种和W. Fruticosa., IRA-SSC边界被嵌入ndhF编码基因,长度为7bp (W. Fruticosa.)至158 bp(l . guilinensis)在爱尔兰共和军地区。其余7种石蒜科植物,ndhF位于IRA-SSC的右侧,在边界的距离为28bp至55 bp。对于所有物种,SSC-IRB边界位于YCF1.基因在IRB区域的长度为1062 ~ 2252 bpYCF1.伪基因在IRA区域中具有相应的长度。的trnH-GUG非编码基因位于IRB-LSC边界69 ~ 75 bp的右侧,距离IRB-LSC边界0 ~ 33 bp。
长重复结构分析
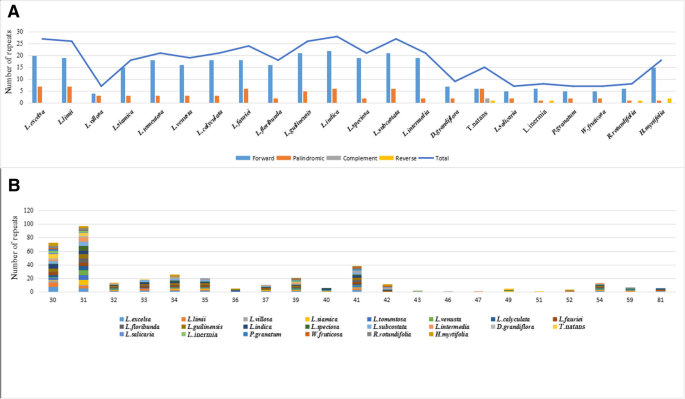
22种石藓科植物有4种类型383个长重复。18种物种只有前向重复和回文重复,而且只有T. Natans.都有四种重复。L. indica.有最多的重复次数,包括22个正向重复和6个回文重复。W. Fruticosa.,P. GANATUM.,l . salicaria和P. GANATUM.只有7次长重复。作为一个整体,H. Myrtifolia.和14lagerstroemia物种有更多长时间的重复比D. Grandiflora.,T. Natans.,l . salicaria,l . inermis,P. GANATUM.,W. Fruticosa.和r . rotundifolia(图。4一个,附加文件4:表S4).复制长度范围为30 bp至81 bp。重复序列为30,31和41占总长度的大部分(图。4b)。
简单序列重复(SSR)分析
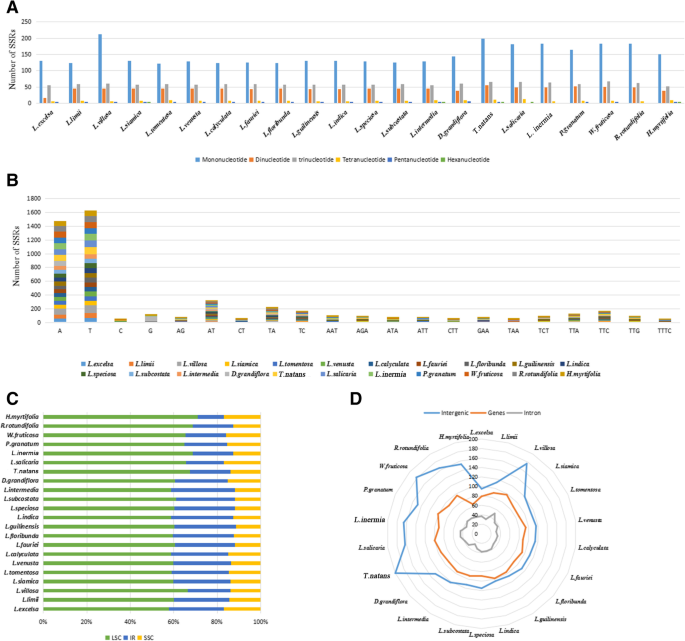
SSRS,也称为短串联重复或微卫星,由长度1-6bp的核苷酸重复单元组成[37.].SSRs在植物分类中起着重要的作用,作为分子标记被广泛应用[38.,39.].每个Lythraceae物种有211 ~ 332个SSRs,长度范围为8 ~ 16 bp(图2)。5,附加文件5:表S5)。发现了六种SSRs:单核苷酸,二核苷酸,三核苷酸,四核苷酸,五核苷酸和六核苷酸和六核苷酸。然而,仅在CP基因组中检测到六核苷酸重复l . siamica,l .媒介物,T. Natans.和L. Salicaria。在每个溶素物种中,单核苷酸重复是最常见的,数字范围为123至212;其次是突项核苷酸,范围为56至68;二核苷酸范围为16至52;四核苷酸范围为6至12;偏核苷酸范围为0至2和六核苷酸,范围为0至1(图。5a).以前发现,单核苷酸重复序列最丰富的是贝母,百合属植物和淫羊藿[22.,40].结果,单核苷酸重复可能在遗传变异中发挥比其他SSR的更重要的作用。
在22种溶素物种中,A / T单核苷酸重复分别占45.30和50.00%。C / G单核苷酸重复分别占1.40和3.30%。其他大多数SSRS由A / T组成,其可能导致覆盖22种溶质物种内的整个CP基因组的高含量的高分内容物(图。5b).同样的偏见也在栎[41.].此外,A / T单核苷酸的数量重复D. Grandiflora.,T. Natans.,l . salicaria,L. Intermis.,P. GANATUM.,W. Fruticosa.,r . rotundifolia和H. Myrtifolia.超过13人Lagerstroemia种类,71 - 103,从71到92。在14日LagerstroemiaA单核苷酸重复数为54 ~ 58个,T单核苷酸重复数为65 ~ 71个L. Villosa..这些结果表明,同一属的A/T单核苷酸重复数相似。而A/T单核苷酸重复的数量L. Villosa.是88/117,远高于其他13岁Lagerstroemia物种。我们可以推断,基因间间隔较长是主要原因。
SSR在LSC地区(62.90%)比IR区(23.20%)和SSC区(13.90%)更常见(62.90%)(图。5c). Lythraceae种cp基因组中的SSRs主要位于基因间间隔区,平均为132。分散在编码基因中的SSRs位居第二,平均为92个。最少的SSRs位于内含子中,平均为37个(图。5d). SSR位点位于31个编码基因(mat,atpI,rpoC2,RPOB.,trns-uga.,RPS14,psaB,psaA,ndhK,accD,YCF4.,cemA,PETA,PSAJ,PSBB,RPOA.,rpl22,RPS19,rpl2.,YCF2.,rrn23,ndhF,rpl32,ccsA,ndhD,ndhA,YCF1.,trnI-GAU,ndhB,YCF2.)和22种溶素的57个亚基族区域。yu等人。发现20个SSR位于9个编码基因中(mat,rpoC1,rpoC2,cemA,ndhD,ndhG,ndhH,YCF2和YCF1)的贝母cp基因(23.].这些结果表明,cp基因组变异较大的SSRs可用于近缘种的鉴定和系统发育研究。
22种Lythraceae种类中的分歧热点
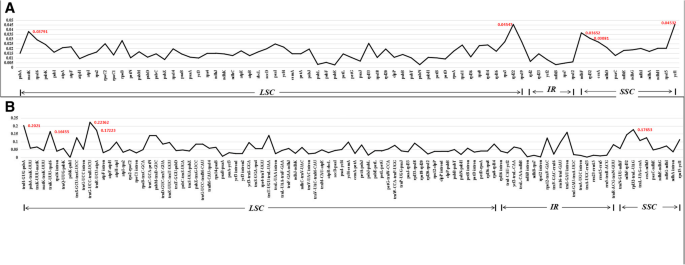
cp基因组上的分歧热点可以用来识别密切相关的物种,并提供系统发育的信息[42.,43.].使用该程序DNASP 5.1计算睫状症1.1的22CC基因组中的编码区和22c基因组的代核区域的核苷酸多样性(PI)值。它可以在图1中看到。6基因间区值高于编码区,说明基因间区分化程度较高。编码区域IR区域的Pi值为0.0029-0.0144,LSC区域的Pi值为0.00261-0.04547,SSC区域的Pi值为0.01254-0.04532。对于基因间区,IR区域的Pi值为0.00232 ~ 0.15964,LSC区域的Pi值为0 ~ 0.22362,SSC区域的Pi值为0.03567 ~ 0.17653(图1)。6,附加文件6:表S6)。选择10个具有高分歧的热点作为潜在的分子标记,以鉴定相关物种并在细胞内检查系统发育。
结合DnaSP和mVISTA的结果,我们评估了10个地区用ML树区分22种Lythraceae的能力。在编码区,4个最易变的基因是ndhF,mat,rbcl.,rpl22.对于基因间区域,trnK-rps16,rpl32-trnL, trnM-atpE,PSBM-TRND.,trnh-psba.和ndhf-rpl32是最多变的。根据其Pi值差异最大的区域与mVISTA程序得到的区域相似。10个发散性热点中,7个分布在LSC区域,3个分布在SSC区域。红外区域是如此的保守,没有发现高度发散的热点。根据ML树,trnK-rps16,ndhF,rpl32-trnL有最高的分辨率。的trnK-rps16基因清晰地将石蒜科的所有属分开,但14Lagerstroemia物种只能分为五个大分支。的ndhF基因也可以将所有属于睫毛内的所有属性分为36-100%的引导值,它分开了所有14Lagerstroemia物种。除了节点细节l . venusta,l .媒介物和L. Speciosa.引导价值为22%,14Lagerstroemia种被分离,bootstrap值为64-100%。的rpl32-trnL基因除以除了Lythrum.和伊米米,和14Lagerstroemia物种只能分为五个大分支。和....相比trnK-rps16和rpl32-trnL,ndhF具有最高的分辨率,是最佳的条形码候选标记。
22例含Cp基因组在Myrtales中的系统发育分析
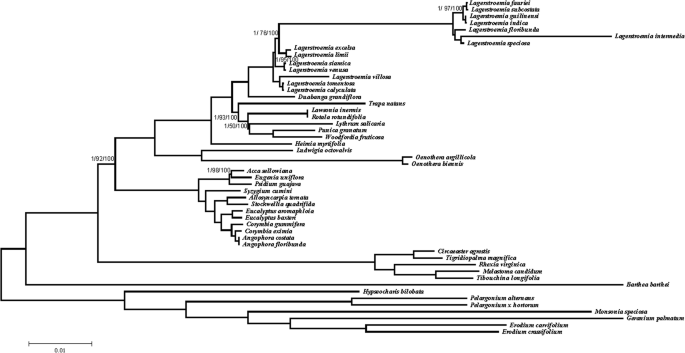
基于50cc基因组的66个共享蛋白质编码基因构建MP,ML和BI树(附加文件7:表S7)。这些cp基因组包括22种石缕藓科植物、12种桃金娘科植物、3种玉缕藓科植物、5种Melastomataceae植物和8种作为外群的植物。包括22种石蒜科植物H. Myrtifolia.,P. GANATUM.,14Lagerstroemia种及6个新测序的种(D. Grandiflora.,T. Natans.,l . inermis,r . rotundifolia,l . salicaria和W. Fruticosa.).
ML树,MP树和BI树的拓扑结构是一致的,四个家族(将含量杆菌,卵巢,Myrtaceae和Myrastomataceae)分为四种单胞菌。此外,Melastomataceae被鉴定为细胞质中的基础组。睫毛的五个亚属植物聚集在一起,展示了这一点P. GANATUM.和T. Natans.以前被认为属于punicaceae,而Trapaceae属于含杆菌。14.Lagerstroemia物种聚集在一起。仅在ML树中仅在90%下的引导值的两个节点。剩余节点的支持值超过92%。MP树中所有节点的引导值达到100%(图。7).结果表明,Melastomataceae是桃金娘家族中最早的分化类群,是桃金娘家族的姐妹科。下一个分化的科是桃金娘科,其次是雌龙科和石缕科。22种石缕藓科植物聚集成一个分支,并与3种石缕藓科植物(八鳃鳗,泥鳃鳗和Oenothera Biennis.).系统发育树显示桃金娘属种间存在明显的亲缘关系。
讨论
22个溶素CP基因组中的每一个具有四个连体结构,并包含110-112个独特基因,包括76-78个编码基因,29-30个TRNA和4 rRNA。基因组长度为152,049至16,0769bp,GC含量为36.41和37.72%。很明显,22cC基因组在基因组大小,结构和组织中高度保守,这也与先前报道的Melastomataceae物种的CP基因组一致[26.].22个Lythraceae CP基因组中的最大变化的位置是在非基因区域,这是CP基因组中的常见现象[10.,44.,45.].
研究结果表明,Lythraceae物种的进化速度较慢,Ka/Ks值较低,均在预期范围内,且不同功能基因组的Ka/Ks值存在差异。光合作用基因(psbN,psbI,psaC,atpH,佩德,psbD和psbM)的进化率最低。的基因rpl2.,rpl20.和rpl23参与复制,rbcl.和psbJ参与光合作用,YCF4.未知功能和其他基因包括ccsAKa/Ks值较高(≥1)。研究还发现,在22种Lythraceae物种中,这7个基因进化得更快辣椒和芝麻纪录物种(23.,46.].有些基因在进化速度上是特定物种的,比如CLPP.基因。虽然它在大多数绿色植物中高度保守,但在某些被子植物中却是迄今为止进化最快的质体编码基因。质体Clp蛋白酶复合物的进化速率是非常不同的[47.].的平均值Ka/KsCLPP.基因含量为0.0395,与某些植物的Ka/Ks比值较高有所不同。威廉姆斯还发现CLPP1经历了非常频繁的序列加速进化,这可能是由于内含子丢失在许多世系,如月见草属.然而CLPP.基因含有22种含量的两种内含子,这可能是其低Ka / Ks的原因。的CLPP.Lythraceae物种的保守长度(591 bp)可能导致负(净)选择。正选择下的基因通常有大量或多或少重复的氨基酸序列基序插入[48.].在阳性选择下的基因也可以在适应新的生态条件后近期多元化率的增加[49.].
四个cp基因组区域之间的边界在某些类群的进化中是重要的[50.].例如,假基因ψycf1要么ψrps19是通过IR地区的收缩和扩展产生的。的ψycf122种Lythraceae中均存在假基因ψrps194种Lythraceae中均未发现假基因。的RPS19基因定位于LSC区域H. Myrtifolia.,W. Fruticosa.和D. Grandiflora..在cp基因组中L. Villosa., 这RPS19基因在IRA中完全复制,在一些Malpighiales物种中也有报道[51.].
在以往的研究中,由于已发表的Lythraceae物种的cp基因组数量有限,缺乏基于完整cp基因组的比较分析,且Lythraceae内部的系统发育关系尚不清楚。P. GANATUM.和T. Natans.单独放在Punicaceae家族和Trapaceae家族中。之间的关系T. Natans.而桃金娘属内的其他物种由于形态上的差异较大而不能被确认T. Natans.,所以DNA数据是必要的,以确定的位置T. Natans.在myrtales。的rbcl.基因,pasA-ycf3垫片,和它的这些序列已被用来建立树和推断Lythraceae的系统发育关系,这些关系被我们的结果所证实。姐妹关系Trapa.和Sonneratia.得到了强烈支持,而姐妹之间的关系Trapa.和Lythrum.是弱的支持。总的来说,位置T. Natans.在系统发育分析中得到了证实。我们的结果进一步表明P. GANATUM.属于含杆菌。
结论
在本研究中,新测序的cp基因组D. Grandiflora.,T. Natans.,l . salicaria,l . inermis,W. Fruticosa.和r . rotundifolia据报道并与16种其他物种的报道,共比较22种含量的CP基因组。22种溶素物种的CP基因组在结构,组成和基因序列中类似,表明它们具有高度保守。三个系统发育树显示,42种Myrtales物种被完全分为四个分支,代表具有高自举值的四个家庭。来自以前存在的CP基因组,Myrtales的进化历史已初步理解。该研究的结果为系统发育研究提供了额外的富含遗传资源,并将在Myrtales的进一步研究中发挥重要作用。
材料和方法
植物材料和测序的DNA提取
在myrtales中六种睫毛的新鲜叶子(D. Grandiflora.,T. Natans.,l . salicaria,l . inermis,W. Fruticosa.和r . rotundifolia),立即用硅胶保存。采用CTAB法提取基因组DNA [52.].使用NanoDrop 2000微分光光度计和安捷伦2100生物分析仪(安捷伦技术,圣克拉拉,CA)来评估提取的DNA的浓度和质量。按照制造商的说明,纯化的DNA被用来建立一个测序库。使用Illumina HiSeq 2000测序仪(Illumina Biotechnology Company, San Diego, CA)获得150 bp的对端(PE) reads [9].
叶绿体基因组组装,注释和结构
Trimmomatic V0.3用于修剪和过滤Reg读取的验收质量得分≤20.Trimmomatic V0.3中的其他参数设置如下:将滑动窗设定为4:15,尾随设置为3,领先的领先设定为3,Minlen设置为50 [53.].CLC版本9.11(QIAGEN公司,Hilden)使用默认参数用于执行De Novo集装件。为每个物种创建了四到八种不同的折叠[54.].使用BLAST算法L. Fauriei.Cp基因组作为参考,以比对所有contigs。每个contig的末端可以重叠50 ~ 80 bp,形成一个大的cp基因组。为了验证基因组,还进行了Re-read mapping。每个基因组的覆盖范围从500倍到900倍不等。使用DOGMA v1.2进行基因组注释[8,9,10.,55.].OgDraw(http://ogdraw.mpimp-golm.mpg.de/)绘制了圆胞蛋白基因组图谱,然后手工编辑[56.].
密码子使用
相对同义密码子使用(RSCU)是特定密码子的频率与预期频率的比率[57.].RSCU > 1.00表示该密码子使用频率高于预期,而RSCU < 1.00表示该密码子使用频率低于预期。RSCU由DAMBE5获得[58.].
基因组比较分析和分子标记识别
比较了总共22种含杆菌物种。服用L. Excelsa.注释作为参考,Lagan模式中的MVISTA用于使22c CP Lythraceae物种基因组中的成对比对进行[59.].
22种溶性物种的77个蛋白质编码区用于评估进化速率变化。DNASP 5.1是计算非同义(KA)和同义替换的率(KS)[60.].获得了13,318 kA / ks;如果ks = 0,则无法计算该值。
Mega 6用于在Bioedit软件中手动调整后对准CP基因组[61.].然后使用dnnasp 5.1分别计算编码序列和非编码序列的Pi值。整个cp基因组、LSC、SSC和IR区域的Pi值也使用DnaSP 5.1计算[62.].
鉴定长重复序列和简单序列重复(SSRS)
声称用于检测四种长重复:前进,逆向,回文和互补重复[63.].参数设置如下:(1)最小重复次数大于30 bp;(2)序列一致性大于90%;(3)汉明距离等于3。Msatcommander 0.8.2.0用于探测SSRs的位置和数量[64.单核苷酸数≥8;二核苷酸≥4;三核苷酸、四核苷酸、五核苷酸和六核苷酸SSRs≥3。
系统发育分析
为了重建系统发育关系并检查细胞膜内睫毛膜的系统发育状态,42种Myrtales物种的完整CP基因组用于分析。具有默认参数设置的Clustal X 2.1软件用于对准66个蛋白质编码基因序列,必要时对对准进行手动调整[65.].系统发育分析中使用的数据矩阵作为补充数据。使用MEGA 6分析最大似然(ML)和最大简约(MP)的进化关系,使用MrBayes 3.1.2分析贝叶斯推断(BI)树[60.,66.].如果节点的引导值等于100%,则它们不会在树上标记。在所有分析中,八种物种被认为是小组。系统发育树是绘制在flogree中的[67.].
数据和材料的可用性
13种溶素物种的完整叶绿体基因组已在加入号MK881626下提交给NCBI数据库(https://www.ncbi.nlm.nih.gov/nuccore/MK881626), MK881627 (https://www.ncbi.nlm.nih.gov/nuccore/MK881627),MK881628(https://www.ncbi.nlm.nih.gov/nuccore/MK881628),MK881629(https://www.ncbi.nlm.nih.gov/nuccore/MK881629), MK881630 (https://www.ncbi.nlm.nih.gov/nuccore/MK881630),MK881631(https://www.ncbi.nlm.nih.gov/nuccore/MK881631), MK881632 (https://www.ncbi.nlm.nih.gov/nuccore/MK881632),MK881633(https://www.ncbi.nlm.nih.gov/nuccore/MK881633), MK881634 (https://www.ncbi.nlm.nih.gov/nuccore/MK881634), MK881635 (https://www.ncbi.nlm.nih.gov/nuccore/MK881635), MK881636 (https://www.ncbi.nlm.nih.gov/nuccore/MK881636), MK881637 (https://www.ncbi.nlm.nih.gov/nuccore/MK881637)及MK881638 (https://www.ncbi.nlm.nih.gov/nuccore/MK881638).分析中使用的其他数据包括在文章和其他文件中。
缩写
- CTAB:
-
十六烷基三甲基铵溴化铵
- di -:
-
二核苷酸
- IGS:
-
基于代际地区
- 美国国税局:
-
反向重复
- 卡:
-
非同义网站
- KA / KS:
-
非同义网站和同义网站的比例
- Ks:
-
同义网站
- LSC:
-
大单副本区域
- ML:
-
最大似然
- 单核细胞增多症-:
-
单核苷酸
- MP:
-
最大限定
- 五-:
-
Pentanucleotides
- PI:
-
核苷酸多样性值
- 核糖体rna:
-
核糖体RNA.
- RSCU:
-
相对同义词密码子用法
- SSC:
-
小单拷贝区域
- SSRS:
-
简单序列重复
- 四):
-
四核苷酸
- 三:
-
三核苷酸
- TRNA:
-
转移rna
参考文献
- 1。
达尔格伦,索恩RF。默特故事集:界限,变异和关系。《安莫Bot Gard》1984;71:633-99。
- 2。
Graham Sa,Crisci J,Hoch PC。基于形态特征的睫毛区菌紫外线菌的层状分析。Bot J Linn Soc。1993A; 113:1-33。
- 3.
Graham SA, Thorne RF, Reveal JL。天麻科植物亚科名称的验证。分类单元。1998;47:435-6。
- 4.
Conti E,Litt A,Sytsma KJ。Myrtales的环保及其与其他ROSID的关系:证据来自rbcl.序列数据。[中国科学(d辑:地球科学)]。
- 5。
等。桃金娘的家族间关系:分子系统发育和形态进化模式。系统机器人。1997;22:629-47。
- 6。
石树华,黄玉玲,谭淑芬,何新军,鲍福德。基于ITS序列的山松科植物及其与石藓科的亲缘关系的系统发育分析。植物学报2000;113:253-8。
- 7。
索恩射频。开花植物的分类和地理:被子植物纲双子叶植物(木兰科亚纲、毛茛科亚纲、石竹科亚纲、蝶叶科亚纲、蔷薇科亚纲)。机器人启66:441 2000;647年。
- 8。
顾Ch,Tembrock LR,Wu ZQ。完整的叶绿体基因组Lagerstroemia媒介物中国云南省西南特有的一种濒危植物。conservation Genet Resour. 2017; 9:357-60。
- 9。
顾长虹,董波,徐丽,Tembrock LR,郑世英,吴志强。完整的叶绿体基因组H. Myrtifolia.Myrtales中的比较分析。分子。2018;23:846。
- 10。
Gu Ch,Tembrock LR,吴ZQ,Simmons MP。完全塑性基因组Lagerstroemia fauriei.内含子的缺失rpl2.基因Lagerstroemia.Plos一个。2016; 11:E0150752。
- 11.
徐C,东路,李文,吕y,谢X,金X等。六个比较分析Lagerstroemia完全叶绿体基因组。前植物SCI。2017; 8:15。
- 12.
蓼科植物的子房、果实和种子形态。acta botanica yunnanica(云南植物研究),2014;
- 13。
李志强,李志强,李志强,等。石龙藓科植物的孢粉学与系统学。2属Haitia通过Peplis。[中国科学(d辑:地球科学)]
- 14。
格雷厄姆A,Graham S,Patel V,Lee S. Palytheracee的Palynology and Systematics。III。Genera Physocalymma通过伍德福德、附录和结论。Am J Bot. 1990; 77:159-77。
- 15。
Baas P, Zweypfenning RCVJ。石蒜科木材解剖。Acta botanica sinica . 1979; 28:117-55。
- 16。
Graham SA, Oginuma K, Raven RH, Tobe hSonneratia.和Duabanga及其系统学意义。分类单元。1993 b; 42:35-41。
- 17。
黄玉玲,史树华。基于叶绿体的石藓科植物系统发育研究rbcl.psaA-ycf3间隔子和核rDNA内转录间隔子(ITS)。植物营养与肥料学报(英文版)。
- 18。
Daniell H, Chan HT, Pasoreck EK。通过叶绿体遗传学的疫苗接种:用于预防和治疗遗传性或传染性人类疾病的负担得起的蛋白质药物。Annu Rev Genet. 2016; 50:595-618。
- 19。
Asaf S,Khan Al,Khan Ma,Waqas M,Kang Sm,Yun BW等。叶绿体基因组的拟南芥halleriSSP。gemmifera和rabidopsis lyrata.SSP。佩特拉亚州:结构和比较分析。Sci众议员2017;7:7556。
- 20.
黄藻叶绿体基因组的初步研究。线粒体DNA Part B. 2019;4:253-255。
- 21.
Rogalski M,Vieira Ln,Fraga HP,Guerra MP。园艺种类的塑性基因组学:植物群体遗传学,演化和生物技术的重要性和应用。前植物SCI。2015; 6:586。
- 22.
ronsted n,法律s,thornton h,fay mf,chase mw。单层的分子系统发育证据贝母和百合属植物(Liliaceae; Liliales)和InfraGeneric分类贝母.mol phylocyet evol。2005; 35:509-27。
- 23.
D'Agostino N,Tamburino R,Cantarella C,De Carluccio V,Sannino L等。十一个完整的塑料序列辣椒基因型:深入了解DNA变异和分子进化。基因。2018;9(10):503。
- 24.
Scarcelli n,Barnaud A,Eiserhardt W,Treier UA,Seveno M,D'Anfray A等。一组100雌噬菌素DNA引物对,用于在单子圈子中研究群体遗传学和系统发育。Plos一个。2011; 6:E19954。
- 25.
高yd,harris aj,他xj。形态和生态分歧百合属植物和Nomocharis在横断山脉和青藏高原可能会用栖息地专业化和杂交产生。BMC EVOL BIOL。2015; 15:147。
- 26.
Reginato M, Neubig KM, Majure LC, Michelangeli FA。Melastomataceae第一个完整的质体基因组在结构上高度保守。同行j。2016;4:e2715。
- 27.
应伟,詹德峰,冼军,梅伟林,戴慧峰,陈晓涛,等。完整的叶绿体基因组序列Aquilaria sinensis(rour。)吉尔格和狂热的进化分析。前植物SCI。2016; 7:280。
- 28.
田楠,韩,陈C,王Z。完全叶绿体基因组序列epipremnu maureum并与8种天南星科植物进行了比较分析。《公共科学图书馆•综合》。2018;13:e0192956。
- 29.
周军,崔勇,陈旭,李勇,徐志彬,等。完整的叶绿体基因组罂粟花rhoeas和东方罂粟花:分子结构、比较分析和系统发育分析。分子。2018;23:437。
- 30.
陈建军,郝志,徐洪,杨林,刘光生,等。残存木本植物叶绿体的完整基因组序列水杉.植物科学学报2015;6:447。
- 31.
顾昌华,Tembrock LR,郑世英,吴志强。完整的叶绿体基因组Catha Edulis.:与相关物种基因组特征的比较分析。国际分子生物学杂志。2018;19:525。
- 32。
钱J,Song J,Gao H,Zhu Y,Xu J,Pang X等。药用植物的完整叶绿体基因组序列萨尔维亚米尔蒂希萨.《公共科学图书馆•综合》。2013;8:e57607。
- 33。
Redwan RM, Saidin A, Kumar SV。MD-2菠萝叶绿体全基因组序列及其与其他9种菊科植物的比较分析。BMC Plant Biol. 2015;15:294。
- 34。
顾Ch,Tembrock LR,张D,吴ZQ。表征完全叶绿体基因组Lagerstroemia多花植物(Lytheraceae),一种狭窄的地方皱纹美丽,原产于东南亚。保守遗传资源。2016B; 9:1-4。
- 35。
杨敏,张旭,刘刚,尹勇,陈坤,云强,等。椰枣叶绿体基因组的完整序列(凤凰dactylifera L)。《公共科学图书馆•综合》。2010;5:e12762。
- 36。
罗布森洛杉矶,Peery R, Chumley TW, Dziubek C, Fourcade HM, Boore JL,等。比较叶绿体基因组学:分析包括来自被子植物的新序列nupharadvena.和毛茛属macranthus..BMC基因组学。2007; 8:174。
- 37。
陈c,ping z,choi ya,shu h,jr fgg。挖掘和表征微卫星柑橘类ests。Al Appl Genet。2006; 112:1248-57。
- 38。
杜杜因,杜杜因等。害虫17个个体的完整叶绿体基因组Jacobaea寻常的:人口和系统发育研究的SNP,微卫星和条形码标记。DNA Res。2011; 18:93-105。
- 39。
他,王Y,Volis S,Li D,Yi T.遗传多样性和人口结构:基于核和叶绿体微卫星变异的野生大豆(甘氨酸Sojasieb.Et Zucc)保护的含义。int j mol sci。2012; 13:12608-28。
- 40。
张勇,杜丽,刘安,陈静,吴丽,胡伟,等。完整的叶绿体基因组序列淫羊藿物种:进入系统发育和分类分析的光。植物科学学报2016;7:306。
- 41.
杨y,周t,段d,杨j,冯l,赵g.比较分析五种完整叶绿体基因组栎物种。植物学报。2016;7:57。
- 42.
杜yp,bi y,杨fp,张mf,陈xq,xue j等。完全叶绿体基因组序列百合属植物:深入了解进化动力学和系统发育分析。Sci众议员2017;7:5751。
- 43.
董开,刘继,俞俊,王L,周S.高度可变叶绿体标志物,用于在低分类水平和DNA条形码下评估植物系统发育。Plos一个。2012; 7:E35071。
- 44.
任t,杨y,周t,liu zl。比较塑性基因组樱草属植物物种:序列分歧和系统发育关系。int j mol sci。2018; 19:1050。
- 45.
Wicke S,Schneeweiss Gm,Depamphilis CW,Muller Kf,Quandt D.在土地植物中塑性染色体的演变:基因含量,基因令,基因功能。植物mol biol。2011; 76:273-97。
- 46.
张辉,李春华,苗洪,熊绍峰。叶绿体全基因组对植物进化的启示芝麻纪录公共科学图书馆。2013;8:e80508。
- 47.
Alissa M,Williams Am,Friso G,Van Wijk KJ,Sloan DB。塑体进化率的极端变化CLP.蛋白酶络合物。工厂J. 2019。https://doi.org/10.1111/tpj.14208.
- 48.
关键词:叶绿体,全基因阳性选择,同义替换率,重复,indel进化CLPP1基因。Plos一个。2008; 3:E1386。
- 49.
PIOT A,Hackel J,Christin Pa,BESNARD G.三分之一的塑体基因在Pacmad草的阳性选择下进化。Planta。2018; 247:255-66。
- 50.
Nazareno AG,Carlsen M,Lohmann LG。完全叶绿体基因组Tanaecium tetragonolobum:第一个Bignoniaceae塑料。Plos一个。2015; 10:E0129930。
- 51.
Menezes APA, Resendemore IRALC, Buzatti RSO, Nazareno AG, Carlsen M, Lobo FP等。叶绿体基因组的Byrsonima.物种(MalpighiAceae):高分歧序列的比较分析及筛选。SCI REP。2018; 8:1。
- 52.
张旭,周涛,坎瓦尔N,赵颖,白刚,赵刚gynostemma布。(葫芦科)叶绿体基因组:表征,对比分析和系统发育关系。前植物SCI。2017; 8:1583。
- 53.
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学。2014; 30:2114-20。
- 54.
Wu Z, Tembrock LR, Ge S.基因组数据集的差异是由于真正的生物变异或基因组装配错误:以两个叶绿体基因组为例。《公共科学图书馆•综合》。2015;10:1-14。
- 55.
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学。2004;20:3252-5。
- 56。
Conant GC, Wolfe KH。基因组Vx:简单的基于网络创建可编辑的圆形染色体图。生物信息学。2008;24:861-2。
- 57。
Cauzsantos LA, Munhoz CF, Rodde N, caet S, Santos AA, Penha HA,等。的叶绿体基因组西番莲岛(西番莲科)从长序列拼接:马尔皮亚目的结构组织和系统基因组研究。植物学报2017;8:334。
- 58。
夏X.AMBE5:用于分子生物学和演化中的数据分析的综合软件包。mol Biol Evol。2013; 30:1720-8。
- 59。
作者简介:陈志伟,副教授,博士,主要研究方向为比较基因组学。核酸学报2004;32:273-9。
- 60。
Librado P, Rozas J. DnaSP v5: DNA多态性数据综合分析软件。生物信息学。2009;25:1451-2。
- 61。
Tamura K,SteCher G,Peterson D,Filipski A,Kumar S. Mega6:分子进化遗传学分析6.0版。mol Biol Evol。2013; 30:2725-9。
- 62。
Rozas J,Sánchez-delbarrio JC,Messeguer X,Rozas R.DNASP,DNA多态性通过聚结剂和其他方法分析。生物信息学。2003; 19:2496-7。
- 63。
Kurtz S,Choudhuri JV,Ohlebusch E,Schleiermacher C,Stoye J,Giegerich R.声誉换句者:重复分析对基因组规模的歧管应用。核酸RES。2001; 29:4633-42。
- 64。
Faircloth Bc。MSATCommander:检测微卫星重复阵列和自动化轨迹的底漆设计。Mol Ecol Resour。2008; 8:92-4。
- 65.
Larkin Ma,Blackshield G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,等。Clustal W和Clustalx版本2.0。生物信息学。2007; 23:2947-8。
- 66.
Huelsenbeck jp,罗魁福特F.Mrbayes:贝叶斯植物发育丛林的推论。生物信息学。2001; 17:754-5。
- 67.
Simmons MP, Cappa JJ, Archer RH, Ford AJ, Eichstedt D, Clevinger CCCelasteae.(Celastraceae)和关系的关系Catha Edulis.(QAT)从形态特征和核和塑性基因推断。mol phylocyet evol。2008; 48:745-57。
致谢
感谢浙江省自然科学基金和国家自然科学基金。我们也感谢编辑和所有有价值的评论,谁帮助提高了我们的手稿的质量。
资金
这项工作研究和出版成本得到了中国(LY17C160003)和中国国家自然科学基金(Ly17C160003)和中国国家自然科学基金(第31770681,31370641)的资金支持。资金机构在设计的实验,分析和解释和写作稿件方面没有作用。
作者信息
从属关系
贡献
GC, WZ和ML设计实验,撰写手稿,修改手稿。ML、CK和WY进行了实验并对数据进行了分析。所有作者阅读并批准了最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
表S1。22个石蒜科叶绿体基因组中含有内含子的基因。(XLSX 50 kb)
额外的文件2:
表S2。22种石蒜科植物的密码子使用及密码子-反密码子识别模式。(XLSX 117 kb)
额外的文件3:
表S3。研究了22种石蒜科植物77个基因的Ka、Ks和Ka/Ks比值。(XLSX 1497 kb)
额外的文件4:
表S4。22种绳藓科植物长重复序列的比较。(XLSX 72 kb)
额外的文件5:
表S5。22种石藓科植物SSRs的比较。(XLSX 525 kb)
额外的文件6:
表S6。蛋白质编码基因和基因间区域的核苷酸变异性(Pi)值。(XLSX 23 kb)
额外的文件7:
表S7。GenBank收录的50个物种用于系统发育。(多克斯17 kb)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
顾超,马磊,吴志强。等等。22种含杆菌种类的叶绿体基因组的比较分析:细胞发育关系推论和MyRtales内的基因组进化。BMC植物BIOL.19,281(2019)。https://doi.org/10.1186/s12870-019-1870-3
收到了:
接受:
发表:
关键词
- Lythaceae.
- 叶绿体基因组
- Phylogenomic
- 桃金娘目