摘要
背景
大豆油是食用油的主要来源,野生大豆的驯化使其油脂含量和成分发生了显著变化。人们已经做了大量的工作来确定与大豆油性状有关的遗传位点。本研究的目的是鉴定与大豆籽油有关的数量性状位点,并比较野生大豆和栽培大豆的脂肪酸组成。
结果
利用特异位点扩增片段测序(slf -seq)方法,从野生大豆ZYD00463 (甘氨酸大豆)和栽培大豆WDD01514 (大豆)进行基因分型。最后,构建了包含20个连锁群(LGs) 11,398个单核苷酸多态性(SNP)标记的高密度遗传连锁图谱。采用基于模型的复合区间映射(CIM)方法,在多个环境中鉴定出24个稳定的种子含油量和成分qtl。在这些qtl中,23个与先前报道的qtl重叠或相邻。一个QTL,qPA10_1(5.94-9.98 Mb)。10是棕榈酸的新位点。在稳定的qtl区间,检测到一些与脂质代谢相关的有趣基因。
结论
从野生大豆ZYD00463与栽培大豆WDD01514杂交中分离到181个ril,并利用SLAF-seq方法构建了该基因的高密度遗传图谱。共鉴定出24个稳定的种子油含量和成分qtl,其中包括qPA10_1在Chr. 10上,一个新的棕榈酸位点。在QTL区域还检测到一些有趣的基因。我们的研究将为科学家们了解野生大豆和栽培大豆脂质代谢的遗传变异提供有用的信息。
背景
大豆(大豆(L. Merr.)是最重要的蛋白质和油料作物之一[1],其产量占2018年世界油籽产量的近60% [2]。大豆油主要由五种脂肪酸组成,分别是棕榈酸(C16:0)、硬脂酸(C18:0)、油酸(C18:1)、亚油酸(C18:2)和亚麻酸(C18:3),它们的平均含量分别约为10%、4%、18%、55%和13% [3.,4]。豆油的质量取决于脂肪酸的组成,它影响豆油的营养价值、风味和稳定性。不饱和脂肪酸在免疫系统调节、血液凝固、神经传递、胆固醇代谢以及脑和视网膜膜磷脂结构中发挥重要作用[5]。然而,不饱和脂肪酸容易被氧化,导致油的味道变差,降低油的保质期[6,7]。因此,目前培育的大豆含有较多的单不饱和脂肪酸(油酸)和较少的多不饱和脂肪酸(亚油酸和亚麻酸),这增加了氧化稳定性,对人体健康也更好[4,8]。
大豆油的含量和组成受多个数量性状位点(qtl)/基因控制,也受环境因素影响[j]。9,10]。迄今为止,与大豆种子油和脂肪酸相关的qtl已被广泛研究[11,12,13,14,15,16,17,18,19]。自从第一次研究试图发现大豆中的油脂qtl以来[10],在SoyBase数据库的所有20条染色体(Chr.)中鉴定出超过322个油质qtl和228个脂肪酸qtl [20.]。在这些QTL中,有一些是稳定的,在不同的双亲本群体和环境中检测到,包括Chr上1.64 ~ 2.09 Mb和33.35 ~ 35.95 Mb的QTL区域。种子含油量为20 [14,21,22,23,24,25], QTL区为44.58 ~ 48.58 Mb。14代表种子亚麻酸[11,13,24,26]。然而,由于与理想QTL等位基因的连锁不平衡不足以及性状的遗传复杂性,这些QTL的选择精度较低,尚未有效地应用于大豆种子油的标记辅助选择(MAS)中。27,28]。
随着大豆cv基因组测序的完成。威廉姆斯82 [29]和新一代测序技术的快速发展,单核苷酸多态性(SNP)标记已被用于构建高密度连锁图谱来鉴定QTL区间[25,30.]。Cao et al.(2017)基于包含2062个SNP标记的高密度遗传图谱,鉴定出一个QTL,qOil-5,对种子油进行了定位,定位到Chr. 05,物理距离为2.5 Mb [30.]。使用Illumina Infinium BeadChip测序平台,Patil等人(2018)在Chr. 02上报告了稳定的油含量qtl。qOil_02),第08段(qOil_08),第15章(qOil_15)及第20 (qOil_20),使用3343个多态snp(3个K-SNP) [25]。特异性位点扩增片段测序(SLAF-seq)技术已被用于构建高密度遗传图谱,是大豆大规模从头发现SNP和基因分型的有效方法[31,32]。Li et al.(2017)使用3541个SLAF标记检测到5种脂肪酸的26个稳定qtl,平均距离为0.72 cM [31]。Zhang等人(2018)创建了包含8597个SNP位点的高密度遗传图谱,平均距离为0.57 cM,其中两个qtl,qOil10-1和qOil10-2,以计算含油量[32]。此外,通过SNP基因分型的全基因组关联研究(genome-wide association studies, GWAS)发现了一些与大豆油含量和组成相关的基因[30.,33,34,35,36,37,38]。这些基因为大豆种子油的改良提供了有用的信息。
在大豆中,与脂质生物合成相关的关键功能基因已被研究,其中包括脂肪酸去饱和酶基因FAD2-1A(Glyma.10 g278000),FAD2-1B(Glyma.20 g111000),FAD3A(Glyma.14 g194300),FAD3B(Glyma.02 g227200),FAD3C(Glyma.18 g062000),FAD7(Glyma.18 g202600和Glyma.07 g151300) [39,40,41]、3-酮酰基acp合成酶II基因(内二世,Glyma.17 g047000和Glyma.13 g112700) [42,43]和二甘油酯酰基转移酶基因DGAT(Glyma.17 g053300) [44]。然而,过表达单个基因不能显著增加脂肪酸的生物合成通量[45]。脂质代谢似乎需要多个相关基因的调控。一些重要的转录因子也被发现通过直接结合脂质生物合成基因的启动子参与脂质积累的调节。例如,过度表达GmNFYA,GmDof4,GmDof11,GmbZIP123,和GmMYB73显著增加转基因植物种子脂质积累[46,47,48,49]。研究这些因素有助于加深对大豆脂质代谢机制的认识。
栽培大豆种子的含油量约为18-22%,而野生大豆种子的含油量约为8-10% [25]。为了鉴定控制大豆种子含油量和组成的基因,需要对栽培大豆和野生大豆的含油量进行QTL分析。本研究以野生大豆ZYD00463(2)的种间杂交获得的181个重组自交系(RILs)为材料,利用slf -seq技术构建了大规模SNP标记,构建了连锁群,并绘制了控制种子油脂和成分的qtl图谱。甘氨酸大豆)和栽培大豆WDD01514 (大豆).本研究结果可以帮助科学家了解野生大豆和栽培大豆在驯化过程中籽油的遗传变异,并通过分子育种进一步提高籽油的数量和质量。
结果
表型变异
2015 - 2016年在湖北武汉和河南许昌对亲本和子代的种子油含量和5种优势脂肪酸组成进行了测定。表格1结果表明,WDD01514的平均含油量为23.76%,约为ZYD00463(11.89%)的2倍。籽油成分方面,WDD01514的油酸含量(20.57%)高于ZYD00463,亚麻酸含量(7.60%)低于ZYD00463,分别为11.39%和16.60%。181个品种的含油量和组成差异较大,含油量为11.36 ~ 13.11%,油酸含量为17.07 ~ 22.68%,亚麻酸含量为11.05 ~ 14.41%。
使用Shapiro-Wilk (w)统计表明,油分、棕榈酸、硬脂酸(2015/X除外)、亚麻酸与P-values > 0.05。然而,油酸和亚油酸并非正态分布(P< 0.05)1;额外的文件1).油酸向ZYD00463倾斜,而亚油酸向WDD01514倾斜1).此外,在棕榈酸、硬脂酸、油酸和亚油酸的后代中观察到海侵分离1;额外的文件1),表明亲本之间发生了全面的等位基因重组。
广义遗传力(h2)在组合环境下,油脂和脂肪酸的变异范围为80.2% ~ 92.0%,这表明遗传变异占观察到的表型变异的主要比例(表2)1).方差分析显示FG × E互作值在各性状(P< 0.001)。然而,F-值小于基因型(表2)2).
在所有四种环境中,油含量与油酸之间呈正相关(P< 0.01),而油分与棕榈酸、油分与亚麻酸、棕榈酸与油酸、油酸与亚油酸、油酸与亚麻酸、油酸与亚麻酸均呈负相关(P< 0.01)(附加文件2).这表明控制这些性状的重要遗传因素是紧密相连的。
利用slf -seq构建SNP标记遗传图谱
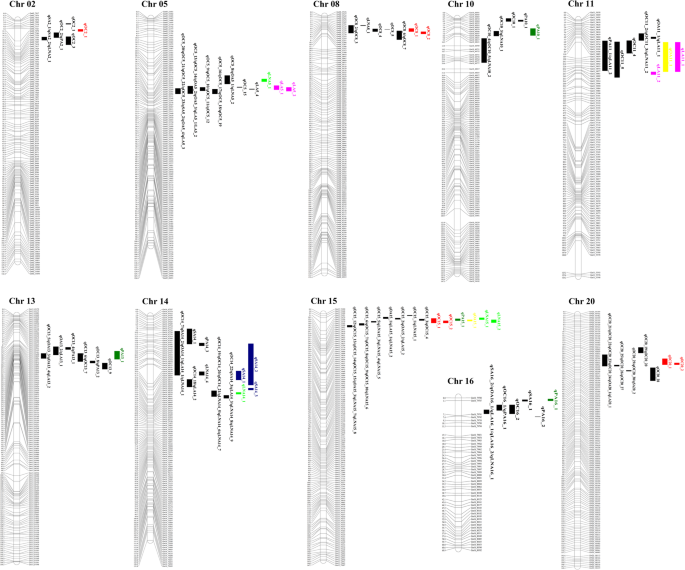
采用SLAF-seq方法开发双亲之间的SNP标记。最终,分布在20个连锁组(LGs)上的11,398个SNP标记被用于构建遗传连锁图谱(附加文件)3.、附加文件4).这些SNP标记覆盖大豆基因组2913.78 cM,标记之间的平均距离为0.26 cM。20个LGs的遗传距离为126.23 cM ~ 226.60 cM (Chr. 03),平均标记间隔为0.15 ~ 0.60 cM。最大的LG (Chr 01)含有891个SNP标记,最小的LG (Chr 13)含有248个SNP标记(附加文件)5).
油含量qtl
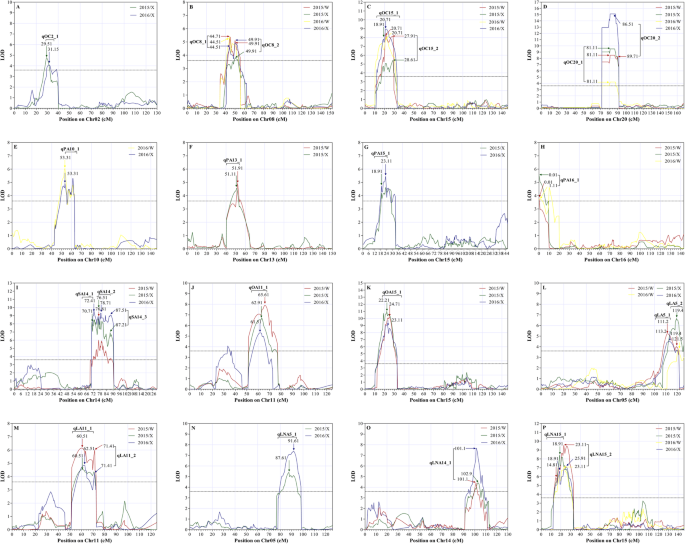
本研究共定位了22个与种子含油量有关的qtl(附加文件)6).其中,在多个环境中鉴定出7个LOD > 3.6的稳定qtl(图2)。1;表格3.),并被映射到第02页(qOC2_1), 08 (qOC8_1和qOC8_2), 15 (qOC15_1和qOC15_2),及20 (qOC20_1和qOC20_2)(附加文件7).QTL的qOC2_1在Chr. 02上平均解释了6.7%的含油量表型变异;qOC8_1和qOC8_2在Chr 08上,平均分别解释了8.9%和6.6%的表型变异;qOC15_1和qOC15_2Chr 15对表型变异的平均贡献率分别为12.9%和11.0%;和qOC20_1和qOC20_2对Chr. 20的平均解释率分别为12.2和19.3%。所有qtl均表现出负加性效应,表明来自野生大豆亲本ZYD00463的等位基因对含油量有负作用。与已报道的QTL区域相比,有6个QTL,包括qOC8_1,qOC8_2,qOC15_1,qOC15_2,qOC20_1,qOC20_2,与之前的qtl重叠(图2)。2;额外的文件8).QTL的qOC2_1,该QTL位于Chr. 02上5.08-6.27 Mb区间内,与定位的QTL相邻,位于6.86-9.67 Mb区间内(图2)。2;额外的文件8).
在这些稳定的QTL区域中鉴定出6个参与脂质代谢的酶基因(附加文件)9),其中包括两个丙酮酸激酶基因(GmPK,Glyma.02 g071000和Glyma.02 g071100的基因区域内qOC2_13-酮酰基acp还原酶基因(GmFabG,Glyma.08 g102100内)qOC8_1,两个3-酮酰基辅酶a合成酶基因(GmKCS,Glyma.15 g042500,Glyma.15 g046300), 3-羟基酰基acp脱氢酶基因(GmFabZ,Glyma.15 g052500内)qOC15-1.此外,还发现了两个可能参与脂质代谢的转录因子基因qOC8_2(附加文件10),包括同源箱亮氨酸拉链基因(GmZIP,Glyma.08 g124400)和核因子Y亚基a基因(GmNF-YA,Glyma.08 g124200).
油成分的qtl
共定位了66个与种子油成分相关的qtl(附加文件)6).在这些qtl中,17个LOD > 3.6的稳定qtl在多个环境中被鉴定出来(图2)。1;表格3.).这些qtl被定位到Chr. 05、10、11、13、14、15和167).对于棕榈酸,有4个稳定的qtl,包括qPA10_1第10页,qPA13_1第13页,qPA15_1在第15章,和qPA16_1在Chr 16上,平均贡献率分别为12.9%、8.4、9.7和8.3%。所有棕榈酸的qtl,除了qPA15_1和qPA16_1,结果表明,野生大豆亲本ZYD00463等位基因对棕榈酸具有负加性效应。对于硬脂酸,Chr. 14上有3个稳定的qtl,分别为:qSA14_1,qSA14_2,qSA14_3,平均分别占表型方差的18.6%、16.0%和17.0%。3个qtl均表现负加性效应。对于油酸,有两个稳定的qtl,包括qOA11_1第11页和第11页qOA15_1在Chr. 15,分别解释了12.3%和19.1%的表型变异。这两个qtl的加性效应为负。对于亚油酸,有4个稳定的qtl,包括qLA5_1和qLA5_25月5日qLA11_1和qLA11_2在Chr 11上,被鉴定和解释的平均表型方差分别为9.9、9.6、10.4和10.0%。除了qLA11_1和qLA11_2,qLA5_1和qLA5_2呈负加性效应。对于亚麻酸,有4个稳定的qtl,分别为qLNA5_15月5日,qLNA14_1月14日,qLNA15_1在第15章,和qLNA15_2在Chr 15上,平均解释了12.5%、10.0%、12.9%和16.1%的表型变异。4个qtl的加性效应均为正,说明这些等位基因对野生大豆亲本ZYD00463的加性效应为正。在17个稳定的脂肪酸组成qtl中,qPA10_1(位于5.94-9.98 Mb范围内)与报道的qtl和数量性状核苷酸(QTNs)没有重叠或不在附近(图2)。2;附加文件8和11).这表明这是一个新的棕榈酸位点。
我们鉴定出21个酶编码基因可能参与稳定QTL区域的脂质代谢(附加文件)9).这些基因包括两个丙酮酸激酶基因(GmPK,Glyma.10 g065000和Glyma.13 g149800)、两个二酰基甘油酰基转移酶基因(GmDGAT,Glyma.13 g106100和Glyma.13 g118300)、三个磷脂酰甘油酰基转移酶基因(GmPDAT,Glyma.13 g108100,Glyma.16 g005800,Glyma.11 g190400)、两个3-酮酰基acp合酶I基因(GmFabB,Glyma.13 g128000和Glyma.05 g218600), 3-酮酰基acp合成酶II基因(GmFabF,Glyma.13 g112700),两个3-酮酰基辅酶a合成酶基因(GmKCS,Glyma.15 g042500和Glyma.15 g046300), 3-羟基酰基acp脱氢酶基因(GmFabZ,Glyma.15 g052500)、两个酰基载体蛋白基因(GmACP,Glyma.16 g011300和Glyma.05 g201300),丙二酰辅酶a:ACP丙二酰转移酶基因(GmFabD,Glyma.11 g164500),一种omega-3脂肪酸去饱和酶3基因(GmFAD3,Glyma.11 g174100),乙酰辅酶a羧化酶基因(GmACCase,Glyma.05 g221100),一个酰基辅酶a合成酶基因(通用汽车金融服务公司(gmac),Glyma.11 g194500),丙酮酸脱氢酶基因(GmPDH,Glyma.14 g186900)和溶血磷脂酸酰基转移酶基因(GmLPAAT,Glyma.15 g034100).此外,还鉴定了9个参与脂质代谢的转录因子基因(附加文件10),包括一个螺旋环螺旋基因(GmHLH,Glyma.05 g200900),一个WRKY蛋白基因(GmWRKY,Glyma.05 g203900),一个C3H蛋白基因(GmC3H,Glyma.05 g224400),两个同源箱亮氨酸拉链基因(GmZIP,Glyma.10 g071700和Glyma.11 g145800),一个B3结构域蛋白基因(GmB3,Glyma.10 g076100)、两个MYB蛋白基因(GmMYB,Glyma.13 g109100和Glyma.16 g007200),以及一个DBB蛋白基因(GmDBB,Glyma.15 g029500).
不同性状的qtl在Chr上也存在共定位。11和15(附加文件7).的经济价值qOA11_1和qLA11_1在Chr。11在11.0-25.6 Mb的物理间隔内。的经济价值qOC15_1,qPA15_1,qOA15_1,qLNA15_2在Chr。在2.80-5.63 Mb的物理间隔内有15个。
讨论
在本研究中,我们鉴定了24个稳定的种子油和成分qtl。通过将他们绘制的区域与先前报道的大豆参考基因组进行比较(图2)。2;附加文件8和11,我们发现了这一点qPA10_1与之前报道的qtl没有重叠或相邻。此外,它不含与GWAS获得的棕榈酸相关的qtn。由于构建了包含11,398个SNP标记的高密度遗传图谱,QTL区域的间隔与之前报道的相比显著缩短。例如,对于含油量,物理距离qOC8_1,qOC8_2,qOC15_2,qOC20_2分别为7.52-9.44 Mb、9.44-10.8 Mb、4.15-5.63 Mb和32.5-33.8 Mb。相比之下,为5.58 ~ 10.28 MbqOC8_1[16], 5.58-10.3 MbqOC8_2[16], 3.23-4.07 MbqOC15_2[17], 27.0-34.3 MbqOC20_2[54]。对于硬脂酸,物理距离为qSA14_1和qSA14_3在我们的研究中分别为32.2-37.5 Mb和42.2-43.4 Mb。相比之下,间隔为16.3-45.9 Mb [56]。对于亚油酸,物理距离为qLA5_1和qLA5_2分别为37.5-39.9 Mb和38.5-40.7 Mb,而间隔为37.6-42.2 Mb [57]。
与之前报道的qtl (http://www.soybase.org)(附加文件8和11), 24个qtl中有23个与先前报道的qtl接近或重叠(图2)。2).例如,QTLqOC2_1Chr. 02的含油量约为5.08-6.27 Mb,与已报道的QTL (6.86-9.67 Mb) [50]。此外,qOC8_18月8日,qOC8_28月8日,qOC15_1在第15章,和qOC15_2在Chr 15上,分别位于7.52 ~ 9.44 Mb、9.44 ~ 10.8 Mb、2.80 ~ 5.63 Mb和4.15 ~ 5.63 Mb,与报道的5.52 ~ 12.64 Mb [16,51,52], 5.52-14.21 Mb [51,52,53], 3.23-4.07 Mb [17],及4.52-5.21 Mb [25),分别。对于棕榈酸,qPA13_1(21.8-26.8 Mb)qPA16_1(0.42-1.19 Mb)分别接近于报道的26.41-29.08 Mb和2.67-5.06 Mb [55,56]。对于硬脂酸,qSA14_1,qSA14_2,qSA14_3分别位于32.2 ~ 37.5 Mb、16.3 ~ 40.5 Mb和42.2 ~ 43.4 Mb,与16.30 ~ 45.90 Mb [56]。对于亚麻酸,qLNA5_1(33.8-35.4 Mb)与31.98-34.65 Mb [18]。虽然这些qtl位于相似的区域,但它们的负责基因是否相同还需要进一步的研究。相比之下,qPA10_1在Chr. 10上定位到5.94 ~ 9.98 Mb的区域,远高于已有报道的0.98 ~ 1.87 Mb [24(图。2;额外的文件8),并没有发现任何经GWAS与棕榈酸有关的qtn [58[附加文件11).结果表明,该QTL是棕榈酸的新位点。
利用SoyBase数据库的基因注释工具对24个稳定QTL区间的所有基因进行注释[20.],鉴定出12个参与脂质代谢的重要酶编码基因(另附文件9).例如,GmACCase(Glyma.05 g221100)编码一种乙酰辅酶a羧化酶,该酶催化乙酰辅酶a生成丙二酰辅酶a,作为脂肪酸从头开始生物合成的直接底物[59]。GmFabD(Glyma.11 g164500)编码一个丙二酰辅酶a:ACP丙二酰转移酶,负责将丙二酰辅酶a的丙二酰基转移到酰基载体蛋白(ACP)。GmACP(Glyma.05 g201300和Glyma.16 g011300)编码了一种酰基载体蛋白,该蛋白在脂肪酸合酶的酶域之间运输不断生长的脂肪酸链[60]。GmFabB(Glyma.05 g218600和Glyma.13 g128000),GmFabF(Glyma.13 g112700)分别编码一个酮酰- acp合成酶I和II,这两个合成酶分别主要用于生产棕榈酰- acp和硬脂酰- acp作为脂肪酸链延伸的凝聚酶。GmFabG(Glyma.08 g102100),GmFabZ(Glyma.15 g052500)编码3-酮酰基- acp还原酶和3-羟基酰基- acp脱氢酶,分别催化3-酮酰基- acp还原和脱水反应[61]。此外,GmFAD3(Glyma.11 g174100)编码了一种omega-3脂肪酸去饱和酶3,该酶催化第三个双键生成亚油酸,从而产生亚麻酸[40,62]。GmDGAT(Glyma.13 g106100和Glyma.13 g118300)编码二酰基甘油酰基转移酶,催化脂肪酸和甘油3-磷酸形成标签[63,64]。这些基因在稳定QTL中的存在表明它们可能参与大豆种子脂质代谢。然而,这些酶编码的基因是否负责相应的qtl需要用转基因方法确认。除了参与脂质代谢的关键酶基因外,一些转录因子基因在调节脂肪酸生物合成中也起着重要作用。我们在24个稳定的QTL区间内鉴定出11个参与脂质代谢的转录因子基因(附加文件)10),包括GmB3,GmC3H,GmDBB,GmHLH,GmMYB,GmNF-YA,GmWRKY,GmZIP.研究了几种参与大豆油脂和脂肪酸生物合成调控的转录因子。例如,过度表达GmNFYA在拟南芥显著增加种子含油量[46]。GmMYB73促进脂质积累转基因拟南芥,可能是通过抑制HD-ZIP的转录因子GLABRA2 [49]。QTL区域转录因子在大豆籽油性状驯化中的作用有待进一步研究。
在新QTL的间隔中qPA10_1研究人员还发现了三个可能参与脂质代谢的候选基因,包括GmPK(Glyma.10 g065000),GmB3(Glyma.10 g076100),GmZIP(Glyma.10 g071700).GmPK(Glyma.10 g065000)编码丙酮酸激酶(PK)。在大豆种子发育过程中,磷酸烯醇丙酮酸羧化酶和丙酮酸激酶活性参与了一个复杂的相互作用,调节糖酵解碳转化为蛋白质和油生物合成前体的代谢流[65]。在拟南芥在种子中,通过破坏丙酮酸激酶β1亚基编码基因来降低可再生丙酮酸激酶活性,导致种子含油量减少60% [66]。GmB3(Glyma.10 g076100)编码一个B3结构域家族的转录因子。在芸苔属植物显著,BnFUSCA3(BnFUS3)突变体,B3结构域转录因子的成员,抑制种子油水平和增加亚油酸水平,可能是由于表达减少ω3 FADESATURASE(FAD3) [67]。在大豆中,B3结构域转录因子GmLEC2a弥补了大豆的缺陷拟南芥atlec2幼苗发育和甘油三酯积累的突变体。的过度表达GmLEC2a在拟南芥与对照种子相比,种子的甘油三酯含量增加了34%,长链脂肪酸组成增加了4% [68]。GmZIP(Glyma.10 g071700)编码同源盒亮氨酸拉链蛋白。Song et al.(2013)报道GmbZIP123(Glyma.06 g010200)提高转基因大豆的脂质含量拟南芥通过促进蔗糖转运基因的表达(SUC1和SUC5)和细胞壁转化酶基因(cwINV1,cwINV3,cwINV6) [48]。在答:芥, bZIP67结合到脂肪酸去饱和酶(FAD3)促进剂FAD3表达,并增加亚麻酸种子含量[69]。
结论
利用slf -seq技术,构建了包含11,398个SNP标记的高密度遗传图谱,并鉴定出24个稳定的qtl,用于测定野生大豆和栽培大豆的籽油含量和脂肪酸组成。在这些QTL中,1个QTLqPA10_1与先前报道的qtl不重叠或不接近,也不包含任何与棕榈酸相关的qtn,表明它是一个新的位点。在QTL区域还发现了一些值得进一步研究的有趣基因。本研究为进一步阐明大豆油脂的生物合成提供了有价值的信息。
方法
植物材料
RIL制图人群包括181名F7由野生大豆ZYD00463 (甘氨酸大豆)和栽培大豆WDD01514 (大豆).ZYD00463 (甘氨酸大豆)及WDD01514 (大豆)由中国农业科学院油料作物研究所(中国武汉)提供。2015年和2016年,在湖北武汉(N30°35′,E114°33′)和河南许昌(N34°02′,E113°81′)两个试验站种植181株rir及其亲本。这两个地点具有不同的气候条件,武汉气温较高,降雨较多,而许昌气温较低,降雨较少。采用完全随机区组设计,设置亲代和子代3个重复。每个地块包括2.5 m的行距,行距1.0 m,相邻植物间距0.5 m。对于每种基因型,在R8生育期(完全成熟期)从每个地块的5株植株上收获种子[70]。将大豆种子风干至定重,然后按如下方法评估性状。
油和脂肪酸的测定
种子含油量和脂肪酸组成根据Wei等测定。71稍加修改。简单地说,每系30颗大豆种子被磨成细粉。将每种粉末状样品的20毫克转移到一个10毫升的玻璃管中。样品中加入硫酸-甲醇(5%,2 mL)、丁基羟基甲苯(BHT) (0.2%, 25 μL)、甲苯(300 μL)和内标(IS)(十七酸甲酯,Sigma Aldrich, St. Louis, USA, 2.5 ~ 5 mg/mL, 100 μL)制备脂肪酸甲酯(FAME),在90-95℃水浴中酯化1.5 h。冷却至室温后,加入氯化钠(0.9%,1ml)和n提取液中加入-己烷(1ml)。所得上清液用于气相色谱分析。
采用毛细管柱(FFAP, 30 m, 0.25 mm id, 0.50-μm膜厚)的气相色谱(Agilent 6890 N, USA)分离酯类。氮气被用作载气,入口压力为25psi。进样口和检测器(FID)的温度分别保持在250℃和260℃,色谱柱的温度程序为:210℃(1 min),以10℃/min (22 min)的速度增加到230℃。用于统计分析的计算机软件为STATISTICA 6.0版本(Statsoft Inc., Oklahoma, USA)。使用脂肪酸甲酯的真实标准,根据其保留时间确定峰。用相对峰面积定量测定脂肪酸的含量。种子含油量计算公式如下:
一个t和一个年代分别为根据停留时间确定的总峰面积和内标峰面积;和m年代和m我是内标和干籽的重量。每行样品测定三次。
统计分析
所有表型数据使用SAS 9.3 (SAS Institute Inc., Cary, NC, USA)的PROC mix程序进行分析。各性状间的Pearson相关系数采用SAS 9.3中的PROC CORR函数计算。
在单一环境下的遗传力估计如下:
不同环境的遗传力计算如下:
在哪里\({\updelta}_{\mathrm{g}}^2 \)为ril间每样性状的基因型方差分量,\({\updelta}_{\mathrm{e}}^2 \)为误差方差,r为性状的重复次数,n为环境的个数[72,73]。对不同环境进行方差分析,以确定基因型、环境及其相互作用的重要性。方差的误差分量(\({\updelta}_{\mathrm{e}}^2 \)),基因型×环境相互作用(\({\updelta}_{\mathrm{gy}}^2 \)),以及基因型(\({\updelta}_{\mathrm{g}}^2 \))采用SAS 9.3 (SAS Institute Inc.)中的一般线性模型程序(PROC GLM)进行分析。所有参数均由方差分析的期望均方估计。
基因图谱构建
采用SLAF-seq方法开发双亲之间的SNP标记[74]。SNP标记利用JoinMap 4.0版软件的Kosambi作图功能构建高密度遗传连锁图谱[75]。根据LOD评分为3.0对SNP标记进行分组,然后根据输入算法进行排序以估计重组频率。将连锁位点之间的重组频率转换为距离(cM) [76]。通过将每个SNP标记的序列与Williams 82的基因组序列比对,分析LGs与大豆参考基因组的共线性[77]。
QTL定位与候选基因预测
使用WinQTL制图师2.5版中引入的复合区间映射(CIM)来检测可加性qtl [78]。对于每个性状,通过1000次重复的排列试验估计LOD > 3.6的显著QTL的鉴定阈值P< 0.05。将辅助因素考虑在内,并在测试间隔周围选择10 cM的窗口大小进行CIM分析。利用MapChart 2.2版本绘制了qtl在遗传连锁图谱上的分布[79]。检测到的qtl用代表性状缩写的一个或多个字母与染色体数目组合表示[80]。将在武汉和许昌两种环境中连续2年重复检测到的qtl定义为稳定qtl。其他组先前报告的qtl被定义为报告的qtl。根据大豆参考基因组(Wm82.a2.v1.1)的注释,从SoyBase数据库中获得稳定QTL区间内的预测基因。使用默认设置的GO网站对预测基因进行基因本体(GO)富集分析[81]。
数据和材料的可用性
支持本文结论的所有数据都包含在本文及其附加文件中。
缩写
- 方差分析:
-
方差分析
- 二叔丁基对甲酚:
-
丁羟甲苯
- CIM:
-
复合区间映射
- 名声:
-
脂肪酸甲酯
- GC:
-
气相色谱法
- 走:
-
基因本体论
- GWAS:
-
全基因组关联研究
- 是:
-
内部标准
- 拉:
-
亚油酸
- 格林:
-
连锁群
- 放大器:
-
亚麻酸
- LOD:
-
概率对数
- MAS:
-
分子标记辅助选择
- 门店:
-
新一代测序
- 办公自动化:
-
油酸
- 度:
-
含油量
- PA:
-
棕榈酸
- QTL:
-
数量性状位点
- 本考察团:
-
数量性状核苷酸
- 瑞来斯:
-
重组自交系
- 山:
-
硬脂酸
- SLAF-seq:
-
特异位点扩增片段测序
- SNP:
-
单核苷酸多态性
参考文献
- 1.
引用本文:金美英,范锴,姜玉军,金坤,李世生。从核苷酸到基因组的大豆驯化历史追踪。品种科学,2012;61:445-52。
- 2.
SoyStats。美国大豆协会(ASA),密苏里州圣路易斯,2019。http://soystats.com/international-world-oilseed-production/.2019年9月10日访问。
- 3.
Lee JD, Bilyeu KD, Shannon JG。大豆籽油中改性脂肪酸谱的遗传与育种。作物科学与生物技术,2007;10(4):201-10。
- 4.
Clemente TE, Cahoon EB。大豆油:功能性和总含量改变的遗传方法。植物生理学报。2009;51(1):1030 - 1040。
- 5.
Abedi E, sahara MA。长链多不饱和脂肪酸来源及其营养和功能特性的评价。食品科学,2014;2(5):443-63。
- 6.
Smouse TH。大豆油还原风味研究进展。石油化工学报,1979;56(11):747-51。
- 7.
mount TL, Warner K, List GR, Kleiman R, Fehr WR, Hammond EG, Wilcox JR.脂肪酸组成改变对大豆油稳定性的影响。石油化工学报,1988;65(4):624-8。
- 8.
Lee JD, Bilyeu KD, Pantalone VR, Gillen AM, So YS, Shannon JG。大豆籽油中油酸浓度的环境稳定性FAD2-1A和FAD2-1B突变基因。作物科学,2012,52(3):1290-7。
- 9.
伯顿JW。数量遗传学:与大豆育种有关的结果。社会科学。1987;16:21 - 47。
- 10.
Diers BW, Keim P, Shoemaker RC, Fehr WR。大豆种子蛋白质和油脂含量的RFLP分析。理论与应用,1992;83(5):608-12。
- 11.
潘塔龙,刘建军,刘建军,等。大豆中亚麻酸的分子标记。石油化工学报,2004;81(6):559-62。
- 12.
Shibata M, Takayama K, Ujiie A, Yamada T, Abe J, Kitamura K.大豆种子脂质含量与亚麻酸浓度的遗传关系。品种科学,2008;58(4):361-6。
- 13.
刘建军,刘建军,刘建军,等。大豆种子油酸含量的数量性状位点定位与比较。作物科学,2009;49(2):433-42。
- 14.
祁忠,吴强,韩旭,孙艳,杜鑫,刘超,姜辉,胡刚,陈强。大豆油含量QTL定位及整合meta分析方法挖掘基因。Euphytica。2011;179(3):499 - 514。
- 15.
孙艳,栾华,齐忠,单丹,刘超,胡刚,陈强,齐忠,吴强,韩旭,孙艳,杜鑫,刘超,姜辉,胡刚,陈强,单丹。大豆含油量综合数量性状位点图谱大豆(l)稳定。,generated using a meta-analysis method for mining genes. Euphytica. 2011;179(3):499–514.
- 16.
毛涛,姜志,韩勇,滕伟,赵鑫,李伟。大豆种子蛋白质和油脂含量的数量性状位点在多遗传背景和环境中的定位。植物育种,2013,32(6):630-41。
- 17.
Pathan S, Vuong T, Clark K, Lee J, Shannon J, Roberts C, Ellersieck M, Burton J, Cregan P, Hyten D, Nguyen H, Sleper D.大豆籽粒蛋白质、油脂含量和籽粒质量性状位点的遗传定位和确定。作物科学,2013,53(3):765-74。
- 18.
哈波,金辉,Velusamy V, Vuong TD, Nguyen HT, Shannon JG, Lee J. PI483463控制亚麻酸浓度的数量性状位点的鉴定(甘氨酸大豆).理论与应用,2014;127(7):1501-12。
- 19.
金正日H, Y,金正日年代,儿子B,崔Y,康J,公园Y,曹Y,崔我分析(法)对种子大小数量性状和脂肪酸成分在大豆中使用重组自交系。生命科学学报,2010;20(8):1186-92。
- 20.
大豆基地:整合遗传学和基因组学以推进大豆研究。爱荷华州立大学,艾姆斯,2017。https://www.soybase.org/.2017年10月10日访问。
- 21.
Csanadi G, Vollmann J, Stift G, Lelley T.早熟大豆种子品质qtl的分子图谱分析。理论与应用,2001;103(6):912-9。
- 22.
王鑫,姜刚,格林明,Scott R,宋强,Hyten D, Cregan P.大豆重组自交系产量、油脂和蛋白质含量数量性状位点的鉴定与验证。生物医学工程学报,2014;39(5):935 - 949。
- 23.
韩勇,滕伟,王勇,赵鑫,吴磊,李东,李伟。大豆种子异黄酮与蛋白或油含量遗传关系的无条件和条件QTL分析。植物育种,2015;134(3):300-9。
- 24.
Reinprecht Y, Poysa V, Yu K, Rajcan I, Ablett G, paul K.低亚麻酸无脂氧合酶大豆种子和农艺QTL (大豆(l)美林)种质。基因组。2006;49(12):1510 - 27所示。
- 25.
Patil G, Vuong TD, Kale S, Valliyodan B, Deshmukh R, Zhu C, Wu X, Bai Y, Yungbluth D, Lu F, Kumpatla S, Shannon JG, Varshney RK, Nguyen HT。利用高密度连锁图谱分析大豆种间定位群体中籽粒蛋白、油脂和蔗糖含量的基因组热点。植物学报。2018;16(11):339 - 353。
- 26.
谢东,韩勇,曾勇,常伟,滕伟,李伟。大豆种子中亚麻酸等脂肪酸含量的ssr和snp相关QTL。动物学报,2012;30(1):169-79。
- 27.
王晓明,王晓明,王晓明,等。利用饱和遗传图谱检测标记-QTL连锁及QTL基因效应和定位。遗传学。1993;134(3):943 - 51。
- 28.
Collard BCY, Mackill DJ。标记辅助选择:二十一世纪精确植物育种的一种方法。中文信息学报。2008;32(1):1 - 4。
- 29.
Schmutz J, Cannon SB, Schlueter J, Ma JX, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J,等。古多倍体大豆基因组序列。大自然。2010;463:178 - 83。
- 30.
曹燕,李松,王忠,常峰,孔健,盖军,赵涛。结合连锁与全基因组关联定位鉴定大豆种子含油量主要数量性状位点。植物科学,2017;8:1222。
- 31.
李波,范松,于峰,陈勇,张松,韩峰,严生,王磊,孙军。基于特异位点扩增片段测序的大豆脂肪酸组成QTL高分辨率定位。理论与应用,2017;130(7):1467-79。
- 32.
张艳,李伟,林艳,张丽,王超,徐锐。大豆高密度遗传图谱的构建及qtl定位(英文)大豆)农艺性状和种子品质性状的特异长度扩增片段测序。生物医学工程学报,2018;19:641。
- 33.
李艳,赖家杰,马艳,洪华,刘志,常锐,邱玲。大豆脂肪酸形成复杂分子遗传学的靶向关联图谱研究。生物医学工程学报,2015;16:841。
- 34.
Bandillo N. Arquin D, Song Q, Nelson R, Cregan P, Specht J, Lorenz a.美国农业部大豆种质资源群体结构及全基因组关联分析。植物学报,2015;8(3):1-13。
- 35.
Sonah H, O 'Donoughue L, Cober E, Rajcan I, Belzile F.大豆8个农艺性状的GBS-GWAS定位及QTL定位验证。植物学报;2015;13(2):211-21。
- 36.
Leamy LJ,张宏,李超,陈云云,宋波。野生大豆种子组成性状的全基因组关联研究(甘氨酸大豆).BMC Genomics. 2017;18:18。
- 37.
吴马方C, Y,年代,刘Z,王Z,杨R, G,周Z, Yu H,张M,潘Y,周G,任H, Du W,燕H,王Y,汉族D,沈Y,刘,刘T,张J,秦H,元J,元X,香港F,刘B,李J,张Z,王G,朱B,田Z全基因组关联研究解剖基因网络的底层在大豆农艺性状。生物医学工程学报,2017;18(1):161。
- 38.
张健,王旭,陆勇,Bhusal SJ,宋强,Cregan PB,颜勇,Brown M,姜刚。大豆种子组成全基因组扫描研究为大豆品质改良及驯化育种提供了新的视角。植物学报,2018;11(3):460-72。
- 39.
刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军,刘建军FAD2大豆基因家族:对古多倍体基因组结构和功能分化的认识。植物学报,2007;47(1):514 - 526。
- 40.
Bilyeu KD, Palavalli L, Sleper DA, beuselink PR.三个微体omega-3脂肪酸去饱和酶基因与大豆亚麻酸水平有关。作物科学,2003;43(5):1833-8。
- 41.
Andreu V, Lagunas B, Collados R, Picorel R, Alfonso M.GmFAD7大豆基因家族:FAD7酶参与去饱和酶活性的新基因和组织特异性构象的鉴定。[J] .生物医学工程学报,2010;61(12):3371-84。
- 42.
黄晓明,黄晓明,黄晓明。大豆种子中3-酮酰基acp合成酶II基因突变与棕榈酸水平升高有关。作物科学,2006;46(6):2453-9。
- 43.
Head K, Galos T, Fang Y, Hudson K.大豆3-酮酰基acp合成酶基因突变与种子棕榈酸水平高相关。动物学报,2012;30(3):1519-23。
- 44.
Roesler K, Shen B, Bermudez E, Li C, Hunt J, Damude HG, Ripp KG, Everard JD, Booth JR, Castaneda L, Feng L, Meyer K.大豆1型二酰基甘油酰基转移酶的改良变体提高了大豆的含油量,降低了可溶性碳水化合物含量。植物生理学报,2016;17(1):878 - 93。
- 45.
Dehesh K, Tai H, Edwards P, Byrne J, Jaworski JG。3-酮酰基酰基载体蛋白合成酶IIIs在植物体内的过度表达降低了脂质合成的速率。植物生理学报。2001;25(5):1103 - 1104。
- 46.
卢鑫,李强,熊强,李伟,毕勇,赖勇,刘鑫,满伟,张伟,马斌,陈生,张杰。大豆种子发育过程中转录组特征揭示了种子性状驯化的遗传基础。植物学报,2016;36(6):563 - 564。
- 47.
王华伟,张斌,郝玉军,黄军,田爱岗,廖勇,张建军,陈世义。大豆dof型转录因子基因;GmDof4和GmDof11,提高转基因拟南芥种子的脂质含量。植物学报,2007;32(4):716 - 729。
- 48.
宋启祥,李秋琴,刘云峰,张飞飞,马波,张文坤,满文奇,杜文伟,王国光,陈世义,张建军。大豆GmbZIP123基因提高转基因种子的脂质含量拟南芥植物。实验学报,2013;64(14):4329-41。
- 49.
刘云峰,李秋琴,卢鑫,宋启祥,林思敏,张文坤,马斌,林琴,满文奇,杜文武,水辉,陈世义,张建军。大豆GmMYB73促进转基因植物的脂质积累。植物学报,2014;14:73。
- 50.
Kabelka EA, Diers BW, Fehr WR, LeRoy AR, Baianu IC, You T, Neece DJ, Nelson RL。大豆引种增产的假定等位基因。作物科学,2004;44(3):784-91。
- 51.
Mansur LM, Lark KG, Kross H, Oliveira A.大豆生殖、形态和种子性状数量性状位点的区间定位大豆l .)。理论与应用,1993;26(8):907-13。
- 52.
梁辉,于燕,王松,连燕,王涛,魏燕,龚萍,刘翔,方翔,张明。大豆异黄酮、油脂和蛋白质含量QTL定位(英文)大豆l .稳定)。中国农业科学,2010;9(8):1108-16。
- 53.
田志强,田志强,田志强,等。大豆种子蛋白质和脂肪含量的定量性状位点分析。品种科学,2003;53(2):133-40。
- 54.
Chung J, Babka HL, Graef GL, Staswick PE, Lee DJ, Cregan PB, Shoemaker RC, Specht JE。大豆籽粒蛋白、油脂与产量的QTL分析[j] .作物科学,2003;43(3):1053-67。
- 55.
王鑫,蒋国良,格林M, Scott RA, Hyten DL, Cregan PB。大豆重组自交系饱和脂肪酸数量性状位点分析。动物学报,2012;30(2):1163-79。
- 56.
李辉,赵涛,王勇,于东,陈松,周锐,盖军。大豆含油量和脂肪酸组分的加性QTL、上位QTL对和未定位的次要QTL组成的遗传结构。Euphytica。2011;182:117-32。
- 57.
Diers BW, Shoemaker RC。大豆脂肪酸含量限制性内切片段长度多态性分析。石油化工学报,1992;69(12):1242-7。
- 58.
Priolli RHG, Campos JB, Stabellini NS, Pinheiro JB, Vello NA。大豆含油量与脂肪酸组分的关联图谱。Euphytica。2015;203(1):83 - 96。
- 59.
奥罗格JB,贾沃斯基JG。脂肪酸合成的调节。植物生理学报。1997;48(1):109 - 109。
- 60.
Chan DI, Vogel HJ。脂肪酸生物合成和酰基载体蛋白的最新研究进展。生物化学学报,2010;30(1):1 - 9。
- 61.
吴国光,薛宏伟。拟南芥β-酮酰基-[酰基载体蛋白]合成酶I是脂肪酸合成的关键酶,在叶绿体分裂和胚胎发育中起重要作用。植物学报。2010;22:3726-44。
- 62.
张建军,张建军,张建军,等。大豆微粒体omega-3脂肪酸去饱和酶基因突变对大豆种子中亚麻酸含量的影响。作物科学,2005;45(5):1830-6。
- 63.
肯尼迪EP。复合脂质的生物合成。联邦公报1961;20:934-40。
- 64.
薛志平,关远平,王立强。大豆二酰基甘油酰基转移酶活性与油脂浓度的关系。石油化工学报,1998;25(7):775 - 781。
- 65.
王晓明,王晓华,王晓华,等。磷酸烯醇丙酮酸羧化酶和丙酮酸激酶在大豆种子发育过程中对蛋白质和油脂合成的影响。作物科学,1989;29(2):349-53。
- 66.
王晓东,王晓东,王晓东,等。大豆种子油合成中丙酮酸激酶复合物的研究进展拟南芥.植物学报。2007;19:06 - 22。
- 67.
Elahi N, Duncan RW, Stasolla C.种子油产量下降FUSCA3甘蓝型油菜突变体植物。植物生理学报。2015;26(6):522 - 522。
- 68.
Manan S, Ahmad MZ,张刚,陈波,Haq BU,杨军,赵军。大豆LEC2调控种子贮藏物质生物合成、分解代谢和种子发育相关基因亚群。植物科学,2017;8:1604。
- 69.
孟德斯A,凯利AA, van Erp H, Shaw E. Powers Sj, Kurup S, Eastmond PJ。bZIP67调节ω -3脂肪酸含量拟南芥激活种子油脂肪酸去饱和酶.植物学报,2013,25(8):3104-16。
- 70.
Fehr WR, Caviness CE, Burmood DT, Pennington JS。大豆发育阶段说明;大豆(l)。美林。作物科学,1971;11(6):929-31。
- 71.
魏峰,高刚,王鑫,董鑫,李鹏,华伟,王鑫,吴鑫,陈辉。超声辅助提取-气相色谱法定量测定少量油菜中油脂含量。超声与光化学,2008;15(6):938-42。
- 72.
邢广宁,周波,王云峰,赵铁军,于达元,陈世云,盖建勇。大豆pypy病抗性的遗传成分及主要QTLLamprosema指示大豆4个RIL群体在多种环境下的遗传多样性。理论与应用,2012,25(5):859-75。
- 73.
李建平,李建平,李建平,等。不同种群间水稻产量的生物统计学研究。刘志强。2006;22(1):1 - 6。
- 74.
孙晓伟,刘元达,张晓峰,李文博,刘宏,洪文文,蒋宝成,关宁,马彩霞,曾惠萍。slf -seq:一种大规模从头发现SNP和使用高通量测序进行基因分型的有效方法。科学通报,2013;8(3):583 - 587。
- 75.
Van Ooijen JW。joinmap4:用于计算实验群体遗传连锁图谱的软件。瓦赫宁根:Kyazma BV,荷兰。2006。
- 76.
从重组值估计地图距离。[j] .中国生物医学工程学报。2009;12(1):1 - 5。
- 77.
刘丹,马超,洪伟,黄磊,刘敏,刘华,曾华,邓东,辛华,宋健,徐超,孙晓,侯晓,王晓,郑华。基于高通量测序数据的高密度连锁图谱构建与分析。科学通报,2014;9(6):98855。
- 78.
王胜,巴斯滕CJ,曾志斌。Windows QTL制图器2.5版。统计学系。罗利:北卡罗来纳州立大学;2012.
- 79.
Voorrips RE. MapChart:用于链接图和qtl的图形化表示的软件。[J] .中华医学杂志。2002;30(1):1 - 8。
- 80.
Asekova S, Kulkarni KP, Kim M, Kim JH, Song JT, Shannon JG, Lee JD。大豆皮483463与‘Hutcheson’杂交饲料品质性状的新数量性状位点。作物科学,2016,56(5):1-12。
- 81.
杜志,周霞,凌云,张志,苏志。agriGO:面向农业社区的GO分析工具。核酸学报,2010;38(S2): 64-70
致谢
我们非常感谢吴丹博士编辑了这篇手稿草稿的英文文本。我们也感谢LetPub (www.letpub.com),感谢其在编写本手稿期间提供的语言帮助。
资金
国家重点研发计划项目(No. 2016YFD0100504)、国家自然科学基金项目(No. 31522042和31371654)、国家转基因项目(No. 2016ZX08004003)、湖北省自然科学基金项目(No. 2016CFA049)、中国农业科学院项目(No. 2016CFA049)资助。Y2017JC14),河南农业大学。资助机构的作用是提供财政支助。资助者在研究设计、数据收集和分析、发表决定或手稿准备方面没有任何作用。
作者信息
从属关系
贡献
y.y.和Y.J.设计了这项研究并撰写了这篇文章。秦永远、郭广德、纪荣、苏正成、纪中、肖立、尚哲进行了数据分析和实验。Y.J.协调了这项研究。所有作者都阅读并批准了最终稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者没有竞争的经济利益。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
补充信息
附加文件1。
四种环境下种子油含量和成分的频率分布。的箭头表明两个亲本系的性状。野生大豆ZYD00463为父本(P1),栽培大豆WDD01514为母本(P2)。四种不同环境下,种子含油量的频率分布如图(A-D)所示,棕榈酸(E-H)、硬脂酸(I-L)、油酸(M-P)、亚油酸(Q-T)和亚麻酸(U-X)。
附加文件2。
四种环境下种子油含量和成分的Pearson相关系数。
附加文件3。
SNP标记构建高密度遗传图谱。x轴和y轴分别表示连锁群数和遗传距离(厘米,cM)。
附加文件4。
大豆参考基因组中20个连锁群的SNP标记位置及其对应的物理位置(bp)。
附加文件5。
高密度遗传图谱中20个LGs的特征描述。
附加文件6。
单一环境下与油脂含量和成分相关的添加剂qtl。
附加文件7。
跨环境遗传连锁图谱上稳定加性qtl的定位。qtl用条形标记。柱长表示QTL的物理间隔。稳定的qtl以不同颜色条表示。红酒吧:含油量;绿色的酒吧:棕榈酸;蓝色酒吧:硬脂酸;黄色的酒吧:油酸;淡红色条:亚油酸;浅绿色条亚麻酸。
附加文件8。
以前报道的大豆籽油含量和成分qtl信息。
附加文件9。
24个稳定QTL区间内参与脂质代谢的候选基因。
附加文件10。
24个稳定QTL区间内参与脂质代谢转录因子的候选基因。
附加文件11。
以前的GWAS检测到的大豆油含量和成分QTNs。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
姚旸,游强,段刚。et al。野生与栽培大豆种子含油量及组成的数量性状位点分析。BMC Plant Biol20.51(2020)。https://doi.org/10.1186/s12870-019-2199-7
收到了:
接受:
发表:
关键字
- 大豆(大豆)
- 油和脂肪酸
- QTL
- SLAF-seq