摘要
背景
桑树是蚕业中养桑蚕最重要的宿主。通过传统的育种方法,培育出了优良的桑树品种。然而,在这些品种的分子特性方面进行的工作并不多。基于聚合酶链反应的随机扩增多态性DNA (RAPD)和小卫星DNA定向扩增(DAMD)方法是分析桑葚遗传多样性的重要手段。这些已经被用来确定9个品种之间的差异桑属国家植物研究所班斯拉研究站,勒克瑙。
结果与讨论
使用用于RAPD的23个任意序列抗溶剂引物分析品种,以及用于该损伤反应的3个小卫星核心序列引物。RAPD和DAMD频段数据(总共200频段)用于根据JACCARD算法确定一对明智的距离。来自这些距离值邻居加入(NJ)分析分别用于RAPD和DAMD数据。发现三倍体品种使用RAPD分析彼此最相似,而使用该DAMD分析更类似的品种S13和S34。近85%的RAPD频段和91%的DAMD带是九个品种的多态性。
结论
利用RAPD和DAMD图谱对桑树品种进行了鉴定。多达五个RAPD引物和一个DAMD底漆产生的曲线可以一起区分所有九个品种的独特频带。
背景
桑(桑属SPP。),是蚕豆中最重要的作物植物作为桑蚕蚕(家蚕都是用它们养大的。桑葚是桑科的一组小乔木或灌木,分布在北半球的温带和亚热带地区。在印度一般有六种,分别是:m·阿尔巴L.(2x,2n = 28),M. indica.lm个roxb。,m .黑质lm . serrataRoxb。(6x, 2n = 84m . laevigata墙。(4x, 2n = 56) [1那2].然而,在物种识别和丹丁明的情况下一些混淆[3.]前三种物种应被视为同义词或品种m·阿尔巴其余三种应作为不同种保留。在过去的40年里,一些物种也从中国和日本引入印度,主要用于养蚕。
桑树熊在同一植物(单一的)或不同植物(雌雄同体)上的男性,女性和双性恋的花朵,往往取决于几种生理和生化因素的表达[4.].桑树的基因型已经评估了许多参数,但很少做的工作对桑树DNA的特性。利用RAPD技术确定了桑树品种、二倍体和多倍体的遗传特性和亲缘关系[5.-7.].香等等。[8.也将RAPD技术应用于该属的系统学研究桑属而这些研究主要集中在不同物种间的差异桑属关于桑树存在的物种内遗传变异很少。此外,在印度生长的桑椹植物中没有报道分子工作。
近年来,基于dna的直接比较两株植物遗传物质的标记被广泛使用。RAPD技术[9.那10],提供了一种方便、快速的评估相关个体遗传组成差异的方法,已被大量应用于植物的遗传多样性测定和评估。另一项技术,小卫星DNA定向扩增(DAMD)是由Heath开发的等等。[11]并揭示由于小型卫星引起的多态性。由于该技术在较高的PCR严格中进行,因此产生的模式具有比RAPD更大的再现性。在本文中,我们将结果从RAPD和DAMD PCR分析中的应用报告,以确定含有九种桑树的差异。这些包括三倍体和选择对易患的二倍体品种。
结果
发现从九个品种分离的DNA是通过琼脂糖凝胶电泳测定的高分子量。这些DNA用于RAPD和DAMD反应。RAPD条件在试验实验中优化,不同浓度的MG2+离子,引物和DNA(数据未显示)。同样,对于三种DAMD底漆,退火温度在50°C到55°C之间变化,以确定最优温度(数据未显示)。为毫克2+滴定,0 mM, 1.5 mM, 2.5 mM, 3.5 mM和4.5 mM氯化镁2被包括在每个反应。类似地0,5,10,15和引物20皮摩尔和0,12.5,DNA模板25,50,100和200纳克。还测试扩增。通常,在Mg的情况下2+离子,由于反应强度的增加,浓度的降低导致很少或没有带。引物浓度增加导致条带数增加。然而,DNA浓度的变化,每次反应至少使用200 ng的DNA,并没有导致图谱的差异。在这些结果的基础上,1.5 mM氯化镁2, 10 pmol的引物和50 ng的模板DNA被认为是产生一致的桑树RAPD图谱的最优条件,因此,在与所有RAPD引物的所有后续反应中都使用。对于DAMD PCR,我们发现退火温度为50℃时,三种引物都能产生不同的谱线。
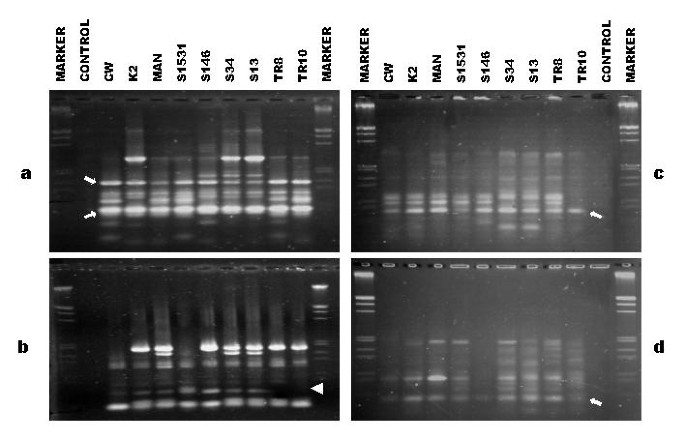
为全面了解桑树品种间的变异情况,采用RAPD和DAMD技术对桑树品种间的变异进行了分析。RAPD反应使用40个任意序列十聚体作为引物。其中3种引物没有给出任何谱图,23种引物(见表)1)给出了一致的(当用至少三种不同的基因组dna制备方法复制这些引物的图谱时)图谱。分别考虑23条引物的RAPD和3条引物的DAMD数据,分析不同品种间的关系。利用RAPD引物OP-G16和OP-G5、DAMD引物33.6和HVR得到的9种桑树dna图谱如图所示。1(分别为A-D)。由这些引物产生的碎片从500-4200个碱基的大小而变化。共有177条带在RAPD型材(其中26%或44条带单态),而23条带中DAMD型材(其中9%或2条为单态)得分进行评分。条带被认为是单态由于相同大小的片段中的所有品种产生的(在图的小箭头所指示的。1 a、c、d).从RAPD和DAMD波段数据计算成对距离,这些值如表所示2.RAPD方法中,品种CW与S34之间的距离最大(0.44),三倍体品种TR8与TR10之间的距离最小(0.24)。同样,在DAMD方法中,S146与两个品种S13和S34之间的距离最大(0.78),S34与S13之间的距离最小(0.13)。
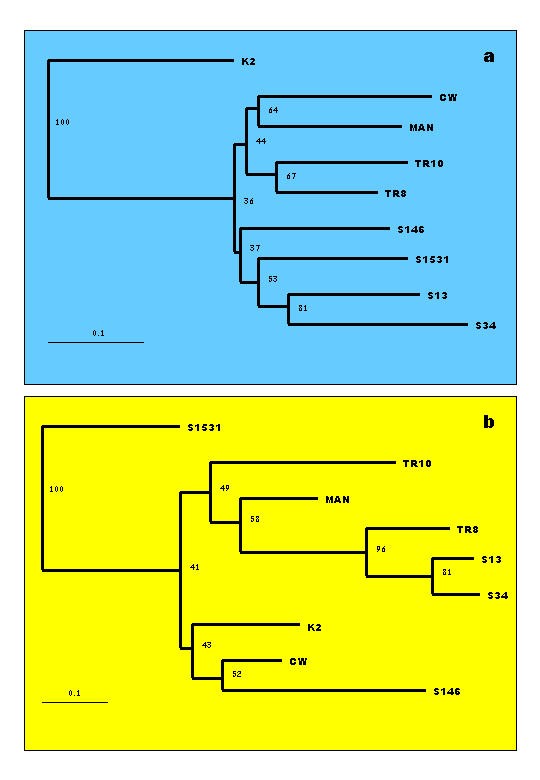
的距离数据被用于产生用于两种方法的NJ树木进行500复制自举试验后。用于RAPD方法NJ树图中给出。2DAMD方法的计算结果如图所示。2 b.在这两种情况下,变种被分组成簇,这样树的拓扑结构对于一些变种簇是大致相似的。在RAPD方法中,NJ树显示两个三倍体品种TR8和TR10之间的距离最近,而K2与其他所有品种之间的距离一般是最远的(图2)。2).将RAPD数据进行500个重复的bootstrap试验,得到432个可能的树。其中最常见的树将本地品种(K2)、外来引种品种(CW和MAN)、三倍体品种(TR8和TR10)和选择品种(S13、S34、S146和S1531)归为单独的簇。结果表明,RAPD方法具有较好的分辨力。由DAMD数据生成的NJ树与RAPD数据生成的NJ树不同。在DAMD数据中,品种S13和S34最接近,而三倍体品种分组不同。有500个重复的自举测试产生了374棵可能的树。其中最常出现的树清晰地显示出上述品种的聚类(图。2 b).
利用RAPD和DAMD数据对桑树品种进行聚类分析。用(a) RAPD方法和(b) DAMD方法得到的NJ树分别用蓝色和黄色图板显示系统图。分叉处的数字是引导百分比值,而每个面板左下角的刻度是基于表中的值的距离刻度2.该品种名称缩写和指示给树木的权利。
讨论
大量的桑树品种在印度的几个地理区域都为他们的食用水果和丝绸产业发展。因为它可以最终对丝绸生产和质量有直接影响的优良品种或承诺桑树品种的鉴定是很重要的。之一的由这些可识别的重要途径是筛选范围和存在于它的遗传多样性的程度桑种质。在最近的时间的种质的这种分析也已进行了使用分子技术,包括RAPD和其他基于PCR的筛选技术如DAMD。与RAPD的帮助下,已被检测的遗传变异,包括,内和植物物种之间。12-14].DAMD-PCR已应用于几种植物的指纹鉴定,包括酵母和真菌[15-18)、小麦(19那20.], 白饭 [21],常见的豆[22]和新人[23].
鉴于此,决定采用RAPD和DAMD技术对桑树等重要植物进行遗传变异分析。选用的9个品种包括2个本地三倍体(TR8、TR10), 4个本地选种(S13、S34、S146和S1531),优秀的本地品种Kanva-2 (K2)和2个外来品种China White (CW)和Mandalay (MAN)。RAPD数据bootstrap测试后的NJ树(图。2)显示出品种TR8和TR10的亲缘关系。此外,当所有选择品种组合在一起时,MAN和CW品种也更接近三倍体品种而不是其他品种(图2)。2).另一方面,NJ树对DAMD数据进行bootstrap检验后发现,品种S13、S34和TR8之间的距离最小,4个选择品种相对于RAPD分析的分组不同。这些品种的分组反映了它们负责RAPD或DAMD谱的基因组区域的相对相似性。其中,后者(DAMD)序列是通过比RAPD序列更严格的PCR条件获得的,因此,在DAMD序列中,品种之间的密切性归因于它们的小卫星序列家族的相似性。
RAPD分析结果表明,该品种可分为选择性品种(S13、S34、S146、S1531)、三倍体品种(TR8、TR10)、外来品种(CW、MAN)和本地品种K2。另一方面,DAMD配置文件导致了不同的聚类。然而,这两种技术并没有显示出与地理来源有关的品种聚类的任何特定偏差。这说明不同品种的RAPD和DAMD分布没有反映出地理差异。在对9个不同参数进行多元分析的基础上,早前也已经表明,地理多样性不一定是桑树遗传多样性的直接原因[24].
三倍体品种的RAPD谱与所有二倍体品种的RAPD谱不同。其中MAN与三倍体品种TR8和TR10的基因图谱比较相似。两个三倍体品种与MAN的距离分别为0.27和0.31,而它们之间的距离仅为0.242, 图。2).三倍体与其他二倍体品种的距离在0.28 ~ 0.41之间。与二倍体相比,三倍体中发现的条带数量并不是特别多,尽管它们有一组额外的染色体。相反,在引物OP-G5的情况下,观察到三倍体品种的dna特异性缺失了一个在所有其他二倍体品种中都存在的条带(如图中小箭头所示)。1 b).就甘蔗而言,Harvey和Botha [25]没有观察到多倍体相对于二倍体RAPD带任何更大数量。在四个选择土著的情况下,各品种的通过两种方法的聚类表明,S34和S13是(由DAMD方法至少距离0.13)最彼此相似。该品种CW和K2似乎是从其他品种更遥远并且不同的相对于他们通过这两种方法(RAPD和DAMD)集群。
结论
多态性的总体程度非常高,RAPD和DAMD数据在区分九个品种之间是有用的,尽管包括显然不同的元素,如三倍体和异国情调的品种。多达五个RAPD引物和一个DAMD底漆产生的曲线可以一起区分所有九个品种的独特频带。这些结果与桑椹已知具有高度杂合品种的事实一致,在英式桑椹父母之间具有大量的天然杂交种[26].支持这一观点的是,多达58个桑树品种的遗传差异被发现很高,如D2多元分析的统计数字[27].本研究中两种方法观测到的最大距离(0.78)和最小距离(0.13)(见表)2)表明,品种相似度在22 ~ 87%之间。两种方法分别显示85% (RAPD)和91% (DAMD)多态性条带。对于一个品种间有广泛的自然杂交和据报道的高遗传差异的植物,这些值因此表明了广泛的多样性。另一种解释是桑树是最近才从它的祖先分类群中分离出来的。然而,目前只有9个品种的数据不足以真正阐明这两种可能性。然而,本研究的一个重要成果是只用少量的RAPD和DAMD引物就能区分不同的桑树品种。显然,这些技术在桑树全种质资源分析中有相当大的应用范围。
材料和方法
植物材料
9个不同的桑树品种,即中国白(CW)、Kanwa2 (K2)、曼德勒(MAN)、S1531、S146、S34、S13、三倍体-8 (TR8)和三倍体-10 (TR10),从NBRI在勒克诺郊区Banthra的生物量研究中心保存的桑树中选择。
DNA的隔离
根据de Kochko和Hamon的方法从新鲜嫩叶中分离出植物总DNA [28]作了一些修改[29].从每一株植物的叶片组织中至少制备了3个独立的DNA。通过比较琼脂糖凝胶上的条带强度,以及使用Hoechst 33258作为荧光色素的荧光测定法(DyNA Quant 200, Pharmacia)来估计DNA样品的数量和质量。
使用RAPD引物的聚合酶链反应
使用试剂盒B, F, G和AP (Operon Technologies Inc., Alameda, California, USA)的十聚体作为引物。DNA基本上是按照威廉姆斯的方法扩增的等等。[9.].首先对不同引物、模板DNA和Mg进行了初步实验2+离子浓度。最终扩增反应包括10 mM的水龙头(pH 8.8), 1.5 mM的氯化镁2, 50 mM KCI, 0.01%明胶,0.2 mM每dNTP, 10 pmol底漆,0.6 UTaqDNA聚合酶(Bangalore Genei, Bangalore, India)和50 ng桑葚DNA模板在25 μl反应体积。在热循环器(Robocycler 40, Stratagene GmbH, Germany)中,在94°C 1 min、35°C 1.5 min和72°C 1.5 min下循环44次。此外,最后一个延长周期允许在72°C孵育5分钟。扩增产物在0.5X TBE缓冲液中,1.0%琼脂糖凝胶电泳(25 mA恒流)分离,Nighthawk可视化成像™用溴化乙锭染色后凝胶文件系统(PDI Inc.,USA)。
利用微卫星核心序列引物的聚合酶链反应用于微卫星DNA的定向扩增
DAMD-PCR基本上按周进行等等。[21].该技术涉及在扩增反应中单独使用微卫星核心序列作为引物。其他工作者也采用了DAMD-PCR技术来确定种间和种内变异[15-23].通过在50℃和55℃两种不同退火温度下进行DAMD-PCR,确定了最佳退火温度。PCR参数如下:第一个周期在95°C的孵化2分钟紧随其后的是35的孵化周期为2分钟95°C,在退火温度为1分钟和72°C 1.5分钟。最后周期允许一个额外的孵化在72°C 5分钟。反应含有100 ng模板DNA, 40 pmoles的底漆,2.5毫米毫克2+离子,200 μM每个dNTP, 0.3 UTaqDNA聚合酶在1×检测缓冲液中,最终体积为25 μl。PCR产物通过1%琼脂糖凝胶电泳分离,并按RAPD图谱进行分析。
数据分析
数据(所有扩增产物的片段大小,通过与标准分子量标记物λ DNA双酶切进行比较,从凝胶中估计后三世和生态RI)记为离散变量,使用“1”以指示存在和“0”以指示不存在一个频段。基因型之间的距离的成对基质用于RAPD(23个引物)和DAMD(3个引物)使用的数据的Jaccard的算法[单独地确定30.]在RAPDistance包[31].根据Saitou和Nei [32],使用freetreree软件包[33]及TreeView [34].
缩写
- 小卫星DNA的DAMD定向扩增:
-
NJ -邻居加入
- RAPD:
-
随机扩增多态性DNA。
参考文献
- 1.
丹丁斯,苏珊·鲍特,兴高采烈的MS,Mallikarjunappa rs,Giridhar K:桑椹氏菌的几种桑椹(桑属属)。印度J.赛瑞。1987年,26:22-31。
- 2.
Basavaiah L,丹丁SB,拉詹MV:孢子六倍体桑属serrataRoxb。细胞学杂志,1989,54:747-751。
- 3.
Dandin SB:促进热带地区可持续生产力的桑树改善项目。见:桑蚕养殖的进展(编辑MC Devaiah, KC Narayanaswamy, VG Maribashetty), CVG出版物,班加罗尔,印度。1999年,51 - 87。
- 4。
生长素:赤霉素平衡-它在桑树性别表达的决定中的作用桑属属)。印度J.赛瑞。1994年,33:188-190。
- 5。
F . Lichun F ., Guangwei Y ., Maode Y ., Yifu K ., Chenjun J ., Zhonghuai Y .:桑树栽培种遗传特性及亲缘关系的研究(桑属通过随机扩增多态性DNA分析。《浙江科学》1996,22:139-
- 6。
娄春芳,张永忠,周建明:桑树亲本及其杂交后代基因组DNA多态性研究桑属。草业科学。1998,38:437-445。
- 7。
张Y,Chengfu L,Jinmei Z,Hongzi Z,Xiaoming X:桑树基因组DNA的多态性研究。浙江农业大学学报。1998年,24:79-81。
- 8.
向智,张智,于敏:RAPD在植物系统学中的应用初报桑属《残业科学》,1995,21:208-
- 9.
Williams JGK,Kubelik Ar,Livak KJ,Rafalski Ja,Tingey SV:由任意引物扩增的DNA多态性可用作遗传标记。Nucl。酸res。1990,18:6531-6535。
- 10.
Welsh J,McClelland M:使用PCR具有任意引物的指纹基因组。Nucl。酸res。1990,18:7213-7218。
- 11.
Heath DD,Iwana GK,Delvin RH:PCR灌注VNTR核心序列产量物种特定模式和高变探针。Nucl。酸res。1993年,21:5782-5785。
- 12.
Esselman EJ, Crawford DJ, Brauner S, Stuessy TF, Anderson GJ, Silva M: RAPD标记在物种间的多样性和差异Dendroseris(Asteraceae:乳酸)。amer。J. Bot。2000,84:591-597。
- 13.
马立新:一年生杂草的遗传结构狗舌草寻常的与生境类型及种群大小有关。生态学杂志,2001,10:17-28。10.1046 / j.1365 - 294 x.2001.01169.x。
- 14.
李Z,尼尔森RL:来自三个国家的RAPD标记测量大豆种质的遗传多样性。农作物科学。2001,41:1337-1347。
- 15.
Lieckfeldt e,Meyer W,送给者T:DNA和PCR指纹酵母的快速鉴定和分化。J基本微生物。1993年,33:413-25。
- 16。
Meyer W,Mitchell Tg,Freedman EZ,Vilgalys R:常规DNA指纹纹杂交探针用作聚合酶链反应中的单引物,以区分菌株隐性球菌新族裔。j .中国。微生物学。1993,31:2274-80。
- 17。
利用单一引物和简单重复DNA序列分析真菌的聚合酶链反应指纹图谱:菌种的变异隐性球菌新族裔。电泳。1995,16:1648-56。
- 18。
Meyer W, Latouche GN, Daniel HM, Thanos M, Mitchell TG, Yarrow D, Schonian G, Sorrell TC:不完全属病原酵母的鉴定假丝酵母通过聚合酶链式反应指纹。电泳。1997年,18:1548年至1559年。
- 19.
小麦基因组特异性探针的研究进展(英文)Triticum aestivum.L. Em Thell),用小卫星核心序列放大。定理。达成。1996, 93: 982-989。
- 20.
引用本文:Bebeli PJ, Zhou Z, Somers DJ, Gustafson JP: pcr引物的微卫星核心序列产生小麦DNA指纹探针。定理。达成。中国科学:地球科学。10.1007 / s001220050560。
- 21.
Zhou Z, Bebeli PJ, Somers DJ, Gustafson JP:利用VNTR核心序列直接扩增小卫星区DNA选用。定理。达成。中国科学:地球科学。10.1007 / s001220050645。
- 22.
Metais I, Aubry C, Hamon B, Jalouzot R, Peltier D:利用RFLP、DAMD-PCR、ISSR、RAPD和AFLP标记评估大豆遗传多样性。Acta长的矮。(伊什)。2001年,546:459 - 461。[http://www.actahort.org/books/546/54662.htm]
- 23.
利用单引物扩增反应(SPAR)技术评估印楝种源的形态差异。现代分子生物学。2002,3:1-10。[http://www.molbio.net/v3/01/01.pdf]
- 24.
多变量分析在桑树育种中的应用。印度,1997,36:111-115。
- 25.
哈维男,博塔FC:用于DNA多样性之间的确定使用基于PCR的方法中的糖品种。acta physica sinica, 1996, 89: 257-265。
- 26.
山楂属植物的地理分布与探索桑属l .(桑科)。见:桑树的遗传资源和利用(编辑K Sengupta和SB Dandin),中央蚕桑研究和培训研究所(CSRTI),迈索尔,印度,1989年,4 - 7。
- 27。
桑树的遗传分化。《科学》,1998,24:180-185。
- 28。
德Kochko A,阿蒙S:从属植物为restrictable DNA的分离的快速和有效的方法现。植物摩尔。BIOL。代表1990,8:3-7。
- 29。
冉纳德SA,库马尔A,哥斯瓦米男,Farooqui N,神志正常PV:苋菜的基因组分析:测定间和种内变化。J. BIOSCI。1997年,22:457-464。
- 30.
雅卡德:《阿尔卑斯和侏罗山脉一段植物分布比较练习曲》。公牛。Soc。Vaudoise科学。1901年,37:547-579。
- 31.
Armstrong JS, Gibs AJ, Peakall R, Weiler G: RAPDistance包ver1.04。澳大利亚国立大学生物科学研究学院,堪培拉,澳大利亚。1996年,(http://life.anu.edu.au/molecular/software/rapd.htm.]
- 32.
王志强,王志强:系统发育树重建的一种新方法。摩尔。杂志。中国科学:地球科学。
- 33.
在距离数据和树的鲁棒性bootstrap / jackknife分析的基础上构建系统发育树的免费软件程序。在该属RAPD分析中的应用弗雷克利亚。叶形线Biologica(行星)。1999年,45:97 - 99。[http://www.natur.cuni.cz/~flegr/freeTree.htm.]
- 34.
TREEVIEW:在个人电脑上显示系统发生树的应用程序。计算机科学与应用。1996,12:357-358。[http://taxonomy.zoology.gla.ac.uk/rod/rod.html]
- 35.
Jeffreys AJ, Wilson V, Thein SL:人类DNA中的高可变小卫星区域。《自然》1985,314:64-72。
- 36.
中村Y,莱珀特男,Mortein C,藤本E,霍夫男,Kumlin E,白K:串联重复(VNTR)标记为人类基因作图的可变数目。科学。1987年,235:1616年至1622年。
- 37.
Winberg BC,Shori Z,Dallas JF,McIntyre Cl,Gustafson JP:小型卫星序列的表征栽培稻。基因组学。1993,36:978-983。
致谢
作者感谢导演NBRI的支持,理事会科学和工业研究(印度)资金支持(SRF),以EB和Bajrang Singh博士,科学家,NBRI,勒克瑙植物材料。我们也感谢裁判的建议有价值的变化,并P.V。博士神志清醒,CSIR名誉科学家,NBRI,对于手稿的批判性阅读。本文是NBRI通信号512。
作者信息
从属关系
相应的作者
作者的原始提交的图像文件
权利和权限
关于这篇文章
引用这篇文章
利用RAPD和DAMD图谱对桑树品种的分子鉴别。BMC植物BIOL.1,3(2001)。https://doi.org/10.1186/1471-2229-1-3
收到了:
公认:
发表:
关键词
- 邻居加入树
- 桑树植物
- 二倍体品种
- 三倍体品种
- 家蚕