摘要
背景
食肉植物Utricularia GIBBA(狸藻属植物)的显著特点是基因组极短,约80兆,大约是其一半拟南芥.狸藻表现出令人难以置信的形式的多样性,围绕着一个明确的主题:微小的,膀胱似的吸力陷阱陆地,附生,或水生植物与不同寻常的营养形式的多样性。Utricularia无根植物的生理特征(呼吸和碳分布)也异常,叶绿体、线粒体和核糖体序列的分子进化速率也高度提高。尽管对该属有极大的兴趣,但没有基因组资源存在Utricularia,替代率增加已获得有限的研究。
结果
在这里,我们描述了对的测序和分析Utricularia GIBBA转录组。调查了三种不同的器官:诱捕器、营养茎体和花序茎。我们还检测了不同胁迫条件下膀胱草的转录组。我们详细介绍了功能分类、组织相似性、氮和磷代谢、呼吸、DNA修复和活性氧(ROS)解毒方面的内容。将质体和线粒体基因组的长拼接以及100个单独的核基因序列与其他植物的序列进行比较,以更好地建立分子进化速率的信息。
结论
的Utricularia转录组提供了一个详细的基因组窗口,了解发生在肉食性植物的过程。它包含了一种复杂的代谢途径的深层表征,该代谢途径刻画了一个假定的最小植物基因组的特征,允许它作为基因组信息的来源来探索该属的结构、功能和进化多样性。从转录组的功能分类来看,营养芽和诱捕器是最相似的器官,诱捕器表达被认为是由细菌编码的消化酶。支持生理数据的是,整体基因表达分析表明,诱捕器显著过表达与呼吸有关的基因,磷吸收可能主要发生在诱捕器中,而氮吸收可能部分发生在营养部位。DNA修复和ROS解毒酶的表达可能表明对呼吸增加的反应。最后,来自Bladderwort转录组的证据,直接测量ROS原位并且细胞细胞基因组和多种核基因的交叉物种比较支持的假设,即在整个植物中增加核苷酸取代速率可能是由于扩增的ROS生产的诱变作用。
背景
食肉植物Utricularia及其妹妹属Genlisea.(Lentibulariaceae)分享两个异常的分子进化特征:在所有三个细胞室,线粒体,塑性和核的基因组上增加核苷酸替代率的速率[1- - - - - -4,以及物种甚至种群水平上基因组大小的动态进化[5,6].一些物种,例如Utricularia GIBBA和Genlisea钻进拥有已知的最小的单倍体被子植物基因组,分别约80和60兆(Mb),是其一半或更少拟南芥(拟南芥),并具有细菌尺寸的染色体,这些染色体在物种之间的数量之间广泛变化[5].矛盾的,Genlisea.也包含基因组高达1500 Mb的物种。这些植物具有许多生理和形态特性,是进一步研究与肉食性有关的植物生理学复杂性、陷阱微生物群落的宏基因组调查、新的植物氮/养分利用途径、猎物吸引生态学、植物生物学、植物生物学和植物生物学等方面的主要对象。全植物和陷阱比较发育,最后,最小被子植物基因组的进化[6].
全世界共有214种,Utricularia是肉食植物最大的属性[7].“膀胱草”这个名字是指一种类似于膀胱的吸力陷阱,用于捕获猎物。膀胱在一个主题内有多种形式,它们在物种间的形态与系统发育分类很好地匹配[1].此外,膀胱可以出现在植物叶茂或非叶状结构的几乎每个表面上,以及代替第一胚胎叶[7,8].在生态学上,这个属主要由小型一年生或多年生草本植物组成,它们以三种生命形式存在:大约60%的物种是陆生的,15%是水生的,其余25%是石生植物和附生植物[7].和其他食肉植物一样,Utricularia通常是营养较差的环境中的居民,并通过捕获和利用猎物来补充正常的光致营养营养,通常是水质甲壳类动物,螨虫,螨虫,旋转器和原生动物[9,10.].以前的研究已经证实了从人工喂养的猎物中得到了营养吸收Utricularia[11.,12.],已知有机碳(C)、氮(N)和磷(P)是肉食性植物消化猎物的主要目标[13.].与其他食肉植物相比,从他们的猎物那里获得碳的碳UtriculariaC被分泌到诱捕环境中[14.],表明提供给捕集器的C有利于相关的大型微生物群落,而来自该群落的N和P以类似陆生植物根际相互作用的方式可供植物吸收[14.,15.].接近于零啊2在水生动物的陷阱中Utricularia物种可能决定了可以生活在陷阱内的生物的类型,其中捕获的血液剥夺的捕食性死亡[16.].在各种诱捕腺体和诱捕液中已检测到消化细胞外酶[17.,18.].已经提出,陷阱液中的相当大比例的酶活性来自生活中的共生生物Utricularia膀胱(15.].然而,酶活性的测定不证明它们的起源,其中一些可能在其中组合Utricularia基因组。
尽管对Lentibulariaceae的生物学有相当大的兴趣,但这些食肉植物的基因组数据是可用的。大规模并行454焦磷酸测序已成为一种可行的新转录组测序方法,具有足够的深度和覆盖范围,可以进行定量差异基因表达分析[19.- - - - - -21.],该方法已经被有效地用于不同植物物种的大规模转录组测序[22.- - - - - -24.].目的是确定Utricularia并对其序列进行了详细分析,对185.5 Mpb的序列进行了测序和组装Utricularia GIBBAests。Utricularia GIBBA(Lentibulariaceae)是一种自由漂浮的水生肉食植物,其小基因组约为80 Mbp [5].这项工作提供了对核基因转录物的第一个广泛的调查Utricularia物种,允许评估其生理学和形态的几个假设。我们详细介绍了美国gibba不同器官和植物在生理胁迫下的转录组。特别关注了N和P摄取相关基因的表达,食物消化过程中表达的水解酶相关基因,以及呼吸和活性氧(ROS)产生和清除相关基因的表达。我们还报道了叶绿体和线粒体基因组的初步测序,并提供了分子进化速率的分析。最后,利用分子进化分析和直接实验方法对Albert的假设进行了评价等等。2010 (6,假设活性氧(ROS)来源于细胞色素的特殊作用c氧化酶占DNA修复后的替代率和基因组大小活力的增加。
结果与讨论
基本分析Utricularia GIBBA转录组
从不同器官提取的RNA产生三个cDNA文库美国gibba植物[陷阱:TrpL,芽:ShtL(营养器官),花序:FlwL(生殖器官)]。此外,还从受多种生理胁迫条件的整个植物中生成了cDNA文库(StsL)(详情见方法)。通过两次454焦磷酸测序,对cDNA文库进行了测序。817,792个屏蔽读取被输入到组装过程中(有关屏蔽读取和组装过程的更多信息,请参阅方法)。使用Newbler Assembler软件(v2.5;cDNA管道)获得了高比例的未组装reads(单链);这个部分约占总屏蔽读(未显示的数据)的四分之一。使用由集群/组装过程组成的不同组装方法,绝大多数屏蔽读(88.27%)被合并到contigs中。生成的集群总数为13,122个,组装成16,551个contig,平均每个contig有66.4个读取。contigs的长度范围为0.1 ~ 3.0 kb,平均长度为707.45 bp,表明相当数量的contigs可能代表了全长cdna。 The presence of multiple contigs in a cluster could be due to possible alternative transcripts, paralogy or domain sharing. All reads that did not meet the match criteria to be clustered/assembled with any other reads during the clustering/assembling process were defined as singlets. The total number of singlets was 95,873 (only 11.72% of total masked reads) with an average length of 215.71 nucleotides. Unique transcripts (UT) from美国gibba由16,551个组装的contigs和95,873个singlets组合而成。
美国gibbaUT是通过使用BLASTX对几种可用的全植物基因组中鉴定的蛋白质进行序列相似性搜索来注释的[拟南芥,白杨,蓖麻,葡萄(dicotyledoneous植物),oryza sativa,高粱双色(monocotyledoneous植物),Physcomitrella patens.(苔藓),衣藻reinhardtii,乌西莫曲霉露西瓜(绿藻),全部从RefSeq数据库下载[25.].使用截止的电子值≤10-05年并且有点得分≥45我们发现60,595(54%)美国gibbaUT对至少一种植物蛋白具有较高的同源性。比例高美国gibba由于对先前描述的蛋白质的相似性的可能性高度依赖于查询序列的长度,因此预期没有显着击中(〜46%)。这由Contig与单线术进行说明,对数据库蛋白质;发现Contigs与90%以上的植物蛋白质具有显着相似性,而大多数(55%)单身孔与任何蛋白质没有相似性。也可能很多美国gibbaUT不能被可靠地注释,因为它们代表非翻译区域(UTRs)或非编码rna (ncRNAs)。比较美国gibba但对美国gibba基因组序列(使用Celera组装; [26.,27.])的结果表明,85.2%的转录本对基因组有显著的命中(比对长度为98%,最小序列一致性为完整比对的90%)。剩余的序列可能无法对齐,因为美国gibba基因组目前以一个相对低覆盖率的初步草案代表(~8x, E. Ibarra-Laclette等,未发表数据)。
我们确定了在其中检测到同源性的植物蛋白的比例美国gibbaUT。同源性为43%拟南芥(33,405人中14,382人),38%杨树(42344人中16202人),40%蓖麻(12,494 of 31,221),55%的葡萄属(23493人中13017人),47%选用(12,652 of 26,940),38%高粱(33005人中有12472人),30%Physcomitrella(35936人中的10789人),30%衣藻(14,503人中有4,441人)和47%Ostreococcus蛋白质(7,603的3,621)。美国gibbaUT非常相似,至多,至16,202个独特的植物蛋白(附加文件1,表S1)。此数字表示最严格低估的最小数量美国gibba发现在本研究中抽样的器官和条件中表达的基因。
基因和基因组的京都百科全书(KEGG)分类[28.]来自最好的植物蛋白质与之相关美国gibba以鉴定具有已知功能的蛋白质。每个KEGG类别的最佳命中比例如图所示1.此外,使用KEGG Atlas资源[29.我们创建了一个全球性新陈代谢地图,结合了119个现有途径,对应于Kegg数据库中引用的16,595个基因拟南芥,杨树,血管,蓖麻雀,oryza,高粱,physcometrella,chlamydomonas和Ostreococcus.这个全球代谢图与为美国gibbaUT,可以分配117个不同的代谢路径(附加文件2,图S1)在Kegg地图集中的119种植物代谢途径中。这些结果表明了美国gibbaUT包含了植物基因组特征的复杂代谢途径的深层表征,允许它们作为基因组信息的来源来探索Lentibulariaceae的结构、功能和进化多样性。
的识别美国gibba转录因子(TF)家族
植物对其基因组编码能力的〜7%的蛋白质对调节转录活动进行调节[30.- - - - - -32.].完整的植物基因组序列分析表明,大多数植物基因组中存在60多个转录因子家族。在拟南芥[33.,34.] 和杨树trichocarpa[35.,36.] 64个TF家庭的大小为1-2名成员到100多名成员。米含含有64个Dicot TF系列中的63个[38.,39.]只缺少SAP1系列,这两种基因都代表拟南芥和p . trichocarpa.约3%(3222人)美国gibbaUT具有显著的同源性(BLASTx;创造价值≤10-05年和先前定义的已知TFS的比分≥45)拟南芥[33.,34.],与920个独特的tf相似。我们研究了已知TF科在维管植物中的分布,并在选定的案例中,分析了TF科的复杂性美国gibbaTF家庭相对于其他植物物种中发现的内容。在血管植物中鉴定的64种TF系列中的至少一个成员是在血管植物中鉴定的美国gibbaUT。在其他植物中存在的低拷贝TF家族中,HRT-like、LFY、Whirly、S1Fa-like和VOZ各有1个成员,BBR-BPC、CCAAT-DR1、CPP、GIF和MBF1基因家族各有2个成员,C2C2-YABBY和EIL基因家族各有3个成员美国gibbaUT。只有SAP1,NZZ和ULT TF系列都没有代表美国gibbaUT(附加文件3.,表s2)。
自美国gibba是一种缺乏根源的植物,涉及根部发育的基因已经丢失,有助于降低基因组大小。虽然转录om永远不会是给定基因组中存在的所有基因的完全代表,但有趣的是,我们发现大多数优先表达的TFS和已知参与根部发育,包括同源蛋白质答:芥ARFS 5,7,19,AUX / IAA蛋白3,7,12,14和17;短根和稻草人(GRAS系列的成员)在美国gibba转录组(37.].这一发现暗示了根部缺失的可能性美国gibba可能不是由于涉及根部发展所涉及的基因的优先损失,而是丧失参与制造根部形成所需的基因表达网络的发展方案。
转录丰富度的变化美国gibba转录组
每个器官特异性转录组显着采样,检测每个器官的读数的数量之间的低视差(FLWL,SHTL的234,963,TRPL的292,970读数258,457读数)。从中获得的转录组美国gibba我们的分析也包括了暴露在不同胁迫下的植物(来自持续的光照、黑暗、低温和干旱条件)(StsL代表,140,507 reads)。大部分reads(88.27%)组装成16551个contigs,每个contigs代表一个不同的基因结构。原则上,在一个特定的contig中组装的读本的数量代表了一个特定基因在一个给定的组织样本中产生的mRNA的丰度。然而,转录本丰度的差异可能反映了取样错误,而不是基因表达的真正差异。因此,读计数必须规范化,以允许在不同样本之间进行表达测量的比较,通常的做法是根据库的总数来衡量基因计数[38.,39.].然而,据报道,据报道,基于普通的基于匹配的程序更加普通的程序,并通过QRT-PCR获得的表达模式值更好地相一致40].因此,我们决定规范化R环境中的读计数[41],使用与Bullard前面描述的类似的分位数规范化过程等等。2010年(40,基于前面描述的微阵列归一化方法[42].创建了一个表达式配置文件矩阵(附加文件4包含由contigs(行)和4个标准化转录组(列)表示的16,551个基因中每个基因的reads数量。标准化读计数范围从0-3500。
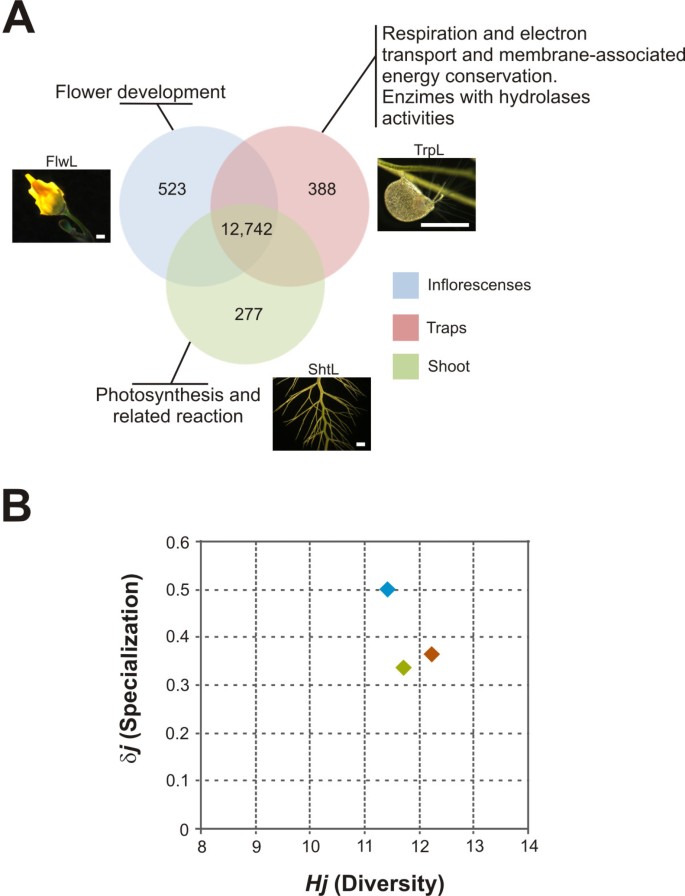
为了评估器官特异性转录组中基因转录本的相对丰度,我们应用了统计学R检验[43].我们认为优先表达基因(PEG)以r≥8(真正的阳性率为〜98%),并且与其他序列组相比,每种器官特异性转录组的读数的最小差异为2倍。共1,181人美国gibbaUT被鉴定为peg;FlwL中有523个,ShtL中有277个,TrpL中有388个,其中一些可以被认为是器官特异性基因,因为这些基因都是由所有的reads构成的美国gibbacontigs来源于单个cDNA组织样本(图)2A和额外的文件5、表S4)。为了识别普遍表达的基因,我们只考虑那些从每个文库中至少读取一次的基因簇。在这种情况下,所有统计测试都需要具有不显著的结果(附加文件6通过比较获得的转录组,鉴定了应激反应基因美国gibba胁迫植物(以StsL表示)与所有器官特定的数据集。按严格程度(R≥8,折叠±2)共200美国gibbaUT被鉴定为响应多种生理应激的差异表达基因(附加文件)7,表S6)。
为了量化器官特定的相似性美国gibba我们使用最近描述的基于香农熵的模型比较了它们的多样性和专门化。多样性(沪江)是通过对香农的转录组频率分布熵公式的调整来衡量的,而专门化(ΔJ.)估计为每个器官中表达的基因的平均特异性[44].这些属性的估计允许识别转录om之间的一般差异,提高对其分布的理解。我们注意到,最专门的器官被取样美国gibba是花序,即使当陷阱,属于属的特征,也是植物王国中最复杂的结构之一,并且是通过哪个器官Utricularia吸引、捕获和消化猎物[10.,45,46].在一个物种多样性相对较低的地区,三个器官类别(茎、花序和陷阱)的多样性测度(图)2B.).然而,根据它们的转录组,芽和陷阱可以被认为是极其相似的器官。然而,这并不奇怪,因为膀胱实际上是“活板门”入口上带有敏感鬃毛的改良过的叶子[45].
差异表达基因的功能分类突出了能量,新陈代谢和水解酶
利用FunCat数据库,根据慕尼黑蛋白质序列分类信息中心(MIPS)将特定器官中的peg按功能类别进行分类[47,48)和一个拟南芥注释如下美国gibbaUT(附加文件8、表S7)。建造一个Venn图表,以显示所选择的超级类别,以及它们在花序,陷阱和射击中的交叉口(图2A).作为这些组织差异表达的一种验证,在花序peg中,MIPS类别“组织分化”通过“花”亚类别显著过度代表(补充表6)。在转录组中选择15个被认为是PEG表达的基因,目的是验证发现的表达模式。总的来说,我们发现了一个很好的相关性(r2= 0.89)9,图S2)。
值得注意的是,在射击和陷阱PEG中,MIPS类别中“能量”的比例过高。在拍摄PEG中,“能量”MIPS类别由“光合作用”和“能量转换和再生”子类别表示,而在捕捉PEG中,这一类别由“呼吸”和“电子传输和膜相关的能量守恒”子类别表示(附加文件)8、表S7)。不出所料,拍出了PEG美国gibba作为SBPase(Sedohepleplulose-Biphastase; AT3G55800)和Rubisco小亚基1B(AT5G38430)注释为在“代谢”MIPS类别中(由子类别'的自身营养有限公司表示2- 修复')(附加文件8、表S7)。这些结果表明,光合作用主要发生在茎部,而呼吸是主要的代谢活动。
关于PEG陷阱,有些美国gibbaUT被标注为水解酶(附加文件)5、表S4)。这些美国gibbaUT为:CL12267contig15708(推定氨基肽酶;类似于AT4G30920), CL3763contig07204(推测的α-葡萄糖苷酶;类似于AT5G11720), CL434contig01978(推测的β-葡萄糖苷酶;类似于AT1G02850), CL6134contig09575(推测的β-己糖苷酶;类似于AT1G65590)和CL851contig02926(假定紫色酸性磷酸酶;类似于AT1G14700)。据报道,从四种水生动物的捕集器收集的液体中也有这五种水解酶的活性Utricularia物种(小叶褐叶藻,南方褐叶藻,金色褐叶藻和美国寻常的)[17.,18.].
氮和磷的吸收美国gibba
氮和磷是植物的两个必需的常量营养素,这通常是陆地和水生生态系统中的植物生长和繁殖的主要限制。植物使用的这些营养素的主要形式是硝酸盐(没有3.-)和磷酸盐(H2宝4-;pi)。已经鉴定了编码转运蛋白和营养采集通道的许多基因并在模型物种中识别和功能性地表征拟南芥和大米(49- - - - - -51].有人提出,从猎物中吸收磷可能比吸收氮更重要[17.].捕集液化学计量(摩尔N:P比约100)以及营养有限的微生物细胞(摩尔N:P比25-61)的存在表明磷而不是氮对营养的重要性Utricularia[15.].此外,在美国寻常的据报道,作为包括陷阱的叶片生物质和叶面积的比例计算的肉毒病变的投资与来自非肉食来源的PI的可用性成反比,而N在肉食中的投资没有显着影响[52].这与猎物对磷的吸收可能比对氮的吸收更重要的观点是一致的Utricularia物种。编码酸性磷酸酶的基因表达量最高Utricularia挂钩(附加文件9Pht1家族中3个高亲和力Pi转运体的编码基因在诱捕器中被鉴定为peg(附加文件)10.,表S8)。由于PHT1家族包含在植物根中强烈表达的高亲和力PI [53- - - - - -58],我们建议在无根UtriculariaPI采用主要发生在陷阱中[8,59].
在高等植物中有两种硝酸盐转运体,分别为NRT1和nrt2(低亲和和高亲和硝酸盐转运体)[60].微阵列实验已用于鉴定硝酸盐/亚硝酸盐同化中涉及的额外基因[61].利用这些信息,我们确定了总共77个美国gibba注释为同源拟南芥参与硝酸盐同化途径的蛋白质(45个成员来自NTR1家族,3个成员来自NTR2和23个硝酸盐/亚硝酸盐同化基因)(附加文件11.、表S19)。这些基因中的大多数被发现在美国gibba的同系物除外拟南芥CHL1基因在芽系peg中被鉴定。CHL1 (AT1G08090)是一种NTR2蛋白,最近有报道称它在植物中起硝酸盐传感器的作用[62].此外,我们发现三个不同的美国gibba注释为δ-尖端(Tonoplast内在蛋白质; AT3G16240)是芽中最高表达的基因。最近被报告为铵(NH)(NH)Δ-尖端(AT3G16240)(NH4)转运体,因为δ-和γ- TIP's(分别为AT3G16240和AT2G36830)补充了酵母中尿素转运体的缺乏[63].狸藻类植物中Utricularia寻常型,据估计,总氮含量的51.8%来自昆虫源氮[12.],然而,食肉植物中动物猎物对氮的贡献是不同的,估计从10%到87%不等,这取决于类群[64].考虑高氨基酸身份(附加文件12.,图S3),从59.2到78.9%Utricularia和拟南芥这些结果表明,在水生植物中Utricularia物种,至少部分地,氮吸收可以在芽(茎/叶)中进行,并且该尿素可能是水生的主要n个来源Utricularia物种。
细胞内基因组块和个体核基因的分子进化率升高
除了转录组的发现之外,我们还测量了塑体和线粒体基因组的大部分Utricularia GIBBA作为我们的一部分Utricularia核基因组测序项目。这为我们提供了前所未有的机会,以评估早期的分子进化率Utriculariaorganellar基因组(1- - - - - -4].从220万年开始美国gibba全基因组霰弹枪(WGS)测序读取(748 MBP,表示估计基因组大小的8倍以上)76,364高质量的读数被鉴定为细胞细胞序列(27.6Mbp)。这些读取使用NewBler汇编程序2.5读数组装,导致来自叶绿体的228个葡萄片和217个葡萄片,其分别具有N50甲簇的线粒体基因组分别为2,146和2,842bp。最大的美国gibba叶绿体contig(长度= 22,577个碱基;FTP:http://www.langebio.cinvestav.mx/utricularia/)对应于部分大的单拷贝区域(LSC;[69,70])。使用多基因组比较和展望工具[65,66],我们从一个细胞器基因组数据库中包含的64个真二联体(玫瑰红和Asterids)被子植物叶绿体基因组中的31个基因组中选择了一个同源区域[67,68,该叶绿体区域共编码28个编码基因。使用GBlocks消除模糊对齐[69,用于识别和删除位置同源性可疑的高度可变的对齐区域(附加文件13.,图S4)。最终的ClustalW对齐[70]包含31个分类单元和8 516个核苷酸特征。对于线粒体基因组,我们使用明确对齐的序列(长度= 4,125个碱基;FTP:http://www.langebio.cinvestav.mx/utricularia/),由4,673个核苷酸的线粒体组成(附加文件14.,图S5)。在这种部分序列中鉴定了总共四种编码基因美国gibba线粒体基因组。由于线粒体基因组的完整序列数有限,使用来自六个eudicot分类的同源区进行系统发育分析。
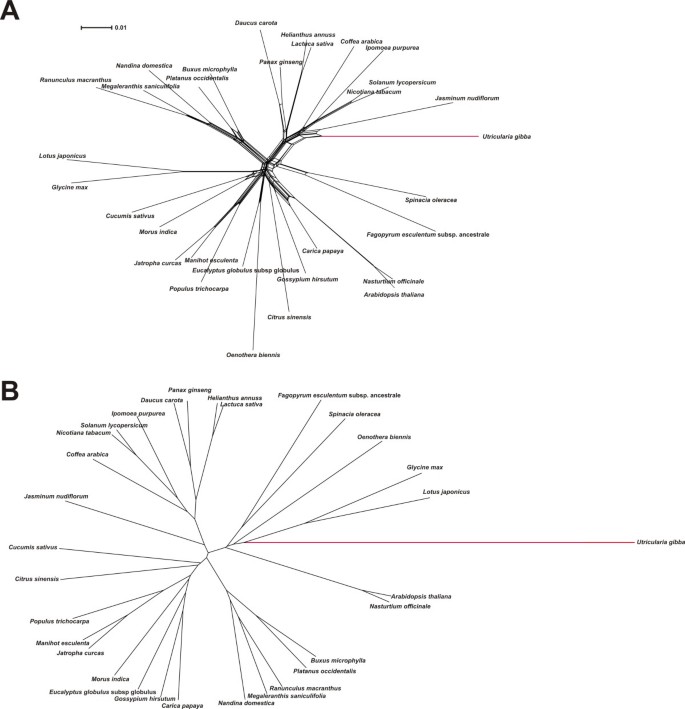
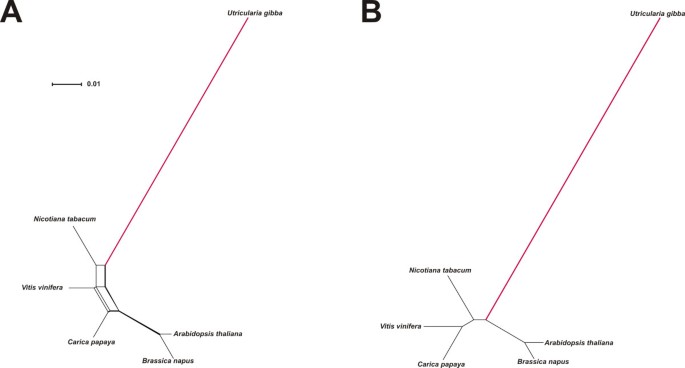
Neignboareet系统发育分析[71用作简单的工具,以说明物种之间的分支长度差异和数据集中的系统发育信号的不协调。分析大块叶绿体LSC序列显示Utricularia GIBBA拥有任何eudicot的最长终端分支(图3A).虽然这个相对比率的差异是轻微的,但在统计上是显著的P< 0.05(使用几个似然模型;参见方法)素馨属(茉莉),属的姐妹属美国gibba,如使用咖啡(咖啡)作为外群体(图3A).升高的进化率美国gibba然而,在一项速率敏感的UPGMA聚类分析中引人注目[72]相同的数据(图3B.).UPGMA假设分子钟在所有物种中同等地运行,因此偏离这一要求在获得的分枝长度方面,可能也在良好的系统发育关系方面,为速率不对称提供了一个有用的测试。因此,质体DNA UPGMA树的位置美国gibba错误地从该物种最接近的小行星类群中分离出来(图3B.).对于线粒体基因组,邻征分析(图4)、相对速率测试(Utriculariavs。烟草,小组葡萄属;P<< 0.001跨几次测试)和UPGMA聚类(图4 b)的现有数据都表明,在中国的替代率大大提高Utricularia.
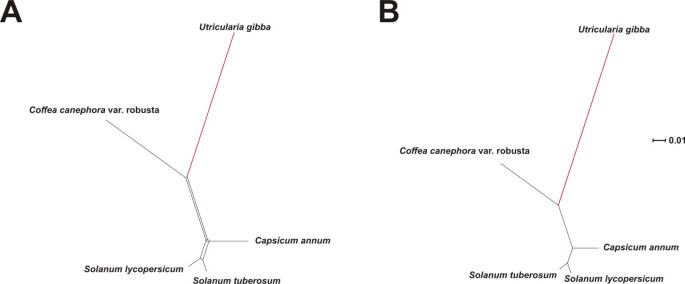
考虑到大量核转录组序列的可用性,我们还分析了一组随机的100个基因的分子进化速率,这些基因与其他几个小行星物种的保守正交基因座(COS II)同源[73- - - - - -75].在这里,我们发现了美国gibba在NeighborNet分析中显示出最长的分支,因此92%的位点具有最高的相对分子进化率。一致地,UPGMA分析确定了美国gibba所有100个数据集、网络和树都可以通过FTP获得:http://www.langebio.cinvestav.mx/utricularia/).包含预期邻征的所有物种的所有基因序列的级联超矩阵(图5)和UPGMA(图5 b)的结果,美国gibba显示出了很高的分子进化速度,在P<< 0.001相对于Coffea阿拉比卡(外围集团Capsicum Annuum.使用与细胞器基因组相同的可能性模型)。
碳、呼吸和活性氧
的分析美国gibbaCholoroplast和线粒体基因组表明,核苷酸取代率升高美国gibba.这些取代率的变化被认为与氧化磷酸化和活性氧(ROS;见下文)。因此,我们分析了射捕PEG的功能分类,以确定它们是否为oxp和ROS相关过程提供分子支持。如前所述,在拍摄和诱捕PEG中,MIPS类别中代表性最突出的是“能量”。在拍摄PEG中,“能量”MIPS类别由“光合作用”和“能量转换和再生”子类代表,而在陷阱PEG中,“能量”类别由“呼吸”和“电子传递和膜相关的能量守恒”子类代表。相应地,UtriculariaBladders的呼吸大使,同时表现出比营养组织更低的光合速率[76,77].有趣的是,“氧和自由基解毒”亚类别在压力PEG中很突出。
线粒体的呼吸链通常与电子传递结合,是细胞获得能量进行各种活动的主要途径之一。电子传输驱动化学渗透泵,使质子在线粒体膜间隙隔离,这些正电荷进入线粒体腔,催化二磷酸腺苷磷酸化成ATP。氧化磷酸化的限速酶是细胞色素c氧化酶(COX),位于ATP合酶之前一步。以往的研究表明,由于在正向达尔文选择下固定的特定氨基酸位置的改变,可能会改变COX的结构和功能Utricularia和它的姐妹属的一些物种,Genlisea.(开瓶器植物)。已经提出假设,其中这些残留物的特异性变化[两个连续的半胱氨酸(c)]可以改变Cox和细胞色素C之间的解离动力学[78]在活动场所可能产生构象变化[79].有人认为,后一个过程可以可逆地将质子泵浦与电子输运解耦[79].以这种方式,可以比作举起直到所需ATP的电容器的电容器,例如,例如,在其烧制后泵出水。然而,储存大量质子可能在形成反应性氧物质(ROS)的后果可能是通过电子传输的备用和泄漏产生的反应性氧物质(ROS)[6].由此可见,增强的ROS产生的诱变作用(具有易出错的修复)可能是一个共同的原因,解释了观察到的高核苷酸替换率Utricularia(上)和慢链菌科基因组大小的动态进化,后者是通过双链断裂的非同源重组实现的[5].
利用FunCat数据库和MIPS分类,我们共识别出18个注释美国gibbaUT(与15同源拟南芥unique proteins)被归类为“DNA重组和DNA修复”MIPS亚类,所有这些都被认为是普遍表达基因(Additional file)15.、表S10)。关于“氧和自由基解毒”亚类别,159美国gibbaUT最多对应91拟南芥其中一些(44.7%)也普遍表达(附加文件16.,表S11)。该MIPS子类别包括参与谷胱甘肽缀合和过氧化物酶反应的35个蛋白,参与超氧化酶代谢和一种过氧化氢酶。同样,这些DNA修复和ROS排毒过程的表达既不是陷阱特异性的也不是陷阱过度表达,但普遍存在。然而,ROS生产不需要在器官中均匀分布,这可以改变活组织中的净修复/排毒能力。
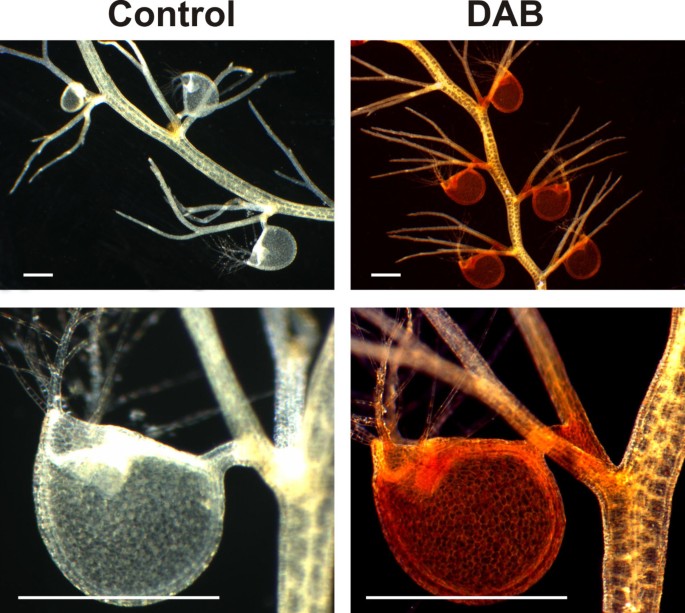
为了评估陷阱的ROS含量与植物有化器官细胞,美国gibba植物被H染色2O2特异性染料3,3-二氨基苯胺(DAB)。在大多数植物用器官(茎/叶子和陷阱)的细胞中检测到DAB染色,在陷阱中具有相当高的强度(图6)对应于在这些结构中观察到的更大呼吸,通过Adamec 2006 [77].给出了普遍存在的表达式模式Utricularia编码解毒酶的基因的器官,较低的相对ROS解毒预期在陷阱中,因此更大的毒性效应,如诱变。因此,肯定有可能观察到的高核苷酸取代率Utricularia细胞器基因组和核基因是由于活性氧的过量生产,面对净效率较低的DNA修复。虽然零期望是所有的核基因都应该平等和随机地产生ros介导的突变,但在100个被调查的基因中,有8%的基因没有证据表明ros介导的突变Utricularia特殊技能的速度增加。分析错误和/或不同物种的基因序列的进化保守差异可以解释这种与100%预期的小差异。未来对这些案例中的非同义与同义替代率的分析可能会揭示后者的现象。关于核基因组的动态,详细的研究,例如搜索猖獗的双链断裂修复的分子特征,必须等待一个高质量的整个序列Utricularia GIBBA基因组。
结论
的Utricularia转录组提供了一个详细的基因组窗口,了解发生在肉食性植物的过程。它包含了一种复杂的代谢途径的深层表征,该代谢途径刻画了一个假定的最小植物基因组的特征,允许它作为基因组信息的来源来探索该属的结构、功能和进化多样性。植物芽和陷阱是其转录组的功能分类最相似的器官,后者表达用于预先被细菌编码的捕食性消化的水解酶。支持生理研究,陷阱显着过表达基因涉及呼吸。其他表达数据表明,虽然氮气吸收可以部分地发生在营养零件中,磷酸盐摄取可能主要发生在陷阱中。DNA修复和ROS排毒酶的表达可以指示对增加的呼吸的反应。最后,来自Bladderwort转录组的证据,直接测量ROS原位,以及细胞器基因组和多个核基因的跨物种比较支持这样一种假设,即整个植物中核苷酸替换率的增加可能是由于放大ROS产生的诱变作用。
方法
植物材料和治疗
Utricularia GIBBA在Umécuaro大坝(Michoacán-México)采集植物,在户外的塑料容器(面积0.1 m2, 10 L)中繁殖,在12.5-15 cm的深度填满大坝的水;通过添加软自来水维持的水位。组织取自茎状结构、诱捕器和包含1 - 4朵花的花序。
为了获得尽可能多的基因表达的证据,额外的几组美国gibba将植物(3至5个体)转移到无菌50mL Erlenmeyer烧瓶中(含有25ml无菌水),然后暴露于不同的生理胁迫条件(光,温度和水剥夺)。对于光线反应,美国gibba在连续黑暗处理48小时后,采用恒光(100 μmol m .)处理-2证券交易委员会-1).将样品在4,8,12和24小时收集;作为一个控制美国gibba植物保持在黑暗中并在同一时间点收集。对于低温响应,美国gibba将植物转移到5℃的冷室,恒定光(约50μ; mol m-2证券交易委员会-1) 48小时。分别于4、8、12、24和48小时采集标本。干旱胁迫处理(缺水)包括从烧瓶中取出水美国gibba在24°C恒定光照下(50 μmol m-2证券交易委员会-1).分别于4、8、12、24和48小时采集标本。作为低温和干旱胁迫处理的对照美国gibba在24°C恒定光照下(50 μmol m-2证券交易委员会-1).此外,我们收集了陷阱,射击(将这些视为茎与寿命的结构)和花序美国gibba目的是探索和比较这些器官的转录组。所有采集的样品立即用液氮冷冻,-70°C保存至使用。
互补脱氧核糖核酸图书馆建设
提取总RNA美国gibba使用TRIZOL (Invitrogen),用研钵和杵在液氮中研磨,对不同胁迫条件下的器官或整个植物进行了研究。在植物受到胁迫的情况下,从每个实验条件中收集5 μg RNA,以获得单个RNA样本。使用Message Amp-II试剂盒(Ambion),按照制造商的建议,用3 μg总RNA混合物合成第一和第二链cDNA。对于三个器官(捕蝇器、嫩枝和花序),每个样本都被分别处理。10 - 12ng合成的cDNA被扩增体外通过Qiagen RNAeasy色谱柱(Qiagen)纯化70 ~ 90 μg反义RNA (aRNA)。以mRNA为模板(20 μg)进行第二轮cDNA合成。除第一链合成阶段采用随机引物(多为六聚体)外,其余cDNA合成均按上述方法进行。该程序产生了大约10 μg的cDNA,并使用DNA Clear Kit for cDNA纯化(Ambion)纯化。用Ampure magnetic particles (agcourt, Beckman Coulter)处理cDNA,得到200 ~ 700 pb的片段。
454 cDNA测序及组装
大约10 μg剪切的cDNA用于454测序。根据制造商的说明,对cDNA样本进行末端修复并连接适配器。链霉亲和素珠富集、DNA变性和乳剂PCR也按照所述程序进行[80].GSFLX测序器运行4.5小时的典型输出约为75 Mb,平均包含约300,000个序列读取c.250个基点。进行了两次FLX焦磷酸测序(微滴度板分为两个扇区),其中1/2序列取自StsL (cDNA文库取自美国gibba多种生理胁迫条件下的植物;1/2为ShtL(芽库),1/2为FlwL花序库),1/2为TrpL(胞果或陷阱库)。总共生成了926,897个读取(FlwL为258,457个,ShtL为234,963个,StsL为140,507个,TrpL为292,970个),估计平均大小为200.18个碱基,共代表185.55 Mpb。使用SeqClean软件管道屏蔽读取[83]消除将导致装配不正确的序列区域。掩蔽的靶标包括多A / T尾,富含Ns(未确定碱基)和低复杂性序列。为了执行装配过程,考虑了817,792次掩蔽读数(总数的88.23%,最小尺寸为100bp)。基于共享序列相似性比较并将掩蔽读取序列成对并分组成簇。结果,所获得的簇包括最可能从相同mRNA衍生的读数。然后将每个群集组装成一个或多个Contig,其来自多个读取对准。使用聚类使用megablast[81],然后使用CAP3组装程序组装得到的簇[82].集群内的Contigs共享在40个核苷酸的窗口内至少90%的同一性。Contigs和Singleters的组合被称为独特的转录物(UT; 16,551个Contigs和95,873单身)。包含序列读取和质量分数的文件被存入国家生物技术信息中心的短读存档(NCBI)[登录号SRP005297]。
注释的美国gibba犹他州
Blastx相似性搜索(E-Value 10-5对Refseq数据库中引用的蛋白质进行比特评分≥45)[25.]来自9个完整的植物基因组[拟南芥,白杨,蓖麻,葡萄(eudicots),oryza sativa,高粱双色(单子叶植物),Physomitrella金属盘(苔藓),衣藻reinhardtii,乌西莫曲霉露西瓜(绿藻)]进行注释美国gibbaUT。我们使用了拟南芥蛋白质注释来定义同源基因美国gibba因为这些注释比来自其他物种的注释更精致。使用基因和基因组的京都百科全书(Kegg)分配给功能类别(Kegg)[83- - - - - -85]和酶委员会(EC)数字[86)有关。
表达谱分析美国gibba转录组
选择在cDNA文库中出现多次以上的转录物(FLWL,SHTL,TRPL和STSL)进行选择在网上分位数归一化和统计检验后的表达式分析[42,43].简而言之,这种方法允许在任意数量的文库中比较基因表达,以识别差异表达的基因。该方法使用一个单一的统计检验,以对数似然比统计描述基因在不同文库间的差异表达程度,该对数似然比统计渐近趋向于一个χ2分布(43].使用genspring GX 7.3.1软件(Agilent Technologies)对结果进行可视化®).
系统发育,簇和分子率分析
质体和线粒体DNA基因组的连接块(用g块定义[75])使用邻吩分析核基因序列[71]和Upgma [72在SplitsTree4包中[87].100个随机选择的核基因数据集通过比对(使用MUSCLE [88]对抗翻译的氨基酸)Utricularia GIBBA互补脱氧核糖核酸片段重叠群和拟南芥在COS II数据库中提供的Asterid Orthologs的基因序列。用于系统发育分析,LOGDET距离[89由于其对基数组成偏差的稳健性,我们使用SplitsTree4中的诊断工具进行了经验观察,因此始终将其作为标准的基于模型的校正。使用HyPhy计算相对替代率差异[90]在若干模型下(F81固定速率,F84固定速率,一般时间可逆或局部参数固定,或全局参数固定,或全局参数、gamma分布和4个速率类)。P报告的值小于在不同模型下获得的最大值。
差异表达基因的功能分类
根据功能目录进行功能基因分类[47,48].采用超几何方法与Bonferroni校正的分析,p值的截止值为0.01。
中存在
Primer设计(Tm, 60-65°C)在Primer3 v.0.4.0 web工具中进行[91].使用逆向特异性引物为每个基因评价的逆特异性引物制备PCR扩增的cDNA模板,并根据制造商的说明用上标III逆转录酶(Invitrogen)处理。每次反应含有10μg总RNA的cDNA模板,1×SYBR Green PCR主混合物(Applied Biosystems)和500μm正向和反向引物。在下面的热循环条件下在ABI棱镜7500序列检测系统(应用生物系统)中进行实时PCR:在95℃下10分钟,然后在95℃下总共40℃,30s分钟为65°C和40s在72°C。对于QRT-PCR,相对于其归一化相对转录物丰度施转录水平。所有计算和分析使用ABI 7500 Software v2.0.1(应用生物系统)和2-ΔΔct方法(92].引物的扩增效率(0.92 ~ 1.01)通过cDNA稀释序列的扩增(1:5)来确定。RT-PCR产物的特异性随后进行熔化曲线分析,并在65-95°C熔体期间连续采集荧光数据。
活性氧的检测
对H202根据Orozco和Ryan (1999, [93])和染色的美国gibba植物被Malamy和Benfey (1997, [94),用SZH10立体显微镜(奥林巴斯)进行分析。
缩写
- UT:
-
独特的成绩单
- 挂钩:
-
优先表达基因
- TRPL:
-
cDNA文库产生美国gibba陷阱
- FlwL:
-
cDNA文库产生美国gibba花序
- ShtL:
-
cDNA文库产生美国gibba芽(茎、叶)
- StsL:
-
从受多种生理胁迫的整株植物中获得cDNA文库
- ROS:
-
活性氧
参考文献
- 1。
Jobson RW,Albert VA:食肉植物姐妹谱系之间的分子速率平行多样化。克莱斯。2002,18(2):127-136。
- 2。
MüllerK,罗宋汤T,Legendre L,Porembski S,Theisen I,Barthlott W:熊属植物和Lamiales的肉毒血症的演变。植物生物学。2004,6(04):477-490。10.1055 / s-2004-817909。
- 3.
国家自然科学基金项目:国家自然科学基金项目,国家自然科学基金项目,国家自然科学基金项目,国家自然科学基金项目。acta botanica sinica, 2010, 30(1): 1 - 5。10.1007 / s00606 - 004 - 0224 - 1。
- 4.
Müller K, Borsch T, Legendre L, Porembski S, Barthlott W:肉食性Lentibulariaceae (Lamiales)进化的最新进展。植物学报。2006,8(06):748-757。10.1055 / s - 2006 - 924706。
- 5.
王志强,王志强,王志强,等:小孢子虫科植物中最小的被子植物基因组及其染色体大小。植物学报。2006,8(06):770-777。10.1055 / s - 2006 - 924101。
- 6.
食肉狸藻(狸藻属,扁豆科):一个膨胀的系统。植物学报,2010,61(1):5-9。10.1093 / jxb / erp349。
- 7.
泰勒Pg,乌特菌属L:一个分类专着。1989年,伦敦:陛下的文具办公室
- 8.
胎生小囊藻胚胎结构的异常,附小囊藻属胚胎进化的评论。中国生物医学工程学报,2010,31(1):69-80。10.1007 / s00709 - 009 - 0084 - 1。
- 9.
王志强,王志强,王志强:一种肉食性狸藻类的摄食生态。南方生态学报,2001,26(6):680-691。10.1046 / j.1442-9993.2001.01149.x。
- 10.
Richards J:Utricularia Purpurea(Lentibulariaceae)的膀胱功能:肉食病重重要。美国植物学杂志。2001,88(1):170-176。10.2307 / 2657137。
- 11.
Lollar AQ,Coleman DC,Boyd Ce:Utricularia inflata的磷吸收的食肉途径。archivfürhydrobiologie。1971,69:400-404。
- 12.
王志强,王志强,王志强,等。水生植物对植物营养的影响[j] .植物营养与肥料学报,2003,23(2):427 - 434。10.1111 / j.1469-8137.1994.tb03946.x。
- 13。
埃里森am,gotelli nj:能量学和肉食植物的演变 - 达尔文在世界上最精彩的植物的植物。实验植物学杂志。2009,60(1):19-42。
- 14。
Sirová D, Borovec J, Santruckova H, Santrucek J, Vrba J, Adamec L:肉食性狸藻的再研究:植物为捕集器提供光合碳。植物学报,2010,61(1):99-103。10.1093 / jxb / erp286。
- 15.
Sirovád,Borovec J,Cernáb,rejmánkováe,adamecl,Vrba J:水生Utricularia物种陷阱中的微生物群落开发。水生植物。2009,90(2):129-136。10.1016 / j.aquabot.2008.07.007。
- 16.
Adamec L:食肉植物Utricularia和Genlisea(Lentibulariaceae)内的氧气浓度。植物学的年。2007,100(4):849-856。10.1093 / AOB / MCM182。
- 17.
引用本文:刘志强,刘志强,刘志强,等:肉食性水族狸藻四种捕蝇器的酶活性研究。植物生态学报,2019,36(3):369 - 372。10.1046 / j.1469-8137.2003.00834.x。
- 18.
Plachno BJ, Adamec L, Lichtscheidl IK, Peroutka M, Adlassnig W, Vrba J:肉食性植物消化腺磷酸酶活性的荧光标记。acta botanica sinica, 2010, 32(6): 816 - 824。10.1055 / s - 2006 - 924177。
- 19.
柳条T,Schlagenhauf E,Graner A,Close T,Keller B,Stein N:454使用大麦复杂基因组进行测试。BMC基因组学。2006,7(1):275-10.1186 / 1471-2164-7-275。
- 20。
Huse S, Huber J, Morrison H, Sogin M, Welch D:大规模平行DNA焦磷酸测序的准确性和质量。生物谷bioon . 2007, 8 (7): R143-10.1186/gb-2007-8-7-r143。
- 21。
Weber APM, Weber KL, Carr K, Wilkerson C, Ohlrogge JB:利用大规模平行焦磷酸测序对拟南芥转录组进行取样。acta photonica sinica, 2007, 44(1): 32-42。10.1104 / pp.107.096677。
- 22。
Jones-Rhoades MW,Borevitz Jo,Preuss D:拟南芥的基因组表达剖面雌性配子体识别小型分泌蛋白质的家庭。Plos Genet。2007,3(10):E171-10.1371 / journal.pgen.0030171。
- 23。
Novaes E, Drost D, Farmerie W, Pappas G, Grattapaglia D, Sederoff R, Kirst M:巨桉高通量基因和SNP发现,一个未特征基因组。中国生物医学工程学报。2008,29(1):1- 4。
- 24。
Vega-Arreguin J, Ibarra-Laclette E, Jimenez-Moraila B, Martinez O, Vielle-Calzada J, Herrera-Estrella L, Herrera-Estrella A:利用焦糖测序高通量策略对Palomero玉米转录组进行深度采样。中国生物医学工程学报。2009,10(1):299-10.1186/1471-2164-10-299。
- 25。
参考序列数据库(Planta)。[ftp://ftp.ncbi.nih.gov/refseq/release/plant/]
- 26。
Myers EW,Sutton Gg,Delcher Al,Dew Im,Fasulo DP,Flanigan MJ,Kravitz Sa,Mobarry Cm,Reinert Khj,Remington Ka,等:果蝇的全基因组大会。科学。2000,287(5461):2196-2204。10.1126 / Science.287.5461.2196。
- 27。
Miller JR, Delcher AL, Koren S, Venter E, Walenz BP, Brownley A, Johnson J, Li K, Mobarry C, Sutton G:与配对配对的焦糖测序序列的积极组装。生物信息学。2008,24(24):2818-2824。10.1093 /生物信息学/ btn548。
- 28。
KEGG:京都基因和基因组百科全书。[http://www.genome.jp/kegg/]
- 29。
导航工具,探索KEGG全球地图。[http://www.kegg.jp/kegg/atlas/]
- 30.
Riechmann JL, Heard J, Martin G, Reuber L, Z C, Keddie J, Adam L, Pineda O, Ratcliffe OJ, et al .拟南芥转录因子在真核生物中的全基因组比较分析。中国科学:地球科学,2000,29(4):591 - 598。10.1126 / science.290.5499.2105。
- 31.
曲丽娟,朱艳霞:拟南芥转录因子家族的研究进展及未来研究的突出问题。植物生物学进展。2006,9(5):544-549。10.1016 / j.pbi.2006.07.005。
- 32.
李志刚,李志刚,李志刚:植物转录相关蛋白的系统发育研究进展。植物营养与肥料学报。2007,27(4):453 - 457。10.1104 / pp.107.095760。
- 33.
郭安华,何凯,刘德华,白淑萍,顾昕,魏丽,罗建军:拟南芥转录因子数据库。生物信息学。2005,21(10):2568-2569。10.1093 /生物信息学/ bti334。
- 34.
拟南芥转录因子数据库。[http://datf.cbi.pku.edu.cn/]
- 35。
朱庆华,郭安英,高刚,钟云峰,徐敏,黄敏,罗静:DPTF:杨树转录因子数据库。生物信息学。2007,23(10):1307-1308。10.1093 /生物信息学/ btm113。
- 36。
杨树转录因子数据库。[http://dptf.cbi.pku.edu.cn/]
- 37。
C, Breton C:转录因子网络。根发展知识的途径。植物生理学报。2004,136(3):3478-3485。10.1104 / pp.104.051029。
- 38。
Marioni JC,Mason Ce,Mane Sm,Stephens M,Gilad Y:RNA-SEQ:对技术再现性的评估和与基因表达阵列的比较。基因组研究。2008,18(9):1509-1517。10.1101 / GR.079558.108。
- 39.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B:通过RNA-Seq定位和定量哺乳动物转录组。王志强,王志强。基于遗传算法的抗肿瘤药物研究进展[j] .中国生物医学工程学报,2008,25(6):641 - 646。10.1038 / nmeth.1226。
- 40.
在mRNA-Seq实验中,标准化和差异表达的统计方法的评价。生物信息学学报。2010,11(1):94-10.1186/1471-2105-11-94。
- 41.
R软件版本2.1.1。[http://www.r-project.org.]
- 42.
Irizarry RA, Bolstad BM, Collin F, Cope LM, Hobbs B, Speed TP: Affymetrix基因芯片探针水平数据摘要。核酸研究。2003,31 (4):e15-e15。10.1093 / nar / gng015。
- 43.
陈志强,陈志强,陈志强:cDNA文库中基因表达的比较。基因组学研究。2000,10(12):2055-2061。10.1101 / gr.gr - 1325 rr。
- 44.
Martínez O, Reyes-Valdés MH:通过信息论定义转录组中的多样性、专门化和基因特异性。中国科学院院刊。2008,25(4):427 - 434。10.1073 / pnas.0803479105。
- 45。
杜松B,Robins R,Joel D:肉食植物。1989年,伦敦:学术出版社
- 46。
危害S:三种食肉水生植物Utricularia(Bladderwort)的猎物选择。archivfürhydrobiologie。1999年,146:449-470。
- 47。
Ruepp A, Zollner A, Maier D, Albermann K, Hani J, Mokrejs M, Tetko I, Güldener U, Mannhaupt G, Münsterkötter M, et al: FunCat,全基因组蛋白质系统分类的功能注释方案。核酸研究。2004,32(18):5539-5545。10.1093 / nar / gkh894。
- 48。
MIPS功能目录数据库。[http://mips.helmholtz-muenchen.de/proj/funcatDB/]
- 49。
Raghothama KG:磷酸转运和信号转导。植物生物学进展,2000,3(3):182-187。
- 50.
Gierth M,MäserP:植物中的钾转运蛋白 - 参与K +采集,再分配和稳态。费用字母。2007,581(12):2348-2356。10.1016 / J.Febslet.2007.03.035。
- 51.
植物中K+通道的活性:基因、调控和功能。中国海洋大学学报(自然科学版)。10.1016 / j.febslet.2007.03.058。
- 52.
奇布里亚,Iwan Jones:寻常狸藻的营养有效性和肉食性习性。水生生物学报。2007,52(3):500-509。10.1111 / j.1365-2427.2006.01719.x。
- 53.
马铃薯磷酸转运体的功能分析及细胞特异性表达。植物学报。1998,206(2):225-233。10.1007 / s004250050394。
- 54.
刘春梅,刘春梅,刘春梅:磷对番茄磷素转运蛋白基因的调控作用。植物生理学。1998,116(1):91-99。10.1104 / pp.116.1.91。
- 55.
赵天杰,刘洪,Harrison MJ:截形紫花苜蓿(Medicago truncatula)中一个磷酸盐转运体(MtPT1)的空间表达模式表明其在根/土壤界面上的磷酸盐转运作用。植物学报。2001,25(3):281-293。10.1046 / j.1365 - 313 x.2001.00963.x。
- 56.
Karthikeyan As,Varadarajan DK,Mukatira UT,D'Urzo MP,Damsz B,Raghothama Kg:调节拟南芥磷酸盐转运蛋白的表达。植物生理学。2002,130(1):221-233。10.1104 / pp.020007。
- 57.
在拟南芥中,磷酸盐转运体Pht1家族的成员在表达分析中发挥了新的作用。植物学报,2002,31(3):341-353。10.1046 / j.1365 - 313 x.2002.01356.x。
- 58.
大麦Pht1;1磷酸转运蛋白基因启动子分析确定了控制根表达和磷酸缺失反应的区域。植物生理学报。2004,136(4):4205-4214。10.1104 / pp.104.045823。
- 59。
Lloyd Fe:食肉植物。MS,USA:沃尔瑟姆,慢性博特纳卡公司。1942年
- 60。
Liu K-H, Huang C-Y, Tsay Y-F: CHL1是一种双亲和的拟南芥硝酸盐转运蛋白。acta botanica sinica(云南植物学报). 1999,11(5):865-874。
- 61。
李建勇,傅玉龙,Pike SM,鲍杰,田伟,张勇,陈长忠,张勇,李慧敏,黄杰,等:拟南芥硝酸盐转运体NRT1.8在木质素处理过程中的作用及其对镉的耐受。《植物细胞》,2010
- 62。
程勋,李尚华,黄恒成,田一芳:CHL1在植物中作为硝酸盐传感器的作用。中国生物医学工程学报,2017,32(6):1143 - 1143。10.1016 / j.cell.2009.07.004。
- 63.
Klebl F,Wolf M,Sauer N:酵母浆膜尿素转运蛋白转运蛋白转运蛋白转运蛋白缺陷由Cpnip1,来自西葫芦(Cucurbita Pepo L.)的Nod26样蛋白质,以及Arabidopsis ThalianaΔ-Tip或[Gamma]]-小费。费用字母。2003,547(1-3):69-74。10.1016 / S0014-5793(03)00671-9。
- 64.
埃里森am,gotelli nj:食肉植物的进化生态。生态与发展的趋势。2001,16(11):623-629。10.1016 / s0169-5347(01)02269-8。
- 65.
M-GCAT:在近缘物种中交互式高效构建大规模多基因组比较框架。生物信息学。2006,7(1):433-10.1186/1471-2105-7-433。
- 66.
多基因组比较软件(M-GCAT)。[http://alggen.lsi.upc.es/recerca/align/mgcat/]
- 67.
o'brien ea,zhang y,wang e,marie v,badejoko w,lang bf,汉堡:gobase:一个细胞器基因组数据库。核酸研究。2009,37(4):D946-D950。
- 68.
器官基因组数据库(GOBASE)。[http://gobase.bcm.umontreal.ca/]
- 69。
Castresana J:从多次对准中选择保守块用于其在系统发育分析中的使用。分子生物学和演化。2000,17(4):540-552。
- 70.
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、特定位置间隙惩罚和权重矩阵选择来提高累进式多序列比对的灵敏度。核酸研究。1994,22(22):4673-4680。10.1093 / nar / 22.22.4673。
- 71.
张志强,张志强,张志强。基于遗传算法的系统发育网络构建方法。分子生物学与进化。2004,21(2):255-265。
- 72.
Sokal RR,Michener CD:一种评估系统关系的统计方法。堪萨斯大学科学公报。1958年,28:1409-1438。
- 73.
中国科学院植物研究所,北京100101;中国科学院植物研究所,北京100101。植物学报,2002,14(7):1457-1467。10.1105 / tpc.010479。
- 74.
Wu F, Mueller LA, Crouzillat D, Petiard V, Tanksley SD:结合生物信息学和系统遗传学鉴定大套单拷贝同源基因(COSII)用于比较、进化和系统研究:一个欧洲小行星植物分支的测试案例。中国生物医学工程学报。2006,34(3):427 - 434。10.1534 / genetics.106.062455。
- 75.
保守的Ortholog Set II(Cosii)标记。
- 76.
Knight SE:普通狸藻类的肉食性成本,及lt;i> macrohiza <生态学报。1998,89(3):348-355。
- 77.
水生狸藻囊和叶的呼吸和光合作用。acta botanica sinica, 2010, 32(6): 765-769。10.1055 / s - 2006 - 924540。
- 78.
细胞色素c氧化酶的适应性进化:一种食肉植物辐射的基础设施。中国科学:地球科学,2004,30(5):531 - 534。10.1073 / pnas.0408092101。
- 79.
Laakkonen L,Jobson Rw,Albert VA:Bladderwort植物中肉食进化的新模型(Utricularia):细胞色素C氧化酶(COX)的适应性变化提供呼吸能力。植物BIOL(斯图特)。2006,8(06):758-764。10.1055 / s-2006-924459。
- 80.
Margulies M, Egholm M, Altman WE, Attiya S, Bader JS, benmben LA, Berka J, Braverman MS, Chen Y-J, Chen Z等:微组装高密度皮升反应器中的基因组测序。自然科学学报。2005,437(4):457 - 461。
- 81.
Zhang Z,Schwartz S,Wagner L,Miller W:一种用于对准DNA序列的贪婪算法。中国计算生物学杂志。2000,7(1-2):203-214。10.1089 / 10665270050081478
- 82.
黄旭,马丹A, CAP3: DNA序列组装程序。中国生物医学工程学报。1999,9(9):868-877。10.1101 / gr.9.9.868。
- 83.
Kanehisa M, Goto S: KEGG:京都基因和基因组百科全书。核酸研究。2000,28(1):27-30。10.1093 / nar / 28.1.27。
- 84。
Kanehisa M, Goto S, Hattori M, aoki kinoshita KF, Itoh M, Kawashima S, Katayama T, Araki M, Hirakawa M:从基因组学到化学基因组学:KEGG的新发展。核酸研究。2006,34(增刊1):D354-D357。
- 85。
Kanehisa M, Goto S, Furumichi M, Tanabe M, Hirakawa M: KEGG用于表达和分析涉及疾病和药物的分子网络。核酸研究。2010,38(增刊1):D355-D360。
- 86。
2000年的酶数据库。核酸研究。2000,28(1):304-305。10.1093 / nar / 28.1.304。
- 87.
Huson DH:Splitstree:分析和可视化进化数据。生物信息学。1998,14(1):68-73。10.1093 / Bioinformatics / 14.1.68。
- 88.
Edgar RC: MUSCLE:多序列比对,高精度,高通量。核酸研究。32(5):1792-1797。
- 89.
Lockhart PJ, Steel MA, Hendy MD, Penny D:在更现实的序列进化模型下恢复进化树。分子生物学与进化。1994,11(4):605-612。
- 90.
池塘SLK, Frost SDW, Muse SV: HyPhy:使用系统发育学的假设检验。生物信息学。2005,21(5):676-679。10.1093 /生物信息学/ bti079。
- 91.
primer3(v。0.4.0)。从DNA序列中挑选引物。[http://frodo.wi.mit.edu/primer3/]
- 92.
利用实时荧光定量PCR和2-[Delta][Delta]CT方法分析基因的相对表达。方法。2001,25(4):402-408。10.1006 / meth.2001.1262。
- 93.
Orozco-Cardenas M, Ryan CA:过氧化氢通过十八烷途径在植物叶片中通过伤害和系统蛋白系统生成。中国科学院院刊,1999,16(11):653 - 657。10.1073 / pnas.96.11.6553。
- 94.
拟南芥侧根的组织和细胞分化。发展。1997,124(1):33-44。
致谢
我们要感谢Daniel Rodríguez-Leal对系统发育分析的宝贵讨论。我们还感谢Octavio Martínez de la Vega通过信息论在多样性和专业化分析方面的帮助,以及Araceli Fernández-Cortes在生物信息学分析方面的帮助。特别感谢Beatriz Jiménez-Moraila, Verenice Ramírez-Rodríguez和他们的同事提供的测序服务。最后,我们感谢LANGEBIO、CINVESTAV的“实验室服务genómicos”成员的帮助。我们也感谢匿名审稿人的积极和相关的评论,这提高了本手稿的质量。EIL感谢CONACyT(墨西哥)的博士奖学金。
作者信息
隶属关系
相应的作者
额外的信息
作者的贡献
EIL进行了汇编、注释、建库、统计分析、稿件撰写等工作。VAA为数据分析、系统发育分析、手稿的起草和编辑做出了贡献。CAPT和MJOE进行RNA提取和cDNA合成。CAPT也参与了ROS染色实验。FMZH进行qRT-PCR实验。AHE和LHE构思了这个项目,并负责指导所有的研究活动,还协助撰写了手稿。所有作者都已阅读并批准了最终提交的手稿版本。
电子辅料
12870 _2011_889_moesm1_esm.zip
附加文件1:表S1 -美国gibba利用BLASTx (e-value≤10-05,a bit score≥45)和蛋白质数据库检索序列相似性,标注UT拟南芥,白杨,蓖麻,葡萄(二榫植物),oryza sativa,高粱双色(单子叶植物植物),Physcomitrella patens.(苔藓),衣藻reinhardtii,乌米莫马里诺拉诺斯(绿藻)(ZIP 13mb)
图S1 -图中所示的代谢途径
额外的文件2:美国gibba独特的成绩单(UT)设置.(一种)结合植物KEGG数据库中已有的通路图和相关基因,构建了全球代谢图谱(黑线)。(b)由此表示的全球新陈代谢图美国gibbaUT(蓝线)。(C)KEGG开花植物代谢全球图与中所表示的代谢图重叠比较美国gibbaUT。(TIFF 2 MB)
12870_2011_889_moesm3_esm.xls.
附加文件3:表S2 -中鉴定的TF家族成员的基因数比较美国gibba一些维管植物(拟南芥,populus trichocarpa和栽培稻(籼/粳稻))。(XLS 27 KB)
12870 _2011_889_moesm4_esm.xls
附加文件4:表S3 - 表达式配置文件矩阵美国gibba基因。对每个特定转录组的读数(原始和标准化)(FLWL; FLWLESCENSE,SHTL;拍摄,TRPL; TRPL; TRAPS和STSL;压力植物)合并到16,551个Contig中的每一个中。(XLS 2 MB)
表S7 -生理胁迫条件(StsL)、花序(FlwL)、茎生(ShtL)和诱捕(TrpL)中功能类别过多
额外的文件8:美国gibba钉子.分类美国gibbaUT通过FunCat数据库根据MIPS分类得到。(XLS 70 KB)
图S2 - peg的qRT-PCR验证
附加文件9:。454测序得到APETALA1, APETALA3, PISTILLATA, AGAMOUS, SEPALATA3, CLAVATA1, MYB21, MYB24, RBCS-1B, SBPase, α-葡萄糖苷酶,ß-己糖苷酶,氨基肽酶,酸性磷酸酶,ß-葡萄糖苷酶的表达模式(一种)和qrt-pcr(b).用QRT-PCR获得的454测序得到的表达水平的相关性(C).(TIFF 1 MB)
12870 _2011_889_moesm11_esm.xls
附加文件11:表S9 -美国gibbaUT被注释为参与硝酸盐同化途径的拟南芥同源基因。(XLS 39 KB)
图S3 - AA序列的对齐
额外的文件12:拟南芥α-和γ-液泡膜固有蛋白(分别为AT3G16240和AT2G36830)以前被认为是尿素转运体[63]和相应的美国gibba鉴定为射击(茎/叶)钉.“NPA”盒子典型的植物提示,由一个带有不连续红线的盒子突出显示。(TIFF 1 MB)
图S4 -的比例
附加文件13:美国gibbaLSC叶绿体区用于系统发育分析.(一种)两个密切相关物种的质体基因组比较(茄属植物lycopersicum和Jasminum Nudiflorum.)和同源地区Utricularia。吉巴LSC地区。(b)在该区域鉴定出质体基因。(C)系统发育分析中使用的非模糊对齐区域(块)。(TIFF 2 MB)
图S5 -
额外的文件14:美国gibba线粒体区域用于系统发育分析.(一种)线粒体基因组比较拟南芥,芸苔,Carica Papaya,尼古里亚安塔巴哈瓦姆和葡萄和同源区域Utricularia GIBBA.(b)在这个区域发现了线粒体基因。(C)系统发育分析中使用的非模糊对齐区域(块)。(TIFF 493 KB)
作者为图像提交的原始文件
权利和权限
开放访问本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
伊巴拉-拉克莱特,E.,阿尔伯特,弗吉尼亚州,Pérez-Torres,加州等等。转录组织和血管曲线曲线中的分子进化率分析(Utricularia),一种基因组极小的食肉植物。BMC植物杂志11,101(2011)。https://doi.org/10.1186/1471-2229-11-101
收到了:
接受:
发表:
关键词
- 线粒体基因组
- 转录因子家族
- 食虫植物
- Organellar基因组
- 分子进化率