抽象的
背景
提高农作物的产量、抗性和环境适应性是育种和研究的永恒挑战。高效黑麦基因组育种策略及遗传潜力的探索(Secale cereale由于缺乏基因组序列信息,阻碍了品种改良。为了克服这一缺陷,我们利用Roche/454 GS FLX技术对5个冬麦自交系的转录组进行了测序。
结果
250多万条reads被组装成115,400个contigs,代表一个综合黑麦表达序列标签(EST)资源。通过序列比较,鉴定了5234个单核苷酸多态性(SNPs),建立了Rye5K高通量SNP基因分型芯片。通过对59个黑麦自交系(包括5个用于测序的品系)、5个大麦、3个小麦和2个小黑麦材料进行基因分型,研究了Rye5K SNP序列的性能。等位基因频率在0.1 ~ 0.9之间呈平衡分布。黑麦自交系的剩余杂合度在4.0 ~ 20.4%之间变化,花粉的平均杂合度高于亲本。
结论
已建立的序列和分子标记资源将改善和促进遗传和基因组研究以及基于基于基于基于基于黑麦的繁殖。
背景
提高农作物的产量、抗性和环境适应性是育种和研究的永恒挑战。关于展望气候变化,对非生物逆境如干旱,土壤肥力低耐受性增强,和极端温度要求在作物改良。异交种黑麦节目中小麦麦最高的抗寒性[1],并对许多生物和非生物胁迫表现出良好的耐受性。了解黑麦抗逆性的功能遗传基础将有助于提高小麦的抗逆性(小麦L.)和大麦(Hordeum Vulgare.l .)。黑麦作为一种遗传研究系统,因其高遗传变异而备受关注。黑麦除了是中欧和东欧的一种重要的经济作物外,作为双倍体小黑麦的亲本,以及作为小麦易位染色体片段的供体,也为其他作物提供了有价值的性状[2].黑麦受益于二倍体,与更广泛的物种小麦和大麦密切相关。虽然草基因组的参考序列已经可用于米[3.那4.],高粱[5.],短柄[6.]和玉米[7.[Rye的序列信息是稀疏的,它妨碍了其遗传势的利用。
黑麦的单倍体基因组大小超过8 GBP [8.]这是禾谷类作物中最大的一个。此外,基因组的92%是由重复序列[9.].与其他资源相比,遗传和基因组资源是有限的麦芽汁.目前,公共数据库中有1 073668个小麦和501 1620个大麦ESTs,而只有9 298个黑麦ESTshttp://www.ncbi.nlm.nih.gov/dbEST/dbEST_summary.html(发布070111)。RYE的公开基因组资源仅限于一个BAC库[10],有限数量的遗传标记http://wheat.pw.usda.gov/GG2/index.shtml,以及标记密度低的遗传图谱[11- - - - - -15].
新一代测序(NGS)技术,如Illumina公司的Genome Analyzer和罗氏公司的454测序平台,为解决大基因组测序(如大麦和小麦)开辟了道路,这是桑格测序无法解决的[16].NGS平台生产几十万序列在大规模并行的方式,是成本和劳动力有效和被证明是可靠和准确的。几项研究都强调NGS的成功和有效性由转录[例如扩展可用基因组资源[17那18和全基因组[]19]测序。此外,NGS已用于基因表达谱[20.],对基因组组织的分析[21] DNA甲基化研究[22,以及分子标记物的发展[23,等等。
面对庞大的基因组大小和缺乏黑麦,识别序列信息和基因组资源和基因基本农艺性状和基因功能和特性变化的认识极大地妨碍有针对性的隔离。本研究的目的是通过第一高通量SNP基因分型阵列的公共黑麦EST资源和发展的庞大的改善,促进黑麦基因组分析。
方法
植物材料,RNA和测序
五个冬季黑麦自交线LO7,LO152,LO225,P87和P105用于cDNA测序。LO7,LO152和LO225由KWS Lochow GmbH(德国)提供,代表来自种子父母和公司杂交黑麦育种计划的花粉父池的线条。P87和P105是在遗传学研究所,明斯克,白俄罗斯遗传学研究所开发的,并且是映射人口P87×P105的父母[24].自交系Lo7、Lo152和Lo225经过6代自交产生,而P87和P105分别经过7次和8次自交产生。此外,对代表这两个育种库的54个自交系进行了研究。来自花粉亲本库的株系经过2 ~ 3代自交产生,而来自种子亲本库的株系经历了5个自交步骤。
为了捕获黑麦转录组的完整部分,我们从一组植物组织中分别在五个发育阶段和三次胁迫处理后获得了每个自交系的20个总RNA样本(附加文件1).每个自交系汇集3株植物,以获得20个RNA样本。在所有的非胁迫处理中,叶片、茎和/或根的组织样品在3 - 4叶期、分蘖期、茎膨出期、抽穗期和收获成熟期收获。胚芽鞘、小花、早期和成熟的穗状花序被采收。3 - 4叶期采用冷胁迫、脱水休克和营养饥饿胁迫处理,以丰富胁迫诱导基因。冷胁迫是将植物置于-15°C的冰箱中引起的。胁迫处理1、3、6 h后,分别采收根、茎、叶组织并汇集。脱水休克试验是通过将水分充足的植物从土壤中移走,并将它们留在Whatman上进行的®3mm纸(Whatman GmbH, Dassel, Germany),室温[25].根,茎和叶组织中3,6和12小时的应力和汇集的后收获。进行密植每自交系三个厂,导致营养饥饿的压力。根和叶组织收集并合并。所有组织样品在液氮中冷冻并储存在-80℃直至使用。总RNA根据使用NucleoSpin RNA植物试剂盒(#740949,马歇雷-Nagel的,Düren的,德国),并与SPECTRONIC GENESYS™10 BIO光谱仪(热电公司,麦迪逊,USA)定量生产商的指示分离。
将每个自交系的20个RNA样本汇集5微克,将每个自交系的总RNA 100微克送vertis Biotechnology AG (Freising, Germany)进行cDNA合成。从总RNA中制备Poly(A)+ RNA。用随机的六核苷酸引物进行cDNA第一链的合成。然后将454个测序适配器A (5'- gcctccctcgcgccatcag3 ')和B (5'-CTGAGCGGGCTGGCAAGGC-3')连接到5'和3' cDNA末端。最后,在20个(Lo152)和21个(Lo7, Lo225, P87, P105) PCR循环中,使用验证酶进行cdna扩增。正常化是通过一个周期的变性和重组cDNA进行的。在羟基磷灰石色谱柱上从ss-cDNA中分离重组ds-cDNA,得到归一化cDNA样品。羟基磷灰石层析后,ss-cDNA样品进行8个PCR循环扩增。从制备的琼脂糖凝胶中分离得到cDNA片段,大小范围为600 ~ 800 bp。作为对照,分离的cdna在1.5%琼脂糖凝胶上进行分析。 Approximately 150 to 250 μg of the normalized, adapter-ligated, and size selected cDNA samples were used for GS FLX 454 sequencing. All 454 sequence raw data were submitted to the EBI sequence read archive (SRA) and are available under the study accession number ERP000274.
美国东部时间资源
从头序列组装
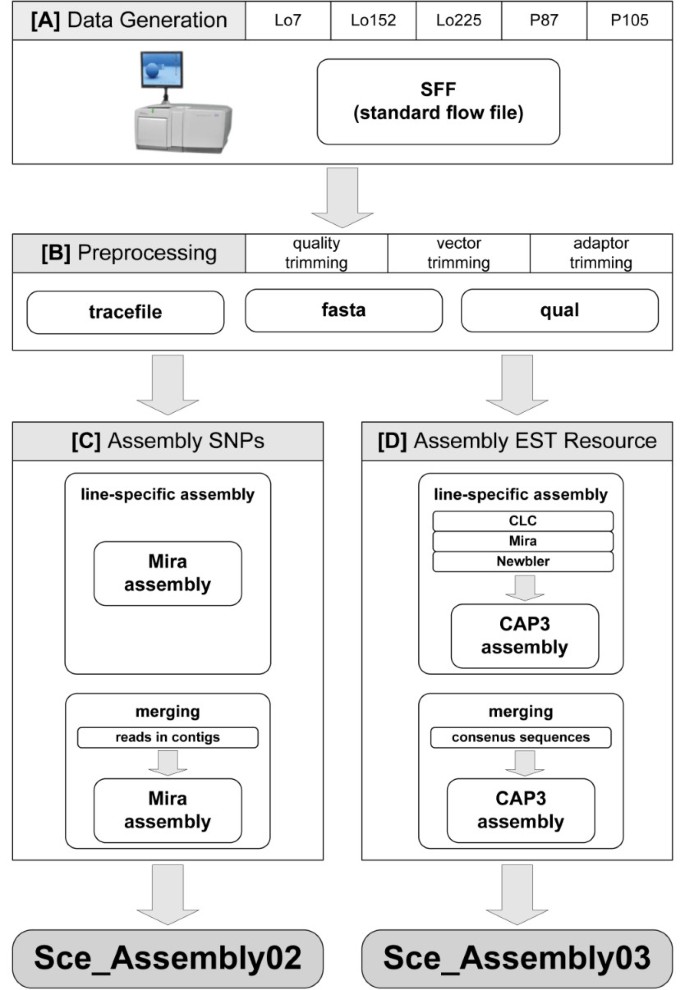
454测序后,对原始序列reads进行质量过滤,去除cDNA合成引物和测序适配器序列。经过预处理后,清洁和修剪的读取进行自交系特定装配。因此,我们采用了Kumar和Blaxter的策略[26用于使用多个组件程序组装转录数据并组合结果以创建更长的重叠群是不太可能in-silico由单一算法产生的人工制品。该策略已被修改以适用于不同的行(图1).我们使用三个独立装配,以实现最可信的共识重叠群序列。最初,所有的读出从每个五行分别组装成一阶重叠群与节目CLC装配单元V3.20http://www.clcbio.com.,mira v3.21 [27和Newbler v2.5 [28].MIRA和Newbler遵循重叠共识布局范式(OLC), CLC则试图在De Bruijn图中找到路径。为了获得特定于行的组装,使用OLC组装程序CAP3合并由三个组装程序构建的所有一阶contigs [29.].我们只考虑线特定的contigs,其成分包括来自所有三个汇编器的一阶contigs。在EST资源生成(Sce_Assembly03)中,我们再次使用CAP3对高置信谱线特异性contigs进行共组装,将来自多个谱线的组分支持的contigs标记为多谱线contigs,仅来自一个谱线的组分支持的contigs视为单谱线contigs。Sce_Assembly03的组装过程已经完成,并筛选了潜在的DNA和外源RNA污染。利用BlastN对大麦(GenBank: NC_008590)和小麦(GenBank: NC_002762)的叶绿体基因组序列、水稻(GenBank: AP011077)、高粱(GenBank: DQ984518)和小麦(GenBank: GU985444)的线粒体基因组序列和小麦(GenBank: GU985444)的质体基因组序列进行分析短柄(GenBank:EU325680)、水稻(GenBank: GU592207)、高粱(GenBank: NC_008602)和小麦(GenBank: AB042240)。通过排除对CDS序列的打击,进一步获得纯度Acyrthosiphon pisum(基因库:ACFK00000000),Buchnera aphidicola(Genbank:AE013218),镰刀菌素graminearum(Genbank:AACM00000000)和草稿顺序叶锈菌在广泛的研究所提供。我们从SCE_AsseMbly03序列集中丢弃了Contigs,该序列集显示了大于E-20的E值,并且所提出的最佳点击率为至少10%的完全Contig大小。已建立的EST资源SCE_ASSEMBLY03可从GABI主数据库中获取[30.],http://www.gabipd.org..
序列比较
五个黑麦近交系之间的序列可能不同的程度,其防止德诺维组装两条线。爆破 [31.进行了不需要严格序列一致性的比较,以分析不同装配体之间的重叠。由CAP3生成的特定行组装与Sce_Assembly03一起用于“全对全”BlastN分析。每个特定于行的程序集以及Sce_Assembly03的多行和单行contigs都被用作主题和查询序列。统计对主题序列的最佳查询命中,以识别各自集合中的同源性。当检测结果超过> = 70%的鉴别性和30 bp覆盖的保守临界值时,被认为是显著的。
Sce_Assembly03与玉米现有的4个蛋白质数据库[ZmB73_v5b.60,http://www.maizesequence.org],稻[RAP2,[32.]],高粱(5.),而短柄[6.],两个大麦和小麦数据库(大麦装配35和小麦装配WK,http://harvest.ucr.edu)和两个大麦全长cDNA (flcDNA)文库数据库[33.]和小麦[34.]用BlastX程序和TBLASTX,分别进行的。当他们超过> 70%的身份和30基点覆盖的保守截止值命中只考虑显著。为了防止命中基于低复杂性序列或重复发现Sce_Assembly03使用RepeatMasker [掩蔽35.]及内部MIPS重复资料数据库[36.].
通过染色体 - 明智的BLASTX分析研究了SCE_Assembly03的基因组分布,比较多线和单线CONTIG短柄蛋白质序列。Sce_assembly03序列被映射到短柄基因组通过使用滑动窗口方法,窗口尺寸为0.5 MB,沿着0.1 MB的偏移短柄染色体。Blastx命中数量和相应的BP覆盖率百分比短柄确定基因。如果超过60%的值将该值设置为零,则校正这些密度值为每个窗口的数量。密度值被推断为基因[6.或每Mb命中数(黑麦)以方便比较。为了可视化映射结果,使用Python matplotlib模块结合jet colormap从密度值创建热图[37.].
功能基因注释
所述Sce_Assembly03的115400个序列功能注释执行与针对非冗余(NR)蛋白质序列数据库Blast2GO默认参数[BLAST搜索38.]后掩蔽重复序列和不包括单身。基因本体论(GO)术语使用B2G4PIPE分配http://www.blast2go.org和一个本地安装的blast2go数据库。注释文件由其各自的Go类别 - 生物过程,蜂窝分量和分子函数扩展 - 使用可根据要求提供的自定义内置的Python脚本。
SSR挖掘和SNP发现
MISA鉴定了338,536个序列内的SSR基序[39.在标准设置下。在5个自交系中,Lo225提供的含有contigs的SSR数量最多,因此选择Lo225作为参考数据集。剩余4行MISA输出与Lo225数据集交叉匹配,检测冗余SSRs。结合Lo7、Lo152、Lo225、P87和P105中检测到的“独特”SSR基序,生成非冗余SSR数据集。单核苷酸重复基序被丢弃,因为在Roche/454数据中,已知单体运行是最常见的测序错误。用于实验验证在网上检测SSR标记的引物侧翼的SSR基序的引物3使用[设计40.].在LO7,LO225,P87和P105中进行片段的扩增,因为它们是两个映射群体的父母。因此,在LO7和LO225和/或P87和P105之间检测到的多态性使得能够成为发现的SSR的遗传映射。PCR在总体积为20μL,包括20ng基因组DNA,1×Hotstar Taq PCR缓冲液(Qiagen,Hilden,德国),250nm的每个引物,200μmDNTPS和0.5 U Hotstar Taq DNA聚合酶(Qiagen,Hilden,德国)。使用触控的PCR曲线,在95℃下15分钟的初始变性步骤随后在94℃下进行45个变性,1分钟,退火1分钟,并在72℃下延伸1分钟。退火温度从65℃至55℃下每循环减少1℃并保持恒定35后的后续循环。最终延伸步骤在72℃下进行10分钟。在1.5%琼脂糖凝胶上检查成功扩增。
为了在组装序列中发现snp,我们采用了第二种组装策略。从所有Reads中选择在特定于行的contigs中组装的Reads,并进行总体组装,合并所有5个基因型的提取的Reads (Sce_Assembly02,图)1).随着维持在约核苷酸覆盖这一策略信息是可靠的SNP发现重要。该Sce_Assembly02在其他文件中描述2并可从GABI主数据库中获得http://www.gabipd.org..的工作流程在网上SCE_Assembly02中的SNP发现到选择高信NestSNP候选者是一个三步步骤:第一,工具千兆v0.4.1 [41.]用在其他文件中给定的参数设定施加3..其次,通过内部实现提取已发现snp的特征,计算候选snp的定义选择标准。通过筛选得到的候选单核苷酸多态性符合以下要求:两个作图群体Lo7 × Lo225和/或P87 × P105亲本间的单核苷酸多态性为双等位和多态。成功的探针设计应与均聚物> 5 bp的距离,与下一个Indel > 60 bp的距离,以及与contig末端> 60 bp的距离。第三,使用EagleView对组装序列中过滤后的SNPs进行人工检测[42.]以确保SNP基因分型阵列的高品质。我们认为假定的测序错误,SNP位在个别读取和单倍型信息。为5234 SNP寡探针设计和Rye5K阵列通过Illumina公司(圣地亚哥,USA),为的Infinium I选择HD定制BeadChip上产生的。为了证明在含有重叠群序列是基因分型阵列SNP表示的SNP的全基因组的覆盖在网上映射兑短柄基因组的BlastN分析。
通过分析59个黑麦自交系(包括5个用于测序的自交系)以及大麦(Barke, Morex, OWB Dom, OWB Rec, Steptoe)、小麦(Chinese Spring, Dream, Mulgara)和小黑麦(Modus,育种品系SaKa3006)的材料,评价SNP阵列的性能。在Illumina iScan平台和Infinium HD分析中,每株植物共使用300 ng基因组DNA进行基因分型。用双通道扫描仪生成带有Cy3-和cy5标记珠的阵列矩阵的荧光图像。使用GenomeStudio软件V2009.1中的基因分型模块(Illumina, San Diego, USA)进行原始杂交强度数据处理、聚类和基因型调用(AA、AB、BB)。通过排除所有丢失数据超过5%的SNP检测,基因型数据得到清理。通过将一个等位基因(AA + 1/2 AB或BB + 1/2 AB)的出现次数除以两倍于每个SNP的检测株数,直接计算A和B等位基因的频率。利用单核苷酸多态性(AB)与单核苷酸多态性的关系,计算了59个自交系的残杂合度。观测值与期望值的显著偏差用精确的二项检验,使用R [43.].通过分析10份非黑麦材料的基因分型数据,探讨黑麦5k SNP序列在其他小粒谷物中的适用性。
结果
Rye EST资源的建立和描述
组装
在364,343和681,787之间产生的五个独立的测序运行,对应于〜87和〜166 MB的原始数据每近交线(表1).随后质量过滤和除去测序衔接和cDNA合成引物的导致〜75至〜145 MB每自交系高质量序列与213和222碱基对之间平均读长。总体而言,2573590高质量具有216个核苷酸的中值长度读取获得,共计548兆。5系特异性cDNA文库组装每行特异性51462个78813的重叠群序列之间分别产生组件,总结到338536个重叠群(附加文件的过滤质量读取2).平均在五个系特异性组件,每个核苷酸覆盖了4.5至6.2的读取。
共有序列由多个组件程序创建的,并通过合并CAP3被用来生成Sce_Assembly03(图1, 桌子2).将89.0%的读入源自来自两个,三个,四个或五个自交系(多线Contig)或从一个自交系(单线Contig)的Contig。SCE_AsseMbly03导致115,400个序列,包括33,352个多线Contigs(占所有读数的77.8%)和82,048个单线Contigs(占所有读数的11.1%)。所有读数的11.0%都未取消质量标准,并从大会中删除。多线CONTIG序列长度从201bp到8,636bp,L50长度为1,070bp。平均而言,每个Contig都是从多线Contig中的六十读取中构建,并在单行Contig中的三个读数中构建。
用于罗氏/454序列读取的组装过程的流水线.数据生成[A]后,提取序列(fasta)、质量(qual)和跟踪文件信息。从原始读取中去除低质量区域、向量和适配器序列[B]。预处理是通过将裁剪后的读交给特定于行的装配来完成的。为了建立SNP资源Sce_Assembly02 [C],只有在特定于行的assembly的contigs中组装的reads,使用Mira对第二个assembly进行合并。为建立EST资源,分别用CLC装配单元、Mira和Newbler计算5个细胞系的Sce_Assembly03 [D]装配,并由CAP3装配合并。将所有品系的一致序列传递给第二个CAP3组装,并在多个品系上组合序列。生成的序列集包括由两到五行一致序列确认的contigs(多行contigs)或包含源自一行的读的contigs(单行contigs)。
序列比较
我们比较了通过CAP3彼此和针对所述多线和Sce_Assembly03的单线共有序列生成的五个系特异性组件(表3.).这表明52.16%到78.72%的命中在特定的线组合之间。针对多线重叠体的线特异性组合的BlastN分析达到87.79%。因此,正如预期的那样,可以得出单线组合之间所代表基因的大量重叠的结论。然而,其余的12.21%显示了明显的序列差异(高多态性基因/等位基因)或基因仅代表(表达)在五个黑麦自交系样本中的一个。
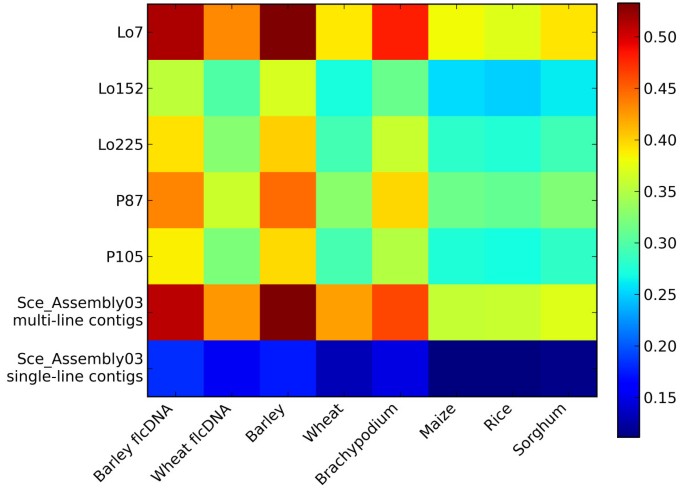
行特定的组件和Sce_Assembly03用的参考基因组之间的序列同源性短柄通过(t)BlastX比对,研究了玉米、水稻和高粱的flcDNA和EST序列,以及小麦和大麦的flcDNA和EST序列2).与大麦序列相比,鉴定了大多数同源物,其次是短柄小麦、高粱、玉米和水稻。Sce_Assembly03的线特异性集合和多线集合的Contig序列与公共序列数据库具有较高的同源性。Sce_Assembly03单线contigs同源性较低。这一发现可以归因于序列长度比多线contigs短约三分之二(表2).Sce_Assembly03的多系拼接产生了超过65%的大麦或小麦flcDNA和HarvEST组合(数据未显示)。通过tBlastX比对Sce_Assembly03基因序列短柄,玉米,高粱和米饭我们能够标记约46.3%,35.9%,37.2%和36.2%的参考基因曲目的碎片。从33,352个多线和82,048个单线Contigs 22,926(68.7%)和23,406(28.5%)揭示了至少一个公共草序列资源。在RYE cDNA文库中包含的基因表明,在将SCE_Assembly03 CONTIG序列比较时,没有偏置黑麦基因组的某个区域。短柄基因组(附加文件4.).在远端区域密集的基因含量短柄染色体以及围绕着丝粒的基因贫困地区广受Sce_Assembly03重叠群序列覆盖。
功能基因注释
在掩蔽Sce_Assembly03的重复序列后,还保留了111,150条序列(32,725条多线序列和78,425条单线contigs序列)用于Blast2GO分析。在这些序列中,有49294条与nr数据库相匹配,随后35356条(71.7%)黑麦杂交序列(16970条多系和18386条单系杂交)被分配到一个或多个GO注释中。在生物过程、细胞成分和分子功能方面,分别获得了35186、38280和51,950个GO项(附加文件)5.).跨越三个GO类别,4997个独特的GO术语进行了鉴定。多在Sce_Assembly03 350个序列与生物和非生物胁迫应答(数据未显示)。
标记发现、SNP阵列设计和高通量基因分型
SSR标记的发展
内线路特定组件的338536个重叠群的12317(3.6%)的重叠群的馏分包含SSR基序。引物序列可以设计这些重叠群5230。限制到二,三,四,五或六核苷酸基序降低SSR候选的数目至3799。交叉匹配分析过滤的最终SSR数据集,其包括1385唯一的,非冗余的SSR(附加文件6.).通过PCR扩增的四个父母基因型LO7,LO225,P87和P105选择随机的155SSRS的随机子集进行实验验证。146引物对(94%)立即扩增预期尺寸的片段,无需进一步优化PCR条件。12个引物组合产生的片段大于预期,表明内含子存在。这些被排除在进一步的分析之外。最后,具有预期片段大小的134个PCR产物中的61(46%)揭示了P87和P105(29)或LO7和LO225(37)之间的琼脂糖凝胶上的裸眼多态性。
SNP的发现
SNP的发现需要高质量序列阅读的充分覆盖,以便区分真正的SNP和测序错误。因此,在合并5个自交系的序列时,执行程序集Sce_Assembly02,从特定的程序集中排除单例。在使用GigaBayes进行的第一步数据挖掘中,在138339个contigs中共鉴定出277,033个推测的多态性,共55mb共识序列。苏格兰民族党候选人的数量被减少到可容纳17917观众通过过滤这些SNP满足选择标准和质量要求,如bi-allelic和多态的父母两个映射之间人口Lo7×Lo225和/或P87×施敏原著,距离均聚物> 5 bp,距离下一个Indel > 60英国石油公司,到contig末端的距离> 60 bp。随后在Sce_Assembly02中进行的人工检查将数据集从3961个contigs减少到5,211个SNP候选数据集。该数据集与在非公开黑麦序列中发现的另外23个SNP被用于设计和生产黑麦5k SNP基因分型阵列。在3961个独特的contigs中,2835个(71.6%)是在网上映射到短柄基因组。在Bd1 ~ 5号染色体上,contigs分别为826、641、688、416和262个4.).Blast2GO对Rye5K阵列上的3,961个contig序列进行分析,分配了2,096个序列,并带有相关的GO标识符(附加文件)7.).
Rye5K SNP阵列的应用
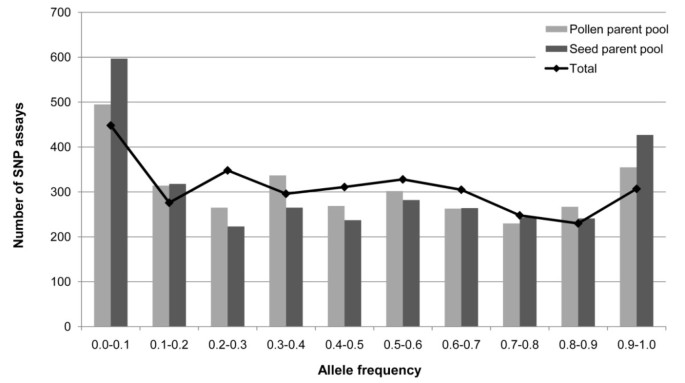
在5个RNA-自交系上测试了Rye5K SNP阵列的性能seq,54个额外的黑麦自交系和10个非黑麦读取。在5,234个SNP中,4,557%(87%)产生的信号和2,970%(57%)和3,148(60%)被成功地呼吁代表杂交黑麦种子父母和花粉父池的59个黑麦自交系(表4.额外的文件8.).根据用于SNP发现的5个自交系的基因分型结果,3%在网上检测到的SNP是假阳性。黑麦的等位基因频率均匀分布在0.1至0.9(图3.).在未用于SNP发现的54个自交系中,有12.3%被称为SNP的小比例是单形的,当单独观察花粉亲本(15.7%)和种子亲本(13.7%)库时,这一比例略有上升。
基因分型数据用于计算黑麦自交系所观察到的残留杂。杂合基因座的每条线所观察到的百分比4.1和在用于454测序五个黑麦自交系4.8%之间,并在4.0从两个杂种优势育种池54个近交系之间到20.4%变化。平均来说,观察到比对种子亲本池(5.5%)的花粉亲池(11.5%)残留的杂合性更高的水平。
研究了Rye5K SNP序列对其他小粒谷物的适用性。在4557个在黑麦中产生信号的SNP检测中,大麦、小麦和小黑麦中分别有63.0%(2871)、75.8%(3452)和84.1%(3831)的SNP检测结果。然而,86.7、91.6和76.5%的单核苷酸多态性在调查的大麦、小麦和小黑麦材料之间未表现出多态性。
讨论
两用转录组测序
在这项研究中,我们报告建立包含115,400个EST序列,1,385个SSR,超过5,000个SNP的Rye基因组资源,以及用于大型基因分型的Rye5k SNP阵列。NGS用于产生五种黑麦自交线LO7,LO152,LO225,P87和P105的转录组序列。本研究的每次测序运行的读数的数量均为在其他研究中获得的甚至超过结果[17那23那44.].由于454测序获得了大量的2.5 Mio读序列德诺维这些数据集的组装仍然是计算和生物信息挑战。遵循两种专用的装配策略,以便首先提供综合的EST资源,并第二位能够发现自交系之间的多态性。五个特定于线组件顶部的第二组件减少了在SCE_Assembly03中创建嵌合伪影的可能性。此外,通过线之间的变化引入的序列冗余。这是通过在接受线特异性核苷酸差异的同时携带相关序列来实现的。相反,这一事实对于SNP检测至关重要,其中仅在特定于行的Contig中预先组装的读取是对SCE_ASSEMBLY02进行的。因此,保留了关于SNP位置处的等位基因覆盖的信息,从而增加了SNP候选者的可靠性。我们研究中的挑战是在没有参考序列的情况下检测SNP。许多SNP检测工具,如GMAP [45.]或MAQ [46.]仅适用于德诺维与引用序列对齐的程序集。这对我们的方法是一个巨大的挑战,我们投入了大量的精力来检测高可信度的snp。对超过10,000个contigs中SNP候选基因的人工检测表明,许多测序错误发生在读取序列的开始,从而导致假阳性。排除在这些读序列区域检测到的SNP候选SNP可能会降低假阳性率,并改进检测多态性的自动化工具德诺维没有参考序列的组装序列数据。
基因组测序已经在模式植物中进展迅速。由于增加了测序吞吐量和降低成本,NGS技术铺平道路,甚至测序大基因组[47.- - - - - -49.].虽然为研究和育种重大意义,黑麦序列资源稀少强加的特征映射,关联研究,并在黑麦功能基因组学严重的局限性。黑麦尤其是对中东和东欧经济市场兴趣,因为它的高耐受非生物胁迫。作为对破译黑麦基因组的第一步骤中,我们的目的是测序黑麦转录的很大一部分。为了实现这一目标,我们各种应激条件下,不同的植物组织和发育阶段下的第一个采样RNA从植物。例如黑麦特异性序列有关胁迫耐受性在本研究中,其是用于在黑麦功能基因组研究必不可少产生。第二,我们减少了通过测序前的cDNA正常化转录的复杂性。cDNA的正常化导致在转录组测序效率显著增加通过均衡在cDNA的群体中高,中的表示和很少表达的转录[50- - - - - -52].由于许多转录本在植物发育过程中是在时间和/或空间上表达的,从不同发育阶段的不同组织汇集的RNA确保了时间和空间特异性转录本的覆盖。
将黑麦连接到草基因组序列资源
为了评估有多少黑麦转录组是由已建立的EST资源所代表的,我们将Sce_Assembly03序列与目前可用的草基因组、flcDNA和EST序列进行了比较。一般来说,在公共数据库中,多行contigs比单行contigs具有显著BlastX命中的序列数量更高。这一发现与Schafleitner等人的结果一致[53]谁比红薯的EST序列(Ipomea甘薯)Uniref100蛋白数据库中包含的序列。
草壳上的整体基因含量Ehrhartoideae(白饭),Panicoideae(玉米,高粱),和Poooideae.[6.]是在类似的范围内。共有25532蛋白编码基因位点被发现短柄[6.与水稻[RAP2, 28,236蛋白编码基因座,[32.],玉米[ZMB73_V5B.60,39,656蛋白质编码基因座,[7.]]和高粱[v1.4,27,640蛋白质编码基因基因座,[5.]]。由于这些模型的基因组密切进化关系,预计黑麦成绩单明显的重叠。针对flcDNA,EST,和基因组序列的Sce_Assembly03的比较揭示了更高的同源性大麦,短柄与玉米、水稻和高粱相比,黑麦在系统发育上与其他成员的亲缘关系更密切Poooideae.玉米、大米和高粱[54那55].该GO注释分析揭示了基因的广谱性在我们的均一化cDNA池从多个组织和发育阶段取样。大量的读出由454测序产生需要在基因的发现,其提供用于前向和反向遗传学的宝贵资源的水平大幅增益在黑麦,以及用于比较基因分析方法。多行重叠群(31%)的比例显著没有给出命中与公众草序列资源。这部分调查结果可以归因于物种特异性和部落的特定基因和基因家族。这Poooideae.包含265个亚家族特异性基因家族,导致亚家族特异性爆发物[6.].由于我们的严格BlastX程序/ TBLASTX截止> 70%序列同一性的值,非保守和非编码如3'-或5'-非翻译区和非编码RNA序列被假定为有助于缺少该馏分同源性,与其他草种。周围的一切黑麦454读取透露命中了MIPS的2%的重复元素数据库[36.,提示反转录转座子的转录活性对样本RNA库有贡献。在两个水稻亚种的转录组测序中,在大约一半的水稻基因中发现了替代剪接模式,以及15000多个新的转录活性区域,其中超过一半在公共蛋白质数据中没有同源物[56].这可能表明,黑麦EST资源包含罕见的,组织特异性和/或没有在近缘种的小麦和大麦的序列资源,尽管他们的广泛EST资源是与压力有关的成绩单。可以预料,黑麦转录序列分析将极大地从参考基因组序列中受益的一构件麦芽汁家庭。大麦的全基因组测序正在进行[49.那57]和小麦[58]和黑麦1rs特异性BAC文库的BAC末端测序[59] 已经被报告了。在网上黑麦est图谱与模式基因组的关系短柄揭示了黑麦转录本的均匀分布,当固定在他们短柄同源染色体。牧草基因组之间的大量同源性将有助于构建代表祖先基因支架的黑麦虚拟基因图。目前正在对黑麦5k序列上的SNPs和从我们的黑麦est中开发的SSRs进行遗传作图,这将导致黑麦和其他禾草之间的精细比较图。由于黑麦基因组的复杂性和高度重复性,一个完整的黑麦基因组序列仍然是无法达到的。然而,在我们的研究中建立的工具,黑麦赶上了其他草基因组资源,更详细地了解黑麦基因组及其进化将是可能的。
黑麦的分子工具箱
五个黑麦自交线的序列信息用于检测转移到1,300个SSR和约5,000个SNPS中的序列变异。已经为一系列作物物种开发了分子标记,并在现代植物育种中发挥重要作用。他们已被用于监测物种内部和中间的DNA序列多样性,以鉴定负责所需特征的基因,以披露遗传变异来源,允许通过引入来自LATERACES和相关草种的有利性状和管理来生产新品种的遗传变异。回复程序[60].与片段长度扩增多态性(aflp)一起,SSRs是目前最流行的谷物标记系统。它们已经被开发用于包括谷物在内的主要作物植物,当应用于育种项目时,这种标记系统预计将加速进展[61].目前,公共黑麦ssr的可用性非常有限。我们的资源显著增加了这一可能有助于评估遗传变异和估计种群间遗传距离的标记资源。除了SSRs外,目前最受关注的标记系统是SNPs [62].单核苷酸多态性在高效指纹图谱、遗传图谱构建、标记辅助选择以及群体和进化遗传学等方面显示出巨大的潜力。Rye5K SNP序列为黑麦分子和基因组中心研究的大规模基因分型提供了一个强大的新资源。最近,全基因组基因分型阵列可以用于农作物和牲畜,并用于全基因组关联研究和调查遗传变异。[63]]。在试点实验中,我们分析了59条黑麦自交系,包括用Rye5k SNP阵列测序的五条线,以估计残留的杂合子程度。两种自我三个或六个循环后的理论期望分别为12.5%,6.3%和1.6%。使用Rye5K阵列的这59条线的基因分型显示杂合度明显(p-Value <0.05)超出了这种理论期望。这可能是由黑麦的同种异体行为解释的部分,导致剩余的杂合性[64].尽管在近交系中产生了强迫自行生成,但由于种子在商业育种计划中作为单耳后代生产,因此不能排除一定程度的交叉授粉。为种子父池观察到的较低水平的残留杂合子与黑麦种子母线(P. Wilde,个人通信)中的更高高级自行生代。对剩余的序列的详细分析,其剩余的杂合子表明序列属于大型基因家族,例如转移酶和羟基酶。检测副病剂或基因家族成员中的SNP可以模拟检测到的杂合子的大部分,从而高估了黑麦自交系中真正的剩余杂合子。
结论
综上所述,Sce_Assembly03提供了一种新的综合EST资源,将黑麦整合到小粒谷物的比较分析中。Rye5K SNP阵列可以对大量个体进行分析,从而获得关联研究的基因分型数据,估计连锁不平衡和群体遗传学方法。我们的基因组资源包括115,400个EST序列,1,385个SSRs,超过5,000个SNPs,以及用于大规模基因分型的Rye5K SNP阵列,这将改善和促进黑麦的遗传和基因组研究以及基于基因组的育种。
参考
- 1.
李利民,傅乐:加拿大草原牧草耐寒性的研究。acta botanica sinica(云南植物科学学报),2017,29(4):593 - 598。10.4141 / cjps87 - 150。
- 2.
高JM、徐BB、徐DY、都GS、Park DS、Kwack YH:生产拥有1BL的新小麦生产线。取自韩国黑麦品种八堂禾密的1RS小麦-黑麦易位。应用计算机学报,2002,14(2):171-176。10.1007 / s00122 - 001 - 0783 - 2。
- 3.
Goff SA,Ricke D,Lan Th,Presting G,Wang R,Dunn M,Glazebrok J,Sessions A,Oeller P,Varma H等:水稻基因组的序列(栽培稻l . ssp。粳稻).中国科学(d辑):地球科学(英文版)。10.1126 / science.1068275。
- 4。
于军,胡舒,王军,黄国康,李松,刘斌,邓艳,戴林,周勇,张旭,等:水稻基因组的一个初步序列栽培稻l . ssp。indic).科学。2002年,296(5565):79-92。10.1126 / science.1068037。
- 5。
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, et al .高粱二色的基因组与草类的多样化。自然。2009,457(729):551-556。10.1038 / nature07723。
- 6.
国际短柄草倡议:模型草的基因组测序和分析Brachypodium distachyon.自然。2010,463(7282):763-768。10.1038 / nature08747。
- 7.
张建平,张建平,张建平,等:玉米B73基因组的复杂性、多样性和动态。中国科学(d辑):地球科学(英文版)。10.1126 / science.1178534。
- 8.
多列热Ĵ,GreilhuberĴ,Lucretti S,梅斯特A,LysákMA,纳迪L,Obermayer R:实验室间的比较:通过流式细胞术植物基因组大小估计。Ann Bot。1998年,82(增刊1):17-26。
- 9。
Flavell RB,Bennett MD,Smith JB,Smith DB:基因组大小和植物中重复核苷酸序列DNA的比例。生物化学遗传学。1974,12(4):257-269。10.1007 / BF00485947。
- 10.
史志强,王志强,王志强,等:黑麦品种的构建。黑麦Alt3耐铝基因的克隆研究进展。王志强。2007,19(4):514 - 514。
- 11.
Hackauf B,Rudd S,Van der Voort JR,Miedaner T,Wehling P:Rye中DNA序列的比较映射(Secale cerealeL.)与水稻基因组有关。理论应用遗传学。2009,118(2):371-384。10.1007 / s00122-008-0906-0。
- 12.
Khlestkina Ek,比MH,Pestsova,EG,Roder MS,Malyshev SV,Korzun V,Borner A:Rye中99个新的微卫星衍生的基因座的映射(Secale cerealeL.)包括39个表达序列标签。理论应用遗传学。2004年,109(4):725-732。10.1007 / s00122-004-1659-Z。
- 13。
Korzun V,Malyshev S,Voylokov AV,伯尔纳答:黑麦的遗传图谱(Secale cerealeL.)组合RFLP,同工酶,蛋白质,微卫星和基因基因座。理论应用遗传学。2001,102(5):709-717。10.1007 / s001220051701。
- 14。
马晓峰,Wanous MK, Houchins K, Milla MAR, Goicoechea PG,王铮,谢敏,Gustafson JP:黑麦分子连锁定位(英文)Secale cerealel .)。理论应用遗传学。2001年,102(4):517-523。10.1007 / s001220051676。
- 15。
森夫特P,Wricke G:一个扩展的黑麦的遗传图谱(Secale cerealel .)。植物育种。1996年,115(6):508-510。10.1111 / j.1439-0523.1996.tb00966.x。
- 16.
Metzker ML:测序技术——下一代。中国生物医学工程学报。2010,11(1):31-46。10.1038 / nrg2626。
- 17.
Meyer E, Aglyamova GV, Wang S, Buchanan-Carter J, Abrego D, Colbourne JK, Willis BL, Matz MV:测序和德诺维利用454 GSFlx分析一个珊瑚幼虫转录组。基因组学杂志。2009,10:219-10.1186/1471-2164-10-219。
- 18.
孙超,李勇,吴强,罗华,孙勇,宋杰,吕爱梅,陈胜:德诺维使用GS FLX钛平台的美国人参根转录组的测序和分析发现涉及人参皂苷生物合成的推定基因。BMC基因组学。2010,11:262-10.1186 / 1471-2164-11-262。
- 19。
Hillier LW, Marth GT, Quinlan AR, Dooling D, Fewell G, Barnett D, Fox P, Glasscock JI, Hickenbotham M, Huang W,等:全基因组测序和变异发现C. Elegans..光子学报。2008,5(2):183-188。10.1038 / nmeth.1179。
- 20.
Torres TT,MetTA M,Ottenwalder B,Schotterer C:通过大规模平行测序的基因表达分析。Genome Res。2008,18(1):172-177。
- 21.
Wicker T, Taudien S, Houben A, Keller B, Graner A, Platzer M, Stein N:一份454个序列的全基因组快照揭示了大麦基因组的组成,并为小麦和大麦基因组大小的平行进化提供了证据。植物j . 2009。
- 22.
Lister R, Ecker JR:找到第五个碱基:胞嘧啶甲基化全基因组测序。基因工程学报。2009,19(6):959-966。10.1101 / gr.083451.108。
- 23.
Novaes E, Drost D, Farmerie W, Pappas G, Grattapaglia D, Sederoff R, Kirst M:高通量基因和SNP发现桉树祖母,未表征的基因组。BMC基因组学。2008年,9(1):312-10.1186 / 1471-2164-9-312。
- 24.
Korzun V,Malyshev S,Kartel N,Westermann T,Weber We,BörnerA:Rye的遗传联系地图(Secale cerealel .)。理论应用遗传学。1998年,96(2):203-208。10.1007 / s001220050728。
- 25。
Oztur Zn,Talame V,Deyholos M,Michalowski CB,Galbraith DW,Gozukirmizi N,Tuberosa R,Bohnert HJ:监测干旱和盐盐型大麦的成绩单丰富的大规模变化。植物mol biol。2002,48(5-6):551-573。
- 26。
Kumar S, Blaxter ML:比较德诺维454个转录组数据的汇编器。基因组学杂志。2010,11:571-10.1186/1471-2164-11-571。
- 27。
Chevreux B, Pfisterer T, Drescher B, Driesel AJ, Muller WE, Wetter T, Suhai S:使用miraEST组装器进行可靠和自动化的mRNA转录本组装和序列est SNP检测。中国生物医学工程学报,2004,27(6):1147-1159。10.1101 / gr.1917404。
- 28.
Margulies M, Egholm M, Altman WE, Attiya S, Bader JS, benmben LA, Berka J, Braverman MS, Chen YJ, Chen Z,等:微组装高密度皮升反应器中的基因组测序。自然科学学报。2005,437(4):457 - 461。
- 29.
Huang X,Madan A:CAP3:DNA序列装配方案。Genome Res。1999,9(9):868-877。10.1101 / GR.9.9.868。
- 30。
Riano-Pachón DM, Nagel A, Neigenfind J, Wagner R, Basekow R, Weber E, Mueller-Roeber B, Diehl S, Kersten B: GabiPD: GABI主要数据库——植物综合“组学”数据库。核酸Res. 2009, D954-959。37个数据库。
- 31。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中国生物医学工程学报,1998,16(3):427 - 434。
- 32。
大米标注项目:大米标注项目数据库(RAP-DB): 2008年更新。核酸学报2008,36 (sup1): D1028-1033。
- 33.
Matsumoto T, Tanaka T, Sakai H, Amano N, Kanamori H, Kurita K, Kikuta A, Kamiya K, Yamamoto M, Ikawa H,等:对来自12个克隆库的24,783个大麦全长cdna进行了综合序列分析。植物生理学。2011,156(1):20-28。10.1104 / pp.110.171579。
- 34.
持田K,吉田T,樱井T,荻原Y,筱崎K:TriFLDB:群集全长编码从小麦序列与应用到比较基因组学草的数据库。植物生理学。2009年,150(3):1135至1146年。10.1104 / pp.109.138214。
- 35.
基因组DNA的重复:挖掘和意义。中国生物医学工程学报。1998,8(3):333- 333。10.1016 / s0959 - 440 x(98) 80067 - 5。
- 36.
Spannagl男,Noubibou O,哈泽d,杨L,贡德拉克H,Hindemitt T,克利K,Haberer G,Schoof H,迈耶KF:MIPSPlantsDB - 综合与比较植物基因组研究植物数据库资源。核酸RES。2007年,D834-840。35数据库。
- 37。
Hunter JD: Matplotlib: 2D图形环境。计算机科学与技术,2007,9(3):90-95。
- 38。
Conesa A,Gotz S,Garcia-Gomez JM,Terol J,Talon M,Robles M:Blast2Go:功能基因组学研究中的注释,可视化和分析的通用工具。生物信息学。2005,21(18):3674-3676。10.1093 / Bioinformatics / BTI610。
- 39。
泰尔T,Michalek W,Varshney RK,格拉纳答:EST环境与开发数据库的大麦发展和表征的基因来源的SSR标记物(Hordeum Vulgare.l .)。应用计算机学报,2003,22(3):441 - 446。
- 40。
Rozen S, Skaletsky H:为一般用户和生物学程序员提供的WWW初级教程。方法:中华医学杂志。2000,132:365-386。
- 41。
Marth GT, Korf I, Yandell MD, Yeh RT, Gu Z, Zakeri H, Stitziel NO, Hillier L, Kwok PY, Gish WR:单核苷酸多态性发现的一般方法。中国生物医学工程学报,1999,23(4):452-456。10.1038/70570。
- 42。
黄W,马斯G:EagleView:一个基因组装配观看者用于下一代测序技术。Genome Res。2008年,18(9):1538至1543年。10.1101 / gr.076067.108。
- 43。
R:统计计算的语言和环境。统计计算基础,奥地利维也纳。2004,ISBN 3-900051-07-0, [http://www.R-project.org]
- 44。
Zagrobelny M, Scheibye-Alsing K, Jensen NB, Moller BL, Gorodkin J, Bak S: 454基于焦糖测序的转录组分析Zygaena filipendulae专注于参与氰基糖苷生物合成的基因。BMC基因组学。2009,10:574-10.1186 / 1471-2164-10-574。
- 45.
GMAP:一种基于mRNA和EST序列的基因组定位和比对程序。生物信息学。2005,21(9):1859-1875。10.1093 /生物信息学/ bti310。
- 46.
李华,阮杰,杜宾:利用定位质量分数定位短DNA测序序列并调用变异。中国生物医学工程学报,2008,18(11):1851-1858。10.1101 / gr.078212.108。
- 47.
黄S,Li R,张Z,Li L,Gu X,风扇W,Lucas Wj,Wang X,谢B,Ni P等:黄瓜的基因组,Cucumis sativus王志强,王志强。基于遗传算法的生物信息学研究[j] .生物医学工程学报,2009,41(12):1275-1281。10.1038 / ng.475。
- 48.
李锐,樊伟,田光,朱华,何磊,蔡军,黄强,蔡强,李斌,白勇,等:序列与序列德诺维巨大的熊猫基因组的组装。自然。2010,463(7279):311-317。10.1038 / Nature08696。
- 49。
Mayer KF, Taudien S, Martis M, Simkova H, Suchankova P, Gundlach H, Wicker T, Petzold A, Felder M, Steuernagel B,等:大麦染色体1H的基因含量和虚基因顺序。植物营养与肥料学报。2009,31(2):491 - 498。10.1104 / pp.109.142612。
- 50.
通过分子选择减少cDNA序列表达的偏倚。核酸学报1994,22(21):4545-4546。10.1093 / nar / 22.21.4545。
- 51。
Emrich SJ, Barbazuk WB, Li L, Schnable PS: LCM-454转录组测序的基因发现与注释。基因工程学报,2007,17(1):69-73。
- 52。
帕坦加利SR,Parimoo S,威斯曼SM:均匀的丰度的构建(标准化)cDNA文库。Proc Natl Acad Sci USA。1991年,88(5):1943-1947。10.1073 / pnas.88.5.1943。
- 53。
Schafleitner R, tinopa LR, Palomino O, Rossel G, Robles RF, Alagon R, Rivera C, Quispe C, Rojas L, Pacheco JA,等:通过重新组装焦磷酸测序和Sanger序列,并挖掘基于基因的微卫星标记建立甘薯基因指数。基因组学杂志。2010,11:604-10.1186/1471-2164-11-604。
- 54。
Bolot S,Abrouk M,Masood-Quraishi U,Stein N,Messing J,Feuillet C,Salse J:谷物基因组的“内圈”。CurrOp植物BIOL。2009,12(2):119-125。10.1016 / J.PBI.2008.10.011。
- 55。
Gaut BS:草基因组的进化动力学。新的植物学家。2002年,154(1):15-28。10.1046 / j.1469-8137.2002.00352.x。
- 56。
陆涛,吕国平,樊东,朱超,李伟,赵强,冯强,赵颖,郭颖,黄晓霞,等:基于RNA-seq的水稻转录组单核苷酸分辨率功能注释。中国生物医学工程学报,2010,20(9):1238-1249。10.1101 / gr.106120.110。
- 57.
Steuernagel B,Taudien S,贡德拉克H,赛德尔男,Ariyadasa R,舒尔特d,彼佐尔德A,费尔德男,格拉纳A,肖尔茨U,等人:德诺维454测序条形码BAC池进行全面调查基因和大麦的基因组的复杂基因组分析的。BMC基因组学。2009年,10:547-10.1186 / 1471-2164-10-547。
- 58.
Paux E,Sourdille P,Salse J,Saintenac C,Choulet F,Leroy P,Korol A,Michalak M,Kianian S,Spielmeyer W等:1-千兆面包小麦染色体3B的物理图。科学。2008,322(5898):101-104。10.1126 / Science.1161847。
- 59.
Bartos J,Paux E,Kofler R,Havrankova M,Kopecky D,Suchankova P,Safar J,Simkova H,Town C,Lelley T等人:对黑麦的第一次调查(Secale cereale通过染色体1R的短臂的BAC末端测序)基因组的组合物。BMC植物BIOL。2008年,8(1):95-10.1186 / 1471-2229-8-95。
- 60.
Korzun V:分子标记及其在谷物繁殖中的应用。工程中的inproceingings“标记辅助选择:植物和动物育种中增加的快速轨道?”2003年10月17日至17日;意大利大学,意大利:Lanteri S 2003,18-22,电子论坛在食品中的电子论坛对电子技术有限公司技术和农业。
- 61.
Gupta PK,Varshney RK,Sharma PC,Ramesh B:分子标记及其在小麦滋生中的应用。植物育种。1999,118(5):369-390。10.1046 / J.1439-0523.1999.00401.x。
- 62.
舒尔曼AH:分子标记,以评估遗传多样性。Euphytica。2007年,158(3):313-321。10.1007 / s10681-006-9282-5。
- 63。
Matukumalli LK,Lawley CT,Schnabel Rd,Taylor JF,Allan MF,Heaton MP,O'Connell J,Moore SS,Smith TPL,Sonstegard TS等:牛的高密度SNP基因分型测定的开发和表征。Plos一个。2009,4(4):E5350-10.1371 / journal.pone.0005350。
- 64。
斯万千瓦,柄DB:杂合性,以平衡在玉米杂交种的关系。遗传学。1959年,44(5):777-786。
确认
我们感谢弗里茨Thümmler(沃提斯AG,弗赖辛,德国)合成和规范的cDNA样品,KWS沃胡夫GMBH提供种子和DNA样本,并克里斯托夫PIETSCH为他的SNP发现管道的前期工作。这项工作是由赠款[0315063A到E,B,0315063B到N.S.,0315063C到K.M.]支持主动教育的德国交通部的“GABI-未来”研究部(BMBF)的框架。
作者信息
从属关系
相应的作者
附加信息
作者的贡献
GH制备测序样品,参与生物信息分析,进行基因分型,并评估基因分型数据。TS,MM和我们进行了454读的处理和组装,并为它们提供了描述性统计数据。KFXM和MS执行BLAST分析,功能注释和序列比较短柄染色体。NS和RZ开发并检测了SSR标记。EB、GH和TS开发了Rye5k SNP序列。CCS、EB、KFXM、NS和US设计了这项研究。EB, GH, MS, RZ, TS起草了手稿。所有作者阅读并批准了最终的手稿。
Grit Haseneyer, Thomas Schmutzer对这项工作做出了同样的贡献。
电子补充材料
12870_2011_914_moesm1_esm.xls.
附加文件1:用于RNA提取的植物组织一套。从暴露于各种应力处理的植物组织中提取每个黑麦自交系的RNA,并在不同的发育阶段收获。(XLS 33 KB)
12870 _2011_914_moesm2_esm.xls
附加文件2:生成的Sce_Assembly02的建立和描述在网上SNP采矿。使用Mira汇编程序V2.9在集成标准设置上以三个步骤进行了三个步骤:首先,原始序列读取超过质量过滤过程,其中454个测序适配器和cDNA合成引物序列以及低质量读数。其次,对清洁和修整的序列读数进行了一种线特定组件,其中每个近交管的读数在单独的组装运行中对齐。在线特定组件中的非对齐读数,即单身人士被拒绝。第三,使用清洁和修剪的读取再次从所有五条自交系中重新开始,将合并到特定于特定于线组件中的Contigs的读取。该策略导致CONTIG序列用于SNP检测,随后用于设计高通量基因分型SNP阵列。关于SNP发现,此组件允许扣除关于等位基因覆盖等SNP位置的关键信息。(XLS 36 KB)
12870_2011_914_MOESM3_ESM.XLS
附加文件3:GigaBayes参数。仅从GigaBayes程序默认设置不同的参数列。(XLS 25 KB)
SCE_Assembly03的多线和单行Contig的关联与
附加文件4:短柄染色体这个Bd1到BD5.每个染色体的四个热图是从描绘的密度短柄在59个黑麦自交系中(从上到下)进行单核苷酸多态性分析短柄染色体在一个滑动窗口中,窗口大小为0.5 Mb,移动0.1 Mb,并确定每个窗口中各自标记基因的数量和bp覆盖率。密度值根据每个窗口的Ns数量进行校正,如果N含量超过60%,则将值设为零并以白色绘制。这个数字被推断为每Mb的数字,以便于比较。热图是使用Python pylab模块结合jet colormap(从蓝色到红色的低值到高值)从密度值创建的。最小、最大和平均基因数/Mb短柄在每个地图的左侧都给出了黑麦的HITS / MB。顶部的标尺给出了Mb中的染色体长度。(PNG 306 KB)
12870 _2011_914_moesm5_esm.png
附加文件5:在Blast2GO第2级的Sce_Assembly03多行和单行contig序列中找到的GO类别。发生率小于0.05%的类别被总结在“其他”中。(PNG 312 KB)
12870 _2011_914_moesm6_esm.xls
附加文件6:在Five File Pround组件的338,536个Contig中检测到SSR主题。丢弃单核苷酸重复基序。混合图案描述了两个关闭SSR图案,其分隔小于100 BP。(XLS 18 KB)
12870 _2011_914_moesm7_esm.xls
附加文件7:Rye5K SNP阵列的描述。在Rye5K SNP阵列上列出了包含contigs的SNP,包括候选SNP位置、提供给Illumina公司(San Diego, USA)的探针设计序列和GO注释。(XLS 2 MB)
的表示两个杂种优势池54黑麦自交系观察到残余的杂合。杂合度为基于与所述Rye5K SNP阵列获得的基因分型数据计算
附加文件8:。来自花粉亲本库的品系属于F代3.到F4.,种子父池的行在第f代中6..(XLS 25 KB)
作者为图像提交的原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
哈塞尼耶,舒穆策,塞德尔,M。et al。从RNA-seq到大规模基因分型-黑麦基因组资源(Secale cerealel .)。BMC植物BIOL.11,131(2011)。https://doi.org/10.1186/1471-2229-11-131
已收到:
公认:
发表:
关键字
- 美国东部时间资源
- 新一代测序
- Secale cerealeL.
- Rye5K SNP数组
- 单核苷酸多态性