摘要
背景
芸苔属种类包括蔬菜和油籽作物,对普通人的日常生活非常重要。同时,甘蓝型物种代表了研究植物生物学众多方面的优异系统,特别是为了分析多倍体的基因组进化,因此对科研也非常重要。现在,基因组Brassica Rapa.已经组装好了,是时候对基因组数据进行深入挖掘了。
描述
Brad是Brassica Database,是一种基于网络的资源,专注于重要的芸苔作物的基因组遗传和基因组数据。Brad基于第一个全基因组序列和芸苔属性种类的进一步数据分析,Brassica Rapa.(chiifu - 401 - 42)。它提供的数据集,如完整的基因组序列B. Rapa.,这是德诺维从Illumina GA II短片段和BAC克隆序列、预测基因和相关注释、非编码rna、转座元件(TE)、B. Rapa.基因的同源性答:芥,以及遗传标记和连锁图谱。BRAD提供了有用的搜索和数据挖掘工具,包括跨注释数据集的搜索、同源或非同源同源词的搜索、特定目标的侧翼区域的搜索,以及BLAST和Gbrowse工具。BRAD允许用户输入几乎任何类型的信息,例如B. Rapa.要么答:芥基因ID,物理位置或遗传标记。
结论
BRAD是一项专注于甘蓝型植物遗传和基因组学的新数据库,它旨在帮助科学家和育种者完全有效地使用甘草植物的基因组数据信息。布拉德将不断更新,可以通过http://brassicadb.org.
背景
芸薹属植物属于芸薹科,共有338属约3700种,其中包括被广泛研究的模式植物拟南芥蒂利亚纳.芸苔属作物既包括蔬菜,也包括油料作物,它们约占世界蔬菜产量的10%,约占世界食用植物油产量的12% [1那2].6种广泛栽培的芸薹属植物的二倍体基因组用著名的“U's triangle”(基因组A, B, C, AB, BC和AC)来描述B. Rapa.那b . oleracea那B. NIGRA.那b . juncea那B. Napus., 和B. Carinata., 分别 [3.].基因组物种,B. Rapa.是亚洲和欧洲的主要蔬菜和油料作物。因为它们作为作物和研究复杂基因组杂交和多倍体的模型的重要性[4.那5.],近年来,对芸苔属的遗传和基因组研究日益深入,产生的芸苔属基因组序列、遗传标记、表达序列标签(ESTs)和数量性状位点(QTLs)等数据集不断增加。
最近完成了整个基因组序列的初始组装B. Rapa.品种线'Chiifu-401'现在可用[6.].基于芸苔属研究界的需要和大量基因组数据的分布,建立了BRAD。它是芸苔属作物全基因组规模遗传和基因组数据及相关资源的重要资料库。BRAD也被设计为其他相关网页和专门数据集的初始访问点。它现在提供了芸苔属A基因组的数据集(B. Rapa., Chiifu-401),包括德诺维从第二代测序技术和BAC末端序列、预测基因、相关注释(InterPro、KEGG2、SwissProt)以及遗传标记和图谱B. Rapa..
在本文中,我们概述了BRAD的主要部分,并介绍了我们开发的关键字搜索工具,以及爆炸和Gbrowse的工具,可以在布拉德中挖掘数据挖掘。
结构和内容
通过对第一批油菜B. rapa基因组序列的分析,建立了Brassica数据库BRAD。在BRAD中有四个主要部分(图1):浏览、搜索、工具和资源。
浏览
在本节中,Brad提供了来自三种人口线的1,160个遗传标记b·拉伯:RCZ16_DH,JWF3P和VCS_DH。这些标记包括758 SSR和402个诱导标记,涵盖所有十种染色体[7.那8.].RCZ16_DH是快速循环系L144与夏型大白菜双单倍体(DH)系Z16杂交而成的群体[9.].在这个群体中有119条DH系。根据亲本L144和Z16的重测序数据,建立了RCZ16_DH标记。将测序数据与组装的chifu401基因组进行比对,获得了L144与Z16之间的26 693个InDel标记,其中402个标记用于锚定德诺维组装到10染色体的支架。另外两个地图,jwf3p和vcs_dh从公共数据库集成在一起http://www.brassica-rapa.org为用户提供更多的选择。
搜索
本节用于注释预测基因,并帮助用户定位特定的基因B. Rapa..完全,在基因组中预测了41,174个基因B. Rapa..每个基因的CDS稍微少一些B. Rapa.与之相比答:芥,虽然每个内含子的大小B. Rapa.比那个更大答:芥(表1).这可能表明,基因组三倍扩增产生的副同源基因B. Rapa.是有区别的6.],在这个过程中丢失了一些编码外显子,并扩大了内含子的平均大小B. Rapa..搜索部分有三个子类别:使用注释,同时基因和侧翼区域进行搜索。
1)注释
这里收集了六个注释数据集:swissprot注释,trembl注释,kegg注释,roce域注释,基因本体和Blastx(最佳击中)B. Rapa.来答:芥.Swissprot和Trembl注释由BLASTP最佳击中(截止电子值:1E-5)产生预测B. Rapa.瑞士 - Prot和Trembl数据库中的蛋白质;B. Rapa.然后,根据来自Swiss-Prot数据库的最佳匹配,将基因映射到KEGG路径图中;InterPro用于注释中的主题和域B. Rapa.使用hmmpfam, fprintscan, ScanRegExp profilescan, blastprodom,和hmmsmart等应用程序对Pfam、PRINTS、PROSITE、ProDom和SMART等公共数据库进行比较。从InterPro结果中提取基因本体信息。我们也使用了同源基因B. Rapa.模型工厂答:芥注释B. Rapa.基因。这些数据集用于根据核苷酸序列、蛋白质和结构域等不同方面注释预测基因。
2)直向同源基因
之间的同步和非同步突出答:芥和B. Rapa.BRAD提供了什么来帮助用户链接B. Rapa.基因信息与研究良好的模式植物的基因信息答:芥.
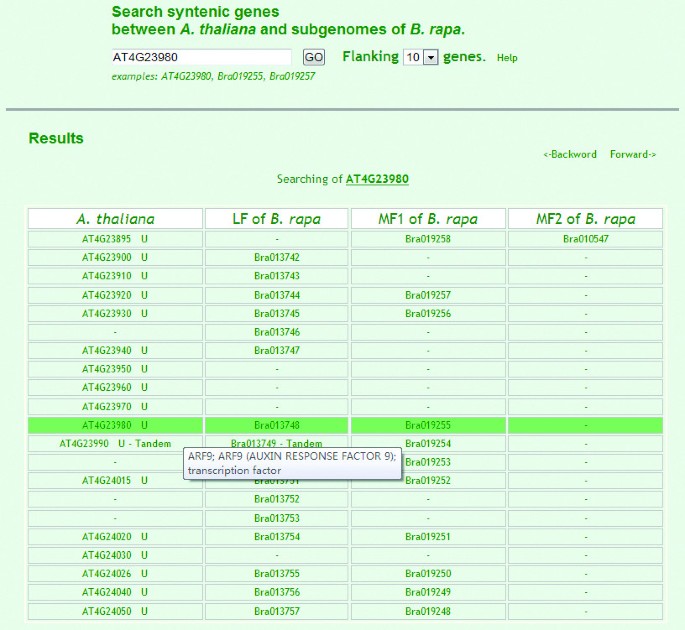
BRAD提供了一组基因,显示出了两种基因之间的保守的同向性答:芥三个亚基因组B. Rapa.(源自基因组三次的三个子宫组),并根据基因的顺序列出答:芥.我们不仅根据基因对序列的纯合度,还根据其侧翼基因的纯合度来确定基因对的同源性。有了这个规则,有30,773对同步对之间B. Rapa.和答:芥获得,9,293,6,683和2,346答:芥有1 2 3个副拷贝的基因B. Rapa.的亚基因组分别为LF、MF1和MF2。LF、MF1和MF2分别是less fraction、more fraction 1和more fraction 2的缩写,表示保留基因多或少的亚基因组。并将三个亚基因组进行了分离答:芥基因组,然后是亚基因组的基因顺序和基因密度[6.].
Non-syntenic基因之间答:芥和B. Rapa.是根据两条规则决定的。首先,应满足BLASTP对准参数:同一性> 70%,覆盖率答:芥基因> 75%,覆盖率B. Rapa.基因> 75%。其次,来自正交对的两个基因不应该是同步基因。完全,有17,159个这样的非同步原理对。
3)侧翼区域搜索
本节的目的是帮助用户找到与感兴趣区域共同定位或侧翼的基因组元素。用户可以输入一个物理位置,例如基因ID或遗传标记,来执行搜索。所有位于搜索区域附近的基因组特征,如基因、转座子、rna (miRNA、tRNA、rRNA和snRNA)被收集并显示在一个表格中。Gbrowse的链接提供了一个选项,可以在染色体的背景下可视化搜索区域。这对于某些研究,如qtl的精细定位,是一个有用的工具。获得qtl后,利用BLAST工具将标记与基因组序列进行比对,获得标记的物理位置。通过搜索这些标记的侧翼区域,可以定位可能是qtl的原因因素的候选基因组元件,如基因或mirna。
随着研究进展的进展,我们将通过添加更多数据集来进一步启用侧翼区域,使其成为对甘草植物感兴趣的分子遗传学家,育种者和所有其他研究人员的一体化和有价值的资源池。
工具
爆炸和基因组浏览(Gbrowse)嵌入(Gbrowse)以帮助用户矿山和可视化基因组数据。
1)爆炸
我们使用标准的wwwblast模块来帮助用户进行序列分析。BLAST数据库,如基因组、基因和蛋白质序列等B. Rapa.,EST序列B. Rapa.,芸苔和十字架在这里提供。
2)基因组浏览(Gbrowse)
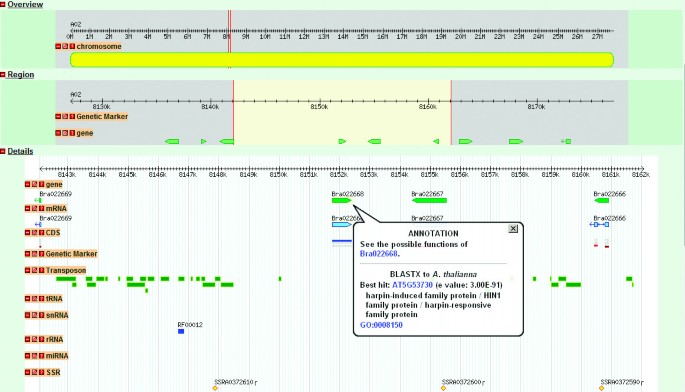
我们使用了基因组浏览器工具,由通用模式生物数据库项目开发,http://gmod.org可视化基因组B. Rapa.[10.].显示三个主要级别:搜索区域的基因组段,侧翼区域和精确的目标。我们现在提供预测的基因,转座子,多种类型的RNA套,Gbrowse中的遗传标记。
资源
除了上述浏览,搜索和工具之外,BRAD还提供批量数据下载,包括基因组和基因序列,基因注释和其他预测的基因组元素。此外,布拉德还可以作为数据或网站链接提供众多可用的社区资源。这些包括专注于Brassicaceae的实验室的其他网站,对Brassica研究人员和关于Brassica育种的遗址集合的会议。
实用程序
使用布拉德的一般指南
浏览基因标记和地图。
使用注释和同时性基因进行搜索。
Gbrowse:基因组病例。
在Browse遗传标记和图谱部分,我们给出了每个标记的遗传和物理位置、引物信息和亲本群体。用户可以在浏览区域中按顺序访问这些数据:染色体选择→种群说明→详细标记信息→点击标记ID查看引物信息。
在使用注释的搜索部分中,用户可以通过提交关键字(例如花或增长)来找到具有感兴趣功能的基因,然后将从六个注释数据集中选择相关的记录,如上所述。单击所选的记录,然后将用户带有与关键字相关的注释的基因。进一步点击基因ID将为用户提供BRAD中该基因的更多信息。
Syntenic基因只能搜索以供使用答:芥和B. Rapa.基因id。在syntenic paralogs的网络中,下拉式“侧翼”菜单有两个选项(10或20个),这意味着它可以从搜索的基因中向上或向下延伸10或20个基因。在表格输出(图2),靶向基因是着色的深绿色。每个答:芥基因对应于1 2或3个基因B. Rapa.亚因素。' - '表明没有发现基因。将光标移动在基因的ID上扩展了功能的功能注释答:芥基因及其同构关系的详细支持信息B. Rapa.基因答:芥.
Gbrowse可视化的功能元件(基因,非编码rna, TEs,遗传标记)的基因组B. Rapa.在一帧下,我们在Brad中的其他应用中,将基因的基因与其他应用进行了联系。通过单击Gbrowse中的基因图标,用户可以获得其注释的链接,最好的Blastx命中答:芥,匹配基因的功能和基因本体(GO),如图所示3..
搜索导航
为了帮助用户快速访问BRAD中感兴趣的基因的所有信息,我们在BRAD的输出表中嵌入了一个javascript对话框作为每个基因ID的导航。通过将多个数据集的访问组合在一个窗口上,该导航可以将用户引导到目标基因的不同资源,从而方便了BRAD的使用。BRAD中有两种类型的基因B. Rapa.和答:芥.为B. Rapa.基因的导航窗口集成了注释、同源或非同源同源、基因序列、基因侧翼区域的功能元件和Gbrowse中的数据可视化等资源。为答:芥基因,导航窗口提供同同或非同同同系词资源的链接,BRAD和TAIR数据库中的注释。
讨论
一些数据库Brassica Rapa.,例如Brassensembl数据库http://www.brassica.info/BrassEnsembl/index.htmlCropStore数据库http://www.cropstoredb.org/brassica/,以及芸苔属基因组数据库http://www.plantgdb.org/BrGDB/,主要专注于基因组数据传播(农作物,芸苔类别数据库)和可视化(BrasseMBL)。BRAD建立了帮助用户从基因组序列中挖掘数据Brassica Rapa.轻松有效地,与现有数据库相比,它具有自己的特定功能和优点。首先,BRAD从模型植物的批量信息中做了准确和有用的链接答:芥到新组装的基因组B. Rapa.并提供细节注释B. Rapa.基因,它提供了同同血缘和非同同血缘之间的特征答:芥和B. Rapa.,主要基因家族B. Rapa.根据答:芥,来自KEGG、InterPro、Swissprot、颤抖等多个注释数据库的基因注释信息。其次,BRAD是最初的基因组数据仓库B. Rapa.,使用的其他数据库或将在BRAD中使用数据作为基本数据来开发其特定功能,我们将改进和不断更新组装的基因组并在布拉德中释放它。
布拉德将包括所有芸苔植物的数据集(例如b . oleracea那B. NIGRA.和B. Napus.)。此外,将首先处理新数据,然后适当地集成或链接到现有数据集。下面列出的数据类型将很快被添加到BRAD中:
-基因家族浏览,如NBS基因家族、生长素基因家族、转录因子基因家族等。
-等位基因数据和频率的遗传标记产生的基因组重测序的不同系B. Rapa..
- 单倍型(来自SNPS映射)的单倍型(来自SNPS映射)B. Rapa.种质收集。
-不同器官转录组数据产生的基因表达水平B. Rapa..
- Synteny浏览器B. Rapa.来b . oleracea.
结论
BRAD是一个专注于甘蓝型植物遗传和基因组学的新数据库。与现有的芸苔属植物数据库相比,Brad具有其特定的功能和优势,特别是其注释和最近组装的基因组的挖掘和深度开采B. Rapa.以及从模型工厂使用信息的使用答:芥.旨在帮助科学家和育种者充分和有效地利用芸苔植物的基因组学和遗传数据集的信息,布拉德将不断改进其应用并将来集成更多可用的数据集。我们建议布拉德将是对比较基因组学,植物演化和分子生物学的科学家的宝贵资源,以及Brassiceae的育种者。
可用性和要求
数据库名称:布拉德
数据库主页:http://brassicadb.org
浏览器要求:该应用程序针对Internet Explorer进行了优化。然而,它还适用于Mozilla Firefox和Safari。
Brad中的数据集是免费提供的。请在Brad Homepage或电子邮件发送“联系我们”,电子邮件博士Xiaowu Wangwangxw@mail.caas.net.cn来请求特定的数据子集。
参考
- 1。
Labana KS, Gupta ML:重要性和起源。油菜育种(编)。Labana, k.s., Banga, S.S. & Banga, S.K.)。施普林格-弗拉格,柏林,1993,1-20。
- 2。
王志强,王志强,王志强:十字花科植物系统发育与毛状体进化。[J] .植物学报,2006,32(6):648 - 648。10.3732 / ajb.93.4.607。
- 3.
联合国:芸苔的基因组分析特别参考B. Napus和奇育的特殊模式.JAP J BOT 1935 ,,7:389-452。
- 4.
Yang TJ, Kim JS, Kwon SJ, Lim KB, Choi BS, Kim JA, Jin M, Park JY, Lim MH, Kim HI, et al: Brassica rapa开花LOCUS C区二倍化过程的序列水平分析。植物学报,2006,18(6):1339-1347。10.1105 / tpc.105.040535。
- 5.
Mun JH,Kwon Sj,Yang Tj,Seol Yj,Jin M,Kim Ja,Lim MH,Kim JS,Baek S,Choi BS等:芸苔属Rapa基因空间的基因组比较分析显示了基因组收缩和差分损失全基因组三重术后重复基因。基因组Biol。2009,10(10):R111-10.1186 / GB-2009-10-10-R111。
- 6.
王X,王H,王J,Sun R,Wu J,Liu S,Bai Y,Mun Jh,Bancroft I,Cheng F等人:中聚百倍作物种类芸苔属rapa的基因组。NAT Genet。2011年。
- 7.
Kim H, Choi SR, Bae J, Hong CP, Lee SY, Hossain MJ, Van Nguyen D, Jin M, Park BS, Bang JW, et al:测序的BAC锚定参考遗传图谱用于协调油菜10条染色体。中国生物医学工程学报。2009,10:432-10.1186/1471-2164-10-432。
- 8.
Choi SR, Teakle GR, Plaha P, Kim JH, Allender CJ, Beynon E, Piao ZY, Soengas P, Han TH, King GJ, et al:国际芥菜基因组测序项目的参考遗传连锁图谱。应用计算机学报。2007,115(6):777-792。10.1007 / s00122 - 007 - 0608 - z。
- 9.
王永强,孙树华,刘波,王华,邓军,廖勇,王强,程飞,王旭,吴军:基于序列的rapa拟染色体组装遗传图谱的参考。基因组学杂志。2011,12:239-10.1186/1471-2164-12-239。
- 10。
Donlin MJ:使用通用基因组浏览器(Gbrowse)。Curr Protoc生物信息学。2009年,第9章:第9章9 9。
致谢和资助
感谢所有在本次工作中给予建议的实验室成员,感谢张中华和陆彩对BRAD服务器搭建的支持。
国家重点基础研究发展计划项目(no . 2006CB101606, no . 2007CB108803, no . 2012CB113901, no . 2012cb11306);国家高技术研究发展计划(no . 2006AA100108);国家国际科技合作项目(no . 2010DFA31730)国家自然科学基金项目(30800753)、欧共体第七期科研、技术开发与示范项目(nue - crop FP7-CP-IP 222645)资助
作者信息
隶属关系
通讯作者
额外的信息
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
XW和FC构思了这项研究。FC处理数据并开发数据库。FC准备了稿件,XW和JW对稿件进行了改进。JW测试了web应用程序和工具,并提供了反馈。LF维护数据库。SL、SS、BL、PL、WH编写了基本数据集。所有作者阅读并批准了最终的手稿。
作者为图像提交的原始文件
权利和权限
开放访问本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
程飞,刘淑芳,吴建军。et al。BRAD,芸苔属植物的遗传学和基因组数据库。BMC植物杂志11,136(2011)。https://doi.org/10.1186/1471-2229-11-136
已收到:
公认:
发表:
关键字
- 双倍单倍体
- 芸苔属
- 芸苔属植物的植物
- 同步基因
- 基因组三份