摘要
背景
目前,甘薯的遗传标记资源存在有限的可用性(Ipomoea Batatas.),这阻碍了对这个物种的基因研究。在甘薯的遗传研究中,有必要开发更多具有潜在应用价值的分子标记。随着新一代测序技术的发展,已经产生了大量的甘薯转录序列,可以通过数据挖掘来识别SSR标记。
结果
在这项研究中,我们调查了181615个EST的SSR标记的识别和发展。总共8294个SSR从7163含有SSR-独特的EST确定的。上的平均,一个SSR是每EST序列的7.1 kb的发现三核苷酸基序(42.9%)是最丰富接着二 - (41.2%),四 - (9.2%),五(3.7%)和六核苷酸(3.1%)的重复类型。前五位的基序包括AG / CT(26.9%),AAG / CTT(13.5%),AT / TA(10.6%),CCG / CGG(5.8%)和AAT / ATT(4.5%)。除去甘薯公布EST-SSR标记的可能重复后,总的非重复7958 SSR图案被确定。基于这些含SSR-序列,1060对高质量SSR引物被设计并用于多态性的一种作图群体的两个亲本(E舒3豪和广2K-30)和八个之间的放大和评估的验证栽培sweetpotatoes的种质。结果表明816个引物对可产生可重现的和强烈的扩增产物,其中195(23.9%)和342(41.9%)的引物对戊戍3豪和广之间表现出多态性2K-30和8个栽培sweetpotatoes之间,分别。
结论
本研究揭示了甘薯EST-SSR标记的频率、类型和分布,为开发栽培甘薯EST-SSR标记提供了依据。这些EST-SSR标记丰富了现有的甘薯群体分子标记资源,对甘薯及其相关群体的定性和定量性状定位、标记辅助选择、进化和遗传多样性研究具有重要意义五爪物种。
背景
地瓜(Ipomoea Batatas.)是六倍倍增物(2n = 6x = 90)Dicot,属于家庭络vulaceae..由于在广泛的环境条件下的高产潜力和适应性,甘薯是世界上重要的粮食作物之一,尤其是在发展中国家。根据粮食和农业组织(FAO)统计,2008年世界甘薯生产的是110多万吨,近80%来自中国,产量约8500万吨约3700000公顷[1].现在甘薯通常用作主食,动物饲料,工业原料和潜在的原料生产醇。此外,高β在许多发展中国家,橙色红薯中的胡萝卜素含量在预防与维生素a缺乏有关的失明和孕产妇死亡方面发挥着关键作用。
尽管甘薯的育种很重要,但这种六倍体作物的遗传复杂性和基因组资源的缺乏限制了它的育种。分子标记在加速选育良种过程中具有巨大的潜力。尽管已经发表了一些甘薯基因图谱[2-4.],现有的地图不具有足够的标志是用于遗传研究非常有用的。因此,对新的标志物的发展有很大的需求。随着新开发的高通量新一代测序技术,已为模式生物中产生,以及经济上重要的大量转录序列的非模式植物。除了提供用于发现基因和转录物轮廓表征的一种有效方法,这些EST序列可以用作分子标记开发,如单核苷酸多态性(SNP),简单序列重复(SSR)具有成本效益的,有价值的来源。
DNA简单序列重复序列,又称微卫星序列,是2- 6bp的DNA核心序列串联重复序列,广泛分布于非编码和转录序列中,通常称为基因组- ssrs和EST-SSRs [5.].SSRs具有基于pcr、可靠、共显性、多等位基因、染色体特异性和高信息性等优点,在植物遗传育种中具有重要的应用价值,如构建高密度连锁图谱、进行遗传多样性分析、进行品种鉴定和标记辅助选择等。虽然基因组ssr具有高度多态性,并在整个基因组中广泛分布[6.那7.],而SSR富集技术的进步也导致许多植物中大量基因组SSR标记的加速发展[8.-14],开发基因组SSR标记仍然是昂贵、劳动密集型和耗时的。相比之下,EST- ssrs可以以较低的成本从EST数据库中快速开发。此外,由于EST-SSRs与编码序列的关联,还可以直接进行基因标记,用于农艺重要性状的QTL定位,提高标记辅助选择的效率[15].此外,EST-SSR标记表明转让的更高层次的密切相关的物种的基因组比SSR标记[13那16-18],并可作为比较作图及进化研究的锚定标记[19那20.].
在甘普托,基因组SSR最初由Jarret和Bowen开发[21]并用于微卫星标记的遗传评价和突变机制[22,亲子关系分析[23[遗传多样性和关系评估[24那25]在栽培甘薯和野生品种中。后来,Hu等人[26]从小插入和富集文库中筛选出79对引物,其中27对在20份甘薯材料中表现出长度多态性。同时,从已发表的EST数据库中鉴定并设计了151对引物,其中75个位点在12个甘薯基因型中表现出长度多态性。最近,利用pyrosequencing和Illumina配对端测序技术产生了大量的est,为开发更有用的甘薯EST-SSRs提供了机会[27那28].为了减少冗余,我们将所有可用序列进行了组合和重组,并进行了大规模est筛选(181,615),目的是:(1)分析栽培甘薯基因组转录区SSRs的频率和分布;(2)从这些序列中设计新的甘薯PCR引物对;(3)对所设计的SSR引物对不同甘薯栽培基因型进行验证和评价。
结果
甘薯est - SSR标记的频率和分布
利用181615条平均长度为548 bp的est序列对SSR基序的存在进行了评价。为了消除冗余序列,提高序列质量,采用tiger Gene Indices Clustering Tools (TGICL) [29]用于从重叠的est聚类中获得一致序列。装配标准包括50 bp的最小匹配,95%的最小同一性重叠区域和20 bp的最大未匹配悬垂。共产生87,492个潜在独特ESTs,包括28,885个contigs和58,607个singleton。对这些装配好的ESTs进行标注,对UniProt数据库进行相似度搜索http://www.uniprot.org.使用BLASTx算法E.价值阈值10-5.结果表明,在87492条est序列中,有53,622条(61.3%)与已知蛋白具有显著的相似性,匹配30,924条独特的蛋白序列。如表所示1使用MISA Perl脚本http://pgrc.ipk-gatersleben.de/misa/,共有8,294个SSRS从7,163个独特的EST鉴定,平均每7.1 KB平均一个SSR。其中,949个EST包含多个SSR,539个具有多于一种重复类型的复合SSR。为了鉴定含有SSR基因座的基因的推定功能,还搜索了7,163个EST序列与UniProt数据库E.-Value截止小于10-5.其中,4,911在此UNIPROT数据库中对已知蛋白质进行了爆炸。
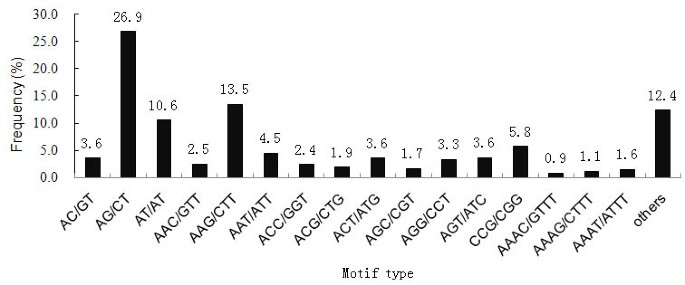
SSR单元大小所占比例分布不均匀。在8294个SSRs中,三、二核苷酸重复基序类型最为丰富(3,554个,42.85%;3,413个,分别为41.15%),其次为四-(762个,9.19%)、五-(311个,3.75%)和六核苷酸重复基序(254个,3.06%)1).如表所示2.SSR的长度主要分布在12 ~ 20 bp之间,占总SSR的84.6%,其次为21 ~ 30 bp的1198个,占14.4%。二核苷酸重复序列(AG/CT)最大值为94 bp。共鉴定出224个SSR基序,其中二、三、四、五、六核苷酸重复序列分别有4、10、31、67和112个类型。在EST-SSRs中,AG/CT双核苷酸重复序列最多(2229个,26.9%),其次是AAG/CTT(1,117个,13.5%)、AT/TA(880个,10.6%)、CCG/CGG(477个,5.8%)、AAT/ATT(375个,4.5%)、AGT/ATC(301个,3.6%)、AC/GT(300个,3.6%)、ACT/ATG(300个,3.6%)、AGG/CCT(276个,3.3%)和AAC/GTT(207个,2.5%)。其余214种母题的频率占22.0%(图)1).
栽培甘薯EST-SSR标记的引物设计与评价
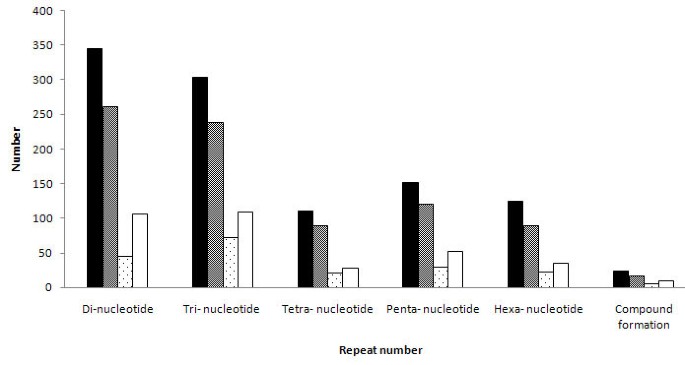
甘薯EST-SSRs以前已被开发[26-28].为了确保新的EST-SSR引物对仅由这些公布微卫星引物针对7163潜在独特含SSR-序列进行比较的设计。共有非重复7958 SSR图案在这项研究进行了鉴定。基于这些含SSR-序列,1060双高质量SSR引物使用Primer总理6.0(PREMIER BIOSOFT国际,帕洛阿尔托CA)设计的。这些设计的引物的,345,303,111,152,125和24分别为二 - ,三 - ,四 - ,分别pena-,六核苷酸重复序列和化合物形成重复序列,(图2).在鄂蜀3号皓和广2K-30中,成功扩增引物897对(84.6%)。其余163条引物在不同退火温度和Mg条件下均未生成PCR产物2+并将被排除在进一步的分析之外。897对工作引物中,811对扩增出的PCR产物符合预期大小,65对扩增出的PCR产物大于预期大小,其余21对扩增出的PCR产物小于预期大小。利用897条引物对8个不同甘薯品种进行进一步验证,其中816条引物在8个不同甘薯品种中均能获得干净、可重复的扩增产物。利用SSR引物扩增鄂蜀3号皓、广2K-30和8个品种的PCR产物示例如图所示3A,3B.附加文件(附加文件)提供了816对引物的标记名称,以及SSR基序、引物序列、包含SSR的序列、Tm(熔化温度)、预期产品长度1表S1)。
栽培甘薯est - SSR标记的多态性分析
首先对鄂蜀3号和广2K-30进行多态性评价。816对有效SSR引物中,195对(23.9%)多态性。在SSR标记上共检测到644个等位基因,根据存在或不存在特定条带的显性评分,平均每个SSR标记的等位基因数为3.30个,范围2 ~ 10个(附加文件)1).可观察到45个二、三、四、五、六核苷酸重复序列和6个化合物形成重复序列的多态性(图)2).BLASTx搜索结果显示,有134个多态性SSR位点与已知或未鉴定的功能基因相关,占68.7%。
在8份红薯栽培材料中进一步分析了816个ssr标记的多态性。结果表明,342条引物(41.9%)具有多态性,共检测到1004个等位基因(附加文件)1).平均每个位点的等位基因数为2.94个,范围为2 ~ 11个。引物GDAAS1073的等位基因最多可达11个。PIC值在0.22到0.88之间变化,平均值为0.35。106个二核苷酸重复序列、109个三核苷酸重复序列、28个四核苷酸重复序列、53个五核苷酸重复序列、36个六核苷酸重复序列和10个化合物形成重复序列(图)2).在342个多态性SSR位点中,有266个对UniProt数据库中已知蛋白有BLAST位点。
讨论
甘薯EST-SSRs的频率和分布
含est的SSR中SSRs的频率可以准确反映基因组转录区SSRs的密度。利用Sanger和下一代测序技术,已经生成了大量的甘薯EST序列。这些序列为我们发现新的基因提供了机会,也为开发标记提供了资源。但由于未归一化cDNA文库和不同研究者的提交,这些EST序列中存在大量的冗余。在本研究中,为了减少冗余,避免对EST-SSR频率的过高估计,在消除冗余后进行SSR搜索。共有87,492条潜在的EST序列(约58.7 Mb)用于SSR的搜索,其中7,163条(8.2%)含有SSR基序,产生8,294条SSR。SSR丰度结果与Hu等(9.1%)的报道一致[26].这两个结果表明,SSR标记为甘薯的EST的丰度比其他物种,例如相对较高的花生(6.8%)〔30.),大麦(3.4%)、玉米(1.4%)、水稻(4.7%)、soyghum(3.6%)、小麦(3.2%)(31],Medicago Truncatula.(3.0%) (17],Epimudium sagittatum(3.4%) (32].在本研究中,EST-SSR的出现频率为每7.1 kb 1个EST-SSR。在以前的报道中,每13.8 kb中就有一个EST-SSR拟南芥,水稻3.4 KB,玉米8.1 KB,大豆7.4 KB,番茄11.1 KB,棉花20.0 KB,杨树14.0 KB [33].然而,由于依赖于SSR搜索标准、数据集大小、数据库挖掘工具和EST序列冗余度,难以直接比较不同报告中SSR的丰度估计和出现频率。
在早期的报道中,三核苷酸重复序列通常是两种单子叶植物中最常见的基序[19]和Dicots [17].在本研究中,三核苷酸重复也被发现是最丰富的重复单元,其次由二 - ,四 - ,五和六核苷酸(表1).如图所示1二、三核苷酸基序类型分别为AG/CT(26.9%)和AAG/CTT(13.5%)。这与最近对栽培花生(arachis hypogaea.l .) [30.],Epimudium sagittatum[32],以及许多双子叶植物[34].拟南芥以往的研究[33]和大豆[35]也提示三核苷酸AAG基序可能是双子叶植物中常见的基序。相比之下,小麦中最常见的三核苷酸重复基序为(AAC/TTG)n,水稻中为(AGG/TCC)n,玉米、大麦和高粱中为(CCG/GGC)n [31那36那37].在谷物物种中大量赞成三核苷酸CCG重复基序的丰度[31那36那38]并且也被认为是单子叶植物基因组中的特定功能,这可能是由于高GC含量和由此产生的密码子使用偏好[5.那39].但有趣的是,在本研究中,第二最主要的三核苷酸重复基序是CCG/CGG(5.8%),其次是AAG/CTT。这一结果与之前在甘薯[26],这也表明CCG重复是高丰度的三核苷酸基序之一。
甘薯EST-SSRs序列的验证与多态性分析
为了去除已发表的甘薯EST-SSR标记中可能存在的重复序列,本研究将已发表的EST-SSR标记中的引物与7163条独特的含ssr序列进行了比较。共发现336对SSR引物与本次调查的含SSR序列相匹配,后续设计引物时将匹配序列排除在外。在336个SSR引物中,王设计的引物有7对等.[27]和7对引物(其中6对由Schafleitner设计等.[28还有一张是胡歌写的等.[26])匹配相同的7个含ssr的序列(表3.).这表明七个SSR引物对放大了与其他七个SSR标记相同的SSR基因座。
基于这些含非重复SSR序列,设计了1,060个引物对,并用于验证甘薯的EST-SSR标志物。其中,897个引物对(84.6%)在我们的绘图人口的两个父母中产生了扩增子。该结果与甘薯的EST-SSR扩增速率类似[26那28]和许多其他研究也报告了60-90%的扩增成功率[37那40-43].在这些研究中(除了[26]),他们还报告了基因组SSRS和EST-SSRS的类似放大率。然而,在甘薯岛,EST-SSRS的扩增效率远高于基因组SSRS的高效率[22那26].EST-SSRs的PCR扩增效率较高,可能是因为引物设计的序列数据来自于相对高度保守的转录区域,而不是全基因组文库中的随机序列。正因为EST-SSRs来自高度保守的转录区域,所以据报道其多态性较低,但在作物植物中比基因组SSR标记具有更高的可转移性和更好的适用性[44-47].816条可扩增的EST-SSR引物将进一步用于野生菌株扩增验证和多态性评估五爪物种。
众所周知,多态SSR标记在遗传多样性、亲缘关系、进化、连锁作图、比较基因组学和基于基因的关联研究等方面具有重要意义。在本研究中,首次在我们的定位群体的两个亲本中检测了SSR引物多态性。在被检测的引物中,195个在两个作图亲本之间具有多态性。这些标记对ssr连锁图谱的构建具有重要意义。在8对工作引物中,342对(41.9%)在8个红薯中表现出多态性。该值低于早期的研究,其中62.5%和67.2%的SSRs在不同的试验集显示多态性[26那28].少量的DNA样本和来自不同地理来源的DNA样本可能导致不同的多态性。例如,当使用的品种数量从38个增加到500个以上时,木薯的多态性水平相对较高[48].另外,来自其它植物和动物物种足够公布的数据证明,三核苷酸SSR位点具有低变化比含有SSR位点[二核苷酸49-51].在我们的结果中,在重复核苷酸基序和多态性水平之间没有发现相关性(如图所示)2).
结论
在本研究中,除了对栽培甘薯的est - SSR标记进行鉴定外,我们在两个亲本中设计并验证了1060个SSR标记。有效引物中,41.9%的引物在8个甘薯品种中表现出多态性。这些开发的SSR标记将为甘薯遗传研究中的遗传多样性、进化、连锁定位、比较基因组学、基于基因的关联研究和标记辅助选择提供有价值的资源。由于这些标记是基于保守表达序列开发的,因此可能对候选基因的功能分析有价值。据我们所知,这是开发甘薯EST数据库和开发大量SSR标记的首次尝试。
方法
植物材料和DNA提取
在本研究中,我们使用了一个定位群体的两个亲本,E Shu 3 Hao和Guang 2k-30,以及8份栽培甘薯材料(见表)4.).每次加入的叶样品通过与来自国家种质苗寨苗圃的6株植物的叶组织混合,位于广东省农作物,广州市农作物,广东广州。使用改性的CTAB方法提取基因组DNA [52].分别用纳米滴分光光度计(Thermo Fisher Scientific Inc., Waltham, MA, USA)和0.8%琼脂糖凝胶电泳测定DNA质量和数量。
数据挖掘的SSR标记
由Schafleitner建立的甘薯基因指数EST序列共181,615条,包括66,418条(31,685个contigs和34733个singleton)等[28, 56516,由王开发等在本研究中使用了58681个在室内产生的。这些ESTs是使用TGICL程序组装的[29].Perl脚本被称为微卫星(MISAhttp://pgrc.ipk-gatersleben.de/misa/)用于挖掘微卫星。在这项工作中,进行搜索的序列,该序列显示为三 - ,五个重复单位的至少六个重复,以及用于四核苷酸的四个重复,排除多元和PolyT重复。SSR的频率是指含有一个SSR的千碱基对EST序列。
引物设计及PCR扩增
为了去除已发表的EST-SSR中可能存在的重复,我们利用370个已发表的EST-SSR标记(75 [26), 19528],100 [27])。每一组序列由NCBI专门的blast程序bl2seq进行比较,使用默认参数,但由于引物长度较短(18-24 bp),词大小算法参数由28更改为16 [53].
采用引物premier 6.0 (premier Biosoft International, Palo Alto, CA)进行PCR引物设计,选择重复最长的序列和可定量引物设计的侧边区域。引物设计的核心标准如下:(1)引物长度为18 ~ 24 bp;(2)熔化温度(Tm)在52 ~ 63℃之间,以60℃为最佳;(3) PCR产物大小在100 ~ 350 bp之间;(4) GC%含量在40% ~ 60%之间,扩增率大于80%。当程序检索到不合适的引物对时,对参数进行修改。当一个EST序列中存在两个不同的微卫星序列时,分别设计引物对。当两个位点在一个序列中接近时,引物对被设计在这些微卫星基因之外。
PCR分析以20μL反应混合物的总体积进行,含有40-50ng模板DNA,1×PCR缓冲液(20mM Tris pH 9.0,100mM KCl,2.0mM MgCl2), 200μM的四种核苷酸,0.2μM的正向和反向引物,和一个单位的Taq DNA聚合酶后骑车简介:1周期5分钟在94°C, 55 - 65°C的退火温度35周期(1分钟在94°C, 30年代在55 - 65°C, 45 s在72°C)和一个额外的10分钟的循环在72°C。对每个引物对进行两次筛选,以确定观察到的条带在每个基因型中的重复性。PCR产物在6%聚丙烯酰胺变性凝胶上分离。对凝胶进行银染,用于SSR条带检测。
引物筛选、评价和数据收集
首先使用E SHU 3 Hao和Guang 2K-30筛选设计的底漆对,以效果扩增预期大小的SSR片段和检测等位基因多态性。从筛选中得到有效底漆对并进一步在以下八种品种上进行评估。每次PCR反应进行两次。为分析的样品计算等位基因频率。样本作为一个整体的遗传多样性的基础上的等位基因每个位点(等位基因总数/基因座的数目),多态位点的百分比数估计(多态位点的数目/位点的总数进行分析)和多态信息内容(pic)。根据SSR基因座的存在或不存在确定多态性。使用公式计算PIC的值P我是由给定的EST-SSR引物对产生的个体基因型的频率,并且求和在N等位基因上延伸。
参考资料
- 1.
粮食及农业组织。[http://faostat.fao.org/]
- 2.
Jim C, G C, Albert K, Kenneth V, Maria A, Robert O, Bryon R:利用多剂量AFLP标记建立甘薯遗传连锁图谱和鉴定同源连锁群。分子育种,2008,21(4):511-532。10.1007 / s11032 - 007 - 9150 - 6。
- 3.
Kriegner A,Cervantes JC,Burg K,Mwanga Rom,张D:基于AFLP标记的甘薯[Ipomoea Batatas(L.)林]的遗传联系地图。分子育种。2003,11(3):169-185。10.1023 /答:1022870917230。
- 4.
李爱霞,刘春春,王启明,张利民,洪志,刘思忠:利用SRAP标记建立甘薯分子连锁图谱。AGRONOMICA学报。2010,36(8): 1286-1295。
- 5.
Morgante M,Hanafey M,Powell W:微卫星优选地与植物基因组中的非重量DNA相关。NAT Genet。2002,30(2):194-200。10.1038 / ng822。
- 6.
Taramino G,Tarchini R,Ferrario S,Lee M,Pe Me:高粱双子石中简单序列重复(SSR)的表征和映射。理论和应用的遗传学。1997,95:66-72。10.1007 / S001220050533。
- 7。
刘志刚,刘志刚,刘志刚:水稻、小麦和大麦基因组和est衍生微卫星标记的非随机分布和频率。BMC基因组学。2005年,6(1):23-10.1186 / 1471-2164-6-23。
- 8。
Edwards K,Barker J,Daly A,Jones C,Karp A:微卫星文库富集植物中的几种微卫星序列。生物技术。1996,20(5):758-760。
- 9.
关键词:微卫星,基因组文库,通用连接和连接方法生物技术。1999,27(3):500-507。
- 10.
文敏,王海霞,邹敏,陆超,王伟:麻疯树EST-SSR标记和基因组ssr标记的开发及其遗传多样性评价。植物遗传资源学报,2010,30(3):442 - 446。
- 11.
Nunome T, Negoro S, Kono I, Kanamori H, Miyatake K, Yamaguchi H, Ohyama A, Fukuoka H:从茄子(Solanum melongenl .) SSR富集文库中提取SSR标记。应用电子学报。2009,119(6):1143-1153。10.1007 / s00122 - 009 - 1116 - 0。
- 12.
iniguez-luy fl,voort av,osborn tc:开发从快速循环芸苔L.基因型的基因组的基因组序列衍生的一组公共SSR标记。Al Appl Genet。2008,117(6):977-985。10.1007 / s00122-008-0837-9。
- 13.
马勉,王志强,王志强,王志强:高羊茅基因组SSR标记的研究进展。应用计算机学报。2006,113(8):1449-1458。10.1007 / s00122 - 006 - 0391 - 2。
- 14.
王永文,吴永强:柳枝稷1030个基因组SSR标记的开发。应用计算机学报,2011,122(4):677-686。10.1007 / s00122 - 010 - 1477 - 4。
- 15.
来自高等植物基因组转录/表达区域的分子标记。功能整合基因组学。2004,4(3):139-162。
- 16.
Scott KD, Eggler P, Seaton G, Rossetto M, Ablett EM, Lee LS, Henry RJ:葡萄est衍生的SSRs分析。应用电子学报。2000,100:723-726。10.1007 / s001220051344。
- 17。
Eujayl I,Slebed Mk,Wange Mk,May Gd,Chekhovskiy K,Zwonitzer JC,Mian Ma:Medicago Truncatula Est-SSRS揭示了Medicago SPP的跨物种遗传标记。Al Appl Genet。2004,108(3):414-422。10.1007 / S00122-003-1450-6。
- 18。
张丽娟,张丽娟,张丽娟,张丽娟。小麦est - ssr基因的克隆与表达分析。acta photonica sinica, 2005, 41(4): 457 - 463。10.1007 / s00122 - 005 - 2041 - 5。
- 19.
植物基因微卫星标记的研究进展。生物技术进展,2005,23(1):48-55。10.1016 / j.tibtech.2004.11.005。
- 20.
Varshney RK, Sigmund R, Börner A, Korzun V, Stein N, Sorrells ME, Langridge P, Graner A:大麦EST-SSR标记在小麦、黑麦和水稻中的种间转移性和比较定位。植物科学进展,2017,36(5):591 - 598。10.1016 / j.plantsci.2004.08.001。
- 21.
黄志强,王志强:甘薯种质资源的简单序列重复序列分析。植物营养与肥料学报。1994,10:9-11。
- 22.
王志强,王志强,王志强,等。二倍体和多倍体甘薯微卫星的序列分析。应用网络。1999,99:123-132。10.1007 / s001220051216。
- 23.
基于微卫星的甘薯多倍性父系分析。中国科学(d辑),2002,27(6):593 - 598。
- 24.
张dp, Carbajulca D, Ojeda L, Rossel G, Milla S, Herrera C, Ghislain M:拉丁美洲甘薯品种遗传多样性微卫星分析。国际马铃薯中心1999-2000年度CIP计划报告,秘鲁利马P295-301。2001.
- 25.
Hwang Sy,Tseng YT,Lo HF:简单序列的应用重复在台湾甘薯多媒繁殖中栽培品种遗传关系。Scientia Horticulturae。2002,93(3-4):215-224。10.1016 / s0304-4238(01)00343-0。
- 26.
胡建军,田志强,田志强:甘薯微卫星标记的研究进展。中国科学:地球科学,2019,38(4):531 - 534。10.1270 / jsbbs.54.177。
- 27.
王志强,方斌,陈建军,张旭,罗志强,黄磊,陈旭,李艳:甘薯根转录组基因重组及ssr标记的构建。基因组学杂志。2010,11:726-10.1186/1471-2164-11-726。
- 28.
Schafleitner R, tinopa LR, Palomino O, Rossel G, Robles RF, Alagon R, Rivera C, Quispe C, Rojas L, Pacheco JA,等:通过重新组装焦磷酸测序和Sanger序列,并挖掘基于基因的微卫星标记建立甘薯基因指数。中国生物医学工程学报。2010,11(1):604-10.1186/1471-2164-11-604。
- 29。
Pertea G,黄X,梁女,安东内斯库V,苏丹娜R,Karamycheva S,李Y,白Ĵ,长楼Parvizi B,等人:TIGR基因指数聚类工具(TGICL):对于大快速聚类软件系统EST数据集。生物信息学。2003年,19(5):651-652。10.1093 /生物信息学/ btg034。
- 30.
梁晓霞,陈旭,洪勇,刘辉,周国刚,李胜,郭斌:est - SSR标记在花生栽培和野生花生中的应用。植物学报。2009,29(9):35-10.1186/1471-2229-9-35。
- 31.
Kantety RV,拉罗塔男,马修斯DE,ME索雷尔斯:数据挖掘在大麦,玉米,水稻,高粱,小麦表达序列标签简单重复序列。植物分子生物学。2002年,48(5-6):501-510。
- 32.
Zeng S ., Xiao G ., Guo J ., Fei Z ., Xu Y ., Roe BA ., Wang Y .: Epimedium sagittatum (Sieb) EST数据集的建立及EST- ssrs特征分析。调查)。的格言。基因组学杂志。2010,11:94-10.1186/1471-2164-11-94。
- 33.
Cardle L, Ramsay L, Milbourne D, Macaulay M, Marshall D, Waugh R:植物物理聚类简单序列重复的计算和实验表征。中国生物医学工程学报,2017,32(2):457 - 461。
- 34.
关键词:双子叶植物,表达序列标签,简单重复序列基因组学杂志。2005,48(6):985-998。10.1139 / g05 - 060。
- 35.
高磊,唐军,李华,贾军:基于计算和实验方法的主要作物微卫星分析。昆虫学报。2003,12(3):245-261。10.1023 /: 1026346121217。
- 36.
Varshney RK, Thiel T, Stein N, Langridge P, Graner A:对某些谷类植物est中微卫星的频率和分布进行了硅分析。中国生物医学工程学报。2002,17(2):537-546。
- 37.
Thiel T,Michalek W,Varshney Rk,Graner A:利用EST数据库,用于大麦(Hordeum Vulgare L)的基因衍生的SSR标记的开发和表征。Al Appl Genet。2003,106(3):411-422。
- 38.
高丽芬,景瑞林,霍娜霞,李艳,李雪萍,周瑞华,常绪平,唐建峰,马志勇,贾建忠:面包小麦EST-SSRs微卫星基因座111个。acta photonica sinica, 2004, 40(7): 1392- 1405。10.1007 / s00122 - 003 - 1554 - z。
- 39.
刘志刚,刘志刚,刘志刚:水稻、小麦和大麦基因组和est衍生微卫星标记的非随机分布和频率。生物医学工程学报。2005,6:23-10.1186/1471-2164-6-23。
- 40.
马勉,王磊,王国强:高羊茅EST-SSR标记在几种牧草上的可转移性。应用计算机学报,2004,109(4):783-791。10.1007 / s00122 - 004 - 1681 - 1。
- 41.
Cordeiro Gm,Casu R,McIntyre Cl,Manirs jm,Henry RJ:来自甘蔗(Saccharum SPP)的微卫星标记物(Saccharum SPP。)ESTS交叉可转移到Erianthus和高粱。植物SCI。2001,160(6):1115-1123。10.1016 / s0168-9452(01)00365-x。
- 42.
余克平,李伟,李文杰,李文杰,李文杰:小麦est - ssr标记的开发与定位。基因组学杂志。2004,47(5):805-818。10.1139 / g04 - 057。
- 43.
利用EST-SSR标记研究面包小麦的遗传多样性。中国生物医学工程学报。2003,27(4):337 - 341。10.1007 / s00438 - 003 - 0921 - 4。
- 44.
Rungis D,Berube Y,Zhang J,Ralph S,Ritland Ce,Ellis,Douglas C,Bohlmann J,Ritland K:来自表达的序列标签的云杉(PiceA SPP)的强大简单序列重复标记。Al Appl Genet。2004,109(6):1283-1294。10.1007 / s00122-004-1742-5。
- 45.
Russell J, Booth A, Fuller J, Harrower B, Hedley P, Machray G, Powell W:大麦基因组转录区和匿名区序列多态性和单倍型含量的比较。基因组学杂志。2004,47(2):389-398。10.1139 / g03 - 125。
- 46.
Aggarwal RK,Hendre PS,Varshney RK,BHAT PR,Krishnakumar V,Singh L:Est衍生的基因微卫星标志物的鉴定,表征和利用,用于咖啡和相关物种的基因组分析。Al Appl Genet。2007,114(2):359-372。10.1007 / s00122-006-0440-x。
- 47.
郭W,王W,周B,张T:Gosypium二倍体衍生EST-SSRS的交叉物种可转移性。Al Appl Genet。2006,112(8):1573-1581。10.1007 / s00122-006-0261-y。
- 48.
Chavarriaga-Aguiregre PP,Maya MM,Bonierbale MW,Kresovich S,Fregene Ma,Tohme J,G K:Cassava的微卫星(Manihot Esculenta Crantz):发现,继承和可变性。理论和应用的遗传学。1998,97(3):493-501。10.1007 / S001220050922。
- 49.
王永文,吴永强:柳枝稷1030个基因组SSR标记的开发。Theor Appl Genet, 2010。
- 50.
Chakraborty R, Kimmel M, Stivers DN, Davison LJ, Deka R:二、三、四核苷酸微卫星位点的相对突变率。美国国家科学院学报。[j] .地理科学进展,2019,37(3):441 - 446。10.1073 / pnas.94.3.1041。
- 51.
Schug MD, Hutter CM, Wetterstrand KA, Gaudette MS, Mackay TF, Aquadro CF:果蝇中二、三和四核苷酸重复的突变率。中国生物医学工程学报,1998,15(12):1751-1760。
- 52.
一种用于RAPD指纹和其他PCR应用的快速CTAB DNA分离技术。生物技术。1993,14(5):748-750。
- 53.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gap BLAST和psp -BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,2017,31(4):531 - 534。10.1093 / nar / 25.17.3389。
确认
感谢广东省农业科学院作物研究所陈晓平博士在SSR位点搜索方面的建议和帮助。国家现代农业产业技术研究体系专项资金(No. nycytx-16-B-5);国家自然科学基金项目(No. 31000737);广东省自然科学基金项目;基金资助:国家自然科学基金资助项目(10151064001000018);广东省农业科学院总统基金资助项目(201009)。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
ZYW构思、组织、策划了研究,并起草了手稿。LJ设计PCR引物,参与DNA提取和SSR实验。ZXL参与了引物设计和SSR实验。LFH参与底漆设计。XLC参与了聚丙烯酰胺变性凝胶的运行。BPF参与设计和协调。YJL参与了稿件的编写和修改。JYC和XJZ为SSR分析提供了植物材料。所有作者阅读并批准了最终的手稿。
作者为图像提交的原始文件
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativeCommons.org/licenses/by/2.0.)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
王志强,李军,罗志强。et al。甘薯est - SSR标记的鉴定与开发Ipomoea Batatas.).BMC植物杂志11,139(2011)。https://doi.org/10.1186/1471-2229-11-139
收到了:
接受:
发表:
关键字
- 多态性信息内容
- 基因组SSR
- 番薯的物种
- 甘薯品种
- 甘薯基因型