抽象的
背景
低再生能力限制生物技术育种方法。基因型在番茄和其他重要作物中的再生反应的影响很高。尽管已经在再生遗传学上进行了各种研究,但甚少关于该过程中涉及的关键基因知之甚少。本研究的目的是本地化影响番茄中再生的遗传因素。
结果
我们开发了两个映射群体(F2和BC.1),源自先前选定的番茄品种(cv。Anl27)的再生能力较低,而野生物种的再生次数较高Solanum Pennellii.(PE-47)。表型分析表明,外植体带芽率和每外植体再生芽数均为显性诱导和加性效应。在5条染色体(1、3、4、7和8)上建立了2个连锁图谱,鉴定出6个qtl1通过区间作图和限制性多QTL作图方法对群体进行定位S. Pennellii.,除了位于染色体8上的次要QTL,由CV提供。ANL27。主QTL对应于在F的染色体1和7上检测到的那些2群体,染色体7上的QTL在与BC中检测到的类似区域上鉴定出类似的区域1人口。在BC的qtl所在区域,该群体存在标记偏析现象1被检测到。此外,我们使用链接到高再生基因的标记物定位两个番茄候选基因:Rg-2(推定的等位基因Rg-1) 和LESK1,编码丝氨酸/苏氨酸激酶,并提出作为再生能力的标志物。结果,我们找到了一个推定的等位基因Rg-2在我们命名为3号染色体的QTL中Rg-3.LESK1它也位于3号染色体上Rg-3.通过对检测到的QTL峰的初步探索,我们发现了几个可能与再生有关的基因。
结论
在本研究中,我们发现了与组织培养再生过程相关的新的qtl。我们还定位了两个候选基因,发现了一个高再生基因的假定等位基因Rg-1在3号染色体的QTL中。所鉴定的qtl对了解这一过程和鉴定其他相关候选基因具有重要意义。这也极有可能促进用于基因分离的分子标记的发展。
背景
体外栽培番茄的再生(Solanum lycopersicum.由于这种作物的商业价值,它一直是人们不断研究的课题。因此,已经发表了大量关于从野生和栽培番茄种质的广泛组织和器官中再生植株的研究[1]这些研究表明,番茄的器官发生是一种常见的再生途径,它受到基因型以及一些物理和化学因素的强烈影响。这些报告还记录了顽抗的存在(部分或完全无法响应体外文化),大大限制了生物技术育种。高再生对于单倍体再生,遗传转化,繁殖,体细胞杂交,突变选择和种质储存等技术的成功至关重要2那3.].例如,番茄转化的低效率与使用的品种的低再生电位相关[4.那5.]此外,在某些品种中,芽可能会被诱导,但不会发育成芽[6.].为了提高低再生能力的番茄品种的再生能力,一些渐渗程序已被记录[7.-10].
这个过程体外射击器官通常涉及体细胞的激素响应,分化细胞的消化率,以获得有机源性能力,响应细胞的细胞分裂以及从新分区的新射击开始和开发新的射击,无论是直接的或间接通过愈伤组织阶段[11那12].因此,许多基因可以参与该复杂过程的不同步骤。例如,cdc2基因表达,编码p34,一个关键的细胞周期调节,已被提出作为一个指标的能力,以分裂[13].编码或调节细胞蛋白和生长素的基因可能会显然影响再生。两种类型的增长监管机构均协同促进细胞划分和拮抗促进来自愈伤组织培养物的芽和根源[14].在拟南芥,一种编码细胞分裂素受体(CRE1/AHK4)的组氨酸激酶(AHK)基因已被鉴定[15那16]并将其他AHKS联系到细胞划分和监管[17].关于芽形成的开始,报告的最表征基因是ESR1,当过表达时,它能在茎中形成不依赖细胞分裂素的芽拟南芥根外植物[18].ESR1编码属于乙烯响应因子(ERF)家族的转录因子,并在亚组VIII-B中分类。这ESR2.编码与之非常相似的蛋白质的基因ESR1似乎具有调节拍摄再生的冗余功能[19].其他拟南芥ERF VIII-B亚组基因的表达模式也可以参与拍摄再生的早期事件[20].
番茄再生的遗传分析表明,显性等位基因决定了高再生能力[7.那21-24].然而,有关涉及的基因数量尚未达成共识。例如,Koorneef等人[25]根据单基因、双基因或三基因模型获得的再生分离率取决于试验番茄品系,尽管这些品系本身都不能从根外植体再生芽。在这项研究中,一个显性等位基因美国peruvianuml . (Rg-1),它决定了番茄根外植体的有效再生,被定位在靠近3号染色体的中间。此外,一个假定的等位基因Rg-1从S..赤仁(Dunal) Reiche (Rg-2Takashina等人报道了)。[9.]和satoh等人。[22]这两个等位基因可能与番茄或野生近缘种的其他等位基因结合作用美国peruvianum或者S. Chilense.[22那25].另一方面,Torelli等人。[26]通过信使rna差异显示技术鉴定出一个cDNALESK1基因,其表达在激素处理下特异性和瞬时增强,导致茎发生(芽诱导)。该基因编码一种假定的丝氨酸-苏氨酸激酶,并被报道为体外番茄中的填充剂标志物[27那28].
尽管正在进行基因控制的研究体外番茄和其他作物中的培养性状,仍然没有足够的信息有关哪种关键基因对低或高再生能力负责,也没有涉及的基因数量。据报道的基因和其他可能被识别的研究和表征可以大大提高我们对番茄不同阶段的分子机制的理解体外器官形成。在本研究中,我们开发了两个映射群体(F2和BC.1)从美国lycopersicum(作为循环父母)和S. Pennellii.Correll(作为再生父代)进行了基于qtl的分析。本文报道了在5条染色体上鉴定的6个qtl。这些qtl具有高显著LOD分数,共同代表了高比例的表型变异。我们也报道了与QTL峰相关的标记。此外,我们还定位了两个候选基因,Rg-2和LESK1,并对位于QTL峰位的基因进行了初步搜索。我们的发现将补充现有的遗传再生知识,并促进分子标记的发展,用于番茄育种和基因分离。
结果
开发人口和再生能力评估
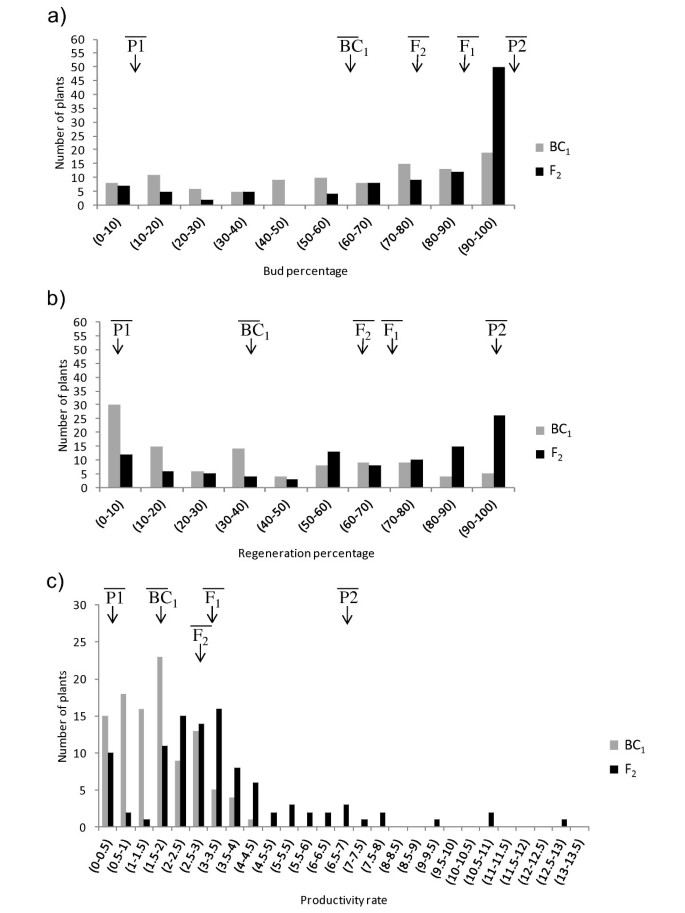
两个映射总体,F2和BC.1从低再生品种番茄(cv.;Anl27)和S. Pennellii.(PE-47)。BC1以番茄品种为轮回亲本获得群体。在第一次试验中,亲本和F1通过叶片外植体在茎部诱导培养基上的培养,鉴定用于获得定位群体的植株。再生发生时,愈伤组织很少发育,可以认为是直接的。正如所料,S. Pennellii.和F.1外植体表现出更高的再生潜力与美国lycopersicum外植体(P <0.001)。芽(b)的外植体的百分比S. Pennellii.100%,而在番茄CV中仅获得10%。ANL27(表1).在f中获得的数据1对于B没有与此获得的B有关S. Pennellii..外植体带芽率(R)和每个带芽外植体的再生植株数(PR)也较高S. Pennellii.和F.1比在简历中。ANL27。但是,对于这些特征(R和PR),F1这些值与S. Pennellii.(桌子1).
F.2和BC.1使用来自父母和F的外植体进行再生的种群进行评估1植物作为控件(表1).表型显示在附加文件中1.对每个特征获得的分布以及该测定中的对照装置的分布如图所示1.F的B,R和Pr的平均值2人口在f之间1番茄(P1),但向F倾斜1.对于公关特质,F2植物的生长范围高于S. Pennellii.父母(P2)。这可以看作是海侵分离。公元前1产生了B、R和PR的平均值,它们介于F之间1和cv。ANL27(图1).
B和R显示高相关(r = 0.88 / 0.79 p <0,001 for f2和BC.1数据分别介绍控制这些特征的常见或链接基因。Pr和B和R两者之间的相关性较低(r = 0.56 / 0.52 p <0,001; 0.66 / 0.66 p <0,001,用于PR和B和R2和BC.1表明不同的基因可能会影响PR的性状,并且/或不同生物样本之间的差异在PR中较高。
联系地图
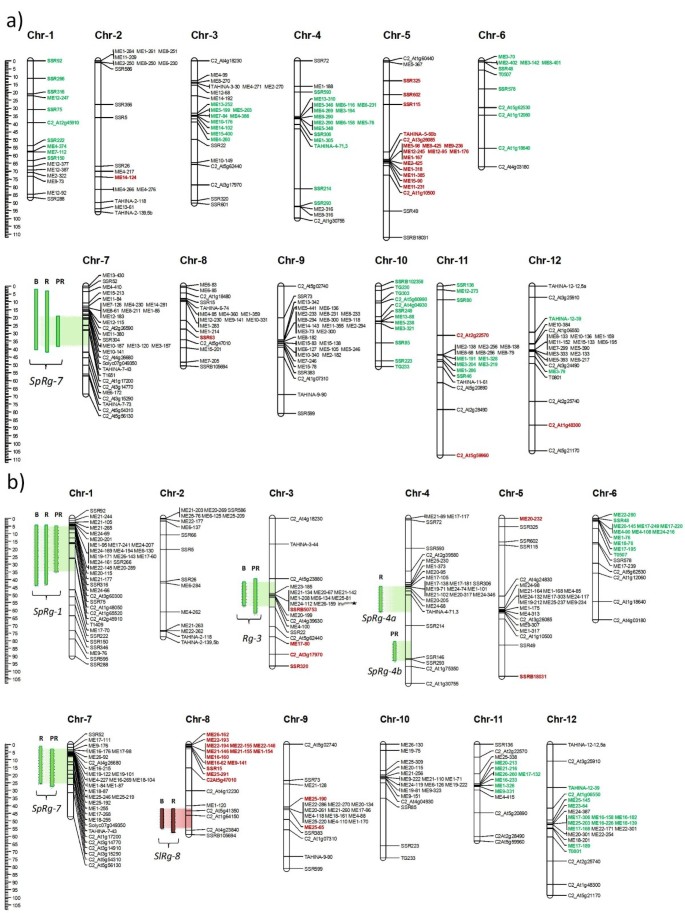
遗传连锁图谱由106个F2和113 BC.1植物与SSR,Cosi,Cosii,Caps和AFLP标记进行基因分型(图2).在149SSR和97其他标记(86 COSII,6 COSI,5章)中,测定,78SSR和59(51 COSII,4 COSI,4章)标记表现出CODOMINANT多态性。这些标志物是从Sol基因组学网络(SGN)网页获得的http://www.sgn.cornell.edu/除了按材料和方法中描述的程序之后设计的60个SSR(见附加文件)2).
a)f的番茄遗传联系地图2人口来源于美国lycopersicum(CV。ANL27)×S. Pennellii.(PE-47)和再生性状的qtl.b)BC的番茄遗传联系地图1人口来源于美国lycopersicum(CV。ANL27)×F1(简历。(Anl27 × PE-47)和rMQM检测到的再生性状qtl。将分离的数据分为12个连锁组,对应于Tomato-EXPEN 2000图谱;斜体表示分离标记显著偏向亲本等位基因(P < 0.05)。颜色表示分离扭曲的方向(红色:标记向栽培番茄等位基因倾斜;绿色:标记偏向于野生亲本的等位基因)。绿色条柱反映来自S。Pennellii.:SPRG-1,RG-3,SPRG-4A,SPRG-4B和SpRg-7; 红色条反映了SlRg-8来自S。Lycopersicum..再生性状:B(芽百分比),R(再生百分比)和PR(生产率)。黑星标记酸转化酶基因(INVpen)标记在3号染色体上Rg-3QTL系列。
对于F.2联系地图(图2a),使用总共246个多晶型基因座,包括151 AFLP,53SSR,35 COSII,3 COSI和4个帽标记。标记物在12个连接基团中对齐,LOD评分≥3.0。每个连杆基团的平均标记数为20,并在所有12个连杆基团上分布出标记物。F.2地图跨越963.85cm,相邻标记之间的平均间隔为3.72厘米。染色体2,4,5和11中有五个间隔> 25厘米。总共268个多晶型基因座用于组装BC的遗传联系地图1(数字2b),包括174个AFLP、46个SSR、43个COSII、3个COSI和2个CAPS标记。标记分布在12个连锁群中,LOD评分≥3.0。每个连锁群的平均标记数为22个。标记间遗传距离为1014.94 cM,相邻标记间平均遗传距离为4.12 cM。标记在12个连锁群中分布良好,在第5和第10染色体中只有2个区间≥25 cM。标记在两种图谱中的分布表明,它们将有助于标记所研究的性状。
SSR标记的订单和安置与美国lycopersicumXS. Pennellii.除TAHINA-6-64 (在Silico.设计),预计将定位在染色体6上(位置64),但在我们的f中定位在染色体8(位置8.85)上2地图。
扭曲的隔离
42.45%的映射标记显着偏离了F的预期1:2:1分离率2P<0.05时的世代(图2a).在染色体1(0.00-63.17cm),3(33.24-38.85cm),4(19.74-92.09cm),5(12.60-72.26cm),6(0.00-55.38cm),6(0.00-55.38厘米)和10(0.00-51.24厘米)。SDM通常由盈余造成的S. Pennellii.纯合子,除了在染色体5上观察到。
在BC1人口(图2b), sdm(30.3%)少于F2,主要观察到染色体6(0.00-6.75cm),8(0.00-15.40cm),11(25.80-27.48cm)和12(28.23-60.63cm)。染色体8上的畸变是由番茄纯合的过剩引起的,而其他染色体的扭曲是由过量的混合基因型引起的。
QTL鉴定
为了识别QTL,我们首先使用的时间间隔映射(IM)分析,导致F中的一个QTL的识别2公元前六个人口和六人1人口(见其他文件3.那4.那5.和6.).在F2中识别的QTL,位于染色体7上,重叠为三个特征。在BC1分析,该QTL也出现了R和PR特征。然而,在这种群体中,在染色体1,3,4(在两个不同的区域:4a和4b)和8上鉴定出另外五个qtls。通过限制多个QTL映射(RMQM)分析来确认所有这些QTLS(图2b、表2)除8号染色体上的QTL外,所有QTL均来自S. Pennellii..这些qtl按其来源命名,SP.为S. Pennellii.或者sl为美国lycopersicum, 其次是rg.(参考再生)和它们位于其上的染色体的数量。
植物再生性状的qtl定位1人口
芽百分比(b)
IM分析确定了染色体1和8上的两个QTL(SpRg-1和SlRg-8;额外的文件4.).SpRg-1最大LOD得分为5.87,由标记SSR316和ME17-70跨越。该QTL解释了B特征的表型变异的22.9%。SpRg-8,最高LOD值为2.8,包括只是C2_At1g64150标记,在B. rMQM分析解释了表型变异的11.7%,使用C2_At2g45910(染色体1)和C2_At1g64150(染色体8)标记作为辅因子,证实了这些的QTL由IM检测并在染色体3上检测到一个新的一个(图2b额外的文件4.).QTL性状见表2.总的来说,这些QTL分别在IM和RMQM中解释了34.6%和48.3%的表型差异。
再生率(R)
IM分析发现了3个qtl,分别位于第1、4和7染色体上SpRg-1, SpRg-4a和SpRg-7, 分别。三个QTL的最大LOD分数为4.20,3.92和3.86,每个QTL的含量约为16-17%的表型变异(见附加文件5.).RMQM分析,使用C2_AT2G45910(染色体1),Tahina-4-71.3(染色体4)和Tahina-7-43(染色体7)标记作为辅助actors,证实了IM检测到的所有QTL并检测到SlRg-8QTL(图2b额外的文件5.、表2).在这种情况下,每个QTL解释的表型变异的百分比为15%SpRg-1,13.3%SpRg-4a,14.9%SpRg-79.3%,SlRg-8.总的来说,这些QTL分别在IM和RMQM中解释了48.7%和52.5%的表型差异。
生产率(PR)
IM检测到之前在第1、3和7号染色体上定位的B和R的qtl(图)2b额外的文件6.),以及染色体4上的另一个QTL,以计值SpRg-4b.PR最大表型变异(17.4%)解释为SpRg-7,最低(11.9%)在染色体上的QTL 3. RMQM分析,使用SSR92(染色体1),ME20-199(染色体3),SSR146(染色体4)和Tahina-7-43(染色体7)标记Cofactors,确认了IM检测到的QTL(表2).
测绘番茄候选基因
我们选择了酸性转化酶基因连接到Rg-2再生基因的S. Chilense.[22)和LESK1基因,在番茄中被描述为体外再生能力(27,作为番茄的候选基因。
酸性转化酶基因标记物(挪威pen)产生不同大小的片段:162bp美国lycopersicum变异系数Anl27和173 bpS. Pennellii.(请参阅附加文件7.).因此,发票pen用于映射BC1人口(图2b,其他文件4.和6.).它位于C2_AT5G23880和SSRB50753标记的C2_AT5G23880和SSRB50753标记之间检测到的QTL,分别在49.9cm和49.93cm之间。出于这个原因,我们将此命名为此QTLRg-3(推定的等位基因Rg-2).
这LESK1基因位于标记C2_AT4G18230和CLPT-5-E7(7-15cm)之间的3型染色体3上的SGN番茄植物200型图中。因此,在我们的BC中1地图,LESK1必须置于C2_AT4G18230和TAHINA-3-44(7-44厘米)之间。因此,该候选基因位于位于Rg-3QTL。
探索QTL
利用国际番茄注释小组在SGN提供的番茄基因组官方注释,对鉴定的QTL峰附近的相关再生基因进行初步搜索。我们发现了一个组氨酸激酶SpRg-7,在所有鉴定的QTL中,几种丝氨酸/苏氨酸激酶,乙烯响应因子(ERF)中的所有QTL除外SpRg-4b细胞周期蛋白在SpRg-1、Rg-3 SpRg-4a和SpRg-7和MADS-boxSpRg-1, SpRg-4a和SpRg-7.
讨论
野生番茄S. Peruvianum,S. Pimpinellifoliuml和S. Chilense.作为再生基因的来源,用于研究体外番茄再生[7.那9.那21].在本研究中,我们使用了S. Pennellii.(PE-47)作为高再生母体[29].这种加入以及先前选择的低再生番茄品种(CV.Anl27),用于开发两个映射群体(F.2和BC.1).使用血栓引入线S. Pennellii.在M82番茄背景[30]由于两个亲本系的再生能力都很高(数据未显示),因此先前已排除进行此分析的可能性。图中的数据1和表格1这与其他研究结果一致,根据所研究的番茄再生性状,也有不同程度的显性[21那22那24那25].B和R特征在群体中表现出高的相关性,表明常见或链接基因控制这些性状。PR和B和R两者之间的相关性较低。这意味着其他基因可能会影响PR的PR特性和/或PR之间的不同生物样品之间的变化(例如,由于在类似的外矫形区域中存在不同芽的发生而导致的发展竞争)。因此,低样本大小也可以是对较低相关的可能解释。
F的一些后裔2种群表现出的PR性状的表型比再生亲本表现的更为极端(图)1).在其他有关植物再生的遗传控制的报告中已经描述了越界分离[31-33],并提出多生遗传[34].这也表明存在促进和抑制的等位基因,体外再生,只有一些等位基因,在同一父母中发生了积极影响[34].事实上,在这项研究中,SlRg-8有助于再生的QTL来自低再生父母。
培养组织的植物再生被认为是在定量遗传学下进行的[34],尽管番茄中有证据[22那25]和其他蔬菜[35-37表明只有少数基因可能负责再生。我们在BC中鉴定了6个qtl1分析,表示在该角色中大量基因的参与。这些QTL位于染色体1,3,4,7和8上(图2b).每个QTL解释的变异百分比在7.4 - 27%之间,与植物组织培养响应QTL遗传定位中最常见的范围(6-26%)一致[34].我们使用了三个特征(B,R和PR)作为测量再生能力,这对于检测在不同时间作用的染色体区域可用于检测。
在F2群体中,所有分析性状仅鉴定出7号染色体的QTL3.);在大多数染色体区域观察到的SDMS在BC中检测到QTL的区域1人口最有可能是原因(图2).在Tomato-EXPEN 2000图谱中也观察到1、3、6、10和11染色体上的SDMs [38].当QTL和SDMS紧密相关时,SDMS会影响QTL的检测力[39],如我们的案件发生。与预期隔离比率的偏差是特异性番茄交叉的共同特征[40].智力:在一个f2人口从美国lycopersicumXS. Pennellii.,de Vicente和坦克利[41]报告的偏斜率高达80%。
在BC1种群中,为B检测了三个QTLS:SPRG-1,SPRG-3和SlRg-8.这些QTL可能与再生的第一阶段相关联,即荷尔蒙感应响应和芽形成。SpRg-1对B的解释率最高(23.9%),对R和PR的解释率也最高。由于芽的形成是产生芽的必要前提,本以为B的这个主要QTL在R和PR上,事实证明是这样的2).对于R和Pr,染色体7上的常见QTL(SpRg-7)也被识别出来。另外,检测到两个QTL(SpRg-4a和SlRg-8)及公共关系(SpRg-4b和Rg-3).所有这些QTL似乎都参与了芽的发展。在该研究中也可以观察到,在不同的研究中报道了与与芽诱导相关的不同再生性状的常见QTL,以及与植物开发相关的特征的QTL的数量QTL。42那43].例如,在拟南芥中,Schianterelli等。[43]发现所有分析的参数中的染色体1的常见区域,染色体4中的峰值4和另一种在染色体5中,当它们分析再生芽的总数时。在小麦,本amer等。[42]鉴定出3个qtl,其中2个影响绿斑起始和芽再生,3个只影响植株形成。
莫里纳和Nuez报道了控制各种类型的外植体的再生频率的部分常见遗传系统[36在甜瓜中。这表明使用不同的外植体用于基因座检测可能导致某些常见QTL的识别,而且可以识别其他新QTL的识别。Koornneef等人使用根外植物。[25]和satoh等人。[22,此时在番茄的3号染色体上定位了2个具有再生能力的等位基因。本研究利用叶片进行表型分析,得到一个QTL (Rg-3)除了影响再生的其他QTL之外,还检测到染色体3的相似区域,并且在染色体1,4,7和8上鉴定了QTL鉴定的根和叶片外植体的差异拟南芥蒂利亚纳[43].
Koornneef等人[25找到了一个显性等位基因美国peruvianum(Rg-1)靠近染色体3的中间部分,它决定了番茄根外植体的有效再生。佐藤等[22]映射了一个推定的等位基因(Rg-2)从S. Chilense.在这条染色体。酸性转化酶基因,被报道为与Rg-2选择映射Rg-2在我们的人口中衍生出来S. Pennellii..在我们的父母中检测到的多态性使我们能够在我们命名的3号染色体上检测到的QTL中绘制该基因Rg-3我们考虑Rg-3成为一个推定的等位基因Rg-2基因。必须确认等位基因。
另一个被选为候选基因是LESK1,它编码一个丝氨酸/苏氨酸激酶,并被报道为一个能力标记体外番茄再生[27那28].该基因定位在3染色体上,但它不位于Rg-3QTL。
最近释放的番茄的全部基因组序列提供了一种用于询问QTL数据的强大工具。在这方面,我们已经采取了位于检测到的QTLS的峰面积的基因,并且可能与器官发生有关。据报道组氨酸激酶作为细胞蛋白受体[15-17].在我们的QTL峰中,只有一个组氨酸激酶位于SpRg-7 QTL。候选番茄基因,LESK1,它被描述为体外编码一个丝氨酸/苏氨酸激酶。我们寻找丝氨酸/苏氨酸激酶,并在所有鉴定的qtl中发现了这类蛋白。其他假定的候选基因可能是ESR1和它的副词,ESR2.,来自拟南芥,这是与再生相关的最佳特征的基因[18那19].这些基因乙烯响应因子(ERF)的代码。我们发现包含AP2域的ERF,在所有分析的QTL中除外SpRg-4b.与细胞分裂有关的细胞周期素[13]被发现SpRg-1、Rg-3 SpRg-4a和SpRg-7.疯了箱基因,与不定增殖诱导和调节相关[44那45],被发现在SpRg-1, SpRg-4a和SpRg-7QTL峰值。
结论
本研究中获得的结果可能很好地代表了理解番茄组织培养和再生反应的过程的目标的重要一步。我们在染色体1,3,4,7和8上位于六QTLSS. Pennellii.和一个来自美国lycopersicum.最重要的QTL是SpRg-1,这很可能与形态发生反应有关SpRg-7,促进芽的发育。在3号染色体上检测到一个QTL,Rg-3,可能包含一个推定的等位基因Rg-1和Rg-2基因,如通过将酸转化酶基因映射到链接的酸转化酶基因所示Rg-2.在染色体8和4上检测到QTL最可能含有影响芽形成和发育的基因。
方法
植物材料和生长条件
S. Pennellii.PE-47,显示出高能力的再生能力[29番茄品种Anl27 (cv.;选取再生能力较低的Anl27)作为测图群体。初步的基因型已经建立体外,在25%商业漂白剂溶液中浸渍10分钟,从种子的灭菌开始(40 g l-1活性氯),然后用无菌去离子水洗涤两次,每次3分钟,然后在含有营养培养基的培养皿中播种(Murashige和Skoog [46盐,包括维生素,2%的蔗糖,0.6%的植物琼脂(DUCHEFA,荷兰)。培养基ph调整至5.8,在121°C灭菌20分钟。培养物在26°C±2°C的生长室中培养,光周期16h,使用Sylvania的冷白光F37T8/CW荧光灯(90 μmol m-2S.-1).获得了各基因型各一株的无性系,并保持了该无性系体外文化。通过用新鲜基础培养基将节点转移到管子(BM:Murashige和Skoog - [46] - 盐,包括维生素,1.5%蔗糖和7升L.-1植物琼脂),每3-4周一次。试管长15厘米,直径22毫米,每管15毫升培养基。
映射人口
一个番茄的克隆和另一个克隆S. Pennellii.被转移到温室以获得f1被重新引进的植物体外通过对种子灭菌的类似程序消毒射击。F2和BC.1获得群体,种子萌发体外如上所述。
F.2作图群体由106个自交个体组成1植物,番茄CV之间的交叉的结果。ANL27(P1)和S. Pennellii.PE-47 (P2)。回车(BC1)由113植物组成的映射群是通过穿过CV而获得的。ANL27和F.1植物。为了使试验得以复制,F1植物与F2和BC.1个人被克隆复制和维持体外如上所述。
评估再生能力
用克隆的P1,P2和F进行第一测定1植物。叶盘(0.6-0.8厘米2) 从...获取体外在与含有Murashige和Skoog Salts的芽诱导培养基(SIM)接触的释放侧,将培养的植物置于与含Murashige和Skoog盐的芽诱导培养基(SIM)接触的植物中46],3%蔗糖,7%植物琼脂和0.2毫克L.-1玉米素核苷(ZR)。该生长调节剂通过过滤灭菌并添加到无菌SIM中。在SIM培养基上培养30天后,将外植体转移到BM上培养20天。在这种培养基中,芽发育成芽。每个基因型每个板5个外植体(90 × 25 mm,培养液40 ml),每个基因型10个重复。在实验结束时,对以下变量进行了分析:
芽率(B):带芽外植体数量× 100/培养外植体总数。
再生百分比(R):分化为完全发育的芽×100 /培养的外植体总数的培养物。
生产力(PR):完全发育的枝条总数/可再生植株的培养外植体总数。
在第二项试验中,F2,BC1,p1,p2和f1如上所述测试植物。在这种情况下,针对每个基因型,每块基因型和每种基因型的4个重复进行评估。获得的102个基因型的再生数据2BC的人口和104种基因型1.QTL分析采用各性状和基因型的平均值。
为了评估基因型对再生能力的影响,来自遗传均匀类的数据(P1,P2和F.1经过对方差(ANOVA)的统一性分析,然后通过Duncan测试分离不同特征的方法。使用Statgraphics Plus 4.0软件计算不同特征之间的相关性。
基因分型
基因组DNA的制备
年轻的叶子从体外收集培养植物并立即用液氮冷冻,然后储存在-80℃。基于Doyle和Doyle的改性CTAB方法制备DNA [47].随后,在0.8%琼脂糖凝胶上用溴化乙啶染色并使用NanoDrop对DNA的质量和数量进行评估®ND-1000分光光度计。
扩增的片段长度多态性(AFLP)程序
在Vos等人之后获得了AFLP。[48] 程序。FINA的十五和十六个选择性组合用于F.2和BC.1分别数量。每个选择组合的代码在表中指定3.. 每个编码后跟每个获得的带(大小以bp为单位)对应的数字用于命名多态性AFLP。PCR产物的电泳使用ABI PRISM 310基因分析仪(美国加利福尼亚州福斯特市Perkinlemer应用生物系统公司)进行。基因扫描™ 600利兹®使用带有荧光团LIZ的大小标准作为分子大小标记。使用GeneScan 3.1.2分析软件(Perkinlemer Applied Biosystems)分析原始数据,并将生成的GeneScan跟踪文件导入Genographer 1.6.0。60至380 bp之间的AFLP片段在Genographer中按存在(1)或不存在(0)进行评分。
微卫星
使用一百四十九个SSR标记来检测P1和P2之间的多态性,其中包括先前报道的89个SSR,并映射到SGN的番茄植物2000上[49那50]以及60个新的SSRS:18来自Comav Research Group“Aprovechamiento de la Variabilidadestraespecíficaenla Mejora del Tomate”,42设计于Genbank(见附加文件)2).利用SSR Primer 3工具从这些序列中设计引物对http://frodo.wi.mit.edu/[51].用于设计引物的标准如下:底漆Tm的范围为55至65℃,GC含量为50%。考虑到来自引物的3'末端(GC夹)的3'末端(GC夹)的最后五个碱基碱基的存在,这有助于促进在3'末端在3'末端促进特异性结合。为了设计SSR,无论如何,基于Frary等人获得的结果选择AT / TA重复。[49].
除以下所列ssr外,所有ssr均采用Schuelke等人描述的M13-tail方法进行标记[52].使用10 ng DNA的样品以15μl的体积进行DNA扩增。反应混合物含有1.5μL10×PCR缓冲液[75mM Tris-HCl(pH9.0),50mM KCl,20mM(NH4.)2所以4.和0.001%bsa],2 mm mgcl2,200μmdntps,0.133μm引物,0.2μm荧光标记的m13底漆和0.3单位Taq.I DNA聚合酶(需要S. L.,瓦伦西亚,西班牙)。使用EPPendorf 5333热循环仪。PCR参数包括以下内容:94°C的初始3分钟;35个循环,每个循环有30秒的DNA变性在94°C;在退火温度(取决于底漆组合Tm)和72℃下的1分钟的延伸,在72℃下的最终延伸10分钟。使用Li-Cor测序凝胶(DNA Li-Cor 4300; Li-Cor Biosciences,Lincoln,Nebraska,Nebraska,USA)可视化扩增的条带;将10μl加载缓冲液(95%甲酰胺,2mM EDTA,0.001%溴苯酚蓝)和5μL去离子水加入到5μLPCR混合物中(2.5μL每次IRDYE 700或IRDDE800标记的)样品,在96°C 8分钟。在50℃的变性条件下在TBE缓冲液中使用6%丙烯酰胺凝胶进行电泳。
SSR356,SSR73,SSR248,SSR46标记,其中多态性在琼脂糖凝胶中可见,其体积为23.32μL,其中10ng DNA,1.6mM MgCl2,171.52μmdntps,0.214μm引物,2.5μl10×PCR缓冲液,和0.6 uTaq.I DNA聚合酶。PCR条件与以前施加的PCR条件相似,除了在这种情况下最终延长30分钟。在100V的TaE缓冲液中,在标准琼脂糖凝胶(1或2%)中以100V的标准琼脂糖凝胶(1或2%)进行扩增的带,并被溴化乙锭染色。
COS (Conserved ortholog set)和CAPS (cleaved amplified polymorphic sequence)标记
96个标记(86个COSII, 6个COSI, 4个CAPS)来自Tomato-EXPEN 2000地图[53那54]和一个已开发的CAPS标记(Solyc07g049350)检测P1和P2亲本之间的多态性。需要时使用的限制性内切酶是SGN数据库中显示的酶。当需要限制酶时,遵循商业产品说明书中描述的方案(Fermentas,约克,英国或Biolabs, Takara,日本)。
使用10ng DNA的样品,在总体积为12μl的PCR反应中进行。反应混合物含有1.5mM MgCl2,200μmdntps,0.25μm引物,1.2μlpcr缓冲液10x,0.3uTaq.I DNA聚合酶。使用EPPendorf 5333热循环仪进行扩增,其如下编程:5分钟在94℃,35℃下,在94℃下,TA(取决于底漆组合Tm)和2分钟在72℃下延伸,最终阶段为10分钟,72°C。在100V的TAE缓冲液中在TAE缓冲液中分离出扩增的条带,并被溴化乙锭染色可视化。
地图结构和QTL映射
使用JoinMap进行映射群体的连接分析®4.0软件(55].LOD≥3的标记被分组为连锁组,除了BC染色体9和10的标记1绘制LOD≥2的种群。顺序是用0.40的重组阈值确定的,距离是用Kosambi映射函数计算的(Kosambi 1944)。遗传图谱构建采用AFLP、SSR、COS和CAPS标记(附加文件)8.).通过Chi-Square试验评估等位基因的分离比,其具有P <0.05的显着性阈值。预期的隔离比为3:1和1:12和BC.1, 分别。使用MapChart软件创建标记映射的可视化表示[56].
F2和BC.1表型数据集用MapQTL进行®6.0软件[57].LOD值的显著性阈值,对应于全基因组错误发现率为5% (p < 0.05),通过使用1000个排列进行全基因组排列试验计算。首先进行IM分析(简单区间映射)。然后,如果IM检测到许多推测的qtl,则将检测到的qtl的可能峰值附近的标记作为rMQM(也称为复合区间作图)分析的辅助因子。
定位候选基因并寻找其他再生相关基因
这S. Pennellii.酸性转化酶基因pen分析并映射为标记物。Harada等人描述的引物。[58使用先前描述的COS和帽标记物的条件用于DNA扩增。使用多功能电泳Qiaxcel系统(Qiagen,Valencia,California,USA)分离扩增的带。我们搜查了所在地的位置LESK1在国际番茄注释组的SGN基因及其最近的标志。该数据库还用于寻找与器官发生相关的基因。
参考
- 1.
Bhatia P,Ashwath N,Senaratna T,David M:番茄组织培养研究(Lycopersicon esculentum).植物细胞组织器官培养。2004, 78: 1-21.
- 2.
Engelmann F:热带植物种质的体外保护。Euphytica。1991,57:227-243。10.1007 / BF00039669。
- 3.
Benson EE:特殊研讨会:体外植物重新分布:介绍。体外细胞驱动Biol植物。2000,36:141-148。10.1007 / s11627-000-0029-z。
- 4.
Hamza S,Chupeau Y:重新评估植物再生和农杆菌介导的番茄转化的病症(Lycopersicon esculentum).J Exp Bot。1993年,44:1837-1845。10.1093 / JXB / 44.12.1837。
- 5.
农杆菌介导番茄转化效率的影响因素研究。植物学报,1996,16(4):457 - 461。
- 6.
佩雷斯LEP,Morgante PG,Vecchi C,Kraus Je,Sluys Mav:从根系和番茄品种和野生相关物种的根和转基因毛状根的拍摄再生能力。植物细胞组织器官邪教。2001,65:37-44。10.1023 /答:1010631731559。
- 7.
Koornneef M,Hanhart CJ,Martinelli L:番茄中细胞培养性状的遗传分析。Al Appl Genet。1987,74:633-641。10.1007 / bf00288863。
- 8.
Wijbrandi J, Vos JGM, Koornneef M:再生能力的转移Lycopersicon Peruvianum.到五味子通过原生质体融合。植物细胞组织器官邪教。1988年,12:193-196。10.1007 / BF00040085。
- 9.
Takashina T,Suzuki T,Emashira H,Imanishi S:与野生番茄种植的高拍摄再生能力有关的新分子标记。品种sci。1998年,48:109-113。10.1270 / JSBBS1951.48.109。
- 10。
关键词:水稻,离体培养,再生能力,基因工程abstractLycopersicon pimpinellifolium磨。进入顽皮的番茄品种。Euphytica。2002,124:59-63。10.1023 /答:1015693902836。
- 11
Sugiyama M:器官发生在体外。植物生态学报。1999,2:61-64。10.1016 / s1369 - 5266(99) 80012 - 0。
- 12
张S,LEMAUX PG:体外芽组织的分子分析。CRIT Rev Plant Sci。2004,23:325-335。10.1080 / 07352680490484569
- 13
Hemerly AS,Ferreira P,de Almeida Engler J,Van Montagu M,Engler G,Inze D:cdc2a在拟南芥中的表达与细胞分裂能力有关。植物细胞。1993,5:1711-1723。
- 14
植物组织培养中生长和器官形成的化学调控体外1957年,11:118-131。
- 15
井上T,东口M,桥本Y,关木M,小林M,加藤T,塔巴塔S,Shinozaki K,Kakimoto T:从拟南芥中鉴定CRE1作为细胞分裂素受体。自然。2001,409:1060-1063。10.1038/35059117。
- 16
Ueguchi C,Sato S,Kato T,Tabata S:Cytokinin-信号通路中涉及的AHK4基因作为直接受体分子拟南芥蒂利亚纳.植物和细胞生理学。2001,42:751-755。10.1093 / PCP / PCE094。
- 17.
Nishimura C,Ohashi Y,Sato S,Kato T,Tabata S,Ueguchi C:编码细胞蛋白受体的拟南芥组氨酸激酶基因的遗传分析揭示了它们在调节芽和根系生长中的重叠生物学功能拟南芥蒂利亚纳.植物细胞。2004,16:1365-1377。10.1105 / TPC.021477。
- 18.
Banno H,Ikeda Y,Niu QW,Chua NH:拟南芥过度表达ESR1诱导芽再生的启动。植物细胞。2001,12:2609-2618。
- 19.
ikeda y,banno h,niu qw,howell sh,chua nh:the芽再生促进剂2拟南芥的基因调节杯形子叶1在转录水平和对照胞内发育。植物细胞生理。2006,47:1443-1456。10.1093 / PCP / PCL023。
- 20.
Mase H, Hashiba M, Matsuo N, Banno H:拟南芥ERFⅷ-b亚群基因在离体芽再生过程中的表达模式及其过表达对芽再生效率的影响生物技术学报。2007,24:481-486。10.5511 / plantbiotechnology.24.481。
- 21。
Faria RT, Illg RD:遗传体外番茄中的植物再生能力。Braz J Genet。1996,19:113-116。
- 22。
Satoh H,Takashina T,Escalante A,Egashira H,Imanishi S:分子标记围绕高拍摄再生能力基因映射Rg-2在Lycopersicon chilense.品种sci。2000,50(4):251-256。
- 23。
Pratta G, Cánepa LN, Zorzoli R, Picardi LA:属离体培养性状的双列分析Lycopersicon.Hortscience。2003,38:110-112。
- 24。
马基奥尼·贝,陈志强,陈志强:番茄离体培养反应的遗传分析。植物营养与肥料学报。2007,28(4):429 - 434。10.1007 / s11240 - 006 - 9195 - y。
- 25。
Koornneef M,Bade J,Hanhart C,Horsman K,Schel J,Soppe W,Vekerk R,Zabel P:番茄中基因控制射击再生的特征和映射。工厂J.1993,3:131-141。10.1111 / J.1365-313x.1993.tb00016.x。
- 26。
Torelli A, Soragni E, Bolchi A, Petrucco S:新的潜在标记体外通过mRNA差异显示鉴定的番茄形态发生。植物mol biol。1996年,32:891-900。10.1007 / BF00020486。
- 27。
Torelli A,Soragni E,Borinato M,Branca C:表达LESK1沿番茄胚轴轴的形态发生标志物与抗芽再生的位置依赖能力联系起来。植物SCI。2004,166(1):179-190。10.1016 / J.Plantsci.2003.09.006。
- 28。
Torelli A,Borinato M,Soragni E,Bolpagni R,Bottura C,Branca C:荷尔蒙治疗的延迟调节表达LESK1,一种编码推定的丝氨酸苏氨酸激酶的基因,番茄中体外填充性的标记物(Lycopersicon esculentum机)。植物科学学报,2004,167(3):607-620。10.1016 / j.plantsci.2004.05.007。
- 29。
Gisbert C, Arrillaga I, Roig LA, Moreno V: Adquisition of a collectionLycopersicon Pennellii.(Corr. D’arcy)转基因植物uid一个和nII标记基因。j hortic sci biotechnol。1999,74(1):105-109。
- 30.
Eshed Y,Abu-Abied M,Saranga Y,Zamir D:Lycopersicon esculentum含有小重叠旋转的线L. Pennellii..《中国科学院院刊》1992年第3期。
- 31。
Komatsuda T,Annaka T,OKA S:量化性状基因座(QTL)的遗传映射,可提高拍摄分化率大麦芽王志强,王志强。
- 32。
Taguchi-Shiobara F,Lin Sy,Tanno K,Komatsuda T,Yano M,Sasaki T,OKA S:绘制与稻米种子愈伤组织再生能力相关的定量特质基因座,奥雅萨苜蓿王志强。1997,19(2):1 - 7。10.1007 / s001220050632。
- 33。
Flores Berrios E,Gentzbittel L,Mokrani L,Alibert G,Sarrafi A:向日葵原生质体部门和再生途径的早期事件遗传控制。Al Appl Genet。2000,101:606-612。10.1007 / S001220051522。
- 34。
Bolibok H,Rakoczy-Trojanowska M:QTL在植物中组织培养反应的遗传映射。Euphytica。2006,149:73-83。10.1007 / s10681-005-9055-6。
- 35
Molina RV, Nuez F:我们可以在我们的解释中找到相关的内容Cucumis梅洛.园艺学报,1989,3:111-118。
- 36
不同外植体来源体外再生能力的相关反应Cucumis梅洛.植物细胞代表1995,15:129-132。10.1007 / BF01690269。
- 37
Molina RV,Nuez F:通过毛皮发生的体外再生能力的性传播Cucumis梅洛L.在高生长素/细胞分裂素比率的培养基中。自然科学进展,1997,19(3):427 - 434。10.1016 / s0304 - 4238(97) 00024 - 1。
- 38
Shirasawa K,Asamizu E,Fukuoka H,Ohyama A,Sato S,·Nakamura Y,·Tabata S,Sasamoto S,Wada T,Kishida Y:番茄中SSR和内肠道多态性标志物的三角形联系地图。Al Appl Genet。2010,121:731-739。10.1007 / S00122-010-1344-3。
- 39
张丽,王胜,李华,邓强,郑安,李松,李鹏,李忠,王军:缺失标记和偏析畸变对玉米QTL定位的影响2人口。Al Appl Genet。2010,121:1071-1082。10.1007 / s00122-010-1372-z。
- 40。
Chaerani R, Smulders MJ, van der Linden CG, Vosman B, Stam P, Voorrips RE:早疫病抗性QTL鉴定(alertaria solani.) 在一个Solanum lycopersicum.XS. Arcanum.叉。Al Appl Genet。2006,114:439-450。
- 41.
陈志强,陈志强,陈志强,等:番茄种间杂交越界分离的QTL分析。遗传学杂志。1993,34:585-596。
- 42.
Ben Amer IM,Korzun V,Worland AJ,Börner A:小麦2B染色体上控制组织培养反应的QTL的遗传作图(小麦与主基因和RFLP标记的关系。应用电子学报,1997,94:1047-1052。10.1007 / s001220050513。
- 43.
Schiantarelli E,De La Pena A,Candela M:使用重组自交系(RILS)来识别,定位和映射涉及的主要基因和定量特质基因座体外再生能力拟南芥蒂利亚纳.Al Appl Genet。2001,102:335-341。10.1007 / S001220051650。
- 44.
Prakash AP,Kumar PP:PkMADS1是一个新的MADS box基因调控不定芽诱导和营养芽发育Paulownia Kawakamii..植物J.2002,29:141-151。10.1046 / J.0960-7412.2001.01206.x。
- 45。
Bielenberg DG,Wang Y,Li Z,Zhebentyayeva T,Fan S,Reighard GL,等:桃子中常绿轨迹的测序和注释[Prunus Persica(L.)Batsch]揭示了六个疯箱转录因子的集群终端芽形成调节的候选基因。树遗传学基因组织。2008,4(3):495-507。10.1007 / s11295-007-0126-9。
- 46。
Murashige T,Skoog F:一种修订的培养基,用于快速生长和烟草组织培养的生物测定。physiol植物。1962,15:473-497。10.1111 / J.1399-3054.1962.TB08052.x。
- 47。
Doyle JJ,Doyle JL:从新鲜组织中分离植物DNA。重点。1990,12:13-15。
- 48。
VOS P,Bleeker M,Reijans M,Van de Lee T,Hornes M,Frijters A,Pel J,Peleman J,Kuiper M,等:AFLP:AFLP:DNA指纹纹理的新技术。核酸RES。1995,23(21):4407-4414。10.1093 / nar / 23.21.4407。
- 49。
Frary A,Xu Y,Liu J,Mitchell S,Tedeschi E,Tanksley S:一组基于PCR的锚标记,包括番茄基因组,评估其遗传和育种实验的实用性。Al Appl Genet。2005,111:291-312。10.1007 / S00122-005-2023-7。
- 50.
穆勒·拉,索洛·TH,泰勒·N, Skwarecki B, Buels R, Binns J,等:SOL基因组网络:茄科生物学及其他领域的比较资源。植物营养与肥料学报。2005,32(4):427 - 434。10.1104 / pp.105.060707。
- 51.
Rozen S, Skaletsky HJ:为一般用户和生物学程序员的WWW入门3。生物信息学方法和协议:分子生物学方法。编辑:Krawetz S, Misener S. Totowa, NJ: HumanaPress;2000:365 - 386。
- 52.
Schuelke M:PCR片段的荧光标记的经济方法。自然生物技术。2000,18:233-234。10.1038 / 72708。
- 53。
Fulton TM,Van der Hoeven R,Eannetta NT,坦克斯利SD:高等植物中对比较基因组学的保守矫形器标记的识别,分析和利用。植物细胞。2002,14:1457-1467。10.1105 / TPC.010479。
- 54。
Wu F, Mueller LA, Crouzillat D, Petiard V, Tanksley SD:结合生物信息学和系统遗传学鉴定大套单拷贝同源基因(COSII)用于比较、进化和系统学研究:欧洲小行星植物分支的一个测试案例。中国生物医学工程学报。2006,34(3):427 - 434。10.1534 / genetics.106.062455。
- 55。
Van Ooijen JW:JoinMap 4,荷兰瓦格宁根实验种群遗传连锁图谱计算软件:Kyazma BV;2006
- 56。
Voorrips RE: MapChart:图形化表达连锁图和qtl的软件。王志强,王志强,王志强,等。10.1093 / jhered / 93.1.77。
- 57。
Van Ooijen JW: MapQTL®6,用于绘制二倍体物种实验居群中数量性状的软件2009.
- 58。
田中,石黑,佐藤:利用分子标记对番茄果实蔗糖积累性状的遗传分析。中国科学:地球科学,2019,37(5):531 - 534。10.1270 / jsbbs1951.45.429。
致谢
CG和CT感谢西班牙语'Ministerio deChearaciónyCencia',分别为Ramóny cajal合同和偷窥者团契。作者承认由O.Julián设计的J.Cañizares,SSRS博士的建议和N. Palacios的技术援助,承认Instituto deCollectaciónyMejorade la Agrodiversidad Valenciana(Comav)的财务支持。另外,A. Monforte和J.卑尔根的修订和英语修订也得到了承认。
作者信息
从属关系
通讯作者
附加信息
作者的贡献
CG得到了映射总体。CT进行了群体表型和基因分型,并参与起草。CT和SV进行图谱构建和QTL作图。CG在表型和基因分型方面合作,构思、监督和起草了手稿。FN构思了这项研究,并对手稿进行了批判性的审查。所有作者阅读并批准了最终的手稿。
Carlos Trujillo-Moya,Carmina Gisbert在这项工作中同样贡献。
电子辅料
叶片外植体的再生反应
附加文件1:。父母叶片外植体的再生反应[番茄(CV.ANL27);S. Pennellii.(PE-47)],f1F2和BC.1培养30 d后转入基础培养基(BM) 20 d。(PDF 276 KB)
12870_2011_925_moesm2_esm.pdf.
附加文件2:电子设计的SSR标记。电子设计的SSR标记的名称、带大小、重复基序、退火温度和引物序列表。(PDF 256 KB)
F的遗传定位和LOD分数剖面
附加文件3:2区间定位法检测7号染色体再生组分的- qtl (SpRg-7).F的遗传定位和LOD分数剖面2-QTLS用于再生组分(芽百分比(B),再生百分比(R)和生产率(PR))。在左侧,QTL的投影作为黑条表示SpRg-7对于B,R和PR特征。垂直虚线表示用于声明QTL(B LOD阈值= 3.7)(R LOD阈值= 3.6)(PR LOD阈值= 4.4)的95%显着的阈值。地图位置(cm)和距离基于本研究开发的遗传联系地图。附表中的QTLS特征。(PDF 294 KB)
BC的遗传位置和LOD评分概况
额外的文件4:1- 在染色体1上检测到芽百分比(b)的qtls(SpRg-1),3(SpRg-3)及8 (SlRg-8).间隔映射(IM)的结果和限制多个QTL映射(RMQM)方法。在左侧,投影为黑条(IM)和灰杆(RMQM)表示范围SPRG-1,SPRG-3和SlRg-8垂直虚线表示声明一个QTL的95%有效阈值(B LOD阈值= 2.7)。水平虚线表示酸性转化酶基因(invpen)包含在染色体3 QTL系列中的标记。地图位置(cm)和距离基于本研究开发的遗传联系地图。(PDF 96 KB)
BC的遗传位置和LOD评分概况
附加文件5:1-QTL用于再生百分比(R),在本研究中检测到染色体1(SpRg-1), 4 (SpRg-4a),7(SpRg-7)及8 (SlRg-8).间隔映射(IM)的结果和限制多个QTL映射(RMQM)方法。在左侧,投影为黑条(IM)和灰杆(RMQM)表示范围SPRG-1,SPRG-4A,SPRG-7和SlRg-8R的QTLS为R.垂直虚线表示用于声明QTL(R LOD阈值= 2.7)的95%显着的阈值。地图位置(cm)和距离基于本研究开发的遗传联系地图。(PDF 102 KB)
BC的遗传位置和LOD评分概况
额外的文件6:1-QTLS在本研究中检测到染色体1的生产率(PR)(SpRg-1),3(SpRg-3), 4 (SpRg-4b)和7(SpRg-7).间隔映射(IM)的结果和限制多个QTL映射(RMQM)方法。在左侧,投影为黑条(IM)和灰杆(RMQM)表示范围SPRG-1,SPRG-3,SPRG-4B和SpRg-7垂直虚线表示声明QTL的95%有效阈值(PR LOD阈值= 2.8)。水平虚线表示酸性转化酶基因(invpen)包含在染色体3 QTL系列中的标记。地图位置(cm)和距离基于本研究开发的遗传联系地图。(PDF 106 KB)
)
附加文件7:多态性酸性转化酶基因标记invpen).扩增条带采用QIAxcel多毛细管电泳系统分离。巷1:美国lycopersicumL.(ANL27),带尺寸(〜162bp)。泳道2:S. Pennellii.PE-47,波段大小(~173bp)。巷3:F1杂交种美国lycopersicumL.(ANL27)×S. Pennellii.PE-47,两个带(〜162bp-〜173bp)。泳道4:负面控制。(PDF 122 KB)
用于F
额外的文件8:2和BC.1人口.SSR,COS,COSII,用于基因分型的帽标记2和BC.1人口。(PDF 171 KB)
作者提交的原始图像文件
权利和权限
开放存取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Trujillo-Moya,C.,Gisbert,C.,Vilanova,S。等等。QTL的本地化体外番茄植株再生。植物生物学11,140(2011)。https://doi.org/10.1186/1471-2229-11-140
已收到:
公认:
发表:
关键词
- 扩增片段长度多态性
- 映射人口
- 组氨酸激酶
- 切割扩增多态序列
- 切割扩增多态序列标记