摘要
背景
现在,深度级的第二代测序(2GS)技术现在正在应用于非模型物种作为桑格测序的可行性和有利的替代品。大规模的SNP Discovery在BlackCurrant中进行(肋条nigrum.L.)使用基于转录组的2GS 454在参考映射群的父母基因型上测序,以产生用于构建高密度连杆地图的大量新型标记。
结果
共产生700,000个reads,共发现7,000个SNPs。选择一个多态snp子集,利用Illumina BeadXpress平台开发384-SNP OPA分析。此外,该数据还能识别3000个新的est - ssr。所选的SNPs和SSRs在不同的区域进行了验证酷栗种质,包括定位居群和其他选定酷栗物种。
基于snp的图谱从两个黑醋栗作图群体中开发出来,分别包含48%和27%的snp。通过BeadStudio分析输出的theta评分的定量性状图谱,我们进一步研究了相对较高比例的视觉单形SNPs,这使得额外的SNPs能够被放置在两个图谱上。
结论
使用2GS技术对于标记的开发优于先前描述的方法,在这些标记的标记数和生物信息中的方法中优于先前描述的方法。虽然读取和组装的折叠的数量与其他非模型物种的类似大小研究相当,但是在这里发现了高比例的新基因,横跨了广泛的推定功能和定位。讨论了在下游育种应用中使用2GS方法开发的标记的潜在效用。
背景
在许多物种中,了解和描述重要性状的主要限制是缺乏足够的遗传标记来发展高密度的遗传图谱和关联研究。需要大量的标记,如简单序列重复(SSRs)和单核苷酸多态性(SNPs),以协助识别遗传变异背后的基因。对于许多作物和园艺品种,现在已经开发了遗传连锁图,数量性状位点(QTL)已经被分配到大的染色体区域,但到目前为止,只有少数的候选基因被确定[1].对更多遗传标记的需求被认可,直到最近一直是一个重大挑战和费用。随着新的测序技术的引入,传统的标记开发方法已经取代了[2].这些技术通常被称为“第二代测序”(2GS),平台包括Illumina基因组分析仪,Roche 454 FLX和应用的生物系统固体系统,所有这些都广泛用于霰弹枪基因组测序和SNP发现[3.-9.].
尽管缺乏用于绘制短读图的参考基因组序列,但深层2GS技术目前正被应用于非模式物种,作为桑格测序的可行和有利的替代方案。表达序列标签(ESTs)来源于基于rna的转录组,已成为非常有用的资源,以协助标记的开发[10而且,通过使用2GS技术,转录本可以进行更深入的测序,使得发现新的基因序列的成本和时间比以前要低得多。这种方法在基因组信息很少的物种中特别有用,允许从广泛的转录本中识别大量的SNPs [11].最近,一些基于高通量转录组测序的此类研究已经在非模式植物物种中进行,包括玉米、葡萄、桉树、橄榄和普通豆[3.那6.那4.那7.那12].
黑加仑(肋条nigrum.L.)在Saxifragaceae中分类分离,目前的基因组学资源极为有限。与许多经济上重要的木质多年生植物一样,繁殖酷栗是一个长期的过程,由于可用的种质高度杂合子和较长的代时间,因此有明显的动机发展标记辅助育种策略,以减少时间范围,选择优越的基因型。此前,我们从发育的果实和芽中构建了cDNA文库,并对数千个ESTs进行了测序[13那14].从这些库中,已缩放四十三个SSR和十六个SNP标记,与AFLP一起映射,与鉴定的关键鉴别和果实质量特征相关的许多标记。尽管这些时间相对较大,但我们仍然能够生成538厘米的稀疏填充框架地图,QTL跨越5到10厘米。2GS技术现在提供有机会产生大量的新型标记,从中构建高密度遗传联系地图。
我们目前的研究的目的是使用2GS 454焦点测序从黑醋栗的基因编码区域进行大规模的SNP发现。一旦确定了SNP,需要一种有效的基因分型手段。以前的研究仅验证了一小部分鉴定的SNP,通常通过Sanger重新测序[4.那15].最近开发了SNP检测的高密度测定,来自Illumina的一个这样的平台使得能够与单个DNA样品同时测定384个标记。因此,在Illumina Beadxpress平台上使用来自黑醋栗的多态SNPS来自黑醋栗的多态SNPS。此外,2GS转录组测序促进了新的EST-SSR的鉴定,其被证明是鲁棒标记类型的[10那16那17].为了促进这些SNP和SSR的验证,测定两个分离映射群体和各种种质,480种样品。
结果
本研究的总体目标是确定2GS技术是否能使重大基因发现肋条nigrum.以及这些简短的阅读是否能被组装起来德诺维用于有效分离和新的遗传标记物的发展。在这项研究中,超过70万的序列读数从cDNA产生从显影亲本基因型的黑醋栗芽衍生得到足够的覆盖面,以检测C..7000个snp,其中一个子集通过Illumina BeadXpress基因分型平台验证。
转录组测序,Contig组装和基因注释
共有712814高质量序列读数从从每个的显影芽提取的合并RNA衍生酷栗筛选亲本S10 (226,248 reads)和S36 (485,566 reads)的适配器序列污染,分别留下225,334 reads (S10)和482,959 reads (S36),然后去除核糖体匹配,分别留下212,104 reads (S10)和314,189 reads (S36)。我们发现,与S10(6%)相比,S36中rrna来源的污染水平显著更高(35%),这被认为是由于加工相关因素,因此进一步运行S36是必要的,以提高从该母体的过滤读取水平。最终组的平均读长分别为214 nt (S10)和230 nt (S36)。这些随后被组装起来德诺维,产生33518个连续的序列(重叠群)和12893个单身,具有407个核苷酸(40个核苷酸到8440个核苷酸范围内)的平均重叠群的长度。这些重叠群和单序列,通过针对从NCBI非冗余蛋白质序列和肽模型运行BLASTX搜索与自己最亲近的同系物的描述注释拟南芥蒂利亚纳从TAIR18那19],分别匹配21,527和17,280个肽段。获得显著BLAST命中(即e值小于10)的装配产品的百分比-10)分别为52%和64%,反映了新基因鉴定的高水平酷栗在这项研究中。爆炸袭击是由搜索造成的拟南芥使用TAIR提供的GO注释提取每次击中的基因本体(GO)术语(附加文件1:图S1)。除了“其他生理过程”这一类别之外,所有GO生物过程的主要类别中都有转录本。除了对组装的contigs进行注释外,我们还将其与本霍普(Ben Hope)品种现有的Sanger序列est(共3327个)进行了比较[20.,在针对Sanger est的BLAST搜索中使用454 contigs作为查询序列。在454次试验的输出中,共有2688个现有的Sanger EST contigs被代表,剩下639个(19%)没有代表,反映了样品之间组织来源的差异。
标记开发:单核苷酸多态性和简单序列重复
一套7,245高信心(p> 0.9)酷栗单核苷酸多态性使用GigaBayes软件发现。亲本基因型也定义和用于大多数情况下,无论是一个父(4239出来的7245)或双亲(2684)是杂合的,并且只有一小部分(202)被发现,其中父母都是纯合的。总共才120情况下,在源自同一母公司的重叠群的所有读取,而这些并没有考虑在这项研究中进一步使用。除了单核苷酸多态性,很多EST序列包含重复图案。利用人造卫星软件[21],鉴定了3,179SSRS,其中超过一半是三核苷酸,第三二核苷酸,少数是四核苷酸重复。
384 SNP检测采用Illumina技术支持(techsupport@illumina.com)进行设计。如方法部分所述,Illumina SNP选择是基于邻近多态性、重复元素或回文的缺失,这些已知对检测的成功有不利影响。
定位群体snp位点的初步分析
从384 SNPS获得,使用BeadStudio软件(版本3.1)识别为映射群体SCRI 9328中的映射。其中,在种子父母中,75杂合子仅在种子父母中杂合,在花粉父母中杂合子杂合,并且在父母双方均杂合。检测单个标志物的分离比在人群中显示出许多SNP的意外基因型,这些群体在群体中出现意外的基因型,并且这些基因型被排除在后续分析之外。基于种子父母杂合的标记物的剩余后代的聚类分析仅显示了没有特定的分组,但基于标记物的聚类分析对于花粉父母的标记物杂合出来的46个后代集群,其中没有其中遗传任何特定于花粉父母的等位基因。用于将这46个后代的分离比率与剩余的261个后代进行比较,用于种子母细合酶的剩余261个后代。这发现分离比率显着不同(P <0.001)72个标记物中的72个标记,其离子比例接近于这46个后代的1:2:1,但剩余后代为1:1。这些结果与这些46个后代是自我的,并且这些结果被排除在联动分析之外。
在MP7群体中,使用BeadStudio软件发现384个SNPs中有118个被分离。其中50株为杂合子。本-芬莱(种子亲本)杂合子35个。heda(花粉本)和33个在双亲中均为杂合子。对MP7群体的聚类分析显示,该群体中有3个品系存在许多SNPs的未预期基因型,这些品系被排除在后续分析之外。聚类分析显示,在这个种群中没有任何自交或其他群体的证据。
SCRI 9328的连锁分析
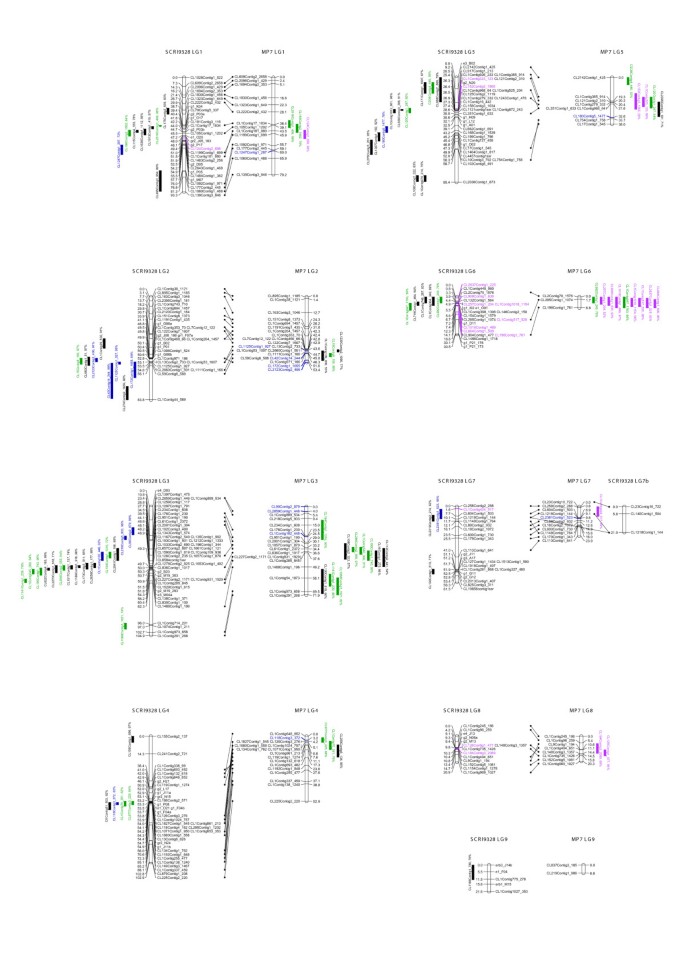
结合SNP和SSR标记进行连锁分析。从这个群体中没有分离出任何标记:所有标记都与至少11个与一个或多个其他标记相连。在lod值为3时,两个连锁基团形成,但其余的标记仅在lod值较高时分离,在7到16之间。这就得到了10个连锁组,其中两个很小,而其余的组有14-46个标记。每个连锁组内的标记被排列在一起,而不是像有时这种类型的杂交所需要的那样将标记从两个亲本分开。根据作者的经验,这种连锁图谱的契合度对于远系物种来说是异乎寻常的好。只有5个标记被忽略了,因为这些标记会导致匹配问题,而JoinMap对结果图谱的平均卡方标准在8个大型连锁群中的每个都低于2.5。数字1显示使用Mapchart 2.1软件制作的连结地图[22].连接基团的编号与[14],利用SSR标记进行鉴定:SSR标记的排列顺序与较小的群体吻合较好。地图总长度为605厘米。
MP7的连锁分析
在这一人群中,有6个SNP标记被排除在外,因为它们具有高度扭曲的比率(p < 0.001)。分离到5个标记,lod为4。其余标记使用lod阈值在5到7之间形成9个连锁组。有两个小组,两个和三个标记,和7个大组,8-21个标记。两个标记被排除在外,因为它们会导致匹配问题。其余吻合良好,所有卡方标准均低于2.5。数字1显示了连锁图谱,标记与SCRI 9328上相应的标记相连。这些显示出这两幅地图很一致。地图总长度为355厘米。
复合频率之间的异质性分析
哪里有对在相应的连接基团之间的共同的SNP时,重组频率可为异质使用JoinMap实现的卡方检验3.共有360对SNP的检查测试。在这些中,有用于339对没有显著异质性(P> 0.05),而15双0.05和0.01,即相似的编号,以对结果因偶然预期之间有显着性。六对多表现为显著的异质性,对LG72双都涉及CL113Contig1_641用P <0.005显著,而在LG5,所有涉及CL754Contig1_7584双,用P <0.001显著。因此重组频率的不均匀性是不这两个十字架之间一个普遍的问题。
该SNP THETA得分得离谱9328人口的QTL分析
对SCRI 9328群体的384个SNP theta评分的检查显示,有15个SNP有超过100个缺失值。这些snp被排除在进一步的分析之外,剩下369个snp,最多缺失15个值。我们还检查了范围:理想的SNP的范围为1,即BB基因型为1,AA基因型为0。范围小于0.05的SNPs被排除在QTL分析之外,总共有310个SNPs进行了theta评分的定位。其中184个snp被定位为清晰的双等位标记,5个snp被分离为双等位标记但被排除在连锁图谱之外,121个snp被BeadStudio认为是未分离的。
利用标记的theta分数进行QTL定位时,可以将184个snp定位到同一个位点。在最显著标记上的theta值的回归解释了theta值的71-99%的方差,较低的四分之一为97%。当以theta评分作为QTL分析时,由于与连锁组不匹配而从连锁分析中删除的5个SNP标记都被映射到同一组,在最近的标记上进行回归,可以解释theta评分方差的90-99%。其中两个标记在双亲中均为杂合子,并被定位到LG2上一个存在分离畸变的区域。其他3个在亲本中为杂合子,但当作为QTL定位时,显示与来自另一个亲本的等位基因相关。
当被映射为QTL时,121个剩余的SNP显示标记关联,其最大百分比方差从0.7%(即无明显关联)到99%。31个SNP的最大百分比差异至少为70%,与也映射为标记的SNP相当。通过排列测试建立了QTL存在的意义阈值[23,对三个不同范围的性状分别进行100个排列,表明这些排列的性状的最大方差解释百分比为6.3%。有36个snp最大百分比方差低于6.3%,属于无显著QTL。然而,我们感兴趣的是存在大量的,而不仅仅是统计上显著的,遗传方差的SNPs,因此我们选择关注由标记回归解释的最大百分比方差大于50%的SNPs。121个snp中有52个在这个范围内。这些SNPs的单lod置信区间,以及5个在连锁分析中不匹配的SNPs,如图所示1.
MP7群体SNP theta评分的QTL分析
在该群体中,有251个SNPs的theta评分范围大于或等于0.05,最多缺失10个值。其中118个被标记,105个被放置在连接地图上。在剩余的133个SNPs中,有36个被定位为QTL,解释方差超过50%,如图所示1.有很好的协议两种人群的SNP标记的位置,是否映射标记或QTL: 15个SNP映射QTL类似职位在同一染色体在两个种群,24个SNP映射作为QTL在一个人口和标记类似的立场相同的染色体。有些只在一个群体中绘制。只发现了一个明显的差异,CL2395Contig1_181。这在SCRI 9328中被定位为连接组LG2的标记。作为一个QTL,它在相同的位点上解释了82%的性状变异,但在LG3和LG5上表现出较小的显著峰(p < 0.001)。CL2395Contig1_181在MP7中未作为标记定位,但作为一个QTL定位到LG5,解释了71%的性状变异。
通过多样性分析SNP的验证
384个单核苷酸多态性检测范围为66个肋条nigrum.栽培品种及5个近缘种。多态性snp的数量与原定位群体相似(207个snp)CF..190个SNP)。对于每个SNP,利用Nei氏无偏期望杂合度,从0.030到不等的0.500最大值,具有0.307的总平均值测量(分集值表1).观察到的和预期的杂合子值相似,平均近距离繁殖系数-0.069(表1).只有22个基因座在小于0.050和47的最小等位基因频率(MAF),MAF小于0.100。在5种相关物种中,几乎一半被评分的人被证明是单组态。
通过制图和多样性分析验证SSRs
利用SCRI 9328作图亲本和黑醋栗种质及近缘种醋栗(R. grossularia和红醋栗(r .石l)。在设计的40个SSR引物中,在测试的所有基因型中和随后用ABI 3730荧光标记和可视化的10SSR的所有基因型中扩增,在分离群体中映射(如图所示)1)和8种种质收集中的多态性。等位基因数量为3至8,平均值为2.9,平均值的预期预期杂合子为0.397(表2).与SNP分析一样,SSRs的观测杂合度和预期杂合度值相似,相近的近交系系数为0.128(表1)2).与野生和栽培品种相比,野生品种的多样性更大酷栗,尽管这与高水平的近亲繁殖有关(意味着F是野生5只,0.432酷栗)的所有位点,这表明空等位基因的野生种质资源的存在。
讨论
所有植物育种计划的核心是鉴定控制经济上重要的特征的基因。传统上,这是通过使用有限数量的分子标记进行遗传图来实现的。随着近期测序技术的进步,现在可以在前所未有的规模上产生标记[10].我们报告使用2GS 454技术从开发黑醋栗芽的cDNA产生超过700,000种读数,允许足够的覆盖范围以确定超过7,000个SNP和3,000个SSR。下面我们讨论组装的Contigs和单身的属性以及SNP和SSR标记的效用,以提供改进的遗传图,以帮助识别对黑醋栗中重要性状的基因识别原因。
在阅读数、组装的contigs和单次序列方面,我们的结果与其他454个非模式物种转录组研究的结果相似[3.那4.那7.那8.那15那24].33,518个Contigs和12,893名单身,52%和64%的公共领域的肽序列的显着爆炸率高于其他树种(包括)桉树祖母(38%)[4.]和Pinus contorta.(32%) (8.].然而,这些相对较低的显着同源性和在我们的Sanger Est系列中未发现的ests的存在[20.反映了在本研究中发现的黑醋栗新基因的比例很高。从多肽同源性和GO注释分析(附加文件1:图S1),很明显,从大范围的基因,相对于推定的功能和定位转录物,已被采样,从而形成新的基因特异的标志物的基础。
第二代测序已用于鉴定一系列植物物种中的SNP [10].在这项研究中,我们确定了7000个SNP从德诺维组装黑醋栗EST数据。除了这种方法进行SNP发现的发展,我们讨论验证,以及是否的问题德诺维仅基于2GS数据的SNP发现可以转化为SNP检测测定,更重要的是有用的标记。我们设计了一种基于Illumina Beadxpress平台的多路复用高通量SNP检测测定,并使用两个隔离群体和多种种质血管检查384个SNP的多态性。虽然所有SNP被选为来自读取对准的多态,但我们无法通过链接映射方法从当前组件确认几乎一半的推定的SNP,因为它们在映射群体中没有清楚地分离。有些SNP没有表现出来的技术原因以及其他人:关闭et al。[25[绅士得分低(小于0.300),描述了一些无法可行的SNP,即使他们选自Sanger测序EST集合。虽然我们的几个SNP落入了这一课程(13%),但其中大多数未经证实的SNP出现在一个高级绅士得分的单一集群中,随后被评为单数。这些单声道SNP可以是伪装成SNP或MIS组装的读数的测序误差,导致来自组装成单个折叠的基因组的不同区域的基因家族成员序列。预期额外的测序将增加转录组空间覆盖率,最终会改善组装的特殊性。最近,我们使用相同RNA的配对Illumina 2gs(未提出的数据)增强了我们的BlackCurrant EST,并发现新组件中不支持导致单声道SNP(〜15%)的454个Contigs中的几个这些Contig的预测的SNP(〜70%)也消失了。这也强调了近2GS的最近的快速技术进步,就可以实现的覆盖率和测序保真程度。实际上,来自多个2GS平台的混合组件通常可以实现最可靠的Contig数据集。RNA-SEQ的替代策略包括基因组减少方法,旨在降低具有大型基因组的物种的GDNA复杂性,如玉米,籽粒苋菜,常见豆豆和大豆[3.那9.那12那26-28].这些方法可从错误组装遭受以下,通过包括独特的非编码序列,不过这样的非核不标记通常不能直接相关的功能。以及在降低初始复杂性,改进德诺维最近开发了组装和SNP识别管道[29那30.].
利用现有的分析软件(Illumina BeadStudio v3.1),我们分别从两个黑醋栗作图群体SCRI 9328和MP7中绘制了184个SNPs(占检测snp的48%)和105个SNPs(占检测snp的27%)。尽管这些水平似乎相对较低,考虑到9328的双亲都被用于SNP发现管道,其他以相同方式(发现、检测和随后的定位)对亲本进行定位的研究发现,玉米遗传图谱上的SNPs数量相似(63%)[27]和两个马铃薯作图群体(43%和48%)[30.].标记与重组频率非常小的异质性之间的标记吻合吻合良好。虽然这些SNP大大改善了我们以前的地图,但我们通过将珠杆菌分析作为定量特征映射到珠粒假性分析来进一步研究单数标记。由于这些分数预计从单一遗传轨迹,以及一些测量误差,我们使用了由单个位置解释的50%的阈值的非常高的阈值。在该阈值下,我们能够在SCRI 9328 MAP和36上放置52个在MP7地图上的36。一般来说,两种群体中的职位之间存在良好的一致性,是否将SNP映射为群体中的QTL,或者在一个人口中的QTL和另一个人口中的标记。通过降低阈值,可以将其他SNP映射为QTL。我们计划进一步调查SNP Theta分数如何最好地用于这种分析。
384 SNP测定也被用于基因分型的一组多样黑醋栗加入,包括育种品系的,以及相关的栽培和野生酷栗物种。超过一半的SNPs具有多态性,平均MAF为0.253,与使用还原文库中的SNPs在鸡(0.280)和猪(0.274)中观察到的结果相似[31那11].妈妈et al。[27]使用MAF作为测量SNP标记的多态性的方法,并且在它们的玉米研究中使用604映射的SNP,80%的MAF> 0.100。在我们对209种多态性SNP的研究中,超过75%的MAF> 0.100。在比较其他研究时,SNP标记也表现良好(平均值E.0.292酷栗相比,HE.鸡肉0.350 [31]),并且正如预期的黑醋栗,没有近亲繁殖的证据,观察到的和预期的杂合度值非常相似。
除SNPS,几项研究已经使用了MINE的类似方法,用于SSR,包括一系列应用,包括映射,系统性,人口和保护遗传学[8.那16那17那32-35].在这些研究中,从几乎所有(97%)微卫星序列(FIASCO富集程序)中鉴定出的SSRs的数量各不相同[17]到几百(单线转录组测序)[33],大多数研究落在两者之间。在这项研究中,我们使用4542GS鉴定了3,000多种新的BlackCurrant EST-SSR,其将为大多数应用提供足够的基于基因的标记。我们研究的分集价值(HE.0.152至0.825)与他人相当(例如,在杜松,0.200至0.900)[34]但是,虽然预期,但是使用基因组SSRS的先前研究略低,值范围为0.184至0.908 [36].然而,开发基因组SSRS所需的努力和时间远远越来越昂贵。此外,我们观察到来自SNP和SSR数据的遗传距离矩阵之间的相同黑醋栗单独的数据(20个常见载体; r2= 0.777,数据未显示),证实了这些标记在一系列应用中的稳健性。
方法
植物材料
Leaf buds were sampled from four-year old blackcurrant plants grown in the field at Invergowrie, Dundee (latitude 56.45, longitude -3.06) of both parents of the reference mapping population SCRI 9328 in February 2008, immediately prior to dormancy break, i.e. as the buds began to visibly swell. Buds were flash frozen in liquid nitrogen and stored at -80°C.
SCRI 9328人口由311 F组成1伪杂交的全同胞后代[38在詹姆斯·赫顿研究所的两个不同的育种系之间的防虫温室中手工制作的[14].此外,第二个F1有95个后代的全同胞mapping群体,指定为MP7,来自黑醋栗cvs之间的杂交。Ben Finlay和Hedda,用于标记物的下游验证。
一系列酷栗种质,包括33条育种线,15种市售品种(Bens)和5种相关野生物种(表1那2)用于测定SNP和SSR标记的多样性。
总RNA提取
使用Plant RNeasy Mini Extraction Kit (RLC buffer, Qiagen)从100 mg冷冻池发育芽材料中提取总RNA,并添加RNA分离辅助剂(Ambion)。用分光光度法检测RNA质量,使用生物分析仪(安捷伦技术)评估其完整性。
基因组DNA的隔离
从两个映射种群的田间种植植物收获幼叶材料(SCRI 9328和MP7)和71酷栗种质过程。使用毫冈(Milligan)描述的方法提取总基因组DNA [39或DNeasy微型提取试剂盒(Qiagen)。采用PicoGreen分光光度法(Invitrogen公司)测定DNA质量和数量。
454测序和质量控制
发育芽的总RNA酷栗父母S10和S36是为标准转录454 FLX(Roche)的RNA-seq的测序分别提交基因库服务设施(爱丁堡大学,英国)。使用任一SMART(Clontech)中或MINT(Evrogen的)的试剂盒如制造商推荐的cDNA被生成。运行样品之前进行破碎和文库制备的建议(Roche)的。所有序列读取已提交给EMBL欧洲核苷酸存档(ENA:http://www.ebi.ac.uk/ena/).所述读取用于每个父筛选从cDNA制备和454个的实验程序都接头序列始发的存在。适配器污染用CROSS_MATCH(掩蔽http://www.phrap.org/phredphrapconsed.html),然后使用自定义perl脚本从读取中进行裁剪。阅读的匹配质量分数也被删除了。任何在中间有适配器污染的读作为可能的嵌合序列被丢弃。在适配器修剪之后,序列被筛选的存在污染核糖体RNA。利用Genbank的关键字检索,构建了一个包含多种植物核糖体RNA序列的小型BLAST数据库。然后在这个数据库中搜索读取的序列,任何与e值大于1e-10的核糖体RNA序列匹配的序列都被丢弃。
序列组装
经过适配器和核糖体序列修剪后,在每个序列的标识符前加上亲本名称(S10或S36),然后使用tgicl套件(http://compbio.dfci.harvard.edu/tgi/software)运行在一台带有四个处理器的CentOS Linux机器上。所使用的程序集参数与HarvEST程序集中使用的“放松”参数相同(http://harvest.ucr.edu.),即CAP3参数-p 75 -d 200 -f 250 -h 90。这些被充分放松,使SNP不会被分离到不同的contigs,从而允许SNP的发现。在组装期间,有19个读取导致从CAP3产生滑移错误消息,因此被删除。
美国东部时间注释
用BLAST (e值截断为1e-10)对其最接近的同系物进行注释,以检索NCBI的非冗余蛋白序列和肽模型拟南芥蒂利亚纳[19].爆炸袭击是由搜索造成的A. Thaliana.通过使用TAIR提供的注释文件(ftp://ftp.arabidopesis.org/home/tair/ontologies/gene_ontological/ath_go_goslim.txt.xt.).然后记录每个GO ID出现的次数,并根据要可视化的最高阶GO类别解析GO ID (ftp://ftp.arabidopsis.org/home/tair/Ontologies/Gene_Ontology/TAIR_GO_slim_categories.txt).
SNP确定
使用来自波士顿学院Gabor Marth实验室的GigaBayes工具,在最终组装中发现了单核苷酸多态性(SNPs)。http://bioinformatics.bc.edu/marthlab/GigaBayes).Gigabayes在装配文件(ACE文件格式)中检测SNP和Indels,并且根据参数设置,也可以输出父母基因型。SNP本身和父母基因型都与贝叶斯概率值相关联,这表明该特征的置信度。参数设置“--crl 6 --cal1 3 - ad2 3 --psl 0.9 --qrl 0 -qal 0 -ioproy二倍体 - sample multiply”用于查找次要和主要等位基因的位置每个组装序列至少存在三次。最小读取基本质量值(--QRL)和最小聚合等位基因质量值(--QAL)标志必须设置为零阈值,因为装配软件使用在存在的位置分配低基本质量分数到共识序列高度的可变性,例如SNPS [40].使用“Tablet”软件包对千兆字节输出和连续序列进行可视化和选择[41],并提交Illumina技术支持(techsupport@illumina.com),用于Illumina GoldenGate SNP检测的设计。Illumina SNP选择是基于邻近多态性(SNPs之间每侧60 bp的侧边序列)、重复元素或回文的缺失,因为已知这些因素会影响SNPs转化为有效分析的比率[42那43].
SSR的鉴定与分析
固态继电器从使用人造卫星程序[组件标识21使用底漆3设计寡核苷酸引物[44].测试引物对的能力,其根据[中描述的协议,用于扩增SSR基因座的能力。36].SSR Loci使用Abi Prism可视化®3730基因分析仪和等位基因使用的GeneMapper进球®应用生物系统公司,沃林顿,英国。多样性的统计数字是根据[45使用Excel微工具箱[]46].赖特近亲繁殖系数的无偏见估计器是,使用FSTAT v. 2.9.3软件计算[47].
Illumina公司基因分型
根据Veracode手册(Illumina VC-901-1001)的Goldengate基因分型测定中推荐进行整个基因分型程序。除非另有说明,否则所有试剂均由Illumina提供。在Beadxpress Reader系统上使用Verascan软件中的默认设置立即扫描示例VBP。
数据提取和解释
使用Illumina Beadstudio数据分析软件(V 3.1)封装在视觉上进行基因型。每个SNP分别得分,并自动确定的集群或手动确定为三个预期组(AA,AB和BB)。
初步数据分析
布伦南et al。[14通过对SCRI 9328花粉亲本AFLP谱带的聚类分析,在SCRI 9328群体的125个原始后代中检测到43个自交后代。利用SNP标记对311个株系的扩展群体进行了重复分析。采用简单匹配系数作为相似性测度,并采用群平均聚类分析构建树状图。为了比较,我们还基于种子亲本分离的SNP标记进行了聚类分析。对MP7子代进行了同样的分析。所有的聚类分析使用Windows 12的Genstat进行[48].
基因映射
联动偏析SNP的映射和SSRS估计两者的参考作图群体SCRI 9328,并且还用于第二MP7人口单独使用JoinMap 3软件[49和Kosambi映射函数。采用JoinMap 3中的卡方检验检验两个种群重组频率之间的异质性。
SNP theta评分的QTL分析
Illumina的数据包括每个SNP的两个强度值(X, Y),测量与SNP的两个等位基因相关的荧光染料的强度。归一化后,强度转化为联合SNP强度R = (X+Y),强度比theta = (2/π)*arctan(Y/X) [50.].根据SNP Theta得分,个体被分类为每个SNP的基因型AA,AB或BB。
384个SNPs均有望在SCRI 9328群体中分离,但据报道,约有一半的SNPs通过BeadStudio软件未被鉴定为分离。另一种方法是把theta分数作为数量性状来分析,把它们看作是由遗传信息和测量误差组成的。利用MapQTL 5.0软件进行QTL区间作图分析[51.].Genstat 12还被用于对标记数据的theta分数进行回归,并估计解释的方差的百分比。
参考资料
- 1.
Mackay I, Horwell A, Garner J, White J, McKee J, Philpott H:重新分析英国品种试验的历史系列,以量化遗传和环境因素对随时间变化的产量趋势和变异性的贡献。中国科学(d辑),2011,32(11):362 - 368。10.1007 / s00122 - 010 - 1438 - y。
- 2.
Wall PK, Leebens-Mack J, Chanderbali AS, Barakat A, Wolcott E, Liang H, Landherr L, Tomsho LP, Hu Y, Carlson JE, Ma H, Schuster SC, Soltis DE, Soltis PS, Altman N, DE Pamphilis CW:下一代测序技术对转录组特性的比较。中国生物医学工程学报。2009,10:347-366。10.1186 / 1471-2164-10-347。
- 3.
Barbazuk WB,Emrich SJ,Chen HD,Li L,Schnable PS:通过454转录组测序SNP发现。植物杂志。2007,51:910-918。10.1111 / J.1365-313x.2007.03193.x。
- 4.
Novaes E, Drost DR, Farmerie WG, Pappas GJ, Grattapaglia D, Sederoff RR, Kirst M:高通量基因和SNP发现桉树祖母,一个没有特征的基因组。基因组学杂志。2008,9:312-326。10.1186 / 1471-2164-9-312。
- 5。
新一代测序技术在功能基因组学中的应用。基因组学杂志。2008,32(4):434 - 434。10.1016 / j.ygeno.2008.07.001。
- 6。
Bellin D, Ferrarini A, Chimento A, Kaiser O, Levenkova N, Bouffard P, Delledonne M:将下一代焦糖测序与微阵列结合用于非模式物种的大规模表达分析。中国生物医学工程学报。2009,10:555-564。10.1186 / 1471-2164-10-555。
- 7。
Alagna F, Agostino ND, Torchia L, Servili M, Rao R, Pietrella M, Giuliano G, Chiusano ML, Baldoni L, Perrotta G:两个橄榄基因型果实发育过程中转录本的454焦糖测序比较。中国生物医学工程学报。2009,10:399-414。10.1186 / 1471-2164-10-399。
- 8。
Parchman TL,Geist Ks,Grahnen Ja,Benkman CW,Buerkle Ca:在生态重要的树种中的转录组测序:装配,注释和标记发现。BMC基因组学。2010,11:180-196。10.1186 / 1471-2164-11-180。
- 9。
Hyten DL, Cannon SB, Song Q, Weeks N, Fickus EW, Shoemaker RC, Specht JE, Framer AD, May GD, Cregan PB:高通量SNP发现,通过深度重排还原文库锚定和定位支架在大豆全基因组序列。基因组学杂志。2010,11:38-46。10.1186 / 1471-2164-11-38。
- 10.
Varshney RK,Nayak Sn,May Gd,Jackson SA:下一代测序技术及其对作物遗传和育种的影响。生物技术的趋势。2009,27:522-530。10.1016 / J.TibTech2009.05.006。
- 11.
拉莫斯AM,Crooijmans RPMA,Affara NA,阿马拉尔AJ,阿奇博尔德AL,Beever JE,本迪克森C,Churcher C,克拉克R,Dehais P,汉森MS:在猪使用鉴定和表征通过单核苷酸多态性的高密度SNP基因分型测定法的设计新一代测序技术。Plos一个。2009年,4:e6524-10.1371 / journal.pone.0006524。
- 12.
Hyten DL,Song Q,Fickus EW,Quigley CV,Lim J-S,Choi I-Y,Hwang E-Y,牧师 - Corrales M,Cregan PB:高吞吐量SNP发现和分析在共同豆中的开发。BMC基因组学。2010,11:475-482。10.1186 / 1471-2164-11-475。
- 13.
Woodhead M,Davies HV,Brennan RM,Taylor MA:从黑醋栗中分离基因组DNA(肋条nigrum.l .)。生物工程学报。1998,9:423 - 427。10.1007 / BF02915797。
- 14.
Brennan R, Jorgensen L, Hackett C, Woodhead M, Gordon SL, Russell J:黑醋栗遗传连锁图谱的开发(肋条nigrum.果实主要品质农艺性状相关区域的鉴定。中国科学:地球科学。10.1007 / s10681 - 007 - 9412 - 8。
- 15.
Trick M, Long Y,孟J, Bancroft I:单核苷酸多态性(SNP)在多倍体中的发现芸苔栗鸟使用SOLEXA转录组测序。植物生物技术杂志。2009,7:334-346。10.1111 / J.1467-7652.2008.00396.x。
- 16。
Hahn Da,Ragland GJ,Shoemaker DD,DDL:基因发现使用大规模平行的焦点测序来开发肉体飞行Sarcophaga Crassippis..中国生物医学工程学报。2009,27(4):429 - 434。10.1186 / 1471-2164-10-234。
- 17。
Santana QC, Coetzee MPA, Steenkamp ET, Mlonyeni OX, Hammond GNA, Wingfield MJ, Wingfield BD:通过富集基因组文库的深度测序发现微卫星。生物技术。2009,46:217-223。10.2144 / 000113085。
- 18。
斯瓦布雷克,韦克思,拉梅斯,贝拉尔迪尼·茨,福尔斯特·H,李东,马勒·R, Ploetz L, Radenbaugh:拟南芥信息资源(TAIR):基因结构和功能注释。核酸研究。2008,36(数据库):D1009-D1014。
- 19。
拟南芥信息资源(TAIR)。[http://www.arabidopsis.org]
- 20.
赫德利PE,罗素JR,乔根森L,戈登S,莫里斯JA,克特CA,Cardle L,布伦南R:用芽休眠释放在黑醋栗相关的候选基因(肋条nigrum.l .)。BMC植物生物学。2010,10:202-10.1186 / 1471-2229-10-202。
- 21.
阿布佳C:斯图尼克。1994年,华盛顿大学分子生物技术,[http://espressosoftware.com/sputnik/index.html]
- 22.
vooorrips Re:MapChart:用于链接地图和QTL的图形演示软件。遗传杂志。2002,93(1):77-78。10.1093 / jhered / 93.1.77。
- 23.
邱吉尔(Churchill GA, Doerge RW):数量性状作图的经验阈值。遗传学。1994,138:963-971。
- 24.
张F, Haas BJ, Goldberg SMD, May GD, Xiao Y, Town CD:测序Medicago Truncatula.使用454生命科学技术表达序列标签。生物医学工程学报。2006,7:272-282。10.1186 / 1471-2164-7-272。
- 25.
关键词:大麦,SNP,高通量,基因分型中国生物医学工程学报。2009,10:582-10.1186/1471-2164-10-582。
- 26.
van Orsouw NJ, Hogers RCJ, Janssen A, Snoeijers S, Verstege E, Schneiders H, van der Poel H, van Oeveren J, Verstegen H, van Eijk MJT:复杂基因组大规模多态性发现的一种新方法。公共科学图书馆。2007,2:e1172-10.1371/期刊。
- 27.
Mammadov JA, Chen W, Ren R, Pai R, Marchione W, Yalcin F, Witsenboer H, Greene TW, Thompson SA, Kumpatla SP:从玉米复杂性降低部分开发高多态性SNP标记[Zea Mays.分子标记辅助育种的应用。中国科学(d辑),2010,31(6):591 - 598。10.1007 / s00122 - 010 - 1331 - 8。
- 28。
莫恩PJ,Yourstone SM,Jellen EN,Udall的JA:通过基因组SNP减少发现,条形编码和454焦磷酸测序苋属植物.植物遗传资源学报。2009,2:260-270。10.3835 / plantgenome2009.08.0022。
- 29。
Ratan A, Zhang Y, Hayes VM, Schuster SC, Miller W:无参考序列的SNPs调用。生物信息学。2010,11:130-142。10.1186 / 1471-2105-11-130。
- 30.
Anithakumari AM, Tang J, van Eck HJ, Visser RGF, Leunissen JAM, Vosman B, van der Linden CG:从EST数据库中高通量检测和绘制SNPs的管道。生物学报。2010,26:65-75。10.1007 / s11032 - 009 - 9377 - 5。
- 31。
Kerstens HHD, Crooijmans RPMA, Veenendaal A, Dibbits BW, Chin-A-Woeng TFC, Dunnen JT, den Groenen MAM:使用第二代高通量测序技术在未测序基因组中发现大规模单核苷酸多态性:应用于土耳其。基因组学杂志。2009,10:479-10.1186/1471-2164-10-479。
- 32.
李鹏,王树华,王丽丽,石英,Wallace R,郭旭,刘铮:东方牡蛎est序列的产生与分析,中国海洋大学学报(自然科学版),Crassostrea virginicaGmelin与微卫星和SNP标记的鉴定。基因组学杂志。2007,8:157-168。10.1186 / 1471-2164-8-157。
- 33.
通过基因组测序,快速、经济地开发物种特异性微卫星标记。生物技术学报。2009,46(5):481 - 484。
- 34.
四倍体微卫星标记的鉴定与特征杜比卡岛柑。利用新一代测序。保护遗传学RESOUR。2010年,1:253-255。
- 35.
Allentoft ME, Schuster SC, Holdaway RN, Hale ML, McLay E, Oskam C, Gilbert TP, Spencer P, Willerslev E, Bunce M:利用高通量(454)序列数据鉴定一种灭绝恐鸟物种的微卫星。生物技术。2009,46:195-200。10.2144 / 000113086。
- 36.
Brennan R,Jorgensen L,Woodhed M,Russell J:SSR标记的开发和表征酷栗物种。分子生态学报。2002,2:327-330。10.1046 / j.1471-8286.2002.00233.x。
- 37.
Brennan R, Jorgensen L, Gordon SL, Loades K, Hackett C, Russell J:一种基于pcr的与黑加仑瘿螨抗性相关的标记的开发(Cecidophyopsis ribis壁虱:瘿螨科)。遗传工程学报,2009,31(4):429 - 434。10.1007 / s00122 - 008 - 0889 - x。
- 38.
Grattapaglia d,Sederoff R:遗传连锁的映射桉树祖母和桉树urophylla利用伪测交作图策略和RAPD标记。遗传学。1994,137:1121-1137。
- 39.
Milligan BG:植物DNA分离。群体的分子遗传学:实用方法。编辑:Hoelzed AR.牛津,英国:IRL出版社;1992:59 - 88。
- 40.
Huang XQ,Madan A:CAP3:DNA序列装配方案。基因组研究。1999,9:868-877。10.1101 / GR.9.9.868。
- 41.
Milne I, Bayer M, Cardle L, Shaw P, Stephen G, Wright F, Marshall DF:平板电脑-下一代序列组装可视化。生物信息学。2010,26:401-402。10.1093 /生物信息学/ btp666。
- 42。
粉丝jb,oliphant a,shen r,kermani bg,garcia f,gunderson kl,hansen m,梯队f,butler sl,deloukas p:高度平行的SNP基因分型。冷泉Harb Symp量子Biol。2003,68:69-78。10.1101 / SQB2003.68.69。
- 43。
李志强,李志强,李志强:基于Illumina GoldenGate分析的多倍体小麦单核苷酸多态性基因分型研究。中国科学(d辑):地球科学(英文版)。10.1007 / s00122 - 009 - 1059 - 5。
- 44.
Rozen S,Skaletsky SJ:Primer3关于General Users和Foribiogrist程序员的WWW。在生物信息学方法和协议中:方法分子生物学。编辑:Krawetz S,Misener S. Totowa,NJ:Humanapress;2000:365-386。
- 45.
Nei M:估计少数个体的平均杂合性和遗传距离。遗传学。1978,89:583-590。
- 46.
Park SDE:西非养牛的锥体作用以及选择的人口生效。都柏林大学;2001年,博士。论文。
- 47.
Goudet J:FSTAT,一个估计和测试基因多样性和固定指数的计划(2.9.3版)。2001年,[http://www2.unil.ch/popgen/softwares/fstat.htm.]
- 48.
Genstat:用于Windows Release 12.1的Genstat。VSN International Ltd, HemelHempstead,赫特福德郡;2009.
- 49.
Van Ooijen JW, Voorrips RE: JoinMap®3.0,用于计算遗传连锁图的软件。国际植物研究组织,荷兰瓦赫宁根;2001.
- 50.
斯塔夫Ĵ,瓦隆ChristerssonĴ,林德格伦阿d,Juliusson G,罗森奎特R,豪格伦德男,博格A,RingnérL:(2008)的Illumina的Infinium全基因组SNP数据的规范化提高拷贝数估计和等位基因强度比。BMC生物信息学。2008年,9:409-10.1186 / 1471-2105-9-409。
- 51.
Van Ooijen JW:MapQTL®5,实验群体中Qualitativetirait基因座的映射软件。Kyazma B.V.,Wageningen,荷兰;2004年。
确认
这项工作得到了苏格兰政府和欧洲区域发展基金的支持(项目号35-2-05-09)。感谢Iain Milne和Gordon Stephen的基因型可视化软件的实现。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
JR帮助构思了这项研究,并协调了分子工作和图谱分析。PH帮助构思了这项研究,为实验设计和分子生物学提供了建议,并促进了2GS程序。MB和LC为2GS数据提供生物信息学支持。CH分析了测绘数据。CB和JAM提供了测序和基因分型支持。RB帮助构思了这项研究,并提供了合适的植物材料。SG收集植物样本进行分析。LJ进行分子工作。JR, PH和RB起草了手稿,所有作者阅读并批准。
电子补充材料
图S1 -基于最接近的衍生同源性的黑醋栗est的GO注释类别分布(蓝色条)
额外的文件1:拟南芥预测肽序列.这些都与从整个GO注解的分布拟南芥基因组(红酒吧)。多克斯(95 KB)
作者的原始提交的图像文件
以下是与作者的原始提交的图像的链接。
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativeCommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
罗素,J.R,拜耳,M,布斯,C。et al。来自黑醋栗的新型转录组标记物的鉴定,利用和映射(肋条nigrum.).BMC植物杂志11,147(2011)。https://doi.org/10.1186/1471-2229-11-147
收到了:
接受:
发表:
关键字
- 定量特质基因座
- 基因本体论
- 连锁集团
- 映射人口
- 数量性状位点分析