摘要
背景
真菌病原体Setosphaeria turcica导致玉米、高粱和相关禾草上的turcicum或北方叶枯病。一种在玉米和高粱这两种主栽作物生长的地方普遍存在的叶面疾病。本研究的目的是寻找控制这种毁灭性植物病原体的宿主防御反应的基因。采用cDNA-AFLP方法鉴定候选序列,通过病毒诱导基因沉默(VIGS)和实时荧光定量PCR分析进一步验证候选序列的功能。进行系统发育分析以处理进化事件。
结果
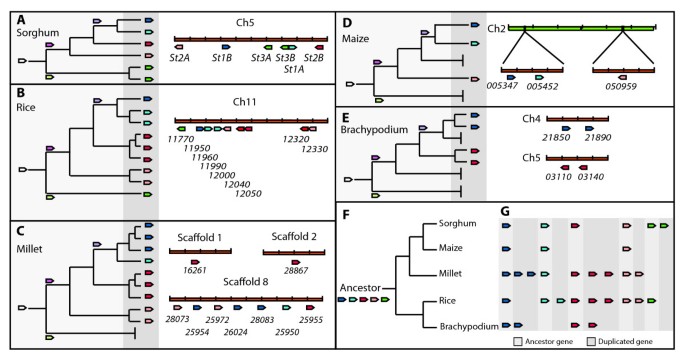
CDNA-AFLP分析在易感和抗高粱和玉米基因型上进行,以鉴定相关抗性序列。一个CC-NB-LRR编码基因GRMZM2G005347是在真菌侵袭后上调的玉米转录本中发现的。新的植物抗性基因命名为圣参考S. Turcica.基因组序列比较显示CC-NB-LRR编码圣基因位于玉米的2号染色体上,高粱的5号染色体上。6圣高粱基因在一个位点上有三对。当高粱圣基因通过VIGS沉默,抗性明显减弱,这是实时PCR的观察结果。数据库检索和系统发育分析表明圣在5千万到7千万年前草亚科分裂之前,基因有一个共同的祖先。如今,高粱中有6个基因,水稻和谷子中分别有9个,玉米中有3个,谷子中有4个Brachypodium distachyon.这圣基因同源物具有所有高度保守的序列,并且通常作为草基因组中的基因对。
结论
抗性基因到S. Turcica,具有CC-NB-LRR蛋白结构域,已在玉米和高粱中发现。VIGS分析表明了其在监测中的重要性S. Turcica在高粱。这圣基因在高粱、水稻、谷子、玉米和短柄草中高度保守,表明一种重要的进化功能。
背景
免疫系统是逐步完善的,它的基本功能帮助祖先的有机体在恶劣的环境中生存下来。以一种特定的方式识别病原体导致在植物中产生一种非常强大的监测系统模式。这种被称为抗性(R)蛋白介导或效应体触发免疫的保护形式,是由植物编码的R蛋白“感知”病原体来源的效应分子(以特异性无毒(Avr)基因产物为代表)的存在而诱导的[1].识别病原体后,在寄主植物中诱导一个或多个信号转导途径,从而防止病原体定植。
大多数特征R蛋白编码核苷酸结合位点(NB)和富亮氨酸重复序列(LRR)。nb - lrr编码基因是植物中发现的最大和最可变的基因家族之一,大多数植物基因组包含几百个家族成员[2-6.].r蛋白的n端主要由TIR (Toll/白介素-1受体)同源结构域或形成一个螺旋-螺旋(CC)基序组成。尤其是单子叶植物,它们的基因组中有大量的CC-NB-LRR蛋白。积累的数据进一步表明,r蛋白的N端可能与一系列病原体来源的蛋白相互作用。然而,LRR域可能决定这种识别的最终结果,从而导致下游信号传递和防御反应的启动[7.].
许多R.-基因位于集群中,或由多个同源序列的副本产生于一个基因家族或共同定位R.- 来自无关基因家族的葡萄项序列[8.那9.].这种基因组组成帮助多种蛋白质通过各种基因和基因间过程被修改,从而使病原体基因组的变化能够快速进化和适应[10.].R.-Genes也可以成对作用[11.那12.].这R.- 基因组位置和蛋白质结构域结构可以不同,但也可以与其与不同病原体分离物的相互作用。
的雌雄异体的子囊菌Setosphaeria turcica(Luttrell)Leonard&Suggs(Anamorph:exserohlium turcicum,前蠕孢菌turcicum)导致Turcicum或Northern叶片枯萎病玉米。这种真菌病原体还攻击高粱和相关草地,例如约翰逊草[13.那14.].Turcicum Leaf枯萎是世界上大多数玉米地区最普遍的叶面疾病之一。该疾病导致与显着产量损失相关的周期性流行病,特别是在中等温度和高湿度的条件下[15.-17.].抵抗S. Turcica主要以玉米为特征。S. Turcica早些时候是名字蠕孢菌turcicum迄今为止,抵抗已经被指定H T并由主要种族特异性基因赋予(Ht1那HT2,HT3要么HtN)通过Welz和Geiger审查的部分抵抗,或通过部分抵抗[18.].在我们的工作中,我们指定新的阻力基因圣参考Setosphaeria turcica.
玉米和高粱是撒哈拉以南非洲(SSA)最重要的主食谷物。玉米是一种引进的作物[19.],高粱被认为是在SSA驯化的驯养盆地或埃塞俄比亚,最近是1000英镑[20.].和许多其他作物一样,高粱也面临着植物病原体,尤其是真菌疾病的严重问题。Turcicum叶枯引起S. Turcica是一个主要问题[21.].这种疾病被认为是乌干达的次要重要性,直到1988年,它在玉米上引起了广泛的产量损失[22.].通过在新品种中引入改良的抗性,这种疾病的威胁随后减少了。严重和零星的郁金香叶枯病现在已在东非重新出现[23.-25.].变化S. Turcica人口已被认为是造成这种疾病模式转变的主要原因。以检测潜在的新变化S. Turcica病原体和火棘叶枯萎病,在乌干达进行了一项调查,以检查高粱 -S. Turcica疾病严重程度和发病率方面的病理系统、种族模式和新的抗药资源[26.].从这些研究可以得出结论,真菌分离自高粱可以侵染玉米。杂交接种后的玉米差异品系表现出不同H T基因,四S. Turcica分离到的菌株分别为1个小种,2个小种,1个对应0个小种和3个小种,10个未分类。来自地区收集的高抗高粱品种也被鉴定。
在本研究中,我们利用cdna扩增片段长度多态性(AFLP)对抗性和敏感的玉米和高粱的基因型进行了分析,以鉴定当受到胁迫时差异表达的基因S. Turcica.随后是使用病毒诱导的基因沉默(Vigs)的所选基因候选的功能评估布罗姆花叶病毒向量。我们发现一个R.-基因簇,包含6个CC-NB-LRR编码基因,共3对,分布在高粱基因组中S. Turcica.基因组数据进一步表明圣基因在单子叶植物中高度保守。
结果
标识上调R.-基因家族对玉米和高粱的响应S. Turcica接种
以确定重要的防御基因S. Turcica,在真菌感染后对易感易感(S)和抗性(R)高粱和玉米基因型进行CDNA-AFLP分析。在我们的情况下,使用乌干达高粱基因型Ga06 / 18(R)和Sila和Sila和玉米A619Ht1(R)和A619(S)系。早期已经评估了高粱材料在各种农艺性状中评估了包括重要真菌疾病的各种农艺性状。除了S. Turcica响应,Ga06 / 18被发现易受尾孢属sorghi,collettrichum sublineolum,而Sila易受C. Sorghi.和抵抗力C. Sublineolum..
总共,使用不同的底漆组合监测大约3000个转录物衍生的片段,范围为50至600bp(附加文件)1).在接种后24、48和72小时采集的与敏感基因型相比,抗性基因型中独特的、上调或下调的转录本被切除、扩增、测序并分析假定的功能。最终的转录组由68个高粱和82个玉米候选基因组成。在这些基因中,根据BLASTP在其他生物体中鉴定的最接近的基因,分别有11个和13个推定与胁迫相关。
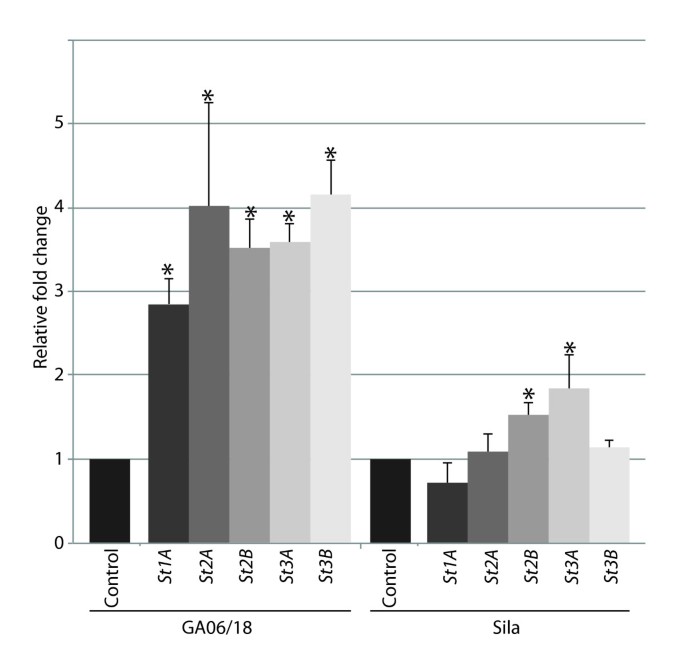
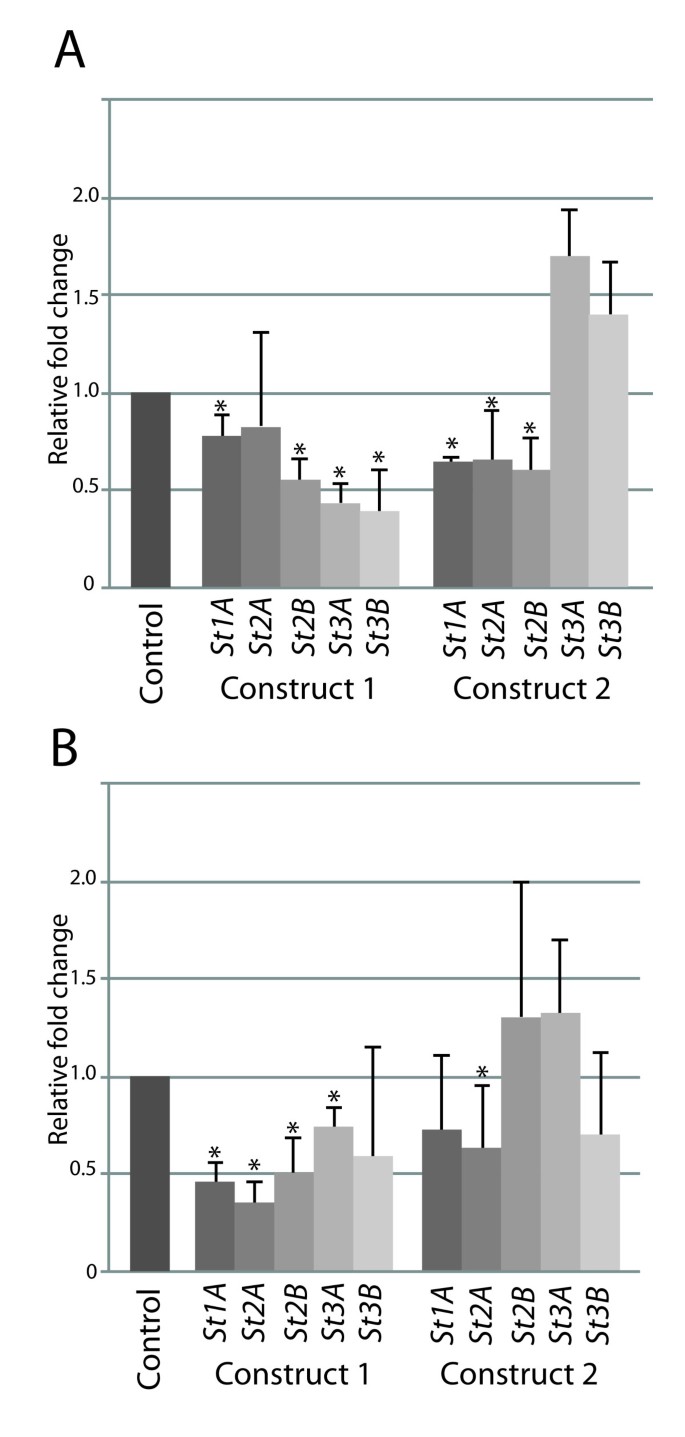
一个CC-NB-LRR编码推定R.- 那烯(GRMZM2G005347),与之同源基因对的成员GRMZM2G005452在2号染色体相同的位点上,并在抗性玉米基因型中唯一表达,进一步研究(图1D).基因组分析显示高粱中存在6个同源基因(图)1A).这六个基因被赋予前缀圣参考S. Turcica并指定St1A(Sb05g008280),St1B(SB05G008140.),St2A(SB05G008350.),St2B(Sb05g008030),St3A(Sb05g008250), 和St3B(Sb05g008270).实时荧光定量PCR进一步证实St1A那St2A那St2B那St3A和St3B)六个中圣当抗高粱抗性GA06 / 18植物攻击时,基因显示出高相对转录水平S. Turcica(数字2).一个基因,St1B,与另一个相比表达了更低的程度圣基因,超出了检测的限度。在尸罗,只有St2B和St3A显示出显著的增长(P.< 0.005)S. Turcica(数字2).
相对QPCR值St1A, St2A那St2B那St3A和St3B高粱GA06 / 18的转录物和接种的硅胶植物S. Turcica现病史,24.在各自的基因型中使用水处理作为对照。误差棒表示三次生物复制之间的标准偏差。*表示显着增加(P.<0.05)与对照水平相比。使用在附加文件中列出的底漆2.
这圣基因在草丛中被保守
6圣高粱的基因在5号染色体上形成了三个基因对,并且拥有共同的祖先(图)1;附加文件2;附加文件3.).圣在搜索米饭,玉米,粪码小米和血清卷曲基因组数据库时,还发现基因直肠。这圣基因编码来自其他草类的蛋白质,与高边支持的高粱St蛋白分组(100)(附加文件2).水稻基因组中含有高粱的同源基因St1A那St1B那St2A那St2B和一个ST3.基因(图1 a, B).这表明水稻和高粱的祖先很可能复制了这些基因。高粱St3A和St3B很可能是最近水稻和高粱物种分裂后基因组复制事件的结果(图1G).水稻基因组也包含多个副本St1A, St2A和St2B同源基因,可能是在该物种从高粱中分离后产生的基因复制。同样,Setaria italica(谷子)基因组包含同源基因St1A那St1B那St2A和St2B由于完整的染色体注释尚未确定(图)1C.).一个ST3.小米中未发现同源物。除了在我们的cDNA-AFLP分析中鉴定的玉米基因对之外,BLASTP和BLASTN搜索还发现了第三个单基因同源物,GRMZM2G050959, St2A玉米染色体2(图1D).另一方面,模型草的基因组,有一对同源的基因St1B在染色体4,一对一St2B在5号染色体上,但缺乏所有其他的同源基因(图1E).这圣在高粱、水稻和谷子基因组之间保持着基因簇,但在玉米和短叶蝉基因组中较小圣位于不同的染色体上的基因。
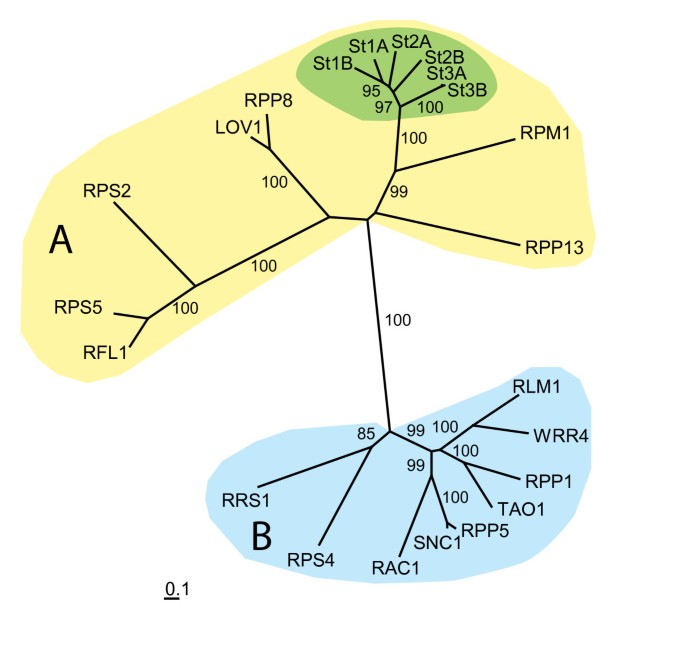
高粱St蛋白与拟南芥CC-NB-LRR编码基因序列也存在同源性(图)3.;附加文件4.).所有6个St蛋白与CC形成一个簇,而不是含有R蛋白的TIR结构域拟南芥表明与预期更近的进化关系。最近的相关拟南芥基因是RPM1,一种介导耐受的基因两隔离表达avrRpml要么AVRB.基因(27.].
从核苷酸结合(Nb)和少氨酸富含重复(LRR)结构域,六种抗性蛋白质的未生根最大似然性PhylographS. Turcica(ST)高粱,与拟南芥NB-LRR抗性蛋白相比,具有已知功能的蛋白质.显示75%以上的LR-ELW值。A. NB-LRR抗性蛋白质,N-末端具有卷曲线圈(CC)域。B. NB-LRR抗性蛋白质在N-末端具有损伤/白细胞介素-1受体(TIR)。单位表示替换/网站。使用的r-proteins列在附加文件中4..
VIGS系统在高粱上的应用
高粱和玉米的遗传转化是可能的,但需要艰苦的努力,需要其他基因型以外的研究才能成功[28.那29.].因此,使用病毒诱导的基因沉默(Vigs)进一步研究了我们的候选基因使用布罗姆花叶病毒BMV系统,以前用来使单子叶植物的基因沉默[30.].真菌接种的重点是评估潜在的防御功能圣基因。在我们手中,在应用于A619HT1玉米基因型时,Vigs程序并未成功。因为这圣在真菌接种时上调基因S. Turcica在我们的高粱GA06/18基因型(图2),我们继续研究高粱材料。
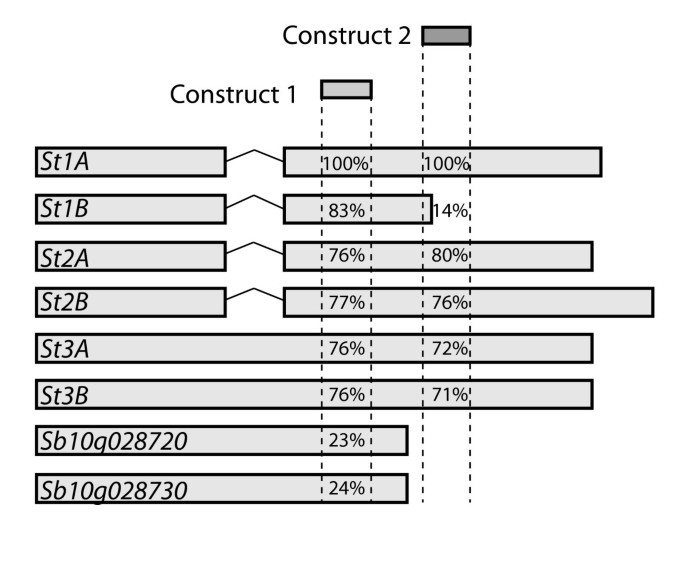
两个重点构建(1和2),高度高度为6圣高粱的基因被设计(图4.),包括检测其脱靶基因沉默能力。最高的非圣高粱基因相似性属于相关的R.基因对,Sb10g028720和Sb10g028730,根据系统发育分析位于不同的亚群(附加文件2),作为脱靶基因沉默的对照。扩增所选序列,在BMV系统中连接到第三个质粒(pF13m),用于侵染高粱植株。
首先优化Vigs程序。在播种之前,高粱种子在播种前灭菌,以最小化其他微生物的额外应力。通过体外转录产生mRNA,加入到接种缓冲液中并直接摩擦到三周大高粱植物的第二片叶子上。使用涉及大麦作为病毒宿主的中间步骤。病毒在整个植物中系统性地蔓延,在接种位点上方的第二和第三叶中沉默,并且很少达到沉默[30.].感染后7天,在第三片叶片上可以看到浅绿色的条纹,表明病毒症状和被病毒感染成功。为了确定沉默的开始,我们对VIGS处理的植物叶片样品进行了实时定量pcr(图)5.).与空白质粒接种的对照植株相比,相对转录水平显著下降,这表明6个靶基因中有5个明显下调,特别是构建1基因在两种高粱基因型中下调。相对转录水平Sb10g028720和Sb10g028730在VIGS治疗中没有受到影响,表明没有脱靶沉默。
沉默的圣基因增加S. Turcica抗性和易感高粱基因型感染
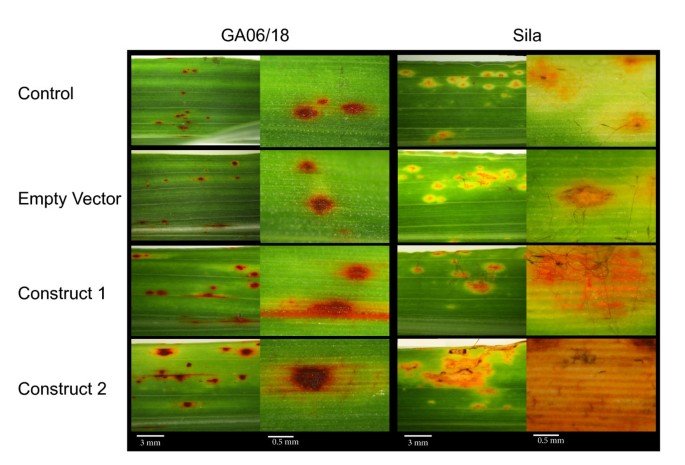
与对照材料相比,接种不同VIGS结构的植物上的真菌定植和生长被仔细监测。图中总结了不同的表型观察结果6.;和附加文件5..进一步评估真菌生长情况的方法是分离受感染的叶子,将它们放在含有潮湿滤纸的培养皿中,然后在25°C的黑暗中培养两天,如Levy所述[31.].通过叶片病变突出的分枝细胞的发展,然后是快速无性孢子发育表明叶片材料的真菌定植,以及易感表型。
抗真菌基因型GA06/18在2 dpi处出现超敏反应(小黑点/红点),而空载体处理的植株则出现稍晚的HR表型3 dpi。在接种真菌之前,将VIGS构建体1应用于GA06/18植株,与对照植株相比,出现了更大、更多的带绿晕的病变。病害病害沿叶片横向传播,真菌分生孢子和孢子在产孢条件下产生。类似地,当构建物2的作用被测定时,疾病病变被观察到2dpi,并向外侧扩散形成产生大量真菌孢子的大病变。在7 dpi时,疾病病灶比构造物1引起的病灶大。在感病的Sila植株上,真菌附着体周围可见明显的病害症状、坏死斑点和褪绿晕。坏死病变凸出的分生孢子上产生大量的无性真菌孢子。当用空的VIGS载体接种Sila植物时,在真菌接种之前,类似的疾病症状出现了2dpi。相比之下,在接种了我们的VIGS结构体1的Sila植株上,与对照植株相比,出现的病变略大且更频繁。当使用construct 2时,疾病症状进一步放大,导致更大的坏死病变和大量的真菌孢子生成。 In order to correlate these observed disease phenotypes with fungal growth, fungal DNA was quantified in the VIGS materials (Figure7.).S. Turcica在Ga06 / 18叶中的DNA增加到1.5±0.4pg / ng高粱DNA,在使用vigs构建体1,以及使用构建体2的3.6±0.9pg / ng高粱DNA,从对照植物中的接近零水平(非凡和空矢量)。一个重要的(P.<0.005)在用构建体1(1.2±0.4pg / ng高粱DNA)的Sila的样品中也发现了真菌DNA的增加,并且与约0.5的对照样品相比,构建2(0.8±0.9pg / ng高粱DNA)。pg / ng高粱DNA。
随着预期的,当接种vigs构建体1或2之前的真菌接种时,抗性GA06 / 18基因型抗性GA06 / 18基因型显示出受损的防御反应。有趣的是,在相应的Vigs治疗中,我们观察到易感淤泥基因型的增强疾病表型。
讨论
高粱(高粱双色(l)芒克]是主要的粮食和饲料资源,特别是在世界干旱和半干旱地区[32.].它主要是自花传粉的二倍体禾草种(2n = 2x = 20),基因组大小为1C = 730 Mbp,约为玉米基因组大小的25% [4.那5.].在高粱基因组中,211 NB-LRR编码R.-基因是存在的,大约一半的数量在水稻中发现,略多于拟南芥[4.].Brachypodium野生草小基因组中NB-LRR编码基因估计有178个[6.].但在更大的玉米基因组中,迄今为止已经确定了95个NB-LRR编码基因[33.].但是,根据搜索程序和阈值设置,略有不同R.-每一种草的基因数量都会被公布。
它提起了大量的R.植物基因组中的-基因及其巨大的序列多样性是抵抗病原菌攻击的监测机制中必不可少的进化因素。抗性基因通过复制、不平等交叉、重组和多样化而进化,从而产生多组副同源基因[10.那34.].的扩散R.-Genes还与基因拷贝的快速营业额相结合,最终导致缺失或扩张,从而动态R.基因簇(33.].在禾草科的不同物种中,抗性基因簇也被发现是保守的[35.],但这样的集群只占少数,71.6%是特定物种[33.].
全基因组重复发生,当草亚毛利彼此分歧和基因组数据进一步时,高粱和稻米的古重复基因对仍然存在于约17%的病例[36.].在水稻染色体11和12上特别发现最近的染色体段,以及高粱中的染色体5和8的相应区域。染色体5,在测序的BTX623高粱基因型中,其中圣基因位于展示最高的丰度(62)R.-Genes [4.].这些Nb-LRR编码基因中的三十六个基于王等人提出的生物信息分析的重复事件受到最近的重复事件的影响。[36.),包括St3A和St3B,这与我们的结果一致(图1;附加文件2).有趣的是,水稻基因组含有直肠的St1A那St1B那St2A那St2B和一个ST3.基因,一体都在一个轨迹中。这表明该基因簇预测水稻和高粱的种类分裂。在草家庭,高粱,玉米和小米属于同一次生(胰岛素),而米饭位于Ehrhartoideae [37.].据估计,这两个亚科起源于5000万至7000万年前的共同祖先,以及短柄草、小麦和大麦所属的普伊德亚科。
在基因组范围内比较拟南芥和A. Lyrata.,进化模式R.-基因可分为两个不同的组,两个物种间序列差异高的正选择(> 50%)和稳定选择的基因(< 30%)[38.].剩下的基因只在一个基因组中发现,而在另一个基因组中缺失。这圣在这项工作中发现的基因经历了很少的序列交换,导致了低分化,因此更类似于稳定选择的基因的描述,尽管5个草基因组之间的拷贝数不同(图)1).nb-lrr编码R.-Genes在不同的草地之间仍然保守,目前被认为是一种常见的现象[33.].
高粱植物,特别是种子颜色为红色的基因型,会积累一系列酚类物质以应对病原体的攻击[39.].在我们的材料中也可以看到大量的红色黄酮类化合物在感染部位诱导,特别是在抗性基因型中。黄酮类化合物是否有助于对抗S. Turcica虽然还没有被阐明,但是在高粱中已经发现了一种遗传联系C. Sublineolum.相互作用,通过存在3-脱氧菁胺(3-脱氧)产生[40].在许多疾病系统中,已经观察到植物细胞在病原体攻击时通过胼胝质沉积而增强。增强的胼胝质沉积也被报道为一种抗性反应S. Turcica在玉米[41.].尽管进行了广泛的染色,但在我们的高粱基因型中均未发现胼胝质积累(数据未显示)。
此外,我们的基因沉默工作导致肉体敏感的敏感反应,我们的易感高粱品种。这种观察可能表明通过瞄准圣在该基因组背景下,揭示了对抗高粱基因型中掩盖的下游信号传导的影响,并且可能构成发现的定量性状的一小部分[41.].这个假设是推测性的,并且仍有待包括在未来的功能研究中圣基因。未来的研究还包括对JGI最近发布的基因组中重要的效应因子的搜索http://www.jgi.doe.gov.并行,来自的序列信息圣基因簇目前已转化为分子标记,用于东非的种质资源评估和育种计划,这是维持该地区高粱和玉米作物生产的重要进展。
结论
我们对敏感和抗性玉米和高粱基因型进行了cDNA-AFLP分析S. Turcica结果在玉米中鉴定到一个CC-NB-LRR编码基因。该基因位于玉米2号染色体上的两个位点。在高粱、6圣正交基因存在于染色体上的三对簇中。在高粱的基因沉默时圣实时荧光定量PCR分析和真菌DNA定量分析均支持这一观察结果。数据库检索和系统发育分析表明圣基因有一个共同的祖先存在于亚家族分裂之前,5000万至7000万年前,这些基因在高粱、水稻、谷子、玉米和短柄草中高度保守。
方法
植物和真菌材料
抗(R)感(S)高粱双色研究中使用来自乌干达,GA06 / 18(R)和Sila和Sila和Sila和Sila的基因型,并通过USDA ARS提供的A619HT1(R)和A619(S)。在22℃下使用12/12 H光周期在生长室(Percival)中生长植物。单个孢子隔离S. Turcica感染高粱(IG1.)或感染玉米(Mb1)来自乌干达的Iganga和Mbale收集,分别用于所有高粱和玉米分析。使用改性的CTAB方法提取真菌DNA [42.].通过使用分析DNAS. Turcica特定ITS1及其2引物(F-GCAACAGTGCTCTGCTGAAA和R-ATAAGACGGCAACACAG)。使用以下条件进行PCR:将10 ng模板DNA加入到由H组成的24μL混合物中。2O, 2.5 mM氯化镁2,2.5μl.Taqbuffer (Fermentas, Helsingborg, Sweden)每个dNTP 0.2 mM, 0.25 μM正向和反向引物,1uTaq聚合酶(Fermentas): 3分钟在94°C, 35周期(在94°C 1分钟,1分钟60°C,和1.5分钟在72°C),最后在72°C扩展10分钟。PCR产品1%琼脂糖凝胶上分离确认片段大小(344个基点)其次是测序(Macrogen Inc .,首尔,韩国)。
植物材料的真菌接种
用25 μl分生孢子悬浮液(5 × 10)接种3周龄幼苗至第3轮5.分生孢子/ml),由Carson描述[43.].将来自三到四种植物的接种叶片在24,48和78小时接种(HPI)的24,48小时,进行CDNA-AFLP分析。在同一时间点收获水处理的对照样品。
RNA提取和cDNA-AFLP分析
使用BioRad RNA分离试剂盒(BioRad, California, USA)从叶片样品中分离总RNA,然后使用mRNA捕获试剂盒(Roche, California, USA)制备mRNA。用Oligo-dT引物和RevertAid™H - M-MuLV逆转录酶(Fermentas)合成cDNA。第二股是用大肠杆菌DNA聚合酶I(Fermentas)。用双链cDNA消化BST.Y1和均方误差1(Fermentas)并连接到各自的适配器,预扩增,后来选择性地使用该适配器选择性地放大BST.yi + n(33.P标记)和均方误差我+ N引物。利用连接cDNA、Taq DNA聚合酶(Fermentas)和特异性非选择性引物进行预扩增BST.易,均方误差i适配器使用25个94°C循环30秒;56℃,1分钟,72℃1分钟。将预扩增的反应混合物稀释600倍,使用5μl用于用24种引物组合进行最终选择性扩增,进行BST.yi + n(33.P标记)引物(附加文件1)及触地放大[44.].选择扩增产物在6%的聚丙烯酰胺凝胶上以100 W分离至4300 Vh。凝胶干燥并暴露于柯达Biomax胶片(Amersham Pharmacia, California, USA) 5-7天。
转录物的分离和序列
从干燥的PAGE凝胶中分离出大约150个与敏感基因型相关的抗性基因型转录本(独特的、上调和下调的),并用H2O和PCR扩增使用非选择性引物在相同的条件下进行预扩增步骤。将产物克隆到pJET 1.2钝性载体(CloneJET™PC, Fermentas)中并进行测序。利用BLASTN和BLASTX程序对序列进行分析[45.],并与NCBI、GRAMENE和PHYTOZOME数据库的序列进行了比较。已鉴定的真菌序列被排除在外。
高粱病毒诱导基因沉默研究
使用的Vigs系统基于单子偶像感染布罗姆花叶病毒(BMV),如前所述[30.],但不包括预先接种大麦。BMV VIGS载体由三个质粒组成,分别含有BMV RNA1 (p1-1)、RNA2 (p2-2)和RNA3 (pF13m,正式形式为pF3-5/13)A / G.),分别。利用抗性GA06/18基因型基因组DNA和基因特异性引物,从高粱候选基因中扩增出长度为246 ~ 253 bp的PCR片段,构建VIGS结构体NCO我和Avr的II使用底漆3版本0.4.0的限制站点http://frodo.wi.mit.edu/primer3/软件(附加文件6.).在PCR扩增之前,我们进行了脱靶基因搜索以设计最佳的VIGS结构(图)4.).酶切后,每个片段克隆到pF13m质粒的相应位点。通过测序验证插入物的身份。P1-1、p2-2和含有不同结构物的pF13m经酶切Spe一世,Psh人工智能和Psh分别AI。通过线性化质粒合成传染性RNA转录本体外根据制造商的说明,使用T3聚合酶(FERMENS)进行转录。将1μl反应产物在1.5%琼脂糖凝胶上进行,以确认存在转录物。
按所述进行了植物接种程序[30.]略有修改。将10μL等分试样的来自每种质粒p1-1,p2-2和pf13m-incord与30μl接种缓冲液组合,并直接用于接种3周龄高粱的第二和第三片叶子和玉米植物。作为对照,植物以与P1-1,P2-2和空PF13M的水或组合的转录物相同的方式接种。玉米和高粱植物受到挑战S. Turcica如前所述,在病毒接种一周后(开始出现淡黄变和静脉清净)评估不同结构的效果。植物被随机化和编码,以减少真菌定植和生长评分的潜在偏差。
定量实时聚合酶链反应
在对经VIGS处理的高粱植株进行真菌接种之前,在每种条件下,从3株独立植株上采集VIGS接种叶片上方约3厘米的第2片叶片,如前所述用于RNA提取。根据制造商的说明,用Oligo-dT引物和RevertAid™H - M-MuLV逆转录酶(Fermentas)从1 μg总RNA中合成第一链cdna。利用iQ5 cycler (Bio-Rad)中的第一链cDNA进行实时PCR。采用Maxima Sybr Green/Fluorescein qPCR Master Mix (Fermentas)进行PCR扩增,总反应体积为20 μl,其中Sybr Green qPCR Master Mix为10 μl,正向和反向引物为0.3 μM, cDNA模板为5 ng。所有pcr均在以下扩增条件下进行3个重复;95°C下10 min,接着95°C下15 s, 60°C下30 s, 72°C下30 s,接着95°C下1 min,进行40个循环,并进行熔化曲线分析。引物序列圣使用底漆3版0.4.0设计基因http://frodo.wi.mit.edu/primer3/软件(附加文件7.).高粱伸长因子1-alpha(SB02G036420)和肌动蛋白(Sb01g010030)作为内参基因,计算相对转录值。所有计算和统计分析都按照Vetukuri等人稍作修改的ABI PRISM 7700序列检测系统用户公告#2(应用生物系统,美国)中的描述进行。[46.].量化的S. Turcica如前所述,在真菌接种后7天对VIGS材料进行DNA检测[41.].合并来自三种植物的大约3厘米的叶片,并提取DNA。分析了每次处理的三个生物样品。使用学生的T检验计算统计显着性。
基因组分析
氨基酸序列St1A(Sb05g008280),St1B(SB05G008140.),St2A(SB05G008350.),St2B(Sb05g008030),St3A(Sb05g008250),St3B(Sb05g008270),利用BLASTN和BLASTP (PHYTOZOME)技术与高粱、玉米、小米、水稻、短穗芥和拟南芥基因组数据库进行比对。使用线圈-线圈预测识别预测域[47.]),lrrfinder [48.]及光碟查册[49.].圣利用基因组观察仪(PHYTOZOME)鉴定了类基因位点。使用Treefinder和最大似然法进行系统发育分析,共10 000个重复[50.].JTT+G模式[51.发现使用Prottest v2.4最适合数据[52.].使用预期的似然重量(LR-ELW)的局部重排来计算自信[53.].系统图使用Treeview 1.6.6绘制[54.].
参考文献
- 1。
Jones JDG,Dangl J:植物免疫系统。自然。2006,444(7117):323-329。10.1038 / Nature05286。
- 2。
Meyers Bc,Kozik A,Griego A,Kuang H,Michelmore Rw:拟南芥中Nb-LRR编码基因的基因组宽分析。植物细胞。2003,15(4):809-834。10.1105 / TPC.009308。
- 3.
刘军,刘旭,戴丽,王刚:植物抗病基因结构、功能及其进化的研究进展。遗传与基因组学杂志。2007,34(9):765-776。10.1016 / s1673 - 8527(07) 60087 - 3。
- 4.
帕特森啊,Bowers Je,Bruggmann R等:高粱双色基因组和草的多样化。自然。2009,457(7229):551-556。10.1038 / Nature07723。
- 5。
Schnable PS,Ware D,Fulton Rs,等:B73玉米基因组:复杂性,多样性和动态。科学。2009,326(5956):1112-1115。10.1126 / Science.1178534。
- 6。
国际短柄草倡议:模型草的基因组序列和分析Brachypodium distachyon.自然。2010,463(7282):763-768。10.1038 / Nature08747。
- 7。
nb - lrr对病原体起着“诱饵和开关”的作用。植物科学进展,2009,14(10):521-529。10.1016 / j.tplants.2009.08.001。
- 8.
在等位基因和单倍型多样性RP1.玉米铁锈抵抗轨迹。遗传学。2004,167(4):1939-1949。10.1534 / genetics.104.029371。
- 9.
Kuang H, Woo S-S, Meyers BC, Nevo E, Michelmore RW:多种遗传过程导致莴苣中主要抗疾病基因的异质性进化率。植物学报。2004,16(11):2870-2894。10.1105 / tpc.104.025502。
- 10。
McDowell JM,Simon SA:最近的见解R.基因进化。植物营养与肥料学报。2006,27(5):437-448。10.1111 / j.1364-3703.2006.00342.x。
- 11.
迪克森MS,Jones Da,Keddie Js,Thomas Cm,Harrison K,Jones JDG:番茄CF-2抗病源位点包括编码富含亮氨酸的重复蛋白的两种功能基因。细胞。1996,84(3):451-459。10.1016 / s0092-8674(00)81290-8。
- 12.
Eitas TK,Dangl JL:NB-LRR蛋白:对,碎片,感知,合作伙伴和途径。植物生物学目前的意见。2010,13(4):472-477。10.1016 / J.PBI.2010.04.007。
- 13。
Aragaki M:致病性的遗传Setosphaeria turcica.植物病理学。1975,65(3):280-283。10.1094 / phyto-65-280。
- 14。
Chiang M,Van Dyke C,Leonard K:潜在的Johansongrass潜在生物控制的地方性真菌评估(高粱霍尔德):筛选和主机范围测试。病害。1989,73(6):459-464。10.1094 / PD-73-0459。
- 15.
Perkins JM,Pedersen WL:疾病发展和与玉米北部叶片相关的产量损失。病害。1987,71(10):940-943。10.1094 / PD-71-0940。
- 16。
关键词:玉米,部分抗性,光照,温度exserohilum turcicum.病害。1994,78(4):408-411。
- 17。
Pratt R,Gordon S:育种玉米叶柄病原体的育种。植物育种评论。2006,27:119-173。
- 18。
魏志强,王志强:玉米叶枯病抗性基因的研究进展。植物育种。2000,119(1):1-14。10.1046 / j.1439-0523.2000.00462.x。
- 19.
Mangelsdorf PC:玉米的起源、演变与改良。1974年,马萨诸塞州坎布里奇贝尔纳普出版社美国
- 20。
金伯CT:驯养高粱的起源及其早期扩散到印度和中国。高粱:起源,历史,技术和生产。编辑:史密斯CW,Frederiksen Ra。2000年,约翰·威利和儿子,纽约,美国,3-98。
- 21。
梁志强,王志强,王志强:高粱炭疽病和叶枯病在不同类型作物混播中的同步时间进程。植物病理学报。2001,91(8):720-729。10.1094 / PHYTO.2001.91.8.720。
- 22。
Adipala E, Lipps EP, Madden LV:发生exserohilum turcicum在乌干达的玉米。病害。1993,77(1):202-205。
- 23。
Ebiyau J, Oryokot OE:高粱(高粱双色(L.)Moench。乌干达农业。2001年,农作物:国家农业研究组织喷泉发报厂:
- 24。
Muiru WM:组织学研究和种族的特征exserohilum turcicum肯尼亚玉米北叶枯叶的因果玉米代理。博士论文。2008年,肯尼亚内罗毕大学
- 25。
Ramathani I:Turcicum叶片枯萎流行病和病原体种群的表征exserohilum turcicum-乌干达高粱病虫害。硕士论文。2009年,乌干达坎帕拉Makerere大学
- 26.
Ramathani I,Biruma M,Martin T,Dixelius C,Okori P:疾病严重程度,发病率和种族Setosphaeria turcica在乌干达的高粱。acta botanica sinica(云南植物学报),2011,31(3):457 - 461。10.1007 / s10658 - 011 - 9815 - 1。
- 27.
格兰特先生,Godiardt L,Straube S,Ashfield T,Lewald J,Satler A,Ines Rw,Dangl JL:结构的结构RPM1双特异性抗病基因。中国科学(d辑):地球科学(英文版)。10.1126 / science.7638602。
- 28.
Opabode J:农杆菌介导的植物的转化:影响效率的新因素。生物技术与分子生物学审查。2006,1(1):12-20。
- 29.
Gurel S, Gurel E, Kaur R, Wong J,孟L, Tan HQ, Lemaux PG:高效,可复制农杆菌利用未成熟胚的热处理对高粱进行-介导的转化。植物学报,2009,28(3):429-444。10.1007 / s00299 - 008 - 0655 - 1。
- 30.
丁xs, Schneider WL, Chaluvadi SR, Mian MAR, Nelson RS: a布罗姆花叶病毒菌株及其作为单子叶寄主基因沉默的载体。植物-微生物分子互作。2006,19(11):1229-1239。10.1094 / mpmi - 19 - 1229。
- 31.
Levy Y:田间分离菌株适合度的变异exserohilum turcicum在以色列。植物病虫害。1991,75(2):163-166。10.1094 / pd - 75 - 0163。
- 32.
《高粱》1988,美国纽约,第2期
- 33。
李家,丁俊,张义,王家,陈俊琦,田D,杨S:禾本科NBS-LRR基因的独特进化模式。Mollucar Genetics&Genomics。2010,283(5):427-438。10.1007 / S00438-010-0527-6。
- 34。
陈志强,陈志强,陈志强,等。植物生物学进展,2005,8(2):129-134。10.1016 / j.pbi.2005.01.002。
- 35。
罗S,Peng J,Li K,Wang M,Kuang H:对比的进化模式RP1.禾本科不同种的抗性基因家族。分子生物学与进化。2011,28(1):313-325。10.1093 / molbev / msq216。
- 36。
王X,王H,帕特森啊:七百万年的同源染色体对的协调演变,并行,在主要的Poaceae谱系中。植物细胞。2011,23(1):27-37。10.1105 / TPC.110.080622。
- 37。
Bolot S,Abrouk M,Masood-Quraishi U,Stein N,Messing J,Feuillet C,Salse J:谷物基因组的内圈。植物生物学目前的意见。2009,12(2):119-125。10.1016 / J.PBI.2008.10.011。
- 38。
陈强,韩振江,田东,杨胜:强正向选择驱动企业快速多元化R.基因在拟南芥亲戚们。分子进化杂志。2010,70(2):137-148。10.1007 / s00239-009-9316-4。
- 39。
高梁酚类化合物和相关酶对高粱品种抗生物和非生物胁迫敏感性的影响。化学学报,2005,31(11):2671-2687。10.1007 / s10886 - 005 - 7619 - 5。
- 40。
对炭疽病叶枯病的植物抗毒素依赖抗性需要功能性黄色种子1在高粱双色.中国生物医学工程学报。2010,31(4):531 - 534。10.1534 / genetics.109.111831。
- 41。
Chungfellow,Walsh Ek,Kerdieh Z,Van Esbroeck G,Balint-Kurti P,纳尔逊RJ:影响菌发病机制明显阶段的抵抗基因座:使用旋毒线进行QTL线条测绘和玉米特征Setosphaeria turcicapathosystem。植物营养与肥料学报。2010,29(6):723 - 732。
- 42。
Okori P, ruaihayo PR, Ekwamu A, Fahleson J, Dixelius C:遗传特征尾孢属sorghi从栽培和野生高粱及其与其他的关系Cercospora.菌类。植物病理学。2004,94(7):743-750。10.1094 / phyto.2004.94.7.743。
- 43。
Calsson ML:玉米感染潜伏期长度的遗传exserohilum turcicum.植物病虫害。1995,79(6):581-585。10.1094 / pd - 79 - 0581。
- 44。
Vos P, Hogers R, Bleeker M, Reijans M, Vandelee T, Hornes M, Frijters A, Pot J, Peleman J, Kuiper M, Zabeau M: AFLP是一种新的DNA指纹识别技术。核酸研究,1995,23(21):4407-4414。10.1093 / nar / 23.21.4407。
- 45。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中国生物医学工程学报,2003,23(3):427 - 434。
- 46。
Vetukuri RR, Avrova AO, Grenville-Briggs LJ, van West P, Söderbom F, Savenkov EI, Whisson SE, Dixelius C: Dicer-like、Argonaute和Histone Deacetylase蛋白参与基因沉默的证据5种.植物病理学杂志。2011,12(8):772-785。10.1111 / j.1364-3703.2011.00710.x。
- 47。
Lupas A, Van Dyke M, Stock J:从蛋白质序列预测螺旋线圈。中国科学:地球科学。
- 48。
LRRfinder:一个用于识别富亮氨酸重复序列和一个完整的toll样受体数据库的web应用程序。发展与比较免疫学。2010,34(10):1035-1041。10.1016 / j.dci.2010.05.004。
- 49。
CD-Search:动态的蛋白质域注释。中国生物医学工程学报。2004,32(4):457 - 461。10.1093 / nar / gkh454。
- 50.
Jobb G,von haeseler a,strimmer k:treefinder:一个强大的图形分析环境,用于分子发育。BMC进化生物学。2004,4:18-10.1186 / 1471-2148-4-18。
- 51.
Jones Dt,Taylor WR,Thornton JM:从蛋白质序列的快速产生突变数据矩阵。计算机应用在生物科学中。1992,8(3):275-282。
- 52.
Abascal F,Zardoya R,Posada D:Prottest:选择最适合的蛋白质进化模型。生物信息学。2005,21(9):2104-2105。10.1093 / Bioinformatics / BTI263。
- 53.
Strimmer K,Rambaut A:推断出可能遗料的基因树的信心集。皇家学会的诉讼程序B:生物科学。2002,269(1487):137-142。10.1098 / RSPB.2001.1862。
- 54.
Page Rd:TreeView:应用程序在个人计算机上显示系统发育树的应用程序。Biosciences的计算机应用程序。1996,12(4):357-358。
- 55.
黄志强,王志强,王志强,等。绿色基因与生命绿色分支的比较基因组学。acta photonica sinica, 2012, 37(2): 372 - 372。10.1016 / j.cell.2007.04.004。
确认
我们非常感谢B. Sarosh对cDNA-AFLP工作的协助。这项研究是由Sida - sarec对东非生物技术网络(BIO-EARN)、Sida swee -2005-453和瑞典农业科学大学的赠款资助的。
作者信息
隶属关系
相应的作者
附加信息
作者的贡献
TM进行了qPCR、基因组分析、绘图,并与MB一起进行了cDNA-AFLP和VIGS分析。IF支持VIGS分析。CD和PO构思了这项研究,并与所有作者一起参与了写作。所有作者阅读并批准了最终的手稿。
电子辅料
基于高粱St蛋白的螺旋螺旋结构域(CC)、核苷酸结合结构域(NB)和富含亮氨酸重复结构域(LRR)的氨基酸序列,以及密切相关的R蛋白,采用JTT+G模型构建最大似然系统发育树
附加文件2:。名称是植物体基因标识符。Physcomitrella金属盘R-protein Pp1s1_327V6作为外组。LR-ELW边缘支持值显示[53.].指示每个网站的替换。(PDF 331 KB)
有关用于拟南芥基因的信息
附加文件4:图3..从TAIR数据库检索的数据.(DOC 40 KB)
12870 _2011_958_moesm5_esm.doc
额外的文件5:高粱叶片疾病表型,监测接种后1-12天(DPI)S. Turcica在耐野生型GA06 / 18和敏感的淤泥品种。在真菌接种之前,用水,空BMV载体,构建体1或构建体2处理植物。数据由每个BMV构建和控制的25-30株植物编译。实验重复2次。(Doc 34 KB)
作者的原始提交的图像文件
权利和权限
本文在BioMed Central Ltd.的许可下发布了这是一个开放的访问文章,根据Creative Commons归因许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
马丁,T.,比鲁马,M.,弗里德堡,I。等等。高度保守的NB-LRR编码基因集群有效Setosphaeria turcica在高粱。BMC植物BIOL.11,151(2011)。https://doi.org/10.1186/1471-2229-11-151
收到了:
公认:
发表:
关键词
- Foxtail小米
- 玉米基因型
- 真菌接种
- 病毒诱导基因沉默
- 高粱植物