摘要
背景
快速且一致的基因分型是许多作物物种中品种鉴定的重要要求。其中,葡萄园品种是多个研究的主题,鉴于许多几个世纪的植物繁殖和交换期间产生的大量同义词和同音异义。简单的序列重复(SSR)标记是优选的,直到现在是由于它们的高水平多态性,它们的Codominant性质及其高型重复性。然而,部分或完全基因组测序方法的快速应用是鉴定成千上万的单核苷酸多态性(SNP),其对于这种目的非常有用。虽然SNP标记是双位等位基因的,因此不像微卫星一样多态性,但是可以复用的大量基因座以及自动化的可能性以及它们在任何分析程序下的高度可重复的结果使它们成为任何选择的未来选择标记基因鉴定类型。
结果
在选择11个基因型中,我们使用重新测序策略分析了葡萄树的基因组中的300 snp。在所识别的多态性中,我们选择了48个SNP,在所有葡萄染色体上分布,等位基因频率足够平衡,以提供足够的信息含量以在葡萄树中遗传鉴定允许良好的基因分型成功率。在全球获得的所选品种组的重复分析中测试标记稳定性,以证明其在遗传鉴定中的有用性。
结论
我们选择了一套48个鉴别能力强、基因组分布均匀的稳定SNP标记(2-3个标记/染色体),提出作为葡萄(vitis ViniferaL.)基因分型。在实验室之间的微卫星等位基因混淆的任何问题或者需要运行参考品种以识别等位基因尺寸的任何问题使用这种类型的标记消失。此外,因为SNP标记是双位等位基因的,所以等位基因鉴定和基因型命名是非常简单的,并且用不同的设备和不同的实验室获得的基因型始终完全可比较。
背景
葡萄树(vitis ViniferaL.)是世界上最宝贵的园艺作物之一。许多广泛种植的品种是非常古老的基因型,这些基因型已被植物乘以几个世纪以来并在全球范围内传播。在许多地方,同样的基因型被重新命名为导致同义词(同一品种的不同名称)以及同音异义(在相同名称下标识的不同品种)。目前,世界上有很大但不精确的葡萄种品种(数千人,[1]):一旦所有品种都适当基因分型并比较,此数字可能会降低。
考虑到遗传识别时,必须满足两种目标:i)足够大量多态性标记的可用性;并且ii)存在公共基因型数据库,允许与先前表征的基因型进行比较。标记应提供高辨别力,并在不同的实验室和检测平台中产生可重复的基因型数据以及随着时间的推移。标记也应该是稳定的,这意味着它们在重复传播品种后产生一致和可重复的结果。这在葡萄叶葡萄酒的情况下尤为重要,其中许多品种在几个世纪以来,由于细胞突变导致一些分子标记显示出某些旧品种的情况下不完全稳定[2].此外,应以低成本易于使用基因分型方法,并且应易于存储在数据库中并公开访问的可比性和基因型数据。
目前基于微卫星标记或简单的序列重复(SSR),这对遗传鉴定非常有用[3.]但还用于父母分析[4.].这些标志物对研究具有一些相关的研究优势,例如它们的共同统治,多位自然和高水平的多态性[5.].但是,使用SSR标记存在许多缺点。最重要的问题与等位基因分布有关:将原始等位基因长度转换为正常表达的等位基因长度的过程[6.].等位基因误叫的问题部分源于基于双核苷酸重复的SSR的广泛使用,以及DNA聚合酶频繁添加一个腺嘌呤核苷酸,导致等位基因大小非常接近,难以区分。根据全基因组序列提供的信息,使用核心重复序列长度为3至5个核苷酸的SSR可以部分解决这个问题[7.].However, even if longer repeat length markers are used, it is also important to take into account the fact that different analytical systems (e.g. DNA sequencers of different brands) could produce different allele sizes and consequently different bins, increasing the hardship of comparing genotype tables produced by different laboratories. To overcome these difficulties, standardization and exchange of information concerning grapevine genetic resources using reference varieties for certain microsatellite markers and alleles have been proposed [6.]并在欧洲项目中讨论,例如Genres 081和Grapegen06,旨在整合由不同实验室获得的基因型信息。
近年来,许多测序项目产生了丰富的序列信息和核苷酸多态性。这些属于两种基本类型:单核苷酸多态性(SNP)和不同长度的插入 - 缺失(Indel)。其中,SNP标记具有以下优点:它们主要是双位等位基因,并且在基因组中非常频繁。虽然SNP多态性信息内容(PIC)低于SSR标记,但是在需要时可以轻松使用数短,数百甚至数千个SNP。在实验室和检测技术中,SNP高度可再现,因为不同的等位基因不是基于它们的尺寸来区分,而是基于在给定位置存在的核苷酸的基础。所有这些功能及其无限制的可用性正在使SNP在许多动物和植物物种中开发识别面板的开发标记[8.-12.].
在这项工作中,我们的特征在于332 snp的遗传特征,以选择适用于葡萄葡萄酒中的品种鉴定的48个标记面板。我们在这里展示了面板具有与一组15个SSR标记相似的歧视电量,并且可以代表一个非常强大的遗传识别系统,没有问题在实验室或检测技术之间存在的等位基因错误。我们还证明标记物具有非常低的基因分型误差率,与SSR相比,新突变的出现低速率,并且可用于在基因型数据库中易于储存。鉴于葡萄树中遗传资源的修订和集成状态,我们的SNP面板可能成为遗传鉴定和基因型在作物中呼叫的快速工具。
结果与讨论
单核苷酸多态性(SNP)检测
基于如前所述的葡萄基因型的选定样品中的重新测序策略进行葡萄基因组中SNP标记的鉴定,如前所述[13.].选择该样品包括与古代原产地的无关葡萄酒和表葡萄品种以及野生加入。基于可用信息,栽培品种对应于不同的遗传群[14.]有属于葡萄葡萄酒中描述的四种主要类型的氯型[15.].以这种方式鉴定了总共270个SNP标记,我们在Csiro验证了一系列基因型中的62个SNP。对于最终的332 SNP,我们开发了基于SnPlex™的基因分型策略。分析葡萄葡萄酒中这些多态性的质量和估计其等位基因频率的第一步是基因型葡萄葡萄树的300种样品,包括葡萄酒和桌葡萄品种以及野生加入(附加文件1,表S1)。这种方法允许丢弃在分析中没有工作的61个SNP,尽管最初鉴定为序列比较中的多晶型,但在分析的样品中表现为单体,或者在100%的样品中被杂合的基因分为型样品中的杂合子重复的基因座。因此,仅考虑进一步分析238个SNP标记(附加文件1,表S3)。
SNP标记的基因组定位
通过对4个葡萄分离子代群体的7个SNPlex™基因分型,我们可以对238个多态性SNP中的大部分进行基因图谱,这些SNP在至少一个子代群体中的一个或双亲中是杂合子的(附加文件)1,表S4和S5)。平均而言,使用7套SNPlex™可以在任何给定的定位群体的共识图谱中包括114个标记:每个祖先(分离类型aaxab和abxaa)有42个标记,29个共同标记(abxab)。
为八个父母品种开发的综合图包括168微卫星和202个SNP(85%的多晶晶型SNP),允许鉴定在同一后代人群中未分离的标记的相对位置(图1,附加文件1,表S3)。由于链接分析不一致,无法映射三个额外的隔离SNP(附加文件1,表S3)。分子标记沿所有19个染色体分布,相邻标记的平均距离为3.4cm(仅在仅考虑SNP时)。集成图的总大小为1204厘米(附加文件1,表S4),类似于发布的其他完整链接地图vitis Vinifera[16.-19.].因为集成图基于平均重组频率[20.]考虑了总共313个后代个体,它应该提供良好的遗传距离估计。然而,分配给每个标记物的遗传位置的准确性受到其偏析的后代的数量的限制,每个后代的分离类型,具有扭曲的分离的标记的存在和可能存在的重组率之间的差异存在祖栽培品种。映射的202个SNP标记中的六十七百分之六十七分在一个以上的映射群体(分别为25%,27%和15%,分别为25%,27%和15%),只有11个SNP显示出较少的信息分离类型
SNP遗传和物理位置.对于每种染色体,左侧(灰色棒)的地图显示了在纯合线附近PN40024的12x葡萄序列上研究了SNP标记的物理位置[40]表示以千碱基;并且右侧(空条)上的地图显示了在厘米中的遗传位置,在厘米中,微卫星(在括号之间)和SNP使用四个隔离后代遗传映射。只有其中一个地图中的具有已知位置的标记以粗体表示:在左侧的地图中,SNP具有无法映射的已知物理位置;在右边SNP上的地图中映射遗传,但具有未知或不确定的身体位置。
序列搜索围绕序列的SNP(附加文件1,表S3)在12×基因组序列中vitis Viniferahttp://www.genosope.cns.fr/externe/genomebrowser/vitis/允许物理地定位大多数研究的SNP(图1,附加文件1,表S3)。238多态性SNP中的两百和二十五可以定位在物理地图上,平均每条染色体平均为12个SNP(从连杆组10,11,16和17的7个SNP,达到连锁组的21个SNP)。物理映射SNP之间的平均距离为1.76 MB。十三个SNP无法物理位置。这可能是由于与12×基因组序列(VV5629和SNP575_128)缺乏显着匹配,鉴定具有相同似然性的不同位置(SNP241_201和SNP1495_148)或其定位对未链接的染色体支架。链接映射允许本地化12的13个SNP中,该13个不能定位在物理地图中(附加文件1,表S3,图1)。无法映射物理或遗传(SNP575_128)的唯一标记对应于在12×基因组序列的搜索中找不到相邻序列的两个SNP中的一个。
在物理和遗传地图之间通常保守标记顺序,尽管在染色体1,3,10,12上发现差异差异,涉及高达7.6 MB和12厘米的差异。此外,对于染色体1,2,4,5,6,7,8,13和19,观察到涉及<1.5mb和<6cm距离的小局部标记逆转,涉及<1.5mb和<6cm距离,观察到(图1)。大多数这些差异可能归因于一些前面提到的因素,影响分配给每个标记的遗传位置的准确性。然而,这些因素中没有任何一个最重要的差异(染色体3和10)存在,这指出了这些区域的当前物理图中的一些问题,并且可能与12×葡萄序列的基因组重排或装配误差有关纯合线附近的PN40024http://www.genosope.cns.fr/externe/genomebrowser/vitis/.例如,标记SNP425_205(包括在SNP用于品种识别的SNP中包含的染色体3上的两个SNP标记之一)在与周围标记的物理和遗传距离之间存在显着的差异,导致该区域的标记顺序差异(图1,附加文件1,表S3)。在当前12×版本的基因组序列vitis Vinifera,该标记距离SNP613_315有1.4 Mb(这是该染色体品种鉴定的48个SNP集中的第二个标记)。然而,基因图谱上的标记顺序与两个SNP位点之间4.4 Mb的基因组序列中的标记顺序(NCBI为8×,数据未显示)以及黑皮诺序列中的标记顺序一致http://genomics.research.iasma.it/gb2/gbrowse/grape/.
选择基因识别SNP设置
目前,鉴于近年来出现了大量的微卫星和SNP标记,葡萄品种的实验室内遗传鉴定并不是一个主要问题[6.那7.那21.-23.].然而,开发一种高效,快速和便宜的系统非常重要,用于识别目前在葡萄树中有几千种品种。这需要仔细设计一组高度多态性和稳定的标记,具有经过验证的质量和再现性,允许构建数据库易于分享不同的实验室。为了开发这种基于SNP标记的系统,考虑了三个选择标准:基因分型成功的高频,高次等位基因频率(MAF)提供更高的照片和良好的染色体分布,最终得到了共有48个SNP分布的每条染色体的2-3只SNP的速率。当这些标准应用于可用的SNP时(附加文件1,表s3和图1),用于设计48 SNP集的选择(表1)获得了。建立了一个完全新的设计,只有所选择的48个SNP集合,彻底评估了它们的遗传识别稳定性和质量。
遗传鉴定SNP稳定性评价
48个SNP标记的稳定性通过分析每个15个品种平均85株的基因型来评估(附加文件)1,表s2)。本研究还允许评分基因分型成功率。15个品种代表葡萄葡萄树中的重要特征的大量表型多样性(葡萄酒,表格和葡萄干),浆果(黑色,红色和白色),成熟时间(早期,中期和晚期),种子的存在(种子和无籽)和其他特征[24.].除了它们不同的地理来源(法国,西班牙,近东,中东),这15个品种也显示出年龄差异:从非常古老的品种,可能超过1000年的历史(例如。“亚历山大的马斯喀特”,“汤普森无籽”),到仅仅起源于几个世纪以前的品种(例如:赤霞珠和20个国家培育的TH.世纪(例如'红衣主教','Crimson无籽')。
使用新设计的48 SNP套装分析了总共1342株植物。表格2显示为每种品种获得的基因型。在任何用于SNP VV1617的任何植物中没有基因型,因此,从分析中排除。然而,这种SNP定期在其他基因分型分析中工作,并包含在进一步的测试中。此外,SNP325_65和VV9227的基因分型完全失败了“Monastrell”品种。在几次分析后,可以获得SNP325_65的基因型,但这不是VV9227(数据未显示)的情况。丢弃该品种纯合无效等位基因的存在,因为它在前一个基因分型与332 SNP集合中呈现该SNP的A / T基因型。
获得完整的基因型(47 snP),得到990株植物,其平均值为每种各种66株植物,范围为54至86株植物(表2,表3.)不包括'Monastrell'。可以为65株植物建立任何基因型。这可能是由于许多情况下的低DNA浓度(17dNA低于4ng / UL的浓度),但在大多数情况下,故障可能是由于阻止扩增的污染物的存在。除了植物(一个SNP)和SNP(65株植物)可以进行基因分型的情况下,平均基因分型率为97.1%(表3.)。标记SNP697_296呈现最高的基因分型成功率,只有两种植物失败。十个SNP标记呈现出高于0.99以上的基因分型成功率,40 snp以上0.95。
关于稳定性分析,所有基因分型植物的99.4%显示了栽培品种的基因型。只有三种SNP在同一品种的植物中显示出不同的基因型:SNP1119_176和SNP581_114(在一个'ohanes'工厂中),SNP1347_100(在一个'火焰无籽'工厂)。为了确定这些变化是由于突变(缺乏稳定性)或基因分型误差,使用相同的DNA提取以及每种植物的独立DNA萃取来重复分析。结果表明,所有差异都相当于基因分型错误。总之,在第58251个为第15个品种的单个SNP基因型中没有发现突变,因此,SNP标记集可以被认为是高度稳定的。
评估基因识别目的的SNP设置
对于48个SNP集合,总共200种葡萄葡萄酒释放,包括在稳定性分析中研究的每种品种的样品。一些access导致相同的基因型,但这些结果始终同意预期;由于它们对应于同义的品种或运动(由自发体细胞突变产生的表型不同品种,并且后来通过切屑繁殖)。通过使用分子标记,运动不会与初始品种不同。这是几个运动证实:“砂浆亚塞琳”,一种无籽运动,没有与“砂轮Blanc”不同。在Pinot组中,'Pinot Blanc'显示了48个SNP设置为“Pinot Noir”,也是“Pinot Meunier”的相同基因型,遗传嵌合[25.[显示出相同的基因型。尽管如此,另一种颜色体育运动,“耻骨Gris”呈现了SNP1229_219的纯合学基因型CC,而该组的其他品种是杂合的CG。这并不奇怪,因为“Pinot”组具有用微卫星标记测量的最大的晶体内变化[26.-29.].
当将本研究中获得的基因型与稳定性分析中的相同品种的基因型(见上文)进行比较时,观察到另一种一位等位基因差异(如上见过),但在“彩色GRIS”的情况下,差异是一致的,并且可以被认为是一个基因突变,在后面的情况下,它们被证明是由于基因分型误差。SNP1119_176的5个品种中观察到差异(表2)。在所有情况下,将错误的纯合基因型(CC)分配给在稳定性分析中研究的植物中,而正确的杂合子(AC)。当板中的大多数样品具有相同的基因型时,这些SNP基因分型错误更频繁,因为对应于每SNP基因座的三种可能基因型的参考基因型云更难以建立。事实上,当使用来自其他板的样品重新分析一些错误的基因分型样品时,将它们分配了正确的杂合(AC)基因型。
建立了非冗余基因型样品,以评估与葡萄栽培品种SNP鉴别力相关的遗传参数。研究了200个牧群,49种基因型,对应于同义词品种,运动和野生植物,被丢弃。在含有151个非冗余品种的所得样品中(附加文件1,确定表S1),等位基因频率和几个遗传参数。MAF是标志物的辨别能力的衡量标记。在双位式标记的情况下,更近的MAF为0.5,更好。在该研究中,19个SNP显示出0.4和0.5之间的MAF,而只有三个SNP的MAF在0.1以下。非偏见的预期杂合性(HE)为0.404,范围为0.107(SNP1399_81)至0.501(SNP581_114,SNP829_281和VV10992)(表4.)。只有三个SNP显示在0.2以下的PIC值,其余包括0.2和0.4之间。这些值表明整个SNP组具有非常高的葡萄品种的辨别能力,并得到了非常低的身份概率(PI):1.4·10-17.这一数值远小于国际葡萄和葡萄酒组织(OIV)在分析57个独特的西班牙基因型(10-7[30.利用9个微卫星对164个欧洲品种(10个-9[31.]),或991种葡萄葡萄树(7·10-12,[23.])。相反,为48个SNP组获得的PI大于2,739个葡萄树的18微卫星获得的值(10-22,[21.]),或在745种中的34间微卫星(10-27年[32.])。这些代表性实例表明,在平均值的情况下,每个微卫星标记的身份的概率在0.06和0.16之间,而这里使用的SNP集合中的平均值为每标记0.445。因此,需要3-4个SNP基因座,以提供葡萄树中一个微卫星基因座的辨别力。相应地,48个SNP集合将提供与14-16微卫星相似的识别功率。
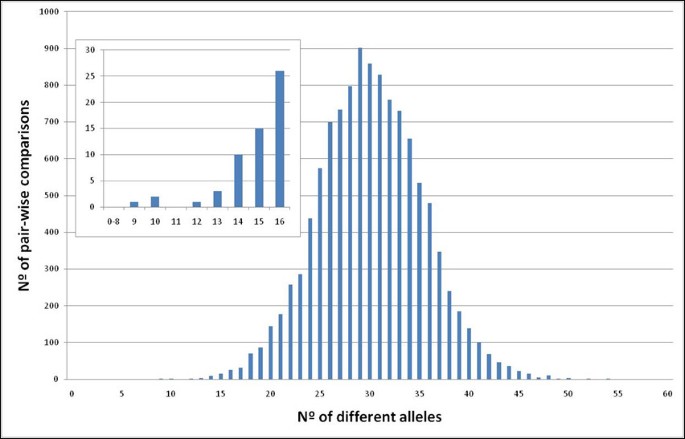
品种鉴定的任务往往与法律问题有关。最重要的是,在技术试验中,任何品种都必须克服在许多国家种植的授权,在这种试验中确定独特性是最重要的问题:如果一个品种能够与所有共同知识的品种明确区分开来,它就被认为是不同的(1991年《国际保护植物新品种联盟公约》;http://www.upov.org/en/publications/conventions/1991/act1991.htm.)。用于建立明显的关键概念是使用形态描述符在物种基础上以物种在物种上建立的最小距离。近年来,已经致力于掺入分子标记[23.].在本研究中,通过它们的成对比较确定具有非冗余基因型的品种的最小距离,并通过不同等位基因的数量测量(图2)。分析品种的平均差异为30个等位基因,总共96个,而最不同的样品在54等位基因中不同。最接近的品种是'Jaénnegra'和'Zalema',其中90个等位基因在90中不同,可以在它们之间比较。这两种品种具有基因型,这些基因型与父母/后代相容,基于微卫星[33.以及本研究中使用的SNP标记。其次发现的品种是“Ciruela Roja”和“Colgar Roja”,它们在研究的96个等位基因中有10个存在差异。这两个品种最近被描述为同一杂交的兄弟姐妹:'Ohanes' × 'Ragol' [34.].与“霞多丽”和“甜瓜”发生同样的情况,它匹配86等位基因,并且具有微卫星基因型,这与一对父母的后代相一致,'pinot'和'gouais blanc'[35.].因此,栽培甚至在基因上近距离研究,目前在各种等位基因的数量上存在大的测量差异。
从数据来看,在最高的品种内变异性(包括这里的体育)之间存在非常清晰的边界,其中1种不同的等位基因和9个不同等位基因的最低距离。因此,对于48个SNP集合建立2至9个等位基因之间的最小距离并不存在任何困难,并且对于在葡萄品种(不包括体育中的明显暗症中,它足够大。仍然需要更广泛的多样性研究来找到更可靠的最小距离,因为它可能在源自与当前表葡萄繁殖中使用的紧密相关的祖细胞的完整兄弟姐妹较短。
在几个先前描述的映射群体中已经确认了这48个SNP标记的孟德尔遗传遗传。此功能还允许对童群和父母/后代关系的遗传检查。使用所选的48个SNP组,找到了151种栽培品种的亲子的总排除概率很高(0.9997),但标记数量太小,无法进行可靠的血统分析。几个Trios获得的赔率(LOD)分数的对数为17至23,这不足以达到最终结论。
结论
一套48个单核苷酸多态性(SNP)已被选择,分布在整个葡萄基因组和测试的遗传鉴定目的。所选标记具有高度稳定性和可重复性,对葡萄品种具有较高的鉴别能力。SNP数据不需要任何等位基因的分类,并允许直接数据库和直接比较来自不同实验室的数据。所有这些特性使我们的标记非常适合建立一个全球公开的葡萄品种基因型数据库。
方法
植物材料和DNA提取
本研究使用了三种不同的品种样品套和四种分离种群。用于测定遗传参数的研究,研究了332个SNP标志物的300种载体样本,包括91个野生戒备以及葡萄酒和表格品种(附加档案1,使用表S1)。这些加入大多是在“Finca elencín”(Imidra,Alcaládehenares,马德里,西班牙)的种质收集。
SNP标记的染色体位置从遗传和生理两方面进行了测定。为了进行遗传测定,使用了IMIDA (Murcia, Spain)发展和维持的4个不同的分离群体:Dominga × Autumn Seedless [36.],Monastrell×Cabernet Sauvignon,Ruby无籽×Moscatuel和Muscat Hamburg×Sugraone。这些映射群体分别包括82,85,71和75个个体。
以15个品种为样本,对48个SNP位点进行了稳定性分析vitis Vinifera物种。在7个不同国家的154个不同样地收集了1277个品种的叶片材料(附加文件)1,表s2)。
在遗传识别方面的所选48个SNP的遗传多样性分析是在200名中进行的,其中大多数均可从Csiro Collection(Glen Osmond)的Imidra的葡萄品种集合中来自于IMIDRA的葡萄品种澳大利亚)(附加文件1,表S1)。
根据Lijavetzky等人的研究,从每个样品的冷冻幼叶中提取总DNA [37.]并储存在-20°C。
SNP鉴定和初始基因分型
如Lijaveztky等人所述接近SNP发现。[13.].SNP基因分型在Centro Nacional de Genotipado进行http://www.cygen.org.使用SnPlex™技术(应用生物系统[38.])。利用300份样本集上的7 48个SNP集研究了332 SNP的有用性。首次基因分型后,剔除基因分型成功率较低的SNP标记和单型SNP,其余标记根据其次要等位基因频率进行分类。
SNP位置的确定
SNP基因组位置是基于遗传和物理信息确定的。使用两阶段策略之后的四个映射群建立了遗传职位。首先,SNP标记定位在使用微卫星标记的每个交叉开发的共识框架地图上。根据Cabezas等人进行分子标记和连杆分析。2006 [36.]使用双向伪测试策略[39.,以及Joinmap 3.0软件[20.].在这种情况下,SNP标记只能在分离为aaxab、abxaa或abxab的后代中被定位。其次,以微卫星染色体作为锚定标记,并包含至少一个子代中分离到的所有SNP,构建了所有子代的完整SNP图谱。集成地图是使用Joinmap 3.0的“组合组用于地图集成”功能构建的[20.].用于重组频率的3.5的值和用于LOD的3作为初始映射阈值。对于具有显示不同连杆地图之间共同的含量的区域的染色体,分别移动到5.0和下降至0,允许地图集成。对于SNP显示在不同后代物理映射信息的联系地图中的重要差异和“固定顺序”功能[20.用来建立标记顺序。丢弃其包含在标记令中导致大扭曲的SNP被丢弃。在IGGP(国际葡萄基因组计划中,分配了染色体名称,http://www.vitaceae.org/index.php/建议。
SNP标记的物理位置通过BLAT搜索其在近纯合的PINOT线PN40024的12×Grapevine基因组序列上的相邻序列[40]和http://www.genosope.cns.fr/externe/genomebrowser/vitis/.在Pinot Noir基因组序列上还检查了遗传和物理位置之间重要差异的标记的位置http://genomics.research.iasma.it/gb2/gbrowse/grape/[41.].
48 SNP遗传鉴定的选择与评估
根据其基因分型成功率,MAF以及它们的遗传和物理位置,从先前显影的332中选择超过48个SNP标记。选择基因识别集合的最后一步是基于设计SnPlex™平台的Plex所需的技术要求。
所选48个SNP集合的稳定性试验的实验设计包括来自10个不同地块(平均)的85株植物的分析。在不同的年份和7个不同国家的不同年份种植(附加档案1,表s2)。由于葡萄葡萄酒是克隆,如果使用的标记是稳定的,则希望在每个植物中获得与其起源,年龄和地点相同各种的每个植物中每个SNP获得相同的等位基因。
通过200种样品评估了用于葡萄葡萄栽培品种鉴定的所选48 SNP鉴定的区分功率。
基因分型和遗传参数估计这些试验。对于每种SNP,在排除在扩增所有SNP的DNA样品之后,计算基因分型成功率。基于在不同分析中获得的结果计算基因分型误差:通过同一植物的基因分型不同DNA提取物;通过基因分型不同植物属于同一品种;或者通过研究特定基因型的已知运动,例如卡射家庭的那些。遗传参数估计了非冗余基因型。次要等位基因频率(MAF),观察到的杂合性(H.o),预期的杂合性(He)使用Identity 1.0工具计算身份(PI)的概率[42.]和Excel microAttellite工具包[43.].家系关系用Cervus 3.0软件分析[44.].在宫颈3.0中实施的父母后代TRIOS的整体似然比的自然对数(LOG到基础E)获得了LOD分数,如Cervus 3.0所实施的。[42.].
缩写
- cm:
-
厘摩
- Ho:
-
观察到的杂合性
- He:
-
预期的杂合性
- IGGP:
-
国际葡萄园基因组计划
- indel:
-
插入删除
- LOD:
-
对数的赔率
- MAF:
-
轻微的等位基因频率
- MB:
-
梅格萨斯
- NCBI:
-
国家生物技术信息中心
- OIV:
-
国际藤和葡萄酒组织
- PI:
-
身份的概率
- 照片:
-
多态性信息内容
- SNP:
-
单核苷酸多态性
- SSR:
-
简单的序列重复
- 苏娃:
-
保护新品种植物的国际联盟。
参考资料
- 1。
这个p,lacombe t,托马斯先生:历史起源和葡萄酒葡萄的遗传多样性。趋势类型。2006,22(9):511-519。10.1016 / J.tig.2006.07.008。
- 2。
Reger F,Hack R,Santiago JL:高度变量vitis.微卫星基因座识别Pinot Noir Clones。血管。2006,45(2):85-91。
- 3.
托马斯Mr,Cain P,Scott NS:DNA键入葡萄藤:用于描述品种和评估遗传相关性的通用方法和数据库。植物mol biol。1994年,25:939-949。10.1007 / bf00014668。
- 4。
宝沃斯耶,梅雷迪思CP:一种经典的酿酒葡萄,赤霞珠的血统。中国生物医学工程学报,1997,16(1):84-87。10.1038 / ng0597 - 84。
- 5。
托马斯先生,斯科特NS:微卫星在葡萄中重复在分析为序列标记的位点(STS)时揭示DNA多态性。Al Appl Genet。1993,86:985-990。
- 6。
这个P,Jung A,Boccacci P,Borrego J,Botta R,Costantini L,Creaspan M,Dangl GS,Eisenheld C,Ferreira-Monteiro F,Grando S,IbáezJ,Lacombe T,Laucou V,Magalhaes R,Meredith CP,Milani N,Peterlunger E,Reger F,Zulini L,Maul E:开发标准的微卫星参考等位基因,用于鉴定葡萄品种。Al Appl Genet。2004,109(7):1448-1458。10.1007 / s00122-004-1760-3。
- 7。
Cipriani G,Marazzo Mt,Di Gaspero G,Pfeiffer A,Morgante M,Testolin R:一组微卫星标记,长核重复优化葡萄(vitis.SPP。)基因分型 - 艺术。没有。127. BMC植物BIOL。2008,8:127-127。10.1186 / 1471-2229-8-127。
- 8。
Allen AR,Taylor M,McKeown B,Curry Ai,Lavery JF,Mitchell A,Hartshee D,Fries R,恐吓RA:北爱尔兰牛群牛身鉴定的信息单核苷酸多态性面板的汇编。BMC遗传学。2010,11。
- 9。
deleu w,esteras c,roig c,gonzalez-to m,fernandez-silva i,gonzalez-ibeas d,Blanca J,Aranda Ma,Arus P,Nuez F,Monforte AJ,Pico MB,Garcia-Mas J:一套甜瓜地图饱和度和品种鉴定的EST-SNP。BMC植物BIOL。2009,9。
- 10。
Ganal MW,Altmann T,Roder MS:植物植物中的SNP鉴定。植物生物学目前的意见。2009,12(2):211-217。10.1016 / J.PBI.2008.12.009。
- 11.
格洛弗卡,汉森MM,Lien S,Als Td,Hoyheim B,Skaala O:SNP和STR基因座的比较划定人口结构并进行个体遗传分配。BMC遗传学。2010,11。
- 12.
Hayden MJ,Tabone TL,Nguyen TM,Coventry S,Keiper FJ,Fox RL,Chalmers KJ,Mather de,Eglinton JK:澳大利亚大麦种质的分子表征的信息套装SNP标记集。作物与牧场科学。2010,61(1):70-83。10.1071 / cp09140。
- 13。
Lijavetzky D,Cabezas Ja,IbánñezA,罗德里格斯v,Martínez-Zapater JM:高吞吐SNP发现和葡萄园基因分型(vitis ViniferaL.)通过结合重新排序方法和SnPlex技术。BMC基因组学。2007,8:424-10.1186 / 1471-2164-8-424。
- 14。
Aradhya Mk,Dangl Gs,Prins Bh,Boursiquot JM,Walker Ma,Meredith CP,Simon CJ:栽培葡萄的遗传结构和差异化,vitis ViniferaL.遗传研究。2003,81(3):179-192。10.1017 / S0016672303006177
- 15.
Arroyo-Garcia R, Ruiz-Garcia L, L博林,Ocete R,洛佩兹,阿诺德C, Ergul, Soylemezoglu G, Uzun嗨,卡贝略港F, Ibanez说J, Aradhya可,Atanassov, Atanassov我Balint年代,Cenis杰,Costantini L, Goris-Lavets年代,Grando女士,克莱恩,麦戈文PE、Merdinoglu D, Pejic我Pelsy F, Primikirios N, Risovannaya V, Roubelakis-Angelakis KA, Snoussi H,Sotiri P, Tamhankar S, et al:栽培葡萄的多种来源(vitis ViniferaL. SSP Sativa)基于叶绿体DNA多态性。Mol Ecol。2006,15(12):3707-3714。10.1111 / J.1365-294x.2006.03049.x。
- 16。
Vezzulli S,Troggio M,Coppola G,Jermakow A,Cartwright D,Zharkikh A,Stefanini M,Grando Ms,Viola R,Adam-Blondon AF,Thomas M,此P,Velasco R:培养葡萄族的参考集成图(vitis ViniferaL.)从三个交叉口,基于283 SSR和基于501个SNP的标记。Al Appl Genet。2008,117(4):499-511。10.1007 / S00122-008-0794-3。
- 17。
Zhang JK,Hausmann L,Eibach R,Welter LJ,Topfer R,Zyprian EM:来自Grapevine V3125的框架地图(vitis Vinifera'Schiava Grossa'×'riesling')×砧木品种“送货”(涟漪Riparia.×血管灰质)本地化遗传决定簇的脑脂氧化根抗性。Al Appl Genet。2009,119(6):1039-1051。10.1007 / S00122-009-1107-1。
- 18。
Troggio M,Malacarne G,CopPola G,Segala C,Cartwright Da,Pindo M,Stefanini M,Mank R,Moroldo M,Morgante M,Grando Ms,Velasco R:葡萄牙胶葡萄酒的致密单核苷酸多态性遗传联系地图(vitis ViniferaL.)锚定皮孔Noir细菌人工染色体Contigs。遗传学。2007,176(4):2637-2650。10.1534 / genetics.106.067462。
- 19。
Lowe Km,Walker Ma:三角形葡萄砧木交叉Ramsey的遗传联系地图(血管蘑菇) ×格洛瓦河(涟漪Riparia.)。Al Appl Genet。2006,112(8):1582-1592。10.1007 / s00122-006-0264-8。
- 20。
van Ooijen JW,Voorres Re:joinmap®3.0,软件计算遗传性联动地图。Wagieningen:植物研究国际;2001年。
- 21。
Laucou V,Lacombe T,Dechesne F,Siret R,Bruno JP,Dessup M,Dessup T,Ortigosa P,Parra P,Roux C,Santoni S,Varèsd,PéroSJP,Boursiquot JM,这个P:葡萄的高吞吐量分析遗传多样性作为种质收集管理的工具。标记理论和应用的遗传学。2011,1-13。
- 22。
Myles S,Boyko AR,欧文斯Cl,Brown PJ,Grassi F,Aradhya Mk,Prins B,Reynolds A,Chia J-M,Ware D,BustAmante CD,Buckler ES:遗传结构和葡萄的驯化史。PROC NAT ACAD SCI USA。2011,108(9):3457-3458。10.1073 / pnas.1019729108。
- 23。
Ibáñez J, Vélez M, de Andrés MT, Borrego J:建立植物营养繁殖特性的分子标记:以葡萄藤为例。应用电子学报。2009,119(7):1213-1222。10.1007 / s00122 - 009 - 1122 - 2。
- 24。
Galet P:DictionnaireencyclopédiqueDscépages。巴黎:Hachette;2000年。
- 25。
Franks Tr,Botta R,Thomas Mr,Franks J:葡萄园的倒眠:对品种身份,祖先和遗传改进的影响。Al Appl Genet。2002,104(2-3):192-199。10.1007 / S001220100683。
- 26。
A:黑皮诺的遗传变异分析(vitis ViniferaL.)用AFLP标记的克隆。Am j enol vitic。2007,58:526-529。
- 27。
Konradi J,Blaich R,Forneck A:'Pinot Noir'的克隆和运动之间的遗传变异(vitis ViniferaL.)。欧洲园艺科学杂志。2007,72(6):275-279。
- 28。
Regener F,Stadlbauer A,Eisenheld C,Kaserer H:卡氏和相关品种之间的遗传关系。Am j enol vitic。2000,51(1):7-14。
- 29。
A:嵌合莫尼耶皮诺(Pinot Meunier)的克隆变异和稳定性分析vitis ViniferaL.)和下行运动。Euphytica。2009,165(1):197-209。10.1007 / s10681-008-9807-1。
- 30.
MartínJP,Borrego J,Cabello F,Ortiz JM:使用序列标记的微卫星位点标记的西班牙葡萄种类多样性的特征。基因组。2003,46:10-18。10.1139 / g02-098。
- 31。
Sefc KM, Lopes MS, Lefort F, Botta R, Roubelakis-Angelakis KA, Ibáñez J, Pejic I, Wagner HW, Glössl J, Steinkellner H:欧洲不同地区葡萄品种的微卫星变异和分配试验评估品种的地理来源。应用计算机学报。2000,100:498-505。10.1007 / s001220050065。
- 32。
Cipriani G,Spadotto A,Jurman I,Di Gaspero G,Creaspan M,Meneghetti S,Frare E,Vignani R,Cresti M,Morgante M,Pezzotti M,PE E,Policriti A,Testolin R:基于SSR的分子曲线1005葡萄园(vitis ViniferaL.)加入揭示了新的同义词和父母,并揭示了各种不同地理起源的巨大综合征。Al Appl Genet。2010,1-17。
- 33。
IbáñezJ,deAndrésMT,莫里诺A,Borrego J:使用微卫星分析的关键西班牙葡萄品种的遗传研究。Am j enol vitic。2003,54(1):22-30。
- 34。
Vargas Am,deAndrésmt,Borrego J,IbánezJ:五十张桌葡萄品种的百分点。Am j enol vitic。2009,60(4):525-532。
- 35。
Bowers Je,Boursiquot JM,这个P,Chu K,Johansson H,Meredith Cp:历史遗传学:霞多丽,野玩和其他葡萄酒葡萄的父母。科学。1999年,285:1562-1565。10.1126 / Science.285.5433.1562。
- 36。
Cabezas Ja,Cervera Mt,Ruiz-Garcia L,Carreno J,Martinez-Zapater JM:葡萄树中种子和浆果重量的遗传分析。基因组。2006,49(12):1572-1585。10.1139 / g06-122。
- 37。
Lijavetzky D,Ruiz-Garcia L,Cabezas Ja,De Andres Mt,Bravo G,IbánezA,CarreñoJ,Cabello F,IbáezJ,Martínez-Zapater JM:浆果颜色变异的分子遗传学曲折。分子遗传学和基因组学。2006,276(5):427-435。10.1007 / s00438-006-0149-1。
- 38。
De La Vega Fa,Lazaruk Kd,罗德MH:Wenz MH:两个灵活和兼容的SNP基因分型平台评估:TaqmanSNP基因分型测定和SnPlex(TM)基因分型系统。突变研究 - 诱变的基本和分子机制。2005,573(1-2):111-135。10.1016 / J.MRFMMM2005.01.008。
- 39。
Gratepaglia D,Sederoff R:遗传联系地图桉树 - 祖母和桉树 - 尿道使用伪testcross - 映射策略和RAPD标记。遗传学。1994,137(4):1121-1137。
- 40。
jaillon o,yury jm,noel b,policriti a,clepet c,casagrande a,choisne n,aubourg s,vitulo n,jubin c,vezzi a,leadeai f,hugueney p,dasilva c,Horner d,云母E,jublot d那Poulain J, Bruyere C, Billault A, Segurens B, Gouyvenoux M, Ugarte E, Cattonaro F, Anthouard V, Vico V, Del Fabbro C, Alaux M, Di Gaspero G, Dumas V, et al: The grapevine genome sequence suggests ancestral hexaploidization in major angiosperm phyla. Nature. 2007, 449 (7161): 463-467. 10.1038/nature06148.
- 41。
Velasco R, Zharkikh Troggio M,卡特赖特哒,Cestaro, Pruss D, Pindo M,菲茨杰拉德LM, Vezzulli年代,里德J, Malacarne G, Iliev D,科波拉G,沃代尔B,米凯莱蒂D, Macalma T, Facci M,米切尔JT, Perazzolli M·G,与P Oyzerski R, Moretto M, Gutin N, Stefanini M,陈Y, Segala C, C达文波特,Dematte L,穆拉兹,等:一个杂合子葡萄品种基因组的高质量共识草案序列。PLoS ONE。acta photonica sinica, 2007,2 (12): e1326-10.1371/journal. 0001326。
- 42。
Wagner HW,SEFC km:身份。维也纳; 1999年。
- 43。
Park SDE:西非养牛的锥体作用以及选择的人口生效。都柏林:都柏林大学;2001年。
- 44。
Kalinowski ST,Taper Ml,Marshall Tc:修改计算机程序颈椎如何适应基因分型错误,提高了父级分配的成功。Mol Ecol。2007,16(5):1099-1106。10.1111 / J.1365-294X.2007.03089.x。
致谢
本研究在经济上支持Grapegen和14322年协议项目从Genoma西班牙以及vin01 de Investigacion y - 025项目从西班牙Tecnologia Agraria y Alimentaria从MICINN(西班牙科学和创新部)和部分CSIRO植物产业和葡萄和葡萄酒的研究发展公司(GWRDC)。我们还感谢MICINN与阿根廷(AR2009-0021)、应用生物系统公司(Applied Biosystems)在48 SNPlex和Centro Nacional de Genotipado的设计中提供的双边合作资助http://www.cygen.org.用于SnPlex基因分型。研究小组参加了成本行动FA1003。我们非常感谢西班牙国家葡萄种种质收集,以“El··伊志安”,Madrid,Madrid,为其植物材料。我们还感谢Enrique Ritter和MónicaHernández(内太科,西班牙)与我们分享他们的数据,他们的数据在后代MNXCS和JoséAntonioChanín(GenaomaEspaña)进行稿件修订。M.D.Vélez由博士学位的博士学位资助来自MadrileñodedintorigaciónyFesarrolloRural,AgraRio Y Alimentario(Imidra)。由于西班牙和国外的众多人和公共机构的合作,这项研究中所需的大型抽样是可能的。我们感谢所有人。具体而言,由于众多监管机构的葡萄酒起源委员会的合作,获得了来自葡萄酒品种的植物材料:Almansa,Calatayud,Campo de Borja,Cariñena,塔拉戈纳,Condado de Huelva,Costers del Segre,Jerez,Jumilla,La Mancha,Méntrida,Monterrei,Montilla-Moriles,Navarra,Penedés,Ribeira Sacra,Ribeiro,Ribera de Duero,Ribera del Guadiana,Rueda,Rioja,Málaga,Utiel-Requena,Valdeorras,Valdepeñas,瓦伦西亚,Vinos de Madrid和Yecla。个人捐赠对抽样的人中有:JoséMarícorado(SupportFrutícolas.a.,穆尔西亚,西班牙);编辑Hajdu(FVMSzölészetiésBorászatiKutatóntézete,匈牙利); Patricio Hinrichsen (Centro experimental La Platina-INIA, Chile); Tim Sheehan (Sheehan Genetics, USA); Jean Satterwhite (National Clonal Germoplasm Repository for Nut Crops, USA); Erika Maul (Institute for Grapevine Breeding, Germany); Jorge Zerolo (Agrovolcán, Tenerife, Spain); Nuria Cid (Estación de Viticultura y Enología de Galicia, Orense, Spain); Peter Allderman (Top Fruit, RSA); Thierry Lacombe (DGPC-Diversité et Génomes des Plantes Cultivées, France); Miguel Lara (CIFA-Centro de Investigación y Formación Agraria, Jerez de la Frontera, Spain); Joaquín Borrego, Paz Fernández, Maite de Andrés, Carlos González, Alba Vargas, Gregorio Muñoz, Cristina Rubio and Mariano Cabellos (IMIDRA, Spain). We apologize for any non-deliberate omission in this list.
作者信息
隶属关系
通讯作者
附加信息
作者的贡献
JAC进行了SNP的物理和遗传映射,参与了SNP选择和起草的手稿。JI开展了稳定性和遗传多样性分析和起草了一部分稿件。DL在选择SNP选择和表征方面发挥作用。MDV参与了稳定性分析。GB,VR,IC和LRG有助于品种和产后的基因分型。AMJ参加了SNP选择。JC产生并维持了大部分后代。MRT协助SNP选择并帮助起草了稿件。JMZ构思了这项研究,在其设计和协调中得到了努力,并帮助起草了稿件。所有作者均阅读并批准最终手稿。
JoséABezas,javierIbáñez同样为这项工作进行了贡献。
电子补充材料
12870_2011_935_moesm1_esm.xls.
附加文件1:补充表S1至S5。表S1:分析了植物样品。表S2:用于48 SNP套件的稳定性研究的植物样品。表S3:分析了238个SNP的基本信息。表S4:遗传贴图功能。表S5:至少一个祖细胞中的杂合标记物的后代数量。(XLS 212 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
卡维萨斯,j.a., Ibáñez, J, Lijavetzky, D。等等。一套48个SNP,用于葡萄栽培品种鉴定。BMC植物BIOL.11,153(2011)。https://doi.org/10.1186/1471-2229-11-153
收到:
接受:
发表:
关键词
- 简单的序列重复标记
- 单核苷酸多态性
- 多态性信息内容
- 标记订单
- 基因识别