抽象的
背景
参与高度复杂和协调系统的植物已经进化出相当程度的发育可塑性,从而最大限度地减少了胁迫造成的损害。MicroRNAs(miRNAs)最近已成为植物基因调控、发育过程和胁迫耐受性的关键调节器。
结果
在本研究中,结合深度测序技术和深度生物信息学分析,鉴定和分析了与干旱、盐和碱度胁迫响应相关的大豆mirna。代表95个miRNA家族的133个保守miRNA在3个处理下的大豆中表达。此外,71、50和45个mirna在干旱、盐度和碱度下分别有独特或不同的表达,这表明许多mirna是可诱导的,并在响应某些胁迫时有差异表达。
结论
我们的研究对进一步鉴定非生物胁迫下的基因调控具有重要意义,并显著有助于完整的miRNAs图谱大豆.
背景
陆生植物面临严重的非生物胁迫(如干旱、盐度、碱性、寒冷、病原体反应和疾病),这些是作物产量下降的主要原因[1].作为世界主要的油料作物之一,大豆面对环境压力所带来的挑战。为了应对环境压力,农作物进化出了复杂的适应反应机制[2].因此,解开大豆的复杂抗性机制将为环境刺激所涉及的生物过程提供基本的见解,这可能会有助于减轻作物损失。
越来越多的证据表明,长度约为21个核苷酸的microRNAs(miRNAs)在植物的基因调控、发育过程和抗逆性中起着关键作用[3.- - - - - -5].mirna的功能是通过切割它们的目标(mrna主要通过RISC)或抑制蛋白质翻译[6,7].事实上,已经有人提出,参与应激反应的许多mirna已经适应了环境挑战。例如,Phillips等人[8]报道miR395、miR397b和miR402参与应激反应。miR393的表达水平在盐和碱胁迫下发生变化,但过表达miR393对植物有害[9].响应于环境应力,MIRNA表达的波动可以通过许多不受控制的因素,例如转录和转录后水平的干旱,盐度和碱度诱导。据报道,硫酸盐饥饿导致miRNA395的上调[7miR398和miR408对水分缺乏反应[10].此外,这些诱导mirna在不同胁迫下表现出不同的特异性。然而,我们对逆境条件下mirna在植物中的作用的了解仍然有限,特别是在全基因组水平上。
近年来,可以通过生物信息学或测序来识别miRNA。例如,各种方法已被用于鉴定米饭,小麦和玉米中的miRNA [11- - - - - -13].许多生物信息学方法和技术已经发展为快速和准确的miRNA检测和分析。最近,深度测序技术显示了在单碱基对分辨率下小RNA发现和全基因组转录组分析的重大前景[14].与微阵列相比,深度测序有几个优势,主要是它在全面鉴定和分析以前未知的小RNA群体方面的应用。深度测序已经在不同的植物、突变体和不同发育阶段的组织中发现了许多小rna [15- - - - - -18].在本研究中,通过高通量测序,鉴定并分析了与应激反应相关的大豆mirna。在3种胁迫处理下,大豆中检测到95个miRNA家族对应的133个已知miRNA。此外,有71、50和45个mirna在干旱、盐度和碱度下分别有差异表达,表明许多mirna是可诱导的,并且在不同的环境胁迫下有差异表达。
结果
不同处理下小RNA转录组的一般特征
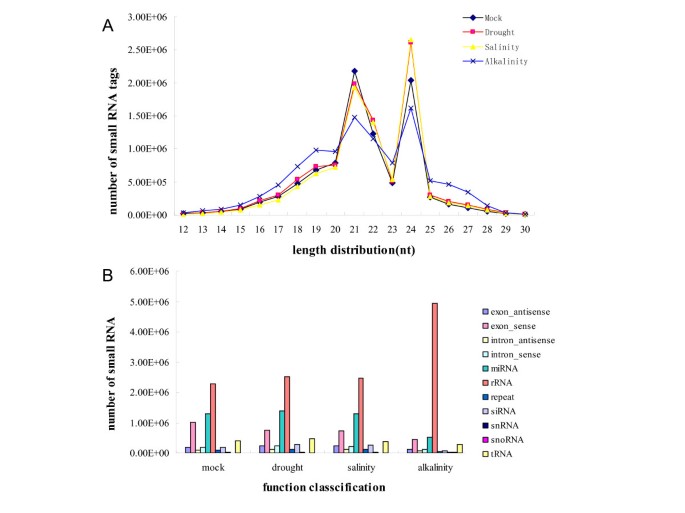
小RNA不仅可以调节一系列复杂的发育事件,还可以调节非生物胁迫下的防御[19,20.].为了探索大豆(模拟、干旱、盐度和碱度)三种胁迫处理的小RNA库,Solexa (Illumina)生成了RNA库并进行了测序。每个文库中大约有900万至1000万原始读取,产生了3600多万个原始测序标签。在丢弃低质量、过滤5'污染物和修剪3'适配器读取后,从模拟、干旱、盐度和碱度处理的数据集分别获得8,500,978、9,357,545、9,003,582和9,223,744个干净读取(参见附加文件1).虽然四个RNA文库的序列reads总数相似,但序列标签的大小分布却有很大的不同(图)1额外的文件2).例如,2 182 055(23.72%的mock clean reads)序列是典型的21 nt小rna,在mock样本的根中小rna最丰富。3个压力库21 nt的净阅读量分别为1 982 765、1 929 505和1 476 829次,分别占干旱净阅读量的19.64%、盐度净阅读量的20.22%和碱性净阅读量的14.33%。小rna在长度和冗余度上差异很大,其中24 nt reads在盐度诱导文库中冗余度最高(27.78%)。24个nt reads在干旱和模拟库中分别占25.90%和22.14%,而在碱度诱导库中仅占15.69%。24个nt reads的相对较低的百分比表明更多种类的mirna参与了反应g·马克斯与其他应力条件下的碱度相比。这些数据突出了不同胁迫条件下小RNA库的总体复杂性。
生成一组参考注释对于探索小RNA类别是非常必要的。每个库中所有相同的Solexa读被分类为唯一的序列标签,以便进一步分析。对齐后,将读取所有序列大豆使用SOAP2的基因组[21],大约70%的读数完美匹配,30%来自未解释的基因组位点,一个错配。例如,在模拟中,7,045,434(75.4%)清洁读入1,609,063个独特的读数与1 115 MB基因组匹配大豆.随后,对于每个库,约60%的清洁小RNA被鉴定为从RRNA,TRNA,SNRNA或其他非编码RNA处理的产品(图1 b).另一部分(约40%)主要来自未注释或重复序列。大部分注释的小rna以非编码rna为主。对于mock组,1 289 824个干净序列,分为1474个独特标签,被认为是潜在的mirna。干旱和盐碱诱导的其他两组分别为13393901(1512个独特标签)和13302431(1503个独特标签)。值得注意的是,在碱性诱导组中,513021个筛选reads(1062个unique tags)为miRNA候选,占前三组miRNA的近一半。据估计,已知的mirna可能是植物防御中转录后水平调控的最丰富的一类小rna。
大豆中已知的mirna
在以前的研究中已经报道了许多大豆的mirna。Kulcheski等人[22]从干旱敏感和耐受幼苗和锈蚀和抗性和抗性大豆中检测到256米,其中尚未报告24个MiRNA。Song等人。[15通过深度测序确定了26种新的miRNA开发大豆种子。Joshi [23]通过测序和生物信息学分析,鉴定出129个mirna,其中42个与大豆或其他物种的已知mirna匹配,87个是新的mirna。在另一项研究中,Chen等人[24,在野生大豆幼苗中报道了15个保守miRNA候选基因,分别属于8个不同的家族和9个新miRNA候选基因,分别属于8个不同的家族。为了鉴定大豆中四种不同处理的已知mirna,将小RNA序列与miRBase 16.0进行比较。经过序列相似性搜索,在大豆中鉴定出与95个miRNA家族对应的133个已知miRNA(附加文件)3.).此外,四种保守的星型mirna (miR156d*、miR157b*、miR162*和miR3630*)也被测序。其中,miR156d*、miR157b*、miR3630*星序列表达较低。然而,在不同处理下,miR162*的丰度从125到220 reads不等。另外,其他星星mirna在四种条件下表达量均较低,分别是miR172b*、miR156h*和miR166g*。其他研究表明,在整个植物中,mirna通常在进化上是保守的[25,26].因此,我们通过比较大豆中所鉴定的mirna的进化保守特性拟南芥,大米,玉米,Medicago truncatula,高粱二色的,小麦,葡萄,芸苔属植物和松果体根据它们的序列相似性(数据未显示)。所鉴定的miRNA家族在多种植物中都是保守的。文献报道了110个miRNA基因大豆,其他23个基因是从已知的同源mirna中检测到的。
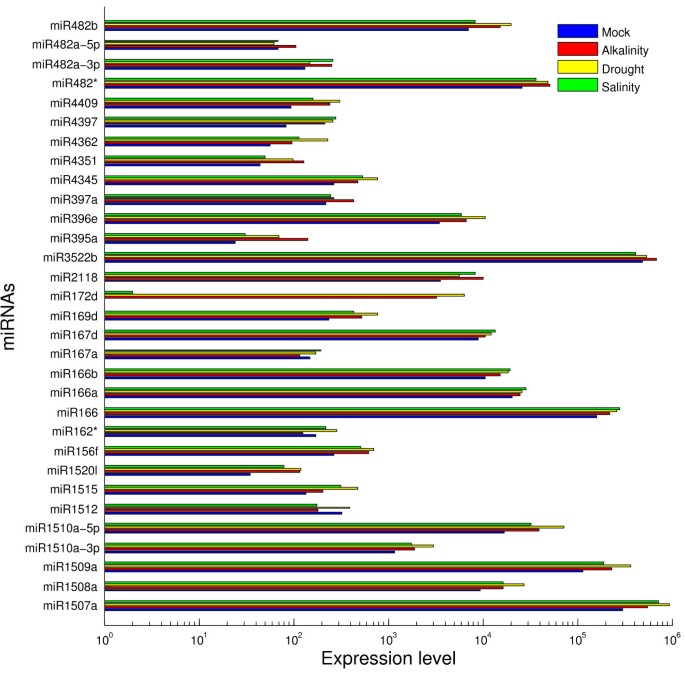
我们以四个文库中mirna的测序频率作为评估133个mirna相对丰度的指标。miRNA的分布频率变化很大,说明这些miRNA在每个文库中都有广泛的表达。3个丰富的miRNA reads (miR166、miR1507和miR3522)平均占表达miRNA标签的79.47%(图1)2额外的文件4和附加文件5).在我们的研究中,所鉴定的miRNA家族在多种植物中都是保守的。例如,miR156、miR1507和miR3522的家族分别在10个、3个和1个物种中被广泛保存(见补充文件)6).在其他植物种类中也检测到大豆中鉴定的大多数成熟miRNA,例如拟南芥[27],葡萄藤[28和白杨树[29].特别是高丰度的miR156、miR166和miR167。其中,miR166数量最多(干旱条件下的序列读值为263 470倍)。以往的研究表明,高表达水平的mirna总是与进化保守相关[25,30.].在本研究中,大多数发生在低频率,读标签不超过100个的mirna,如miR408和miR1517,保守性较差。而序列reads最少的mirna,包括miR169g、miR171b和miR393b,测序了几十次,但分别在9种、17种和8种植物中被保守(图)3.).在mock中表达的MiR171b和在干旱中表达的miR393b分别测序21次和0次。这些结果表明,保守的microrna可能在控制植物的基本细胞和发育途径(如细胞周期)中起重要作用。
为了通过深度测序验证mirna的表达模式,我们随机选取10个mirna (miR156f、miR167d、miR169d、miR393a、miR394a、miR482、miR1507a、miR1508b、miR4369、miR4397)进行qRT-PCR验证。比较了三种胁迫(干旱、盐度和碱度)诱导样品的表达丰度模式。在三种胁迫诱导条件下,mirna的上调频率最高的分别是miR167d、miR169d、miR482、miR1507a、miR1508b和miR4369。只有miR393a与Solexa的结果不一致。MiR394a在两种方法中均下调,且表现出相同的模式。两种方法的结果高度一致,表明qRT-PCR验证表明两种方法具有良好的一致性(图)3.).
大豆中的新型mirna
从4个小RNA库中筛选出102个miRNA作为大豆miRNA候选基因。为了支持新mirna的存在,我们利用它们的发夹结构和自由能来评估这些候选mirna。我们鉴定了50个新的mirna,其中10个高表达的候选mirna列于表中1,其他则在附加文件中7.这些mirna的能量范围从70.8千卡/mol (gamma -050)到-24.2千卡/mol (gamma -013)。这些候选基因的表达水平范围广泛,从数千个序列计数到单个序列计数。大多数成熟序列都是由dicer样酶介导的5'和3'步进环结构产物。在发夹的3′和5′端均发现了新的mirna,包括Gma-m0004、Gma-m008、Gma-m009、Gma-m011和Gma-m030。与3'标签相比,5'读标签显示很小的读计数。Gma-m045, Gma-m046, Gma-m030和Gma-m050显示了来自miRNA前体的两条臂的序列读取几乎相同的数量。包括Gma-m006在内的11个miRNA,来自5'臂的序列阅读数高于包含3'臂的注释成熟miRNA,这表明DCL处理的大多数miRNA基因在植物中具有链偏向性。
与这些保守的miRNA相比,所有的新miRNA标签在四个文库中的读计数都很低,最高的5'端只有4 830个(Gma-001)。最小的在3'和5'末端只有一个(例如Gma-011, Gma-023, Gma-025, Gma-026, Gma-037, Gma-039, Gma-040, Gma-047),平均读计数为318。众所周知,保守mirna的表达频率高且普遍存在,而非保守mirna则不然。需要进一步的实验来确定这些新的mirna是否是应激诱导的。
干旱、盐度和碱度下的mirna表达模式
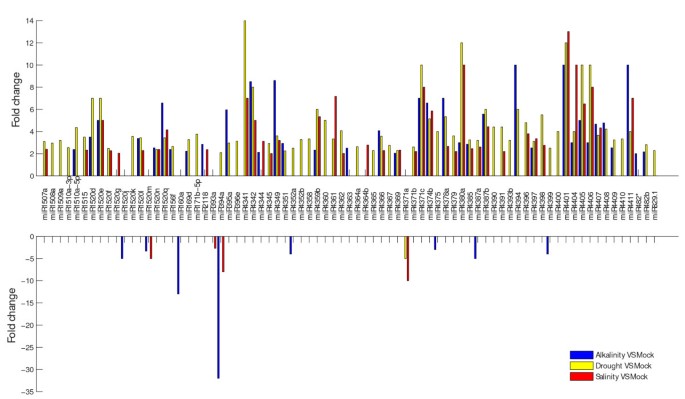
为了深入了解大豆对环境的适应,我们研究了干旱、盐度和碱度条件下常见和独特的miRNA表达模式。如图所示4, miRNA的表达随胁迫诱导条件的不同而不同。这些基因被鉴定为抗胁迫的功能调控因子。观察到的miRNA表达谱显示,一小部分miRNA (miR434a, miR157b*和miR171a)表现出应激特异性的表达模式。此外,这3种mirna的表达丰度均较低。大量的mirna在两到三种胁迫条件下表达。例如,miR156d*、miR160a、miR394a、miR1520j、miR4341、miR4387a、miR4399、miR1520c、miR1520r出现在三种胁迫条件下,miR169g、miR1517、miR3630*出现在两种胁迫条件下。因此,有的miRNA表达中间数(如miR160a、miR394a),有的miRNA只有几个reads(如miR-156d*、miR169g、miR393b)。
绝大多数差异表达mirna在三种胁迫条件下或两种胁迫条件下均表现出不同的表达模式。其中,78个miRNAs的表达有显著差异(fold change > 2;p < 0.05)(图4),它们在三种应激条件下均得到一致或差异的调节。在三种胁迫条件下,有27个mirna(如miR1520d、miR1520n和miR4407)的表达水平均高于mock。例如,miR4407的表达水平在干旱、盐度和碱度下分别变化了3.67、4.33和4.67倍。51种mirna在不同的诱导条件下(miR394a、miR4361、miR4396、miR4308)表现出不同的表达趋势,说明在不同的胁迫条件下,个体mirna可能具有不同的表达模式。例如,miR394a在干旱条件下表达上调(倍变化= 2.09),在盐度条件下表达下调(倍变化= -8)。在不同条件下,干旱、盐度和碱性条件下,分别有70、46和37个mirna上调,> 2(如miR169d)倍数发生变化,1、4和8个mirna下调,> -2(如miR393a)倍数发生变化。表达谱强烈表明,不同的miRNA调控模式可能完全或部分解释了不同处理之间的胁迫调控。
microrna的目标预测
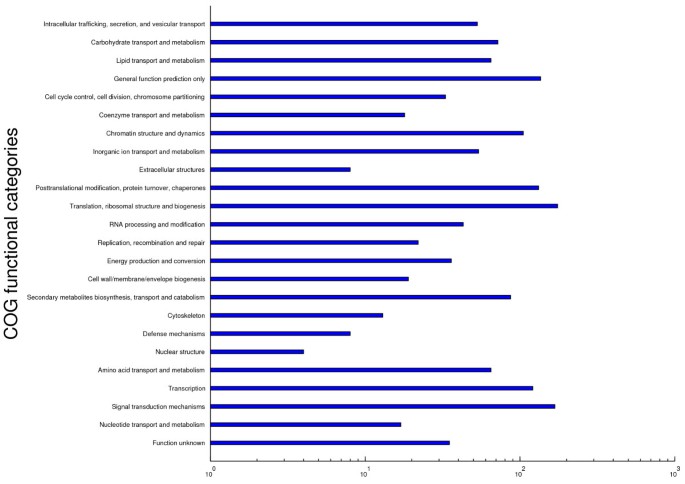
研究mirna的目标mrna可以帮助我们理解它们的生物学作用[31,32].在之前的研究中,Katara等人[33],预测了数据库中69个成熟miRNA序列中44个的573个目标。对受影响蛋白质的研究表明,更多的目标蛋白质产物参与多种生理过程,例如光合作用[34].Joshi [23]用计算方法预测了129个鉴定出的mirna的推定靶基因,并使用5' RACE方法验证了这些靶点子集的体内预测的切割位点。此外,作者还通过与Solexa cDNA测序数据的比较,研究了miRNA丰度与各自靶基因丰度的关系。在Song等人的研究中[15], 145个被鉴定为38个已知mirna和8个新mirna和25个基因的靶标。GO分析表明,许多鉴定的miRNA靶点可能在大豆种子发育中起作用。为了了解大豆中4种处理中鉴定的miRNA与之前发表的mRNA之间的关系,我们利用psRNATarget程序预测miRNA的mRNA靶点。1 219个mrna被预测为126个miRNAs的靶标(附加文件)8额外的文件9)最后,989个基因被分为24种类型,用COG注释(图5).大多数mrna的功能是翻译、核糖体结构和生物发生以及信号转导机制。此外,在核苷酸的运输和代谢、转录、防御机制等方面涉及多种生物学功能,这将为mirna对不同耐受性的调节作用提供有用的信息。这些结果表明,大部分靶标都属于转录调控的范畴,说明这些靶标编码转录因子(如miR169d靶标:CBF-B/NF-Y转录因子)。一些mirna,如gamma - mir156f和gamma - mir172d,具有多个靶位点,表明这些mirna在功能上是分散的。此外,单个基因可能被多个mirna靶向,比如多酚氧化酶,它是由gamma - mir157b和gamma - mir3522b调控的。
northern blotting检测成熟miRNA
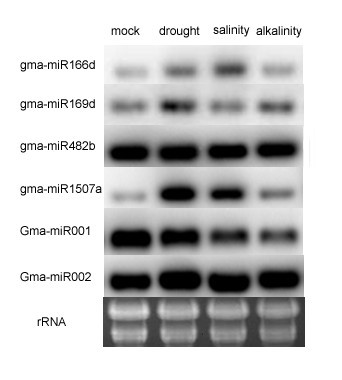
为了证实和验证从Solexa文库获得的结果,我们检测了4个已知mirna和2个新mirna的表达模式。这6个mirna (miR166b, miR169d, miR482b, miR1507a, Gma-m001和Gma-m002)分别被单独选择并通过northern blotting杂交进行实验验证。反义RNA探针的序列列在附加文件中10.将Solexa测序的miRNA与northern杂交的结果进行比较,鉴定出3个胁迫应答miRNA (miR166b、miR169d和miR1507a)具有相同的表达模式。MiR166b和MiR1507a在干旱、盐度和碱性条件下上调。MiR169d在干旱和碱性条件下表达上调(图)6).而通过northern blotting检测,miR482b和Gma-m002的表达模式在三种胁迫条件下均保持不变。然而,根据Solexa的结果,这些基因在干旱胁迫下上调。根据northern blot分析,与Solexa测序相比,Gma-m001在盐胁迫下表达量下降,但在干旱和碱度下的表达量与Solexa测序的表达量相同(图)6).因此,RNA印迹分析得到的表达模式可能反映了深度测序的结果。
讨论
如今,MiRNA在植物应激反应中的重要作用的表征是一个活跃的研究领域。虽然许多研究表明,植物miRNA在发育和形态发生过程中作为重要调节剂,但更多的报告表明植物MiRNA也参与了环境压力耐受性[7].
由于非生物胁迫是世界范围内作物损失的主要原因之一,揭示植物抗逆的复杂机制具有深远的意义。最近,新开发的测序技术,如Illumina基因组分析仪(GA)与传统方法相比,罗氏/454 FLX系统和ABI-SOLiD系统在提高吞吐量和显著降低成本方面取得了进步。目前,高通量测序技术的应用引起了广泛的研究兴趣,如全套miRNA的鉴定,这为miRNA的作用提供了新的见解通过这种方法,许多大豆miRNA已经得到了很好的注释[34].与微阵列不同的是,高通量测序使我们能够全面调查与应激相关的mirna。迄今为止,人们对mirna在非生物胁迫反应中的作用知之甚少大豆.
在本研究中,我们在深度测序的基础上,对三种处理下的大豆小RNA进行了测序和分析。对小RNA的研究表明,gma-miR1507a(936627个序列标签)从下一代测序数据中获得了133个已知的miRNA和50个新的miRNA。通过在干旱、盐度和碱性胁迫条件下表达丰富的miRNA库,发现许多miRNA在库和库之间具有广泛的表达水平这种miRNA表达变异的特征可能是由于miRNA的成熟加工[35和/或与压力相关的调节[2,36].我们推测这些mirna可能具有功能意义,表明它们可能参与了植物的胁迫反应。高度丰富的mirna似乎也表现出类似的保守。例如,miR2188和miR3522b在所有四个文库中都表现出高表达水平,并在许多物种中保持保守。这些观察结果支持了之前的结果,即最丰富的mirna是系统遗传保守的[37].
mirna和星型mirna都是由step-loop发夹结构生成的。mirna是稳定的,通过结合或锚定在mRNA序列的编码区参与mRNA的翻译抑制或切割[4].Khvorova等人[38从比例释放到预-miRNA茎时,这些链通常被破坏的明星miRNA的显着低丰度。恒星miRNA序列的低表达水平,例如miR156D *,miR157b *和miR3630 *,进一步支持miRNA合成假设。下一代测序是检测miRNA和星形miRNA的强大工具[[15,39]和[40]]。星型miRNA与其灵活表达的相关性可能揭示其特殊的调控功能。MiR162*和miR482*可能参与调控应激。单个发夹的两臂可能与不同的生物学活性有关,导致不同序列的RNA功能分离。在我们的研究中注释的小的新mirna,如Gma-004和Gma006的5'和3',都是从预测的发夹结构中衍生出来的。
据报道,植物mirna具有强烈的倾向调节对非生物胁迫的反应,包括脱水、冷冻、盐度、碱度和转录因子或蛋白质的其他胁迫[7].环境胁迫诱导的mirna表达水平不同。因此,它们可能在靶向应激调控基因方面发挥关键作用。有报道称,应激反应mirna普遍存在于杨树[41],大豆[22和其他植物。先前的研究报道,miR167、miR319和miR393的成员在应激耐受性中受到类似的调控[9,42,43].在本研究中,miR1520n、miR4374b和miR4396的成员在三种胁迫下同时上调,这表明它们可能针对的是逆境耐受负调控基因。此外,此前有报道称,在硫酸盐饥饿条件下,靶向硫酰化酶和ASP1基因的盐诱导大豆品系中miR395的表达上调。因此,我们推测miR395可能参与了非特异性盐诱导应答途径,如维持能量供应[7,13].此外,miR166在大麦中对脱水胁迫有响应[44],在脱水条件下大豆幼苗中含量丰富且上调。MiR393a靶向F-box蛋白和一种碱性螺旋-环-螺旋家族蛋白,在冷、脱水、盐或ABA胁迫下上调[7],并在碱性胁迫下下调。这些响应性mirna参与应激反应过程中的转录后调控。
小RNA转录组的深度测序产生了令人难以置信的大量数据,从这些数据中,我们不仅可以确定已知的mirna,还可以成功地以高精度和高效率探索新的mirna。首先,在这项研究中,我们鉴定了133个已知的和50个新的miRNAs大豆,说明了miRNA表达的多样性大豆,揭示了比之前所知的更多的mirna的存在。此外,深度测序技术结合生物信息学分析,使我们能够深入了解miRNA的表达模式,进一步了解miRNA的功能,阐明其潜在的分子机制和多种生理途径。其次,比较不同诱导条件下的miRNA表达谱,发现miRNA的调控模式存在显著差异,在干旱、盐度和碱性条件下,分别有71、50和45个表达模式发生改变。本研究获得的差异表达mirna可作为进一步鉴定耐胁迫作用的基础大豆.
结论
在本研究中,结合深度测序技术和深度生物信息学分析,鉴定和分析了与干旱、盐和碱度胁迫响应相关的大豆mirna。代表95个miRNA家族的133个保守miRNA在3个处理下的大豆中表达。此外,71、50和45个mirna在干旱、盐度和碱度下分别有独特或不同的表达,这表明许多mirna是可诱导的,并在响应某些胁迫时有差异表达。本研究对进一步鉴定非生物胁迫下的基因调节并显着贡献了米兰斯的完整概况大豆.
材料和方法
样本收集和处理
本研究以非生物胁迫敏感大豆之一的HJ-1自交系为材料。每个自交系用乙醇处理均匀种子10分钟,然后用无菌蒸馏水冲洗多次。这些种子在1 × Hoagland的营养液(4 ml/L隔离铁,6 mM K+和4 mM Ca2+)中培养。当达到4个叶期时,我们开始对其进行盐(120 mM NaCl)、碱度(70 mM NaCl和50 mM NaHCO)胁迫处理3.)干旱(2%PEG)胁迫48小时,以未受胁迫的植株为模拟对象,然后收集120株幼苗的根系,并在液氮中冷冻备用。
小RNA测序文库构建
分离的RNA样品在15%的PAGE凝胶上纯化,用于大小选择。使用T4 RNA连接酶,将小于30个碱基的小RNA与一对Solexa测序适配器引物(5'-PucGuugccGuugcUgidt-3'和5'-GuucagagUuCuacaguCgacAuc-3')连接。将连接的RNA在10%琼脂糖凝胶上进行大小分级,并将70-90个nt级分扩增15个周期,以将RNA转化为cDNA以产生测序文库。根据制造商的说明,使用Solexa sequencer(美国加利福尼亚州圣地亚哥Illumina)将含有约20 mg小RNA的纯化文库用于簇生成和测序分析。所有短读物均存放在国家生物技术信息中心(NCBI),可在短读物档案(SRA)中访问,登录号为SRA045367.1。
生物信息学分析
Solexa测序后,通过过滤低质量标签和消除适配器序列污染,从原始reads中提取高质量的小RNA reads。得到的具有相关读计数的唯一序列集视为干净序列标记。然后使用SOAP 2.0程序对Rfam数据库中的非编码rna (rRNA, tRNA, snRNA,和snoRNA)进行匹配序列查询http://soap.genomics.org.cn/.从进一步的分析中排除了对这些序列的任何小RNA读取匹配。接下来,我们将所有序列与MiRBase16.0对齐http://mirbase.org/使用SOAP 2.0搜索允许不匹配的已知mirna(或> 90%身份)。为了比较四种不同处理下的miRNA表达数据,首先,将每个鉴定的miRNA读计数归一化到每个给定样本的读总数。然后,采用贝叶斯方法评价统计显著性(P值)。经过贝叶斯检验,如果P值≤0.01,且归一化序列计数变化超过2倍,则认为特异性miRNA表达差异。
与上述任何数据库不匹配的读数被标记为未解式。为了鉴定新的miRNA预测,过滤匹配miRBase和RFAM的小RNA标签,并与剩余的标签与剩余的标签对齐大豆基因组。为了分析匹配序列是否能形成合适的发夹(小RNA前体的二级结构),提取匹配序列周围的序列。第二个结构由RNAfold预测http://rna.tbi.univie.ac.at/cgi-bin/RNAfold.cgi.随后,利用华大基因(北京基因组研究所)开发的MIREAP程序鉴定了新的mirna。http://sourceforge.net/projects/mireap/)和mirTools [45].使用psRNATarget预测miRNA候选靶点http://bioinfo3.noble.org/psRNATarget/.通过与NCBI的COG进行比较,注释了目的基因的COG (Clusters of Orthologous Groups)项http://www.ncbi.nlm.nih.gov/COG.
real-time PCR MiRNA定量
用总RNA (1 μl)用One Step PrimeScript合成逆转录本®miRNA cDNA Synthesis Kit (Takara, Japan), 20 μl反应混合物。反应在37°C下进行60分钟,在85°C下按照制造商的说明进行5秒。用SYBR进行RT-PCR®prex Ex Taq II™(Takara,日本)。附加文件中设计的引物11用大豆5srrna作为内源对照,加入Uni-miR-qPCR引物作为普通反向引物,在最终体积为25μl(含12.5μl SYBR)中进行qRT-PCR反应®Premix EX Taq™,1 μl正向和反向引物,2 μl模板。为了估计应力诱导样品中mirna的相对丰度,直接比较Ct值,并将其转化为褶皱变化差异。这些反应使用ABI7300 (Applied Biosystems 7300 Real-Time PCR System)进行。
MiRNA通过Northern Blot验证
对于MiRNA的定量,使用High Sensitive MiRNA northern blot Assay Kit (Signosis, USA)进行northern杂交。每个样品的总RNA 30 μg在15%的聚丙烯酰胺凝胶上电泳,并转移到膜上(由Signosis提供)。5'端标记的反义RNA生物素(Invitrogen公司,中国)用于杂交探针。绿色网络®二丙烯酰胺凝胶中的II染色(Biotech,中国)RRNA条带显示为负载控制。
参考文献
- 1.
Boyer JS:植物生产力和环境。中国科学:地球科学。
- 2.
Shinozaki K, Shinozaki K:细胞反应和对脱水和冷胁迫的耐受性的转录调控网络。植物学报,2006,57:781-803。
- 3.
Bartel B, Bartel DP: MicroRNAs:在植物发育的根?植物营养与肥料学报。2003,32(2):429 - 434。
- 4.
Bartel DP: microrna:基因组学、生物起源、机制和功能。acta photonica sinica, 2011, 40(3): 441 - 446。
- 5.
赖ec: microrna:基因组的矮子断言自己。中国生物医学工程学报。2003,13 (23):R925-936。
- 6.
RNA:引导基因沉默。中国科学:地球科学,2011,32(5):591 - 598。
- 7.
植物microrna及其靶点的计算鉴定,包括胁迫诱导的miRNA。acta photonica sinica, 2004, 40(6): 489 - 494。
- 8。
Phillips JR, Dalmay T, Bartels D:小rna在非生物胁迫中的作用。中国科学(d辑),2007,26(6):514 - 514。
- 9。
高鹏,白雪梅,杨丽,吕东,潘晓霞,李艳,蔡华,纪伟,陈强,朱艳:osa-MIR393:盐碱胁迫相关的microRNA基因。Mol Biol众议员2010。
- 10.
Trindade I, Capitao C, Dalmay T, Fevereiro MP, Santos DM: miR398和miR408在苜蓿水分亏缺反应中上调。植物学报。2010,231(3):705-716。
- 11.
Zhu QH, Spriggs A, Matthew L, Fan L, Kennedy G, Gubler F, Helliwell C:发育中的水稻颗粒中多种多样的microrna和类似microrna的小rna。中国生物医学工程学报,2008,18(9):1456-1465。
- 12.
金伟,李宁,张斌,吴飞,李伟,郭安,邓铮:小麦(Triticum aestivum) microRNA的鉴定与验证。植物保护学报,2008,32(3):441 - 446。
- 13.
丁丁,张磊,王浩,刘志,张志,郑毅:玉米根系中miRNAs在盐胁迫下的差异表达.安博特.2009,103(1):29-38。
- 14.
McCormick KP, Willmann MR, Meyers BC:小RNA测序实验的实验设计、预处理、标准化和差异表达分析。[j] .中文信息学报,2011,2(1):2。
- 15.
宋庆新,刘艳芳,胡晓燕,张文坤,马斌,陈淑英,张金生:大豆种子发育miRNAs及其靶基因的深度测序分析。生物技术学报。2011,11:5。
- 16.
Szittya G, Moxon S, Santos DM, Jing R, Fevereiro MP, Moulton V, Dalmay T: Medicago truncatula短rna高通量测序鉴定出8个新的miRNA家族。基因组学杂志。2008,9:593。
- 17.
赵长忠,夏辉,Frazier TP,姚媛媛,毕艳萍,李青青,李明杰,李春生,张波林,王小军:深度测序鉴定花生(Arachis hypogaea L.)中新型保守microrna。生物谷bioon . 2010, 10: 3。
- 18.
周丽丽,李晓明,刘强,赵凤琴,吴建云:基于下一代测序技术的小RNA转录组研究。J麝猫基因组学。
- 19。
草食对烟草小rna转录组和植物激素信号转导的影响。美国国家科学院学报。2008, 105(12): 4559-4564。
- 20.
miR393/AFB3调节模块调控拟南芥根系统结构。美国国家科学院学报。acta photonica sinica, 2010, 37(9): 1405 - 1405。
- 21.
Li R, Yu C, Li Y, Lam TW, yism, Kristiansen K, Wang J: SOAP2:一种用于短读对齐的超快工具。生物信息学。2009,25(15):1966-1967。
- 22.
Kulcheski FR, de Oliveira LF, Molina LG, Almerao MP, Rodrigues FA, Marcolino J, Barbosa JF, Stolf-Moreira R, Nepomuceno AL, Marcelino-Guimaraes FC,等:参与非生物和生物胁迫的新型大豆microrna的鉴定。基因组学杂志。2011,12:307。
- 23.
Joshi T, Yan Z, Libault M, Jeong DH, Park S, Green PJ, Sherrier DJ, Farmer A, May G, Meyers BC, et al: Glycine max中新的mirna和相关靶基因的预测。生物信息学。2010,11(增刊1):S14。
- 24.
陈R,Hu Z,张H:野生大豆(甘氨酸Soja)中microRNA的鉴定。J Intent Plant Biol。2009,51(12):1071-1079。
- 25.
Lagos-Quintana M, rahutt R, Lendeckel W, Tuschl T:小表达rna新编码基因的鉴定。中国科学:地球科学,2011,32(5):591 - 598。
- 26.
Floyd SK,Bowman JL:基因调控:植物中古老的microRNA靶序列.自然.2004,428(6982):485-486。
- 27.
杨晓霞,张浩,李丽:基于深度测序数据的拟南芥microRNA表达的全局分析。基因组学杂志。2011,98(1):40-46。
- 28.
Pantaleo V,Szittya G,Moxon S,MIOZZI L,Moulton V,Dalmay T,Burgyan J:使用高通量测序和降低分析鉴定葡萄微稻草及其目标。工厂J.2010,62(6):960-976。
- 29.
Klevebring D, Street NR, Fahlgren N, Kasschau KD, Carrington JC, Lundeberg J, Jansson S:杨树小rna的全基因组图谱。基因组学杂志。2009,10:620。
- 30.
Zhang B, Pan X ., Cobb GP, Anderson TA:植物microRNA:一个具有重大影响的小调控分子。中国生物医学工程学报。2006,27(1):3-16。
- 31.
生物燃料模型植物柳枝稷中的microrna及其靶标的鉴定。植物生理学杂志。2010,167(11):896-904。
- 32.
Carlsbecker A, Lee JY, Roberts CJ, Dettmer J, Lehesranta S, Zhou J, Lindgren O, Moreno-Risueno MA, Vaten A, Thitamadee S, et al:细胞信号通过microRNA165/6调控基因剂量依赖的根细胞命运。自然科学进展。2010,465(7296):316-321。
- 33.
Katara P, Gautam B, Kuntal H, Sharma V:对Glycine max中miRNA靶点、影响蛋白及其同源物的预测。生物信息学。2010,5(4):162-165。
- 34.
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD, Muehlbauer GJ, Nelson RT, Grant D, Specht JE,等:大豆转录组指南Glycine max的RNA-Seq图谱。植物生态学报。2010,10:160。
- 35.
wang J, Jung S, Keller S, Gregory RI, Diederichs S:许多通往成熟的道路:microRNA的生物发生途径及其调控。细胞生物学杂志。2009,11(3):228-234。
- 36.
朱建平:拟南芥微rna和其他小rna的研究进展。植物学报。2004,16(8):2001-2019。
- 37.
在大量不同植物物种中保守microrna的硅质鉴定。BMC Plant biology . 2008, 8: 37。
- 38.
Khvorova A, Reynolds A, Jayasena SD:功能性sirna和miRNAs表现出链偏。细胞科学学报,2003,22(2):209-216。
- 39.
Git A, Dvinge H, Salmon-Divon M, Osborne M, Kutter C, Hadfield J, Bertone P, Caldas C:微阵列分析、实时PCR和下一代测序技术测量差异microRNA表达的系统比较。RNA。2010,16(5): 991-1006。
- 40.
Evan Johnson W, Welker NC, Bass BL:用于下一代测序数据中mirna识别的动态线性模型。生物识别技术》2011。
- 41.
孙玉华,李丽,李丽,蒋VL:拟南芥中缺失的白杨胁迫响应微rna。植物学报,2005,17(8):2186-2203。
- 42.
孙卡R,Chinnusamy V,朱J,朱JK:小RNA在植物非生物胁迫反应和营养缺乏中的重要作用。趋势植物科学。2007,12(7):301-309。
- 43.
Girginova PI, Daniel-da-Silva AL, Lopes CB, Figueira P, Otero M, Amaral VS, Pereira E, Trindade T:二氧化硅涂层磁铁矿颗粒磁性去除水中的汞(2+)。胶体界面科学。2010。
- 44.
Kantar M, Unver T, Budak H:大麦mirna在脱水胁迫下的调控与靶基因表达相关。功能基因组学杂志,2010,10(4):493-507。
- 45.
Zhu E, Zhao F, Xu G, Hou H, Zhou L, Li X, Sun Z, Wu J: mirTools: microRNAprofiling and discovery based on high-throughput sequencing。核酸Res 2010, 38 Web服务器:W392-397。
确认
转基因植物研究专项(No. 2008ZX08010-002, No. 2011ZX08010-002);国家自然科学基金(No. 30971804);教育部新世纪优秀人才支持计划资助项目(批准号:ncet -08-0693);山东农业大学作物生物学国家重点实验室(批准号:ncet -08-0693)2010 kf02)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
YYD和JYW进行数据分析。HYL、JYW和YYD撰写了手稿。HYL和XKL构思了这项研究。HLY参与northern blot验证。NW和JY准备样品。XML和YFW参与了qPCR验证。所有的作者都通过了最终的手稿。
李海燕、董元元对这项工作的贡献不相上下。
电子补充材料
12870 _2011_964_moesm1_esm.doc
附加文件1:读取丰富的各种分类的小rna。阅读丰富的各种分类的小rna在模拟和三个胁迫,干旱,盐度,和碱度。(医生33 KB)
12870 _2011_964_moesm2_esm.pdf
附加文件2:小rna的长度大小分布。小rna的长度大小分布分别在模拟、干旱、盐度和碱度。(PDF 493 KB)
已知的miRNAs
额外的文件3:大豆.共鉴定出95个miRNA家族对应的133个已知miRNA大豆在模拟和三种压力下。(医生290 KB)
12870 _2011_964_moesm4_esm.pdf
附加文件4:miRNA读取的频率分布。丰富的miRNA阅读频率分布在模拟和干旱、盐碱三种胁迫中。(PDF 109 KB)
12870_2011_964_MOESM5_ESM.DOC
附加文件5:表达的miRNA标签的分布。表达的miRNA标签数量分布在模拟和干旱、盐度和碱度三种胁迫下。(医生82 KB)
12870 _2011_964_moesm6_esm.png
附加文件6:保守的miRNA分布在其他物种中。大豆的miRNA序列在其他物种中保守。(PNG 97 KB)
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
李慧,董永强,尹慧。et al。应力相关的MicroRNA的表征大豆通过深度测序。BMC植物杂志11,170(2011)。https://doi.org/10.1186/1471-2229-11-170
收到了:
接受:
发表:
关键字
- mirna表达
- 深度测序
- 阅读数
- microrna的家庭
- 植物microrna