抽象的
背景
常见的豆是一种重要的豆科作物,只有用传统方法制成的中等数量的短表示序列标签(EST)。该研究的目标是使用全长cDNA技术来开发与开放阅读框架的开始重叠的EST,因此可用于基因组序列的基因注释。还构建了图书馆以代表在干旱,低土磷和高土铝毒性下表达的基因。我们还对共同豆类进行了全长cDNA库对两个以前的非全克隆EST集的比较。
结果
构建了两种全长cDNA文库:一种用于干旱耐耐受性MesoAmerican基因型BAT477,另一个用于已选择用于基因组测序的酸性土壤耐受性Andean基因型G19833。使用经过干旱受到干旱的深根汽缸生长在三种土壤类型中生长,并从根部和地上采集无干旱胁迫和组织。机器人上共选择了20,000个克隆,从每个文库中有一半。然后,将来自G19833库的近10,000个克隆进行测序,平均读取长度为850个核苷酸。共有4,219个unigenes鉴定为2,981个Contigs和1,238名单身。这些在功能上用基因本体论术语注释并置于Kegg途径中。与常见豆中的其他EST测序努力相比,大约一半的序列是新的或代表已知基因的5'末端。
结论
目前的全长cDNA文库增加了普通菜豆可用的技术工具箱,我们对这些克隆的测序大大增加了普通菜豆基因组可用的独特EST序列的数量。这对大豆基因或普通大豆全基因组序列的功能基因注释、剪接位点变异分析以及内含子/外显子边界的确定具有重要意义。此外,该文库还包含大量的转录因子,对发现和验证干旱或非生物胁迫相关基因具有重要意义。
背景
豆类家族是谷物后作为人类食物来源的第二个最重要的作物家庭,并提供了其他产品,包括饲料和原料,有价值的木材,植物油,生物燃料,重要药物甚至毒品[1].豆科植物具有固氮、与其他植物竞争、排斥食草性和在一系列环境中的酸性土壤上生长的能力,在退化土地的稳定和造林方面是无与伦比的[2].许多豆类是国际贸易的主要成分,因为它们价值高,是蛋白质、热量和油的来源。在豆科植物中,普通豆类(菜豆)是人类直接消费的最重要的作物,总产量仅次于大豆(大豆L.)和花生(落花生hypogeal .)。然而,与这些物种不同,豆类主要在小于中等规模的农场上生长,并不用于工业处理[3.].
表达的序列标签(EST)是转录基因的部分序列,并且代表不同组织中的基因表达,并且通常根据提取mRNA的植物处理和发育阶段的植物处理和发育阶段而不同的基因型。4].已知的ESTs来源于转录后的mRNA,将其克隆到cDNA文库中,然后进行测序集体.因此,为构建主要豆类作物(如大豆)的许多不同的cDNA文库进行了大量的工作[5]和模型豆类种类如莲花japonicus.[6和枪管医生,Medicago truncatula[7].
为所有植物物种发现的EST数量超过2100万次序列。对于豆类植物,总共有超过300万次序列,豆豆(150万)和模型豆类桶(280,000)和莲花(242,000)。这与超过600万次序列相比禾本科近300万十字花科.
米饭和米饭和综合图书馆拟南芥蒂利亚纳例如(8].在豆科植物中,与模型豆科植物和大豆相比,在作物豆科植物中发现的est相对较少。在较小的豆类作物中,只有最近在豇豆(Vigna Unguiculata.)由Mandero等。[9接近20万株无害环境技术的临界值,而普通蚕豆只有这一临界值的一半左右。
在常见的豆中,CDNA克隆或EST测序的大规模努力很大程度上努力,截至2010年12月,目前的EST数量是114,139。常见豆类的编制开始于来自CIAT的群体的中等数量的GenBank条目开始和联庭[10- - - - - -12]哥伦比亚,巴西和墨西哥的组织分别显示了这一作物对拉丁美洲的重要性。
美国的一些大学,如明尼苏达大学,也对其他的ESTs进行了测序或分析[13和密苏里大学[14].在这些研究中,由Melotto等人进行第一中等大小的系列。[11Ramírez等[12]分别包含5243个和15333个ESTs或unigenes。然而,这些分别代表了3个和5个不同的cDNA文库的EST测序。
从普通豆中取样的组织主要代表了Melotto等人的一组库中的病苗组织[11然后是Ramírez等人的一系列组织,从结节和结节状根到叶子和豆荚。[12].此后,Thibivilliers等人发表了一份包含37,919个未修剪的EST的大规模EST集[14]由染上锈病的豆子(Uromyces Appendiculatus.)和来自两个根库的一套额外的est [15].此外,在近缘种的胚柄细胞中已开发出大量的est序列391,150个p . coccineus由加州大学生。最后,在大学的加拿大团体。萨斯喀彻温省已经测量了10,272个estsP. angustsisimus,常见豆的另一个亲戚。
在其他热带豆类中,鸽子豆(Cajanus Cajan.L.),已经有一个大约10000个序列的EST项目[16]由于与常见豆的密切关系及其适应与亚湿润的条件豆类相同的豆类的紧密关系,这是感兴趣的。栽培花生,落花生hypogea在美国,有86,935条ESTs,加上两个祖先种的花生,每个都有大约32,000条ESTs,是唯一被强调的其他热带豆科植物。
这些EST系列,只有一个来自Ramírez等人的集合。[12另一种来自Blair等人[15]已经代表了对非生物胁迫的耐受性,两个研究小组都强调在低磷条件下根中表达的基因。然而,一些研究已经做出了评估代谢途径和克隆转录因子的努力[17[序列来自干旱处理的组织的差异表达CDNA [18,19].因此,需要在共同的豆类和其他热带豆类中进行额外的EST测序,特别是对于受干旱和土壤或天气应力影响的组织,这对这些作物的生产力非常重要的问题[20.].
在豆科植物尤其是普通豆类中,转录组分析和EST测序的一个方面一直缺失,那就是全长cDNA克隆的克隆。这项技术,首先由Seki等人描述[21和Carnici等[22],包括通过其5'帽捕获mRNA,并在连接到合适的载体和从poly A尾部逆转录时稳定完整的转录[23].已经为大量拟南芥组织进行了全长cDNA文库[24,25以及几种富含淀粉的作物[26,27]但除大豆外,豆类少少[28].
全长cDNA文库对于转录组分析、比较基因组学和基因组序列验证极为有用,因为它们代表整个转录单位,而不是像大多数其他cDNA文库那样代表部分基因序列[24].它们特别有价值,因为它们揭示了大多数基因的转录起始位点,对其5'端进行EST测序,揭示了未翻译区域和蛋氨酸编码、ATG密码子、翻译起始信号。然后,它们可以与非全长cDNA测序克隆一起使用,覆盖整个基因序列,让科学家确定开放阅读框的开始和结束,并将所有信息锚定到基因组序列。
这些特性使全长cDNA序列在发现可变剪接模式和启动子区域方面发挥重要作用[23].在某些情况下,已用于构建微阵列以表征转录因子与基因的5'UTR中的启动子元素的结合[25].全长CDNA还通过作为在[23].有几个例子是通过使用这些克隆体来确定3D晶体结构[29- - - - - -32].
此外,全长cDNA克隆具有在不同物种中表征基因结构的作用。例如,它们的5'和3序可以用于比较5'UTR(未翻译区域)与ORF(开放阅读框架)和3'UTR区域中的GC内容和折叠容量进行比较[27].
最后,与其他类型的EST一样,全长cDNA克隆测序可用于开发许多类型的遗传标记,包括简单的序列重复(SSR),其倾向于在5'UTR序列中的单一核苷酸多态性(SNPS)特别适用于ORF的不同部分[33,34].在构建全长cDNA文库时,重要的是使用标准基因型,如基因组测序工作中的基因型,因为基因组与基因的比较在这种情况下变得更加直接。综上所述,全长cDNA技术对于基因注释、转录组测序和比较基因组学具有非常重要的意义
因此,本研究的目的是制备全长cDNA文库,该文库可用于共同豆类中的基因发现,测序基因型的基因组注释以及对作物中的非生物胁迫耐受性的理解。取样多种处理,包括不受顽固的,干旱,低磷和铝胁迫植物,以增强转录组机械的激活,并自然地标准化MRNA的取样。此外,在该初始FL-cDNA文库结构中使用了两种基因型,该基因型已知是耐旱(BAT477),另一个是全长基因组测序的对象(G19833)。从第二个文库中产生了近10,000个EST,以显示该技术在确定基因结构方面的效用。
该EST测序项目是作为育种项目的一部分进行的,以发现亚撒哈拉以南非洲的边缘地区的共同豆类中的分子标记,并讨论了来自全长cDNA序列的标记发现的过程。我们还旨在将全长CDNA库中的EST与共同豆类的两个大型EST集团进行比较,并表明这项技术在这种较低的研究中具有基因组工具开发的优势。
方法
植物材料
基于它们的应力抗性和在基因组研究中使用的属性来选择两种精英常见的豆类基因型。首先,基于其深生生根能力和已知的干旱耐受性选择中间酵母基因池高级线BAT 477 [35[第二,基于对Al毒性和低磷土壤应激的耐药性选择Andean基因库基因型G19833 [36,37].根据Schlueter等人绘制的物理图谱,选择后一个基因型进行全基因组测序[38]并由Cordoba等提炼。[39].Blair等人所描述的DOR364 × G19833制图群体[40还用于确定有趣基因的位置,例如红色种子颜色的位置[41.]和对BCMV的隐性抗性[42.]以及营养性状的QTL [43.].
处理、实验条件和采样次数
将基因型进行干旱和灌溉(对照)条件作为主要处理。在不同位置收集三种具有特定性质的土壤,包括Palmira(高度压实),Darién(低P含量)和QuiliChao(高Al含量),总共六种处理,如附加文件所示1.试验是在温室条件下进行的,采用0.8 m的塑料pvc管插入非半透明套管,填充特定的土壤和土砂比2:1。所有干旱处理均在种子萌发后10 d停止灌溉,模拟自然干旱胁迫。在2个重复的裂区设计中,对照处理在整个试验过程中对每种土壤类型进行正常灌溉。采集气生组织和根组织,立即用液氮清洗并冷冻,用于随后的总RNA分离。每5天采集一次组织,直到干旱期达到35天,并从植物的每个发育阶段取样:幼苗(子叶和芽),生长阶段(叶、茎、芽和根),生殖阶段(花和小豆荚)。他们小心翼翼地用水流把沙土混合物洗掉,然后在塑料浴缸里漂洗。灌溉对照只在萌发后15、30和45天采集组织,这代表了生长和开花阶段(见附加文件)2为解释组织收获的时间进程和深根的照片,圆柱体培养系统)。
总RNA分解动作
冷冻组织用液氮机械研磨成细粉末。分别处理气生组织和根组织及其相应处理和采样次数。使用TRIzol进行总RNA分离®试剂(表达载体,猫。# 15596-018)并遵循制造商指南。总RNA微球再次悬浮于无RNA水中,用分光光度法定量。定量后,在每个目标基因型内分别汇集干旱和灌溉处理的每个采样时间获得的RNA数量。同时也分别收集了空气组织和根组织的总RNA。采用含甲醛变性琼脂糖凝胶(1.5%),溴化乙啶染色,测定RNA质量。分别对BAT477和G19833来源的mrna采用两种不同的引物标记,对分离的cDNA克隆进行基因型鉴定。
图书馆施工和EST测序
图书馆建设在日本的瑞肯学院进行,采用以下缩写方法,使用池中的MRNA在单独的反应中的每个基因型:Poly(a)+用手动方案后用μMACSmRNA分离试剂盒(Miltenyi Biotec)制备RNA。从生物素化的聚(a)构建全长cDNA文库+用CAP诱捕法和海藻糖热激活逆转录酶检测RNA。合成的双链cdna被酶切BAM嗨XHO.我,并连接到BAM嗨萨尔基于Lambda的pflciii-cDNA载体的I位点[21].在第一链CDNA合成期间,将个体化标签引物序列掺入每个基因型的文库中:由其在聚尾部之间的放置而鉴定的G19833的BAT477和5'-GTCATACG-3'中的5'-CTGATACG-3'。一种你以后我(GGCCNNN·NGGCC)网站。
一旦转换成Eschereschia杆菌从G19833库中克隆的细菌,由一个自动化的机器人菌落采摘器采摘到总共10,000个克隆(每个库各取一半)。在密苏里州圣路易斯的华盛顿州立大学测序中心,总共有9984个克隆(约占文库的一半)被送往测序中心,从两端分别对384个克隆进行了大小和测序,以确定它们插入的大小和质量。应用生物系统公司的全自动毛细管DNA测序仪(ABI 3730×)对甘油样品共26个测序板进行了测序。采用M13-21引物(5'-TGTAAAACGACGGCCAGT-3')对全长cDNA插入的5'端进行测序。尽管我们制作了两个文库(一个是来自G19833的安第斯文库,一个是来自BAT477的中美洲文库),但由于资金限制和G19833的相对重要性,我们只从第一个文库中进行了测序(S. Jackson, pers.)。沟通)。
数据组件
为低质量和矢量污染修剪序列读数。这是针对每个全长EST序列完成使用PHRED软件的[44.],以消除100个碱基中N大于1的低质量区域或Phred质量分数< 20的一段碱基。这些通常出现在序列的3'端,被丢弃。同时,通过TrimVector (Sequencher, Ann Arbor, MI)使用NCBI发现的载体数据库消除载体序列,然后使用geneih (Biomatters Ltd, Auckland, New Zealand)软件程序进行人工确认,以检查不良序列。Poly-N拉伸被掩盖和小插入克隆(< 100)或点击大肠杆菌序列被忽视。如果随后是载体序列,则在其相邻的碱中鉴定并修剪聚尾。软件程序BLAST2GO来自[45.],它是Blast 2.2.23的一部分[46.[用于除去核糖体,叶绿体和线粒体序列以及非植物击中的任何命中。软件CAP3来自[47.[默认参数(间隙惩罚因子,n> 6;段对分数截止,n> 40,重叠长度分数截止n> 80等)用于组装来自全长cDNA克隆的序列,使我们能够组装新创建和清洁全长的EST,进入Contigs和Unigenes。与Ramírez等人的全长cDNA文库中清洁序列分布的分布进行了比较。[12和Thibivilliers等人[14].
基因注释和比较基因组学
Blast2Go再次与EST序列的组装一起使用,以确定每个UNIGENE和CONTIG从全长cDNA数据库的顶部击中分布。针对相应的非冗余(NR)数据库进行搜索,其粘附肌肌菌对所有更高的植物蛋白质。然后用高分段对(HSP)截止为33的高分段对(HSP)截止值和1E的电子值阈值来确定E值和正对准长度分布-3.基于Harris等人分配基因本体(GO)。[48.并通过Uniprot、TAIR、GR-protein和Ecosys数据库进行评估。然后在四个基因本体论水平上评估基因可能的分子细胞功能、细胞定位和参与。通过比较BLAST2GO报告中已知蛋白的起始点,并使用e值阈值将其与每个单基因的起始密码子进行匹配,从而对5'非翻译区(UTR)和开放阅读框(ORF)边界进行基因注释-6截止到1E-55还与Ramírez等人的序列进行了比较[12和Thibivilliers等人[14],并附有大豆的注释(G. Max.)基因组和大豆est序列测定内含子/外显子边界,并确定可能的起始密码子。此外,KEGG注释还用于确定KO本体,确定以上描述的全组装在各种生化途径中的unigenes和singleton的位置,并使用定向无环图(DAG)确定基因关系。最后,根据5'UTR或ORF内的位置,首先使用软件程序RepeatFinder在全长序列中识别简单序列重复[49.],然后更确切地说是SciRoKO [33].
结果
利用cap -trap技术成功构建了中美洲耐旱基因型BAT477和安第斯耐酸土壤基因型G19833的全长cDNA文库。6个不同灌溉×土壤处理对这两个基因型分别获得3.789 mg和4.258 mg高质量总RNA文库,足以满足高度复杂的polyA mRNA全长选择过程。图书馆,共有20000个克隆选择机械(一半从每个基因型)和5和3的初步排序'ends的克隆库板(384)被证明在长度和平均1.5 kb non-chimeric序列主要与多聚腺苷酸尾3 'end。额外的文件3.以总长度表示初始无性系的分布。
在此初始测试之后,从Andean G19833库中测序近10,000个克隆。该序列均为5',平均读取长度为850个核苷酸(NT)。其中,9626超过了N的初始阈值,并且平均读取长度为781 nt。在载体修剪时,平均插入长度为564nt,修整和低质量序列消除后的清洁序列的数量为7039.序列被提交给GenBank(条目号JK037067-JK044145),并由每个Contig中的单身和独立克隆组成。桌子1通过Ramírez等人对全长cDNA文库和非全长文库序列的比较,显示了最近三次对普通豆科植物EST的测序成功率[12和Thibivilliers等人[14].该图书馆的测序成功率(71%)与Ramírez等人的75%相当。[12].所有全长cDNA克隆组装后,共鉴定出4219条unigenes。其中1238个单基因(占29.3%的ungene和17.5%的序列)和2981个contigs(占70.7%的ungene和42.1%的序列)由CAP3组装而成。平均序列重叠群内的数量是3.3,平均长度的重叠群678元。与此同时,单例的平均长度是568元。大量的单例和大量的重叠群的数量相对于序列表示为图书馆低冗余。这并不奇怪,因为用于提取RNA的植物是在三种不同的土壤处理、干旱胁迫和灌溉条件下生长的,而且整个植株(地上部分和地下部分)分别在7个和3个时间点收获,用于cDNA的制备。
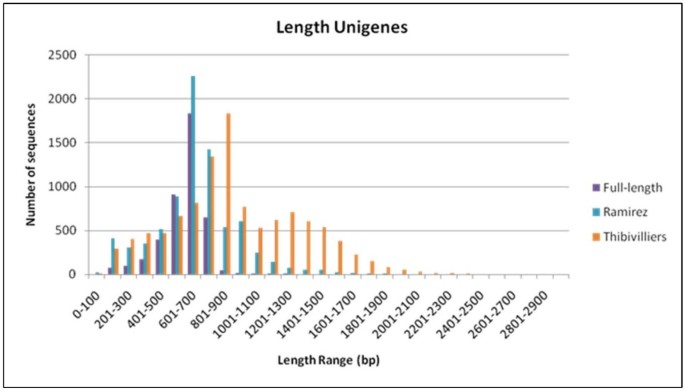
将全长unigenes与Ramírez等人的其他EST测序结果进行了比较[12和Thibivilliers等人[14在常见的豆中,鉴定的unigenes的长度,与中间长度(606nt)相比,其在第一研究中的中间长度(606nt)中的中等和良好分布,但是对于制造unigenes的第二研究(1024nt)的平均值更高每个克隆的5'和3'序列。这为Thibivilliers等人创造了支架或全长基因。[14]库中较长的单基因在图右侧的双峰显示1对于那些unigenes。在本研究中或Ramírez等人。[12]中包含3′端序列,解释了单基因长度较短的原因。
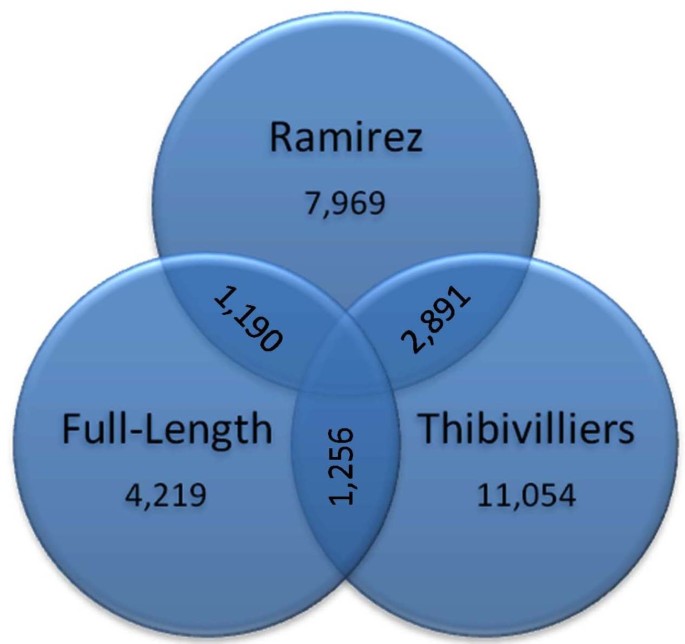
在Ramírez等人的EST测序努力的全长cDNA文库组装比较。[12和Thibivilliers等人[14我们发现,大约一半的序列是新的,其中相当多的序列代表了之前从其他EST研究中发现的基因的5'部分,如图所示2和3..在本研究中,与Ramírez等人的研究相比,共发现3029个全长cDNA unigenes。[12虽然1190是相似的。对于Thibivilliers等人的工作。[14],其中1256项相似,2963项新颖。在Ramírez等人的研究中[12和Thibivilliers等人[14]只有2891个类似的unigenes和est序列的重叠。在该分析Thibivilliers等人的分析的情况下。[14与他们报告10581 unigenes的报告相比,ESTS给出了11054个unigenes。同时,近78%的个体全长的全长unigenes对已知或未知功能的大豆基因具有同源性(4219个Centig或Singleton序列中的3280次),仅基于任何击中的豆类特异性工厂数据库。基于使用1E的BLASTN搜索,来自Medicago(1221个unigenes,28.9%)或拟南芥(147人未进,3.5%)的同源物(147人,3.5%)-30临界点。
在我们在全长cDNA文库中发现的未发现的Unigenes的功能分析中,我们发现Blast2Go发现了从我们的低阈值为1×10的命中范围-10年可达1 × 10-175相似性值从40到100%在一系列核苷酸窗口内对齐。获得全长基因最多的物种是大豆(超过1500个)和葡萄(超过500个)。其次是用苜蓿、杨树和蓖麻分别击中250至400次。
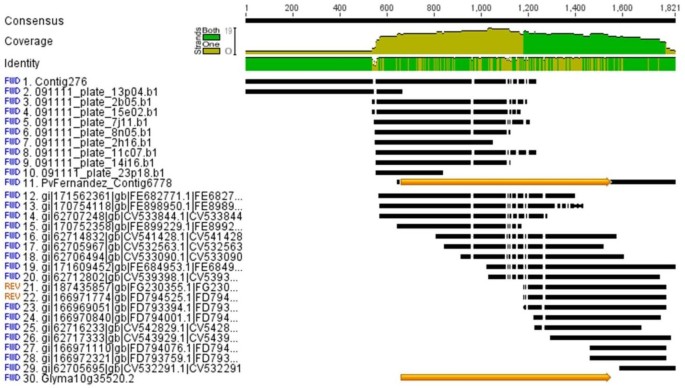
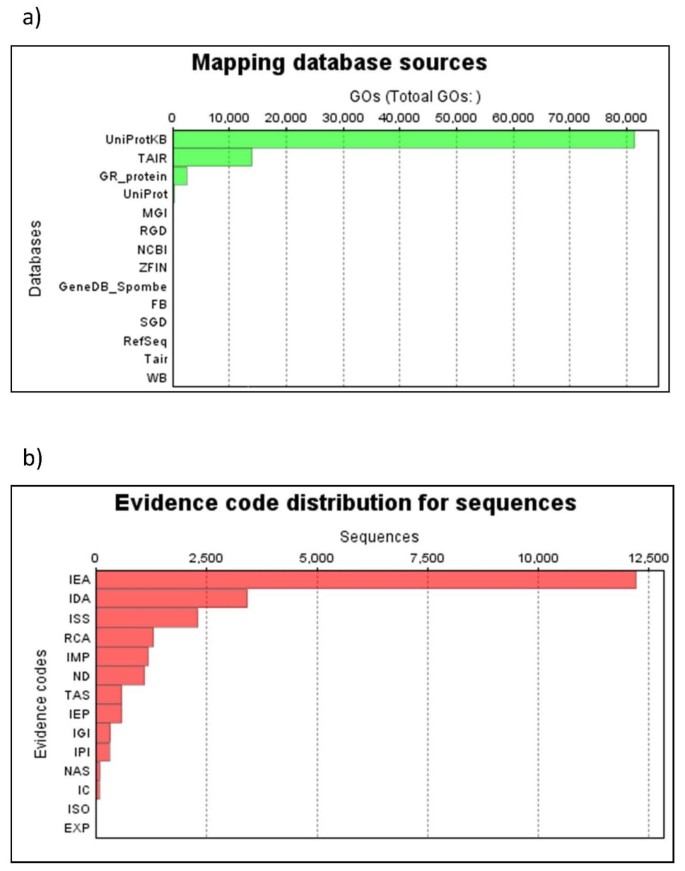
只有100个最高点击率是直接使用普通豆子基因的。这些命中数既反映了物种的相似性,特别是在普通豆豆的情况下,也反映了非冗余的UniProtKB或TAIR数据库下GenBank中存在的unigenes的数量,这些unigenes是最常用于unigene本体的映射(图)4).表达最频繁的基因通常是管家基因,但确实反映了植物的非生物胁迫条件,从其全长文库制作和测序(附加文件)4),包括寻找水通道蛋白的倾向,这对渗透胁迫下细胞的水分吸收有用(图3.).实际上,分布147和1001分别表示含有19和20个EST的尖端和覆盖物型水蛋白。
在基因本体论分析(表2),评估各种水平,并在2级,用于生物过程的四分之一基因各自落入细胞(28.4%)或代谢(26.7%)类别。对于分子函数,基因几乎均匀地分成结合(42.5%)和催化功能(37.8%),然后进行转运蛋白活性(5.8%)。细胞组分分为细胞,细胞器和较小程度的细胞膜和细胞外区域。与我们实验室中最近制作的根本的值相比,这些值与多样性不同[15].例如,全长库中的响应,生物调节,发育过程和刺激反应的基因百分比较高。由于全长文库是由鸟类制成的,并且预期这种性质的地面差异。
本文中对全长cDNA文库的分析与Ramírez等人的文库的分析还发现了其他差异[12]对于叶组织和Thibivilliers等人。[14].这表明全长cDNA文库在不同水分处理和土壤生长条件下的根、茎和叶组织的混合情况下具有高度的标准化。对非生物应激、广义应激、化学刺激、细胞和大分子代谢、初级代谢、氧化/还原和分解代谢过程的反应在生物过程的第三层次中都很重要(数据未显示)。水通道蛋白在文库中的出现频率可能反映了半数用于构建全长cDNA文库的组织来源对干旱胁迫的适应。
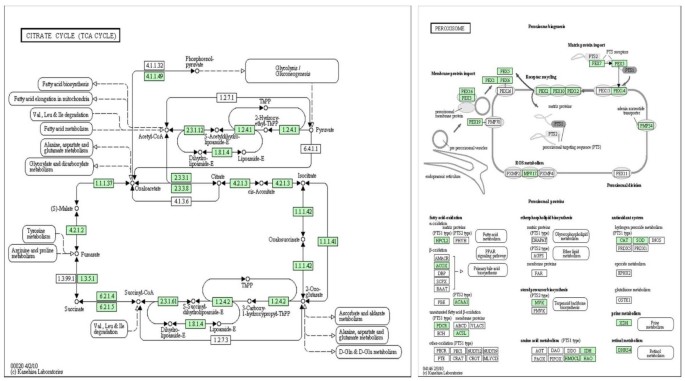
通过将unigenes与全长cDNA文库的Go注释与基因和基因组(Kegg)的京都百科全书进行比较,我们能够提出一个KO注释,其中主要类别下降顺序:翻译,能量新陈代谢,碳水化合物新陈代谢,转录,共同代谢,复制和修复,氨基酸代谢,信号传导机制以及未分类的基因。用于柠檬酸盐循环和过氧缺组函数的Kegg途径的实例如图所示5其中,全长cDNA文库中的基因被突出显示。作为一个例子给出了过氧物酶体通路的通路受到各种压力,如采样在图书馆的建设(干旱、低磷、高影响根和地上组织),而柠檬酸循环,同时受到这些压力的影响,充满了更多的本构基因。
就用于产生全长cDNA文库的两种基因型的比较(G19833与BAT477)的比较,这受到仅在初始阶段使用的初始阶段使用的初始阶段。此外,鉴于基因型的单个标签在3'末端,SNP识别需要3°序列。因此,不可能进行我们最初计划的两种基因型的单核苷酸多态性分析比较,尽管我们能够鉴定使用与三核苷酸的两个不同搜索引擎重复的大量简单序列重复,而不是比di更常见- 核苷酸重复(表3.).SSRS的标记识别因RepealFinder鉴定只有175个(2.5%的EST),但Sciroko共发现1,932名(24.3%的EST)。
讨论
本研究的主要成果是在三种非生物胁迫条件下,从两种重要的普通菜豆基因型中构建了两个全长cDNA文库,并对文库进行了约7000个ESTs的初步测序,平均长度为564个核苷酸。EST测序工作的成功率相当于Ramirez等人[12],但略低于Thivibilliers等人[14[鉴于riken到大学的图书馆运动是预期的。华盛顿的排序。每HQ序列的高率在为迄今为止为普通豆立的cDNA文库中具有相当独特的,并且代表用于创建全长cDNA文库的组织的广泛表示及其5'dend的主要覆盖。
Umezawa等人采取了类似的方法。[28]当构建大豆全长cDNA文库时,它们共有七种胁迫(干旱,盐,冷冻,低温,P饥饿,洪水和线虫)以及三种专业组织类型(花蕾,结节和发展种子)用于他们的RNA提取物。我们使用了类似的策略,并在低于地面和以上的所有器官上方的所有器官,在低磷,铝毒性和干旱的重要常见豆胁迫下,高达35天的增长[2,20.].我们没有包括正在发育的种子或豆荚,因为在圆筒盆栽下生长的植物繁殖阶段的组织质量低于营养阶段的组织。我们仔细地从土壤中分离根部,广泛取样。
除了图书馆建设本身之外,EST测序现在现在将常见豆类的总数带到近120,000个序列中。考虑到最先前的est在500至600nt的范围内,并且来自常规cDNA文库,仅代表从基因序列中的随机点(通常在ORF中)开始的特异性组织和部分序列,这项工作具有提供更多的优点物种的不同EST。此外,我们获得了多个EST读取(从700到900 NT长),主要代表5'UTR加上最重要的起始密码子和ORF的第一个外显子,而不是3'典型的常规EST项目。这是全长cDNA文库构建的真正益处,并使我们能够在与大豆的基因组序列进行比较CDNA序列时发生外显子和内含子的适当基因建模。预计全长cDNA序列将有助于对基因组的注释。我们在EST测序中努力的另一个优点来自我们发现的多组织采样,与其他文库相比,我们发现的多组织采样增加了某些基因本体类别中未成年人的比例。例如,非常值得注意的是,全长库的总未进剂的数量差不多是ramírez等人的数量。[12尽管测序工作只完成了一半,但这表明混合组织库和全长cDNA构建作为基因发现策略的成功。
在RIKEN开发的全长cDNA文库和帽捕获的这种优势基于生物素组的化学引入真核mRNA帽结构的二醇残留物。然后通过RNase I的消化方法,核糖核酸酶可以在任何位点切割单链RNA,并通过选择全长cDNA。通过该方法产生的文库含有非常高的全长CDNA比例,并在不涉及PCR扩增的情况下产生优异的产量,这可能引入克隆的代表性中的偏差。Carninci等。[22还发现,通过在60℃的高反应温度下将二糖海藻糖引入逆转录酶反应,导致合成甚至更长的全长CDNA,以及文库中长全长CDNA的更高表示。总之,通过MRNA帽的生物素化和链霉抗生物素蛋白捕获选择全长CDNA的方法,然后使用海藻糖 - 热稳定的逆转录酶,使得可以制备更长的全长CDNA,同时去除非全长CDNA [24,25].该方法已被证明是构建各种作物高含量全长cDNA文库、分析结构域顺序和确定起始密码子的理想方法。幸运的是,我们现在有了普通豆类,这是继大豆之后第二个拥有这些库的豆类[28].
最近,由于下一代测序技术的出现,对EST测序的重视已经减弱,下一代测序技术可以快速分析转录组。然而,全长cDNA序列仍将有助于发现可变剪接位点和解开基因家族中的同源基因。对于普通豆的全长cdna,这保留了更多的相关性,因为它是一个基础成员Phaseoleae含有其他重要物种,如大豆,豇豆和Pigeonpea [1.50,[51]]。二倍体常见豆类和祖先四倍体大豆的EST的比较可能是重要的,以发现基因的精确拷贝是直脑,以这种方式利用每个物种作为另一个模型。目前,与其他豆类部落(Cicereae,Dabergieae,Fabeae,Galegeae,Loteae和Trifoliea)相比,Pumplace of T部部落中的EST的数量是豆类(2.2米的读数)中最高的豆类(2.2米)。的确,热带属Cajanus,甘氨酸,菜豆和豇豆属所有代表经济上重要的食物或工业庄稼,而在凉爽的季节豆类中,在较重要的较小的模型或饲料种类中已经进行了大部分rest测序[50].最后,不断增加的序列数量Phaseoleae与CaesiLpinoids和MimoSid家族相比,反映了Papilionoideae豆类的偏见,每个含有较少的ESTS。
相关进化枝中可用序列的数量与每个植物物种或进化枝中任何一个成员标记发展的容易程度日益相关。我们已经发现全长cDNA文库对豆科植物重要途径(如非生物胁迫反应或营养积累)中各种基因的5'UTR和启动密码子非常有用。在本工作中,我们开始寻找一套新的EST-SSR,考虑到我们已经获得的est中5'UTR序列的比例很高,可能会有新的类别和更多的多态性ssr。到目前为止,我们已经应用了两个程序,从175到1400个ssr中发现,并正在考虑在开始初级设计之前使用其他软件进行更完整的分析。已知无害环境技术是SSRs的丰富来源[52].在第一个软件时发现2.5%的EST有SSR。在其他未来的工作中,我们计划利用我们制作的两个图书馆,这些图书馆在3'末端标记,其中两个不同的标签,区分我们用于图书馆结构的每个基因型。我们计划它从已经测序的全长克隆的3'end序列序列,以构建每个基因的支架,然后将CDNA克隆的群体与单个核苷酸多态性(SNPs)进行比较。我们开始评估全长cDNA序列的组件,常规EST序列来自常见的豆类和相关物种,通过Andean与Mesoamerican的比较来识别可能的SNP或p .寻常的与p . coccineus组装的ests。由于迄今为止普通豆的许多EST图书馆,这是可能的,从Andean Snap Bean(早期加仑)到中间酵母干酪(Negro jamapa)代表一系列品种。
此外,我们很快将具有Andean Landrace G19833基因型的基因组序列和Mesoamerican育种线BAT93基因型,用于这些比较。我们的计划是在即将到来的纸张中从两个文库中逐步排序,以便确定每个克隆的基因型,并在BAT477和G19833彼此比较中使用这些对单核苷酸多态性的生物信息分析。用测序的BAT93基因组。我们感兴趣的基因组注释的另一个方面是通过对这些文库的更多序列进行易于衡量的基因组注释是通过将它们区分为5'ends的揭示多基因家族,并在表达中确定促进剂序列转录物。在这种情况下,我们被识别为交替剪接的示例的水素是有趣的,以进一步表征。研究如何完全克隆的全长克隆覆盖的个体的单独克杆途径也有趣,因为它给出了在压力期间导通的途径的证据(例如,例如PerixoSime途径),或者已被证明是构成相关的对特定应力(例如柠檬酸盐循环途径,这对于在铝应力期间产生有机酸是重要的。另一种活性是确定代表性的全长cDNA克隆是如何从全基因组序列表达的所有基因。
结论
全长cDNA文库的价值在其实用性中,因为它们的基因组序列的正确注释和基因的功能分析由于其大多数基因的5“UTR和全部或全部或全部或全部或全部或者的全部或全部或者展示来源(SEKI ET)al。2002)。当从5'开始SEQECUECECENCECENCENCED 3'时,他们可以用来创造明确地确定转录物长度的物理脚手架。由于它们是每个独特的克隆,它们更有用来确定替代剪接位点。这是第二个为豆科植物和谷物,木薯或拟南芥之外的第一组全长cDNA图书馆。因此,利用该图书馆收集的信息是重要的,以进行标记发育和可用于植物育种的基因组特征。
最后,目前的全长cDNA文库大大增加了普通大豆基因组的独特EST序列的数量,特别是提供了更独特和更有用的基因鉴定的5'端序列。这些EST标签在功能基因注释、剪接位点变异分析、内含子/外显子测定、基因同源性评估或KEGG通路确认等方面都很有用,特别是在未来全基因组序列可用的情况下。
参考
- 1.
Varshney RK,Close TJ,Singh Nk,Hoiseton Da,Cook Dr:孤儿豆科作物进入基因组学时代!CurrOp植物BIOL。2009,12:202-210。
- 2.
Graham pH值,Vance CP:更新豆类利用,豆类:重要性和约束更大的使用。植物理性。2003,131:872-877。
- 3.
Broughton WJ,Hernandez G,Blair Mw,Beebe S,Gepts P,Vanderleyden J:Beans(菜豆SPP。) - 模型食品豆类。植物和土壤。2003,252:55-128。
- 4.
Hatey F,Tosser-Klopp G,Clouscard-Martinato K,Muleant P,Gasser F:基因的表达序列标签:综述。evol evol enecet。1998年,30:521-524。
- 5.
Vodkin Lo,Khanna A,Robin Sheay R,Steven J Clough SJ,Gonzalez Do,Philip R,Gracia Zabala G,Thibaud-Nissen F,Sidarous M,StrömvikMW,Shoop Eschmidt C,Retzel E,Erpelding J,Shoemaker RC,Rodriguez-Huete Am,Polacco JC,Coryell V,Keim P,Gong G,Liu L,Pardinas J,Schweitzer P:全局表达的微阵列构建,具有低冗余组27,500测序CDNA,代表了一种发育阶段阵列和生理条件。大豆植物。BMC基因组学。2004,5:73-
- 6.
Asamizu E,Nakamura Y,Sato S,Tabata S:特征莲花japonicus.从大规模表达序列标签(EST)分析中推断的基因曲目。植物mol biol。2004,54:405-414。
- 7.
张F, Haas BJ, Goldberg SMD, May GD, Xiao Y, Town CD:测序Medicago truncatula使用454生命科学技术表达序列标签。中国生物医学工程学报,2006,27 (4):414 -
- 8.
Asamizu E,Nakamura Y,Sato S,Tabata S:拟南芥CDNA的大规模分析:来自归一化和尺寸选择的cDNA文库的12,028个非冗余表达序列标签。DNA Res。2000,7:175-180。
- 9.
Bhat Pr,Fenton Rd,Wanamaker S,Pottor M,Hearne S,Cisse N,Fatokun C,Ehlers JD,Roberts Pa,Close TJ:Cowpea的共识遗传地图[Vigna Unguiculata.(l)沃尔普。]和基于EST衍生的SNP的同步。PROC NATL ACAD SCI。2009,27:18159-18164。
- 10.
Blair MW, Muñoz-Torres M, Giraldo MC, Pedraza F:安第斯起源、基于基因的普通豆子微卫星的发展和多样性评估(菜豆l .)。BMC植物生物。2009,9:100-
- 11.
普通菜豆表达基因的比较生物信息学分析(菜豆)幼苗。基因组。2005,48:562-570。
- 12.
Ramírez M, Graham MA, Blanco-López L, Silvente S, Medrano-Soto A, Blair MW, Hernández G, Vance CP, Lara M:普通豆子est测序和分析:为功能基因组学奠定基础。植物物理学报。2005,37(4):457 - 461。
- 13.
Graham MA, Ramírez M, Valdés-Lopez V, Lara, Tesfaye M, Vance CP, Hernández G:磷胁迫诱导候选基因的鉴定菜豆通过多种植物物种的聚类分析。Func Pl Bio。2006,33:789-797。
- 14。
Thibivilliers S,Joshi T,Campbell Kb,Scheffler B,Xu D,Cooper B,Nguyen HT,Stacey G:生成菜豆对他们的监管的估计和调查Uromyces Appendiculatus.感染。BMC Plant Bio. 2009, 9: 46-
- 15.
Blair MW, Fernandez AC, Pedraza F, Muñoz-Torres MC, Kapu Sella N, Brown K, Lynch JP:对普通豆类高磷和低磷适应的两个cDNA文库的ESTs进行平行测序。植物基因组,2011。
- 16.
Raju NL,Gnanesh Bn,Lekha P,Jayashree B,Pande S,Hiremath PJ,Byregowda M,Singh Nk,Varshney RK:PigeO发现的基因发现和标记开发的第一组EST资源(Cajanus Cajan.l .)。BMC Plant Bio. 2010, 10: 45-
- 17.
Hernándezg,ramírezm,瓦尔德斯 - 洛普·卢比·卢达姆Ma,Czechowski T,Schlereth A,Wandrey M,Erban A,Cheung,Wu HC,Lara M,Town CD,Kopka J,Udvardi Mk,Vance CP:磷常见豆类的压力:根转录和代谢反应。植物物理。2007,144:752-767。
- 18.
Barera-Figueroa是,Peña-Castro JM,Acosta-Gallegos Ja,Ruiz-Medrano R,Xoconostle-Cázaresb:在干旱耐麸质豆根中隔离上调基因(菜豆简历。PINTO VILLA)在早期水缺陷应力下,以及第3组LEA基因的新成员的表征。Func植物Biol。2007,34:368-381。
- 19.
黄志强,王志强,王志强,等:紫穗槐叶片响应相关基因的鉴定菜豆干旱压力。mol品种。2008,21:159-172。
- 20.
Graham Ph,Rosas JC,Estevez de Jensen C,Peralta E,Tlusty B,Acosta-Gallegos J,Arraes Pereira PA:解决豆类生产的辅助约束:Bean / Cowpea CRSP项目的角度来看。现场裁剪。2003,82:179-192。
- 21.
Seki M,Carninci P,Nishiyama Y,Hayashizaki Y,Shinozaki K:生物素化帽捕集器的高效克隆全长cDNA。工厂J. 1998,15:707-720。
- 22.
Carninci P,Shabata Y,Hayatsu N,Itoh M,Shiraki T,Hirazane T,Watahiki A,Shibata K,Muramatsu M,Hayashizaki Y:全长,帽子被困CDNA的平衡尺寸和长尺寸克隆到新型Lambda-FLC系列允许增强基因发现率和功能分析。基因组学。2001,77:79-90。
- 23.
Seki M,Shinozaki K:使用Riken Arabidopsis Thaliana全长CDNA的功能基因组学。j植物res。2009,122:355-366。
- 24.
Seki M, Narusaka M, Kamiya A, Ishida J, Satou M, Sakurai T, Nakajima M, Enju A,秋山K, ooono Y, Muramatsu M, Hayashizaki Y, Kawai J, Caninci P, Itoh M, Ishii Y, Arakawa T, Shibata K, Shinagawa A, Shinozaki K:拟南芥cDNA全长文库的功能注释。中国科学:地球科学。
- 25.
Seki M,Satou M,Sakurai T,Akiyama K,Iida K,Ishida J,Nakajima M,Enju A,Narusaka M,Fujita M,Shinosaki K:Riken Arabidopsis全长(RAFL)cDNA及其在非生物下表达概况的应用压力条件。J Exp Bot。2004,55:213-223。
- 26.
Ogihara Y, Mochida K, Kawaura K, Murai K, Seki M, Kamiya A, Shinozaki K, carinci P, Hayashizaki Y, Shin IT:六倍体小麦幼穗cDNA文库的构建及其表达序列标签的大规模测序。中国生物医学工程学报。2004,29(4):429 - 434。
- 27。
Sakurai T,Plata G,Rodríguez-zapata F,Seki M,Salcedo A,Toyoda A,Ishiwata A,Tohme J,Sakaki Y,Shinozaki K,Manabu Ishitani M:Shinazaki K,Manabu Ishitani M:Shomabu Ishitani M:Shomabu Ishitani M:Shomabu Ishitani M:Shomabu Ishitani M:Shomabu Ishitani M:Cassava的20.000个全长cDNA克隆的测序分析显示谱系特定基因家庭与压力反应相关的扩展。BMC植物生物。2007,7:66-
- 28.
Umezawa T,Sakurai T,Totoki Y,Toyoda A,Seki M,Ishiwata A,Akiyama K,Kuotani A,Yoshida T,Mocida K,Kasuga M,Todaka D,Maruyama K,Nakahsim K,Enju A,Mizukado S,Ahmend S,Yoshiwara K, Funatsuki H, teraishi M, Osaki M, Shinano T, Akashi R, Sakaki Y, Yamaguchi-Shinosaki K, Shinozaki K: Sequencing and analysis of approximately 40,000 soybean cDNA clones from a full length enriched cDNA library. DNA Res. 2008, 15: 333-346.
- 29.
山崎裕K, Kigawa T,井上M, Tateno M,山崎裕T, T城市,青木M,塞其E,松田T, Nunokawa E,不能Y,田农T,是M,书T,田中,塞其M, Shinozaki K, Yokoyama年代:锌结合主题揭示了解决dna结合结构域的拟南芥SBP-family转录因子。中国生物医学工程学报,2004,32(6):643 - 648。
- 30。
Yamasaki K, Kigawa T, Inoue M, Yamasaki T, Tateno M, Yabuki T, Aoki M, Seki E, Masuda T, Tomo Y, Hayami N, Terada T, Shirouzu M, Osanai T, Tanaka A, Seki M, Shinozaki K,横山S: WRKY dna结合域的解结构。acta botanica sinica(云南植物研究),2017,34(4):457 - 461。
- 31。
Yamasaki K, Kigawa T, Inoue M, Yamasaki T, Yabuki T, Aoki M, Seki E, Matsuda T, Tomo Y, Terada T, Shirouzu M, Tanaka A, Seki M, Shinozaki K,横山S:拟南芥乙烯INSENSITIVE3-LIKE3主要dna结合域的解结构。中国生物医学工程学报,2003,31(4):457 - 461。
- 32.
横山S, Hirota H, Kigawa T, Yabuki T, Shirouzu M, Terada T, Ito Y, Matsuo Y, Kuroda Y, Nishimura Y, Kyogoku Y, Miki K, Masui R, Kuramitsu S:日本结构基因组学项目。自然资源学报。2000,7:943-945。
- 33.
SciRoKo:一种用于全基因组微卫星搜索和研究的新工具。生物信息学。2007,13:1683-1685。
- 34.
Galeano CH, Fernández AC, Gómez M, Blair MW:基于SNP和Indel标记的普通菜豆单链构象多态性的遗传作图和同源性分析(菜豆l .)。中国生物医学工程学报。2009,10:629-
- 35.
Sponchiado BN, White JW, Castillo JA, Jones PG: 4个普通豆品种在不同土壤类型环境下根系生长与耐旱性的关系。农业试验。1989,25:249-257。
- 36.
Yan X,Liao H,Beebe Se,Blair MW,Lynch JP:QTL映射根毛和酸渗出性状及其与普通豆磷吸收的关系。植物和土壤。2004,265:17-29。
- 37.
López-Marín HD, Rao IM, Blair MW:铝胁迫下普通菜豆根系形态性状的数量性状位点(菜豆l .)。acta photonica sinica, 2009, 39(4): 439 -458。
- 38.
Schlueter JA, Goicoechea JL, Collura K, Gill N, Lin J-Y, Yu Y, Vallejos E, Muñoz M, Blair MW, Tohme J, Tomkins J, McClean P, Wing R, Jackson SA: bac端序列分析和普通菜豆物理图谱(菜豆l .)基因组。热带植物学报,2008,1:40-48。
- 39。
CórdobaJM,Chavarro MC,Schleuter JJ,Jackson Sa,Blair MW:通过微卫星标记的常见豆类基因组的物理和遗传地图整合。BMC基因组学。2010,11:436-
- 40。
Blair MW, Pedraza F, Buendia HF, Gaitán-Solís E, Beebe SE, Gepts P, Tohme J:开发一种普通豆子的全基因组锚定微卫星图(菜豆l .)。Al Appl Genet。2003,107:1362-1374。
- 41。
Bassett MJ,Miklas Pn,Caldas GV,Blair MW:Garnet棕色种子外套的主要基因Rk'dorado'常见豆类和映射的轨迹Rk联动组1. Euphytica。2010,176:281-290。
- 42。
mirlas PN, Larsen RC, Riley R, Kelly JD:潜在的标记辅助选择bc-1(菜豆普通花叶病毒抗性)。acta physica sinica, 2000, 32(4): 457 - 461。
- 43。
黄小豆种子铁、锌含量的遗传研究(菜豆l .)。生物学报。2009,23:197-207。
- 44.
Ewing B, Hillier L, Wendl MC, Green P:使用phred自动音序器追踪的基础呼叫。即精度评估。基因组学报,1998,8:175-185。
- 45.
Conesa A,Götzs:Blast2Go:植物基因组学中功能分析的综合套件。Internat J植物基因组。2008,12-第12条ID 619832
- 46.
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华生物医学杂志,1996,16(3):413 - 417。
- 47.
Huang X,Madan A:CAP3:DNA序列装配方案。Genome Res。1999,9(9):868-877。
- 48.
Harris MA, Clark J, Ireland A, Lomax J, Ashburner M, Foulger R, Eilbeck K, Lewis S, Marshall B, Mungall C:基因本体(GO)数据库和信息学资源。核酸学报2004,32(4):497 - 503。
- 49.
Volfovsky N,Haas BJ,Salzberg SL:DNA序列中重复分析的聚类方法。基因组生物学。2001,2:0027.1-0027.11。
- 50.
豆科植物比较基因组学研究进展。三个排序的豆科植物基因组和许多作物物种:丰富的机会翻译基因组学。植物营养与肥料学报。2009,29(4):429 - 434。
- 51.
Varshney RK,Singh NK,Kulwal PL,Penmetsa RV,Saxena RK,Datta S,Rosen B,Farmer广告,Dubey A,Saxena KB,Fakrudin B,Singh Mn,Wanjar Kbi,Kirtian A,May Gd,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,McCombie R,厨师博士:Pigeonpea基因组学倡议(PGI):改善Pigeonpea作物生产力的国际努力(Cajanus Cajan.l .)。生物学报。2009,23:1-16。
- 52。
Kantety RV, La Rota M, Matthews DE, Sorrells ME:大麦、玉米、水稻、高粱和小麦表达序列标签中简单重复序列的数据挖掘。植物科学学报,2002,48(4):451 - 457。
致谢
我们感谢José Polonia和Idupulapati Rao组织了温室实验,感谢Natalia Hurtado在GenBank序列提交方面的帮助,华盛顿大学EST世代测序中心,以及世代挑战计划通过比尔和梅林达·盖茨基金会赠款资助热带豆科I项目。我们也要感谢日本政府和理研所提供的相应资金。
作者信息
从属关系
相应的作者
附加信息
作者的贡献
MWB获得了资金并起草了论文。MI和MWB计划与DM一起进行实验。ACF和SA进行了生物信息学分析。ACF准备的数字和编辑表。DM进行RNA提取物。MS和SS和构建全长cDNA库,并进行了初步测序和分析。所有作者均阅读并批准了手稿。
电子补充材料
12870 _2011_949_moesm1_esm.docx
附加文件1:表格显示了用于开发基因型G19833组织全长库的实验中使用的土壤和灌溉条件。试验包括两种灌溉(充分灌溉vs.极端干旱)处理×三种土壤(Darién, Palmira, Santander de Quilichao)。对BAT477和G19833进行了类似的实验。(多克斯14 KB)
12870_2011_949_moesm2_esm.docx.
附加文件2:图显示了用于收集组织的实验的设计,用于G19833组织的全长文库构建。在从图中右侧的土壤和幼苗,生长尖端,茎,茎,芽,花,花,花,鲜花,小豆荚,茎,花,花,鲜花,小豆荚和根部)进行了组织收集的土壤 - 圆筒生长的植物在摄影中所示的右侧水胁迫下三根土管的插入。对BAT477和G19833进行了类似的实验。(DOCX 16 KB)
12870 _2011_949_moesm3_esm.pdf
附加文件3:图显示了对GenBank数据库的(a)e-value和(b)序列相似性分布的分布。基于从全长cDNA文库测序项目中全部收集到的unigenes。(PDF 90 KB)
12870 _2011_949_moesm4_esm.pdf
附加文件4:表显示全长库中表达的频率(EST计数)的40个顶部基因。所显示的每次Contig的基因同源性。(PDF 136 KB)
作者为图像提交的原始文件
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativeCommons.org/licenses/by/2.0.)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
布莱尔,m.w.,费尔南德斯,a.c., Ishitani, M。等等。豇豆干旱胁迫cDNA文库的构建及EST序列测定(菜豆l .)。BMC植物BIOL.11,171(2011)。https://doi.org/10.1186/1471-2229-11-171
已收到:
接受:
发表:
关键词
- 基因本体论
- 菜豆
- 平均读取长度
- 常见的豆类基因型
- 剪切位点变异