摘要
背景
一组(苏(var) 3 - 9、Enhancer-of-zeste Trithorax)结构域是约130-150个氨基酸的进化上保守的序列,并且构成赖氨酸甲基(KMTs)的催化位点。KMTs执行许多重要的生物学功能via染色质组蛋白甲基化。组蛋白甲基化标记的不同解释取决于组蛋白类型(即H3或H4)、赖氨酸位置(如H3K4、H3K9、H3K27、H3K36或H4K20)和添加的甲基数目(即me1、me2或me3)。例如,H3K4me3和H3K36me3与转录激活有关,而H3K9me2和H3K27me3与基因沉默有关。kmt的底物特异性和活性是由蛋白的SET域和其他区域的序列决定的。
结果
在这里,我们从最近的测序中鉴定了49个SET-domain蛋白芜菁基因组。我们对这些蛋白以及双子叶的set结构域蛋白进行了序列相似性和蛋白结构域分析拟南芥蒂利亚纳,单科科斯栽培稻和Brachypodium distachyon,和绿藻欧斯特激热粉花。我们发现,植物SET结构域蛋白可以分为6个不同类别,即KMT1,KMT2,KMT3,KMT6,KMT7和SET。除了SET类,其具有中断的SET结构域和可能参与非组蛋白甲基化,其它类具有显示出不同的底物特异性的组蛋白甲基转移酶的特性:用于KMT1 H3K9,KMT2为H3K4,KMT3为H3K36,KMT6对于H3K27和KMT7也H3K4。我们还提出了植物SET结构域蛋白一致的,合理的命名。序列相似性和同线性的的比较b·拉伯和答:芥设定域蛋白显示出一些KMTS的最近基因重复事件。
结论
本研究首次对KMT的set结构域蛋白进行了表征b·拉伯.系统发育分析数据使得在植物中这一重要的蛋白质家族的命名具有一致性和合理性,就像在动物中一样。本研究结果将为其他植物kmt的命名提供依据,并有助于这些重要表观遗传调控基因的功能鉴定芸苔属植物作物。
背景
表观遗传调控通过基因组功能的可遗传变化而发生,而不改变DNA序列。一个著名的表观遗传机制是通过组蛋白的翻译后共价修饰;这些修饰包括乙酰化、甲基化、泛素化等,并形成基因调控的“组蛋白代码”的基础[1].组蛋白赖氨酸甲基化在包括异染色质形成、转录调节、亲代印迹和细胞命运决定等广泛的细胞过程中起着关键作用[2].在组蛋白H3(K4,K9,K 27,K36,K79)和一个ON H4(K20)上,至少六个赖氨酸残基,五个赖氨酸残基在H4(K O 2)上进行。每个赖氨酸可以携带一种,两个或三个甲基残基,分别称为单,二 - 和三甲基化。通常,H3K4和H3K36的二甲基化与转录激活相关,而H3K9的二甲基化和H3K27的三甲基化与植物和动物中的基因沉默相关[2那3.].
除H3K79甲基化外,所有已知的赖氨酸甲基化修饰都是由甲基转移酶进行的,甲基转移酶包含一个进化上保守的SET结构域,以三个SET命名果蝇基因(苏(VAR)那E(z),和Trithorax) [4.].设定结构域包括大约130-150个氨基酸,形成结的结结构,构成赖氨酸甲基化的酶催化位点[5.].除了SET结构域外,侧翼序列、更远处的蛋白结构域,可能还有一些辅助因子对酶的活性和特异性也很重要。编码set域蛋白的基因是古老的,存在于原核生物和真核生物中,但已经增殖和进化出与真核生物的出现有关的新功能[6.].
第一个被遗传学鉴定的植物基因编码的set结构域蛋白是卷曲的叶子(CLF),美狄亚(意味着)在拟南芥蒂利亚纳[7.那8.].植物set结构域蛋白的染色质结合特性和组蛋白甲基化活性在烟草NtSET1和NtSET1中首次被报道拟南芥KRYPTONITE(KYP)9.那10].植物SET结构域蛋白的系统发育分析已被证明有助于指导这一大家族蛋白的遗传和分子研究[11那12].迄今为止,一些人拟南芥SET-domain家族成员在开花时间控制、细胞命运决定、叶片形态发生、花器官发生、亲本印记和种子发育等多个过程中发挥着关键作用[3.那13-15].
除了模型植物外,植物种类越来越多的基因组序列(拟南芥蒂利亚纳那栽培稻,和Brachypodium distachyon),亦已完成。其他芸苔属植物物种由于其农业经济的重要性和与之密切的关系而特别引起人们的兴趣拟南芥,从而为最近的SET-domain基因扩增在进化芸苔属植物物种。在这里,我们鉴定并分析了最近完成的49个SET-domain蛋白芜菁全基因组序列[16].我们的数据提供了一个平台,未来的功能表征这些重要的表观遗传调节基因芸苔属植物物种。
结果
中set结构域蛋白的鉴定b·拉伯基因组
使用BLASTp和tBLASTn与已知的全部补体拟南芥和水稻SET-domain蛋白作为查询,我们鉴定了49个编码不同SET-domain蛋白的基因b·拉伯基因组(http://brassicadb.org/brad).我们最近提出了赖氨酸甲基转移酶(KMTS,[17),并为新确认身份的人命名b·拉伯基于我们对其相应蛋白序列的系统发育分析(见下文)。除了1 BrKMT1B;和BrKMT1B; 2 b基因的染色体位置尚不清楚,其余47个基因分布在10个基因上b·拉伯染色体,与1 - 7国民党每条染色体的基因1).
b·拉伯SET-domain蛋白可分为六类
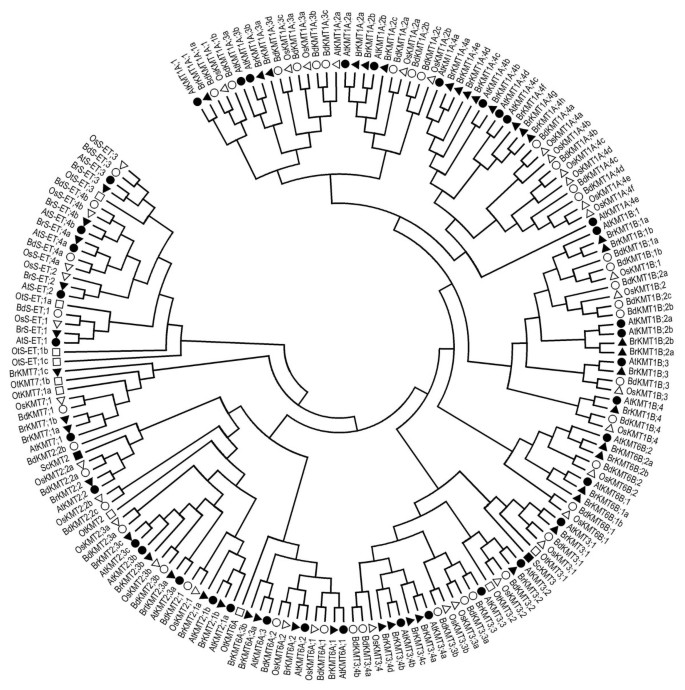
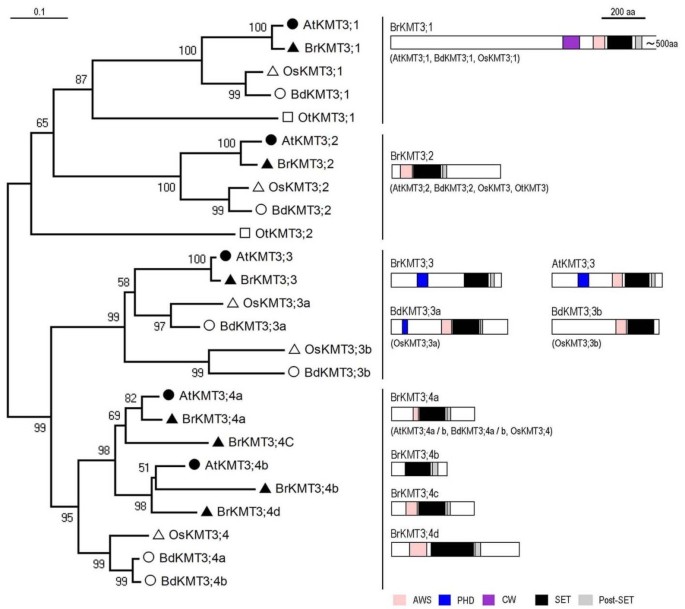
分析b·拉伯SET-domain蛋白序列,我们从其他几个绿色谱系物种中提取了SET-domain蛋白,包括37个答:芥, 36种蛋白质来自o .漂白亚麻纤维卷,从41个蛋白b . distachyon和10个蛋白质Ostreococcus金牛座的(表1).我们还包括酿酒酵母ScKMT2/Set1和ScKMT3/Set2蛋白,分别是H3K4-和h3k36特异性的KMTs [18那19],从进化的角度可以用来代表古代真核生物的SET-domain蛋白。对上述175个set结构域蛋白的系统发育分析表明,它们可以分为6个不同的类,即KMT1、KMT2、KMT3、KMT6、KMT7和S-ET类(图)1).这里使用的前四类编号与先前建议的酵母类和动物类的名称一致[17].此外,KMT1和KMT6被鉴定为两个植物特异性亚类(A和B)。每个类/子类的代表成员可以在答:芥那b·拉伯那o .漂白亚麻纤维卷和b . distachyon.所述SET类成员包含一个中断的SET结构域和可能参与非组蛋白,例如甲基化RUBISCO亚基;然而,它们的生物学功能仍是未知。此后,我们专注于中涉及的组蛋白甲基化国民党类/子类。
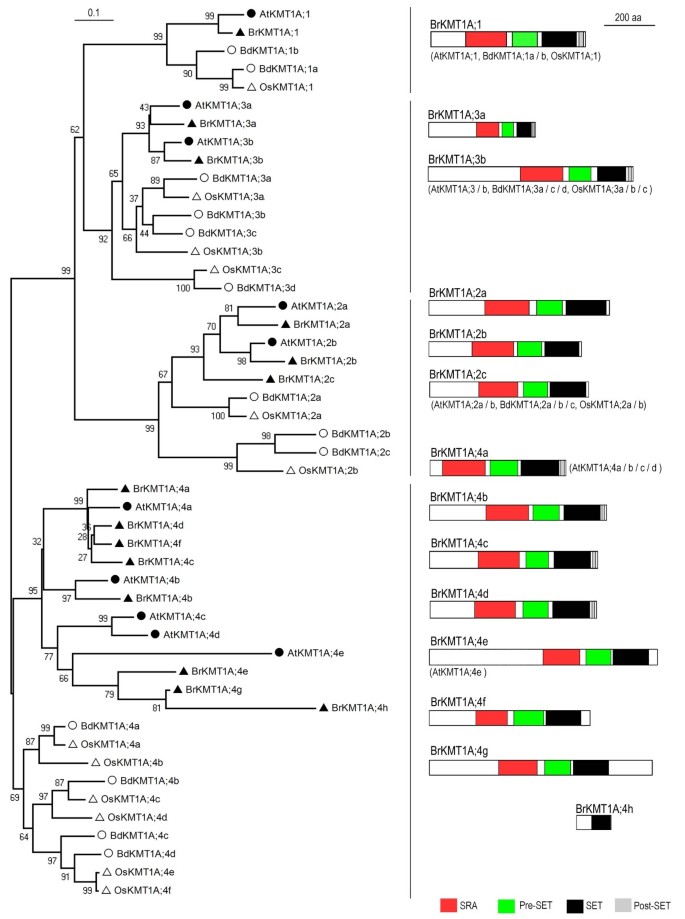
KMT1A亚类蛋白
KMT1A子类是最大的类,可以进一步分为4个不同的组(图)2).在每一组中,双子叶植物(b·拉伯和答:芥)和单像(o .漂白亚麻纤维卷和b . distachyon)明显属于不同的分支,这表明它们源自一个共同的祖先基因,但在单子叶/双子叶分离之前就分化了。前三组的关系相对简单,基因数量也较少,但第4组更复杂:每个植物物种有4-8个成员,它们在进化的不同时期分化。在…的情况下b·拉伯, Group-4的8个成员中,BrKMT1A;4a, BrKMT1A;4c, BrKMT1A;4d, BrKMT1A;4f与拟南芥AtKMT1A; 4 / SDG32 / SUVH1;与AtKMT1A BrKMT1A; 4 b; 4 b / SDG19 / SUVH3;和BrKMT1A;4e, BrKMT1A;4 g和BrKMT1A2).对两者一致性的检查b·拉伯和答:芥(http://brassicadb.org/brad/searchSynteny.php)透露了这一点4 BrKMT1A;那BrKMT1A; 4摄氏度和BrKMT1A;四维但不是BrKMT1A; 4 fsyntenic有AtKMT1A;图4a / SDG32 / SUVH1,和BrKMT1A; 4 h但不是BrKMT1A; 4 e也没有BrKMT1A; 4 g是同步的AtKMT1A; 4 e / SDG11 / SUVH10.由此可见,无论是在染色体段还是单基因规模上,都发生了多次重复事件,导致了较近期的Group-4基因扩增b·拉伯分离后答:芥在进化过程中。与之前的研究一致拟南芥,米饭和玉米[11那12,则很少有内含子存在BrKMT1A基因(额外的文件1:图S1)。最多BrKMT1A基因以ESTs表示,但有些基因在当前数据库中没有任何ESTs(补充文件2:表S1)。我们的RT-PCR分析显示,确实有两个基因缺乏ESTs,2 BrKMT1A;和BrKMT1A; 2摄氏度,非常弱。检测到强烈的表达BrKMT1A; 4,但检测到的表达相对较弱BrKMY1A;四维表达是无法检测的BrKMT1A; 4摄氏度(附加文件3.:图S2)。总之,这些数据表明不同的表达水平BrKMT1A基因差异很大,因此这些基因可能在不同程度上调节基因组功能。
植物KMT1A亚类蛋白与动物KMT1蛋白在SET结构域和周围区域(即Pre-SET和post-SET结构域)具有高度的序列相似性。此外,大多数植物蛋白都含有一个特定的结构域SRA(与SET和RING相关)。与之前研究的相似拟南芥蛋白质(12那20.],大多数BRKMT1A蛋白还包含SRA,预先设定,设定和后域(图2).这些结构域在一些Group-4蛋白中缺失;例如,BrKMT1A。4.e, BrKMT1A;4f and BrKMT1A;4 g lack a Post-SET domain, and BrKMT1A.4 h lacks SRA, Pre-SET and post-SET domains (Figure2).有报道称SRA结构域具有多种功能,包括与组蛋白H3的n端尾部结合和DNA胞嘧啶甲基化[21].AtKMT1A;3a/SDG9/SUVH5的晶体结构揭示了SRA识别CG和CHH序列的甲基化状态[22].预设结构域包含9个保守的半胱氨酸。后设定结构域是一种富含富含半胱氨酸的区域,通常在设定结构域的C末端侧找到。已经显示出预先设定的和置网后结构域来影响设定结构域的组蛋白甲基转移酶活性[23那24].
植物KMT1A亚类的成员,就像动物KMT1蛋白一样,可能负责H3K9甲基化,这是一种涉及异染色质形成和基因沉默的表观遗传标记。与此一致的是,分析AtKMT1A; 1 / SDG33 / SUVH4 / KYP那/ SDG3 / SUVH2 AtKMT1A; 2那ATKMT1A; 3A / SDG9 / SUVH5和AtKMT1A; 3 b / SDG23 / SUVH6已经揭示了它们在H3K9甲基化、异色基因沉默和H3K9与DNA甲基化之间的交叉对话中的重要作用[9.那21那22那25-28].在水稻上的研究也证实了这个亚类的几个成员参与了H3K9甲基化和转座子沉默[29-31].的一些BrKMT1A基因可能也有类似的功能。
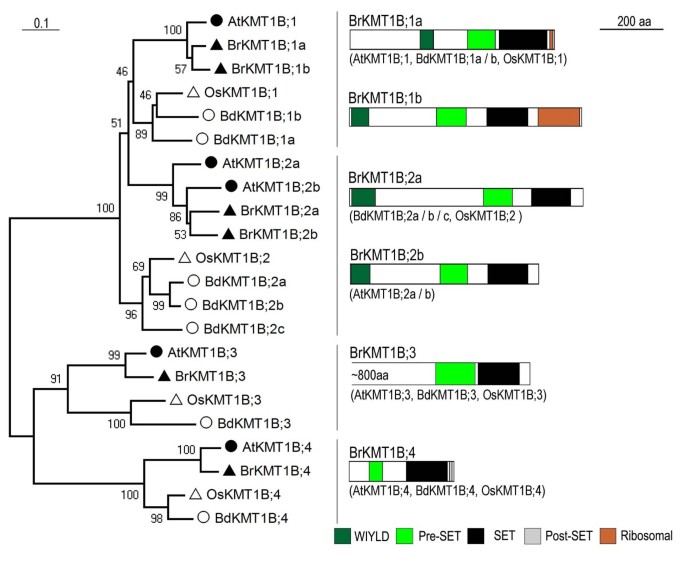
KMT1B亚类蛋白
六个b·拉伯蛋白属于KMT1B亚类,该亚类可进一步分为4类(图1)3.).第1组包含两个b·拉伯蛋白(BrKMT1B;1a和BrKMT1B;1b),其基因与AtKMT1B; 1 / SDG31 / SUVR4.此外,序列分析显示BrKMT1B;1b与AtKMT1B;1/SDG31/SUVR4具有高度相似的蛋白结构域,说明BrKMT1B;1b与AtKMT1BBrKMT1B; 1 b更保守和1 BrKMT1B;在进化中相对较晚的时候分化拉伯/。芥分离。组2有两个b·拉伯,两个答:芥蛋白质,o .漂白亚麻纤维卷蛋白质,和三个b . distachyon蛋白质。这两个2 BrKMT1B;和BrKMT1B; 2 b显示同线性AtKMT1B; 2 b / SDG18 / SUVR2.第3组和第4组各有一个代表成员,在四个被调查的高等植物物种。
KMT1B亚类与KMT1A亚类在蛋白质结构域上存在差异;具体来说,这些蛋白质缺乏SRA结构域(图)3.).最近的一项研究表明,AtKMT1B;1/SDG31/SUVR4具有h3k9 -甲基转移酶活性,并与泛素结合,将H3K9me1转化为H3K9me3沉积在转座子染色质上[32].值得注意的是,与泛素结合的WIYLD结构域在BrKMT1B;1a, BrKMT1B;1b, BrKMT1B;2a和BrKMT1B;2b中是保守的3.).有报道称AtKMT1B;3/SDG6/SUVR5/AtCZS参与了花期的调控,可能是通过花期抑制因子H3K9甲基化的沉积FLC[33].到目前为止,KMT1B子类的其他成员的功能仍未确定。
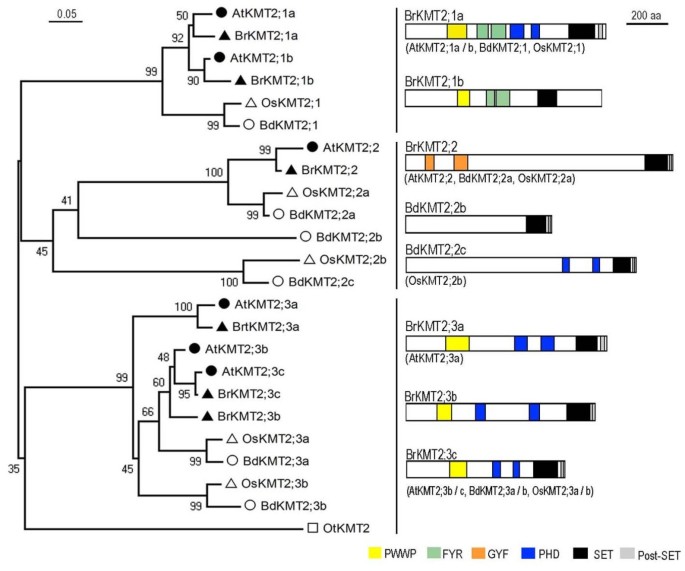
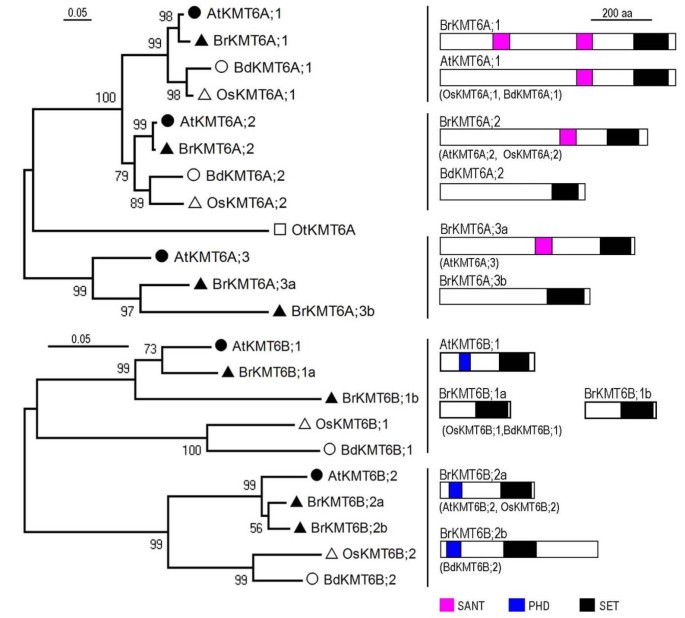
该KMT2类蛋白质
KMT2级包括6个b·拉伯和六个答:芥3组蛋白质(图4.).该类具有高度保守的SET和Post-SET结构域,具有酵母h3k4 -甲基转移酶ScKMT2/Set1。然而,一些植物蛋白在进化过程中获得了特定的结构域,如PWWP、PHD、FYR和/或GYF。PWWP结构域也存在于真核蛋白中,参与DNA甲基化、DNA修复和转录调控[34],并通过介导蛋白-蛋白相互作用调节细胞生长和分化[35].PHD结构域在许多染色质相关蛋白中被发现,并被认为参与了对多蛋白复合物组装非常重要的蛋白质-蛋白质相互作用[36].动物BRPF1蛋白的PWWP结构域结合H3K36me3 [35]和PHD域也是蛋白质的重要模块,其读取组蛋白修饰[37].FYR域由FYR-C和FYR-N端子部分组成,其通常彼此靠近,但也可以分离[38].GYF结构域被认为参与蛋白质相互作用中脯氨酸丰富序列的识别[39].
KMT2组1成员包含一个PWWP,一个FYR和两个PHD结构域。只有属于第1组一名成员在被发现o .漂白亚麻纤维卷或b . distachyon,但两个成员被发现b·拉伯和答:芥.我们的考试显示1 BrKMT2;与之同步AtKMT2; 1 / SDG27 / ATX1,和BrKMT2; 1 b与AtKMT2;图1b / SDG30 / ATX2,这表明它们来自于两种不同的祖先副本拉伯/。芥分离。与此一致的是atx1突变植物表现出强烈和多效性缺陷[40),但atx2突变体植物具有正常的表型[41].的atx2突变可以增强atx1还原反应时的开花阻抑基因的表达FLC通过降低H3K4me3水平FLC基因座[42].
PWWP和FYR结构域在Group-2成员中不存在,PHD结构域仅在一些单子叶蛋白中发现(图)4.).在Dicot物种中只发现了一个组2代表成员b·拉伯或答:芥,而是单花椰菜o .漂白亚麻纤维卷有两个成员b . distachyon有三个成员。的b·拉伯和答:芥蛋白质,以及来自o .漂白亚麻纤维卷和b . distachyon在蛋白质的n端包含一个GYF结构域(图4.).这个结构域在KMT2保守的事实;从所有四个高等植物物种2个蛋白表明GYF域的获取单子叶/双子叶植物分离之前发生,并且可以具有在高等植物的保守功能。遗传分析表明,AtKMT2; 2/SDG25 / ATXR7在防止开花早[必要43那44].重组AtKMT2; 2 / SDG25 / ATXR7蛋白显示出甲醇组蛋白H3在体外和AtKMT2的耗尽; 2 / SDG25 / ATXR7在足底略微降低H3K4和H3K36的甲基化FLC染色质(43那44].OsKMT2;2b/SDG732和BdKMT2;2c包含两个PHD结构域,但BdKMT2;2b与酵母蛋白ScKMT2/Set1一样,不包含可识别的PHD结构域。未来对这些单子叶蛋白的研究可能会对KMT2蛋白的结构域进化提供更深入的理解。
-3-3 kmt2蛋白有一个域组织,更类似于组-1,除了他们缺乏Fyr域(图4.).b·拉伯和答:芥两种植物都有三个第三类成员,但其他两种高等植物都只有两个第三类成员。Synteny被观察到BrKMT2; 3 c与AtKMT2; 3 c / SDG29 / ATX5但不与ATKMT2; 3B / SDG16 / ATX4,建议ATKMT2; 3B / SDG16 / ATX4是由相对较近的一次重复事件衍生出来的。这与之前的研究结果一致ATKMT2; 3B / SDG16 / ATX4和AtKMT2; 3 c / SDG29 / ATX5共线重复吗AtKMT2; 1 / SDG27 / ATX1和AtKMT2;图1b / SDG30 / ATX2[12].到目前为止,没有一个Group-3蛋白的功能特征。
KMT3类蛋白质
KMT3类包含5个成员答:芥但是7个成员b·拉伯,这些可以进一步分为四组(图5.).其他组每个植物物种只包含一个成员,但第4组包含2个成员答:芥和4个成员b·拉伯.我们的检查表明brkmt3; 4a.和BRKMT3; 4C.syntenic有4 AtKMT3;/SDG7 / ASHH3,和BRKMT3; 4B.和brkmt3; 4d.与AtKMT3; 4 b/SDG24 / ASHH4.在当前数据库中找到的ESTs符合所有四个BrKMT3; 4基因(额外的文件2表S1),因此不允许我们区分每个基因的表达。我们的RT-PCR分析表明brkmt3; 4a.和BRKMT3; 4C.在不同的检测器官/组织中表达水平更高,范围更广,而BRKMT3; 4B.和brkmt3; 4d.在一些器官/组织(附加文件3.:图S2)。
KMT3类植物蛋白具有高度的序列相似性,并具有AWS (Pre-SET的一个子域,[45]), SET和Post-SET结构域与酵母h3k36 -甲基转移酶ScKMT3/Set2(图5.).组-1蛋白具有长序列并含有特异于该组特异的额外CW结构域。最近显示ATKMT3; 1 / SDG8 / ASHH2 / EFS / CCR1的CW结构域以结合H3K4ME1 / ME2 [46,表明植物中H3K4和H3K36甲基化之间存在新的联系。AtKMT3;1/SDG8/ASHH2/EFS/CCR1是H3K36me2和H3K36me3沉积所需的主要h3k36 -甲基转移酶,激活包括FLC和加年代(47].AtKMT3;1/SDG8/ASHH2/EFS/CCR1缺失导致多效表型,包括开花早、器官大小减少、枝条分枝增加、育性和类胡萝卜素组成受到干扰,以及植物对病原体的防御能力受损[47-54].另一类KMT3植物蛋白序列较短,不含CW结构域;有趣的是,AtKMT3;2/SDG26/ASHH1的缺失导致了晚开花表型的升高FLC表达式[47].除BdKMT3;3b和OsKMT3;3b/SDG707外,Group-3 KMT3蛋白含有一个PHD结构域;3/SDG4/ASHR3被报道可能通过介导H3K4me2和H3K36me3沉积参与花粉和雄蕊发育[55那56].Group-4蛋白的功能至今仍未被研究。这组人在考试中b·拉伯可能是一个挑战,因为基因增殖和更分散的序列(图5.).
KMT6A亚类蛋白
KMT6A子类包括4个成员b·拉伯和3个特色鲜明的成员答:芥1/SDG1/CLF, AtKMT6A;2/SDG10/EZA1/SWN和AtKMT6A;3/SDG5/MEA,代表三个不同的组(图6).ATKMT6A; 1 / SDG1 / CLF和AtKMT6A; 2 / SDG10 EZA1 / SWN在营养和生殖发育的调控中广泛表达和部分冗余,而ATKMT6A; 3 / SDG5 / MEA似乎专门在配子体和种子发展中发挥作用[7.那8.那57].这三个拟南芥蛋白质可以作为进化保守的Polycomb压抑复合体2 (PRC2)的关键组成部分,它三甲基化H3K27参与转录抑制[57].AtKMT6A、1/SDG1/CLF和AtKMT6A、2/SDG10/EZA1/SWN分别代表类群1和类群2,各类群在不同的高等植物物种中都有同源性。3/SDG5/MEA属于group 3,在单子叶中没有同源物,但在中有两个同源物b·拉伯.这两个3 BrKMT6A;和BrKMT6A; 3 bsyntenic有ATKMT6A; 3 / SDG5 / MEA和BrKMT6A;3b相比,BrKMT6A;3a与AtKMT6A;3/SDG5/MEA具有更相似的蛋白结构域组织BrKMT6A; 3 b可能是在答:芥/b·拉伯在进化过程中分离。
SANT (SWI3, ADA2, N-CoR和TFIIIB dna结合)结构域存在于大多数植物KMT6A亚类蛋白中。该区域也存在于许多其他具有多种活性的染色质重塑蛋白中,如dna结合、组蛋白尾结合和蛋白-蛋白相互作用[58].然而,目前尚不清楚SANT结构域在KMT6A蛋白中的确切作用。值得注意的是,BrKMT6A;3b不包含SANT域。如预期的ATKMT6A; 3 / SDG5 / MEA在生殖过程中只有少数细胞表达,两者均表达3 BrKMT6A;和BrKMT6A; 3 b在检查的组织中勉强可检测到(附加文件3.:图S2)。这将是有趣的调查3 BrKMT6A;和BrKMT6A; 3 b并检查这两个基因在功能上是否重要。
KMT6B亚类蛋白质
中的KMT6B子类包括两个成员答:芥那b . distachyon和o .漂白亚麻纤维卷,四名成员b·拉伯,它们可以分为两个不同的群体(图6 b).从两名成员答:芥1/SDG15/ATXR5和AtKMT6B;2/SDG34/ATXR6在首次基因组分析中被归类为三胸相关拟南芥SET-domain蛋白(11].然而,我们的研究以及之前两项包含更完整的植物set -domain蛋白集的研究清楚地表明,AtKMT6B;1/SDG15/ATXR5和AtKMT6B;2/SDG34/ATXR6属于KMT6B亚类(图)1) [12那20.].与此相一致的是,功能分析显示AtKMT6B;1/SDG15/ATXR5和AtKMT6B;2/SDG34/ATXR6参与了H3K27的单甲基化[59].它们似乎是冗余的,因为H3K27单甲基化的耗尽仅在ATXR5 ATXR6.双突变体(59].kmt6a介导的H3K27me3主要存在于正色区,对基因沉默起重要作用[58而kmt6b介导的H3K27me1则存在于异染色质中心,对异染色质凝聚和复制起重要作用拟南芥[60].
不同于含有SANT结构域的KMT6A蛋白,许多植物KMT6B亚类蛋白都含有PHD结构域(图)6.).既AtKMT6B的PHD结构域; 1 / SDG15 / ATXR5和AtKMT6B; 2 / SDG34 / ATXR6强烈结合未甲基化的H3尾部的肽(氨基酸1-21),并且该结合带负通过甲基化上H3K4 [受影响60].这种结合偏好可能有助于确保这些KMT6B蛋白不是针对常染色质和富含H3K4甲基化的活性基因。值得注意的是,组1和组2成员都是重复的b·拉伯.这两个1 BrKMT6B;和BrKMT6B; 1 bsyntenic有AtKMT6B; 1 / SDG15 / ATXR5,和两个2 BrKMT6B;和BrKMT6B; 2 b与AtKMT6B; 2 / SDG34 / ATXR6.的表达1 BrKMT6B;那2 BrKMT6B;和BrKMT6B; 2 b在不同的组织中检测到,但我们没有检测到BrKMT6B; 1 b表达式(附加文件3.:图S2)。推测是合理的1 BrKMT6B;那2 BrKMT6B;和BrKMT6B; 2 b可能有冗余功能。
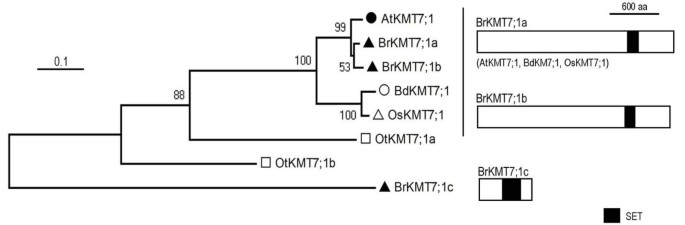
KMT7类蛋白质
KMT7类包含一个成员答:芥那o .漂白亚麻纤维卷和b . distachyon,但三个成员b·拉伯(图7.).虽然拟南芥在以前的一些研究中,1/SDG2/ATXR3被认为与KMT2类成员有关[11那20.],在施普林格及其同事的系统发育树分析中,它被定位在任何类之外[12],我们的分析显示,它与其他一些绿色谱系蛋白聚在一起,形成植物KMT7类(图1).与上面描述的不同,植物和动物的KMT7类并不聚类,尽管它们在H3K4甲基化中具有相似的功能。动物KMT7类的代表只在哺乳动物中发现,包括人类SET7/9,它使H3K4单甲基化,也使许多非组蛋白甲基化[17].植物KMT7蛋白与人类SET7/9序列没有最高的相似性,AtKMT7蛋白缺失;1/SDG2/ATXR3导致H3K4me3蛋白整体减少,导致孢子体和配子体发育中多性缺陷[61那62].这两个1 BrKMT7;和BrKMT7; 1 b但不是BrKMT7; 1 c有同线性AtKMT7; 1 / SDG2 / ATXR3,系统发育分析显示BrKMT7;1a与AtKMT7;1/SDG2/ATXR3更接近。RT-PCR分析表明BrKMT7; 1 b表达在高于1 BrKMT7;(附加文件3.:图S2)。鉴于……的重要作用AtKMT7; 1 / SDG2 / ATXR3,这将是有趣的研究角色1 BrKMT7;和BrKMT7; 1 b组蛋白甲基化与植物发育的关系b·拉伯.
讨论
在过去的10年里,一些SET-domain基因在拟南芥并且在水稻中发挥了关键的基于染色质的功能via植物生长发育过程中的组蛋白甲基化[3.那15].然而,植物set结构域蛋白的命名仍然很复杂,许多植物有多个同义词拟南芥蛋白(表1),这可能导致这一重要领域具有相当大的混乱。基于序列相似性的命名有几个优点,通知其组蛋白赖氨酸残基的KMT酶底物特异性,并在其全基因组序列可用后,在生物体中提供KMT类型的全球视图。然而,SDG命名法未能提供有关酶衬底特异性的信息,并且“SDG”之后的数量可能是长而难以记住的[12那63],例如,第一个SDGb·拉伯(ID = 197)将被命名为SDG19701。虽然Baumbusch及其同事的命名法提供了与动物蛋白同源性的信息,但不完整的列表拟南芥SET-domain蛋白和(当时)只有一个植物物种进行全基因组分析的限制限制了本研究中系统发育分组的精确性和正确性[11].此外,国民党的动物命名也不连贯;最近才提出一个合理的命名法[17].因此,我们在此提出的命名符合该领域的最新进展。
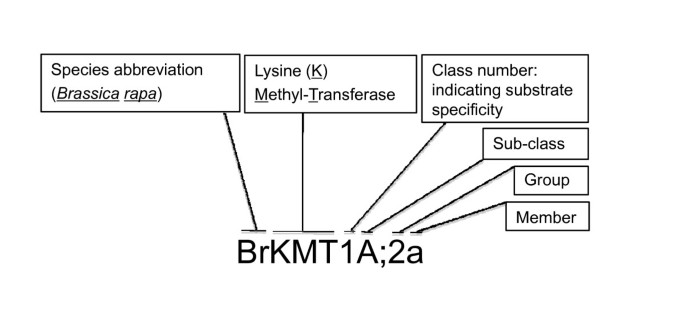
按照植物基因委员会命名委员会的指导方针[64],植物kmt的命名是由种首字母定义的(例如:Br为Brasica拉伯)KMT,其后是类号(图之前8.).类数是基于表明酶底物特异性的酵母和动物系统,即KMT1表示H3K9, KMT2表示H3K4, KMT3表示H3K36, KMT6表示H3K27, KMT7也表示H3K4 [17].多个子类由大写字母表示(例如KMT1A和KMT1B),类/子类内的不同组由阿拉伯数字后缀表示(例如KMT1A;1)。组内成员用小写字母表示(例如KMT1A;1a和KMT1A;1b)。亚群目前还没有定义,但在未来可能会被指定为功能分析,越来越多的测序基因组显示出物种之间的序列保存或一个确定的国民党群体的几个成员具有不同的功能。特定亚群后缀的使用应表明几个物种之间高度相似的序列或等价的功能角色。新的命名法可能很难被采纳拟南芥因为许多SET-domain蛋白的原始名称对研究人员来说是很熟悉的,但由于对国民党的巨大兴趣,对不同物种的一个连贯和合理的命名是重要和有用的。这里提出的指导方针对新发现的set结构域蛋白的命名特别有用,随着基因组序列可用于其他植物物种,这些蛋白正以指数级增长的速度被发现。
我们从最近完成的全基因组序列中鉴定了49个SET-domain蛋白b·拉伯.其中,5个蛋白质属于可能参与非甾酮蛋白甲基化的S-ET类,20蛋白属于潜在涉及H3K9甲基化的KMT1级,6个蛋白质属于潜在参与H3K4甲基化的KMT2类,7个蛋白质属于KMT3阶级可能涉及H3K36甲基化,8种蛋白质属于潜在涉及H3K27甲基化的KMT6类,并且3属于KMT7类,也可能参与H3K4甲基化。这一点在硅片这一研究对今后对这一重要的表观遗传调控家族的功能分析是有帮助的芸苔属植物.H4K20甲基化检测拟南芥利用抗体(28那65,但所涉及的催化酶尚不清楚,目前的系统发育分析也无法预测涉及H4K20甲基化的特定国民党类。有可能是上述国民党类的一些成员催化H4K20甲基化。国民党的总人数b·拉伯(49)略高于在答:芥(37)o .漂白亚麻纤维卷(36)和B。distachyon(41)。然而,我们不能排除在现有的基因组序列中可能会少一些国民党的可能性b·拉伯.
基因复制是基因组和遗传系统进化的主要驱动力之一,被认为是建立新基因功能和产生进化新颖性的主要机制[66那67].与预期的染色体数量复制相反b·拉伯相比答:芥,国民党基因的数量b·拉伯(49) much than…的数量的两倍答:芥国民党(74)。许多重复的基因与它们的基因表现出同步性答:芥同系物,这表明它们是从染色体/基因组节段的重复的。三种替代的结果可以在复制基因的进化发生:(i)一种拷贝可以简单地变得被退行性突变(nonfunctionalization)沉默;(ii)一个拷贝可以获取新的,有益的功能和成为由自然选择(neofunctionalization)保存;(ⅲ)两个副本可以通过突变变成部分受到损害,使得它们的总容量增加了到单拷贝的祖先基因(subfunctionalization)的容量[66].这些不同的结果可能适用于不同的国民党复制基因,从他们的表达模式判断3.:图S2)。的表达BrKMT1A; 4摄氏度和BrKMT6B; 1 b是无法检测到的,说明它们可能是非功能化的。重复的对2 BrKMT1B;和BrKMT1B; 2 b那BRKMT3; 4B.和BrKMT3; 4 d,或1 BrKMT7;和BrKMT7; 1 b在植物器官中有差异表达,表明它们可能获得了不同的组织特异性功能。最后,一些重复基因的表达,例如。2 BrKMT1A;和BrKMT1A; 2摄氏度那1 BrKMT1B;和BrKMT1B; 1 b,或3 BrKMT6A;和BrKMT6A; 3 b显示出类似的模式,表明它们可能是次功能化的和/或具有冗余功能。
在显示基因重复的组中b·拉伯,值得注意的是拟南芥那ATKMT6A; 3 / SDG5 / MEA对亲本基因印迹和种子发育至关重要[8.那68],AtKMT6B; 1 / SDG15 / ATX5和ATKMT6B; 2 / SDG34 / ATX6是重要的异染色质凝聚和复制拟南芥[59那60],和AtKMT7; 1 / SDG2 / ATXR3对于两个孢子体和配子体发育必不可少[61那62].这将是极大的兴趣来研究这些基因组为他们的调节和功能的染色质组织,植物生长和发育b·拉伯.
结论
我们的研究表明,植物的set结构域KMT蛋白可以被系统划分为不同的类别,而涉及组蛋白甲基化的类别可以按照动物和酵母set结构域KMT的命名法进行命名。在不同的生物体中这样一个连贯和合理的命名将有助于避免因同一蛋白质或基因的多个名称的存在而引起的混淆。提供的信息b·拉伯KMTs也将有助于进一步阐明表观遗传调控机制芸苔属植物作物。
方法
SET-domain蛋白质识别
来源的SET-domain蛋白序列答:芥那o .漂白亚麻纤维卷那o .金牛座的和酿酒酵母从染色质数据库中分别从染色体数据库中检索物种数据库中的关键词SDG(ChromDB,http://www.chromdb.org).这些序列,主要来自答:芥和o .漂白亚麻纤维卷,被用作搜索的疑问b . distachyon基因组(http://www.brachypodium.org)和b·拉伯基因组(http://brassicadb.org/brad/index.php),使用BLASTp和tBLASTn工具(http://blast.ncbi.nlm.nih.gov).Expect阈值设置为1.0,其他参数设置为默认值。我们没有使用严格的e值阈值;相反,我们检查每个结果命中是否存在SET或S-ET域,以收集以前未识别的序列。使用在线查看器工具(http://brassicadb.org/brad/index.php).est序列的b·拉伯SET-domain蛋白基因从芸薹属植物数据库(http://brassicadb.org/brad/index.php)及NCBI (http://blast.ncbi.nlm.nih.gov), Expect阈值为1,最小序列长度为50 bp。
蛋白质结构域分析
利用NCBI-CD搜索技术对蛋白序列进行结构域分析(http://ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi).关闭低复杂性过滤器,并将Expect值设置为1.0,以检测该分析中保守性较低的短域或区域。根据SMART数据库(http://smart.embl-heidelberg.de/).
系统发育分析
使用ClustalW比程序[进行SET结构域序列的多重序列比对69].结果文件使用MEGA4.0程序进行系统发育分析[70].树木采用以下设置构建:树推理作为邻居加入;为每个类分析的总序列分析和完整的删除选项包含作为成对删除选项的站点。替代模型:泊松修正;和引导测试1,000用于内部分支的可靠性。
rt - pcr分析
b·拉伯植物在18-22°C条件下生长,光照时间为12 h (10,000 Lx)/12 h暗光周期。叶片采自2周、4周、6周、8周或10周的植株;根和茎采自6周龄的植株;花蕾采自10周大的植株。使用Trizol试剂(Invitrogen,美国)从收集的约100 mg植物组织中提取总RNA。RNA制备用dnase (Promega, USA)在37°C处理30分钟,然后在65°C孵育5分钟酶失活。使用RT-PCR试剂盒(RevertAid™First strand cDNA Synthesis Kit, Fermentas, CA)制作第一链cDNA。含有第一链cDNA的rt溶液保存在-80℃。用于RT-PCR反应的引物列于附加文件4.:表S2。PCR反应的条件如下:94℃3分钟;然后30 s,50-63℃的30℃,30℃,72℃持续30℃,1分钟;最后72℃持续8分钟。PCR产物以1.5%(w / v)琼脂糖三硼酸盐/ EDTA缓冲凝胶分离,并被溴化乙锭染色可视化。
参考文献
- 1.
斯特拉尔BD,Allis CD:共价组蛋白修饰的语言。自然。2000,403:41-45。
- 2.
马丁C,张Y:组蛋白赖氨酸甲基的多种功能。NAT Rev Mol Cell Biol。2005年,6:838-849。
- 3.
刘超,吕芳,崔旭东,曹旭:高等植物组蛋白甲基化的研究进展。植物营养与肥料学报,2010,31(6):643 - 646。
- 4.
Tschiersch B, Hofmann A, Krauss V, Dorn R, Korge G, Reuter G:编码的蛋白质果蝇位置效应杂色抑制基因苏(var) 3 - 9结合了抗拮抗基因复合物的拮抗稳压因子。Embo J. 1994,13:3822-3831。
- 5.
钱超,周明明:集结构域蛋白赖氨酸甲基转移酶的结构、特异性和催化作用。中国生物医学工程学报。2006,31(6):755- 762。
- 6.
Alvarez-Venegas R, Sadder M, Tikhonov A, Avramova Z:细菌SET域基因的起源:垂直还是水平?生物科学进展。2007,24(4):482-497。
- 7.
古德里奇Ĵ,Puangsomlee P,马丁男,龙d,迈耶罗维茨EM,科普兰德G:阿多梳组基因调节在同源异型基因表达拟南芥.自然。1997,386:44-51。
- 8.
王志强,王志强,王志强:胚胎发生的母系控制美狄亚,一个多梳群基因拟南芥.《科学》,1998,28(4):446-450。
- 9.
KRYPTONITE组蛋白H3甲基转移酶对CpNpG DNA甲基化的控制。自然资源学报。2002,416:556-560。
- 10.
Shen WH:NTSET1,含有植物域域蛋白的新鉴定的亚组的成员,是染色质相关的,其异位过表达抑制烟草植物生长。工厂J. 2001,28:371-383。
- 11.
鲍姆布施,托尔斯坦森,克劳斯V,菲舍尔A,诺曼K, Assalkhou R, Schulz I, Reuter G,艾伦RB: The拟南芥蒂利亚纳基因组包含至少29个编码SET结构域蛋白的活性基因,这些基因可划分为4个进化保守类。核酸学报2001,29(4):419 -433。
- 12.
施普林格NM, Napoli CA, Selinger DA, Pandey R, Cone KC, Chandler VL, Kaeppler HF, Kaeppler SM:玉米和玉米SET结构域蛋白的比较分析拟南芥揭示了在单子叶和双子叶分化之前的多重重复。植物生理学报。2003,32(6):927 - 934。
- 13.
Pien S, Grossniklaus U: Polycomb组和三胸组蛋白拟南芥.Acta botanica sinica(云南植物研究),2007,34(6):643 - 648。
- 14.
沈文辉,徐丽:染色质重塑在干细胞维持中的作用拟南芥蒂利亚纳.植物学报。2009,2:600-609。
- 15.
沈文辉:植物发育过程中转录激活的组蛋白修饰。生物化学学报,2011,37(4):569 - 572。
- 16.
太阳王X,王H,王J, R,吴J,刘年代,白Y, Mun JH,班克罗夫特,程F,黄年代,李X,华W,王J,王X, Freeling M,皮雷JC,帕特森啊,Chalhoub B, B,海沃德,夏普AG)、公园BS, Weisshaar B,刘B, B,刘B,通C, C, C杜兰,彭C, C耿,Koh C, C林,爱德华兹D,μD,沈D, Soumpourou E, F, F弗雷泽,柯南特G, Lassalle G,国王GJ, Bonnema G,唐H, H, Belcram H,周H, Hirakawa H,安倍H,郭H, H,金H,帕金IA, Batley J, Kim JS J,李J,徐J,邓J,金正日是的,李J, Yu J,孟J,王J, Min J, J,也不会王J, K畠山直哉,吴K, L,方L,技巧,链接毫克,赵M,金米,Ramchiary N, Drou N,伯克曼PJ,蔡问,李黄Q, R, Tabata年代,程年代,张,张年代,黄,佐藤年代,太阳年代,Kwon SJ,老崔,李TH,风扇W,赵X, X,徐X, Y,邱Y,阴Y, Y, Y Du,廖Y, Y Lim, Narusaka Y, Y小王,小王Z,李Z,王Z,熊Z,张Z: mesopolyploid作物物种的基因组芜菁.NAT Genet。2011年,43:1035-1039。
- 17.
Allis CD, Berger SL, Cote J, Dent S, Jenuwien T, Kouzarides T, Pillus L, Reinberg D, Shi Y, Shiekhattar R, Shilatifard A, Workman J, Zhang Y:染色质修饰酶的新命名。电池学报。2007,31(4):633-636。
- 18.
布里格斯SD,Bryk男,施特拉尔BD,祥WL,戴维JK,登特SY,温斯顿楼艾利斯CD:组蛋白H3赖氨酸4甲基化是由SET1介导的和细胞生长和其rDNA沉默中所需要酿酒酵母.基因工程学报,2001,15(4):388 - 388。
- 19.
Strahl BD, Grant PA, Briggs SD, Sun ZW, Bone JR, Caldwell JA, Mollah S, Cook RG, Shabanowitz J, Hunt DF, Allis CD: Set2是一个核小体组蛋白h3选择性甲基转移酶,介导转录抑制。中国生物医学工程学报。2002,22(4):593 - 598。
- 20.
王涛,王涛,王涛。植物SET结构域蛋白的结构、功能和调控。Acta botanica sinica(云南植物研究),2007,34(6):743 - 748。
- 21.
SRA甲基胞嘧啶结合区域连接DNA和组蛋白甲基化。生物谷学报。2007,17:379-384。
- 22.
Rajakumara E, Law JA, Simanshu DK, Voigt P, Johnson LM, Reinberg D, Patel DJ, Jacobsen SE: 5mC识别的双翻转机构拟南芥SUVH5 SRA结构域及其对DNA甲基化和H3K9二甲基化的影响在活的有机体内.基因工程学报,2011,25:137-152。
- 23.
Rea S, Eisenhaber F, O’carroll D, Strahl BD, Sun ZW, Schmid M, Opravil S, Mechtler K, Ponting CP, Allis CD, Jenuwein T:位点特异性组蛋白H3甲基转移酶对染色质结构的调控。自然科学。2000,406:593-599。
- 24.
Tachibana M, Sugimoto K, Fukushima T, Shinkai Y: Set domain-containing protein, G9a,是一种新型的偏爱赖氨酸的哺乳动物组蛋白甲基转移酶,对组蛋白H3的赖氨酸9和赖氨酸27具有高活性和特异性。中国生物医学工程杂志。2001,27(6):593 - 598。
- 25.
Malagnac F, Bartee L, Bender J: An拟南芥SET结构域蛋白是维持DNA甲基化所必需的,而不是建立DNA甲基化所必需的。国家自然科学基金青年项目,2001,21(4):491 - 498。
- 26.
bbs ML, Bartee L, Bender J:通过SUVH6和SUVH4甲基转移酶的联合作用,H3赖氨酸9甲基化维持在转录的反向重复序列上。中国生物医学工程学报。2005,25(4):457 - 461。
- 27。
Ebbs ML, Bender J:基因座特异性控制的DNA甲基化拟南芥SUVH5组蛋白甲基转移酶。acta botanica sinica, 2017, 36(4): 497 - 503。
- 28。
Naumann K, Fischer A, Hofmann I, Krauss V, Phalke S, Irmler K, Hause G, Aurich AC, Dorn R, Jenuwein T, Reuter G:关键作用AtSUVH2在异色组蛋白甲基化和基因沉默拟南芥.Embo J. 2005,24:1418-1429。
- 29。
Wang X, Wang X, Su L, Zhai J, Cao S, Zhang D, Liu C, Bi Y, Qian Q, Cheng Z, Chu C, Cao X: SDG714,一种组蛋白H3K9甲基转移酶TOS17水稻DNA甲基化与转位。植物学报。2007,19:9-22。
- 30。
丁B,朱Y,卜ZY,沉WH,俞Y,董AW:SDG714通过H3K9二甲基化调控特定基因表达,进而影响植物生长。植物营养与肥料学报,2010,29(6):643 - 646。
- 31。
秦福军,孙启伟,黄丽梅,陈新新,周德兴:水稻SUVH组蛋白甲基转移酶基因在染色质修饰和反转录转座子抑制中的特异功能。植物科学学报,2010,33(3):773-782。
- 32。
VEISETH SV,Rahman MA,YAP KL,Fischer A,EGGE-JACOBSEN W,REUTER G,周MM,AALEN RB,Thorstensen T:SUVR4组蛋白赖氨酸甲基转移酶结合泛素并将H3K9ME1转化为拟南芥转座染色质的H3K9ME1至H3K9ME3。Plos Genet。2011,7:e1001325-
- 33.
Krichevsky A, Gutgarts H, Kozlovsky SV, Tzfira T, Sutton A, Sternglanz R, Mandel G, Citovsky V: C2H2锌指- set组蛋白甲基转移酶是一种植物特异性染色质修饰剂。Dev Biol. 2007, 303: 259-269。
- 34.
邱C,Sawada K,Zhang X,Cheng X:哺乳动物DNA甲基转移酶DNMT3B的PWWP领域定义了一种新的DNA结合折叠系列。NAT STRUCT BIOL。2002,9:217-224。
- 35.
Vezzoli A,Bonadies N,Allen MD,Freund SM,Santiveri Cm,Kvinlaug Bt,Huntly BJ,GOTTGENS B,Bycroft M:BRPF1的PWWP领域的组蛋白H3K36Me3识别的分子基础。NAT STRUCT MOL BIOL。2010,17:617-619。
- 36.
Aasland R, Gibson TJ, Stewart AF: PHD手指:染色质介导的转录调节的含义。生物化学学报。1995,20:56-59。
- 37.
云美,吴杰,Workman JL,李波:组蛋白修饰的读者。细胞生物学杂志,2011,21(4):564-578。
- 38.
Schultz J, Copley RR, Doerks T, Ponting CP, Bork P: SMART:一个基于网络的研究基因移动域的工具。核酸学报2000,28(4):431 - 434。
- 39.
Kofler MM, Freund C: GYF域。中国海洋大学学报(自然科学版),2006,33(2):437 - 441。
- 40.
alvarez - veneas R, Pien S, Sadder M, Witmer X, Grossniklaus U, Avramova Z:ATX-1,一个拟南芥同族体的trithorax,激活花的同源基因。黄志强。2003,13:627-637。
- 41.
saleh a,alvarez-venegas r,yilmaz m,le o,hou g,vadder m,al-abdallat a,xia y,lu g,Ladunga i,avramova z:高度相似拟南芥同系物的trithoraxATX1和ATX2编码具有不同生化功能的蛋白质。acta botanica sinica(云南植物研究),2008,29(5):531 - 534。
- 42.
Pien S, Fleury D, Mylne JS, Crevillen P, Inze D, Avramova Z, Dean C, Grossniklaus U:拟南芥TRITHORAX1动态调整对象开花轨迹C通过组蛋白3赖氨酸4三甲基化激活。acta botanica sinica, 2017, 36(5): 569 - 572。
- 43.
Berr A, Xu L, Gao J, Cognat V, Steinmetz A, Dong A, Shen WH:设置域GROUP25编码组蛋白甲基转移酶并参与开花轨迹C开花的激活和抑制。植物营养与肥料学报。2009,31(4):591 - 598。
- 44.
Tamada Y Yun JY, Woo SC, Amasino RM:拟南芥TRITHORAX-RELATED7是组蛋白H3的赖氨酸4的甲基化所必需的,并用于转录活化开花轨迹C.acta botanica sinica, 2017, 36(4): 457 - 461。
- 45.
Doerks T,Copley Rr,Schultz J,Ponting CP,Bork P:系统鉴定与核职能相关的新型蛋白质领域家庭。Genome Res。2002,12:47-56。
- 46.
Hoppmann V,Thorstensen T,克里斯坦森PE,Veiseth SV,拉赫曼MA,Finne K,阿伦RB,奥斯兰R:该CW域,在染色质蛋白新组蛋白识别模块。EMBO J. 2011,30:1939年至1952年。
- 47.
Xu L, Zhao Z, Dong A, Soubigou-Taconnat L, Renou JP, Steinmetz A, Shen WH:组蛋白H3赖氨酸36的二甲基化和三甲基化而不是单甲基化标志着参与开花时间调控和其他过程的基因的活跃转录拟南芥蒂利亚纳.中国生物医学工程学报。2008,28(4):457 - 461。
- 48.
Kim SY, He Y, Jacob Y, Noh YS, Michaels S, Amasino R:建立春化反应,冬季-年度习惯拟南芥需要推定的组蛋白H3甲基转移酶。植物细胞。2005,17:3301-3310。
- 49.
赵智,于宇,Meyer D,吴超,沈文华:通过表达对早花的抑制作用开花轨迹C需要组蛋白h3k36的甲基化。细胞生物学杂志。2005,7:1256-1260。
- 50.
董刚,马丹平,李军:组蛋白甲基转移酶SDG8调控植物分枝拟南芥.Biochem Biophys Res Communce。2008,373:659-664。
- 51。
Cazzonelli CI, Cuttriss AJ, Cossetto SB, Pye W, Crisp P, Whelan J, Finnegan EJ, Turnbull C, Pogson BJ:类胡萝卜素组成和分枝的调控拟南芥通过染色质修饰组蛋白甲基转移酶,SDG8。植物细胞。2009,21:39-53。
- 52。
Grini PE, Thorstensen T, Alm V, Vizcay-Barrena G, Windju SS, Jorstad TS, Wilson ZA, Aalen RB: ASH1 HOMOLOG 2 (ASHH2)组蛋白H3甲基转移酶是胚珠和花药发育所必需的拟南芥.公共科学图书馆。2009,4:e7817-
- 53。
Berr A,McCallum EJ,Alioua A,Heintz D,Heitz T,Shen Wh:拟南芥组蛋白甲基转移酶设定结构域组8介导尿酸盐/乙烯途径基因的诱导植物防御反应对坏养殖真菌。植物理性。2010,154:1403-1414。
- 54。
Palma K,Thorgrimsen S,Malinovsky FG,FIIL BK,Nielsen HB,Brodersen P,Hofius D,Petersen M,Mundy J:AutoImunity拟南芥acd11是由免疫受体的表观遗传调节介导的。PLOS POAROG。2010,6(10):E1001137-
- 55。
Cartagena JA, Matsunaga S, Seki M, Kurihara D, Yokoyama M, Shinozaki K, Fujimoto S, Azumi Y, Uchiyama S, Fukui K拟南芥SDG4通过对成熟花粉中组蛋白H3赖氨酸4和36的甲基化调控花粉管生长。[j] .环境科学学报,2008,31(4):457 - 461。
- 56。
Thorstensen T,Grini Pe,Mercy是,Alm V,Erdal S,Aasland R,Aalen RB:the拟南芥SET结构域蛋白ASHR3参与雄蕊发育和交互与bHLH转录因子ABORTED孢子(AMS)。植物分子生物学。2008年,66:47-59。
- 57.
郑斌,陈旭:组蛋白H3赖氨酸27三甲基化在植物发育中的作用。植物营养与肥料学报。2011,14:123-129。
- 58.
acta physica sinica, 2017, 36(4): 553 - 558 .张丹,马志强,陈志强,等。SANTA结构域在真核生物中应用的研究进展。生物信息学。2006,22:2459-2462。
- 59.
雅各布y,feng s,勒布朗卡,伯纳拉维琴领yv,stroud h,cokus s,johnson lm,pellegrini m,jacobsen se,michaels sd:Atxr5和Atxr6是染色质结构和基因沉默所需的H3K27单甲基转移酶。NAT STRUCT MOL BIOL。2009,16:763-768。
- 60。
陈志刚,陈志刚,陈志刚:组蛋白H3 -赖氨酸27甲基转移酶对DNA复制的调控作用。自然科学进展。2010,466:987-991。
- 61。
Berr A, McCallum EJ, Menard R, Meyer D, Fuchs J, Dong A, Shen WH:拟南芥SET DOMAIN GROUP2H3K4三甲基化需要,对孢子体和配子体发育至关重要。植物细胞。2010年,22:3232-3248。
- 62.
郭玲,于勇,Law JA,张旭:组蛋白H3赖氨酸4三甲基转移酶的主要结构域GROUP2拟南芥.美国国家科学院学报。2010年,107:18557 - 18562。
- 63.
aqua F, Vega A, Timmermann T, Poupin MJ, Arce-Johnson P:葡萄藤SET结构域家族的全基因组分析。植物营养与肥料学报,2011,30(4):457 - 461。
- 64.
价格加利福尼亚州:植物基因委员会命名委员会。植物MOL BIOL REP。1993,11:273-274。
- 65.
异染色质蛋白与异染色质基因沉默的调控拟南芥.植物营养与肥料学报。2006,29(6):741 - 746。
- 66.
通过亚功能化保存重复基因的概率。遗传学报。2000,54:459-473。
- 67.
Cannon Sb,Mitra A,Baumgarten A,Young Nd,5月G:节段性和串联基因重复在大型基因家族中的演变中的作用拟南芥蒂利亚纳.植物学报。2004,4:10-
- 68.
Luo M, Bilodeau P, Koltunow A, Dennis ES, Peacock WJ, Chaudhury AM:控制受精不依赖种子发育的基因拟南芥蒂利亚纳.美国国家科学院学报。1999年,96:296-301。
- 69.
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、特定位置间隙惩罚和权重矩阵选择来提高累进式多序列比对的灵敏度。核酸学报1994,22(4):493 - 498。
- 70.
Tamura K, Dudley J, Nei M, Kumar S: MEGA4:分子进化遗传学分析(MEGA)软件版本4.0。生物科学进展。2007,24:1596-1599。
致谢
我们感谢王小武博士,吴武和芬辰,从中国农业科学院蔬菜和鲜花研究所提供序列,帮助同时分析。本工作得到了中国国家基础研究计划(973计划,2012CB910500)和中国国家自然科学基金(NSFC31071129和NSFC31071455)的支持。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
YH进行了大部分实验并起草了手稿;CL参与了RT-PCR实验,参与了手稿的起草;WHS和YR构思并指导了这项研究,并撰写了最终版本的手稿。所有作者阅读并批准了最终的手稿。
作者为图像提交的原始文件
以下是与作者的原始提交的图像的链接。
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
黄,Y.,刘,C.,沉,WH。等等。系统发育分析与分类芜菁SET-domain蛋白质家族。BMC植物杂志11,175(2011)。https://doi.org/10.1186/1471-2229-11-175
收到:
接受:
发表:
关键词
- 染色质
- 组蛋白
- 赖氨酸甲基化
- 设置域
- 基因复制
- 命名法