摘要
背景
非生物胁迫,如水分亏缺和土壤盐度,导致葡萄生理、养分利用和营养生长的变化,最终影响酿酒葡萄浆果的产量和风味,葡萄L.生成、策划和分析大规模表达序列标签(ESTs),以确定负责应激适应反应的主要遗传决定因素。尽管根是许多类型的非生物胁迫的第一个感知和/或损伤部位,但迄今为止,暴露在非生物胁迫下的酿酒葡萄根组织的EST测序非常有限。为了克服这一限制,对暴露于多种非生物胁迫的根组织进行了大规模EST测序。
结果
从受非生物胁迫的葡萄树的叶片、浆果和根组织中获得了62236个表达序列标签(ESTs),并与从20个公共cDNA文库中测序的32286个ESTs进行了比较。策展,以纠正注释错误,聚类和装配浆果和叶子est与目前可用的诉酿酒用葡萄全长转录本和ESTs共获得13278个独特序列,其中2302个单基因,10976个已映射序列诉酿酒用葡萄基因模型。其中739个转录本在胁迫叶片和浆果中有显著差异表达,其中250个基因之前未被描述为非生物胁迫响应基因。在第二次分析中,从暴露于多重短期非生物胁迫的根的标准化根cDNA文库中提取16,452个ESTs,根据其在根中相对于其他组织的EST丰度,鉴定出135个具有根富集表达模式的基因。
结论
在不同的非生物胁迫条件下,来自葡萄酒葡萄叶片、浆果和根组织的23个不同cDNA文库的相对EST频率的大规模分析揭示了不同的、组织特异性的表达模式,以前未被识别的胁迫诱导基因,以及许多具有根丰富mRNA表达的新基因,这有助于提高我们对葡萄酒葡萄根生物学和砧木性状操作的理解。基于EST文库富集表达模式的mRNA丰度估计显示,微阵列和定量、实时逆转录-聚合酶链反应(qRT-PCR)方法之间只有适度的相关性,突出了对深度测序表达分析方法的需求。
背景
酿酒葡萄基因功能的研究(葡萄由于两个黑皮诺品种(无性系115和PN40024)的全基因组序列的可用性,已经从根本上提高了[1,2]以及基于bac的物理地图[3.].为了研究葡萄酒葡萄基因的功能,已经开发了多种转录组学方法[4,5],包括表达序列标签[6],大规模并行签名测序(MPSS) [7],小RNA深度测序[8], Illumina测序[9],以及多个寡核苷酸微阵列平台[10- - - - - -13].
大多数诉酿酒用葡萄品种对盐度胁迫的敏感程度中等[14- - - - - -17与Cl-阴离子毒性对生长和葡萄健康的影响最大[18].相比之下,诉酿酒用葡萄相对耐水分亏缺胁迫。调亏灌溉可用于抑制葡萄生长,而不会对果实产量产生显著影响。据报道,调亏灌溉可通过提高包括花青素和原花青素在内的多种代谢产物来改善葡萄品质[19- - - - - -22].酿酒葡萄浆果在发育过程中和对非生物胁迫效应的响应中mRNA和酶的表达谱已被深入研究[11,12,23- - - - - -30.].更多的研究检查了叶片和芽组织中响应非生物胁迫的mRNA表达模式[10,31],植物-病原体相互作用[13,32,33],以及相关事件葡萄属芽内休眠[34- - - - - -36].
陆生植物的根是获取水和必需矿物质的重要器官。因此,对于许多类型的非生物胁迫,包括缺水、盐碱化、营养缺乏和重金属,根是感知和/或损伤的第一个部位[37- - - - - -39].葡萄属根还积累了许多独特的二苯乙烯和低聚二苯乙烯防御化合物,这些化学物质在种子或其他富含植物抗毒素的组织中没有发现[40,41].尽管根的重要性,研究诉酿酒用葡萄与对浆果组织的研究相比,根组织的研究相当有限。在一项比较EST研究中,Moser和同事从诉酿酒用葡萄简历。并发现它们富含在初级代谢和能量功能方面的基因[42].使用12 K CombiMatrix定制阵列,Mica和同事分析了microRNAs (miRNAs)的表达,这种小的(19-24 nt)非编码rna在多个器官中转录后负性调节基因表达。本研究表明,根中有9种和4种miRNAs相对于叶片和早期花序的相对丰度分别显著增加或减少[8].利用抗性和易感杂交,还为酿酒葡萄开发了一个框架物理或遗传图谱,以定位与根病原根瘤蚜抗性相关的遗传决定因素[43].EST转录谱分析最近被用于鉴定可能与抗药有关的基因根瘤菌葡萄在半抗葡萄属杂交'Tamnara' [44].
在葡萄中,已经产生和分析了超过35万个EST序列,以确定与葡萄浆果发育等广泛过程相关的基因表达[30.,45]和table grape [46]、组织特异性基因表达[6,42],以满足休眠葡萄芽的冷却需求[34],以及对病原体抗性的特征,如Xylella fastidiosa[47),根瘤菌葡萄[44].为了辨别稳态转录本积累在多种环境胁迫处理下的变化,我们从受到非生物胁迫(例如,盐度、冷、热、水分亏缺和缺氧)的葡萄树的叶片和浆果组织中总共生成了45784条ESTs。这些结果与来自20个图书馆的32286份ESTs进行了比较,这些ESTs来自公共数据库中保存的树叶和浆果组织。叶片和浆果est的聚类和组装与所有可用诉酿酒用葡萄全长转录本和ESTs共返回了13278个独特序列,其中2302个单例和10976个簇映射到已知的基因模型。在这10976个独特的聚类中,发现739个转录本在所检查的文库中有显著的表达差异。的比较在网上通过Affymetrix获得的转录丰度估计的数字表达分析葡萄属GeneChip®基因组微阵列和定量实时逆转录聚合酶链反应(qRT-PCR)表明EST频率计数与微阵列或qRT-PCR分析基本一致。鉴于葡萄根组织中可用的ESTs相对缺乏,从年轻葡萄藤(10厘米长)的根中测序了16,452个ESTs,这些葡萄藤生长在非胁迫条件下,以及在寒冷、盐度和水分不足胁迫下。确定了根组织中表达的主要基因类别,鉴定出135个具有根特异性或高度富集的根表达模式的基因。
结果
植物非生物胁迫组织EST文库分析葡萄
cDNA文库来源于植物非生物胁迫叶片(文库ID 10208)和浆果(文库ID 12435)诉酿酒用葡萄简历。对霞多丽(Chardonnay)的基因进行测序,分别生成24,400和21,384条ESTs1).此外,从品红盒生长的cv根组织中合成的归一化cDNA文库中,共测序了16452条ESTs。赤霞珠(Cabernet Sauvignon)暴露在受控、水分亏缺、寒冷和盐度胁迫条件下(详见方法部分)(库号22274)。总共生成了66236个表达序列标签(ESTs)(表1).叶子和浆果库在前面的花和浆果发育的背景下已经描述过[6].此外,UniGene数据库中还有5个非胁迫叶文库,共代表8642条ESTs; 13个全浆果,其种子文库来自于浆果发育的不同阶段的非胁迫源组织,共代表31840条ESTs; 2个根文库,共代表1657条ESTs [48]进行编译(表1).这些EST集合被用作工具,以识别编码叶片和浆果中的非生物胁迫响应转录本以及根特异性或根富集转录本。
为了创建最新的注释,每个EST与对应的“暂定共识”(TC) contig序列相匹配葡萄基因指数(VvGI,第6版,2008年7月30日,达纳法伯癌症研究所)[49]并从Genoscope 8.4X中预测肽序列葡萄简历。黑比诺(GSVIV)基因组组装,2007年8月8日[1].在进行这项分析之后,发布了一个更新的VVGI版本(7.0,4/17/2010)。然而,该版本与6.0版本基本相似,包含同样的25,497个来自NCBI RefSeq源的基因模型,只有4851个额外的ESTs,预计不会显著改变所提出的结果。更新的12X覆盖草案葡萄基因组也变得可用。然而,在这一12X草稿中注释的一些基因模型被发现含有更多频率的内含子-外显子剪接,而EST证据不支持(数据未显示),因此,这一12X草稿没有被使用。由于混合胁迫归一化根文库是使用一种归一化技术生成的,实际上,这将减少最丰富的转录本的表观表达,而且由于很少有其他非胁迫根ESTs可用于比较,因此根EST文库中的基因的特征在单独的分析中进行。
识别EST冗余
从EST频率推断的基因表达模式估计,EST频率是基因x转录本的次数我与所有基因的随机观察总数(x我/∑x)时,从5'和3'两个方向测序的单个克隆的est必须精确计数一次,以避免高估基因频率。cDNA文库测序策略因来源不同而不同,一些ESTs仅由单次5'或3'读取生成,而其他文库则对所选克隆进行双向和/或同向重测序。在非生物胁迫叶文库(文库ID 10208)中,2802个ESTs被测序两次(代表1401对reads)。另外2250份ESTs测序3次(750个三胞胎)。消除这种冗余使EST总数从24,400个ESTs减少到21,499个唯一克隆(表2)1).将配对克隆末端的GSVIV基因标识符与来自同一转录本的多个ESTs的基因标识符一致的期望进行比较。从该非生物胁迫叶文库中获得的1401对无性系中,有114对(7%)与不同基因的注释相匹配,保留了置信度最高的ID。同样,该文库中750个三复制克隆中只有60个(8%)在基因身份上存在分歧。来自胁迫浆果文库(文库ID 12435)的ESTs的总克隆冗余度和基因分配错误率相似。在这个EST集合中,2402个ESTs被测序了两次(代表1201对),1821个ESTs被测序了三次(607个三胞胎),两个克隆各被测序了四次。消除这种冗余使EST总数从21,384个ESTs减少到18,963个唯一克隆(表1).在1201对和607个三拷贝中,分别有107对(9%)和68对(11%)不一致。
这种冗余消除方法被扩展到UniGene数据库中获得的非胁迫叶片和浆果文库的双向测序克隆(表2)1).在叶子库(Library id: 12752、12753、12948和12949)和浆果库(Library id: 12754、13015、13016和13017)的注释组合中发现了许多错误。所做的错误和修正如下图所示1并汇总于表2.对于赤霞珠叶片文库CA48LN(文库ID为12948),我们能够将1486个ESTs组织成743对reads。在这些对中,> 68%(509)不能归属于同一基因。同样,在表中列出的其他库中也发现了较高的分歧率2.由于这些错误率高于从非生物胁迫的叶片或浆果库中成对读取的错误率,因此进一步研究了这些较高错误率的原因。
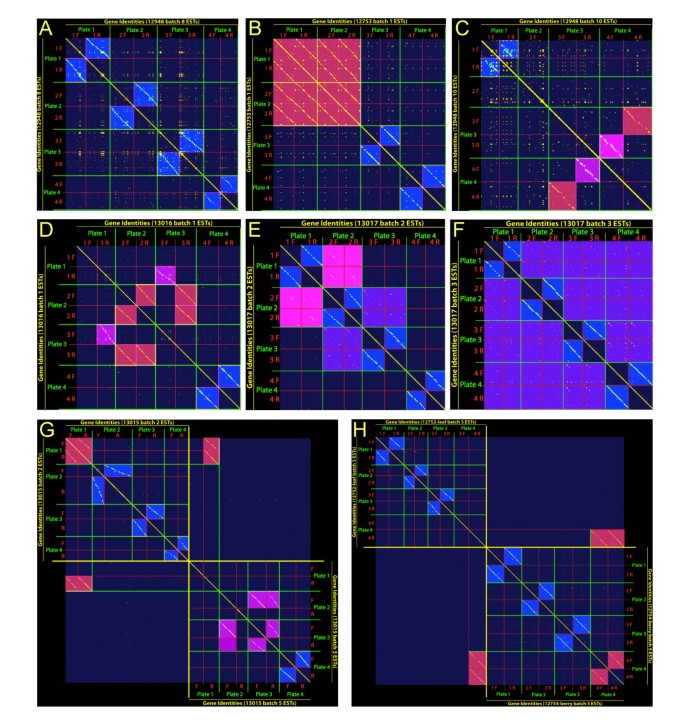
用点图纠正双向序列叶和浆果库中的错误EST标识.从双向测序文库中分配给ESTs的Contig名称在二维上绘制,以识别类似于点图序列比对的“自相似性motif”。从dbEST提交的文件中,将测序批、板序和井位汇总为顺序列表,排列为1f 1r 2f 2r 3f 3r 4f 4r,并在x轴和y轴上与自身相对。一)对角线表示编号为12948的四组图版,第8批已命名并配对正确(蓝色);B)库号12753,第一批,所有板的组合1f 1r 2f而且2 r是重复的(鲑鱼色),盘子3和4配对正确(蓝色);C)库ID 12948,第10批板1f匹配1r(蓝色),板2f和2r不匹配,板3f匹配4r(鲑鱼色),板4f匹配3r(洋红色);D)Berry Library ID 13016, batch 1, plate 3r匹配2f和2r (salmon), 1r匹配3f(品红),1f没有匹配,plate 4配对正确(蓝色);E)Library ID 13017, batch 2, plate 1和2显示部分匹配(粉色),plate 2和3也部分匹配(紫色);F)Berry Library ID 13017 batch 3,四个板块部分匹配(紫色);G)Berry Library ID 13015, batch 2, plate 1匹配batch 5 plate 1r (salmon);右下象限(洋红色)也有明显的板匹配错误;H), Leaf Library ID 12752,第5批,4r plate匹配Berry Library ID 12754,第5批,4fr (salmon)。
cDNA文库见表2是双向测序的,并有注释,允许部分重建工作流,它们最初是通过工作流准备和测序的[50,并将克隆名称存入NCBI,如“CA48LN09IF-A9, 5'end”。该注释标识了“CA48LN”库,批号(“09”),该批中的板(I), 96孔板(A9)上的位置和方向(5’)。一个文库中的所有ESTs共享文库茎,批一般包含4个板(I-IV), 80%的板从5'和3'两个方向测序。将ID 12948文库的正向和反向ESTs对按其96孔板孔序(A1,A2,…,A12,…,H1,…,H12)排列,识别出各种“井滑”模式,其中A9(5’)孔的基因ID与A10(3’)孔的基因ID匹配,A10(5’)孔的基因ID与A11(3’)孔的基因ID匹配,等等。这些“井滑”的距离既不均匀也不一致。
为了确定井对不正确的所有ESTs对,设计了一种方法,可以可靠地识别非均匀距离的“井滑”,类似于局部核苷酸序列比对的点图方法[51].在这种方法中,est的基因id从A1-H12排列为每个5'和3'板,并沿两条轴绘制,基因id相同的地方用点表示(图2)1).点阵图被证明可以有效地识别带有“井滑”的板中的正反向配对,如图所示1,其中ID 12948叶库中“09批次”的四块正、反板沿×轴和y轴依次为1f、1r、2f、2r、3f、3r、4r。等分图的主对角线,其中有序列表与自身相同,两侧是四条偏移对角线,说明正向和反向板对匹配的位置(1f≈1r, 2f≈2r,等.).通过ID 12948库中的变量“well slip”,匹配清楚地区分了对板。
对表中所列库的所有印版批次(通常为每批4个正版和4个反版)重复此匹配过程2以及库ID 12948中出现的“well slip”之外的其他错误类型。一些板块被复制,如图所示1 b,其中四个单独板的正、反组合均匹配于berry Library ID 12753 (1f≈1r≈2f≈2r)。如果这些错误没有被识别出来,1号板和2号板的ESTs都将被加到其中基因的频率总数中(即,计算两次应该只计算一次的基因),导致文库中这些转录本的频率被高估。其他成对的板块显示出不太完整的复制模式,如图中板块1和板块2(粉红色)之间以及板块2和板块3(紫色)之间的早期对角线1 e,以及图中所有四个板(紫色)1 f.在其他情况下,一个板不匹配注释的反向,而是一个不同的板,例如图中Library ID 12948 (3f≈4r和4f≈3r)的对交换1 c和berry Library ID 13016的三倍(2f≈2r≈3r) /错配(3f≈1r)(图1 d).一旦发现,这些部分重复和不匹配的板被处理,就像完全重复一样,减少EST计数以反映所涉及的独立克隆的真实数量。
然后,同样的分析方法被扩展到将库中的每个板与该库中的所有其他板进行比较。发现了另一种意外匹配的情况,其中来自一个批次的板与同一库中不同批次的板匹配(图1克).最后,我们扩展了该方法,将每个库中的每个板与所有其他库中的所有板进行比较,甚至是那些注释为来自不同组织的板。由此,发现了一个单一实例,其中叶子库ID 12752 (plate 4r)中的一个板与浆果库ID 12754中的一对板相同(图1 h).这些板上编码的基因与成熟浆果文库中发现的基因一致(例如细胞壁蛋白、成熟相关蛋白和无光合作用基因),但没有叶片文库,这导致了cDNA文库错配的结论,并导致这些数据从我们的分析中排除。为了发现其他可能的文库分配错误,我们将本研究中所有文库中的每个板与所有其他文库(例如,芽组织、叶柄、花和病原体感染的叶片)进行了比较,这些文库未被考虑用于我们的非生物胁迫分析,但没有检测到进一步的虚假配对(数据未显示)。在详尽地识别所有可观察到的错误模式后,将5' est与其3'伙伴配对,并统计每个文库中唯一的克隆(表21).从表中列出的库中,总共发现了23351个ESTs中的5558个(24%)的识别/注释错误2.
EST频率估计基因表达
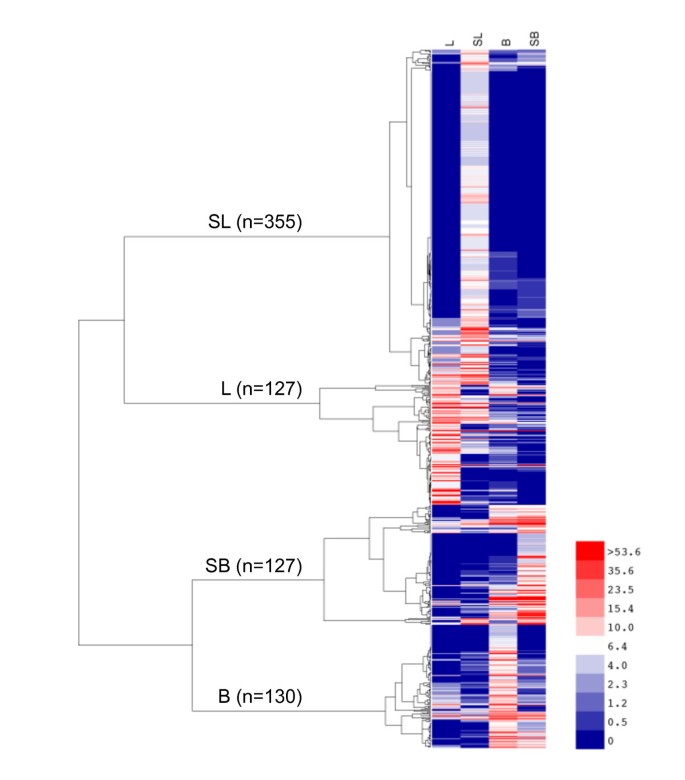
为了测量胁迫和非胁迫叶片和浆果之间基因表达模式的差异,计算每个GSVIV基因ID(或UniGene ID,在没有GSVIV基因模型的情况下)内的EST频率为每个叶片、浆果、胁迫叶片和胁迫浆果库。5个叶片库的EST频率和13个浆果的EST频率均采用加权平均值[52].然后使用IDEG6 web工具,使用基因的EST频率联合计数来计算差异基因表达[53].卡方检验(χ2)作为检验统计量,这是在进行两个以上组的统计比较时所建议的[54].在p值临界值< 0.001时,比较的文库中估计有739个基因存在差异表达。然后使用Pearson相关系数函数作为距离度量和平均聚类方法对739个基因进行分层聚类组织(图2).基因组首先聚集在组织类型之间,如树突图中的第一个分支所示,然后按对照或非生物应激条件聚集,如接下来的两个分支所示。在这个距离上,四个簇通常与单一文库类型的转录本丰度分布相关,最大的簇有355个转录本,对应于胁迫叶片(SL)组织。叶片集群(L)含有127个基因,而胁迫浆果集群(SB)和非胁迫浆果集群(B)分别含有127和130个基因。所有739个基因的注释、基因模型和相对频率在附加文件中按集群列出1,2,3.而且4.在胁迫叶片簇中存在的大量转录本可能反映了该文库的测序深度,这些源组织所遭受的非生物胁迫的多样性,以及在非生物胁迫下葡萄叶片转录组中表达的转录本的多样性[10].
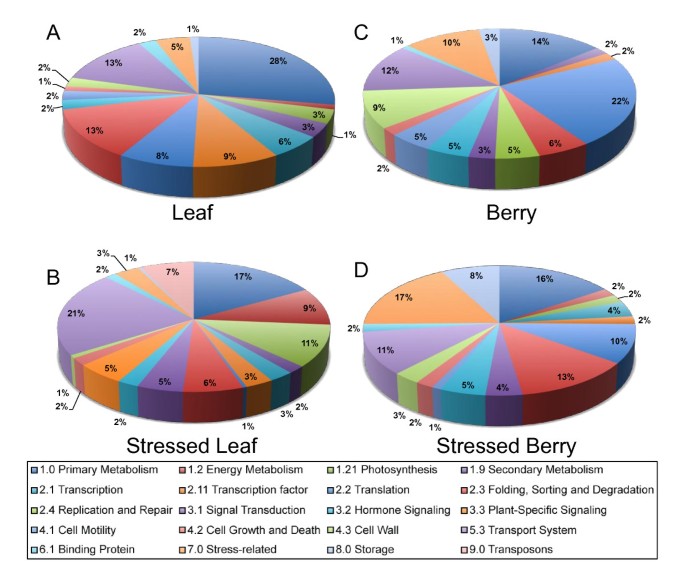
在这739个cDNA文库簇之间存在差异表达的基因中,有637个成功匹配到GSVIV基因/蛋白质标识符,然后将其与相关的注释文件进行匹配葡萄属净(55].葡萄属网状网络按其主要网络分类,代谢网络分为初级代谢、光合作用、次生代谢和激素生物合成,后者与激素信号类归为一类。“网络外”的基因id,但是在葡萄属网络主列表也被纳入功能类别的指定。在图3.,显示了在四个主要簇中鉴定的基因的功能类别。
EST频率分析鉴定差异表达转录本的功能类别.在四个主要的差异表达基因簇中发现了基因的功能分配。在选择的层次深度/距离上,四个簇在很大程度上对应于其中的最大频率一个叶,B)受压叶,C)浆果,和D)胁迫Berry cDNA文库。作业是根据在葡萄属网http://www.sdstate.edu/aes/vitis/pathways.cfm[93].图表颜色从顶部顺时针排列。
在没有过度解释的情况下,每个器官/疾病中突出的基因功能类别之间的一些关键差异是很明显的。例如,无应力叶(图3)的主要特征是初级代谢基因的比例很大(28%),而一些光合作用基因,如RUBISCO小亚基和可塑性光合电子传递成分的比例极其高。非特异性脂质转移蛋白、金属硫蛋白、早期光诱导蛋白(ELIP1)和几个未知基因的转录本也与23S rRNA(附加文件)在该簇中高度代表2).在胁迫叶片中,11%的转录本编码光合作用相关功能,包括可塑性ATP合酶和电子传递链亚基,这表明在胁迫下可能会发生更高的要求和/或损伤,必须修复(图3 b).与这一观点一致的是几个低分子热休克蛋白家族的过度代表性。在非生物胁迫下的叶片表达了更大比例的特定转运基因(21%)(附加文件)1).有趣的是,在受胁迫的叶片中,转座子的活性明显被解除抑制,这可以从着丝粒特异性类反转录转座子的优势(7%)来判断。在茄科植物的非生殖系组织中,已经描述了类似的逆转录因子的非生物诱导,在Tnt1A启动子的aba诱导拟南芥[56].未受胁迫的浆果簇具有编码基因的转录本,这些基因的功能涉及初级代谢、翻译、细胞壁相关蛋白(9%)和运输(12%)(图)3 c,附加文件4).相比之下,受胁迫的浆果簇(图3 d)具有最高比例的基因注释为“应激反应”(17%),包括编码木葡聚糖内转葡萄糖酶/水解酶、DEAD box RNA解旋酶、种子存储蛋白(包括白蛋白和球蛋白)和几种高度丰富的未知蛋白(附加文件)3.).
与微阵列数据的相关性
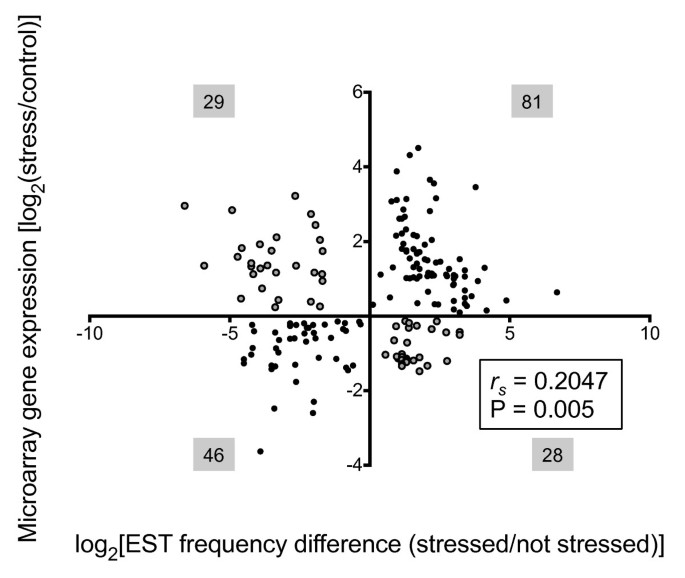
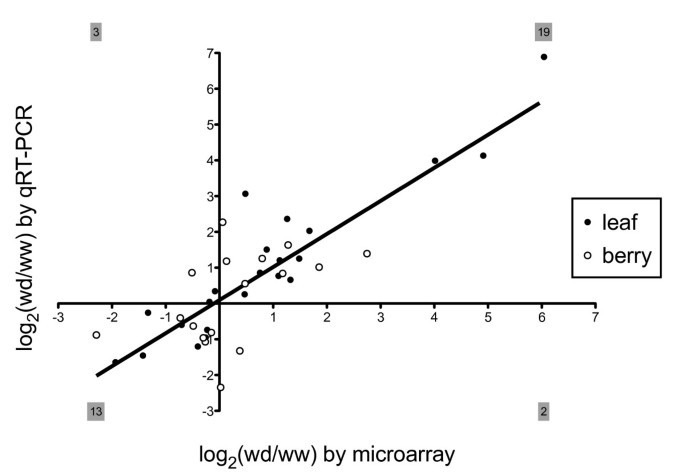
接下来,通过EST频率估计的转录本表达模式的差异与第二个平台Affymetrix进行比较®葡萄属GeneChip®微阵列。在上面描述的739个转录本中,可以为其中的489个分配微阵列探针标识符。收集了所有来自微阵列实验的差异表达基因,在这些实验中施加了类似的压力。对于叶片组织,其中我们的胁迫叶片库包括干旱、NaCl、热和光胁迫组织的混合物,我们使用两个实验作为微阵列数据的来源:一个实验中,干旱和盐胁迫施加超过16天的时间[10]以及一项分析渗透胁迫(甘露醇)、NaCl和低温暴露下基因表达快速变化(≤24小时)的实验[31].对于浆果库,来自霞多丽和赤霞珠干旱胁迫浆果时间过程实验的微阵列数据[27]与EST频率数据进行比较。以van Ruissen及其同事为例[57],然后将探针组表达值与EST频率进行比较,仅使用在原始微阵列实验中在应力组织和非应力组织中观察到显著差异的探针组。使用这种方法,绘制了184个显著不同变化的比较(图4).微阵列和基于频率的表达测量之间的总体相关性是适度的。非参数斯皮尔曼秩相关为适度正相关,在(r年代= 0.2),但P < 0.005,这表明这种相似性虽然不大,但极不可能仅仅是由于偶然。Pearson相关性相似(r= 0.21)。在将微阵列与EST或类似的基于标签的技术进行比较的其他研究中,观察到适度的Spearman和Pearson相关性[58].以Li及其同事为例,确定了184个芯片或EST采样检测方法共有的显著基因的定向一致性,即在应激处理下增加或减少相对转录丰度的定向一致性。在将SAGE标签与多个人体组织中的微阵列进行比较时,这些作者发现重要基因之间有75%的方向一致性[58].同样,在我们的184个共有基因中,方向性一致性为69%,或者说两个以上的一致性对应两个以上的差异。
为了验证微阵列分析确定的基因表达率,对附加文件中列出的基因进行qRT-PCR5.这些基因是随机选择的,代表了在叶片或浆果组织中优先表达的基因。分别在干旱胁迫和水分充足的浆果组织和叶片组织中检测了17和22个转录本的相对mRNA表达。对数的线性回归2这些基因的-比值发现,通过微阵列和qRT-PCR方法测量的转录丰度之间存在很强的相关性(Pearson相关,r= 0.85)和高度的方向一致性(34/39基因或87%)(图5).
根系富集基因的鉴定
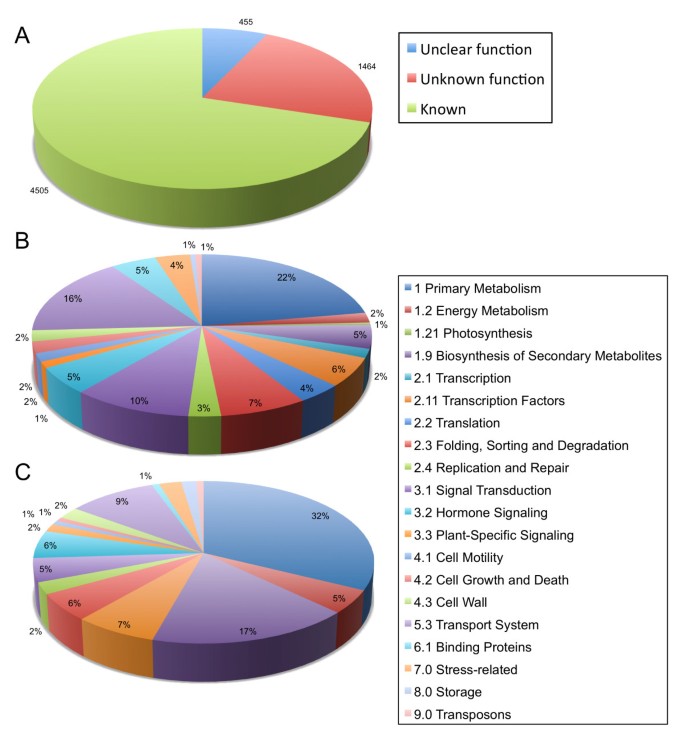
从归一化非生物胁迫赤霞珠根cDNA文库(VVM)中测序得到的16452个ESTs序列与它们进行了匹配Vv胃肠道版本。6一致性序列contigs [59],并在可能的情况下,将8.4X基因组GSVIV基因/蛋白质标识符,并与相关的注释文件匹配葡萄属净(55],结果识别出6424个非冗余转录本。其中,6002例成功映射到8.4X GSVIV基因模型,而其余307例和115例为单例VvGI contigs与GSVIV基因模型不匹配。cDNA文库归一化方法成功地生成了一个高度复杂的文库,其中3449个(54%)唯一的转录本由EST单子表示。6424个非冗余根转录本的注释显示4505(70%)具有已知功能,455(7%)与先前注释的基因模型匹配,但功能不清楚,1464(22.8%)具有未知功能,与任何先前描述的基因没有同源性匹配(图6).对4505个已知功能的转录本进行了功能分类6 b).总的来说,VVM标准化库包含了高度多样性的转录本,其中初级代谢、信号转导和运输系统的功能类别得到了很好的代表(图6 b).
VVM根cDNA文库和富根亚群中基因的功能分类.对赤霞珠根EST文库VVM的基因进行了功能分配葡萄属净注释。A) VVM中功能不明确、未知或已知的基因比例葡萄属净注释;B) VVM中上述“已知”类别中所有4505个基因的功能分类;C)根据Audic-Claverie测试估计的135个转录本在根组织中差异表达的功能分配[60].
接下来,16452个VVM赤霞珠根ESTs加上来自两个赤霞珠根库的1657个ESTs(库号14445,16696;表格1)进行根特异性或根富集转录本分析。这些根cDNA文库与NCBI UniGene数据集的114个文库中的291233个ESTs进行了比较http://www.ncbi.nlm.nih.gov/UniGene/lbrowse2.cgi?TAXID=29760[48的五个EST库除外在体外或细胞培养(库号10498,15513),混合器官(例如,根和叶一起)cDNA文库(库号20007,20010),或扩增片段长度多态性(AFLP) cDNA文库(库号20099)。相对EST频率计数的计算方法与前面描述的一样,使用“根”或“非根”组库组合的加权平均值。在一个或两个文库集中具有两个或两个以上ESTs的基因的EST频率计数(去除单例),并使用IDEG6 web工具使用Audic-Claverie统计量(AC)计算相应的差异基因表达模式。p-value < 0.01。Bonferroni多重测试校正仅用于考虑p-values < 3.0 × 106[53,60].将根ESTs与所有非根ESTs进行比较,初步鉴定出255个具有遗传特性的基因p-低于显著性阈值的值。此外,AC统计确定了135个“根富集”转录本,与表中所列的非根组织相比,这些转录本在根组织中表现出更高的频率3..此外,255个基因中有119个被鉴定为在非根文库中富集。因为分析的是一个归一化的根cDNA文库,所以这119个基因没有被进一步考虑,因为归一化过程预计会导致高度丰富的根转录本的系统性不足。对135个根富集基因的功能类别进行评估表明,与整个根EST集合相比,参与初级和次级代谢以及转运过程的基因数量更多(图6摄氏度).
根系富集基因的验证
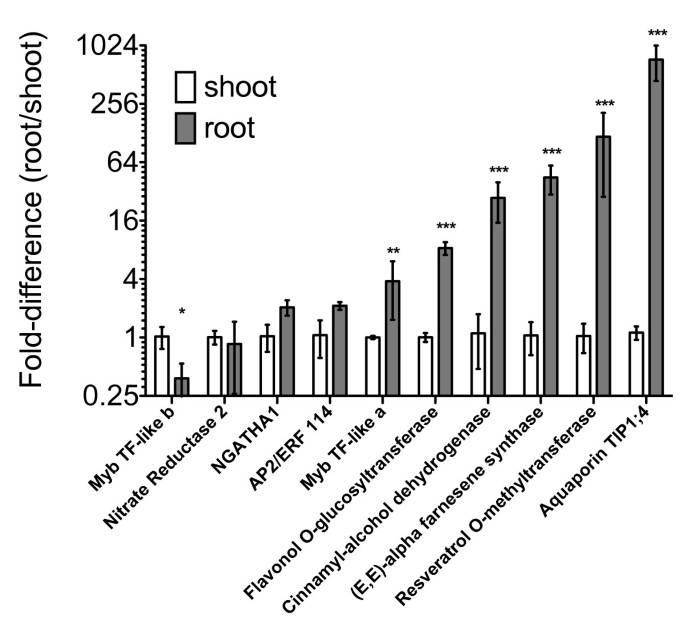
为了确认EST频率估计的根表达模式,选择一组假定的根特异性或根富集基因的表达进行qRT-PCR验证。基因特异性引物设计用于135个高根富集转录本中的10个。基因的选择不仅适用于那些根EST计数非常高的基因,也适用于那些频率较低但仍被认为具有统计学意义的基因。所使用的基因特异性引物列在附加文件中6.每个基因的相对转录丰度在赤霞珠的根和茎组织中进行测试(图7).采用基因和组织双因素方差分析,均有统计学意义(P < 0.0001)。方差分析后,计算根和茎组织间每个基因的Bonferroni校正t统计量。在这10个转录本中,有6个转录本在根中含量显著高于芽中(p < 0.01)。根中的转录丰度比芽中的转录丰度高3.8- 730倍。
赤霞珠根和芽中候选根特异性基因的表达.qRT-PCR分析茎部组织(白色条)和根部组织(灰色条)10个转录本。从三个生物重复中获得的转录丰度被归一化为肌动蛋白内参基因,折叠差异被标准化以获得表达值。误差条表示标准误差。双因素方差分析(基因,组织)后进行bonferroni校正t -统计数据。基因表达的显著差异(根与茎的比较)用星号表示。*表示p < 0.05;**表示p < 0.01;***表示p < 0.001。折痕差异以对数尺度绘制。测试的基因按照从左到右在图中出现的顺序列在下面,括号中是根ESTs与非根ESTs的比较。Myb家族转录因子样b,(7比1);硝酸还原酶2(9比3);NGATHA1转录因子,(5比0); (AP2/ERF transcription factor, 6 compared with 0); Myb family transcription factor-like a, (5 compared with 0); Flavonol 3-O-glucosyltransferase, (10 compared with 1); Cinnamaldehyde dehydrogenase, (9 compared with 1); (E, E)- α -法内烯合成酶(23比0);白藜芦醇o -甲基转移酶(30比0);水通道蛋白TIP1;4,(57比2)。
根含量最高的转录本编码了一种未鉴定的葡萄属在根组织中检测到的转录本丰度是芽组织的730倍。这与EST频率估计的表达有很好的相关性,在根中发现它的频率为每万分33.6个标记(tp10k),而在非根组织中为0.1 tp10k(57个根ESTs与2个非根ESTs)。qRT-PCR检测到白藜芦醇o -甲基转移酶(ROMT, GSVIVP00018661001)在根中(30个根EST与0个非根EST相比)的表达频率为17.7 tp10k,其在根中的表达量是茎部的120倍。类似地,萜烯合酶(TPS)基因(E, E)-法尼烯合酶[61], qRT-PCR检测其在根中的含量为13.6 tp10k(23个根EST, 0个非根EST),在根中的含量是茎部的44倍。肉桂醇脱氢酶(9个根EST与1个非根EST相比)在根中的表达量比在芽中高27倍。黄酮醇3- o -葡萄糖苷转移酶(10个根EST比1个非根EST)在根中的丰度比在芽中的丰度高8.3倍。最后,测试Myb转录因子样基因(5根EST与0根EST相比)以评估选择的显著性截断。该转录本在根中的丰度是芽中的3.8倍(显著,p < 0.05)。相比之下,三个测试基因(如AP2/ERF114, NGATHA1,硝酸还原酶2)未能通过多重测试校正t统计量测量出显著差异,并且一个单一转录本,第二个Myb转录因子样b基因(7个根EST与1个非根EST相比),被确定在根中的丰度比在芽中低2.6倍(p < 0.05)(图)7).对于所有测试的10个基因,基因表达的两种测量方法(EST频率与qRT-PCR相比)之间的Spearman秩相关较高(r年代= 0.78, p = 0.005)。虽然只测试了10个基因,但通过EST频率估计转录丰度显然可以有效地识别具有根特异性表达的基因,尽管大多数根ESTs来自标准化文库来源。
讨论
数据挖掘发现葡萄压力适应基因
为了识别对多种环境压力处理有反应的新转录本,我们对我们生成的EST库和那些来自公共资源的EST库进行了仔细的策划和挖掘,以获得基于EST频率的转录本丰度估计。分别从混合非生物胁迫叶片和水分亏缺胁迫浆果组织的非归一化cDNA文库中获得21,499条和18,963条独特的ESTs,与从非胁迫叶片和浆果组织的cDNA文库中获得5277条和24,953条独特的ESTs进行了比较1).基于标签频率的差异表达基因检测是一种完善的ESTs方法[53,60,62],圣人[57],及MPSS [7],并继续成为“下一代”转录组深度测序时代的重要工具[63].除了从单个cDNA克隆的双向和/或同方向重测序中去除冗余ESTs之外,在数据管理过程中遇到的主要问题之一是在镀克隆库内部和跨镀克隆库中发现各种类型的命名错误。借助一种类似于局部核苷酸序列比对的简单点图方法[51],基因id可以对齐并易于可视化,以发现含有“井滑”命名错误的不正确配对的板(或板的部分),由于同一文库中的板的重复测序,这些板可能高估了实际存在于特定感兴趣的cDNA文库中的ESTs的数量2,图1 g).该技术的应用还允许发现从叶子cDNA文库到浆果cDNA文库的EST错误分配板,这一错误将混淆EST计数对于特定组织的准确性(图1 h).
将混合胁迫或水分亏缺胁迫叶片和浆果组织的cDNA文库的EST频率计数与非胁迫叶片和浆果组织的cDNA文库的EST频率计数进行比较,共鉴定出739个转录本,并将其聚类为4个主要簇(图2)2、附加文件1,2,3.而且4).其中,637份(86%)转录本可以被注释并分配到功能类别(图3.).每个聚类包含不同的功能基团,清楚地反映了组织类型和治疗条件。例如,编码cbl相互作用蛋白激酶10 (CIPK10)的转录本在胁迫叶片(SL)和胁迫浆果(SB)簇中都有过多的表达。CIPK10参与钙调神经磷酸酶b样(CBL)钙传感器蛋白- cipk网络,该网络解码响应环境扰动的钙信号[64].的拟南芥CIPK10以GFP融合表达时定位于细胞核和细胞质烟草benthamiana叶子(65].CBL-CIPK相互作用对盐胁迫和其他形式的环境胁迫中离子稳态的调节至关重要,不仅在质膜和液泡体中,而且在细胞质和细胞核中[65].在这些胁迫特异性cDNA文库中,CIPK10转录本丰度的增加表明CIPK可能在胁迫适应中发挥作用葡萄属树叶和浆果。其他几个胁迫特异性转录本似乎在两个胁迫文库中都有过多的代表,包括RD22,葡萄浆果中的盐、脱水和aba响应基因[66](附加文件1而且3.).除了前面讨论的在胁迫浆果(SB)簇中富集的基因之外,还有几个致病相关(PR)蛋白,如三个thaumatin基因,一个IV类几丁质酶基因,两个渗透蛋白基因,以及snake -1,一种富含半胱氨酸的肽,表现出广谱抗菌活性在体外以及对真菌和细菌病原体的抗性在活的有机体内[67],在这个聚类中也很丰富。使用EST频率计数方法鉴定这组PR蛋白清楚地说明了它在发现生物和非生物应激反应的重要遗传决定因素方面的实际用途。大量的未知基因与离散的,集群特定的表达模式也被确定,特别是在胁迫叶(SL)集群。这些未知基因可以作为未来对基因功能进行详细研究的主要目标。
微阵列分析EST频率计数的验证
为了验证EST频率计数方法的有效性,在Affymetrix上可以识别出739个转录本中的489个®葡萄属GeneChip®从而比较使用这两种截然不同的技术方法。其余250个转录本没有匹配,因此,可能没有被描述为非生物应激反应葡萄属.在这两个平台之间,可以比较184个转录本的表达数据,使用这两种技术都观察到基因表达模式的显著差异。就像之前比较标签和杂交措施的报告一样[63],一个谦虚的(r= 0.21),但两个平台之间存在显著相关性(图4).进一步比较两种方法,发现方向性一致性为69%,这表明两种平台在总体基因表达趋势方面有更大程度的一致性。什么可以解释这些相当适度的相关性呢?首先,这些低相关性可能部分与所报道的转录丰度增加或减少幅度的差异有关。然而,在这两个平台上,每两个被报道显著增加或减少的基因中,就有一个基因在相反的方向上发生了显著变化(图4).因此,规模只能解释部分分歧。其次,使用高度多样化的公共数据集,可能会在基因表示方面引入偏差。在早期挖掘公共数据集的研究中,例如在对两个猪组织中454焦磷酸测序生成的EST reads与微阵列mRNA谱进行比较时,观察到四比一的一致性比率(160比38)[63]或在一组人体组织中SAGE标签与微阵列mRNA谱的比较中,观察到三比一的一致性比率[58].在本研究中,虽然公共数据集中的主要系统错误被纠正,但试图获取非应力叶和浆果库的正确频率计数(图1;表1,2),这些公共数据集包含葡萄品种、年龄、发育阶段、季节、风土和样品制备方面的巨大差异,这可能会导致基因表达的偏差。第三,我们的混合胁迫叶片文库的相对复杂性可能是偏差的来源,因为该文库的来源组织包括来自紫外线和热处理叶片的RNA,这些处理的相应微阵列数据无法进行比较。存在强烈或完全受紫外线或热胁迫调控的基因有望促进est显著转录本的数量,而相应的微阵列数据无法与之进行比较。
基于est的基因发现葡萄属根
为了弥补可用的葡萄根序列数据的相对缺乏,从赤霞珠根组织暴露在寒冷、盐度和水分亏缺胁迫下构建的标准化cDNA文库(VVM)中生成了超过16,000个ESTs(表2)1).在其准备过程中,该文库进行了标准化,目的是增加已识别的不同的低丰度根基因的数量[68].16452个ESTs组合成6424个唯一转录本,其中3449个(>53%)只出现一次。由于归一化文库有偏差,导致大量转录本计数不足,罕见转录本计数过多,因此违背了随机抽样的假设,因此通常不考虑用于基因表达的标签频率分析[6,42].认识到库归一化至少会低估大多数根转录本的真实相对表达,因此尝试通过EST频率计数来识别特定根或丰富根的EST。共18,109条根源性ESTs与来自114个非根cDNA文库的291,233条ESTs进行了比较。该分析结果鉴定出135个“根部富集”转录本,根据AC统计值确定,根系中的EST频率明显高于其他组织(表2)3.).通过qRT-PCR对10个具有不同程度明显根富集的候选根基因进行验证,证实有6个基因在葡萄根中明显多于在枝条中(图2)7).估计EST频率与qRT-PCR表达率之间的相关性很强(r年代= 0.78),显著性(P = 0.005)。用芽组织广泛但不全面地证实表达模式是根增强的。确认这些候选基因的根特异性表达模式将需要在基因的基础上测试额外的非根组织类型(例如,茎,花,浆果等)。
在qrt - pcr验证的根基因中,最主要的是一个编码水通道蛋白/膜体固有蛋白1的基因;4 (VvTIP1;4)在根中的表达量是在芽中的730倍。VvTIP1;4先前已从基因组序列中被两组[69,70],但尚未被功能特征。另一个富含根系的基因,通过qRT-PCR显示,其在根中的mRNA丰度比在芽中的mRNA丰度高120倍,编码一种推测的白藜芦醇- o -甲基转移酶(ROMT),与已知的rmt相同78%,相似88%葡萄属ROMT [71].Schmidlin及其同事观察到ROMT将白藜芦醇的o -甲基化分子加倍转化为pterostilbene,这是一种5-10倍大的植物抗逆素在体外真菌毒性比白藜芦醇强[71].这种根表达的ROMT在结构上也与最近在红浆果中发现的ROMT不同。红莓ROMT转录本在赤霞珠红葡萄中比白霞多丽更丰富,仅在véraison后2周在红色品种中达到峰值表达[72].一种萜烯合酶(TPS)在根中高度表达,根中相对丰度比茎中高44倍。Martin和同事发现TPS是一种(E, E)- α -法尼烯合成酶在一个彻底的调查特征诉酿酒用葡萄TPS基因[61].该TPS在39种被鉴定的TPS中表现出独特的活性,仅产生(E, E)- α -法二烯(FPP),而不是多种产物的混合物。另一种肉桂醇脱氢酶(CAD)基因在根中的含量也被证实是嫩枝的27倍。CAD基因在木材形成过程中对木质素化合物的合成至关重要,但某些CAD基因可能具有其他活性或功能。在拟南芥时,一些AtCAD基因启动子的活性已在cad介导的木质化似乎没有发生的细胞中观察到,包括年轻的根尖[73].最后,一个udp -葡萄糖o -葡萄糖基转移酶(UGT)基因在根中的含量是在芽中的8.3倍。与NCBI的保守域数据库(CDD)中发现的位置特定评分矩阵(PSSMs)相比[74],该UGT与PLN02554组UGT最为相似,这些UGT被归类为黄酮醇3- o -糖基转移酶(EC 2.4.1.91)。然而,确定UGT的确切催化活性通常需要生化表征,因为即使是UGT蛋白中单个氨基酸的变化也会改变区域选择性(例如,哪个羟基被糖基化)或udp -糖底物偏好[75,76].另外四个候选基因也被调查,但没有一个被发现表现出显著的、根富集的mRNA表达(p < 0.01)。
结论
非生物胁迫,特别是水分亏缺胁迫,对葡萄生长和浆果发育有重大影响,最终会影响葡萄酒的品质。在这里,利用EST频率计数来识别mRNA表达谱被非生物胁迫改变的候选基因,方法是将从遭受混合非生物胁迫的葡萄树收获的叶子和浆果中制备的cDNA文库中的大量EST集合与从非生物胁迫的葡萄树收获的相同组织中公开获得的EST集合进行比较。该分析在非生物胁迫的叶片和浆果中鉴定出739个表达差异显著的转录本。将这些基因的EST频率计数与现有的微阵列表达数据进行比较,确定了184个基因,这些基因也显示出应激和非应激组织之间的显著差异。虽然表达模式的相关性充其量是适度的,但69%的基因表现出方向性一致性。此外,EST频率计数方法鉴定了许多新的候选基因,这些基因的应激诱导mRNA表达模式以前没有被描述过。在…中优先或专门表达的基因葡萄属从赤霞珠的标准化、非生物胁迫cDNA文库中鉴定了16452个EST。将这些EST与公开的非根组织EST集进行比较,鉴定出135个根富集转录本,通过qRT-PCR验证,其中大多数显示出根优先的mRNA表达。这些富含根系的EST集合不仅为未来研究根系内的非生物胁迫反应网络提供了丰富的资源,而且还为未来的基因分型工作提供了丰富的资源葡萄属在耐盐性或耐旱性方面不同的砧木,或用于酿酒葡萄中根茎性状的操纵。
方法
植物材料
从非生物胁迫中提取总RNA诉酿酒用葡萄简历。开花后8、9、11、13、15、16周的霞多丽叶和浆果组织)使用修改的Tris-LiCl协议,如前所述[77].根组织从10cm高处采集诉酿酒用葡萄简历。赤霞珠扦插在热压灭菌,77毫米× 77毫米× 97毫米(宽×长×高)的无菌品红GA-7盒(品红公司,芝加哥,伊利诺伊州),含有80毫升1%植物组织培养琼脂(#A111,植物技术实验室,肖尼Mission, KS), Murashige & Skoog改性的基础培养基,含Gamborg维生素(#M404,植物技术实验室),1.5%蔗糖,pH值5.7 [78,79]在提供50 μmol m光子通量密度的荧光灯下生长-2年代-1光照16小时(24°C)/暗8小时(18°C)循环。将根系从非胁迫植物中分离出来,并将其置于对照条件(如上所述的液态MS介质中)、暴露于空气中的水分亏缺胁迫条件(2和4小时)、低温(1.5°C)和150 mM NaCl(如上所述的液态MS介质中)胁迫2、4和6小时。不使用6小时的水分亏缺胁迫暴露时间点,因为4小时胁迫后无法从根组织中恢复完整的RNA。
叶片和浆果cDNA文库的构建、测序和处理
叶(文库编号10208)和果(文库编号12534)cDNA文库的制备已在前面描述[6].将冷冻后的霞多丽叶和浆果组织均质于含有200 mM Tris-HCl、pH = 8.5、1.5% (w/v)十二烷基硫酸锂、300 mM LiCl、10 mM EDTA钠、1% w/v去氧胆酸钠和1% v/v NP-40的缓冲液中。高压灭菌后,在使用前立即加入2mm金内羧酸,20mm二硫代糖醇(DTT), 10mm硫脲和2% w/v聚乙烯聚吡咯烷酮。在乙酸钠沉淀和异丙醇沉淀后,样品以25:24:1苯酚:氯仿:异戊基萃取一次,然后以24:1氯仿:异戊基萃取两次,然后进行LiCl沉淀以去除DNA污染。根据制造商说明,使用Micro-FastTrack™2.0 mRNA分离试剂盒(Invitrogen, Inc., Carlsbad, CA)从500 mg总RNA中纯化Poly(A)+ RNA。根据制造商推荐的方案(Stratagene, La Jolla, CA),使用Lambda Uni-Zap-XR cDNA合成试剂盒从1-5 μg poly(A)+ RNA合成cDNA。将定向克隆(EcoRI/XhoI)的cDNA文库大量切除在活的有机体内得到的质粒(pBluescript II)在大肠杆菌SOLR宿主菌株。使用TempliPhi DNA测序模板扩增试剂盒(Amersham生物科学公司,皮斯卡塔韦,新泽西州)扩增含有插入的单个cDNA克隆,并在应用生物系统公司3700自动化DNA测序系统上使用Prism™Ready Reaction Dyedeoxy™Terminator循环测序试剂盒(应用生物系统公司,珀金-埃尔默,福斯特城,加利福尼亚州)上使用双脱氧链终止法进行测序。T3引物(5'- GGGAAATCACTCCCAATTAA-3')和T7引物(5'- gtaatacgactcactatagggc -3')分别用于cDNA克隆的5' reads和3' reads。Oligo-dT引物(T22M)用于含有poly-A尾的cDNA克隆的3'测序reads。
从Geospiza Finch服务器检索原始单次序列数据,并下载到EST分析管道(ESTAP) [80]进行清洗和分析。去除矢量序列和低质量序列后,所有长度≤50 bp的序列都被丢弃。其余序列使用d2_cluster [81]和CAP3算法[82]使用为ESTAP建立的默认参数。
根cDNA文库构建
利用冷、亏水、150 mM NaCl胁迫和对照条件下根系的总RNA构建了第三个混合cDNA文库(VVM),文库ID 22274。提取不同处理的总rna,等量汇总,然后进行mRNA筛选。Poly(A)+ mRNA使用Oligotex Direct mRNA试剂盒(Qiagen, Valencia, CA)从总RNA中分离。cDNA合成采用5'- aactggaagaattcgcggccgctcgcattttttttttttttttttttttttttv -3' (V = A,C,G)引物和Superscript III逆转录酶(Invitrogen)将poly(A)+ mRNA转化为双链cDNA。双链cdna大小选择(大于600bp),用EcoRI适配器(AATTCCGTTGCTGTCG - Promega #C1291)修饰,用NotI消化。然后将cdna定向克隆到EcoRI-NotI消化的pBluescript II SK+噬菌体载体(Stratagene, Inc., La Jolla, CA)。扩增前白色菌落形成单位(cfu)总数为3.0 × 106.蓝色菌落(空载体)在盘子上的菌落总数中不到10%。从主要文库中纯化的质粒DNA被转化为单链圆形,并作为模板,使用克隆cDNA插入物两侧的T7 (5'-TAATACGACTCACTATAGGG-3')和T3 (5'-AATTAACCCTCACTAAAGGG-3')引物位点进行PCR扩增,如前面所述[68].纯化的PCR产物,代表整个克隆cDNA种群,被用作标准化的驱动程序。单链文库(50 ng)与PCR产物(500 ng)在30°C下杂交44小时。将未杂交的单链DNA环与部分双链杂交DNA分离并电穿孔大肠杆菌DH10B单元生成标准化库。总插入无性系数为1.6 × 106cfu。空克隆的背景水平低于10%。cDNA文库归一化和构建由伊利诺伊大学香槟分校Roy J. Carver生物技术中心的W.M. Keck比较和功能基因组学中心进行。通过对原库和归一化库中的96个和285个克隆进行随机采样和测序,并比较其冗余率,验证了归一化效率。
根EST测序和数据分析
标准化根cDNA文库的EST测序使用T7测序引物(5'-TAATACGACTCACTATAGGG-3')在基因组服务公司Beckman Coulter, Inc.的应用生物系统3700自动化DNA测序系统(应用生物系统部门,perkins - elmer)上进行(丹佛斯,马萨诸塞州;前身为agcourt生物科学公司。在安卡拉大学生物技术研究所中央实验室的Beckman CEQ8000和CEQ8800测序仪(Beckman Coulter Inc., Brea, CA)上。对序列色谱进行了处理phr[83]用于高质量的基本调用,并屏蔽/屏蔽省略矢量序列使用cross_match(-minmatch 10 -minscore 20 -masklevel 100)对抗NCBI的UniVec,增加了克隆适配器策略特异性的筛选和删除序列。为了精确地识别并完全掩码插入cDNA片段的5’载体/适配器区域,即“典型适配器区域”(5’-TTGTAAAACGACGGCCAGTGAATTGTAATACGACTCACTATAGGGCGAATTGGGTACCGGG
CCCCCCCTCGAGGTATAAGCTTGATATCGAATTCCGTTGCTGTCG-3')、“2variant39”(5'- gcttgatatcgaattccgttctaattccgttgcctgtgtcg -3')、“3variant51”(5'- gcttgatatcgaattccgttgtcgccgctgtcg3 ')和“4variant39”(5'- gcttgatatcgaattccgttgctgtcgccgctgtcg3 ')序列被添加到矢量屏幕文件中。为了检测和掩码tgcga标记的/NotI/向量区域3'到插入的EST,“pB SK- at NotI site”(5'- tgcgagcggccgccaccgcggtggagctccagcttttgttccctttagtgagggttaatttcga
GCTTGGCGTAATCATGGTCATAGCTGTTTCC-3')和变体(5'- gatcagcggccgccaccgcggtggagctccagcttttgttccctttagtgagggttattttcga
将GCTTGGCGTAATCATGGTCATAGCTGTTTCC-3')序列添加到矢量屏幕文件中。设计了一组Perl程序来处理序列的最小长度(>100 nt)、嵌合体去除、poly-A尾信号识别、基本局部对齐搜索工具(BLAST)注释和dbEST提交。提交的高质量est被临时给予了与之比较的每个顶级BLAST命中的注释nr(11.06.2007版)[84].库VVM中的根ESTs被提交给dbEST,并被分配到Genbank id FC054794-FC071210和FC072669-FC072703。库作为“VVM”提交给dbESThttp://www.ncbi.nlm.nih.gov/UniGene/library.cgi?ORG=Vvi%26LID=22274[85].
使用的数据集,聚类分析和注释
所有可用的诉酿酒用葡萄序列(包括ESTs、表达转录本以及NCBI数据库中其他可用的DNA序列)通过NCBI的Batch Entrez从GenBank中提取(http://www.ncbi.nlm.nih.gov/sites/batchentrez) [86].cDNA文库的其他信息来自NCBI UniGene葡萄数据库(http://www.ncbi.nlm.nih.gov/UniGene/UGOrg.cgi?TAXID=29760) [48].然后将ESTs序列与其对应的“暂定一致”(TC) contig序列相关联诉酿酒用葡萄基因索引(VvGI,版本6,达纳法伯癌症研究所,http://compbio.dfci.harvard.edu/tgi/cgi-bin/tgi/gimain.pl?gudb=grape) [49,59].所分析的库列在表中1.然后将组装的TC序列或无法分配TC的单个单例与Genoscope 8.4X的预测肽序列进行比较诉酿酒用葡萄简历。黑皮诺葡萄(GSVIV)基因组组装(http://www.genoscope.cns.fr/externe/Download/Projets/Projet_ML/data/) [1,87].使用BLAST进行数据库搜索,使用Tera-BLAST™P算法,开放惩罚11,扩展惩罚1,双仿射Smith-Waterman窗口50,最大e值截断1 × 103TimeLogic decrypher硬件(Active Motif, Inc., Carlsbad, CA)。如果没有找到包含开放阅读框的基因模型,则每个EST与其UniGene模型相关联或被列为单例[88].
鉴定差异表达的转录本
从UniGene下载了单个ESTs和来源文库的所有可用EST信息,以匹配来自单个克隆来源的配对EST读取。通过从dbEST提交文件中解析的匹配克隆信息识别出冗余表示的克隆(例如,来自同一克隆的两个或多个ESTs),并使用DotPlot(版本2.1.1)插件(http://sourceforge.net/projects/dotplot/) [89]用于Eclipse(3.4.2版)软件开发环境(http://www.eclipse.org/) [90]用他们的8.4X基因模型的唯一名称在两个轴上绘制板,以验证克隆对。然后调整EST总量以反映正确的总量[91].
通过EST计数除以文库大小,计算每个文库中每个基因的频率。正如Haverty及其同事所描述的那样,相同类型的多个库(例如,多个未强调的浆果库)的EST频率通过加权平均值合并为单个频率项[52].使用网络工具“鉴定差异表达基因6”(IDEG6;(http://telethon.bio.unipd.it/bioinfo/IDEG6_form/) [53]使用推荐的卡方检验进行多库比较,p值临界值< 0.0001。有了这些设置,IDEG6会计算每个基因的频率分布的概率,并报告低于临界值的基因的频率(转录本/10,000)。使用Cluster软件包对差异表达基因进行分层聚类[92],采用函数(1 - Pearson相关系数)作为成对距离度量,采用平均聚类方法。这些差异表达的基因与在Affymetrix上发现的探针相匹配葡萄属GeneChip®微阵列(55],然后通过Spearman秩相关与多个Affymetrix微阵列实验中显著变化的基因表达数据进行比较,这些实验在多个时间点测试了非生物胁迫条件[10,27,31].对于微阵列探针组的表达值,以折叠变化最大的时间点/条件进行比较,不考虑对胁迫反应矛盾的探针组(一种条件下表达显著增加,另一种条件下表达显著降低)。然后使用路径、网络和网络外注释分配功能注释葡萄属网络软件http://www.sdstate.edu/aes/vitis/pathways.cfm[55,93].在单独的分析中,VVM库序列与非根EST库进行了比较,同样使用IDEG6 web工具(http://telethon.bio.unipd.it/bioinfo/IDEG6_form/) [94]使用推荐的Audic-Claverie (AC)统计量进行配对比较,p值< 0.01,采用IDEG6软件确定的Bonferroni多重测试校正调整(已调整)p-value cutoff < 3.0 × 106) [53,60].
定量实时逆转录pcr
用研钵和研杵在液氮中研磨冷冻的叶片和芽组织,使用Qiagen RNeasy植物迷你试剂盒(Qiagen Inc., Valencia, CA)从冷冻粉末中提取总RNA,并根据制造商的说明进行柱上DNase处理。使用Qiagen RNeasy Plant Midi试剂盒提取冷冻浆果和根组织RNA,除了添加2%聚乙二醇(MW > 20,000 kD, Sigma-Aldrich, Inc.,圣路易斯,密苏里州)以减少多酚污染外,制造商的说明进行了修改[77].RNA完整性经1.5%含甲醛琼脂糖凝胶电泳证实。cDNA使用iScript cDNA Synthesis Kit (Bio-Rad Laboratories, Inc., Hercules, CA)按照制造商的说明书进行合成,逆转录的RNA/反应量为1 μg。在NCBI使用Primer-BLAST选择用于实时qRT-PCR的基因特异性引物http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome[95使用RefSeq诉酿酒用葡萄文本作为输入,与所有其他文本进行筛选诉酿酒用葡萄RefSeq序列,以及以下Primer3 [96设置:Tm范围58-60°C,产品尺寸= 50-150 bp,底漆尺寸= 13-25 nt,最大poly-X = 3, G/C含量= 30-80%。根据qRT-PCR仪器的建议,选择引物对进行抗gc钳夹,使最后5个3'核苷酸中不超过2个是G或C。采用Fast SYBR制备实时定量RT-PCR反应®使用ABI PRISM进行绿色Master Mix®7500序列检测系统(应用生物系统公司,福斯特城,CA)。使用ΔΔCt方法对三个重复生物重复进行表达测定,参照eIF4a内源控制基因(GSVIV基因模型,GSVIVP00034135001)进行叶和浆果比较,参照肌动蛋白7内源控制基因(NCBI位点ID, LOC 100232968)进行茎和根比较[97].本研究中设计和使用的引物以及同源基因描述在其他文件中列出5而且6.
参考文献
- 1.
Jaillon O, Aury J, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C, Vezzi A, Legeai F, Hugueney P, Dasilva C, Horner D, Mica E, Jublot D, Poulain J, Bruyère C, Billault A, Segurens B, Gouyvenoux M, Ugarte E, Cattonaro F, Anthouard V, Vico V, Del Fabbro C, Alaux M, Di Gaspero G, Dumas V,等。自然科学学报,2007,29(3):344 - 344。10.1038 / nature06148。
- 2.
Velasco R, Zharkikh A, Troggio M, Cartwright D, Cestaro A, Pruss D, Pindo M, Fitzgerald L, Vezzulli S, Reid J, Malacarne G, Iliev D, Coppola G, Wardell B, Micheletti D, Macalma T, Facci M, Mitchell J, Perazzolli M, Eldredge G, Gatto P, Oyzerski R, Moretto M, Gutin N, Stefanini M, Chen Y, Segala C, Davenport C, Demattè L, Mraz A,等:一个杂合子葡萄品种基因组的高质量共识序列draft。PLoS ONE。2007, 2: e1326-10.1371/journal.pone.0001326。
- 3.
Scalabrin S, Troggio M, Moroldo M, Pindo M, Felice N, Coppola G, Prete G, Malacarne G, Marconi R, Faes G, Jurman I, Grando S, Jesse T, Segala C, Valle G, Policriti A, Fontana P, Morgante M, Velasco R:高杂合子基因组的物理图谱:黑皮诺葡萄品种的物理contig图。中国生物医学工程学报,2010,30(4):344 - 344。
- 4.
罗秀珍,刘志刚,李志刚。功能基因组学:转录组分析的封闭系统方法。葡萄遗传学、基因组学与育种“,”编辑:亚当-布朗登A-F,马丁内斯-萨帕塔J,科尔C. 2011,纽约,纽约:CRC出版社,270-298。
- 5.
蒂利特R,库什曼J:葡萄属功能基因组学:转录组分析的开放系统。葡萄遗传学、基因组学与育种“,”编辑:亚当-布朗登A-F,马丁内斯-萨帕塔J,科尔C. 2011,恩菲尔德,新罕布什尔州:科学出版社,235-269。
- 6.
da Silva F, Iandolino A, Al-Kayal F, Bohlmann M, Cushman M, Lim H, Ergul A, Figueroa R, Kabuloglu E, Osborne C, Rowe J, Tattersall E, Leslie A, Xu J, Baek J, Cramer G, Cushman J, Cook D:葡萄转录组的特征。分析多个表达序列标签葡萄属浆果发育过程中基因表达的种类和编制。植物科学学报,2005,29(3):344 - 344。10.1104 / pp.105.065748。
- 7.
Iandolino A, Nobuta K, da Silva F, Cook D, Meyers B:葡萄的比较表达谱分析(葡萄)基于est和MPSS特征的频率分析。中国生物医学工程学报,2008,8:53-10.1186/1471-2229- 853。
- 8.
Mica E, Piccolo V, Delledonne M, Ferrarini A, Pezzotti M, Casati C, Del Fabbro C, Valle G, Policriti A, Morgante M, Pesole G, Pe ME, Horner D:修正:高通量方法揭示了原始microRNA转录本的剪接和成熟microRNA的组织特异性表达葡萄.中国生物医学工程学报,2010,11:109-10.1186/1471-2164-11-109。
- 9.
Zenoni S, Ferrarini A, Giacomelli E, Xumerle L, Fasoli M, Malerba G, Bellin D, Pezzotti M, Delledonne M:草莓发育过程中转录复杂性的表征葡萄使用RNA-Seq。中国生物医学工程学报,2010,29(3):357 - 357。10.1104 / pp.109.149716。
- 10.
Cramer G, Ergül A, Grimplet J, Tillett R, Tattersall E, Bohlman M, Vincent D, Sonderegger J, Evans J, Osborne C, Quilici D, Schlauch K, Schooley D, Cushman J:葡萄水分和盐度胁迫:转录本和代谢物谱的早期和晚期变化。中国生物医学工程学报,2007,27(3):344 - 344。10.1007 / s10142 - 006 - 0039 - y。
- 11.
griplet J, Deluc L, Tillett R, Wheatley M, Schlauch K, Cramer G, Cushman J:葡萄浆果组织特异性mRNA表达谱分析。中国生物医学工程学报,2007,8:187-10.1186/1471-2164- 8187。
- 12.
Chervin C, Tira-Umphon A, Terrier N, Zouine M, Severac D, Roustan J:乙烯对葡萄果实膨胀的刺激及其对相关基因转录物的影响。物理学报,2008,34(3):344 - 344。10.1111 / j.1399-3054.2008.01158.x。
- 13.
Polesani M, Bortesi L, Ferrarini A, Zamboni A, Fasoli M, Zadra C, Lovato A, Pezzotti M, Delledonne M, Polverari A:霜霉病易感(葡萄)和一个电阻(诉锐利)葡萄品种。中国生物医学工程学报,2010,11:117-10.1186/1471-2164-11-117。
- 14.
杨晓明,杨晓明,杨晓明,等。盐胁迫对葡萄光合特性的影响。中国植物学报,2004,28(3):359-374。10.1071 / PP9810359。
- 15.
Prior L, Grieve A, Cullis B:灌溉田种植苏丹纳葡萄中氯化钠与土壤质地的相互作用。1.产量与果实品质。中国植物科学,2002,23(4):457 - 457。
- 16.
Stevens R, Harvey G, Partington D, Coombe B:不同生育期的盐水灌溉对土壤、营养生长和产量的影响。中国农业科学,1999,30(4):344 - 344。10.1071 / A98077。
- 17.
辛克莱C,霍夫曼AA:监测盐胁迫在葡萄:测量植物性状变异有用吗?应用生态学报,2003,40:928-937。10.1046 / j.1365-2664.2003.00843.x。
- 18.
马林斯MG,波束A,威廉姆斯LE:葡萄生物学。1992年,剑桥大学;纽约:剑桥大学出版社
- 19.
卡斯特林S, Pfeiffer A, Sivilotti P, Degan M, Peterlunger E, DI Gaspero G:季节性水分亏缺对葡萄成熟果实花青素生物合成的转录调控。植物生态学报,2007,30:1381-1399。10.1111 / j.1365-3040.2007.01716.x。
- 20.
黄志刚,李志刚,李志刚,等:灌溉对红毛牛皮肤花青素组成的影响(英文)葡萄l)葡萄果实在成熟过程中。中国农业科学,2001,29(4):489 - 497。10.1002 / 1097 - 0010 (200103) 81:4 < 409:: AID-JSFA830 > 3.0.CO; 2 h。
- 21.
肯尼迪JA,马修斯MA,沃特豪斯AL:成熟度和葡萄藤水分状态对葡萄皮和酒黄酮的影响。中国科学(d辑),2002,30(4):344 - 344。
- 22.
Castellarin S, Matthews M, Di Gaspero G, Gambetta G:水分亏缺加速葡萄果实成熟并诱导调节类黄酮生物合成的基因表达变化。植物学报,2007,27(3):344 - 344。10.1007 / s00425 - 007 - 0598 - 8。
- 23.
沃特斯·德尔,霍顿·塔,阿莱特·EM,斯莱德·李·L,亨利·RJ:成熟葡萄酒葡萄果实皮转录组。植物科学学报,2006,27(3):344 - 344。10.1016 / j.plantsci.2006.03.002。
- 24.
Deluc L, Grimplet J, Wheatley M, Tillett R, Quilici D, Osborne C, Schooley D, Schlauch K, Cushman J, Cramer G:赤霞珠葡萄果实发育的转录组和代谢产物分析。中国生物医学工程学报,2007,8:429-10.1186/1471-2164-8-429。
- 25.
陈建勇,温凤芬,孔伟峰,潘庆华,万寿生,黄卫东:葡萄果实发育过程中苯丙类代谢相关酶的变化及亚细胞定位。植物营养学报,2006,29(3):344 - 344。10.1016 / j.jplph.2005.07.006。
- 26.
戴维斯C,罗宾逊S:差异筛选表明在葡萄浆果成熟过程中mRNA谱发生了巨大变化。编码假定细胞壁和应激反应蛋白的cdna的克隆和鉴定。植物科学学报,2002,22(3):344 - 344。10.1104 / pp.122.3.803。
- 27.
Deluc LG, Quilici DR, Decendit A, Grimplet J, Wheatley MD, Schlauch KA, Merillon JM, Cushman JC, Cramer GR:水分亏缺改变了影响赤霞珠和霞多丽葡萄浆果重要风味和品质性状的不同代谢途径。中国生物医学工程学报,2009,10:212-10.1186/1471-2164- 10212。
- 28.
Pilati S, Perazzolli M, Malossini A, Cestaro A, Demattè L, Fontana P, Dal Ri A, Viola R, Velasco R, Moser C:葡萄浆果成熟的全基因组转录分析揭示了一组基因在三个季节中相似地调节,并在vèraison发生氧化爆发。中国生物医学工程学报,2007,28(4):428-10.1186/1471-2164- 828。
- 29.
Waters DLE, Holton TA, Ablett EM, Lee LS, Henry RJ:发育葡萄cDNA微阵列分析(葡萄简历。西拉子)浆果皮。中国生物医学工程学报,2005,30(5):344 - 344。10.1007 / s10142 - 004 - 0124 - z。
- 30.
Terrier N, Glissant D, Grimplet J, Barrieu F, Abbal P, Couture C, Ageorges A, Atanassova R, Leon C, Renaudin J, Dedaldechamp F, Romieu C, Delrot S, Hamdi S:等基因特异性寡核苷酸阵列揭示葡萄浆果中基因表达的多面变化(葡萄l .)发展。植物学报,2005,22(3):344 - 344。10.1007 / s00425 - 005 - 0017 - y。
- 31.
Tattersall E, Grimplet J, DeLuc L, Wheatley M, Vincent D, Osborne C, Ergül A, Lomen E, Blank R, Schlauch K, Cushman J, Cramer G:转录本丰度谱显示,与渗透和盐度胁迫相比,葡萄对低温胁迫的响应更大更复杂。中国生物医学工程学报,2007,7:317-333。10.1007 / s10142 - 007 - 0051 - x。
- 32.
Figueiredo A, Fortes A, Ferreira S, Sebastiana M, Choi Y, Sousa L, acolii - santos B, Pessoa F, Verpoorte R, Pais M:葡萄转录和代谢分析(葡萄L.)叶片揭示了可能的对病原真菌的先天抗性。应用物理学报,2008,29(3):379 - 379。10.1093 / jxb / ern187。
- 33.
Rotter A, Camps C, Lohse M, Kappel C, Pilati S, Hren M, Stitt M, Coutos-Thevenot P, Moser C, Usadel B, Delrot S, Gruden K:葡萄与真菌病原体易感相互作用的基因表达谱Eutypa拉塔病:为葡萄藤扩展MapMan本体。中国生物医学工程学报,2009,29(4):344 - 344。
- 34.
Keilin T, Pang X, Venkateswari J, Halaly T, Crane O, Keren A, Ogrodovitch A, Ophir R, Volpin H, Galbraith D, Or E:葡萄芽EST序列的数字表达谱分析为葡萄芽休眠释放过程中的分子事件提供了新的见解。植物科学学报,2007,43(3):344 - 344。10.1016 / j.plantsci.2007.07.004。
- 35.
Mathiason K, He D, Grimplet J, Venkateswari J, Galbraith D, Or E, Fennell A:转录分析在葡萄属锐利在冷却要求的实现中,基因表达模式与优化的破芽相协调。中国生物医学工程学报,2009,29(3):344 - 344。10.1007 / s10142 - 008 - 0090 - y。
- 36.
Sreekantan L, Mathiason K, Grimplet J, Schlauch K, Dickerson JA, Fennell AY:长、短光周期下葡萄花发育和基因表达差异表明花基因在休眠过渡中的作用。中国生物医学工程学报,2010,29(3):344 - 344。10.1007 / s11103 - 010 - 9611 - x。
- 37.
李志刚,李志刚:植物抗逆性的基因组研究。中国植物学报,2000,3:457 - 457。10.1016 / s1369 - 5266(99) 00052 - 7。
- 38.
朱建科,朱建科,朱建科:植物耐盐性的认识与提高。作物科学,2005,45:437-448。10.2135 / cropsci2005.0437。
- 39.
杨晓明,王晓明,杨晓明,杨晓明,铅在植物根系中积累的研究进展(英文)Lathyrus巨大成功L.)植物在芽中引发基因表达的系统性变异。环境科学学报,2009,27(3):344 - 344。10.1016 / j.chemosphere.2009.07.058。
- 40.
黄克胜,林敏,程广峰:白藜芦醇抗炎四聚物的研究进展葡萄属amurensis以及一些低聚二苯乙烯中七元环的构象。植物化学学报,2001,28(3):357-362。10.1016 / s0031 - 9422(01) 00224 - 2。
- 41.
朱玉娟,Agbayani R, Jackson MC, Tang CS, Moore PH:葡萄二苯乙烯合成酶基因VST1在木瓜中的表达增强了木瓜对植物病害的抗性疫霉palmivora.植物学报,2004,29(3):344 - 344。10.1007 / s00425 - 004 - 1343 - 1。
- 42.
Moser C, Segala C, Fontana P, Salakhudtinov I, Gatto P, Pindo M, Zyprian E, Toepfer R, Grando M, Velasco R:不同器官表达序列标签的比较分析葡萄中国生物医学工程学报,2005,30(3):344 - 344。10.1007 / s10142 - 005 - 0143 - 4。
- 43.
张军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军。葡萄夏瓦·格罗萨(Schiava grossa) ×雷司令(Riesling) ×波尔纳(Borner)葡萄属锐利x葡萄属灰质)定位根瘤蚜根系抗性的遗传决定因素。应用物理学报,2009,29(3):344 - 344。10.1007 / s00122 - 009 - 1107 - 1。
- 44.
崔玉杰,尹洪红,朴克辉,卢俊华,许艳雨,金世胜,金德伟,李海杰:est对转录谱分析的响应根瘤菌葡萄取自“Tamnara”葡萄藤(葡萄属sp)。中国植物学报,2010,37(4):344 - 344。10.1016 / j.jplph.2010.02.005。
- 45.
小梗N, georges A, Abbal P, Romieu C:葡萄浆果不同发育阶段EST的生成。中国植物科学,2001,29(4):457 - 457。10.1078 / 0176-1617-00566。
- 46.
冯鹏,K Reid, N Liao, Schlosser J, Lijavetzky D, Holt R, Martínez Zapater J, Jones S, Marra M, Bohlmann J, Lund S: est的生成葡萄酿酒葡萄(赤霞珠)和鲜食葡萄(汉堡马斯卡)以及在浆果发育中具有潜在作用的新候选基因的发现。基因学报,2007,402:40-50。10.1016 / j.gene.2007.07.016。
- 47.
林海燕,王晓霞,杨晓明,杨晓明:葡萄对烟草胁迫响应的est序列分析Xylella fastidiosa感染。中国生物医学工程学报,2007,27(3):344 - 344。
- 48.
葡萄-UniGene Build #12。[http://www.ncbi.nlm.nih.gov/UniGene/UGOrg.cgi?TAXID=29760]
- 49.
DFCI -葡萄基因指数。[http://compbio.dfci.harvard.edu/tgi/cgi-bin/tgi/gimain.pl?gudb=grape]
- 50.
Iandolino AB, da Silva FG, Lim H, Choi H, Williams LE, Cook DR:来自葡萄的高质量RNA, cDNA和衍生EST文库(葡萄l .)。植物分子生物学学报,2004,22:269-278。10.1007 / BF02773137。
- 51.
Gibbs AJ, McIntyre GA:比较序列的图表方法。它用于氨基酸和核苷酸序列。生物化学学报,2000,16:1-11。10.1111 / j.1432-1033.1970.tb01046.x。
- 52.
Haverty PM, Hsiao LL, Gullans SR, Hansen U, wz:三种全局基因表达方法之间的有限一致性突出了对非全局验证的要求。生物信息学,2004,20:3431-3441。10.1093 /生物信息学/ bth421。
- 53.
Romualdi C, Bortoluzzi S, D'Alessi F, Danieli GA: IDEG6:多标记采样实验中差异表达基因检测的网络工具。中国生物医学工程学报,2003,29(3):344 - 344。
- 54.
Romualdi C, Bortoluzzi S, Danieli GA:在多个标记采样实验中检测差异表达基因:统计检验的比较评价。胡文杰。2001,10:2133-2141。10.1093 /物流/ 10.19.2133。
- 55.
格里姆雷特J,克莱默GR,迪克森JA,马蒂亚森K,范赫默特J,芬内尔AY:葡萄属网络:“组学”整合通过葡萄藤分子网络。PLoS ONE。2009, 4: e8365-10.1371/journal. peer .0008365。
- 56.
Grandbastien MA, Audeon C, Bonnivard E, Casacuberta JM, Chalhoub B, Costa AP, Le QH, Melayah D, Petit M, Poncet C, Tam SM, Van Sluys MA, Mhiri C:应激激活和基因组影响Tnt1茄科逆转录转座子。中国生物医学工程学报,2005,29(4):344 - 344。10.1159 / 000084957。
- 57.
van Ruissen F, Ruijter JM, Schaaf GJ, Asgharnegad L, Zwijnenburg DA, Kool M, Baas F:利用SAGE和Affymetrix GeneChips估计基因表达数据的相似性评价。中国生物医学工程学报,2005,6:91-10.1186/1471-2164-6-91。
- 58.
李松,李艳红,魏涛,苏万伟,Duffin K,廖B:数据太多,但互换性很少:从挖掘基因表达组织特异性公开数据中得到的教训。生物工程学报,2006,1:33-10.1186/1745-6150-1-33。
- 59.
Pertea G, Huang X, Liang F, Antonescu V, Sultana R, Karamycheva S, Lee Y, White J,张F, Parvizi B, Tsai J, Quackenbush J: TIGR基因指数聚类工具(TGICL):用于大型EST数据集快速聚类的软件系统。生物信息学,2003,19:651-652。10.1093 /生物信息学/ btg034。
- 60.
Audic S, Claverie J:数字基因表达谱的意义。基因组学报,1997,7:986-995。
- 61.
Martin D, Aubourg S, Schouwey M, Daviet L, Schalk M, Toub O, Lund S, Bohlmann J:葡萄功能注释、基因组组织和系统发育(葡萄)萜类合成酶基因家族的基因组组装,FLcDNA克隆,酶测定。中国生物医学工程学报,2010,30(4):326 - 326。
- 62.
Stekel DJ, Git Y, Falciani F:多个cDNA文库中基因表达的比较。基因组学报,2000,10:2055-2061。10.1101 / gr.gr - 1325 rr。
- 63.
Hornshoj H, Bendixen E, Conley LN, Andersen PK, Hedegaard J, Panitz F, Bendixen C:利用高通量技术对两种猪组织的转录组和蛋白质组学分析。中国生物医学工程学报,2009,10:30-10.1186/1471-2164-10-30。
- 64.
李文杰,李文杰。CBL-CIPK Ca2 +-解码信令网络:功能与展望。中国生物工程学报,2009,34(4):344 - 344。10.1111 / j.1469-8137.2009.02938.x。
- 65.
Batistic O, Waadt R, Steinhorst L, Held K, Kudla J: cbl介导的CIPKs靶向促进来自不同细胞存储的钙信号解码。植物学报,2010,29(3):344 - 344。
- 66.
Hanana M, Deluc L, Fouquet R, Daldoul S, Léon C, Barrieu F, Ghorbel A, Mliki A, Hamdi S:葡萄rd22脱水响应基因的鉴定与鉴定(葡萄l .)。中国生物医学工程学报,2008,31(2):369 -578。10.1016 / j.crvi.2008.05.002。
- 67.
Almasia N, Bazzini A, Hopp H, azquez-Rovere C: snake -1基因的过表达增强了抗病性辣椒而且Erwinia carotovora在转基因马铃薯植株中。中国植物病理学杂志,2008,9:329-338。10.1111 / j.1364-3703.2008.00469.x。
- 68.
Bonaldo M, Lennon G, Soares M:归一化和减法:促进基因发现的两种方法。中国生物工程学报,1996,6:791-806。10.1101 / gr.6.9.791。
- 69.
傅凯,李丽娟,李丽娟,李丽娟。葡萄水通道蛋白的鉴定及其在果实发育中的表达分析。植物细胞学报,2008,27:1541-1550。10.1007 / s00299 - 008 - 0566 - 1。
- 70.
shellen M, Howitt S, Kaiser B, Tyerman S:葡萄水通道蛋白的鉴定和功能特征,葡萄.植物学报,2009,36:1065-1078。10.1071 / FP09117。
- 71.
Schmidlin L, Poutaraud A, Claudel P, Mestre P, Prado E, Santos-Rosa M, Wiedemann-Merdinoglu S, Karst F, Merdinoglu D, Hugueney P:一种胁迫诱导白藜芦醇o -甲基转移酶,参与葡萄中紫檀二苯乙烯的生物合成。中国生物医学工程学报,2008,29(3):344 - 344。10.1104 / pp.108.126003。
- 72.
Deluc LG, Decendit A, Papastamoulis Y, Merillon JM, Cushman JC, Cramer GR:赤霞珠浆果水分亏缺增加二苯乙烯代谢。食品化学学报,2010,59:289-297。
- 73.
金世杰,金文伟,赵兆明,陈志伟,陈志伟,陈志伟:肉桂醇脱氢酶在肉桂醇脱氢过程中的表达拟南芥成长和发展:数据库注释的经验教训?植物化学学报,2007,29(3):344 - 344。10.1016 / j.phytochem.2007.02.032。
- 74.
Marchler- bauer A, Anderson JB, Chitsaz F, Derbyshire MK, dewese - scott C, Fong JH, Geer LY, Geer RC, Gonzales NR, Gwadz M, He S, Hurwitz DI, Jackson JD, Ke Z, Lanczycki CJ, Liebert CA, Liu C, Lu F, Lu S, Marchler GH, Mullokandov M, Song JS, Tasneem A, Thanki N, Yamashita RA, Zhang D, Zhang N, Bryant SH: CDD:使用保存域数据库的特定功能注释。核酸研究,2009,37:D205-210。10.1093 / nar / gkn845。
- 75.
何晓珍,王霞,Dixon RA:紫花苜蓿糖基转移酶UGT71G1的突变分析揭示了控制(iso)黄酮糖基化区域选择性的残基。中国生物医学工程学报,2006,28(1):441- 447。10.1074 / jbc.M605767200。
- 76.
郭波,李志刚,李志刚。植物糖基转移酶供体特异性的单点突变研究。生物化学学报,2004,29(3):349 - 349。10.1016 / j.abb.2004.06.021。
- 77.
Tattersall EAR, Ergul A, AlKayal F, DeLuc L, Cushman JC, Cramer GR:从葡萄叶片中分离高质量RNA的方法比较。中华医学杂志,2005,29(4):344 - 344。
- 78.
Murashige T, Skoog F:烟草组织培养快速生长和生物测定的修订培养基。植物生理学杂志,2003,15:473-497。10.1111 / j.1399-3054.1962.tb08052.x。
- 79.
Gamborg OL, Miller RA, Ojima K:大豆根细胞悬浮培养的养分需求。中国农业科学,2015,30(4):457 - 457。10.1016 / 0014 - 4827(68) 90403 - 5。
- 80.
毛C, Cushman JC, May GD, Weller JW: estap - EST数据分析自动化系统。生物信息学,2003,19:1720-1722。10.1093 /生物信息学/ btg205。
- 81.
杨晓明,陈晓明,王晓明,等。一种有效的EST和全长cDNA聚类方法。中国生物医学工程学报,2003,29(4):344 - 344。10.1101 / gr.9.11.1135。
- 82.
黄X, Madan A: CAP3: DNA序列组装程序。基因组学报,1999,9:868-877。10.1101 / gr.9.9.868。
- 83.
Ewing B, Hillier L, Wendl MC, Green P:使用phred的自动序列跟踪的基调用。一、准确性评估。中国生物工程学报,1998,18(4):379 - 379。
- 84.
Altschul S, Gish W, Miller W, Myers E, Lipman D:基本的局部对齐搜索工具。中国生物医学工程学报,2000,26(3):344 - 344。
- 85.
cDNA文库22274 [葡萄].[http://www.ncbi.nlm.nih.gov/UniGene/library.cgi?ORG=Vvi&LID=22274]
- 86.
- 87.
/externe/Download/ projects /Projet_ML/data索引。[http://www.genoscope.cns.fr/externe/Download/Projets/Projet_ML/data/]
- 88.
Pontius JU, Wagner L, Schuler GD: UniGene:转录组的统一观点。NCBI手册。2003,贝塞斯达(MD):国家生物技术信息中心
- 89.
DotPlot |从SourceForge.net免费下载DotPlot软件。[http://sourceforge.net/projects/dotplot/]
- 90.
Eclipse——Eclipse基金会开源社区网站。[http://www.eclipse.org/]
- 91.
Journet EP, van Tuinen D, Gouzy J, Crespeau H, Carreau V, Farmer MJ, Niebel A, Schiex T, Jaillon O, Chatagnier O, Godiard L, Micheli F, Kahn D, Gianinazzi-Pearson V, Gamas P:探索模型豆科植物的根系共生程序Medicago truncatula使用EST分析。中国生物医学工程学报,2002,30:579- 592。10.1093 / nar / gkf685。
- 92.
Eisen MB, Spellman PT, Brown PO, Botstein D:全基因组表达模式的聚类分析和显示。美国国家科学研究院。1998, 95: 14863-14868。10.1073 / pnas.95.25.14863。
- 93.
- 94.
IDEG6软件主页。[http://telethon.bio.unipd.it/bioinfo/IDEG6_form/]
- 95.
底漆设计工具。[http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome]
- 96.
Rozen S, Skaletsky H:为一般用户和生物学家程序员提供的万维网上的Primer3。方法分子生物学杂志,2000,132:365-386。
- 97.
柯瑞德,李志强,李志强,李志强,李志强。葡萄果实发育过程中RT-PCR内参基因的定量分析。中国生物医学工程学报,2006,6:27-10.1186/1471-2229-6-27。
确认
美国国家科学基金会NSF (DBI-0217653)和内华达大学农业实验站(给GRC和JCC)支持了这项工作。作者要感谢Kitty Spreeman宝贵的技术支持。我们还要感谢Brian Anspach和Mary Ann Cushman分别在图形设计和关键手稿阅读方面的专家帮助。我们感谢伊利诺伊大学香槟分校Roy J. Carver生物技术中心的W.M. Keck比较和功能基因组学中心和Agencourt Biosciences, Inc.提供的cDNA文库构建和高通量EST测序服务。国家研究资源中心(NCRR)的NIH IDeA生物医学研究卓越网络(INBRE, RR-03-008)计划为内华达基因组学、蛋白质组学和生物信息学中心提供支持,NIH拨款号P20 RR-016464也使本出版物成为可能。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
RLT完成了所有EST数据分析、Perl编程、mRNA提取、qRT-PCR,准备了所有的图表和表格,并撰写了最初的手稿草稿。AE完成了所有根EST测序、原始数据分析和提交。RLA进行了所有根组织的准备和mRNA的提取,并进行了根cDNA文库构建的数据分析。KAS对稿件进行了层次聚类分析和数据解释定稿。GRC参与组织研究和定稿工作。JCC构思并组织研究,进行EST数据分析、跟踪提交、数据解读,最终确定数据和撰写稿件。所有作者都阅读并批准了最终的手稿。
电子辅助材料
胁迫叶簇内基因列表(SL, n = 355)
附加文件1:。基因SL不同表达的标记的群集用它们列出葡萄属Net-derived注释基因描述和功能分类。EST频率(f,每10,000个标签)显示为每个库类型:叶子f(L),强调叶子f(SL),浆果f(B),强调浆果f(SB)。基因id用于对应的8.4X草图基因组标识符或NCBI UniGene模型。相应Affymetrix葡萄属GeneChip®如果可用,还会显示探测集标识符。(doc 540kb)
叶簇内基因列表(L, n = 127)
附加文件2:。基因l不同表达的标记的群集用它们列出葡萄属Net-derived注释基因描述和功能分类。EST频率(f,每10,000个标签)显示为每个库类型:叶子f(L),强调叶子f(SL),浆果f(B),强调浆果f(SB)。基因id用于对应的8.4X草图基因组标识符或NCBI UniGene模型。相应Affymetrix葡萄属GeneChip®如果可用,还会显示探测集标识符。(docx38kb)
应激浆果集群基因列表(SB, n = 127)
附加文件3:。基因某人不同表达的标记的群集用它们列出葡萄属Net-derived注释基因描述和功能分类。EST频率(f,每10,000个标签)显示为每个库类型:叶子f(L),强调叶子f(SL),浆果f(B),强调浆果f(SB)。基因id用于对应的8.4X草图基因组标识符或NCBI UniGene模型。相应Affymetrix葡萄属GeneChip®如果可用,还会显示探测集标识符。(docx35kb)
Berry B簇基因列表(B, n = 130)
附加文件4:。基因B不同表达的标记的群集用它们列出葡萄属Net-derived注释基因描述和功能分类。EST频率(f,每10,000个标签)显示为每个库类型:叶子f(L),强调叶子f(SL),浆果f(B),强调浆果f(SB)。基因id用于对应的8.4X草图基因组标识符或NCBI UniGene模型。相应Affymetrix葡萄属GeneChip®如果可用,还会显示探测集标识符。(docx38kb)
12870 _2011_878_moesm5_esm.docx
附加文件5:用于芽和浆果基因实时qRT-PCR表达的引物列表生成基因实时qRT-PCR引物,与微阵列和EST频率结果进行比较。显示了基因名称、基因模型或contig标识符、正向引物(FP)和反向引物(RP)序列以及产物大小。(docx25kb)
12870 _2011_878_moesm6_esm.docx
附加文件6:用于根基因实时qRT-PCR表达的引物列表利用实时qRT-PCR方法对EST频率估计的根富集基因表达进行验证。显示了基因名称、NCBI基因位点标识符、正向引物(FP)和反向引物(RP)序列以及产物大小。(docx24kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
蒂利特,R.L., Ergül,阿尔比恩,R.L.et al。葡萄组织特异性、非生物胁迫响应基因表达模式的鉴定(葡萄L.)基于大规模EST数据集的管理和挖掘。BMC植物生物学11,86(2011)。https://doi.org/10.1186/1471-2229-11-86
收到了:
接受:
发表:
关键字
- 互补脱氧核糖核酸库
- 大规模并行签名测序
- 酿酒葡萄
- 贝瑞发展
- 紫檀芪