摘要
背景
真核生物DNA甲基化是研究最多的表观遗传过程之一,因为它导致外部刺激引发的直接和可遗传的共价修饰。与哺乳动物不同,植物DNA甲基化不仅发生在CG位点,还发生在CNG (N表示A、C或T)和CNN(不对称)位点。DNA甲基化的全基因组分析拟南芥CNN甲基化优先集中在转座子基因和非编码重复元件上。我们特别感兴趣的是研究具有更大和更复杂基因组的植物物种的表观遗传学拟南芥,特别是关于由非生物应激引起的相关改变。
结果
我们描述了跨越cnn甲基化的外胚轴的存在Asr1这是一种来自番茄植物的非转座子蛋白质编码基因,在番茄中缺乏同源对应物拟南芥。此外,为了验证表观遗传修饰与作物对非生物胁迫的适应之间存在联系的假设,我们详尽地探索了叶片中胞嘧啶甲基化状态Asr1DNA,我们体内的一种模式基因,是由于番茄植株缺水造成的。我们发现,干旱条件导致110个不对称位点(CNN)中约75个甲基标记被去除,同时抑制H3K27me3表观遗传标记减少,并在RNA水平上大量诱导表达。当精确定位这些位点时,我们观察到去甲基化主要发生在内含子区域。
结论
这些结果证明了CNN甲基化的一种新的基因组分布,即在蛋白质编码的非重复基因的转录区域,以及由水分胁迫引起的表观遗传标记的变化。这些发现可能代表了体细胞获得新的外胚轴的一般机制,它是调节植物基因表达的关键。
背景
表观遗传学是指基因调控和功能在有丝分裂和减数分裂上可遗传的变异,这种变异不能用DNA序列的变化来解释,而是由酶介导的DNA及其相关染色质蛋白的化学修饰引起的[1]。在过去的十年中,表观遗传学研究主要集中在哺乳动物身上,而植物受到的关注较少,尽管有相当数量的关于某些植物模型的信息,如拟南芥(2,3.],大米[4]和玉米[5]。
动物基因组的甲基化主要发生在调控区域,而动物基因组的甲基化主要发生在调控区域拟南芥在转录序列中,不仅在典型的CG位点,而且在CNG (N表示A、C或T)和CNN(不对称)位点上也有发现。后一种位点在重复元件和转座子中优先甲基化[6,7]。
通过对突变体的化学分析已经确定,哺乳动物DNMT1 (DNA甲基转移酶1)的同源酶MET1维持CG位点的DNA甲基化[8]。另一方面,植物特异性甲基转移酶CMT3维持CNG位点的DNA甲基化[7]同时与组蛋白H3甲基转移酶KYP相互作用[9]。最后,先锋突变体分析证明了第三种植物胞嘧啶甲基化(CNN,称为“不对称”)是由甲基化酶DRM2引起的[10],是哺乳动物的同源物新创甲基转移酶DNMT3。DRM2与内源性小干扰rna一起,也维持CNN位点的DNA甲基化[11,一种研究较少的表观遗传修饰。
我们的研究集中在番茄植株(茄属植物lycopersicum)、可食用的植物作物(http://mips.helmholtz-muenchen.de/plant/tomato/index.jsp)具有重要的经济意义,其基因组几乎是人类基因组的10倍拟南芥很少有关于表观遗传学的研究[12]。利用该模型系统,我们研究了不同环境下的胞嘧啶甲基化状态,以及胞嘧啶甲基化在水稻中基因内的分布Asr1LEA超家族的非转座子、蛋白质编码、水分胁迫诱导基因[13在植物界中是保守的,但在植物中缺乏同源的对应物拟南芥。这个基因已经被我们和其他DNA研究小组广泛研究过了。14], RNA [15]和蛋白质[16,17]水平和生理功能方面[18]和进化[14]。这个1199 bp的基因有一个非常简单的组织,由153和358 nt的外显子1和外显子2组成,由688 nt的内含子分开。我们选择叶子作为基因组DNA的来源,因为它是一个器官Asr1在水分胁迫下表达最大[15]。
我们工作的第二个方面是处理表观遗传学和植物胁迫之间的有趣联系[19- - - - - -21]。应激诱导的生理反应拟南芥被认为依赖于DNA甲基化的改变[22]。为了通过实验验证这一假设,我们研究了在番茄植株施加水分胁迫条件下,模型基因上胞嘧啶甲基化标记的增加和减少。
结果
番茄基因组整体非cg甲基化
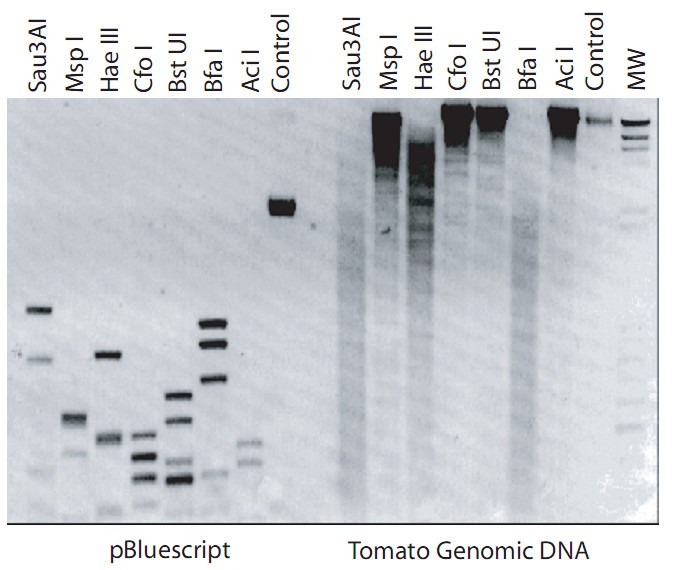
为了探索番茄叶片DNA甲基化的一般特征,我们首先使用几种限制性内切酶观察了CG和CNG甲基化的全景。甲基化敏感酶和甲基化不敏感酶之间的比较提供了对总体CG甲基化的评估。这个低分辨率但说明性的分析(图1)1)显示出明显的典型CG甲基化水平和明显的整体CNG甲基化程度(图2)1(Msp I处理),这种修饰通常(尽管不是唯一)与重复和/或转座因子相关。
非cg甲基化Asr1基因体
受上述结果的启发,我们希望深入了解胞嘧啶背景下的甲基化事件,而不是众所周知的CpG。为此,我们仔细检查了Asr1在树叶里。
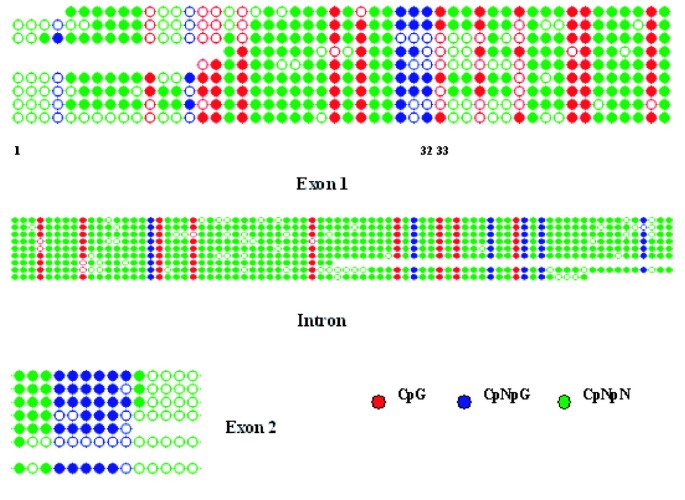
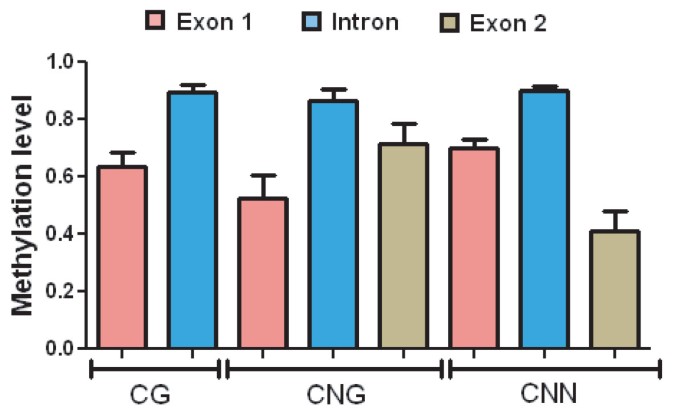
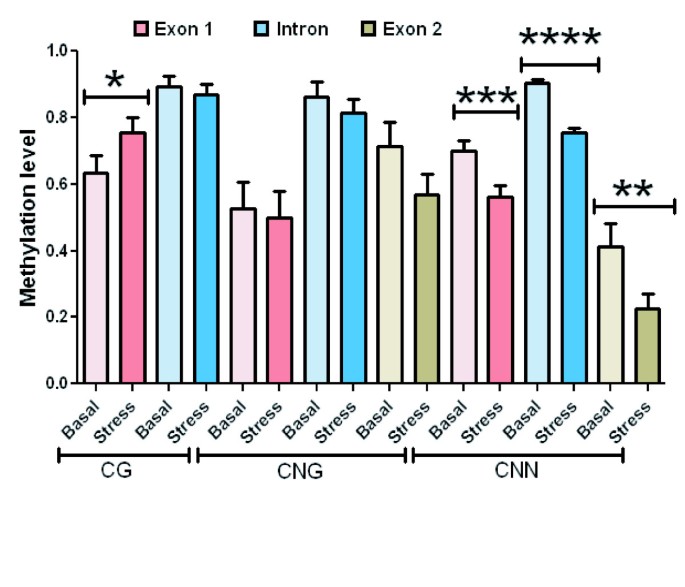
在这个分析中,我们使用了亚硫酸法[23],它允许更高的分辨率,因为它能够检测到所有的胞嘧啶残基(图2)2),而不仅仅是限制性内切酶识别的位点内的残基。在汇总了每种甲基化类型的数据并按基因区域分组后(图2)3.),我们得出结论,在非水分胁迫条件下,三种类型的甲基化(CG, CNG和CNN)水平显著。我们惊讶地发现CNN,因为Asr1是一个没有重复元件的非转座子基因,因此构成了这种类型甲基化位点的新位置。在这种情况下,CNN被证明优先集中在内含子中。
为了解决方法学上的问题,我们对未甲基化或体外甲基化的质粒DNA进行了平行亚硫酸反应,并获得了预期的结果(见方法)。此外,我们通过使用专门设计用于扩增转化模板且无法退火为自然模板的引物,排除了由于最终亚硫酸盐转化效率低下而导致的胞嘧啶甲基化的无意高估。此外,没有理由相信某些胞嘧啶残基(与引物互补的残基)实际上被转化,而同一纯DNA样本中的其他残基则没有被转化。
CNN在水胁迫下的去甲基化
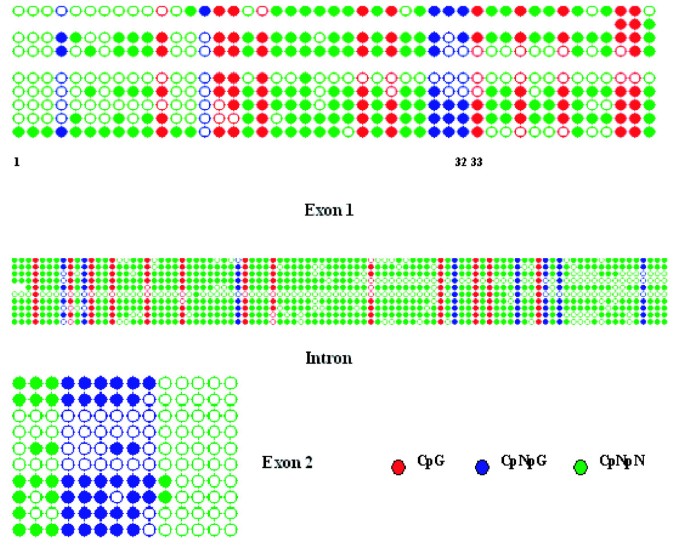
为了了解植物适应非生物胁迫的分子机制,我们验证了胁迫诱导的表型依赖于表观遗传变化的假设。考虑到这一目标,在通过根部干燥对整个番茄植株施加缺水胁迫后,对叶片DNA进行了类似的实验分析。我们发现干旱模拟条件导致外显子1 CG位点的甲基化(p < 0.08),并且分析的110个不对称(CNN)位点中75个甲基标记同时去除。这种去甲基化场景在整个基因体中具有统计学意义:外显子1 (p < 0.005),内含子(p < 0.0001)和外显子2 (p < 0.05)(图2)4和5).
这些结果与直接(即,没有先前的亚克隆)测序获得的甲基化状态数据一致Asr1基因组DNA经亚硫酸处理后的PCR产物(数据未显示)。
值得注意的是,具有不同模式的克隆可能是由不同的细胞类型(例如,表皮细胞和保护细胞)在检查的叶子样本中一起产生的,每个细胞都表现出不同的表观遗传行为。
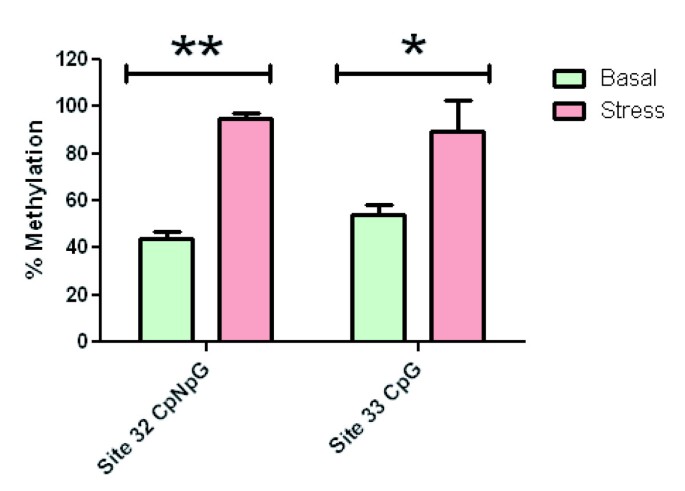
为了进一步验证亚硫酸盐方法,我们通过甲基化敏感和不敏感的限制性内切酶测量了单个CCGG位点(显然包含CG和CNG背景)的甲基化程度;选择的地点是C32C33GG,属于外显子1。结果(图1)6)与亚硫酸盐在基础条件和应激条件下获得的特定胞嘧啶残基的结果一致(图2)2和4).
在这一点上,有必要澄清图中所示的甲基化趋势3.和5反映了每个基因区域中所有胞嘧啶位置的平均行为,因此可能不一定与图中所示的单个胞嘧啶残基的表观遗传情况相匹配6。
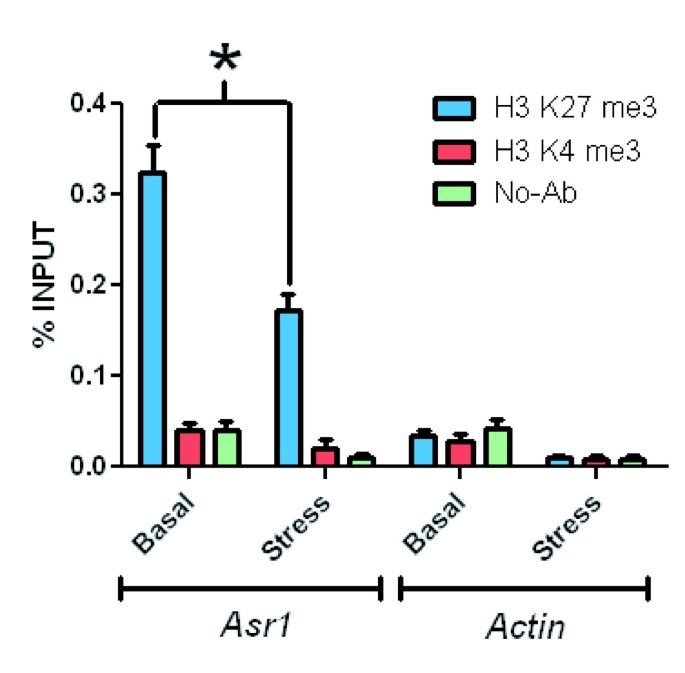
由于基因表达也可以通过翻译后组蛋白修饰来调节,而组蛋白修饰反过来又可能与胞嘧啶的甲基化相互作用,因此我们决定探索H3K27me3和H3K4me3,这两个组蛋白标记丰富拟南芥(24]。我们发现Asr1基因的表达水平与H3K27me3紧密相关,H3K27me3是基因表达的主要抑制标记。这种共价修饰在数量上似乎随着水分胁迫而减少(p < 0.05)(图2)7).相比之下,H3K4me3作为基因激活的标志,在任何条件下都没有被显著检测到Asr1(图7).
水分胁迫下的Asr诱导
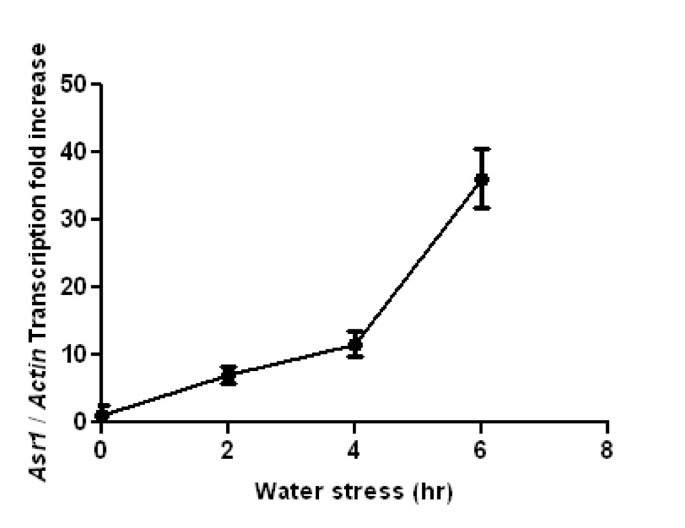
为了确定任何类型的甲基化(CG, CNG和CNN)与模型基因表达之间的最终相关性,我们在基础和应激条件下进行了qRT-PCR。结果(图8)表示7倍的感应Asr1在水分胁迫2小时后,叶片mRNA水平达到了36倍的诱导,在6小时时,在根和叶中观察到的显著萎蔫表型仍然是可逆的(数据未显示)。
讨论
在动物基因组中观察到的启动子区域内典型的CG甲基化也在某些植物基因座中被发现[25]。然而,全表观基因组的调查拟南芥已经发现转录区域也能够被甲基化,但与转座子相比,甲基化程度较低,并且甲基化仅限于CG位点[26]。一个这样的例子来自对矮牵牛花的研究,该研究表明,在内含子的CG位点转基因诱导的rna定向DNA甲基化(RdDM)后,c类花同源基因得以表达[27],这也揭示了基因体中的DNA甲基化与沉默并不一定像在动物中那样相关。Zhang等人报道了另一个类似的例子。[28他发现许多管家基因在编码区被甲基化,实际上表现出更高的表达水平。根据这些数据,我们发现应激引起模型基因第一外显子的CG甲基化水平升高,同时基因表达增强。
另一方面,Jacobsen小组已经积累了串联重复序列中非cg甲基化的证据[29]以及它在基因组复制区域的保守性[30.]。在我们的工作中,我们在一个新的位置检测到广泛的不对称CNN甲基化:一个非重复转录区域。此外,我们发现这种表观遗传修饰与表达不良相关,这与之前的研究结果一致[31]。类似地,据报道,一个零DRM2突变体可以阻断非cg甲基化,从而允许FWA基因完全去沉默,导致开花晚的表型[32]。
目前的模型表明,甲基胞嘧啶结合蛋白通过其SRA (SET和ring相关)结构域连接DNA和组蛋白甲基化事件[33]。事实上,DNA甲基化可以通过募集甲基胞嘧啶结合蛋白(如KYP、H3K9甲基转移酶和VIM1)诱导染色质重塑,从而诱导异染色质化[34]。DRM2对CNN甲基化的自我执行也由SUVH9介导,SUVH9没有可检测到的组蛋白甲基转移酶活性,但可以结合甲基化的CNN位点,从而促进DRM2进一步进入甲基化区域[35]。
蛋白质编码基因体内的胞嘧啶甲基化可能在连续几代中以很高的频率丢失,这似乎很重要[36],这与我们的结果一致,表明在基础条件下外胚轴是异质的。在自然重复的RNA基因中也发现了类似的变异情况[37]。
有趣的是,基因内DNA甲基化机制正成为调控基因表达和植物发育的重要修饰[1],但这些机制如何运作仍然是一个重要的问题。遗传证据提供了一个额外的分子参与者存在的例子拟南芥突变体在数千个基因中经历了CNG甲基化的异位沉积[38]。这些数据表明,有一组尚未发现的基因组保护因子在阻止甲基转移酶修饰含有非cg位点的基因区域(可能包括CNN位点)中发挥作用。
在这一点上,值得一提的是,据我们所知,关于不同植物甲基化酶在特定底物位点上的分配的结论,完全是通过分析突变体的反向遗传学得出的[7,10,39而不是来自纯化酶的体外实验。目前尚不存在这样做的生化方法,但如果发展起来,将令人信服地验证当前的假设。此外,生物化学将有助于建立新的模型,以了解DNA复制过程中CNN甲基化的体内维持,这是难以想象的,因为在新生的互补链中没有局部胞嘧啶残基需要甲基化。
就植物表观遗传学与胁迫之间的引人注目的联系而言,我们在番茄植株上的发现与Kovalchuk小组强调的假设是一致的[22在拟南芥,并在水稻中得到实验支持[40],至少一些应激诱导的表型依赖于DNA甲基化的改变。
关于染色质结构,H3K27me3在基础条件下而不是在应激条件下导致与基因Asr1表达水平相关并不奇怪,因为它是一个主要的抑制标记,至少在拟南芥(24]。
总之,本文提供的数据显示了植物中CNN甲基化的一个新位置,即在一个没有重复序列的模式基因体内,该基因受水分胁迫调节。这些发现可能代表了胁迫驱动的表观遗传标记的获得或损失的替代和一般机制,这些表观遗传标记调节着植物中其他基因的表达拟南芥它们的基因组更大、更复杂。这些新获得的外胚轴在受影响的体细胞中迅速出现,再加上植物在发育后期产生种系细胞的独特能力,可能允许其跨代遗传[41,42]和最终的积极选择,从而促进适应性进化。
结论
1)空气中存在明显的CNG甲基化茄属植物lycopersicum基因组,一种典型的,但不是唯一的,与重复和/或转座因子相关的修饰。
2)发现了一个异源的外胚轴群体Asr1基因在基础和水分胁迫条件下的表达。
3)我们在一个新的位置发现了广泛的不对称CNN甲基化:一个蛋白质编码的非重复基因的转录区域,与低表达相关。
4)干旱条件导致模型基因第一外显子的CG甲基化水平升高,CNN位点(主要在内含子区域)甲基标记的去除,同时该基因的表达增强。
5)干旱条件导致模型基因H3K27me3表达减少,同时该基因表达增强。
方法
植物材料
商品番茄(茄属植物lycopersicum)种子在20 g/l次氯酸钠溶液中浸泡30分钟,进行漂白。处理后,将种子放在潮湿的吸墨纸上,在黑暗中放置72小时。将幼苗置于23°C的生长室中,光照12小时/黑暗12小时,培养5天,然后移栽到花盆中。然后将植物放回生长室,每周浇水两次,直到进行实验。
水分胁迫条件
从花盆中取出四株三周大的植物,仔细清洁它们的根。两株植物的叶片被切下并冷冻在液氮中(非胁迫植物)。将另外两种植物的根放在吸墨纸上,在白炽灯下放置约2小时,直到出现萎蔫表型(证明是可逆的)。叶子被切下来并立即冷冻(受胁迫的植物)。
DNA提取
Peralta和Spooner的协议[43之后又做了一些修改。该程序包括使用CTAB作为洗涤剂,而不是SDS,由于其糖和多酚含量高,因此适用于番茄植株。采用分光光度法测定DNA质量260/一个280比率。只提供带有A的样品260/一个280比值在1.7 - 2.0之间。
用限制性内切酶估计全基因组甲基化
总基因组DNA (100 ng)用对胞嘧啶甲基化表现出明显敏感性的限制性内切酶处理(表2)1).所有病例均以20 μl(终体积)加5u酶在37℃条件下孵育4小时。
用限制性内切酶分析特定位点的甲基化
甲基化状态的检查进行了一对相邻的胞嘧啶位于外显子1Asr1(GenBank登录号L08255)通过同分异构体(HpaII和MspI;识别位点5'-CCGG-3')对甲基化表现出不同的敏感性,这取决于核苷酸的背景;HpaII对内部胞嘧啶的甲基化敏感(表明CpG甲基化),而MspI对外部胞嘧啶敏感(CpNpG甲基化探针)[44]。
每次酶促反应结束后,采用实时荧光定量PCR对切割位点两侧引物产生的169 bp扩增子进行定量:正向引物5'- atggaggaggagaaaccc -3'和反向引物5'-GATTATATCAACGTACCAAGGC-3'。为了进行PCR,我们使用Taq DNA聚合酶(Invitrogen) (0,625 U), 3 μM MgCl2分别为0.2 μM dNTPs和0.2 μM引物。加入5 ng (1 μl)模板。最终反应体积为25 μl。使用的设备是带有SybrGreen的MJ Engine Opticon (BioRad)®在以下条件下:变性(94°C, 30秒)、退火(67°C, 30秒)和延伸(72°C, 45秒)40次循环。在70 ~ 95℃每隔0.5℃制作熔化曲线。所有PCR反应重复运行,包括4个非模板阴性对照。并制作标准曲线验证PCR的线性范围、灵敏度和检出限。扩增数据分析使用PCR生产厂家提供的Opticon Monitor软件。板和盖由氧提供,寡核苷酸从IDT公司购买。
如果观察到长片段的后期扩增,则推断由于缺乏甲基化而发生近完全切割。相反,甲基化,因此未切割的DNA允许在相同条件下早期扩增长片段。C(t)值归一化为缺乏限制性位点的非相关扩增子。为此,我们使用了一对肌动蛋白(GeneBank登录号AB199316.1)引物(正向:5'- gggatgatatggagaagatgg -3'和反向:5'-AAGCACAGCCTGGATAGC-3'),在相同的循环条件和试剂浓度下扩增了一个185 bp的扩增子。计算每种酶的ΔC(t)值(酶处理与未处理):甲基化程度越高,ΔC(t)值越低,反之亦然。所有病例均采用熔融曲线分析和2%琼脂糖电泳检测PCR产物的特异性。
亚硫酸氢盐过程
我们使用Clark等人描述的方案。[23做了一些修改。DNA用Bfa I(5’-CTAG-3’)在37℃下通宵消化,得到平均长度约为2000 bp的DNA片段,用苯酚:氯仿(1:1)萃取部分纯化。然后用亚硫酸处理总基因组DNA (1 μg);转化步骤在55℃下进行16小时。然后使用商业向导DNA清理系统试剂盒(Promega)纯化处理过的DNA。
Post-bisulphite PCR
的Asr1基因(GenBank登录号L08255)使用先前设计的引物扩增[45具有最高的C+G含量,有利于退火到模板,并且具有最高含量的由亚硫酸转化的胞嘧啶残基衍生的胸腺嘧啶残基,特别是在3'端,这有利于转化分子的选择性扩增。引物设计使用Beacons Designer软件(http://www.premierbiosoft.com/molecular_beacons/index.html).
选择半巢式规则PCR,以尽量减少扩增非转化DNA的风险。对于第一个反应(1044 -bp扩增子),用Taq DNA聚合酶(Invitrogen)在MJ Research PTC-100 (MJ Research Inc.)上扩增5 μl亚硫酸处理产物,程序如下:变性(94°C, 30秒),退火(50°C, 30秒)和延伸(72°C, 1.30分钟)40个循环;采用以下引物进行PCR:正向引物5′-ATAGAGGATTTGATAAGATTATATTTG-3′和反向引物5′-CTTTTTTCTCATAATACTCATAA-3′。对于第二个反应,使用一个在第一个反应内部的正向引物,如下:正向5'- ggaggaggagaaatattattattat -3'。
作为反向引物,在相同的循环条件下使用相同的引物。最终获得的扩增子长966 bp,包括整个外显子1、内含子和外显子2的前104 nt。在这两个PCR反应中,我们使用了0.625 U的Taq DNA聚合酶,6 μM MgCl2每个引物分别为0.2 μM dNTPs和0.2 μM,终体积为50 μl。
亚硫酸盐转化效率的验证
为了评估完全转化,质粒DNA (pBluescript SK+, Stratagene)首先线性化,然后通过甲基转移酶mHaeIII (5'- ggmc -3')在体外甲基化(New England Biolabs)。将酶处理的质粒和未处理的质粒在与基因组DNA样品相同的条件下用亚硫酸盐孵育,然后用专门为本实验设计的引物进行PCR,正向引物5'-TTGTTATTATGTTAGTTGGTGAAAGG-3'和反向引物5'-CCCAAACTTTACACTTTATACTTCC-3'。
得到的383-bp扩增子与BfaI (5'-CTAG-3')孵育;当质粒之前没有经过亚硫酸处理时,酶将扩增子切割成两个预期的片段,分别为212和171 bp,但当质粒经过亚硫酸处理时,该位点丢失(现在为5'-TTAG-3'),酶无法切割。此外,当质粒先前被mHaeIII甲基化时,由于将5'-GGCCAG-3'序列修饰为5'-GGCTAG-3',新位点的创建通过凝胶检测得到285和98 bp的剪切后的两条预期条带得到证实。
亚克隆和测序
在pGEM-T“简易载体”(Promega)上进行亚克隆。使用GeneJET质粒迷你准备试剂盒(Fermentas)从随机选择的插入阳性菌落(每种生物情况10个)中处理质粒迷你准备。对SP6和T7通用引物进行Sanger测序。
甲基化数据分析
Kismeth软件[46) (http://katahdin.mssm.edu/kismeth/revpage.pl)用于分析甲基化数据。一旦收集到每个站点的数据,GraphPad软件就会被用于统计分析。根据基因区域(1外显子、2外显子、1内含子)和甲基化类型(CpG、CpNpG、CpNpN)对数据进行分组。统计学分析采用Mann-Whitney检验,显著性水平为95%。
亚硝酸盐后PCR产物的直接甲基化分析
PCR产物采用凝胶电泳和QIAquick gel Extraction Kit (Qiagen)进行纯化,使用与PCR相同的引物进行测序,无需预先进行亚克隆。用VarDetect分析色谱图[47来估计胞嘧啶和胸腺嘧啶信号的比例。统计学分析采用Mann-Whitney检验,显著性水平为95%。
染色质免疫沉淀(ChIP)用于组蛋白修饰
我们跟着里卡尔迪等协议[48],但使用dynabead蛋白A (Invitrogen)代替琼脂糖珠。我们用2 μg的DNA作为输入,每次处理8 μg。为了保持这些数量不变,体积根据DNA的初始浓度而变化。抗h3k4me3和H3K27me3抗体购自Abcam。使用相同的引物进行定量Real-Time PCRAsr1和肌动蛋白,用限制性内切酶进行甲基化分析(图2)6).我们使用与限制性内切酶实验相同的循环条件和试剂浓度(图2)6),但使用Maxima热启动DNA聚合酶(Fermentas)。
表达分析(RNA提取、逆转录和qRT-PCR)
用Trizol (Invitrogen)从100 mg灰浆磨碎的叶片中提取总RNA,然后用12.5 U DNAsaI (Invitrogen)孵育。用2 μl RNA、50 U MMLV-RT (Promega)和50 pmol oligo-dT (25 μl终体积)在42℃下逆转录1小时。为了防止RNA降解,加入10 U的RNAseOUT (Invitrogen)。在上述相同条件下,使用以下引物进行Real-time PCR:
Asr1 337 bp5“-CAGATGGAGGAGGAGAAACAC-3”5“-TAGAAGAGATGGTGGTGTCCC-3”
肌动蛋白185 bp5“-GGGATGATATGGAGAAGATATGG-3”5“-AAGCACAGCCTGGATAGC-3”
获得的数据Asr1在比较不同应激处理之前,在每个应激时间将mRNA归一化为肌动蛋白mRNA。
作者的信息
RMG和MMR持有阿根廷国家调查委员会Científicas y tsamcnicas (CONICET)的博士奖学金。NDI是CONICET的独立研究员。
缩写
- Asr1:
-
脱落酸胁迫与成熟
- 背景:
-
脱氧核糖核酸
- RNA:
-
核糖核酸
- MET1:
-
甲基转移酶1
- DNMT1:
-
DNA甲基转移酶
- CMT3:
-
Chromomethylase 3
- KYP:
-
氪石组蛋白3赖氨酸9甲基转移酶
- DRM2:
-
结构域重排甲基转移酶
- 李:
-
胚胎发生晚期丰富
- 聚合酶链反应:
-
聚合酶链反应
- 存在:
-
定量实时聚合酶链反应
- 澳洲公平工作委员会:
-
开花瓦赫宁根
- VIM1:
-
甲基化变异
- SUVH9:
-
SU (Var) 3-9同源物
- CTAB:
-
十六烷基三甲基溴化铵
- SDS:
-
十二烷基硫酸钠
- RdDM:
-
rna导向的DNA甲基化。
参考文献
- 1.
冯绍平,李建平,李建平,等。表观遗传重编程在植物和动物发育中的应用。科学通报,2010,33(3):622-627。10.1126 / science.1190614。
- 2.
Reinders J, Paszkowski J:拟南芥表观基因组的解锁。遗传学报,2009,4:557-563。10.4161 / epi.4.8.10347。
- 3.
张敏,Kimatu JN,徐坤,刘波:植物发育过程中DNA胞嘧啶甲基化。基因工程学报,2010,37(1):1-12。10.1016 / s1673 - 8527(09) 60020 - 5。
- 4.
秋本,金海军,小川E,谷野明,和田Y,谷野H:水稻的表观遗传。中国生物医学工程学报,2007,25(1):391 - 391。10.1093 / aob / mcm110。
- 5.
王X,我和我的朋友AA,李X,李N,彭Z, G,太阳H,气Y,刘XS,邓XW:全基因组和表观遗传学修饰瀑特异性景观及其关系在玉米mRNA和小RNA转录组。植物学报,2009,21(1):1053-1069。10.1105 / tpc.109.065714。
- 6.
陈敏,吕生,孟勇:植物的表观遗传表现者。[j] .科学与技术,2010(2):555-566。10.1111 / j.1440 - 169 x.2010.01192.x。
- 7.
植物DNA甲基化系统和靶点。2月。
- 8.
Finnegan EJ, Peacock WJ, Dennis ES:拟南芥DNA甲基化降低导致植物发育异常。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.93.16.8449。
- 9.
Jackson JP, Lindroth AM, Cao X, Jacobsen SE: KRYPTONITE组蛋白H3甲基转移酶对CpNpG DNA甲基化的控制。自然科学,2002,16(4):556-560。10.1038 / nature731。
- 10.
曹霞,Jacobsen SE:拟南芥DRM甲基转移酶在新DNA甲基化和基因沉默中的作用。中国生物医学工程学报,2002,12(2):368 - 368。10.1016 / s0960 - 9822(02) 00925 - 9。
- 11.
张欣:植物的表观遗传景观。科学通报,2008,32(3):489-492。10.1126 / science.1153996。
- 12.
李建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军。番茄果实发育过程中DNA甲基化和核内重复水平的组织依赖性。植物学报,2008,28(2):391-399。10.1007 / s00425 - 008 - 0743 - z。
- 13.
杨建军,杨建军,李建军,等。一种新型的氨基酸与氨基酸的结合。植物生理学报,2008,28(4):663 - 668。10.1104 / pp.108.120725。
- 14.
[3]刘建军,刘建军,刘建军,等Asr基因家族。中国生物医学工程学报,2006,32(3):744 - 744。
- 15.
Maskin L, Gudesblat GE, Moreno JE, Carrari FO, Frankel N, Sambade A, Rossi MM, Iusem NDAsr番茄基因家族(Lycopersicon esculentum).植物科学,2001,31(1):739-746。10.1016 / s0168 - 9452(01) 00464 - 2。
- 16.
[3]张志强,张志强,张志强,等ASR1蛋白质和渗透物甘氨酸甜菜碱。植物学报,2008,27(2):1213-1219。10.1007 / s00425 - 008 - 0693 - 5。
- 17.
马金龙,陈建军,陈建军,陈建军,等。dna二聚体的研究进展ASR1一种小的亲水性蛋白质,大量存在于遭受水分损失的植物组织中。中国生物医学工程学报,2007,35(2):831-835。10.1016 / j.bbrc.2006.11.115。
- 18.
Bermudez-Moretti M, Maskin L, Gudesblat G, Correa-García S, Iusem ND:Asr1这是一种应激诱导的番茄蛋白,可以保护酵母免受渗透胁迫。植物物理学报,2006,27(1):111-118。10.1111 / j.1399-3054.2006.00664.x。
- 19.
芬尼根EJ:外等位基因——压力时期随机变异的来源。植物学报,2002,5(5):101-106。10.1016 / s1369 - 5266(02) 00233 - 9。
- 20.
李建军,李建军,李建军,等。植物逆境响应的表观遗传调控。环境化学,2008,49(4):61-72。10.1002 / em.20347。
- 21.
朱建军:植物逆境响应的表观遗传调控。植物学报,2009,32(2):393 - 398。10.1016 / j.pbi.2008.12.006。
- 22.
杨建军,杨建军,杨建军,杨建军,杨建军,杨建军。拟南芥在逆境胁迫下的DNA甲基化和基因表达。科学通报,2010,5 (5):951 - 951 /journal.pone.0009514。
- 23.
李建军,李建军,李建军,李建军,李建军,李建军,李建军。自然科学学报,2006,31(1):2353-2364。10.1038 / nprot.2006.324。
- 24.
冯绍平,李建平,李建平,等。植物表观遗传修饰的研究进展。植物学报,2011,14(4):779 - 781。10.1016 / j.pbi.2010.12.002。
- 25.
Berdasco M, Alcázar R, García-Ortiz MV, Ballestar E, Fernández AF, Roldán-Arjona T, Tiburcio AF, Altabella T, buine N, Quesneville H, Baudry A, Lepiniec L, Alaminos M, Rodríguez R, Lloyd A, Colot V, Bender J, Canal MJ, Esteller M, Fraga MF:未分化拟南芥细胞启动子DNA超甲基化及其基因抑制。科学通报,2008,31 (3):3306-10.1371/journal.pone.0003306。
- 26.
Lister R, O'Malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Millar AH, Ecker JR:拟南芥表观基因组的高度集成单碱基分辨率图谱。Cell. 2008, 33(3): 523-536。10.1016 / j.cell.2008.03.029。
- 27.
Shibuya K, Fukushima S, Takatsuji H: rna定向DNA甲基化诱导植物转录激活。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.0809294106。
- 28.
张晓燕,孙德良,陈海燕,陈海燕,陈海燕,陈海燕。拟南芥DNA甲基化的全基因组定位和功能分析。细胞学报,2006,26(6):1189-1201。10.1016 / j.cell.2006.08.003。
- 29.
Henderson IR, Jacobsen SE:拟南芥内源SDC上游串联重复序列招募非cg DNA甲基化并启动siRNA扩散。基因工程学报,2008,22(2):597- 596。10.1101 / gad.1667808。
- 30.
魏德曼N, Jacobsen SE, Pellegrini M:拟南芥DNA甲基化的保守性。遗传学报,2009,4(4):119-124。10.4161 / epi.4.2.8214。
- 31.
杨建军,刘建军,刘建军,等。细胞嘧啶甲基化受体位点的基因表达与细胞嘧啶甲基化的关系。生物医学工程学报,1997,25(3):581-588。10.1007 / s004380050360。
- 32.
Greenberg MV, Ausin I, Chan SW, Cokus SJ, Cuperus JT, Feng S, Law JA, Chu C, Pellegrini M, Carrington JC, Jacobsen SE:拟南芥新生DNA甲基化所需基因的鉴定。遗传学报,2011,6:344-354。10.4161 / epi.6.3.14242。
- 33.
Johnson M, Bostick M, Zhang X, Kraft E, Henderson I, Callis J, Jacobsen SE: SRA甲基胞嘧啶结合结构域与DNA甲基化的关系。中国生物医学工程学报,2007,17(7):379-384。10.1016 / j.cub.2007.01.009。
- 34.
吴海涛,彭志强,李志强,等。甲基西橼酸结合蛋白VIM1的研究进展。基因工程学报,2007,21(2):367 - 367。10.1101 / gad.1512007。
- 35.
Johnson LM, Law JA, Khattar A, Henderson IR, Jacobsen SE: sra结构域蛋白在drm2介导的从头DNA甲基化中的作用。科学通报,2008,29 (4):11000280 - 11371 /journal.pgen.1000280。
- 36.
Vaughn MW, Tanurdzic M, Lippman Z, Jiang H, Carrasquillo R, Rabinowicz PD, Dedhia N, McCombie WR, Agier N, Bulski A, Colot V, Doerge RW, Martienssen RA:拟南芥表观遗传自然变异。中国生物医学工程学报,2007,35 (5):574 - 571 /journal.pbio.0050174。
- 37.
吴海华,李志强。拟南芥核糖体RNA基因甲基化的自然变异。植物生态学报,2008,28(2):559 - 564。
- 38.
王晓明,王晓明,王晓明,等。拟南芥jmjC结构域蛋白对DNA甲基化的影响。中华医学杂志,2009,28:1078-1086。10.1038 / emboj.2009.59。
- 39.
Lindroth AM, Cao X, Jackson JP, Zilberman D, McCallum CM, Henikoff S, Jacobsen SE: CpXpG甲基化维持的CHROMOMETHYLASE3要求。科学通报,2001,29(2):2077-2080。10.1126 / science.1059745。
- 40.
王文伟,潘玉军,赵小强,Dwivedi D,朱丽华,阿里娟,傅碧,李志康:水稻干旱诱导位点特异性DNA甲基化及其与抗旱性的关系[J]。栽培稻l .)。[J] .中国生物医学工程学报,2011,32(2):551 - 561。10.1093 / jxb / erq391。
- 41.
李建军,李建军,李建军,等。植物表观遗传研究进展。自然科学,2007,44(4):418-424。10.1038 / nature05917。
- 42.
王晓明,王晓明,王晓明,等。植物基因组DNA甲基化动力学。生物化学学报,2007,17(6):276-286。
- 43.
Peralta IE, Spooner DM:野生番茄(Solanum L.)颗粒结合淀粉合成酶(GBSSI)基因系统发育Wettst。分段Lycopersicon)。[J] .中国生物医学工程学报,2001,31(2):588 - 592。10.2307 / 3558365。
- 44.
Takamiya T, Hosobuchi S, Asai K, Nakamura E, Tomioka K, Kawase M, Kakutani T, Paterson AH, Murakami Y, Okuizumi H:使用同分异构体(MspI/HpaII)进行DNA甲基化分析的限制性标记基因组扫描方法。电泳,2006,27:2846-2856。10.1002 / elps.200500776。
- 45.
王晓明,王晓明,王晓明,等。甲基化研究中引物设计方法的研究进展。生物医学工程学报,2008,(1):54-10.1186/1756- 05001 -54。
- 46.
grundman E, Qi Y, Slotkin RK, Roeder T, Martienssen RA, Sachidanandam R: Kismeth:亚硫酸氢盐测序法测定植物甲基化状态。生物信息学杂志,2008,9:371-10.1186/1471-2105-9-371。
- 47.
Ngamphiw C, Kulawonganunchai S, Assawamakin A, Jenwitheesuk E, Tongsima S: VarDetect:一种核苷酸序列变异检测工具。生物信息学,2008,9(增刊12):S9-10.1186/1471-2105-9-S12-S9。
- 48.
Ricardi MM, González RM, Iusem ND:番茄染色质免疫沉淀(ChIP)方案的微调。植物方法,2010,6:11-10.1186/1746-4811-6-11。
致谢
这项工作得到了阿根廷布宜诺斯艾利斯大学(UBA)、阿根廷国家机构Promoción Científica y Tecnológica (ANPCyT)和阿根廷国家调查委员会Científicas y Tecnológicas (CONICET)的资助。我们感谢Ignacio E. Schor博士提供抗h3k27me3抗体。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
RMG完成了所有的实验工作,生成了数据,并与NDI一起广泛修改了手稿。MMR支持日常的实验任务,并在整个工作过程中提出了宝贵的建议,特别是修订版本所需的ChIP实验。NDI介绍理论框架,协调项目,起草稿件。所有作者都阅读并批准了最终的手稿。
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
González, r.m., Ricardi, M.M. & Iusem, N.D.非典型位置的非典型表观遗传标记:非重复番茄基因体内不对称(CNN)位点的胞嘧啶甲基化。BMC Plant Biol11,94(2011)。https://doi.org/10.1186/1471-2229-11-94
收到了:
接受:
发表:
关键字
- 表观遗传学
- 不对称的甲基化
- Asr1
- 水的压力
- 番茄