摘要
背景
突变扫描技术已被用于培育具有优良性状的作物品种。改进筛选产量和敏感性将有助于作物的定向突变育种。高分辨率熔解(HRM)分析的技术创新使基于临床的人类疾病基因多态性筛查成为可能。我们研究了两种HRM修饰,COLD-PCR和QMC-PCR在桃基因突变扫描中的应用,Prunus Persica.目标基因是假定的花的调节基因PpAGAMOUS和PpTERMINAL花我.
结果
人力资源管理分析PpAG和PpTFL1在36个桃品种的编码区中,每个基因都有一个多态性位点。PpTFL1和PpAGSNPs被用来检验增加人力资源管理吞吐量的方法。在12种基因型的池中可以可靠地检测到SNP的栽培品种。研究发现,冷pcr提高了混合样品HRM分析的灵敏度,但在小扩增物中效果最好。对QMC-PCR的检查表明,用于进一步分析的初级PCR产物可以由基因组DNA的可变水平产生。
结论
通过来自东南美国育种计划的品种的HRM分析发现了目标桃基因外显子的天然SNP。为了在较大的群体中检测天然或诱导的SNP,通过通过冷PCR和QMC-PCR等方法增加样品池和模板产生,可以提高HRM效率。为改善临床诊断而开发的技术进步可以在庄稼的目标突变繁殖中发挥作用。
背景
通过筛选候选基因诱导的突变来开发具有改善性状的作物[1- - - - - -5].几种方法已被用于筛选化学物质诱变的植物群体,如甲基磺酸乙酯(EMS)。例如,通过SSCP分析筛选ems诱变的烟草株系[1].尼古丁n -去甲基化酶基因突变的烟草基因型(ntabcyp82e4.)已经被鉴定出显著降低了去甲尼古丁的水平。采用分蘖法对一个小麦品种进行ems诱变株系筛选Wx-B1三个中的一个蜡状淀粉生物合成的同源基因[2].诱发的小麦基因型Wx-A1和Wx-D1突变被检测出来,然后杂交产生wx-a1 / wx-b1 / wx-d1低直链淀粉的谷物。第三种突变扫描方法是高分辨率熔解(HRM),用于鉴定ems诱导的调控果实品质和耐旱性候选基因突变的番茄品系[3.].
改善筛选吞吐量和敏感性的修改将加快筛选成千上万的天然突变或诱导突变的基因型。例如,高通量毛细管电泳通过SSCP促进了突变分析[1]和TILLING [6,7].将人力资源管理用于人类疾病基因的临床筛选,鼓励了改进的发展,使其更敏感、更方便使用和更经济有效。我们检测了两种HRM修饰的应用,COLD-PCR [8]及QMC-PCR [9,用于植物基因的突变筛选。
增加人力资源管理吞吐量的一个方法是通过汇集样本进行分析。Gady等等。[3.]发现人力资源管理可以可靠地分析番茄系的四种基因型,但8倍的池增加了假阴性的频率。采用5倍池法对ems诱变玉米进行HRM分析[10].人力资源管理的吞吐量对医疗诊断很重要[如。,但更常见的问题是检测细胞中的突变,这些细胞只占正常组织样本的一小部分[12].增加人力资源管理的敏感性将改善异质性组织样本的突变分析,以及汇集的个体。
冷PCR是一种PCR改性,通过有利于使用DNA Mismatch的扩增子的产生来增加突变筛选的敏感性[13].PCR是在异双链DNA优先变性和扩增的变性温度下进行的。基因突变检测的灵敏度®,通过富集变体序列,通过富集,在耕地中使用的特定于抗匹配的内切核酸酶增加了多个级别[13].COLD-PCR已与HRM联合使用,以识别野生型DNA背景中低至0.1%的基因突变[8].我们检验了COLD-PCR是否可以用于增加人力资源管理分析的样本汇集深度。
另外,突变分析的效率可以通过修改DNA模板生产从大群体中提高。诸如NEATTILL等技术[14]及QMC-PCR [9加快DNA模板的制备。开发QMC-PCR用于改进福尔马林固定石蜡包埋组织DNA突变的HRM分析,该组织受DNA降解和交联的影响。在QMC-PCR中,最初的多重反应产生模板,这些模板在二次反应中使用嵌套引物来扩增每个模板的多个区域。在检测野生型DNA背景突变方面,QMC-PCR被证明与COLD-PCR一样敏感,是Sanger测序的8倍[9].为了验证这一方法,我们用嵌套引物测试了基因组DNA模板水平对初始PCR扩增子及其产物HRM的影响。
作为一个实验系统,我们针对两个调控桃开花的基因,Prunus Persica.桃子直接同源的无性生殖的(PpAG),终端花1(PpTFL1)已被描述,并可获得基因组序列数据[15- - - - - -17].2010年桃(http://www.rosaceae.org)将促进新的基因发现。然而,桃中的功能和翻译基因组学术中的重新分析对遗传转化的限制。桃是靶向突变育种的候选者,因为它的紧凑型二倍体基因组(220 Mbp),自相容性和木质植物短的少年阶段(2-3岁)。在这项研究中,来自东南美国繁殖计划的桃子以天然多态性的HRM筛选PpAG和PpTFL1.利用在这些基因中发现的单核苷酸多态性(SNPs),研究了两种提高人力资源管理吞吐量的方法:(1)增加样本池和(2)使用PCR产品作为模板进行进一步的PCR和人力资源管理分析。
结果
桃花基因snp的HRM检测
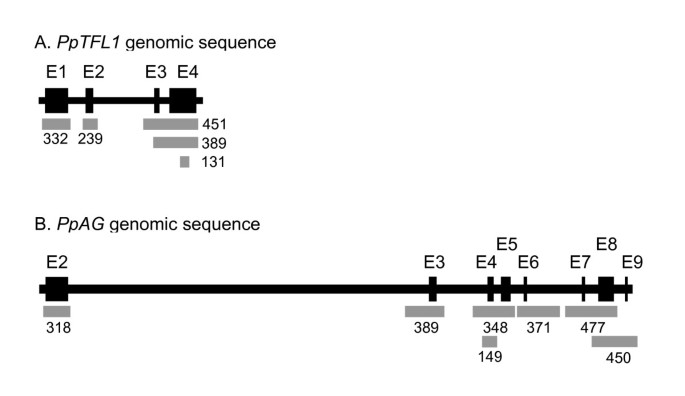
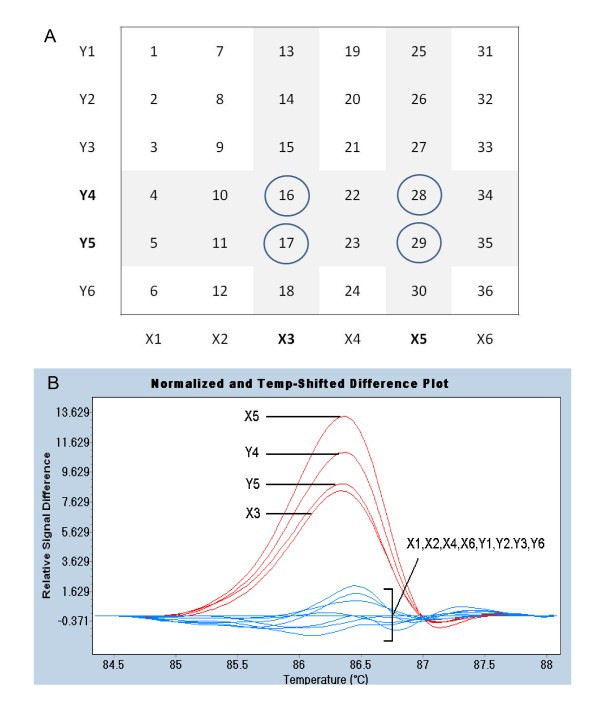
外显子的PpTFL1和PpAG通过对准基因组和cDNA序列的对准来鉴定,并开发了引物套,即扩增的外显子区(图1)。从36个桃子栽培中分离的基因组DNA在六组中二维汇集(图2)。除了提高产量外,样品池还促进了通过提供野生型DNA进行不匹配产生的纯合突变的检测。用Lighycler 480(Roche诊断)进行PCR和HRM。通过LC480基因扫描软件(1.5版)分析DNA熔化数据,其在数据标准化和温度换热后,使用专有算法具有类似熔化模式的分组品种。
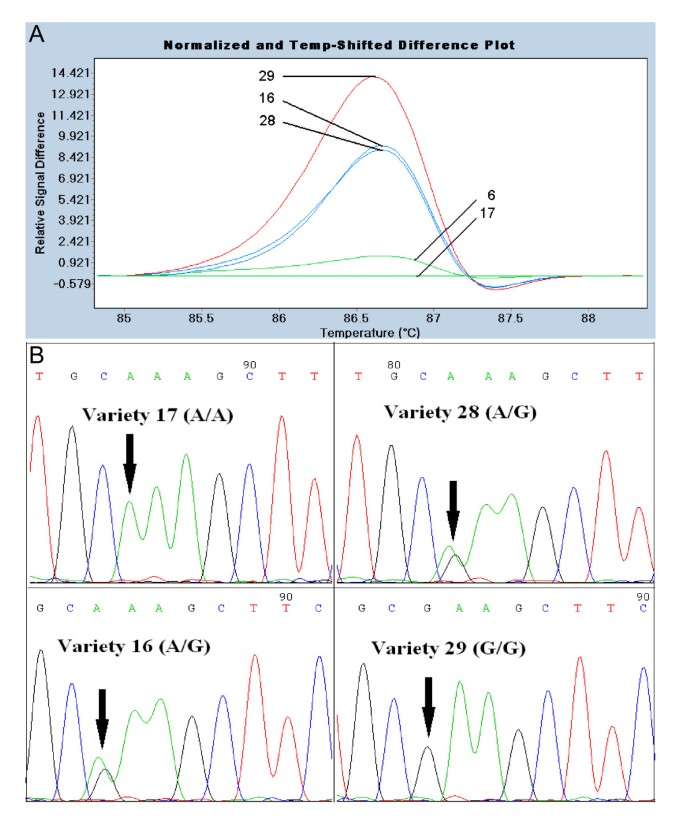
人力资源管理分析PpTFL1外显子1和2在12个样本中没有发现DNA熔化谱的差异(未显示)。相比之下,当跨越外显子3和4的扩增子被分析时,四个DNA池显示出改变的DNA熔化曲线(图)2 b)。这些池中共有的四个品种被独立检查,其中三个品种的熔化谱表明DNA不匹配(图)3)。DNA测序结果显示,品种16、28和29具有相似的多态性(1202A > G)PpTFL1外显子4为野生型(图17)3 b)。品种29在该位置具有纯合的SNP,而栽培品种16和28具有杂合子SNP。由于两者都引起的更大熔化曲线变化,栽培品种29分开与其他SNP系列分开进行分开进行分组PpTFL1等位基因在1202位形成错配。
DNA序列PpTFL1品种16、17、28和29的外显子3被发现是相同的(未显示)。的顺序PpTFL1其他5个具有野生型HRM谱的品种的外显子3和4在这个区域没有发现多态性(附加文件)1)。所有没有栽培品种16、28和29的池在该地区具有相似的野生型人力资源管理模式(图)2)。每人PpTFL1所有36种桃子品种的外显子也被HRM单独检查,没有检测到在汇集样品中确定的SNP(附加档案2)。这些结果表明,1202A > G的单多态性位点PpTFL1HRM可检测到编码区,36个品种中有3个含有该SNP。
的八个翻译的外显子PpAg在六个相应的PCR扩增子中检测在310-480 bp之间(图1)。跨越外显子4和5的分析识别熔化型材改变的六个池(图4),共包含8个品种。当单独检查时,4个品种的熔融曲线表明多态性(图4 b)。测序结果证实,4个品种均为外显子4 (4757G > a) SNP杂合子PpAgSNP (X2和X5)分别从有一个SNP和没有SNP的池中分组。其他五个扩增子覆盖了PpAG编码区显示36个品种间DNA熔解无差异(未显示)。表格1的外显子中发现的单核苷酸多态性PpAG和PpTFL1.在两个基因中没有含有SNP。对于两个基因,SNP导致同义突变。
使用标准PCR和COLD-PCR对混合样品进行HRM分析
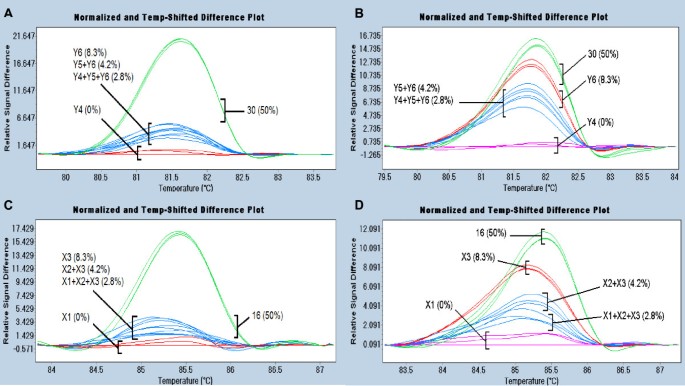
具有多态性的基因型PpAG或PpTFL1在从六个桃子中汇集的DNA中检测到。我们检查了SNP是否可以在样品池中识别,这是两个或三倍的样本池。品种30(PpAGSNP)和品种16(PpTFL1SNP)每组汇集在6,12或18个基因型中,含有麦类的野生型伴有所检查的基因。对于这两个基因,LC480基因扫描软件区分三个池中含有野生型序列的品种池中的SNP(图5和5度)。然而,含三种SNP的池彼此不区分。超过300bp的扩增子影响1:18稀释度的SNP检测的可重复性,但不是1:6或1:12稀释度(如。额外的文件3.)。这些数据表明,在桃树中,将基因库扩大到12个基因型是可行的。
标准PCR和冷PCR后混合样品HRM比较.a . 131 bp扩增子的标准PCR/HRM分析PpAG外显子4.在六(Y4),12(Y4 + Y5)和十八(Y4 + Y5 + Y6)基因型中,检查栽培品种30。池中的轴承率的百分比显示在括号中。B. 411bp扩增子的冷PCR / HRM分析PpAG外显子4。C.标准PCR/HRM分析来自149bp扩增子PpTFL1外显子4。品种16在6个(X1)、12个(X1 + X2)和18个(X1 + X2 + X3)基因型库中进行了检测。D.来自149bp扩增子的COLD-PCR/HRM分析PpTFL1外显子4。每个池子被检查了一式三份。用LC480基因扫描软件对颜色进行分组。
使用COLD-PCR优先扩增错配DNA,作为一种提高人力资源管理分析汇集样本的灵敏度的手段。T米通过LC480基因扫描软件检测,在85.7°C的温度下,扩增出的单核苷酸多态性片段PpTFL1和81.8°CPpAG.临界温度(Tc)的最佳变性温度范围约小于1°C的T米的扩增子。Tc84.5°CPpTFL1和80.7°CPpAG导致PCR扩增产物与DNA错配(图)4 B和4 d)。对于这两个基因,在混合样本中检测SNPs的敏感性相对于单独含有SNPs的品种(绿线)增加。经冷pcr后,LC480基因扫描软件可以区分1:6池(红色)和更大池的SNPs熔化谱。COLD-PCR结果与小于150 bp的扩增子一致(图)5 b和5 d),但检测的扩增子超过300 bp(未显示)时则没有。
DNA模板数量和质量对人力资源管理分析的影响
QMC-PCR的两个重要特点是:(1)利用不同可用性的基因组模板生产初始PCR产物;(2)利用得到的PCR产物作为模板,使用嵌套引物分析多个DNA区域。
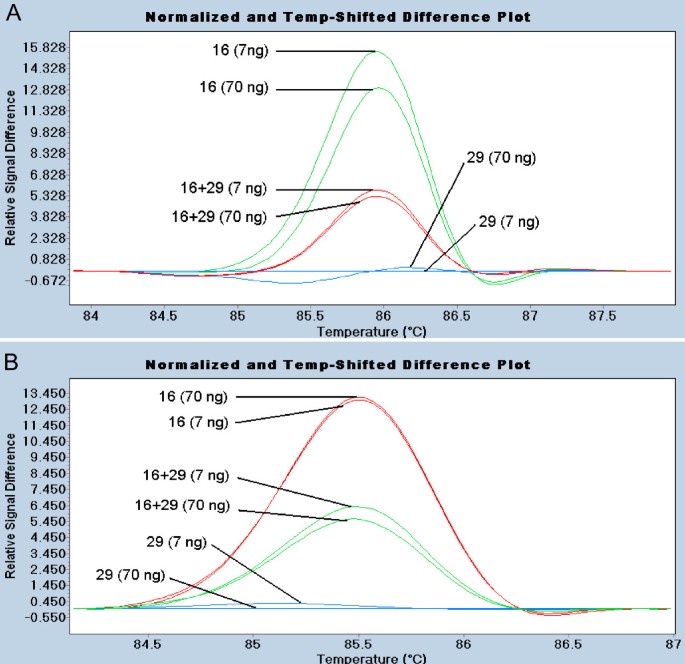
首先使用带有(品种16)和不带有(品种29)SNP的基因型检测了基因组模板的10倍差异TFL1外显子4。当这些品种被单独和一起分析时,7和70 ng模板浓度的HRM结果相似(图6)。
来自上述实验的PCR产物用作用嵌套引物扩增的内部区域的模板。HRM结果与基因组DNA获得的结果类似,不同之处在于较短扩增子的熔化温度较低约0.5°C(图6 b)。这些结果表明,PCR产物可以作为HRM的模板,初始模板浓度的10倍差异并不影响SNP检测。像QMC-PCR这样的技术可以减少模板数量和质量的变化,从而简化从大型植物种群中提取DNA的过程。
讨论
人力资源管理分析PpTFL1和PpAG36个桃品种的等位基因在编码区均有一个多态性位点。共鉴定出7个具有SNPs的品种,每基因型筛选3374 bp序列。来自个体和汇集样本的野生型熔化剖面与14个扩增子的测序结果相一致,因此不太可能有额外的SNPsPpTFL1和PpAG外显子。比较完整PpTFL1桃品种Lovell和nemarared的基因组序列发现内含子1存在SSR变异,但外显子序列未发现多态性[17].
以桃为对照,对25个品种的杏仁(李属dulcis)在编码序列中检测到大量SNPs,平均频率为1:57 bp [18].橄榄(Olea Europea.),一种异交种如杏仁,在307 bp的区域内变异巴经人力资源管理委员会审核[19].38个橄榄品种中有16个品种在该区域的一个或两个多态性位点上有snp位点。观察到的低水平的遗传变异PpTFL1和PpAG可能是桃子中的自相容性的后果,美国东部的品种狭窄的遗传基础[20.,21].此外,可能有针对编码区突变的选择PpTFL1和PpAG,是桃的单基因[16,17].
PpTFL1和PpAGSNPs被用来检验增加人力资源管理吞吐量的方法。通过对12个品种的DNA库进行标准PCR, HRM分析一致地鉴定出含有snp的品种。这个基因库的规模比以前报道的四或五种基因型的基因库要大[3.,10,可能是由于仪器或基因组大小的差异。HRM可以检测到在野生型DNA中以1:200的比例稀释的变异序列[12].然而,HRM对不同个体汇集的DNA的敏感性低于用单一来源的DNA稀释的变异序列[3.].
Cold-PCR增加了合并样品的HRM分析的敏感性PpTFL1和PpAG单核苷酸多态性。在COLD-PCR之后,混合样本的熔化谱更接近于单个包含snp的基因型的熔化谱,可能是通过对序列变体的富集。在稀释实验中,HRM with COLD-PCR的检出限在1%以下[8].在这项研究中,尽管超过18种基因型的样本库没有进行检查,但检测到的变异序列占汇集DNA的2.8%。由于扩增子大小的限制,冷pcr在基因分型方面可能比突变扫描更有用。冷pcr已获批用于医学诊断和进一步研究[如。可以扩大这项技术的适用性。]
尽管原始PCR反应中的基因组模板存在10倍差异,但HRM结果是由PCR衍生的模板生产的嵌套产品一致。这表明QMC-PCR等方法可以降低来自高通量样品制备的高度纯化的DNA。QMC-PCR捕获固定档案组织中的完整目标区域的可变水平,其中DNA降解是有问题的[9].具有人DNA的稀释实验发现,QMC-PCR可以检测存在于野生型DNA背景的2.5%的变体序列。
与QMC-PCR和COLD-PCR相比,Sanger测序检测不到总DNA中20%以下的突变[8,9].然而,下一代测序具有相当大的潜力,可以作为一种突变筛查工具,当采用区分突变和测序错误的策略,并使用样本汇集来提高成本效率时[23].例如,罗氏454测序被用于在汇集的番茄样本中识别ems诱变的候选基因[24Petunia [25].直接比较焦磷酸和冷PCR或QMC-PCR增强的HRM,发现改良的HRM分析具有相同或更低的检测限[26,27].像人力资源管理这样检测不匹配DNA的诊断方法可以作为测序的替代或补充。
结论
HRM突变扫描可以识别出基因外显子中的SNPsPpAG和PpTFL1在一小组桃品种中。利用这些基因中具有SNPs的品种,可以确定在12个基因型库中可靠地检测到多态性。研究发现,冷pcr提高了混合样品HRM分析的灵敏度,但在小扩增物中效果最好。对另一种HRM修饰QMC-PCR的检验表明,用于进一步分析的初级PCR产物可以从不同水平的基因组DNA中产生,这为简化高通量DNA分离提供了一种方法。临床诱变筛选技术的发展可以在作物的定向诱变育种中发挥作用。
方法
基因序列和引物
的PpAG基因组序列为GenBank FJ184275PpTFL1基因组序列来自洛弗尔品种[17].内含子/外显子结构PpAG和PpTFL1通过使用蜘蛛侠对齐计划来确定[28比较它们的基因组序列PpAGmRNA (GenBank AY705972)和MdTFL1mRNA(Genbank AB366643)分别。信标设计器7软件(Premier Biosoft)用于设计寡核苷酸引物以扩增外显子区(附加文件4)。引物由MWG Operon (Huntsville, AL)合成并hplc纯化。
基因组DNA分离及PCR模板制备
36个桃品种的叶子(附加文件5)在美国农业部东南果树坚果研究实验室(Byron, GA)采集。使用DNeasy Plant试剂盒(Qiagen)分离总DNA,并使用NanoDrop 800分光光度计(Thermo Scientific)进行定量。从单个品种或样本库中提取的DNA共30 ng用于PCR。将6个品种的主库进行组合,检测12个和18个品种的大库。在QMC-PCR实验中,利用引物TE3MF和TE4R分别对16和29个品种的基因组DNA进行7 ng或70 ng的PCR反应。以PCR产物为模板,用ddH 1:100稀释扩增产物2O,稀释2 μl,用引物TE4F和TE4R扩增内片段。
PCR和COLD-PCR
PCR用Mastercycler (Eppendorf)进行,反应体积为20 μl,包含30 ng DNA,引物0.2 μM,氯化镁2.5 mM2和2x roche hrm master mix(用qualight dye)。将反应在95℃下变性3分钟,然后在20s,55℃下的45℃,55℃,72℃持续30s,最终延伸,在72℃下进行5分钟。冷PCR在含有已知野生型的其他品种的游泳池中用含有品种16或30进行的。对于Ptfl1.外显子4 SNP分析,包括X3、X3 + X2和X3 + X2 + X1(图)1)。分析对PpAG外显子4 SNP使用了Y6、Y6+Y5和Y6+Y5+Y4组合。冷pcr条件Ptfl1.外显子4为:95°C 3分钟;20个95℃的20℃,20s,20s,72℃的30s,30s;异络通过94°形成30秒和70℃的8分钟;和20s,20s,20s,61℃的20个循环,72℃,25℃。冷pcr条件PpAG除了最终20次循环之外,外显子4类似于:80.7℃,20s,20s,72℃,25℃。
高分辨率熔化分析和扩增子测序
在LightCycler 480 (Roche Diagnostics)上,PCR产品在95°C条件下变性1分钟,冷却到40°C条件下变性1分钟,然后以0.02°C/秒的速度加热到95°C,同时持续测量荧光,获得25个数据/°C。DNA熔化数据通过LC480基因扫描软件进行分析,灵敏度设置为0.3和5。所有PCR/HRM实验均至少重复3次。测序时,PCR产物通过琼脂糖凝胶电泳分离,使用PureLink™Quick gel Extraction kit (Invitrogen)纯化。DNA样本由MWG操纵子测序(Huntsville, AL)。
缩写
- SSCP:
-
单链构象多态性
- 耕种:
-
靶向诱导基因组的局部病变
- COLD-PCR:
-
降低变性温度-PCR的共扩增
- QMC-PCR:
-
quick-multiplex-consensus-PCR
- neattill:
-
从排列组织中提取核酸用于TILLING
参考资料
- 1.
Julio E, Laporte F, Reis S, Rothan C, de Borne FD:通过诱变育种降低烟草中去硝烟碱含量。植物营养学报。2008,21:369-381。10.1007 / s11032 - 007 - 9138 - 2。
- 2.
董超,王志强,王志强,等:一种改良的小麦耕作方法。植物学报。2009,2:39-47。10.3835 / plantgenome2008.10.0012。
- 3.
Gady Alf,Hermans FWK,Van de Wal MHBJ,Van LoO en,Visser RGF,Bachem CWB:在新应用中实施两种高通量技术:检测大EMS突变植物群中的点突变。植物方法。2009,5:13-10.1186 / 1746-4811-5-13。
- 4.
McCallum CM, Slade AJ, Colbert TG, Knauf VC, Hurst S:番茄多半乳糖醛酸酶活性的降低是由多半乳糖醛酸酶基因的非转基因突变引起的。美国专利7393996。2008
- 5.
Dahmani-Mardas F, Troadec C, Boualem A, Leveque S, Alsadon AA, Aldoss AA, Dogimont C, Bendahmane A:使用TILLING方法改良瓜果货架期。PLoS ONE。2010年,5:e15776 - 10.1371 / journal.pone.0015776。
- 6.
Suzuki T, Eiguchi M, Kumamaru T, Hikaru Satoh H, Matsusaka H, Moriguchi K, Yasuo Nagato Y, Kurata N: mnu诱导的突变库和高性能TILLING能够在水稻中发现任何基因突变。中国生物医学工程学报。2008,29(6):513 - 514。10.1007 / s00438 - 007 - 0293 - 2。
- 7.
Cross MJ, Waters DLE, Lee LS, Henry RJ:内部标记的核内溶酶突变分析(EMAIL)。电泳。2008,29:1291-1301。10.1002 / elps.200700452。
- 8.
Milbury CA, Li J, Makrigiorgos GM: cold - pcr增强的高分辨率熔化能够快速和选择性地识别低水平的未知突变。临床化学杂志。2009,55:2130-2143。10.1373 / clinchem.2009.131029。
- 9.
Fadhil W, Ibrahem S, Seth R, Ilyas M:快速多重共识(QMC)-PCR接高分辨率熔解:一种用于福尔马林固定石蜡包埋组织突变检测的简单而可靠的方法。临床病理杂志。2010,63:134-140。10.1136 / jcp.2009.070508。
- 10.
李捷,楚茨兹,刘XG,静兴,刘yg,郝越:一种使用植物中的Evagreen染料的高分辨率高分辨率熔化方法,植物中的DNA多态性检测和基因分型。j int植物biol。2010,52:1036-1042。10.1111 / J.1744-7909.201010.01001.x。
- 11.
Xiao J, Bastian RW, Perlmutter JS, Racette BA, Tabbal SD, Karimi M, Paniello RC, Blitzer A, Batish SD, Wszolek ZK, Uitti R, Hedera P, Simon DK, Tarsy D, Truong DD, Frei KP, Pfeiffer RF, Gong S, Zhao Y, LeDoux MS:原发性肌张力障碍TOR1A高通量突变分析。中国生物医学工程学报。2009,10:24-
- 12.
Bastien R, Lewis TB, Hawkes JE, Quackenbush JF, Robbins TC, Palazzo J, Perou CM, Bernard PS:使用高分辨率荧光熔化曲线分析和自动突变呼叫的TP53基因高通量扩增子扫描乳腺癌。基因工程学报。2008,29:75-764。
- 13.
李家,王L,Mamon H,Kulke MH,Berbeco RGM,Makrigiorgos GM:用冷PCR替代PCR富集变体DNA序列并重新定义基因检测的敏感性。自然医学。2008年,14:579-584。10.1038 / nm1708。
- 14.
Sreelakshmi Y, Gupta S, Bodanapu R, Chauhan VS, Hanjabam M, Thomas S, Mohan V, Sharma S, Srinivasan R, Sharma R: NEATTILL:一种从阵列组织中提取核酸的简化程序,用于TILLING和其他高通量反向遗传应用。植物学报。2010,6:3-10.1186/1746-4811-6-3。
- 15.
Martin T, Hu M, Labbe H, McHugh S, Svircev A, Miki B:PpAG1,同源际无性生殖的,表达桃花和水果。可以j bot。2006,84:767-776。10.1139 / b06-031。
- 16.
Tadiello A, Pavanello A, Zanin D, Caporali E, Colombo L, Rotino GL, Trainotti L, Casadoro G: A满桃类基因参与心皮的形成和随后向肉质果实的转化。中国生物医学工程学报。2009,30(6):651-661。10.1093 / jxb / ern313。
- 17.
Zhebentyayevaa T梁H, Olukolua B,王尔德D, Reighard GL, Abbott:比较基因的染色体区域包含一个秩序终端花1杏和桃的同源性揭示了被子植物间的微同系。植物科学学报,2010,33(6):751 - 756。10.1016 / j.plantsci.2010.06.018。
- 18.
Wu Sb,Wirthensohn Mg,Hunt P,Gibson JP,Sedgley M:杏仁SNP的高分辨率熔化分析衍生自ERES。Al Appl Genet。2008,118:1-14。10.1007 / S00122-008-0870-8。
- 19.
Muleo R, Colao MC, Miano D, Cirilli M, Intrieri MC, Baldoni L, Rugini E:利用高分辨率DNA熔解技术对橄榄种质进行突变扫描和基因分型。基因组学杂志。2009,52:252-260。10.1139 / g09 - 002。
- 20。
黄志刚,李志刚:美国东部砂石桃品种的近交系和共祖先及其对桃种质改良的启示。中国科学:地球科学。
- 21.
Aranzana MJ,Abbassi Ek,Howad W,ArúsP:遗传变异,人口结构和桃子商业品种中的联系不平衡。BMC遗传学。2010,11:69-
- 22.
Milbury CA, Li J, Makrigiorgos GM: Ice-COLD-PCR能够对低丰度的未知DNA突变进行快速扩增和稳健富集。核酸研究,2010
- 23.
Gilchrist E,Haughn G:反向遗传技术:植物中的工程损失和基因功能的增益。功能基因组学的简报。2010,9:103-110。10.1093 / BFGP / ELP059。
- 24.
Rigola D, van Oeveren J, Janssen A, Bonne A, Schneiders H, van der Poel HJA, van Orsouw NJ, Hogers RCJ, de Both MTJ, van Eijk MJT:使用KeyPoint™技术的诱导突变和自然变异的高通量检测。PLoS ONE。2009年,4:e4761 - 10.1371 / journal.pone.0004761。
- 25.
Stuurman J:具有特定突变的植物的方法。美国专利申请。美国2010/0212043
- 26.
Ibrahem S, Seth R, O’sullivan B, Fadhil W, Taniere P, Ilyas M:焦磷酸测序和QMC-PCR结合高分辨率熔解用于KRAS/BRAF突变检测的比较分析。[J] . acta physica sinica, 2010, 32(5): 457 - 461。10.1111 / j.1365-2613.2010.00733.x。
- 27.
Pinzani P, Santucci C, Mancini I, Simi L, Salvianti F, Pratesi N, Massi D, De Giorgi V, Pazzagli M, Orlando C:BRAFV600E通过COLD-PCR,黑色素瘤的检测得到了极大的提高。中华医学杂志。2011
- 28.
蜘蛛侠:信使rna到基因组比对的工具。基因组Res. 2001, 11: 1952-1957。
确认
我们要感谢Tetyana Zhebentyayeva博士和Bert Abbott博士(克莱姆森大学)提供的PpTFL1基因组序列。我们感谢汉中奥曼·奥姆曼博士和威廉奥(Usda东南水果和树坚果研究实验室),用于来自桃子的叶片。对YC的博士后支持由UGA研究基金会和农业和环境科学学院提供。
作者信息
从属关系
通讯作者
附加信息
作者的贡献
YC设计并进行了实验。YC和HDW分析了数据。HDW构思了这项研究。HDW和YC促成了手稿准备,并读取并批准了最终手稿。
电子辅料
外显子4序列
额外的文件1:PpTFL1外显子4序列.PCR产品跨越PpTFL1对9个桃品种进行了外显子3和4的序列分析。仅显示第4外显子中多态性位点侧翼的序列(箭头);其余的序列是相同的。对16和29个品种的PCR产物进行了亚克隆,并进行了测序,确定了包含snp的等位基因。(PDF 37 KB)
36个桃品种的个体人力资源管理分析
附加文件2:。PCR产品跨越PpTFL1每个品种在不同的反应中扩增外显子3和4,并通过HRM进行分析。当重复人力资源管理时,品种16、28和29显示了融化模式的改变,但品种21没有。(PDF 21 KB)
人力资源管理分析
附加文件3:PpAG外显子4 + 5.在六(y4),12(Y4 + Y5)和十八(Y4 + Y5 + Y6)线上的池中检查栽培品种30。348BP扩增子跨越熔化谱的相对差异曲线PpAG显影外显子4和5。组名称是指图2A所示的汇集策略。HRM分析以三份和线颜色进行,表示通过LC480基因扫描软件进行分组。12倍池的重复始终如不地区分野生型线(Y4),但是18倍的池。(PDF 44 KB)
作者为图像提交的原始文件
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
陈永强,陈永强,王志强,桃花基因的突变分析。BMC植物杂志11,96(2011)。https://doi.org/10.1186/1471-2229-11-96
收到:
接受:
发表:
关键词
- 高分辨率熔化
- 融化的概要文件
- 高分辨率熔化分析
- 粪床
- 桃品种