抽象的
背景
许多非常有益的性状(例如疾病或非生物胁迫性)通过与野生亲属的交叉转移到庄稼中。13种公认的番茄(茄属植物部分番茄)与彼此密切相关,野生物种基因已广泛用于改善作物,Solanum lycopersicum此外,地理屏障的缺乏使得自然杂交成为可能S. lycopersicum.它最近的相对茄属植物pimpinellifolium在厄瓜多尔,秘鲁和智利北部。为了更好地了解模式S. lycopersicum.diversity, we sequenced 47 markers ranging in length from 130 to 1200 bp (total of 24 kb) in genotypes ofS. lycopersicum.野生西红柿S. Pimpinellifolium.,茄属植物奥秘,Solanum Peruvianum.,Solanum Pennellii.和茄属植物habrochaites。与六至十基因型比较每个标志物分析。几个标志物此前被假设为内携带野生种等位基因S. lycopersicum.,即隐性基因渗入。
结果
每个标记都有高置信度(e<1 x 10)-30)使用Blastn对番茄全基因组霰弹枪染色体(SL2.40)数据库的单个基因组位置。邻近的树木表现出高平均举射支持(86.8±2.34%),以区分从绿色炸弹的分类群中的红色粉末38个标记。杂交和剖视分裂网络,标记的基因组地图相对于记录的血栓引入的基因组地图,以及历史起源的历史起源用于解释九个标记的进化模式,具有令人血栓的等位基因。
结论
在这项研究调查了47个遗传标记,4人参与渗入繁殖过程中9号染色体上连锁累赘,而等位基因在5个标记显然源于自然杂交S. Pimpinellifolium.并与原始基因型相关联S. lycopersicum.。作物种类中血栓碱基因的阳性鉴定如S. lycopersicum.将有助于作物种质多样性的保护和利用,例如,有助于消除不良连锁阻力或开发新的有利等位基因。
背景
渐渗是指一个物种的基因通过杂交转移到另一个物种的基因库中。作为一种现象,它已成为动植物遗传学研究的一个重要课题。例如,基因渗入已经与现代人类的适应有关[1],并引起保育生物学家的关注,因为野生鸟类和哺乳动物的种群已丧失完整性[2,3.].在植物中,血栓球是通过转基因(GM)作物对天然群体污染风险的关键概念。更常见的是作物,野生亲属的有利基因被故意转移成品种发育的育种线。这对于遗传多样性相对较低的作物物种特别有价值。
根据作物渐渗育种综述[4从野生物种转移的有利性状的主要功能类别是抗或耐非生物胁迫或疾病、产量、杂种生产的细胞质雄性不育或育性恢复以及品质性状。在19世纪末,野生作物近亲的开创性使用中th早期的20th几个世纪是将疾病抵抗转移到葡萄(葡萄) [5]和甘蔗(Saccarum OfficinArum.) [6].1986年对23种作物的回顾估计,美国每年总经济价值的6%是由作物的野生亲缘关系贡献的[7].对于全球重要性的13个主要作物,据估计,释放品种已经用于46种野生物种,而且,血液育种方法正在增加[4].系谱信息的缺乏、私营部门内未发表的活动和分类的变化是造成作物渗入育种集体影响的不确定性的一些因素[4].
美国最早的番茄血液血液繁殖可能是通过法国品种的Merville desMarchés间接和不知疲倦地进行的。最近为Merville desMarchésPI109834收集的表型数据显示它在水果尺寸和平滑度下变化(http://www.ars-grin.gov/cgi-bin/npgs/acc/display.pl?1129442);它的基因型是分离的,表现出群体混合,并且与许多其他的遗传距离是一个离群值S. lycopersicum.登记入册(8,9].我们假设这些是S. Pimpinellifolium.在其祖先中(这一观点在当前的研究中得到了验证)。的镰刀菌素抗枯萎处理品种Marvel [10)选自20世纪初的Merville des Marchés, Marvel是1925年发行的《Marglobe》的母公司[11,而在20世纪30年代至50年代后期的许多重要品种的系谱中都可以发现这种差异(H.M. Munger的番茄系谱图由E.D. Cobb提供,康奈尔大学,2012)。在美国,番茄与野生物种的直接渗透始于20世纪30年代,与此同时,人们也开始了对地理起源中心的采集。第一个释放的品种,由Marglobe xS. Pimpinellifolium.,被恰如其分地命名为泛美[12].基因渗入育种番茄努力增加全球第二次世界大战后,涉及范围广,性状的筛选和所有野生番茄品种[13].今天使用精致的工具,这种努力仍然优先考虑,例如基因发现的速降图书馆等血统图书馆[14,15].
在11个品种(木薯、小麦、小米、水稻、玉米、向日葵、生菜、香蕉、马铃薯、花生、番茄)的96个品种中,番茄(Solanum lycopersicumL.);在水稻和土豆中发现了下一个最高数量,每次具有12个特征[4].强调和的基因渗入在番茄育种成功涵盖若干个因素,包括其固有遗传基础狭窄,相对容易交叉的与几个野生类群,生产需求根据生长条件和市场定位,其易感性害虫和病原体,其对非生物灵敏度因素。除了抗性或耐受性几十个细菌,病毒,真菌,昆虫,线虫病原体,数百个非生物胁迫抗性,花和果实性状,产量和工厂结构有利基因或数量性状位点(QTL)已被映射在野生番茄品种[16],因而具有可开发的潜力。
血液育种繁殖携带成本,即通过重复的回复而消除的非靶向基因座的遗传联系。尽管回复,Linkagage Drag可以在基因组中持续存在,特别是如果抑制重组。使用分子标记量化番茄和其他作物中的联动阻力的几个例子[17- - - - - -21].连锁累赘可以表示该变得无意中掺入育种品系或品种有利,有害的或中性的等位基因。
在这项研究中,我们使用了术语“隐渐渗”[22]描述潜在的遗传变异S. lycopersicum.来源于野生番茄品种它的起源可以被引出各种各样的场景,从连锁拖拽,野生之间的杂交S. lycopersicum.以及野生近亲,通过风或昆虫媒介与渗入的品种的花粉在开放授粉的种群中进行杂交[23].在美国农业部、农业研究局(USDA, ARS)植物遗传资源单位(PGRU)保存的种质资源中,隐渐渗是很有意义的,因为它可以显示可供最终用户利用的新的遗传变异,或者相反地,揭示在作物改良方面的不利或不良等位基因。
在以前的报告中,我们假设在番茄种质资源面板中重测序的DNA标记中有5%到10%的隐性渐渗检测[9,24,25].本研究的目的是通过重测序和分析几个野生番茄种质和一个杂草种质中的相同标记来收集这些等位基因的新证据S. lycopersicum.(桌子1).虽然野生物种基因库的变异使得试图发现100%相同的同源等位基因变得不可行,但假设基因渗入与特定野生物种的等位基因的关系更密切,而不是与它们的等位基因的关系S. lycopersicum.同源染色体。为了鉴定渐渗等位基因,我们还使用了来自标记定位、表型描述、株系和亲本历史起源的证据。
结果与讨论
标记和在网上映射
对于本研究中使用的47个标记(附加文件1表S1),用于PCR和测序的核苷酸引物最初是针对S. lycopersicum.除Conserved Ortholog Set II (COSII或C2)和unigene (U)标记外的序列[27,28].分子标记在野生番茄远亲种间的转移成功率较高S. lycopersicum.和美国pennellii(例如见[29- - - - - -31])。在目前的研究中,有些引物对在每个野生番茄样品中都没有扩增或给出干净或同源的reads。COSII和U标记在成功生成高质量序列数据方面并不优于其他标记,因为它们大多含有内含子,有时在杂合条件下携带小的内含子。这些杂合子是导致读取质量差和数据丢失的主要原因。
在我们当前的种质小组(表1),S. lycopersicum.和S. Pimpinellifolium.没有遗漏的标记;在最终的47个标记中,4个绿果类群中至少有2个序列可用。美国peruvianumLa1537,美国peruvianumG32592,美国秘密,美国pennellii和美国habrochaites分别给出32、42、40、35和38个标记点的数据。后两个品种通常是自交不亲和的,在自交系中所携带的多态性标记数比自交系多美国peruvianum和美国秘密这是抽样的。跨越多态性标记的平均SNP频率为0.0127(n= 17)美国habrochaites0.0082 (n= 17)美国pennellii0.0126 (n= 5)美国peruvianumLA1537, 0.0058 (n= 2)美国peruvianumg32592和0.0078(n= 7)秘鲁野生。TA496 Tomate和美国秘密在任何标记中都没有多态性位点。
S. Pimpinellifolium.多态性异常高,为0.0055 (n= 17)相对于其他自相容类群。利用SSR (simple sequence repeat, SSR)基因分型分析,将该新加入群体归类为混合群体S. Pimpinellifolium.人口结构(32,所以自然代表两种不同S. Pimpinellifolium.基因组。因为种子来源可追溯到美国沃恩种子公司和加拿大安大略省园艺实验站[33,另一种可能性是S. lycopersicum.被其中一个实体纳入了它的谱系。这一点可以通过比较以前的估计得到证明D,每kb的突变数,[34]在两个物种之间[35,大约是我们下面报告的估计的四倍。
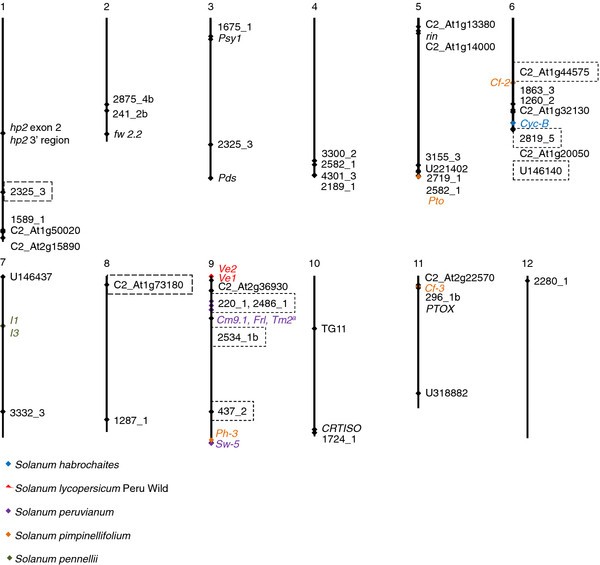
大多数标记被高度可靠地定位到基因组内的单个地图位置(图)1额外的文件1:表S1)。这是第一份全基因组序列[36]和基于网络的工具可用来完成基于表达序列标记(EST)的标记[24].据此,对12个est标记进行了新定位,并对437_2、2189_1和2819_5标记进行了修改,以验证限制性片段长度多态性(RFLP)标记的一致性[25].所有染色体均由47个标记表示;每种染色体的标记数范围从染色体12-8上的一个染色体6.在染色体上观察到小的间隙,但仔细检查显示这些是富含Poly A或Poly T.的掩蔽区域,只有标记1675_1才能最初提供任何命中。一个缩小的严格程度为1 x 10-30随后找到了可能的位置。
四个标记有两个BLASTN每次击中。它们在染色体1和3,3,2582_1的染色体1和3,5,2和5的2325_3上的两个紧密连接(2,849nt分开)序列的1260_2,以及2819_5的两个连接(14,114 nt分开)染色体6.检查引物区域发现在次级击中的预测引物结合位点进行多种错配;假设这些被扩展为单拷贝。对于标记2819_5,前后底漆具有单个不匹配,并且反向没有错配。失配区域中的预测扩增子与参考TA496序列相同的86%。当Mega簇分析中包含不匹配序列时,它与87%的引导支持的所有其他(野生和栽培)等位基因分开,平均值D序列中增加了8倍(D= 1.5n= 10个等位基因对D = 12 forn= 11个等位基因)。因此,这种类比不太可能放大和混淆结果。本实验室已对所有标记进行了单带扩增和内高纯合序列的筛选S. lycopersicum.[24,25].的结果在网上作图证实,这套标记在单拷贝取样中提供了可靠的结果S. lycopersicum.基因。所有序列存入欧洲分子生物学实验室(EMBL),欧洲核苷酸存档(ENA)数据库,登录号HE977919-HE978211。
类群间的聚类模式和差异估计
PI 99782番茄被选择用于当前的研究,代表一个“前渐渗育种”基因型。它的果实小,略具棱纹,未改良,有疤痕和开裂(http://www.ars-grin.gov/cgi-bin/npgs/acc/display.pl?1127604.)并且是纯合的普通S. lycopersicum.已测序的50个标记中的48个为单倍型[25].38个标记对红果分枝有较高的自举支持度S. lycopersicum.包括Tomate和S. Pimpinellifolium.等位基因(附加文件1:表S2)。Bootstrap值的范围从54%到100%(平均±标准误差= 86.8±2.34%)。这些从100%中减去的引导值可以被解释为I型错误概率的估计,即错误地接受一个不是由数据表示的集群(见[37]讨论)。
结果强调了二者之间的密切关系S. lycopersicum.来S. Pimpinellifolium.因为他们的等位基因经常相同或高度相似。绿果种相对于红果种的位置在不同位点之间存在差异。这可能是不完整的谱系分类或基因渗入造成的[38].一个例子是美国habrochaites附近S. Pimpinellifolium.标记符TG11(附加文件2:图S1);Nesbitt和Tanksley也报道了这个例子[35].
在不支持红果进化支的9个标记中,有7个先前被假设为携带基因渗入(附加文件)1:表S2)。这些是下面讨论的。另外两个标记,1287_1和2280_1,显示了包含的模式美国peruvianum在红果进化枝内(附加文件2:图S1)似乎是由于缺乏分辨率。在标记1287_1美国peruvianum单倍型只有一个或两个来自tomate的突变台阶。这些突变未与任何其他分类征分享。标记2280_1只观察到四个独特的单倍型。这些曾经是美国habrochaites美国pennellii美国秘密和{TA496 Tomate,S. Pimpinellifolium.,秘鲁野生,g32592,la1537}。对于支持红炸弹的人的38个标记,通常支持番茄物种中的接受分类间关系[39,40].
散度的估计(D由于突变率和重组率的差异、选择限制以及配子随机抽样和人口统计等各种因素的影响,位点之间的差异与进化时间的高方差有关。平均D4301_3标记为5个突变,C2_At1g44575标记为52个突变/ kb(平均±标准误差= 16.5±1.29)。在总尺度上,平均值D±标准误差为:TA496D= 0.001±0.0005,秘鲁野生D= 0.002±0.0006,S.醋栗d = 0.002 ± 0.0004,美国秘密D = 0.020 ± 0.0022,美国peruvianumLA1537D = 0.020 ± 0.0025,美国peruvianumG32592D = 0.022 ± 0.0024,美国habrochaites D = 0.021 ± 0.0024 and美国pennellii D = 0.022 ± 0.033.
缺乏精确解析区别分类群的聚类,或平均散度Tomate的green-fruited类群是共享的功能多态性在许多情况下,例如,在24标记至少一个单核苷酸多态性(SNP)在一个物种也对其他物种之间的隔离。这在其他关于作物物种及其近缘野生亲缘物种的研究中已被观察到([41]及其参考文献)。此外,随机采样导致分辨率低,例如:美国peruvianum单倍型似乎是基于少数无信息的SNP标记物在从1287_1衍生TOMATE。
渐渗现象的证据
在以前的研究中,我们报道了9种标志物(见表)2)内有高度分化的等位基因S. lycopersicum.并假设这是由于野生物种的渗入。分别为220_1,437_2,2325_3,2534_1R(重新设计为2534_1b), 2486_1 [24],2819_5,C2_AT1G73180 [25],U146140和C2_AT1G44575 [9].在目前研究的47个标记中,这9个标记中有7个显示出分枝图的模式,没有将红果分枝的成员聚在一起(附加文件)2:图S1)。杂交网络(附加文件2:图S1)、已记录的抗疾病等位基因(例如[16,26,42- - - - - -44])被用作总证据,以将分歧标记分类为推动的联系阻力在血液育种期间与自然交叉相比S. Pimpinellifolium.(桌子2).该分类并未构成自然杂交的证据与血液繁殖。然而,这是一个有用的概念,从中综合独立证据,可以作为两种情况的未来假说测试的基础。
在TA496携带等位基因的4个标记中,至少有3个标记与红果分支的所有其他成员存在高度的连锁阻力。所有4个染色体都被定位到9号染色体,长度从5.77 MB到54.71 MB2).9号染色体上的抗病基因座渐渗[42] 包括VE1(0.06 MB)VE2(0.05 MB)来自秘鲁野生,FRL.(实体地图位置未标注),TM-2一个(13.62 MB)和SW-5(67.30 MB),所有三个从美国peruvianum, 和Ph-3(66.71-66.78 MB)来自S. Pimpinellifolium.(图1、表2).在9号染色体5.77 ~ 17.00 MB的3个标记的分支图中,LA1537是与TA496最接近的等位基因TM-2一个位于13.62 MB的位置。LA1537是一种跨交配的加入,它来自PI 128650,原始来源TM-2一个(桌子1).
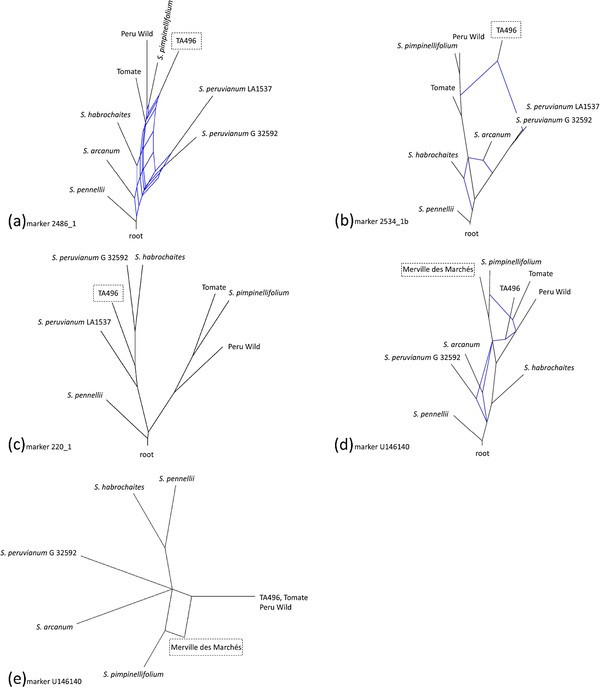
杂交网络放置TA496在各种位置的每个周围的三个标记的TM-2一个:靠近红磨的等位基因,返回标注物美国peruvianum标记2486_1(图2a和附加文件2:图S1),在红果等位基因和美国peruvianum标记为标记2534_1b的标记物(图2b和附加文件2:图S1),并且在绿果野生物种之间的等位基因内美国peruvianum标记220_1无网格(图2c和附加文件2:图S1)。尺寸TM-2一个通过在隔离F中绘制来估计狭窄的区域2人口(19].9号染色体位置50.41 MB的RFLP标记TG101与(≤1 cM)紧密相连TM-2一个。的染色体位置TM-2一个已经被描述为非常靠近Centromere的非常靠近极度压抑的重组[47].因此,220_1 (5.77 MB)、2486_1 (5.90 MB)和2534_1b (17.00 MB)可能是TA496基因导入片段的一部分。
在标记处分裂网络的例子与在美国lycopersicum。虚线框表示具有渐渗等位基因的加入(a-d:以标记U146437作为对照的杂交网络,e: parsimony split网络);一个)TA496显示了网状化美国peruvianumLA1537等绿果品种,b) TA496与红果和绿果品种呈网状结构;c)TA496嵌套绿色结实物种中,dMerville des Marchés PI 109834与Merville des Marchés PI 109834密切相关S. Pimpinellifolium.,与Tomate PI 99782呈网状,e)Parsimony分裂网络仅使用标记内的保守,全局信号来说明Merville desMarchésPI109834的混合起源。
TA496等位基因在标记437_2处(54.71 MB)与dna序列无明显相关性美国peruvianum而它的起源就更难解释了。该进化枝图将TA496置于两个进化枝之间的中间位置,即{S. Pimpinellifolium.,TOMATE,美国秘密、秘鲁,美国peruvianum}和{美国habrochaites美国pennellii}(附加文件2:图S1)。杂交网络呈网状美国habrochaites和红果等位基因(附加文件2:图S1)。番茄品种常见的几种基因起源于美国habrochaites(Cf-4TM-1Ol-1Cyc-B和▽)或美国pennellii(我1和我3.),但这些都不能映射到9号染色体[43,45].表中列出的染色体9个血栓竞争2被判断为基于对在线种子目录(未发表意见)的非正式调查的非正式调查以及对番茄育种的全面评论(例如[16,26,42- - - - - -44])。
TA496中437_2的链接阻力Ph-3(66.71 MB)或SW-5(67.30)被拒绝,因为它们来自S. Pimpinellifolium.和美国peruvianum, 分别。根据BLASTN搜索TA496标记437_2的针对茄科PlantGDB组装唯一Transcipts(放置)在茄科基因组学资源数据库(http://solanaceae.plantbiology.msu.edu/), TA496和美国habrochaites在我们重新开始的所有其他等位基因中缺席的核苷酸位置46处共享次异性。这提供了暂定但不确定的证据表明迟滞之间的直接关系美国habrochaites。作为替代证据,FM6203, TA496的前身,据说携带Asc抵抗(pers comm. S. Loewen, University of Guelph, 2005)美国pennellii已在番茄担任的原始来源之一第3染色体[48].
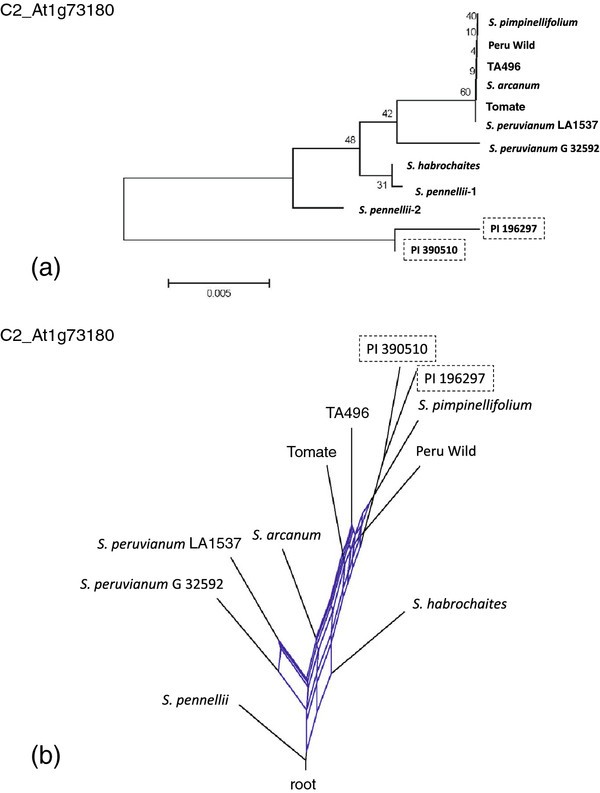
其余5个等位基因分化的标记表现出与遗传渐渗一致的模式S. Pimpinellifolium.在自然种群。在2325_3、C2_At1g44575、2819_5和U146140四个标记上,分化等位基因与红果类群的亲缘关系更近,bootstrap值为82% ~ 100%1虽然标记2819_5和U146140不支持红果等位基因的单系性(附加文件2:图S1)。杂交网络显示红结实品种的等位基因是不同的从绿结实物种等位基因2325_3和C2_At1g44575,而2819_5和U146140显示红结实和绿色结实物种之间的连接(附加文件2:图S1)。标记U146140很好地说明了S. lycopersicum.xS. Pimpinellifolium.Merville des Marchés PI 109834的杂交起源(图2d2e)。标记C2_AT1G73180在PI 196297(以及具有相同等位基因,表的三个额外的辅助方案)显示了一种不寻常的模式2)和PI 390510从集群分析中的所有其他等位基因分析,具有99%的引导支持(图3.a和附加文件2:图S1)。这暗示了一种潜在的类比。然而,没有观察到杂合子,标记没有映射到一个以上的基因组位置,而COSII标记被设计用于扩增高度保守的单拷贝基因[27].
标记C2_At1g73180分化严重。一个)在标记聚类C2_At1g73180表明PI 129026和PI 196297的独特用99%的自举支撑,b)的分裂网络显示PI 129026和PI 196297的结合渐渗(其中见表)2) 和S. Pimpinellifolium.并保留了网状直至根部的古老多态性;后者支持基因搭便车和拒绝选择性中立。
变异的C2_At1g73180等位基因不太可能来自绿色果种,因为8号染色体不携带任何渐渗抗病等位基因[16,42- - - - - -44].杂交网络将PI 196297和PI 390510描述为从S. Pimpinellifolium.,在红果分支的基部有复杂的网状结构,它延伸到绿果分类群,一直延伸到美国pennellii(图3.b)。在PI 196297和PI 390510携带的七种独特多态性中,两个是非保守氨基酸取代,两个是同义词,三个是内血清。一种可能性是在该轨迹处或附近的选择已经导致古代多态性被保留。大量的HKA测试[49] (χ2 = 7.36,P = 0.007) strengthened this interpretation. Therefore, diversity at this marker showed patterns of both natural selection and introgression.
这五个标记所代表的额外证据S. Pimpinellifolium.自然种群的渐渗包括三个来自厄瓜多尔的物种的地理起源,在那里两个物种广泛杂交[50]的原始收集,可追溯到1935年和1938年(可能排除了直接渗透育种的影响),原始果实表型的fasciation(4个材料)或野生樱桃番茄(1个材料,n.b.,樱桃番茄被描述为混合S. lycopersicum.和S. Pimpinellifolium.由(35PI 196297的基因渗入S. Pimpinellifolium.通过瑞克[23].在估计人口结构时[51] PI 109834,PI 129128,PI 258474和PI 258478所有在推断的两个人群中都显示出高概率S. lycopersicum.与种间杂交一致的基因型[9].
结论
这对描述内部的隐渐渗是有帮助的S. lycopersicum.进入纺织萎缩的育种与自然杂交相比S. Pimpinellifolium.,尽管这一分类并不是明确的,应予以进一步审查。野生番茄物种标记的序列在其物理地图位置的背景下加强了我们以前对检测驯化番茄中基因渗入等位基因的解释[24,25].用于精确分辨作物物种中渗透区域的基因组工具越来越多。比较基因分型验证基因渐渗的优点和缺点已经在这里用茄属植物部分番茄分类单元。
在目前的研究中,基因导入的连锁阻力对表型的影响尚不清楚。至少有一个标记(2534_1b)编码一种参与果实成熟的酶(见表)2).在品种或育种线将更加有用估计基因组港口连锁累赘的比例。这应该使用生物信息学可给出的大量公共EST序列数据的这一行和野生番茄种(TA496是可行的http://solgenomics.net/tools/blast/dbinfo.pl.截至2012年6月,报道了323465个番茄红素mrna)。重要的是,对连锁阻力的理解将有助于区分它与作物改良中的选择。先前拒绝中性检测的6个标记中有4个(437_2、2486_1、2534_1b和2819_5)(见表2)2在[25])被发现迟滞而不是选择。预计下一代测序将用于更快地消除作物中的联动阻力[52].
天然杂交物将携带高比例的野生等位基因,使它们稍微易于使用大量分子标记检测。天然又种质的内在价值在共同豆中识别出来(phoudolusulus vulgaris.)作为新等位基因的来源,以获得抗病能力等特性[53].在S. lycopersicum.如果血液凝血的园艺效应是微妙的,那么诸如Merville desMarchés的牧场可能是筛选新等位基因的主要来源。搜索作为当前研究的一部分的加入文献(表2)发现PI 129128表现出较高的番茄红素含量,类似于含有色素突变的品系og.c, hp和dg[54].
最后,值得注意的是,里克·[23报道了自然杂交的潜力S. lycopersicum.与Solanum Chilense.美国habrochaites(以前Lycopersicon hirsutumf。glabratum),Lycopersicon Peruvianum.(现在被修订为四种,[55在智利、厄瓜多尔和秘鲁地区,同域种群密切接触生长,尽管他没有发现基于表型和严重合子后障碍众所周知的证据。从这些地区取样的群体的基因分型将提供证据,以重新检查这些野生番茄物种是否渗入S. lycopersicum.在作物的进化史上扮演了重要角色。
方法
植物材料
本研究中使用的标记基因型以前有报道茄属植物奥秘和S. lycopersicum.[9,25或从5个野生番茄和1个杂草中各新收集S. lycopersicum.加入秘鲁野生。的具体加入美国habrochaites美国pennelliiS. Pimpinellifolium.秘鲁野生历史上服务于番茄品种的重要疾病抵抗等位基因(表1).这两个美国peruvianum选择亲本是因为已知它们是自交系,并且预测它们是高度纯合的。其中的一个,美国peruvianumLA1537,来源于accession PI 128650TM-2一个[56,57].6份供试材料各取2株作为幼苗进行基因组DNA分离和标记测序。本研究中包括的3个样本之前曾发表过标记的序列[25] (桌子1).这些是 - 一种繁殖系列,其血统在其血统中记录了多种TM-2一个(TA496) [57,早于番茄渐渗育种(Tomate, PI 99782)美国秘密(g32591)一种以前归类为自然自受精的植物Lycopersicon Peruvianum.。对于几个标记,从额外发布的序列S. lycopersicum.PI 109834 Merville des Marchés, PI 129026, PI 129128, PI 196297, PI 258474, PI 258478, PI 390510, Table1)被包括在分析中,因为它们以前报告为携带高度发散的等位基因,即推定的血栓引入,在那些基因座中[9,25].
DNA序列
基因组DNA从幼苗中提取,PCR扩增和双通测序如Labate等人所述。[28].最初,来自Labate等人的50个标记的49个。[9测序。这些代表随机基因座,包括表达基因(表达序列标签,EST基),高度保守基因(COSII和U)和任意基因座。标记1523_4被排除在没有测试的情况下,因为它往往会在内部提供差的质量序列S. lycopersicum.。在这项研究期间,标记175_1和1909_2被删除,因为它们并未始终放大S. lycopersicum.野生番茄种类中的同源物,留下47个标记,总共约为24 KB(附加文件1:表S1)。软件包Phred,Phrap和Consed [58,59]和staden [60用于汇编和基本读取调用。Pregap4(版本。(1.5)的Staden包配置为应用一个不同于phred的基调用算法“Estimate Base accuracy”,以独立验证数据。对序列数据进行修剪以去除引物结合位点和低质量末端(phred<40),并在BioEdit中手动对齐[61].所有snp和杂合位点均由两人通过肉眼检查痕迹文件确认。如果来自同一登封者的两种植物序列不同,它们就保持不同;如果它们是相同的,则作为该加入的单一代表性序列处理。杂合位点使用IUPAC核苷酸歧义码手工编辑。GeneSeqer(版本。08十月2008)[62[习惯于将所有标记的外显子和内含子预测与以前的注释进行比较[28].
所有序列使用BLASTN [映射63]对番茄全基因组猎枪染色体(SL2.40)数据库的e值阈值为1 × 10-40在SGN网站[64].使用ITAG2.3 Release:基因组注释,在SGN基因组浏览器中识别标记和邻近区域的基因模型。对于标记437_2,将番茄序列与进化外群体(马铃薯,茄属植物tuberosum)由密歇根州立大学茄科基因组学资源数据库的BLASTN搜索(http://solanaceae.plantbiology.msu.edu/).
统计分析
对47个标记中的每一个,首先采用neighbor-joining (NJ)聚类方法检测基因型(也称为分类群)之间的关系[65],在MEGA 4.0.2中实现[66] 1,000 Bootstrap复制。遗传距离和平均进化分歧(D)是用Jukes-Cantor方法估算的[34];在成对序列比较中消除了对准间隙或缺失数据的位置。对于11个标记中的每一个,这些标记不支持基于Mega结果的红磨的思工,Phylip Ver产生共识树。3.69使用SEQBoot通过Bootstrap重新采样产生100个数据集,DNAdist使用Jukes-Cantor方法来估计遗传距离,邻居生产UnoRoted NJ树和由大多数规则共识树方法计算共识树[67].SplitsTree4版本。4.12.3 [68]用于创建来自DNA序列或NJ树的分裂网络[69].杂交分裂网络的创建使用标记U146437的共识树作为控制,高度支持红果分支(99%)加上标记的共识树正在测试一个渗入,与平衡的类群集(每个树中的相同类群)。中立性测试[49标记C2_At1g73180在DnaSP v. 5.10中检测[70以标记物TG11为对照美国秘密作为外群体。
缩写
- 农业研究所:
-
农业研究服务
- COSII:
-
保守正交集2
- EMBL的:
-
欧洲分子生物学实验室
- ENA:
-
欧洲核苷酸存档
- 美国东部时间:
-
表达序列标签
- NJ:
-
邻居加入
- 通用汽车:
-
转基因
- PGRU:
-
植物遗传资源小组
- 放:
-
PlantGDB组装独特的替代
- QTL:
-
数量性状位点
- RFLP:
-
限制性片段长度多态性
- SGN:
-
索尔基因组学网络
- 如果:
-
自交不亲
- SNP:
-
单核苷酸多态性
- 你:
-
Unigene.
- USDA:
-
美国农业部。
参考文献
- 1.
Evans PD, Mekel-Bobrov N, Vallender EJ, Hudson RR, Lahn BT:证据表明大脑大小基因的适应性等位基因从一个古老的人属渗入到智人。美国国立科学院。中国科学(d辑):地球科学(英文版)。10.1073 / pnas.0606966103。
- 2.
Randi E:检测野生物种和驯化物种之间的杂交。生态学报,2008,17(1):285-293。10.1111 / j.1365 - 294 x.2007.03417.x。
- 3.
Tosi AJ, Detwiler KM, Clifford SL: x染色体突触为猴的基因渗入提供了一种非侵入性测试。中国生物医学工程学报,2006,27(5):531 - 534。10.1007 / s10592 - 005 - 9072 - y。
- 4.
利用野生近缘种进行作物改良:近20年来的研究进展。acta physica sinica, 2007, 36(1): 1- 11。10.1007 / s10681 - 007 - 9363 - 0。
- 5.
渴望C:麝香葡萄繁殖。j hered。1917,8(9):409-424。
- 6.
褐色G:糖基团内一些物种杂交种的细胞学调查。genetica。1923,5(2):97-148。10.1007 / BF01489607。
- 7.
Prescott-Allen C, Prescott-Allen R:第一资源:北美经济中的野生物种。纽黑文:耶鲁大学;1986.
- 8.
Baldo AM, Francis DM, Caramante M, Robertson LD, Labate JA: AlleleCoder:用于编码PCA分析共显性多态性数据的PERL脚本。植物遗传资源研究。2011,10:1-3。
- 9。
中国地理科学(英文版):多样性与群体结构。作物科学,2011,51(3):1068-1079。10.2135 / cropsci2010.05.0305。
- 10。
加德纳M:印第安纳植物疾病,1920年。在印第安纳州科学的诉讼程序中,第30-31卷。印第安纳州的Breeze F. Fort Wayne编辑:韦恩印刷公司堡;1920:187-208。
- 11.
Boswell VR, Pearson OH, Work P, Brown HD, MacGillivray JH, Seaton HL,Starr GE, Bayles JJ, Friend WH, Hawthorn LR等:美国主要番茄品种的类型描述。In: Misc Pub No 160。华盛顿特区:美国农业部;1933:176 - 206。
- 12.
portte WS, Walker HB:泛美番茄,一个新的红色品种高度抗枯萎病。华盛顿特区:美国农业部Circ 611;1941.
- 13.
陈志强,陈志强:近缘野生植物在番茄改良中的应用。Acta photonica sinica, 1995, 412: 21-38。
- 14.
Zamir D:通过异国情调的遗传文库改善植物育种。NAT Rev Genet。2001,2(12):983-989。10.1038 / 35103589。
- 15.
Zamir D:植物育种家回归自然。中国生物医学工程学报。2008,40(3):269-270。10.1038 / ng0308 - 269。
- 16.
番茄基因组定位与分子育种。acta botanica sinica(云南植物学报). 2007,29 (4):497 - 503
- 17.
Haanstra JPW,LaugéR,梅杰-Dekens酒店女,Bonnema G,德威特PJGM,Lindhout,P:对CF-ECP2基因与,但不是,染色体的短臂的CF-4 / CF-9集群的一部分1番茄。Mol Gen Genet。1999年,262(4/5):839-845。
- 18.
基于品种间RFLPs的番茄抗病基因定位策略:1号染色体上抗枝孢杆菌Cf9。中国科学(d辑):地球科学(英文版)。
- 19。
Young ND, Zamir D, Ganal MW, Tanksley SD:利用等基因系和同步探针技术鉴定番茄Tm-2a基因紧密连接的DNA标记。中国生物医学工程学报。
- 20.
基于标记辅助背景选择技术的小麦基因快速定向导入研究。公共科学图书馆。2009,4 (6):e5752-10.1371/journal. pon.0005752。
- 21.
Klein RR, Mullet JE, Jordan DR, Miller FR, Rooney WL, Menz MA, Franks CD, Klein PE:高分辨率基因分型揭示的热带高粱转化和近交系发育对基因组多样性的影响。作物科学,2008,48(4):457 - 461。
- 22.
杂交、渐渗和连锁进化。植物生态学报。2000,42(4):497 - 503。10.1023 /: 1006340407546。
- 23.
瑞克厘米:自然杂交在西南西部耕种番茄衍生中的作用。econ bot。1958,12:346-367。10.1007 / BF02860023。
- 24.
Labate JA,多山AM:番茄SNP发现的EST挖掘和重新排序。mol品种。2005年,16:343-349。10.1007 / s11032-005-1911-5。
- 25.
Labate JA, Robertson LD, Baldo AM:多位点序列数据揭示了驯化番茄(Solanum lycopersicum L.)广泛偏离平衡。遗传。2009,103:257-267。10.1038 / hdy.2009.58。
- 26.
罗伯逊LD,Labate Ja:番茄(Lycopersiconescentum Mill的遗传资源。)和野生亲戚。在Solanaceouscorops,番茄,体积2.由Razdan Mk,Mattoo Ak编辑的遗传改进。恩菲尔德,NH:科学出版商;2007年:25-75。
- 27.
Wu F, Mueller LA, Crouzillat D, Petiard V, Tanksley SD:结合生物信息学和系统遗传学鉴定大套单拷贝同源基因(COSII)用于比较、进化和系统研究:一个在Euasterid植物分支的测试案例。中国生物医学工程学报。2006,34(3):427 - 434。10.1534 / genetics.106.062455。
- 28.
Labate JA、Robertson LD、Wu F、Tanksley SD、Baldo AM: EST、COSII和任意基因标记对栽培番茄(Solanum lycopersicum L.)核苷酸多样性的估计相似。应用电子学报。2009,18:1005-1014。10.1007 / s00122 - 008 - 0957 - 2。
- 29.
Fulton TM,Van der Hoeven R,Eannetta NT,坦克利S:高等植物中对比较基因组学的保守地基因组标记的识别,分析和利用。植物细胞。2002,14(7):1457-1467。10.1105 / TPC.010479。
- 30.
Jiménez-Gómez JM, Maloof JN:三种番茄物种的序列多样性:snp、标记和分子进化。acta botanica sinica(云南植物学报),2009,29(1):1- 5。
- 31。
白泽K,Asamizu E,福冈H,大山A,佐藤S,中村Y,Tabata的S,Sasamoto S,和田T,岸田Y,等人:SSR和在番茄内含子多态性标记的种间连锁图谱。Al Appl Genet。2010年,121(4):731-739。10.1007 / s00122-010-1344-3。
- 32。
利用SSR标记绘制龙葵遗传多样性和群体结构,开发核心种质资源。植物遗传资源:鉴定与利用。2012
- 33。
关键词:番茄,细菌斑点病,番茄假单胞菌,抗性Acta长的矮(ish)。1980年,100:379 - 382。
- 34。
Jukes TH, Cantor CR:蛋白质分子的进化。《哺乳动物蛋白质代谢》,Jukes TH, Cantor CR. Munro H.编辑,纽约:学术杂志;1969:21-132。
- 35。
Nesbitt Tc,Tanksley SD:Lycopersicon属的比较测序:对栽培西红柿驯化中果实大小演变的影响。遗传学。2002,162(1):365-379。
- 36.
番茄基因组联盟:番茄基因组序列提供了深入了解肉质水果的进化。自然科学进展。2012,32(4):457 - 461。10.1038 / nature11119。
- 37.
soltis ps,soltis de:在系统发生重建中施用自血针。stat sci。2003,18:256-267。10.1214 / ss / 1063994980。
- 38.
周云飞,Abbott RJ,蒋志勇,Du FK, Milne RI,刘建强:中国东南部两种松林重叠分布的基因流和物种划分。地球科学进展。2010,64(8):2342-2352。
- 39.
罗德里格斯楼吴楼ANE C,坦克斯利S,斯普纳DM:做土豆和西红柿有一个单一的进化历史,以及基因组支持多大比例这段历史?BMC EVOL BIOL。2009年,9(1):191-10.1186 / 1471-2148-9-191。
- 40.
基于AFLP和两个核基因序列的茄属植物番茄切片的分类和系统发育关系。植物遗传资源学报,2009,56(5):663-678。10.1007 / s10722 - 008 - 9392 - 0。
- 41.
朱强,郑旭,罗静,葛胜:水稻及其野生近缘种核苷酸变异的多位点分析:水稻驯化过程中的瓶颈。中国生物化学学报,2007,24(3):875-888。
- 42.
白伊,Lindhout P:驯鹿和繁殖西红柿:我们获得了什么,未来我们可以获得什么?Ann Bot。2007,100(5):1085-1094。10.1093 / AOB / MCM150。
- 43.
cause M, Caranta C, Saliba-Colombani V, Moretti A, Damidaux R, Rousselle P:通过分子标记提高番茄遗传资源。农业科学。2000,9:197-210。
- 44.
沙蚕RC,Radwanski ER,雅恩L:茄科内抗病性的比较遗传学。遗传学。2000,155:873-887。
- 45.
Riaño-Pachón D, Nagel A, Neigenfind J, Wagner R, Basekow R, Weber E, Mueller-Roeber B, Diehl S, Kersten B: GabiPD: GABI初级数据库-植物综合“组学”数据库。核酸学报2009,37(数据库版):D954-D959。
- 46.
马志强,陈志强,陈志强:1987番茄(Lycopersicon esculentum)的连锁图谱。TGC报告。1987,37:5-34。
- 47.
黄志强,王志强,王志强:番茄Tm-2a区高分辨率遗传图谱的构建及YAC-contigs的构建。应用电子学报。1996,93(1-2):228-233。
- 48.
Van der Biezen EA,Glagotskaya T,Outduin B,John H,Nijkamp J,Hile J:抵抗alteraria alternata f的继承和遗传映射。sp。Lycopersici在Lycopersicon Pennellii。Mol Gen Genet。1995,247:453-461。10.1007 / bf00293147。
- 49.
Hudson RR, Kreitman M, Aguadé M:基于核苷酸数据的中性分子进化测试。中国生物医学工程学报。
- 50。
野生番茄物种的空间遗传学揭示了安第斯地理在人口历史中的作用。植物学报。2011,98(1):88-98。10.3732 / ajb.1000272。
- 51。
高辉,威廉姆森S,布斯塔曼特CD:人口结构的联合推理和多位基因型数据近亲繁殖率的MCMC方法。遗传学。2007年,176:1635年至1651年。10.1534 / genetics.107.072371。
- 52。
Berkman PJ, Lai K, Lorenc MT, Edwards D:下一代测序技术在小麦作物改良中的应用。中国生物医学工程学报。2012,31(2):353 - 356。10.3732 / ajb.1100309。
- 53。
Islam FMA, Beebe S, Munoz M, Tohme J, Redden RJ, Basford KE:利用分子标记评价安第斯基因库中普通菜豆数量属性的渗入效应。应用计算机学报,2004,108(2):243-252。10.1007 / s00122 - 003 - 1437 - 3。
- 54。
汉森PM,杨R,吴俊,陈家,莱斯玛D,TSOU SCS,Lee TC:番茄抗氧化活性和抗氧化剂的变化。J AM Soc Hortic SCI。2004,129(5):704-711。
- 55。
关键词:番茄,植物分类,番茄属,外类群,番茄属acta photonica sinica, 2008, 40(4): 457 - 462。
- 56。
霍尔TJ:番茄TM-2位轨的抗性到番茄马赛克病毒。Euphytica。1980,29(1):189-197。10.1007 / bf00037266。
- 57。
坦克斯利SD,Bernachi D,Beck-Bunn T,Emmatty D,Eshed Y,Inai S,Lopez J,Petiard V,Sayama H,Uhlig J等人:在一对加工番茄系上的产量和质量评估几乎是雌性的TM-2A基因用于抵抗烟草叶片病毒。Euphytica。1998,99:77-83。10.1023 /答:1018320232663。
- 58。
Ewing B, Green P:使用phred自动测序器追踪的基础呼叫。2错误概率。基因组学杂志。1998,8:186-194。
- 59。
Ewing B, Hillier L, Wendl MC, Green P:使用phred自动音序器追踪的基础呼叫。即精度评估。基因组学报,1998,8:175-185。
- 60.
Staden R: Staden序列分析包。生物技术学报,1996,5(3):233-241。10.1007 / BF02900361。
- 61.
大厅TA:BioEdit:适用于Windows 95/98 / NT的用户友好的生物序列对齐编辑器和分析程序。Nucl酸Symp Ser。1999年,41:95-98。
- 62.
Schlueter SD,Dong Q,Brendel V:GeneseQer @ PlantGDB:基因结构预测植物基因组。核酸RES。2003,31(13):3597-3600。10.1093 / nar / gkg533。
- 63.
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gap BLAST和psp -BLAST:新一代蛋白质数据库搜索程序。核酸学报1997,25:3389-3402。10.1093 / nar / 25.17.3389。
- 64.
Bombarely A, Menda N, Tecle IY, Buels RM, Strickler S, Fischer-York T, Pujar A, Leto J, Gosselin J, Mueller LA: Sol Genomics Network (solgenomics.net): using Perl种植番茄。核酸学报2011,39(数据库版):D1149-D1155。
- 65.
斋藤N,NEI玛:邻接法:用于重建系统发生树的新方法。mol Biol Evol。1987年,4:406-425。
- 66.
Tamura K, Dudley J, Nei M, Kumar S: MEGA4:分子进化遗传学分析(MEGA)软件版本4.0。生物科学进展。2007,24:1596-1599。10.1093 / molbev / msm092。
- 67.
Felsenstein J: PHYLIP—系统发育推断包(3.2版)。演化论。1989,5:164-166。
- 68.
Huson DH,科比d:在进化研究演化网络中的应用。mol Biol Evol。2006年,23:254-267。
- 69.
陈志强,陈志强,陈志强:基于基因树的网状网络重建。《计算生物学研究》,《计算机科学讲义》,第3500卷。Miyano JM S, Kasif S,Istrail S, Pevzner P, Waterman M. Berlin:施普林格;2005:233 - 249。
- 70.
Librado P, Rozas J: DnaSP v5: DNA多态性数据综合分析软件。生物信息学。2009,25:1451-1452。10.1093 /生物信息学/ btp187。
致谢
我们感谢S. Sheffer和P. Kisly为优秀的技术支持和E. Cobb促进讨论。这项工作得到了1910-21000-019-00D的CRIN项目资助。USDA是一个平等的机会提供者和雇主。
在本出版物中使用贸易、商号或公司名称是为了提供信息和方便读者。这种使用不构成美国农业部或农业研究服务局对任何产品或服务的官方认可或批准,而排除其他可能合适的产品或服务。
作者信息
从属关系
通讯作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
日本航空和LDR构思并设计了这项研究。日航获取、分析和解释了数据,并起草了手稿。LDR最终批准了将要出版的版本。两位作者都阅读并批准了最终的手稿。
电子补充材料
表S1。
附加文件1:47个基于序列的标记的虚拟映射。从WUR 2.40版本的番茄全基因组猎枪染色体中获得47个标记,截至2012年1月10日。表S2。47个标记的分类群中的进化关系。在野生和栽培的番茄加入中测序的47个番茄标记的引导支持和进化分歧。以粗体标记名称先前被假设为携带血栓增量。(XLS 40 KB)
12870_2012_1075_moesm2_esm.pdf.
附加文件2:番茄基因型的聚类和网络分析。47个标记的邻居连接树和9个标记从野生和栽培番茄取样的杂交网络。(PDF 2 MB)
作者为图像提交的原始文件
权利和权限
本文由BioMed Central Ltd授权发表。这是一篇基于知识共享署名许可协议(http://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
番茄隐性基因渗入的证据(Solanum lycopersicum以野生番茄种等位基因为基础。BMC植物杂志12,133(2012)。https://doi.org/10.1186/1471-2229-12-133
已收到:
接受:
发表:
关键字
- 神秘的渐渗现象
- 链接拖
- 繁殖
- DNA序列
- 茄属植物物种

注释
查看存档评论(2)