摘要
背景
植物细胞壁(PCW)合成相关基因的鉴定是一个非常重要和具有挑战性的问题。尽管已经投入了大量的精力来研究这个问题,但绝大多数与PCW相关的基因仍然未知。
结果
在这里,我们提出了一个计算机研究的重点鉴定的新PCW基因拟南芥基于在351条条件下收集的转录组数据的共表达分析,使用双聚类技术。我们的分析在一些实验条件下鉴定了217个高度共同表达的基因簇(模块),每种实验条件含有至少一种作为PCW附带的基因,根据PURDUE细胞壁基因家族数据库相关。这些共表达模块覆盖349个已知/带注释的PCW基因和2,438名新候选者。对于每个候选基因,我们注释了所涉及的特定PCW合成阶段,并预测详细功能。此外,对于每个模块中的共同表达基因,我们预测并分析了它们的独联体启动子中的调控基序使用我们的基序发现管道,提供了强有力的证据,表明每个共表达模块中的基因是转录共调控的。从所有共表达模块中,我们推断出108个模块使用三种互补的方法与四种主要的PCW合成成分相关。
结论
我们相信我们的方法和数据将有助于进一步鉴定和鉴定PCW基因。所有预测的PCW基因、共表达模块、基序及其注释都可以在一个基于网络的数据库中找到:http://csbl.bmb.uga.edu/publications/materials/shanwang/cwrpdb/index.html..
背景
植物细胞壁(PCW)主要由多糖和木质素组成,形成植物生物质的主要成分。了解哪些基因参与PCW的形成和重塑是非常重要的,因为它们在植物生长过程中发挥了许多关键作用,包括细胞分化,细胞间粘附和通信,水运动控制,防御害虫和病原体的侵袭[1- - - - - -4],更不用说它是纤维素生物燃料研究的焦点。据估计,参与PCW合成、重塑和翻转的基因可能占到全部26500个蛋白编码基因的15%左右拟南芥基因组(4,5,即约4000个基因。到目前为止,只有1000名左右拟南芥根据普渡细胞壁基因家族数据库(以下简称普渡数据库),基因已被描述或预测为PCW相关基因[6].因此,绝大多数PCW相关基因拟南芥基因还有待鉴定。
PCW相关基因的实验阐明主要是通过前进遗传筛查进行的[7,8,这既费时又昂贵。通过快速积累基因组规模的基因表达数据,可以通过共表达分析计算预测PCW相关基因。其基本观点是,被认为在多种条件下共同表达的基因往往是功能相关的[9- - - - - -11];因此,与已知PCW基因共同表达的基因也可能与PCW相关。利用这种或类似的观点,已经开展了许多关于PCW相关基因推断的研究。例如,棕色et al。和佩尔森et al。发表首两项通过微阵列数据分析预测PCW相关新基因的研究[12,13],其中纤维素合成(CESA)基因,CESA4,CESA7和CESA8用作“种子”以鉴定具有类似的表达模式的额外基因.两项研究中预测与PCW相关的基因中有很大比例后来被实验证实确实参与了PCW生物合成[14- - - - - -16[这表明了共表达分析在识别潜在的PCW基因时,为进一步的实验验证提供良好的候选者。
本文对PCW相关新基因的预测进行了研究拟南芥基于在351种条件下收集的已发表的基因表达数据的基因组规模[17].与以往的类似研究相比,我们的研究的一个独特之处在于,我们的目标是在多个但不一定是所有条件下,找到与已知PCW相关基因共同表达的基因。与已发表的研究相比,这使得我们的策略在检测PCW相关基因方面更加敏感和特异性[12,13].但这也提出了一个非常具有挑战性的技术问题:如何确定351个条件中的哪些子集应该被考虑?很明显,把这两个全部做完是不现实的351至少有一定大小的子集,以寻找这样的共同表达基因。
为了克服这个问题,我们应用了一种新的广义聚类技术,称为双聚类[18- - - - - -20.[搜索在351条件的一些(待鉴定)下共同表达的基因组。我们专门使用QUBIC,这是一个用于解决这种类型的广义聚类问题的双聚类算法,这是我们最近开发的[21.].
我们已经实现了基于QUBIC的计算管线,以使用已知/带注释的PCW相关基因(下文所述已知的PCW基因)作为种子以产生共表达基因模块的基因组拟南芥.然后通过识别保守的识别来计算预测的共表达基因模块以转录共同调节独联体同一模块中基因启动子的调节基序。使用这种方法,我们鉴定了2,438个候选基因,其在具有高统计学意义的某些条件下用349个已知的PCW基因表示。候选基因上的功能分析显示了这些基因在PCW合成和重塑中的更详细的功能作用。我们已经进行了含有与四个主要PCW合成组分相关的基因的共表达模块的详细功能分析,其可能编码具有类似功能的生物途径,而是在不同的条件下表达。我们认为,我们的总体分析程序将有助于基因表达数据分析,以阐明植物中的其他生物途径。
结果与讨论
PCW共表达基因推断的计算管道
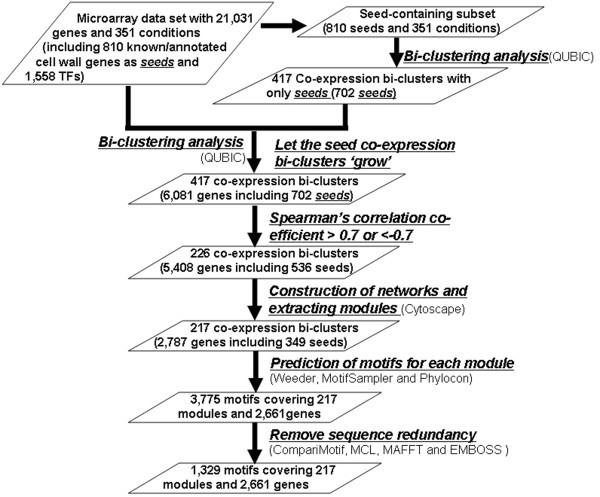
为了鉴定与已知的PCW基因共同表达的基因,我们开发了一个计算管道(图 1).该系统包括以下步骤:(1)利用双聚类程序QUBIC识别已知PCW基因之间的共表达基因;(2)扩展双簇,以包括在相同条件下未知的与PCW相关的其他基因;(3)基于扩展的双聚类,构建包含已知PCW基因和新招募基因的共表达基因网络;(4)提取子网络,命名为co-expression模块,在每个网络;(5)预测、整合和注释各模块共表达基因启动子区域的保守基序。
PCW基因共表达模块
在810个已知的PCW基因中,共鉴定出217个共表达模块,其中包含349个已知的PCW基因(附加文件)1:表S1)。这些基因覆盖了普渡数据库中与PCW合成相关的6个关键阶段中的5个,即底物生成;多糖合成酶和糖基转移酶;组装、架构和增长;分化和次生壁形成;和信号和响应没有任何基因参与分泌和瞄准这可能是因为目前已知只有少数基因参与了这一阶段。另一个原因可能是它与其他阶段的低基因表达相关性,因为它的机制是与细胞骨架动态耦合的[22.].
我们评估了含有已知的负责二次壁纤维素的CESA基因的预测模块的质量,即CESA4, CESA7和CESA8,这些已经被广泛研究和注释[12,13,23.].我们使用评估结果在这些基因上作为217个预测模块的整体质量的指标,因为所有这些模块的质量评估由于大多数人缺乏地面真理信息,这一点在这一点上不可以。
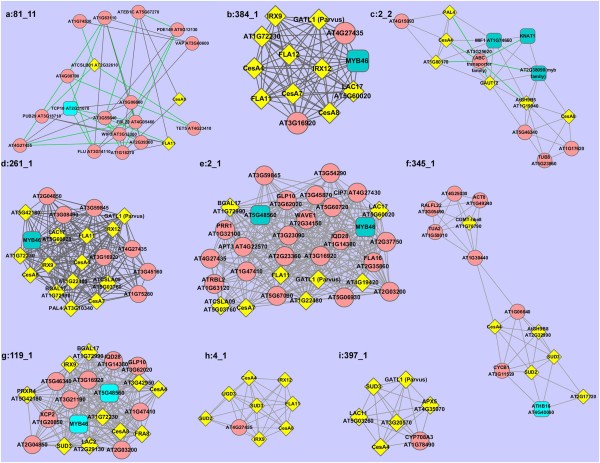
我们指出,9个模块各自含有三个CESA基因中的至少一种(图 2).这些模块中的每一个还包含先前报告的许多基因或预测与CESA基因共同表达[12,13,17,24.],如GT8家族基因gatt1 /Parvus(在模块中261_1,384_1,4_1,397_1)和Gaut12 / IRX8(在模块中2 _2), GT47家族基因FRA8(在模块中119_1)、GT43家族基因IRX9(在模块中119_1, 261_1, 384_1, 4_1)和木质素合成相关基因IRX12(在模块中261 _1 384 _1 4 _1)[13].此外,MYB46的转录因子(TFs)在模块中2_1, 119_1, 261_1, 384_1),称为二级细胞壁合成的总开关[25.,最近被报道与CESA基因共同表达[23.].
9个共表达模块包含CESA4、CESA7和CESA8的CESA基因。每个方块代表一个TF;每颗黄色的钻石代表一个已知的PCW基因,一个红色的圆圈代表一个新的PCW基因。在任何可能的情况下,都使用基因名称而不是TAIR的AT编号。CESA4: AT5G44030、CESA7: AT5G17420、CESA8: AT4G18780、IRX9: AT2G37090、GAUT12/IRX8: AT5G54690、IRX12: AT2G38080、FRA8: AT2G28110、gat1 /Parvus: AT1G19300、SUD2: AT5G59290、SUD3: AT2G28760、PAL4: AT3G10340、UGD3: AT5G15490、COMT-like8: AT1G76790。
PCW候选基因的功能分布
我们的双聚类分析预测了2,438个候选PCW基因,这些基因在217个模块中与349个已知PCW基因共同表达1:表S2和S3)。其中190个模块(88%)包含不超过40个基因2:图S1)。217中的74个模块含有从一个PCW合成阶段的种子基因。其中33种超过10%的基因是种子基因。预计这33个模块中总共201个新型PCW基因将参与特定的合成阶段(附加文件1:表S4)。
为了评估与每个模块相关的实验条件,我们提取与每个模块中的共表达条件相关联的组织/器官信息(附加文件1:表S2, S3)。这些模块涵盖了351种疾病中的317种,与62种不同的组织/器官有关。我们进行了植物本体(PO)解剖富集分析[26.),并保留PO富集量最高的5个组织/器官(附加文件1:表S2)。所有模块的种子的PO解剖学富集结果和所有模块中的总基因分别涉及26和20种不同的组织/器官。对于覆盖每个模块中最多基因的组织/器官,145个含有超过90%的总基因在其目前的模块中。这些信息可能有助于选择合适的实验条件来研究同一模块的基因之间的共表达关系。
为了获得2438个候选基因的更详细的功能,使用了几个软件工具(附加文件1表S5): 181个基因编码CAZy蛋白[27.];正如GolgiP所预测的那样,有269个基因编码以高尔基体功能为目标的酶[28.];根据TransportDB, 161个基因编码转运体[29.];657个基因编码至少有一个跨膜结构域的蛋白质,基于TMHMM [30.].此外,在217个模块中的102个中共发现144个转录因子,属于45个蛋白家族(表)1),其中大部分可能是相应模块的主要转录调控因子(附加文件1:表S6)。许多这些转录调节因子已经被实验证实可以调节次级细胞壁的合成或生物量的形成[25.,31.- - - - - -33.],例如MYB、NAC和WRKY家族的成员。
识别和功能诠释独联体监管图案
对于每个共表达模块,我们使用CGMD管道,通过检查这些基因的启动子区域是否共享作为潜在调控元件的保守序列基序,检测了模块中的基因是否可以转录共调控(见方法)。总的来说,预测了1329个非冗余motif实例(附加文件1:表S7),涵盖2,661个基因的启动子(附加文件1表S8),代表1329个高度保守基序组(见方法)。在217个模块中,有197个(91%)每个模块中至少有80%的基因共享一个保守motif(附加文件)1:表S2和附加文件2图S2a),提供了一个强有力的证据,证明同一模块中的大多数基因是转录共调控的。对于每个模块中的所有主题,我们还使用BOBRO(附加文件1:表S2) [34.].
与已知序列比较独联体PLACE和AGRIS数据库中的调控基序显示,1,329个基序中有769个(58%)与1,009个注释基序中的622个(61%)匹配良好,表明我们的预测质量很高(附加文件)1:表S9)。在1329个主题中,20个是回文(附加文件)1:表S7)。对于具有注释PCW阶段信息的201种新基因,它们在同一阶段中与已知的PCW基因共享273个保守的主题(附加文件1:表S4)。所有这些都证明了我们预测的共表达基因模块的高质量。
评估预测特异性,检查一个零假设已知的主题在AGRIS和地点的数量匹配我们的预测主题本质上是相同的,这种图案的数量匹配预测主题基于任意选定的基因从整个组拟南芥基因组使用Chi-Square测试(参见附加文件1:表S10为详细)[35.].该测试用p值,2.8e-05拒绝了零假设,表明我们预测的主题的高统计显着性。
我们还检查了1329个预测基序是否存在于其相应的同源基因的启动子序列中杨树,使用fuzznucEMBOSS软件包的程序[36.].我们鉴定了1,489对之间的正交基因拟南芥和杨树(附加文件1:表S5),占2,787个基因的53%(已知349个候选PCW基因)。我们扫描了1,489个基因的启动子序列杨树使用1,329个预测的主题。我们的搜索发现,1,329个图案中的374个(29%)在1,234对的原始基因中排除在1,234对(附加档案1表S7),其中已知PCW基因149个,候选基因1085个。因此,我们得出结论,这374个基序更可能是功能基序,而相应的1,085个基序拟南芥基因可能代表PCW基因最可靠的预测。
预测基序启动子区域的位置偏好和丰度
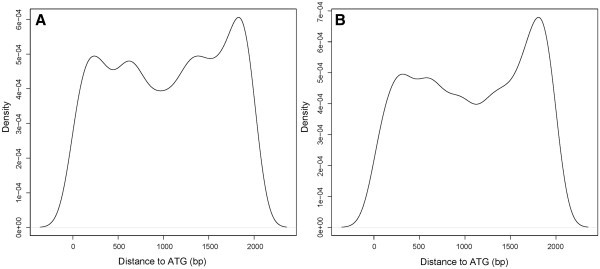
在1329个保守motif中,预计172个为MYB相关转录因子的结合位点,23个为木质素生物合成相关转录因子的结合位点(补充文件)1:表S7)。众所周知,不同的独联体监管主题在启动子的位置可能具有不同的偏好[37.- - - - - -40].这里我们使用AC元件相关基序作为案例研究,已知存在于大多数木质素生物合成基因的启动子中[32.,41].我们发现在我们识别的motif中有11个和10个分别对应AC-I和AC-II元素。对于每一个木质素合成基因,我们分别计算了翻译起始位点与AC元件之间的距离;然后画出距离的分布。除了AC-II元素相关基模在离ATG 1750至2000 bp之间的百分比高于AC-I元素相关基模之外,这两种分布基本相似(图)3.).这可能表明两个AC元素在启动子区域中的位置之间的潜在差异。
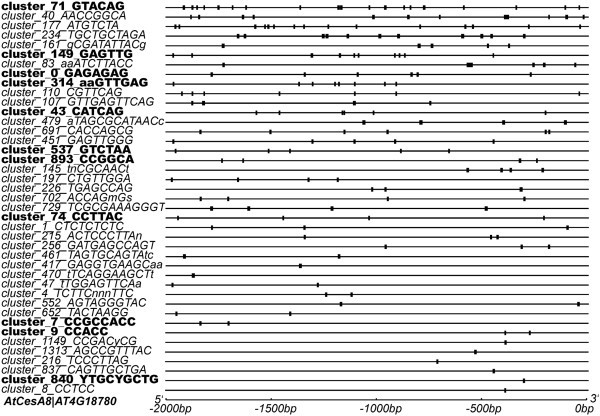
我们还注意到,一个基因的启动子区域可能有多个独特的基序,一个基序可能在同一个启动子中出现多次(例如非常相似的情况)。我们注意到,大多数注释/预测的PCW基因每个都至少有五个不同的基序(附加文件1:表S8)和图案序列可以在所考虑的基因的启动子区域中最多可包含50份副本(附加文件2:图S2B-C)。一个基因启动子区域的基序的位置分布和丰度可能在基因表达的调节中起重要作用[42- - - - - -45].例如,图 4结果表明,AtCesA8 (AT4G18780)在其上游区域有41个独特的motif,每个motif有1 ~ 24个副本;其中11个(图中加粗字体)4的CesA8同源基因启动子中也发现杨树,表明这些母序在进化过程中被保存下来。具体来说,主题cluster_71_GTACAG拥有最多的副本,并且在两者中都保存了拟南芥和杨树.该motif与PLACE中与干旱、低温或高盐胁迫相关的ABRE3 motif (GCCACGTACA)相匹配(附加文件)1:表S9)。此外,cluster_9_CCACC在CESA8的上游是由MYB TFS结合的AC元素的变体[46].
共表达式模块的功能预测
对于每个识别的共表达模块,我们根据其已知的PCW基因的功能注释推断了整个模块的一般功能。具体地,我们专注于包含与PCW合成有关的四组种子基因的模块,即木质素合成途径,木质素合成途径的基因,核苷酸二磷酸糖(Ndp-糖)合成的基因,以及所选GT系列的基因(GT8,GT31,GT34,GT37,GT43,GT47)[47].结果,我们确定了108个这样的模块:30个模块与纤维素合成有关;28与NDP-糖相互转化相关;38与木质素合成相关;49与GT系列相关(附加文件1:表S11)。在这108个模块中,有56个包含tf,它们可能在调控某些特定PCW组分的合成中发挥作用(附加文件)1:表S2)。
我们还比较了108个模块的功能注释与其预测的功能注释的一致性水平独联体模块的管理主题。具体来说,我们首先在每个模块中选择了三个最可靠的主题(附加文件1表S2),基于所覆盖的基因数量和基因间的保守拟南芥和杨树,比较这些motif的功能注释是否与模块的功能注释一致,即检查每个motif是否属于上述MYB或木质素合成相关motif中的一个(附加文件)1:表S7)。在108个模块中,37个(34%)具有来自两个来源的一致功能注释(附加文件1:表S2)。例如,在木质素合成相关模块中86 _1,最可靠的主题是cluster_11cluster_56., 和cluster_284,分别注释为已知的AC元素和两种MyB结合位点,以前涉及对木质素生物合成的调节[46,48,49].在纤维素合成相关的模块中119_1是三个图案中的一个,cluster_149.,被注释为R2R3型MYB TF的结合位点,已知参与纤维素合成[50,51].此外,在108个模块中,有30个模块有未注释的主题。通过排除这些,两个注释源之间的总体一致性是47%,这表明我们的函数推断通常是可靠的。
在108个模块中,我们确定了PCW合成所必需的“热链接”。这个想法是,与周围不太活跃的相互作用相比,一些高活性的相互作用可能主导整个遗传网络的生化活动[52].在108个模块中,68个模块中有119组“热点链接”;52个这样的“热点链接”组包含上述4个组的98个种子基因1表S12)和293候选基因。这些“热点链接”代表了108个模块中最主要的共表达关系。例如,CESA4, CESA7和CESA8以前曾被报道可形成用于纤维素合成的蛋白质复合物[12,17,23.].
结论
共表达分析已被广泛用于鉴定功能基因。在这项研究中,我们预测了与PCW相关的新候选基因拟南芥在基因组规模。与之前的研究相比,这项研究有几个新奇和优势。首先,我们使用了双聚类技术来分析了多种病症的转录组数据,这代表了传统聚类的替代方法,用于鉴定在一些但不一定是所有提供的条件下的共同表达基因。其次,我们使用与所有已知的PCW基因的共表达关系为种子(而不是少数)以识别新的候选基因,这导致了与以前的研究相比鉴定了更大的新候选者。第三,我们使用了基于网络拓扑的方法来识别每个网络内的高度共同表达的基因模块,这使得我们的预测更可靠。第四,使用三个图案预测工具的组合,我们的主题预测更可靠,这是通过我们的功能预测一致性评估所证明的。最后,通过使用三种互补分析方法,我们在单个基因水平和模块级别的功能预测是信息性的,可靠性。每个分析步骤的统计验证确保了我们计算分析结果的整体质量。我们预计我们在此指出的方法和数据将用于研究基因表达数据分析和PCW合成的其他研究人员。
方法
数据收集和处理
的标准化转录组数据集拟南芥蒂利亚纳从Aragennet下载[17],其中包含在351个非冗余条件下收集的基因组级基因表达数据。原始数据集是Affymetrix Ath1拟南芥TAIR的微阵列数据集(22,810个探针组× 1,428个ATH1微阵列)http://www.Arabidopsis.org).该数据集中的探针组代表21,031拟南芥(a)1,558的基因是DATF数据库的带注释转录因子(数据库拟南芥转录因子)[53]和(b)根据PURDUE数据库的810匹配的双型毕因已知的PCW基因[6]除了四个GT家族43基因。基因组序列拟南芥(版本9),杨树(版本2.0)和大米(version 6.1)和相关注释,包括蛋白编码序列和基因间区域,从TAIR、Phytozome (http://www.phytozome.net/poplar)和RGAP (rice.plantbiology.msu.edu)。基本的数据处理是使用内部Perl脚本完成的;使用R软件包进行统计分析(http://www.r-project.org).
基因表达数据的双聚类分析
为了识别与已知PCW基因共表达的基因,我们采用两步双聚类方法对上述微阵列数据集进行分析,该数据集表示为21031 × 351矩阵,这是QUBIC程序所要求的格式[21.].Qubic程序的关键算法思想基于微阵列数据集的图形表示,将双群集问题转换为图形问题[21.].
从该基质中提取含种子的基质(810×351),其中810是已知的PCW基因的数量,称为种子, 351为实验条件数。在第一步中,我们在含种子的矩阵上运行QUBIC来识别种子基因之间的共表达双簇。在第二步中,我们在大矩阵(21031 × 351)上运行QUBIC,在种子矩阵上生长已识别的双簇,即招募在相同条件下与种子双簇共同表达的额外基因。
大多数微阵列分析程序采用离散数据矩阵来降低计算复杂性。我们还将所有表达值离散到三个级别,-1,0,1中,分别代表下调,缺点和上调。QUBIC在离散化表达式范围内的灵活性,从-k到+ k,对于任何固定的正整数k [21.].我们发现K = 1适合我们的研究。qubic使用参数C[0,1]作为控制双簇内共同表达基因表达模式一致性水平的阈值。找到一个合适的C值,我们进行了模拟研究,这表明C0.7和0.98之间的值应该为我们的双聚类分析提供最佳性能结果;因此,我们使用0.05作为增量的0.05在此范围内的最佳C值进行了基于网格的搜索。特别是我们搜索了两个值(C1C2)组合,提供最佳AUC (曲线下的区域)值为接受者操作特性(ROC)曲线分析[54,55](见附加文件1:表S13, S14和支持信息的详细信息)。
Co表达网络和模块的构建
双簇基因在351个实验条件的一个子集下共同表达。为了评估检测到的共表达双聚类的相似性水平,我们检测了同一双聚类中每对基因的表达模式之间的相关性。具体地说,对于每个双聚类,我们计算了斯皮尔曼相关系数ρ在与双簇相关的条件下,每对基因的表达模式之间。注意,这里我们使用的是实际的表达式值,而不是离散的数据(即- 1,0和1)ρ> 0.7(共表达阳性)或< - 0.7(共表达阴性)视为明显发生.这种截止物已被许多公布的论文使用[11,56,57].如果一个双聚类的基因对都不满足这一截断,那么它就不再需要进一步的考虑。
对于每个通过这个测试的双聚类,我们构造一个co-expression网络使用cytoscape [58]的表达式如下所示:网络中的每个节点代表一个唯一的基因,每条边代表上面两个基因表达模式相似的基因ρ在当前双簇的条件下的阈值。应该注意的是,并非所有基因都同样在网络内表达;并且每个网络通常由多种高表达基因的多种簇组成,而簇间的共同表达关系往往较弱,因此具有稀疏边缘。为了识别网络内的所有高度共同表达基因的集群,我们应用了一种流行的基于图形的聚类算法“分子复数检测”(MCODE)[59,以识别所有(非重叠)高共表达基因簇,每一簇称为aco-expression模块。具体来说,每个模块都是一个连接的子网络,与子网络与其他网络之间的密度相比,子网络内部的边密度要高得多。MCODE中的默认评分参数已经过优化,以很好地适应平均网络,因此我们使用了它们(详细信息请参阅MCODE手册)。注意,并不是网络中的所有基因都被分配到一个共表达模块。是特定的密度水平决定了哪些基因被选择或不被选择。实际上,我们使用这种策略是为了摆脱对共同表达基因的意外预测。在设置密度阈值时,我们故意将其设置得足够高,以便尽可能多地排除此类意外预测,这也可能排除一些真正的共同表达基因。
最后一组共表达式模块是由所有表示上述双簇的网络导出的。由于一些双簇可能有重叠,即一些基因可能在不同条件下与不同的基因集共同表达。因此,最后一组共表达式模块可能有重叠。这些信息使我们能够推断包含重叠基因的共表达模块之间的细胞水平功能关系。
保守主题预测
为了确定在同一个模块中共同表达的基因是否在转录上共同调控,我们检查了它们是否共享保守独联体它们的启动子中的调控元素。为此,我们实现了一个新的管道,共表达基因motif discovery (CGMD),通过整合多种算法的预测结果,识别相关基因启动子序列中的保守序列motif,具体如下。
为了获得共表达模块中每个基因的启动子序列,我们从翻译起始位点提取了一个2000 bps的上游区域;我们没有使用转录起始来达到这个目的,因为目前对转录起始位点的预测往往不是很准确。此外,我们使用一个2000 bps的序列作为核心启动子,因为植物启动子的长度通常是1000 bps,加上一个5 '非翻译区域的长度拟南芥可以达到我们的数据显示的1000个bps(额外的文件2:图S3a)。
对于motif预测,我们使用了以下三个预测程序:WeederTFBS 1.4.2 [60, MotifSampler 3a [61,62]和PhyloCon 3.2 [63].这些项目之所以被选中,是因为它们具有公认的强劲表现,以及项目之间的互补性[64].WeederTFBS允许motif长度为6、8、10或12 bps长,并且每次运行输出15个得分最高的motif;假设待识别的基序出现在所有潜在序列中;每个主题在一个序列中可以出现不止一次。MotifSampler使用先验概率来寻找motif,并将预测motif的默认长度设置为8 bps。PhyloCon需要系统发育信息来预测其基序(其他两个不需要),所以我们需要提供每个相关的同源性拟南芥基因杨树和大米,我们使用了双向最佳命中方法[65]并预测每个术语,这些主题在三种外贸序列中保存。对于其他两个基因组中的启动子序列,我们为每个提取了2,000bps的上游序列杨树每个水稻基因的上游序列为4000 bps。原因是,对于Rice基因组,一个5 '非翻译区域可能长达3000 bp,而对于杨树,它的5 '未翻译区域不超过1,000 bps(附加文件2:图S3b-c)。
我们使用了CompariMotif [66]来整合三个程序的所有预测基序,特别是在共表达模块之间高度相似的预测。具体来说,每对预测的基序的相似度评分是用匹配位置的数量除以两个基序之间最大可对齐位置的长度来计算的。基于这个分数,我们使用MCL v10-201 [67[将所有预测的图案集聚到组中,每个预测的主题具有高于预定阈值的相似性得分(MCL的粒度参数设置为4)。然后,我们使用Mafft v6.603b对齐每个组(或群集)内的图案[68,并从无间隙多序列比对中计算出一致序列缺点EMBOSS v6.2.0的程序[36.],并以这种一致序列作为每个基序组的代表。
为了说明这些基序的功能,我们将上述分析得到的基序与两个植物基序数据库AGRIS [69]及地点[70]通过使用CompariMotif。对于两个数据库中的母题,我们也像上面所做的那样,集成了每个集群中最具代表性的母题。对于每对比较基序,如果它们的相似度评分为> 4,匹配位置的百分比为>80%,则认为它们本质上是相同的基序。
为了评估预测的共识主题的统计学意义,我们已经使用了使用两种不同方法与预测的基序匹配的AGRIS中已知基序的数量进行了比较,这些方法是基于共表达基因和任意选择的基因的组整个基因组拟南芥.具体来说,我们创建了1000个任意基因组,其大小与所有正在考虑的共表达模块的平均大小相同。对于每个这样的基因组,我们使用上述程序预测基序(仅WeederTFBS)。为了保持一致,我们仅为了比较目的,使用WeederTFBS对共同表达的基因进行了motif预测。我们的零假设是,在共同表达基因中,已知基序与预测基序匹配的比例与任意选择的基因相同。采用卡方检验来检验这一假设[71].根据给定数据集上的卡方检验p值,可以拒绝或接受假设。
缩写
- PURDUE数据库:
-
普渡细胞壁基因家族数据库
- 中国极限运动协会:
-
纤维素合成
- 已知的物理化学加工基因:
-
已知/注释的PCW相关基因
- TFs:
-
转录因素
- 博:
-
植物本体
- NDP-sugar:
-
核苷二磷酸糖
- 物理化学加工:
-
植物细胞壁
- DATF数据库:
-
数据库拟南芥转录因子
- AUC:
-
曲线下的面积
- 中华民国:
-
接受者操作特性
- CGMD:
-
共表达基因基序发现
- GT:
-
糖基转移酶
- GATL1:
-
半乳糖酰基转移酶样1
- Parvus:
-
Polygalacturonate 4-alpha-galacturonosyltransferase
- GAUT:
-
半乳糖核糖基转移酶
- IRX:
-
不规则的木耳
- Fra:
-
脆弱的纤维
- ABRE:
-
脱落酸反应元件
- MYB:
-
成髓细胞病病毒癌基因同源物
- AC元素:
-
激活元件。
参考文献
- 1.
Braam J:如果墙壁可以说话。CurrOp植物BIOL。1999,2(6):521-524。10.1016 / s1369-5266(99)00029-1。
- 2。
Jones DA, Takemoto D:植物先天免疫-对一般和特定病原体相关分子的直接和间接识别。中国免疫学杂志。2004,16(1):48-62。10.1016 / j.coi.2003.11.016。
- 3.
糖基转移酶和细胞壁生物合成:新颖的角色和见解。植物生态学报。2004,27(3):285-295。10.1016 / j.pbi.2004.03.006。
- 4.
植物细胞壁多糖组成在抗病中的作用。植物科学进展,2004,9(4):203-209。10.1016 / j.tplants.2004.02.005。
- 5.
Carpita N,Tierney M,Campbell M:植物细胞壁的分子生物学:寻找定义结构,架构和动态的基因。植物mol biol。2001,47(1-2):1-5。
- 6.
Yong W, Link B, O’malley R, Tewari J, Hunter CT, Lu CA, Li X, Bleecker AB, Koch KE, McCann MC, et al .:植物细胞壁生物发生基因组学。植物学报。2005,221(6):747-751。10.1007 / s00425 - 005 - 1563 - z。
- 7.
Jones L,Ennos AR,Turner SR:不规则XYLEM4(IRX4)的克隆和表征:拟南芥的严重木质素缺乏突变体。工厂J. 2001,26(2):205-216。10.1046 / J.1365-313x.2001.01021.x。
- 8.
特纳SR,Somerville Cr:映析拟拟合拟南芥的表型鉴定义纤维素沉积在二次细胞壁中的突变体。植物细胞。1997,9(5):689-701。
- 9.
STUART JM,SEGAL E,Koller D,KIM SK:一种基因 - 用于全球保护遗传模块的基因共存网络。科学(纽约,纽约。2003,302(5643):249-255。10.1126 / Science.1087447。
- 10.
Ihmels J,Levy R,Barkai N:酿酒酵母代谢网络中转录控制原则。NAT BIOTECHNOL。2004,22(1):86-92。10.1038 / NBT918。
- 11.
陈志强,陈志强,陈志强:基于基因共表达网络的植物遗传信息提取方法。植物生理学报。2007,48(3):381-390。10.1093 /卡式肺囊虫肺炎/ pcm013。
- 12.
Persson S,Wei H,Milne J,GP,Somerville CR:通过公共微阵列数据集的回归分析鉴定纤维素合成所需的基因。Proc Natl Acad Sci U S A. 2005,102(24):8633-8638。10.1073 / pnas.0503392102。
- 13。
棕色DM,Zeef La,Ellis J,Goodacre R,Turner SR:使用表达分析和逆向遗传识别拟南芥中参与二次细胞壁形成的新型基因。植物细胞。2005,17(8):2281-2295。10.1105 / TPC.105.031542。
- 14。
Brown DM, Goubet F, Wong VW, Goodacre R, Stephens E, Dupree P, Turner SR: 5个木聚糖合成突变体的比较揭示了木聚糖合成机制的新见解。植物学报,2007,52(6):1154-1168。10.1111 / j.1365 - 313 x.2007.03307.x。
- 15.
周国康,周国康,周国康,周国康。拟南芥不规则木质素8和不规则木质素9的生物合成研究。植物学报,2007,19(2):549-563。10.1105 / tpc.106.049320。
- 16.
Persson S, Caffall KH, Freshour G, Hilley MT, Bauer S, Poindexter P, Hahn MG, Mohnen D, Somerville C:拟南芥不规则xylem8突变体缺乏葡萄糖醛酸氧聚糖和同半乳糖醛酸,这对次生细胞壁的完整性至关重要。植物学报。2007,19(1):237-255。10.1105 / tpc.106.047720。
- 17.
Mutwil M,Ruprecht C,Giorgi FM,Grantmann M,Usadel B,Persson S:拟南芥中与细胞壁相关基因的转录布线。莫尔植物。2009,2(5):1015-1024。10.1093 / MP / SSP055。
- 18.
Hartigan JA:数据矩阵的直接聚类。中华医学杂志。1972,67:123-129。10.1080 / 01621459.1972.10481214。
- 19.
Cheng Y, Church GM:表达数据的双聚。分子生物学智能系统国际会议论文集;ISMB。2000年,8:93 - 103。
- 20。
基因芯片数据的双向聚类分析。中国科学(d辑:地球科学),2000,19(4):531 - 534。10.1073 / pnas.210134797。
- 21.
李国强,马强,唐华,徐勇:一种用于基因表达数据分析的定性双聚类算法。中国生物医学工程学报,2009,37 (15):e101-10.1093/nar/gkp491。
- 22.
植物纤维素的生物合成:从基因到莲座。植物生理学报。2002,43(12):1407-1420。10.1093 /卡式肺囊虫肺炎/ pcf164。
- 23.
Ruprecht C,Mutwil M,Saxe F,Eder M,Nikoloski Z,Persson S:大规模的共同表达方法,用于在植物物种中对次级细胞壁形成分析。植物科学的边疆。2011,2:
- 24.
Wu LF, Hughes TR, Davierwala AP, Robinson MD, Stoughton R, Altschuler SJ:利用重叠转录簇大规模预测酿酒酵母基因功能。中国生物医学工程学报,2002,31(3):255-265。10.1038 / ng906。
- 25。
叶志华:植物生物量生产的调控。植物生态学报,2010,13(3):299-304。
- 26。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT,等:基因本体:生物学统一的工具。基因本体论联盟。中国生物医学工程学报,2000,25(1):25-29。10.1038/75556。
- 27.
Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B:碳水化合物活性酶数据库(CAZy):糖基因组学的专家资源。核酸学报2009,37(数据库版):D233-D238。
- 28.
Chou Wc,Yin Y,Xu Y:Golgip:预测植物中的高尔基居民蛋白。生物信息学。26(19):2464-2465。
- 29.
IT: TransportDB:细胞质膜运输系统和外膜通道的综合数据库资源。核酸学报2007,35(数据库版):D274-D279。
- 30.
Krogh A,Larsson B,Von Heijne G,Sonnhammer El:用隐藏的马拉可夫模型预测跨膜蛋白拓扑:应用于完成基因组。J Mol Biol。2001,305(3):567-580。10.1006 / jmbi.000.4315。
- 31。
赵强,狄克森:木质素生物合成的转录网络:比我们想象的更复杂?植物科学进展,2011,16(4):227-233。10.1016 / j.tplants.2010.12.005。
- 32。
Zhong R,Ye Zh:木质素生物合成的转录调控。植物信号表现。2009,4(11):1028-1034。10.4161 / PSB.4.11.9875。
- 33.
王H,AVCI U,Nakashima J,Hahn Mg,Chen F,Dixon Ra:Wrky转录因子的突变引发了髓次壁的形成,并增加了茎生物质在二坐子植物中。Proc Natl Acad SCI U S A. 2010,107(51):22338-22343。10.1073 / pnas.1016436107。
- 34.
李克,刘湾,马Q,徐义:识别原核生物中的CIS-Scientatory icif的新框架。核酸RES。39(7):E42。
- 35.
Fisher RA, Yates F.E,经作者和出版商许可:《生物、农业和医学研究统计表》第6版。Oliver &博伊德
- 36.
米P,Longden I,Blasby A:浮雕:欧洲分子生物学开放的软件套件。趋势类型。2000,16(6):276-277。10.1016 / s0168-9525(00)02024-2。
- 37。
Hughes JD,Estep PW,Tavazoie S,Church GM:与酿酒酵母中功能相关基因组相关的顺式调节元素的计算鉴定。J Mol Biol。2000,296(5):1205-1214。10.1006 / jmbi.2000.3519。
- 38。
啤酒ma,tavazoie s:从序列预测基因表达。细胞。2004,117(2):185-198。10.1016 / S0092-8674(04)00304-6。
- 39.
协调增强子在果蝇基因组中具有共同的组织特征。中国科学(d辑:地球科学),2004,51(11):3851-3856。10.1073 / pnas.0400611101。
- 40。
Elemento O,Tavazoie S:使用非对准方法的保守监管元素的快速和系统基因组发现。基因组Biol。2005,6(2):R18-10.1186 / GB-2005-6-2-R18。
- 41。
Raes J,Rohde A,Christensen JH,Van de Peer Y,Boerjan W:拟南芥中的紫花形工具箱的基因组。植物理性。2003,133(3):1051-1071。10.1104 / pp.103.026484。
- 42。
发现调控DNA序列基序的实用策略。中国生物医学工程学报,2006,27(4):457 - 461。
- 43。
Remenyi A, Scholer HR, Wilmanns M:基因表达的组合控制。中国生物医学工程学报。2004,11(9):812-815。10.1038 / nsmb820。
- 44。
Bulyk ML:转录因子结合位点位置的计算预测。中国生物医学工程学报。2003,25 (1):201-10.1186/gb- 2005-1-201。
- 45.
Creux NM, Ranik M, Berger DK, Myburg AA:拟南芥、杨树和桉树同源纤维素合成酶启动子的比较分析:被子植物中保守调控元件的证据。植物营养学报,2008,29(3):427 - 434。10.1111 / j.1469-8137.2008.02517.x。
- 46.
关键词:PAL2启动子,转基因烟草,顺式序列,基因表达植物学报,1995,7(6):859-876。10.1046 / j.1365 - 313 x.1995.07060859.x。
- 47.
Cosgrove DJ:植物细胞壁的生长。NAT Rev Mol Cell Biol。2005,6(11):850-861。10.1038 / NRM1746。
- 48.
Patzlaff A, McInnis S, Courtenay A, Surman C, Newman LJ, Smith C, Bevan MW, Mansfield S, Whetten RW, Sederoff RR,等:松树MYB调控木质素的特征。植物学报,2003,36(6):743-754。10.1046 / j.1365 - 313 x.2003.01916.x。
- 49。
Patzlaff A, Newman LJ, Dubos C, Whetten RW, Smith C, McInnis S, Bevan MW, Sederoff RR, Campbell MM:来自松树木质部的一种R2R3-MYB Pt MYB1的特征。植物学报,2003,53(4):597-608。
- 50.
Lesnick ML, Chandler VL:玉米花青素基因a2的激活是由许多花青素启动子中保守的一个元素介导的。植物营养与肥料学报。1998,17(2):437-445。10.1104 / pp.117.2.437。
- 51。
Tuerck JA, Fromm ME:花青素B/C1或酞菁P调控基因转位所需的玉米A1启动子元件。植物学报。1994,6(11):1655-1663。
- 52。
网络生物学:理解细胞的功能组织。自然评论。[j] .环境科学学报,2004,5(2):101-113。10.1038 / nrg1272。
- 53。
郭安华,何凯,刘德华,白淑萍,顾昕,魏丽,罗建军:拟南芥转录因子数据库。生物信息学(英国牛津大学)。2005,21(10): 2568-2569。10.1093 /生物信息学/ bti334。
- 54。
Fawcett T:ROC分析介绍。模式识别出菜。2006,27:861-874。10.1016 / J.Patrec.2005.10.010。
- 55。
Hanley Jaam BJ:接收器操作(ROC)曲面特征下该区域的含义和使用。放射学。1982,143(1):29-36。
- 56。
Usadel B, Obayashi T, Mutwil M, Giorgi FM, Bassel GW, Tanimoto M, Chow A, Steinhauser D, Persson S, Provart NJ:植物生物学共表达工具:假说生成的机会和警告。植物学报。2009,32(12):1633-1651。10.1111 / j.1365-3040.2009.02040.x。
- 57。
Mentzen Wi,Wurtele es:Reginon of Arabidopsis组织。BMC植物BIOL。2008,8:99-10.1186 / 1471-2229-8-99。
- 58.
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T: Cytoscape:一种生物分子相互作用网络集成模型的软件环境。中国生物医学工程学报,2017,32(11):1498 - 1405。10.1101 / gr.1239303。
- 59.
在大的蛋白质相互作用网络中寻找分子复合物的自动化方法。生物信息学。2003,4:2-10.1186/1471-2105-4-2。
- 60.
paesi G, Mereghetti P, Mauri G, Pesole G: Weeder Web:在一组共调控基因序列中发现转录因子结合位点。核酸Res. 2003,32 (Web服务器版):W199-W203。
- 61.
Thijs G, Lescot M, Marchal K, Rombauts S, De Moor B, Rouze P, Moreau Y:高阶背景模型改进了Gibbs抽样对启动子调控元件的检测。生物信息学(英国牛津大学)。[j] .地球科学进展,2001,17(12):1113-1122。10.1093 /生物信息学/ 17.12.1113。
- 62.
thjs G, Marchal K, Lescot M, Rombauts S, De Moor B, Rouze P, Moreau Y:一种检测共表达基因上游区域过度代表基序的Gibbs抽样方法。中国计算机科学(英文版),2002,9(2):447-464。10.1089 / 10665270252935566。
- 63。
Wang T,Stormo Gd:将系统发育数据与共调基因组合以识别调节基序。生物信息学(英国牛津大学)。2003,19(18):2369-2380。10.1093 / Bioinformatics / BTG329。
- 64。
d'haeeeleer p:DNA序列如何序列工作?NAT BIOTECHNOL。2006,24(8):
- 65。
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ: gap BLAST和psp -BLAST:新一代蛋白质数据库搜索程序。中国生物医学工程学报,2017,31(4):531 - 534。10.1093 / nar / 25.17.3389。
- 66。
Davey NE, Edwards RJ, Shields DC: SLiMDisc服务器:蛋白质中短的线性motif发现。核酸学报2007,35 (Web服务器版):W455-W459。
- 67。
Enright AJ,Van Dongen S,Ouzounis Ca:一种高效检测蛋白质家族的高效算法。核酸RES。2002,30(7):1575-1584。10.1093 / nar / 30.7.1575。
- 68。
Katoh K,Misawa K,Kuma K,Miyata T:Mafft:一种基于快速傅里叶变换的快速多序列对齐的新方法。核酸RES。2002,30(14):3059-3066。10.1093 / NAR / GKF436。
- 69。
Palaniswamy SK, James S, Sun H, Lamb RS, Davuluri RV, Grotewold E: AGRIS和AtRegNet。一个将顺式调控元件和转录因子连接到调控网络的平台。植物营养与肥料学报。2006,27(3):429 - 434。10.1104 / pp.105.072280。
- 70。
Higo K, uawa Y, Iwamoto M, Korenaga T:植物顺式调控DNA元件(PLACE)数据库:1999。核酸学报1999,27(1):297-300。10.1093 / nar / 27.1.297。
- 71。
Nikulin MS:正态性的卡方检验。国际维尔纽斯概率论和数理统计会议论文集。1973,2:119-122。
确认
我们承认美国能源部(Grant #de-PS02-06ER64304)和国家科学基金会(Deb-0830024)的支持。生物能源科学中心得到了在科学职业办公室生物和环境研究办公室的支持。我们感谢川周在中国山东大学数学学院,以便计算预测主题的P价值。
作者信息
隶属关系
相应的作者
附加信息
相互竞争的利益
作者声明没有竞争利益。
作者的贡献
SW策划并执行整个项目,分析数据,撰写稿件。YY发起并监督项目,分析数据,修改稿件。QM策划了双聚类分析,并为使用QUBIC和BOBRO程序提供了技术支持。XT为微阵列数据矩阵的离散化提供了技术支持。卫生署提供支持和指导。YX提供支持和指导,启动项目,分析数据,修改稿件。所有作者阅读并批准了最终的手稿。
山王,yanbin yin贡献了这项工作。
电子辅料
12870_2012_1082_moesm2_esm.xls.
附加文件2:支持信息。ROC曲线分析细节及补充图S1, S2, S3。(XLS 3 MB)
作者为图像提交的原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
王帅,尹勇,马强。et al。细胞壁相关基因的基因组尺度鉴定拟南芥基于共表达网络分析。BMC植物杂志12,138(2012)。https://doi.org/10.1186/1471-2229-12-138
已收到:
接受:
发表:
关键字
- 植物细胞壁
- 拟南芥
- Co-expression网络分析
- Bi-clustering
- CIS.监管图案