摘要
背景
R基因是植物和生物营养细菌之间遗传相互作用的关键组成部分,并调节对细菌入侵的抗性。最常见的R蛋白包含一个核苷酸结合位点和一个富亮氨酸重复序列(NBS-LRR)域。大豆基因组中一些NBS-LRR基因也被报道具有抗病功能。本研究发现,NBS-LRR基因的数量与这些基因侧边的抗病数量性状位点(QTL)的数量相关。NBS-LRR基因与抗病QTL共定位。该研究还探讨了NBS-LRR基因最近复制的区域的抗病功能冗余,以及NBS-LRR基因在BLP诱导的大豆转录组中的表达。
结果
在大豆基因组中,共鉴定出319个推测为NBS-LRR基因。各染色体上NBS-LRR基因的数量与NBS-LRR基因2-Mb侧翼区抗病QTL的数量高度相关。另外,新近发现的重复区域含有重复的NBS-LRR基因和抗病QTL,且每侧NBS-LRR基因数量不均匀或均匀。抗病近等基因系(NIL)与易感近等基因系(NIL)接种后NBS-LRR基因表达差异显著黄axonopodispv。甘氨酸支持NBS-LRR基因在大豆基因组中具有抗病功能的猜测。
结论
各染色体2-Mb侧翼区NBS-LRR基因数量与抗病QTL显著相关,且最近重复的多个含有NBS-LRR基因的区域具有正反两方抗病QTL。此外,blp抗性和blp敏感的NIL对细菌感染的反应中,NBS-LRR基因的表达也存在显著差异。这些结果表明,NBS-LRR基因可能有助于大豆抗病。此外,我们提出了NBS-LRR基因复制的模型,并对每个NBS-LRR基因簇应用Ks值。
背景
R基因是植物与生物营养细菌之间基因相互作用的关键组成部分,通常具有调节对细菌入侵的抗性的功能[1].最近的研究提出了“之字形模型”来描述在入侵者和宿主共同进化的背景下植物的抗性[2].植物防御的第一阶段是病原体分子模式触发免疫(PTI),通过植物的免疫系统识别广泛的保守分子模式的病原体,从而赋予非寄主抗性。在第二阶段,效应触发免疫(ETI)通过细菌的III型分泌系统(TTSS)检测注入植物细胞的效应因子。通常,ETI会导致放大的PTI反应,也称为超敏反应(HR)。
在已知的R蛋白类型中,最常见的是含有核苷酸结合位点和富含亮氨酸重复结构域的R蛋白[3.].在拟南芥Col-0基因组中,预测149个蛋白具有NBS-LRR结构域,其中约三分之二包含在一个至少有一个已知R蛋白或R蛋白的Col-0同源的亚群中[4].在大豆中,虽然对具有多种疾病抗性的R蛋白的研究还比较缺乏,但有报道称具有螺旋基序的NBS-LRR基因(CC-NBS-LRR)与该基因共分离Rpg1-b提供抗性菌株的位点两pv。glycinea[5].
值得注意的是,一种与Toll/白介素-1受体同源的NBS-LRR基因(TIR-NBS-LRR)被报道抑制大豆结瘤[6].虽然结瘤是一种共生互作,而不是病原菌与寄主之间的互作,但这可能表明R基因控制着微生物进入大豆植株。因此,定位于整个大豆基因组的NBS-LRR基因可能参与识别病原体的存在并最终赋予抗性。
植物感知细菌入侵被认为是由植物监测系统完成的,该系统识别特定的病原体产生的效应物,并触发植物细胞进行HR [2].转录组分析是研究R基因在抗病中的作用的有力工具。最近,随着GS-FLX 454 Titanium和Illumina-GA等新一代测序仪的出现,在可以获得参考基因组序列的情况下,可以评估特定植物中所有预测基因的表达水平。大豆全基因组草图序列在Phytozome (http://www.phytozome.net/soybean.php),可以检索所有预测基因的大豆转录组数据[7].此前的一项研究提出了大豆叶片脓疱(BLP)之间的近等基因系(NILs)的转录组分析黄axonopodispv。甘氨酸(Xag抗性和敏感品系的基因表达差异Xag可能有助于大豆抗BLP [8].
在这项研究中,我们研究了NBS-LRR基因数与每种染色体上的抗病率QTL数量之间的相关性和共定位,并调查了港口NBS-LRR已知最近重复的地区的抗病性基因的功能冗余基因。此外,我们调查了效果Xag通过接种敏感和抗性NILs进行基因表达的时间过程实验,发现与BLP抗性相关的QTL上的NBS-LRR基因的表达显著增加。
结果
基因组NBS-LRR基因的分布和抗病性QTL
为了确定大豆基因组的NBS-LRR基因,我们利用了Phytozome (http://www.phytozome.net/soybean.php)[7].基于Pfam、Panther、KOG、KEGG等蛋白家族数据库,Phytozome根据Gmax_109_annotation_info.txt对46628个基因进行了注释。为了在大豆基因组中检索到推测的NBS-LRR基因,我们选择了LRR Pfam id为PF00560、PF07723和PF07725的基因,并对其进行进一步细化,筛选出含有NBS结构域(PF00931)的基因。在大豆鉴定的1286个LRR基因中,319个基因被确定为NBS-LRR推测基因。共有314个NBS-LRR基因位于大豆染色体上(见表)1),而另外5个NBS-LRR基因被放置在支架上。利用与已分类的拟南芥NBS-LRR基因的同源性,进一步对NBS-LRR基因的n端结构域进行了分类,包括116个tir -NBS-LRR、20个cc -NBS-LRR和183个其他NBS-LRR(见附加文件)1).
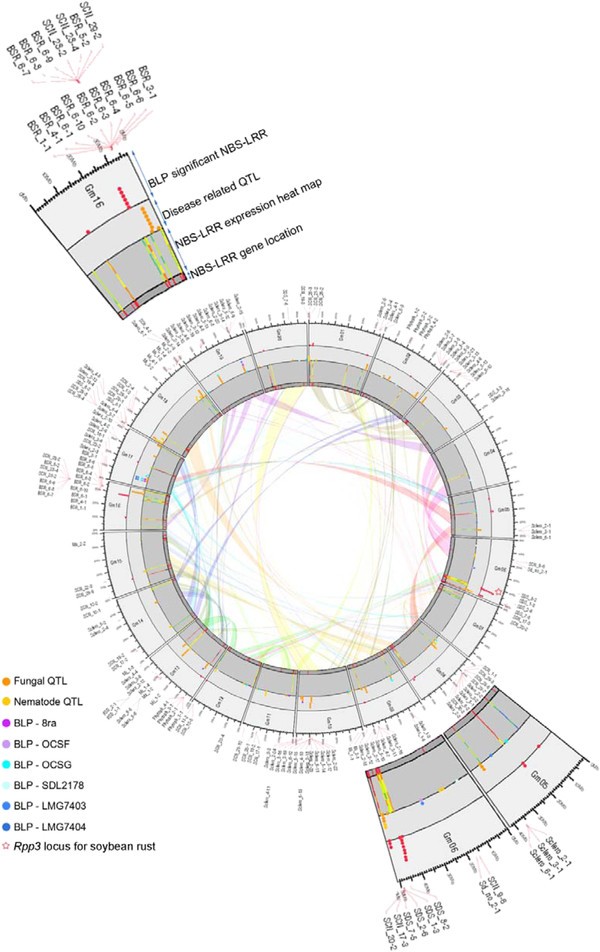
为了研究大豆基因组中NBS-LRR基因的分布,在最内层的圆形基因组图上绘制基因的染色体位置(图1).NBS-LRR基因在染色体(Chr) 3、6、13、15、16和18上呈偏聚性分布。314个NBS-LRR基因中超过一半(178个)位于这6条染色体上,提示NBS-LRR基因簇是重复事件的结果(见表)1).
利用相应标记信息所鉴定的抗病QTL位置也存在高度偏倚,与NBS-LRR基因的位置相似。296个报道的大豆抗病QTL (http://soybase.org/),通过相应的标记信息选择175 QTL。数字1从最内层到第三层描述了抗病QTL的定位。抗病QTL倾向于定位在NBS-LRR基因物理位置侧翼的2mb区域,特别是高度聚集的区域(见附加文件)2).例如,NBS-LRR基因在Chr 6上的高度聚集区包含4个真菌抗性QTL和3个线虫抗性QTL。在Chr 16上聚类了40个NBS-LRR基因,其中14个真菌抗性QTL和5个线虫抗性QTL位于聚类基因附近。以Chr 18为例,有8个抗真菌QTL和4个抗线虫QTL位于包含32个NBS-LRR基因簇的2-Mb侧翼区域(见表)1,图1).
尽管并非所有疾病抵抗QTL都位于NBS-LRR基因附近,但大多数QTL和NBS-LRR基因倾向于共定。大约63%的疾病相关QTL定位在NBS-LRR基因的2 MB侧翼区域内(表1).线性回归分析表明,NBS-LRR基因的数量与NBS-LRR基因侧翼2 mb区域内的抗病QTL数量显著相关R2值为0.520和aP-value小于0.001(表1,请参阅附加文件3.).此外,选择大豆“小叶状”性状的QTL,以了解NBS-LRR基因与不同类型QTL之间的相关性,而不是与疾病相关(R2= 0.073,P = 0.251) (see Additional file4).共有667个QTL归属于大豆性状项,分别为:SOY:0000099, (http://soybase.org)被选择来调查NBS-LRR基因是否有其他假定的影响(见附加文件5).从中筛选出345个QTL,定位于NBS-LRR基因2-Mb侧翼区,多数性状为大豆形态解剖性状(SOY:0001398)和大豆抗逆性性状(SOY:0001400)。这表明一些NBS-LRR基因可以影响许多不同的数量性状或抗病与其他重要农艺性状的互作。
最近含有基因组区域的NBS-LRR重复
为了进一步支持NBS-LRR基因与抗病QTL的功能相关性,我们最近研究了大豆基因组中的重复区域,这些区域可能包含根据其性质执行冗余功能的基因。数字1将最近复制的大豆区域描述为圆形基因组图上的透明彩色带。表格2提供了最近重复区域2 Mb侧翼内的抗病QTL列表,并检索了最近重复区域内的NBS-LRR基因。在10个最近重复的基因组区域中共有91个NBS-LRR基因,这些区域包含重复的抗病QTL。在最近重复的区域中,有4个区域(id: 10176678、8358327、11590712和10856314)具有相同数量的NBS-LRR基因。另外4个重复区域(id分别为10176681、10176680、18934087和18159398)显示只有一个基因存在差异。在这些区域中,NBS-LRR基因在每一侧的拷贝数相似。
在这八个重复的区域中,10176681,1176680,10176678和11590712具有疾病性QTL,具有不同疾病的特异性(表2).例如,最近复制的区域10176681,复制了Chr 1的nss - lrr基因Glyma01g01400和Glyma01g01420,复制了Chr 9的Glyma09g33570、Glyma09g34360和Glyma09g34380。线虫抗性QTL定位在Chr 1上,真菌抗性QTL定位在Chr 9上。这些例子表明,NBS-LRR基因在复制后可能保留了其生物抗性功能,但也可能获得了新的特异性。其他重复区域(8358327、10856314、18934087和18159398)具有抗病QTL。
然而,两个最近复制的区域(22835159和18934088)具有不同数量的NBS-LRR基因,其可以由串联复制在重复区域的一侧上独立发生的串联复制产生。与另一侧相比,这些重复区域的一侧含有较高数量的NBS-LRR基因和另外的抗病率QT1。例如,最近重复的区域18934088在CHR 9和15串联重复的基因(Glyma16G32320至Glyma16G34110)上仅具有一个重复的NBS-LRR基因(Glyma09G29050)在CHR 16上。若干疾病QTL也如下分离:4 QTL对CHR 9的抗性位于CHR 9上,而在CHR 16上发现了13 QTL用于抗真菌的抗棕色茎腐(BSR)和5 QTL抗大豆囊肿线虫(SCN)。
提供细菌抗性的BLP电阻QTLXag也与最近重复的NBS-LRR区域重叠。Chr 8 (ID 8358327)上最近复制的一个区域,两侧各有两个nws - lrr基因Glyma08g12990和Glyma05g29880(表)2).抗BLP和sclo OCS-G分离物的抗病QTL位于复制区域的一侧,抗BLP和sclo LMG7403分离物的抗病QTL位于复制区域的另一侧[9].另一个最近的例子重复区域(ID 10856314),双方存在NBS-LRR基因,包含对OCS-G BLP抗性QTL, SDL2178和SDS 7隔离的一侧重复区域和抗病QTL对BLP 8 ra隔离和Sclero另一边重复区域。
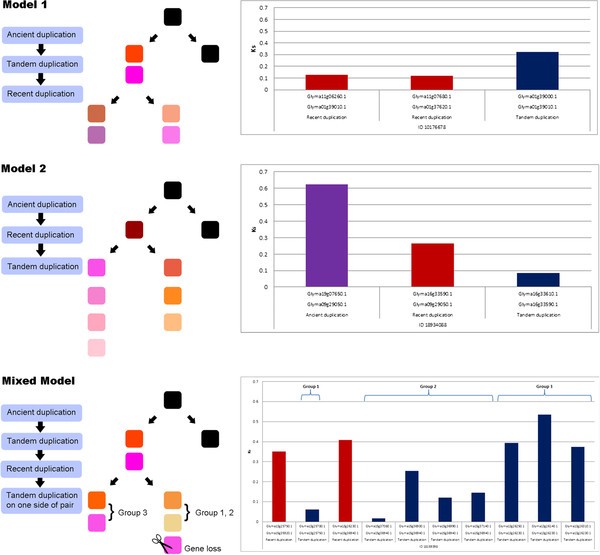
最近重复区域的NBS-LRR基因的同义替换率(Ks)在0.016 ~ 3.722之间。这些值可能反映了每个重复事件的顺序,在此我们提出了解释重复顺序的模型。我们建议用两个模型来解释复制顺序(图2).在第一个模型中,NBS-LRR基因首先被串联复制,然后在一个重要的进化时期受到选择压力,最后被复制到另一个染色体上。符合这一模型的基因在连续复制的基因之间表现出高的k值,而在最近复制区域的平行对数之间表现出低的k值。在第二个模型中,一个NBS-LRR基因首先复制到另一个染色体上,然后独立地串联复制。这将产生一个与第一种模式相反的模式。
解释NBS-LRR基因复制过程的模型。在模型1中,串联复制发生在最近的复制之前。在模型2中,古老的复制事件首先发生,最近的复制事件将一个基因或区域复制到另一条染色体上,串联复制独立发生。在混合模型中,串联复制发生在最近一次复制之前,独立串联复制在最近一次复制之后再次发生。图的右栏显示了最近复制区域之间的k值柱状图。ID 10176678与模型1匹配,其中串联复制发生在最近的复制之前。ID 18934088与模型2相匹配,其中首先发生一个古老的复制,然后是最近的复制,然后是串联复制。ID 18159398与Mixed Model匹配;第13染色体上第3组的串联复制首先发生,最近的重复发生在第1组和第2组的独立串联复制之前,后者发生在第13和15号染色体上。
为了确定这两个模型中的哪一个更有可能,我们通过估计ParaLogs和Tandemly重复的区域之间的KS值来估计最近复制和串联复制事件的顺序(参见附加文件6).ID 10176681和10176678近期重复序列的平均Ks值分别为0.17和0.12,与近期重复序列的平均Ks值(≈0.13)相似[7].串联重复区域的k值较高;因此,我们根据第一种模型推断这两个区域发生了复制。值得注意的是,在一个最近重复的区域(ID 18934088)上,我们发现了一个古老的重复区域,其Ks值为0.62,这与之前的报告(Ks≈0.59)的值一致[7].副重叠区和串联重复区Ks值分别为0.26和0.08。该区域在一次古复制事件后,可能经历了符合第二种模式的复制过程。ID 22835159的相似性区域的Ks值为0.35,串联复制区域的k值为0.50,其他两个区域的k值为0.16,表明两种模式是混合模式。重复区ID 18159398的k值分别为0.35和0.40;0.06为Chr 13上的串联复制区域(组1);Chr 15平均为0.13(组2);Chr 13的平均值为0.43(组3)(图2).组3的ks值比组1和2的ks值支持的混合模型首先发生组3的串联重复,然后在Chr 13和Chr 15之间的最近重复,然后最后串联重复组12。
NBS-LRR基因的转录组分析
为了进一步探索nbs - lrr的抗病功能,我们利用了此前生成的blp处理的NILs的转录组数据[8].圆环图中显示了抗性NIL和敏感NIL之间差异表达的NBS-LRR基因的位置(图)1).共35个NBS-LRR基因分布在12条染色体上(Chrs 1、2、5、6、8、9、12、13、15、16、17和20),在抗性和敏感NILs之间表达变化显著。在35个NBS-LRR基因中,有5个NBS-LRR基因聚在Chr 6末端,具有猝死综合征(SDS)和SCN抗性QTL。此外,大豆抗锈病基因座RPP3.据报道,围绕该地区(图1)[10.)(表3.).值得注意的是,2个nbs - lrr聚集在Chr 5上,BLP抗性QTL也位于2-Mb的侧翼区域。
为了验证Illumina-GA的转录组数据,我们对4个NBS-LRR基因和1个RIN4-like基因进行了qRT-PCR(见附加文件)7和8).表达谱与RNA-Seq RPKM值和qRT-PCR值一致。qRT-PCR也证实了NILs之间的基因表达差异显著(见附加文件)8),表明nbs - lrr的转录组分析是可靠的。
讨论
NBS-LRR染色体分布
NBS-LRR基因分布在大豆基因组的多条染色体上。其中,Chr 16具有最多的NBS-LRR基因,抗病QTL数量最多。nbs - lrr偏多的其他染色体也有携带抗病QTL的倾向。NBS-LRR基因数量与基因2-Mb侧翼区域抗病QTL数量显著相关。在其他植物中也有类似的相关报道。应用NBS分析技术鉴定和定位苹果抗性类似物(RGAs)的结果显示,在43个NBS分析衍生的标记中,有25个标记与一个主要QTL接近[11.].与已知抗病基因座共分离的大麦RGAs [12.].在马铃薯中,来自BAC文库的NBS-LRR基因和38个与tir -NBS-LRR共定位的抗性位点,其中大部分位于QTL [13.].因此,nbs - lrr与抗病位点或QTL的关联可能是这些作物的共同特征。
位于NBS-LRR群附近的QTL不是针对某一种疾病的,而是针对多种病原体和害虫(如真菌、细菌和线虫)的,而且往往密切相关。有趣的是,有报道称细菌基因组具有致病岛,功能相似的基因聚集在一起[14.].这些簇被认为是入侵策略机制进化的结果。这种进化背后的主要驱动力将是串联复制和可移动的dna样转座酶和整合酶[15.,16.].值得注意的是,突变- 状元素(骡子)转座酶和MuDR(突变家族转座酶结构域位于Chr 16上的NBS-LRR基因Glyma16g23790内。此外,Glyma16g23790的这个区域聚集了最多的NBS-LRR基因。这一发现与之前对花生的一项研究一致,该研究发现在含NBS-LRR基因的BAC无性系中存在高密度的转座元件。这些转座子可能是染色体内复制和宿主特异性扩大的原因[17.].
最近的大豆基因组复制和抗病功能的冗余
整个大豆基因组测序(var. Williams 82)揭示了最近重复区域的实际位置[7].到目前为止,在大豆的几种性状中已经报道了重复区域之间的功能冗余,包括种子蛋白、油脂和BLP抗性[18.,19.].根据我们的分析,抗病能力在最近重复的区域之间也是重复的。有趣的是,一些重复的基因组区域包含相同数量的NBS-LRR基因,而另一些重复的基因组区域包含完全不同数量的NBS-LRR基因2).
基因家族的序列差异可能发生在复制过程之后。具体来说,R基因经常在整个基因组中包含大量的拷贝,很可能是不平等杂交的结果[4,20.].从提出的模型来看,串联复制和染色体间复制结合的可能性表明,NBS-LRR基因的复制历史可能是这些基因附近抗病QTL多样化的因素之一(图)2).然而,并非所有具有NBS-LRR基因的区域都具有抗病QTL,这可能是由于缺乏足够的抗病QTL或与抗病功能无关的NBS-LRR基因所致。
blp抗性NIL集上NBS-LRR基因的转录组分析
转录组分析aXag结果表明,2个NBS-LRR基因的表达在blp抗性和blp敏感的NIL基因之间有显著的倍数变化,与之前报道的blp抗性QTL接近。23个NBS-LRR基因均不接近抗病QTL。Chr 17上的一个抗BLP主基因可能影响不同类型NBS-LRR基因的表达。也有可能RNA-Seq分析可能对检测不相关基因的表达很敏感。
在几个NBS-LRR基因中,显示出抗性和易感型的含量明显不同的表达水平,六个基因(Glyma01G01420,Glyma06G46800,Glyma06G46800,Glyma08G42930,Glyma08G42930和Glyma09G34360)被称为RPM1(耐抗两pv。maculicola1)据报道,据报道,通过从入侵细菌分泌的效应器改变后保护RIN4蛋白。在一个RIN4基因和由RNA-SEQ数据鉴定的四个RIN4样基因中,只有一个(Glyma15G06090.1)显示了日志2折叠变化1.76,在易感和抗性的NIL之间。该基因有一部分拟南芥RIN4和效应识别的保守域(表4).尽管转录谱并不反映该蛋白的实际功能,但这一结果表明,部分RIN4基因可能参与了与RPM1共同作用的抗性反应。在番茄中,只有一个效应结合域的部分蛋白的存在被认为是“诱饵模型”的一部分,例如PTO和RCR3 [21.,22.].
还分析来自几种NCBI沉积数据的转录om以通过各种疾病攻击弄清楚差异表达的NBS-LRR基因(见附加文件9).大豆品种威廉姆斯82的RNA-SEQ数据接种Phakopsora pachyrhizi.(MS06-1) (SRP008837,http://www.ncbi.nlm.nih.gov/sra/?term=SRP008837)威廉姆斯82和PI 194639的cDNA阵列数据接种sclerotinia sclerotiorum.(GSE15369)被使用[23.].由于大豆的共生关系Bradyrhizobium日本血吸虫我们还利用了SS2-2,超结瘤大豆突变体对该细菌(GSE10340)的响应的cDNA阵列数据[24.].
在每个转录组合组中的差异表达的NBS-LRR基因中,Glyma01g04000在所有四个转录om和Glyma05G09440和Glyma06G40980在三个转录组中差异表达,表明那些NBS-LRR基因可以针对广泛的病原体攻击工作。一些NBS-LRR基因对不同的接种来源具有特定的反应。
结论
我们分析比较了NBS-LRR基因和抗病QTL在大豆基因组中的位置,以确定NBS-LRR基因与模式植物抗病功能之间的关系。NBS-LRR基因与抗病QTL在NBS-LRR基因侧翼2mb区域的相关性足以支持这些基因参与抗病的观点。由于最近复制的NBS-LRR基因也保留了抗病QTL,其抗病功能相关性更有说服力。我们还提出了一个NBS-LRR基因重复顺序的模型,包括最近的重复和串联重复事件。此外,在大豆抗blp和不感blp的NIL集的RNA-Seq数据中Xag,我们观察到NBS-LRR基因表达的显着折叠变化。两种差异表达的NBS-LRR基因位于先前报告的BLP抗性QTL区域中,表明NBS-LRR基因可能位于下游Rxp对BLP产生抗性的位点[25.].通过开发NBS-LRR结构域的分子标记,可以整合对多种病原菌的抗病能力,形成优良的大豆品种。
方法
数据生成
从Phytozome (http://www.phytozome.net/soybean.php截至2011年12月)。该信息包含域的注释和列表拟南芥同源物和他们的描述[7,26.].为了进一步研究和确定NBS-LRR结构域,我们使用Pfam结构域信息和带有NBS和LRR结构域的基因,分别用Pfam id PF00560、PF07723和PF07725筛选LRR结构域,用PF00931筛选NBS结构域。最近的复制信息也从Phytozome下载。大豆抗病QTL从大豆基地(http://soybase.org,截至2011年12月)的真菌和线虫抗性,以及此前对blp抗性QTL的研究[9].这些QTL的物理位置是根据标记信息的物理位置推断出来的,这些标记信息以大豆地图4.0版的形式发布在SoyBase上,本分析使用的是唯一一个具有定位标记的QTL [27.].在Microsoft Excel中分析了使用NBS-LRR基因与疾病相关QTL之间使用线性回归的共定位。
为了确定NBS-LRR基因的邻近性,将基因的2-Mb侧翼区域视为连锁区域的一部分,这可能会影响QTL的统计。因此,我们检索到每个NBS-LRR基因的2mb侧翼区域,这些区域包含的QTL如图所示1.为了研究大豆基因组最近重复的抗病冗余效应,筛选了最近重复区域内的QTL和2-Mb侧翼区域。所有这些工作都是用Python的简短脚本实现的。分析的数据使用Circos可视化软件包进行解析和处理[28.].
从Phytozome下载最近重复区域和串联重复区域的编码序列(CDS),用SeqinR, R软件包软件计算Ks值[7,29.].
转录组数据分析
使用了Kim等人发表的关于blp敏感和blp耐药的NILs的转录组数据集[8].利用blp抗性NIL和blp敏感NIL之间的四倍变化截断,将差异表达的基因缩小到显著表达的基因。利用Circos将表达显著的NBS-LRR基因定位在圆形地图上。
qRT-PCR验证RNA-Seq实验
我们进行了qRT-PCR验证RNA-Seq分析。检测4个NBS-LRR基因和1个rin4样基因,利用Primer3软件设计引物(http://frodo.wi.mit.edu/primer3/)(请参阅其他文件6和7).使用Bio-rad Iscript TM cDNA合成试剂盒(Cat.170-8891,Hercules,Ca,USA)合成cDNA后,对LightCycler 480系统进行了实时定量(Roche Diagnostics,Laval,QC,加拿大)和Bio-rad IQ™Sybr Green SuperMix套件(Cat。170-8882)。PCR混合物含有200ng cDNA,500nm底漆,18μl无菌水,以及25μl的IQ™SYBR绿色超混(Bio-rad),总体积为50μL。放大条件在95℃和95℃的40℃下为5分钟的变性,10秒,60℃,20秒,72℃10秒。扩增后,在95℃的三段循环中产生熔化曲线,在连续采集模式下在95℃的三段,64℃下,64℃,为0秒。将每个靶基因的量化表达水平归一化为管蛋白的对照。
参考文献
- 1。
Flor HH:亚麻锈病的寄主-寄主相互作用及其遗传学意义。植物病理学。1955,45(12):680-685。
- 2。
植物免疫系统。自然科学学报。2006,444(7117):323-329。10.1038 / nature05286。
- 3.
植物感染后的综合防御反应。自然科学学报。2001,32(4):591 - 598。10.1038 / 35081161。
- 4.
Meyers BC, Kozik A, Griego A, Kuang HH, Michelmore RW:拟南芥nbs - lrr编码基因的全基因组分析(vol 15, pg 809, 2003)。植物学报。2003,15(7):1683-1683。
- 5.
Ashfield T, Bocian A, Held D, Henk AD, Marek LF, Danesh D, Penuela S, Meksem K, Lightfoot DA, Young ND, et al:大豆Rpg1-b抗病基因的遗传和物理定位揭示了一个包含NBS-LRR基因几个紧密相连家族的复杂位点。植物营养与肥料学报,2003,16(9):817-826。10.1094 / MPMI.2003.16.9.817。
- 6.
朱海英,杨思敏,唐芳,高MQ, Krishnan HB: R基因控制豆科植物-根瘤菌共生寄主特异性。中国科学(d辑:地球科学),2010,30(4):531 - 534。10.1073 / pnas.1011957107。
- 7.
宋强,陈建军,程军,等:古多倍体大豆基因组序列分析。自然科学学报。2010,463(7278):178-183。10.1038 / nature08670。
- 8.
Kim Kh,Kang Yj,Kim DH,Yoon My,Moon JK,Kim My,Van K,Lee Sh:RNA-SEQ分析大豆近代含有细菌叶脓疱和-Uscrectules的抗菌性和-Uscrecteles。DNA Res。2011,18(6):483-497。10.1093 / DNARES / DSR033。
- 9.
黄志强,黄志强,黄志强:大豆抗黄单胞菌基因的SSR定位。甘氨酸。遗传学报。2004,26(1):47-54。
- 10。
Hyten DL,Smith Jr,Frederick Rd,Tucker ML,Song QJ,Cregan PB:使用Goldengate测定的膨胀分析分析来定位赋予大豆生锈的RPP 3基因座。农作物科学。2009,49(1):265-271。10.2135 / cropsci2008.08.0511。
- 11.
Calenge F, Van der Linden CG, Van de Weg E, Schouten HJ, Van Arkel G, Denance C, Durel CE:通过nss -profiling方法鉴定的抗病基因类似物与苹果抗病的主效基因和QTL接近。应用计算机学报。2005,110(4):660-668。10.1007 / s00122 - 004 - 1891 - 6。
- 12.
Madsen LH,Collins NC,Rakwalska M,Backs G,Sandal N,Krusell L,Jensen J,Waterman EH,Jahoor A,Ayliffe M,等:NBS-LRR类的大麦疾病基因类似物:识别和测绘。Mol Genet基因组学。2003,269(1):150-161。
- 13。
Bakker E,Borm T,Prins P,Van der Vossen E,Uenk G,Arens M,De Boer J,Van Eck H,Muskens M,Vossen J,等,Nb-LRR疾病抵抗基因座的基因组族遗传图谱在土豆。Al Appl Genet。2011,123(3):493-508。10.1007 / s00122-011-1602-z。
- 14.
骇客J,肉毒杆菌E:生态适应度,基因组岛和细菌致病性。对微生物进化的达尔文主义观点国家自然科学基金项目,2001,21(5):457 - 461。
- 15.
作者简介:罗思,Benson N, Galitski T, Haack K, Lawrence JG, Miesel L:细菌染色体重排的形成和应用。大肠杆菌和沙门氏菌:细胞和分子生物学。1996年,2:2256 - 2276。
- 16.
串联复制的基因聚类选择。微生物学进展。2004,58:119-142。10.1146 / annurev.micro.58.030603.123806。
- 17.
Wang X, Li J, Compton RO, Rainville LK, Lemke C, Kim C, Tang H, Paterson AH:花生NBS-LRR基因簇的比较分析表明重复结构域的进化创新和基因微共体的侵蚀。植物营养学报,2011,19(1):104 - 108。10.1111 / j.1469-8137.2011.03800.x。
- 18.
动态重排决定了大豆基因组的结构和有用性状。植物营养与肥料学报。2009,31(3):369 - 372。10.1104 / pp.109.141739。
- 19.
Shoemaker RC, Polzin K, Labate J, Specht J, Brummer EC, Olson T, Young N, Concibido V, Wilcox J, Tamulonis JP,等:大豆(Glycine subgenus soja)基因组复制。中国生物医学工程学报。
- 20.
Meyers BC,Chin DB,Shen Ka,Sivaramakrishnan S,Laplle Do,张Z,Michelmore Rw:生菜中的主要抗性基因簇是高重复的,跨越几个兆级。植物细胞。1998,19,10(11):1817-1832。
- 21。
青年杂志,Shindo T, Gu C, Kaschani F, Pansuriya T, Chintha R, Harzen A, Colby T, Kamoun S, van der Hoorn RA:真菌效应蛋白AVR2靶向番茄防御相关的cys蛋白酶。植物学报。2008,20(4):1169-1183。10.1105 / tpc.107.056325。
- 22。
范德胡恩,李志刚:从防御到诱骗:一种感知植物病原效应的新模式。植物学报。2008,20(8):2009-2017。10.1105 / tpc.108.060194。
- 23。
Calla B,Radwan O,Vuong T,克劳SJ,Hartman GL:基因表达剖析大豆茎组织早期反应巩膜肺刺菌和硅映射与抵抗标志有关。植物基因组。2009,2(2):149-166。10.3835 / Plantgenome2008.02.0008。
- 24。
Brechenimacher L,Kim My,Benitez M,Li M,Joshi T,Calla B,Lee MP,Libault M,Vodkin Lo,Xu D等,Bradyrhizobium japonicum的大豆瘤的转录剖析。莫尔植物微生物。2008,21(5):631-645。10.1094 / mpmi-21-5-0631。
- 25。
王涛,李绍华,王涛。大豆细菌性脓疱Rxp调节反应的分子定位。王志强,王志强,王志强,等。10.1093 / jhered / 92.3.267。
- 26.
Finn Rd,Mistry J,Tate J,Coggill P,Heger A,Pollington Je,Gavin OL,Gunasekaran P,CercerC G,Forslund K,等:PFAM蛋白质家族数据库。核酸RES。2010,38(数据库问题):D211-D222。
- 27.
Grant D, Nelson RT, Cannon SB, Shoemaker RC: SoyBase,美国农业部大豆遗传和基因组数据库。核酸Res. 2010, 38(数据库版):D843-D846。
- 28.
Krzywinski M, Schein J, Birol I, Connors J, Gascoyne R, Horsman D, Jones SJ, Marra MA: Circos:比较基因组学的信息美学。中国生物医学工程学报,2009,19(9):1639-1645。10.1101 / gr.092759.109。
- 29.
Charif D, Lobry JR: SeqinR 1.0.2:一个贡献的包,用于统计计算的R项目,致力于生物序列检索和分析。序列进化的结构方法:分子,网络,种群。Bastolla U, Porto M, Roman HE, Vendruscolo M.编辑。纽约:施普林格Verlag;2007:207 - 232。
确认
本研究得到了大韩民国农村发展管理局的下一代生物生物21分(No.PJ008117)的补助金。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
YJK设计了该研究,并使用生物信息学工具分析了序列。KHK进行RNA-SEQ并产生转录组数据。SRS有助于鉴定抗毒性QTL的毒性QTL。Myy通过QRT-PCR验证了重要的基因表达。SS有助于接种黄axonopodispv。甘氨酸并证实了由这种细菌引起的疾病症状MYK, KV和SHL帮助设计了这项研究,并起草了手稿。所有作者都已阅读并批准了最终稿。
电子辅料
12870 _2012_1115_moesm1_esm.xlsx
附加文件1:cc - nbs - lrr、tir - nbs - lrr和其他nbs - lrr的2mb侧翼区抗性QTL。(XLSX 26 KB)
12870 _2012_1115_moesm3_esm.docx
附加文件3:NBS-LRR基因数量与NBS-LRR基因2 mb侧翼区域抗病QTL数量的相关性。(多克斯17 KB)
12870_2012_1115_moesm5_esm.xlsx.
附加文件5:NBS-LRR基因2-Mb侧翼区域的QTL数量。(XLSX 11 KB)
12870_2012_1115_moesm6_esm.xlsx.
附加文件6:NBS-LRR基因最近重复区域之间和串联重复区域之间的Ks值。(XLSX 13 KB)
作者为图像提交的原始文件
权利和权限
开放访问本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
姜玉杰,金金衡,沈淑珍。等等。NBS-LRR基因的基因组映射及其与大豆抗病性的关系。BMC植物杂志12,139(2012)。https://doi.org/10.1186/1471-2229-12-139
收到了:
接受:
发表:
关键词
- 基因组重复
- NBS-LRR
- 大豆
- 转录组分析