摘要
背景
下一代测序(NGS)技术为加速许多物种的精细定位和基因分离提供了新途径。到目前为止,这些努力主要集中在具有容易获得的全基因组序列信息的二倍体生物体上。在这项研究中,作为概念证明,我们测试了NGS在四倍体小麦系中发现SNP的使用,这些小麦系与之前克隆的粒蛋白含量(GPC)基因不同GPC-B1.使用散装分离分析(Bulked separant analysis, BSA)定义候选基因区域内的假定snp子集,然后用于精细定位GPC-B1.
结果
我们使用Illumina配对端部技术对近等基因系的mRNA (RNAseq)进行测序,测序间隔为~ 30cmGPC-B1轨迹。在对两个同源小麦基因组进行snp鉴别和额外的质量筛选后,我们在亲本系之间的小麦unigenes中鉴定出了品种间snp。这些SNPs的相对频率由RNAseq在两个由GPC表型不同的纯合子重组系组成的散装样品中检测。进一步评估了在相应池中富集至少3倍的SNPs(占所有SNPs的6.5%)。标记分析是针对富集的SNPs的一个子集设计的,并使用来自每个批量个体的DNA进行绘制。39个新的SNP标记,对应于67%的验证SNP,映射在12.2 cm的区间,包括GPC-B1.这转化为每0.31 cM 1个SNP标记GPC-B1在同位谷物基因组中,基因变异在13-18个基因之间,在小麦中变异在0.4 cM之间。
结论
这项研究证明了RNAseq在多倍体物种中用于SNP发现,并支持使用BSA作为一种有效的方法,将SNP定位到特定的遗传区间,以精细定位未测序基因组中的基因。
背景
小麦是一种主要的主食,提供了人类消耗的大约20%的卡路里和蛋白质[1].两种主要的栽培小麦品种是四倍体硬粒小麦(小麦属植物turgidumssp。硬质),通常用于制作意大利面,以及六倍体面包小麦(t . aestivum),用于制作面包和饼干。这两种多倍体物种的基因组大小都很大(每个单倍体基因组5.3 Gb) [2],重复元素比例高(~85-90%)[3.],低基因密度,缺乏一个组装的全基因组序列。这使得通过正向和反向遗传方法鉴定基因成为一项耗时且具有挑战性的任务。
为了鉴定小麦中的基因,通常采用传统的定位克隆项目,尽管它们的通量很低,而且要持续很多年。最初的基因发现阶段包括全基因组调查,在数量性状的情况下,与随后使用近等基因系对最相关的候选区域进行验证相耦合[4,5].基因分离的后续步骤包括利用与已测序谷物基因组的同位关系,通过增加目标区域的标记密度来进行精细定位(短枝稻,水稻,高粱二色的) [6- - - - - -10].最后的步骤包括制作物理地图[11,12],在许多情况下使用专用的BAC库[13],以及候选基因验证[14,15].该策略已成功应用于若干小麦重要基因的克隆及QTL(综述于[16])。然而,这些例子都是孤立的案例。在多倍体小麦中,基因定位和克隆的速度和效率需要新的方法来逐步改变。
下一代测序(NGS)技术为加速性状的遗传分析提供了新的途径。NGS的一个应用是使用全基因组重测序来帮助精确定位和识别因果多态性。模型工厂最近发表了几种策略拟南芥并被统称为“NGS-enabled genetics”[17].SHOREmap方法[18]使用单一的NGS反应对大量突变F进行全基因组重测序2允许在感兴趣的基因中鉴定致病单核苷酸多态性(SNP)的个体。奥斯汀和同事们[19]提出了这种方法的修改版本(称为“下一代映射”),它需要更小的F2与SHOREmap相比,使用统计数据定性地描述SNP频率。另一种被称为“快进遗传学”的方法,将散装分离技术与基因组捕获技术结合起来,以识别候选基因[20.].这三种方法突出了NGS的力量,以及不同的方法如何结合起来,在提供完整的基因组序列和详细的SNP目录的情况下,加速二倍体模式生物中基因的精细定位和克隆。
加速多倍体小麦基因的精细定位和克隆的能力将对我们理解这一重要作物物种的基础生物学产生巨大影响,并将使小麦育种者能够直接获得编码重要农艺性状的基因变异。非测序多倍体物种转录组SNP检测方法的最新进展[21- - - - - -23]提供了使用“ngs基因”的机会[17为了这个目的。为了解决这个问题,我们评估了NGS在SNP发现和散装分离分析(BSA)中的使用[24]在多倍体小麦中精确定位先前克隆的基因(GPC-B1) [14].利用mRNA样本上的NGS (RNAseq),我们在亲本系之间鉴定了超过3500个假定的SNPs,并在两个表型相反的重组系的大样本中检测了它们的频率。我们在12.2 cM区间(每0.31 cM 1个SNP)绘制了39个新的SNP,并进行了精细映射GPC-B1到约0.4 cm和13-18个基因之间的共线谷物基因组。
结果
Illumina的赋值读取参照小麦unigenes
两个近等基因系为GPC-B1选择籽粒蛋白含量的位点来鉴定该染色体区域的snp。第一个系是四倍体硬粒小麦兰登(LDN),它携带一个无功能的等位基因GPC-B1.第二个系是四倍体重组取代系65 (RSL65),该系由LDN和野生二聚体(小麦属植物turgidumssp。dicoccoides) 6B染色体替代系[LDN (DIC6B)][4].RSL65携带至少30厘米的6B染色体野生二聚体片段,其中包括一个功能性二聚体GPC-B1等位基因,但应该是等基因的LDN以外的区间。为了减少生长条件的变化,亲本LDN和RSL65在同一个花盆中一起生长。从第5叶期的叶片中提取总RNA,并在Illumina GAIIx上制备非归一化样品进行mRNA-seq(120个循环,配对端(PE),每个亲本一车道)。完整过程的流程图可在附加文件中找到1:图S1。
除去适配器污染和低质量区域的低质量读取和修剪后,LDN和RSL65分别获得了256和3170万对末端120碱基读取。目前还没有小麦可用的参考基因组序列,因此每个亲本的个体读数都与NCBI小麦转录组相匹配[25],由40,349个单基因序列组成,共计31,671,110个碱基。近50%的LDN原始reads(2550万)映射到参考unigenes,其中78.6%是成对映射的。对于RSL65, 47%的原始reads(2990万)映射到NCBI Unigene参考,其中67.2%成对映射。总的来说,在Illumina产生的1.146亿份reads中,48.3%映射到NCBI Unigene参考。
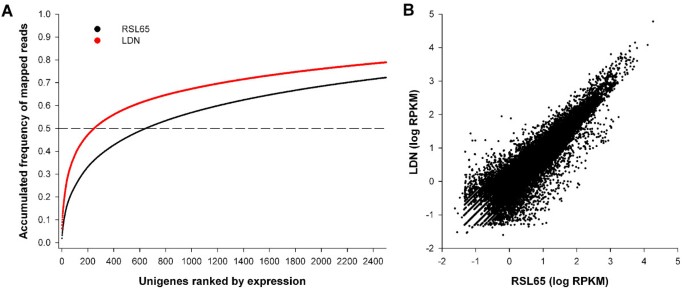
在本研究中,RNA没有被归一化,因为目标是发现SNP和随后鉴定散装样品之间的等位基因频率。为了检验这种方法对高表达转录本测序的影响,通过计算以每千碱基每百万映射reads (RPKM)表示的转录本丰度来估计unigenes的相对表达水平[26].根据Unigenes的RPKM从高到低进行排序,并计算映射reads的累积频率(图1).对于LDN数据集,14.8%的映射reads对应于10个表达最多的unigenes(包括与光合作用和重复元素相关的基因),一半的映射reads对应于248个表达最高的unigenes。在RSL65中,这些值略大,50%的映射reads对准了647个高表达的unigenes。图中曲线的斜率1在两个亲本系的50%值后减少。这表明规范化的缺乏导致了少数转录本的极端过度表达(在LDN和RSL65中,50%的reads分别映射到所有有映射reads的unigenes的0.98%或2.47%)。
尽管使用了不完整的参考文献,但在单个Illumina lane中有足够的测序深度来将reads(至少8倍覆盖)分别与LDN和RSL65中的24083和25080个unigenes进行比对。这代表了亲本数据中大约60%的单基因参考集。比较亲本系之间的单基因转录水平(RPKM)显示出较高的相关性(R2= 0.85,图1 b)表明所采用的方法对于SNP发现和后来描述的散装分离分析都是有效的。
四倍体小麦近等基因系SNP的发现
在多倍体中,SNP的发现被两种类型SNP的存在所混淆。第一个对应于发生在纯合子个体中的同源基因组之间的多态性。这些SNPs通常在四倍体(A和B基因组)和六倍体小麦(A, B和D基因组)中发现,因为同源基因组具有~96-98%的序列一致性[27].这些snp被称为同源多态性(IHP) [21].第二种多态性对应于个体之间的品种SNPs,代表了传统上所说的等位基因变异。这种类型的SNP很少出现,现代小麦品种在相应的同源位点上有99.9%相同。由于参考小麦单基因中的转录本组合代表了来自共同组装的a、B和D基因组的一致序列(即没有模糊代码,就像单基因一样),因此不能通过简单的SNP发现管道来区分这两种类型的SNP。因此,基于先前针对多倍体油菜开发的方法,对snp进行了检测和评分芸苔属植物显著[21,28].
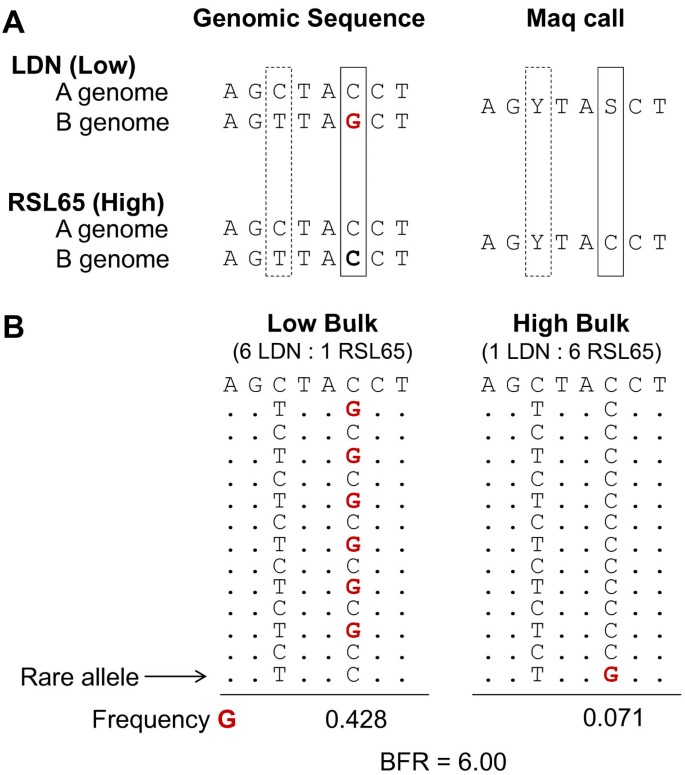
在这种方法中,预期来自同源基因(四倍体小麦中的A和B基因组)的reads将被映射到相同的单基因参考。Maq(默认参数)[29]用于分别调用每个亲本系的参考SNP,从而生成两个SNP集。接下来是第二步,使用自定义Perl脚本派生两个父SNP集的对称差异。同源基因组之间的多态性应该在亲本系中产生相同的歧义代码,并且对于两个SNP集都应该是共同的(图2, C/T生成Y码)。另一方面,LDN和RSL65之间的品种SNP应该只为一个亲本产生模糊码,因此对相应的SNP集是唯一的(图2,全线盒装SNP)。这种位于同一位点的品种snp被Trick等人称为“半snp”。[21].在只表达一个基因组或单拷贝基因的情况下,snp可以被识别为传统的“简单snp”,每个亲本由一个碱基表示。
多倍体基因组中SNP鉴定和牛血清白蛋白的示意图.一)a和B基因组之间同源SNP的示意图(C/T,虚线矩形)和LDN和RSL65之间的品种间SNP(或半SNP) (C/G,全线矩形)。同源SNP在双亲中产生相同的Maq歧义码(C/T = Y),而半SNP在带有信息性碱基的亲本系中产生唯一的歧义码(S)(红色粗体G)。数字改编自[21].B紧密相连的一个SNP的等位基因组成的示意图GPC-B1在低和高的散装。参考A基因组表示在顶部,附加读数在下面对齐。同源SNP (C/T)和半同源SNP (C/G)显示。每个批量中LDN和RSL65植物之间的比率被显示(LDN:RSL65),作为信息基G的计算BFR。
在LDN和RSL65之间的2520个unigenes中,这个两步管道的实施确定了3963个假定的品种SNPs。所发现的snp绝大多数(80.8%)为半snp型,表现为模糊编码,表明同源基因共表达,仅在亲本一方具有特定的核苷酸多态性。在这种情况下,半snp成为等位基因,并作为遗传上可控制的标记。
在研究非测序和复杂的基因组(如小麦的基因组)时,一个值得关注的问题是存在密切相关的副基因或假基因,它们可能会混淆SNP的发现和下游标记的设计和定位。有证据表明,同源同源序列被映射到相同的参考序列中是具有高SNP密度的unigenes。因此,我们计算了至少含有一个SNP的2520个单基因的SNP密度,并检查了它们的频率分布。平均SNP密度为1.80 SNPs/kb(±1.46),超过3个SNPs/kb后迅速衰减。这一值与通过对4个基因的外显子和utr的6.8 kb进行测序而经验计算的结果(2.2 SNPs/kb)相似GPC-B1地区(14].基于这些结果,消除了SNP密度高于5 SNPs/kb的unigenes,以避免可能的同源,但仍为一些变异留有空间。大约13%的假定snp被丢弃,将2427个unigenes的假定snp数量减少到3427个(1.80 SNPs/kb)。
使用修改的对齐和SNP调用标准发现SNP
对几个snp的目视检查显示,Maq不能正确地调用发生在同源snp附近或在10 bp滑动窗口内的snp。这意味着一些IHPs被称为品种半snp,因为它们很接近。因此,通过在工作流的每个步骤中将不匹配的碱基的最大质量总和分数设置为120(默认为70)(以下称为Maq-120),放宽了缺省Maq参数。这使得发生在120个碱基读取内的SNP单倍型能够与更高的映射质量相匹配,并减少这些错误。
Maq-120分析在映射到单基因参考的总读数方面产生了一些改进(双亲系均为55.2%,而Maq-default为48.3%),尽管成对映射的读数的百分比保持不变(表12)1).这些reads与LDN和RSL65的Unigene覆盖至少8倍,分别达到25,262和26,180(约占NCBI Unigene集的63%)。在3195个unigenes中共鉴定出6035个假定snp(2.41个SNPs/kb),与Maq默认管道相比有相当大的增加。同样,半snp是最主要的,超过89%的snp被分配给这种类型。在筛选> 5 SNPs/kb的unigenes后,大约四分之一的假定SNPs被丢弃(在226个unigenes中有1605 SNPs)。Maq-120分析的最终输出是2969个unigenes中的4430个假定snp (1.90 SNPs/kb)。
通过散装分离分析鉴定假定的链接snp
28个纯合子系,在约12cm的间隔内发生重组事件(Xwms508-Xwms193) [5]被用来组装散装,以对比表型(籽粒蛋白质浓度)。这些线首先被表征为三个标记,跨越相邻的250kb区域GPC-B1来确认他们的基因型。除RSL135外,所有的系都产生了预期的结果,RSL135对所有标记都是杂合的,因此被排除在下游工作之外。来自14个先前被分类为高蛋白质含量的个体的等量总RNA被混合以产生高蛋白RNA块,而15个重组系用于低蛋白质块(这些系的详细描述见“材料和方法”)。对于每个批量,构建两个平均插入大小略有不同的文库(250-bp和400-bp),并在单独的Illumina车道(80个循环,成对端)中测序。
reads的质量控制评估与亲本系相似。对于Maq-default和Maq-120分析,与250bp库相比,在400 bp库中获得了更高比例的映射到参考集的读取(前者多25%),尽管成对对齐的读取数量较少(表2 -1)2).结合这两个库,分别为高批量和低批量产生了5340万和6120万对终读。同样,与Maq-default分析(41.8%)相比,Maq-120分析更成功地将更高百分比的读取与参考数据对齐(47.6%)。这些值与仅比较等效的400 bp库时在亲代行中获得的值相似。
从散装样品中测序RNA的目的是比较两个散装样品中亲本SNPs的等位基因频率。在一个二倍体生物体中,一个与感兴趣性状的基因相一致的SNP应该在两个群体中通过信息性的碱基频率趋向于1.0或0.0来揭示,这取决于亲本来源。然而,在多倍体物种中,1.0的上限更低。例如,对于LDN (C/G)中相对于RLS65 (C)识别的半snp,来自信息性碱基(G)的信号被同源(非信息性)碱基(C)部分掩盖(图2 b).因此,假设两个同源转录本定量共表达,在四倍体物种中,信息性碱基的上限频率趋向于0.5。当相对转录水平偏离平价时,该值将发生变化。对于每个bulk,在每个SNP位置计算信息基的频率,然后确定每个SNP的bulk之间的比率。这种体积之间的比率被称为体积频率比(BFR)。对于从LDN中获得信息性碱基的半snp, BFRs是通过将低块频率除以高块频率来确定的。对于来自RSL65的半单核苷酸多态性,这些值是相反的。因此,BFR提供了相应亲本等位基因在适当体积(LDN为低体积,RSL65为高体积)富集程度的相对测量。
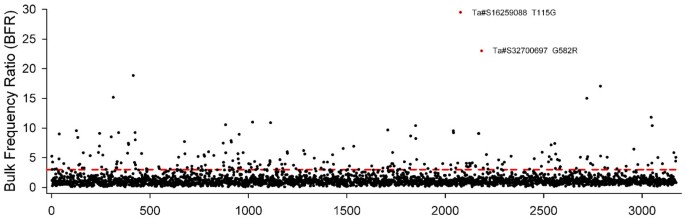
该方法只关注在初始实验中先前在LDN和RSL65之间确定的假定snp,而不寻求在复杂的体RNA混合物中识别snp。在Maq-default分析中识别的3427个snp(2427个unigenes)中,有1619个snp被恢复,并且在两个bulk中序列覆盖率至少为8倍(附加文件)2:表S1)。在本分析中,每个单基因仅使用一个SNP来估计BFR。因此,snp的回收率仅为47.2%,尽管这代表了unigenes的66.7%。单基因Ta#S16259088中的简单SNP T115G被鉴定为最高的BFR,其富集量为28.5倍。LDN等位基因仅在3.4%的高容量序列中存在,而96.5%的低容量reads携带该等位基因。这些值与紧密相连的简单SNP的预期频率一致。该单基因与BrachypodiumBradi3g03340,水稻LOC_Os02g04500,高粱Sb04g003030,均位于小麦染色体臂6BS对应的共线区域,其中GPC-B1地图。
Maq-120假定snp(2969个unigenes中的4430个snp)的等位基因频率也在大块中进行了检测。在这个新的管道中,单个单基因中多个snp的频率和比例是独立估计的,因此考虑了所有snp。超过71%的SNPs(3172)具有至少8倍的覆盖率,并且可以在两个bulk中检测到(图2)3.,附加文件3.).同样,假定BFR最高的SNP是单基因Ta#S16259088中的T115G,尽管准确的值在Maq-120管道中略有变化(富集29.5倍)。假定BFR第二高的SNP是ta# S32700697中的RSL65半SNP G582R。信息性碱基(A = R = G/A)仅在低批量reads中出现1.7%,而在高批量reads(23倍富集)中出现39.7%。这些值与紧密连接的半snp的预期一致。同样,该单基因与小麦染色体臂6BS共线区域内的谷物基因同源(Bradi3g03530, LOC_Os02g04660, Sb04g003160)。
SNP验证
两种分析中BFRs的分布被用来确定哪些假定的snp需要验证和映射(表3.).大约85%的snp的BFR低于2.0,这表明LDN和RSL65等位基因在两个批量中表达水平相对相似。在两种分析中,随着BFR阈值的增加,snp的数量相对减少。目的是使用一个足够低的阈值来检查这种方法的敏感性,同时将待验证的snp数量保持在一个可管理的数量内。因此,根据经验,BFR≥3.0作为阈值,在253个unigenes中共产生270个SNPs(在两种分析中共有41个SNPs)。
为了验证这些假定的多态性,选择每个单基因的单个SNP进行标记发育。首先检验了Maq-default分析(BFR高于3.0)中的99个假定snp。5×中国春小麦基因组序列的高命中数(> 100命中,e值1E-50)决定了15个snp要么位于距离可用序列末端太近的位置,要么其指定的单基因重复[30.].因此,将这15个snp从进一步分析中移除。共开发了84个分析,其中82个成功扩增了LDN和RSL65。采用单链构象多态性(SSCP)进行PCR扩增,48个unigenes(58.5%)检测到目标多态性,34个单型(41.5%)检测到目标多态性。单型扩增子中假定的SNPs的缺失由PCR产物的直接测序证实(附加文件)1:图S2)。
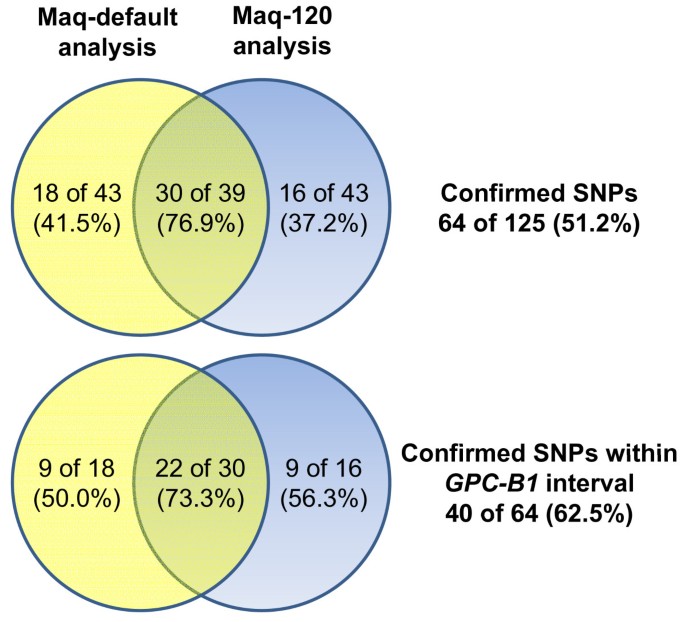
随后的Maq-120分析共产生了212个BFRs高于3.0的假定snp, 41个snp(19.3%)与Maq-default分析相同。对这些常见的假定snp的检查表明,在之前开发的39个成功的检测中,30个在LDN和RSL65之间具有多态性(76.9%),而只有9个是单态的。相对于初始Maq-default分析中的平均多态性率(58.5%多态性),这是一个很大的增加(图4).
为了评估在所有Maq-120假定SNP中验证率的增加是否一致,对另外43个仅在Maq-120分析中确定的假定SNP进行了SNP测定。与普通snp相比,Maq-120唯一snp的验证率(16个多态,37.2%)较低,与Maq-default唯一snp的验证率(41.5%)相当。从Maq-120分析中,共成功筛选出82个假定snp(39个常见snp, 43个独特snp),其中46个snp被验证为多态性(56.1%),36个snp被确认为单态snp。这些结果表明,Maq-120分析能够以与Maq-default分析(58.5%)相似的转换率(56.1%)识别假定SNP,但两种分析之间共有的SNP产生了最高验证率(76.9%)的假定SNP集。
综上所述,在总共125个假定snp中,有64个snp(18个是Maq-default独有的,16个是Maq-120独有的,30个是两种方法共有的)在亲本系中被确认为多态性(51.2%)。
已验证snp的映射
在包含高蛋白和低蛋白的个体RSLs中筛选64个验证的snp。通过SSCP(42个SNPs)和PCR产物直接测序(6个SNPs)筛选Maq-default分析的snp,而通过KASPar法(16个SNPs)筛选Maq-120分析中确认的snp(不包括常见的Maq-default分析)。所有确认的SNP都可以在29个RSLs和亲本系中被评分,除了一个SNP (Ta#S32606580),它很难被SSCP明确评分,因此从进一步的分析中删除。
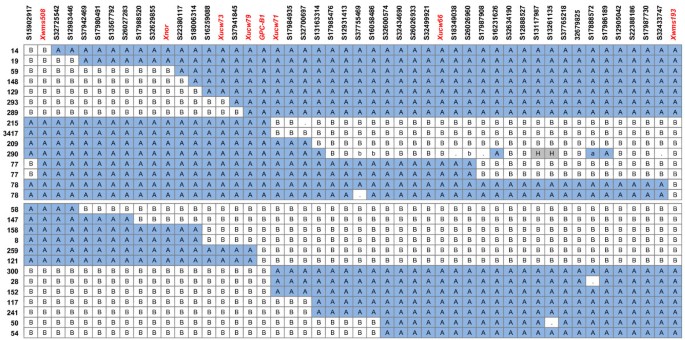
共40个SNPs映射到12.2 cM区域GPC-B1,定义为Xwms508-Xwms193(图5,附加文件1:图S3)。这意味着约63%的已验证snp映射到由RSLs确定的目标区域。24个snp没有被立即映射到该区域,尽管其中有11个snp相互连接。在40个相连的snp中,有一个位于Xwms508,而在12.2 cM目标区间内映射的SNPs有39个5).这相当于该区域每0.31 cM有一个SNP标记的平均标记密度,其值从图的远端每0.59 cM有一个SNP标记(Xwms508-GPC-B1),到近端每0.19 cM有一个SNP标记(GPC-B1-Xwms193).该地区几乎所有的重组事件都被发现了,包括以前没有解决的其他事件。未识别的重组事件与侧翼的重组事件相对应GPC-B1基因在两边,中间Xucw79而且Xucw71.这40个snp的组成与整体Maq-default和Maq-120分析相似,半snp的数量(33个)高于简单snp(7个),来自每个亲本的比例相似(16个LDN和17个RSL65 snp)。SNP标记的详细信息在附加文件中提供4.
所测序的40个小麦unigenes中有32个具有独特的同源体Brachypodium和水稻基因组,而31个unigenes在高粱中有独特的同源体。根据所建立的共向关系,其中22、18和20个正交配位在相应的共向区域Brachypodium分别是大米和高粱。这意味着56%(大米)和69% (Brachypodium)所映射的unigenes是同程的。这些共线区域介于5.5 - 6.8 Mb之间,包括665、989和607个基因Brachypodium分别是大米和高粱。研究了同源同源谷物基因的分布,以估计测序基因组中每个SNP之间的基因数量。平均来看,同源共线基因分别为32个、59个和32个Brachypodium分别是大米和高粱。这一平均值包括相邻基因上的标记和中间隔最多103个基因的标记Brachypodium高粱和水稻中的248个基因。其余的unigenes在这些已测序的草基因组的其他地方具有同源同源,除了两个在小麦中连接并在小麦中映射紧密的unigenes之外,没有明显的模式Brachypodium(Bradi1g67540和Bradi1g67570),大米(LOC_Os03g15390和LOC_Os03g15350)和高粱(Sb01g040520和Sb01g04530)。
的GPC-B1利用先前发表的RSLs表型信息对基因进行了定位[5,14]在0.4 cM的间隔内,由侧翼位点Ta#S37941845和Ta#S17984935定义4,图5).这些标记要么完全链接,要么与Maq-default和Maq-120分析中BFRs最高的snp只有一个重组(ta# S16259088和ta# S32700697,图3.).这些标记的同源性Brachypodium,大米和高粱提供了一个直接的位置GPC-B1在一个区域内分别包含大约18、16和13个基因。表格4显示了这些标记的特征及其共位关系。这些结果,以及SNP标记在小麦中的分布GPC-B1基因图谱,表明通过BSA富集在识别紧密间隔的标记方面非常有效,能够将基因定位到较窄的遗传区间。
为了进一步明确这一立场,我们检查了亲本SNP数据,以确定共线谷物基因没有相应的小麦单基因的情况,或者评估候选基因中的亲本SNP是否在散装中未被识别。我们分析了23个与基因同源的小麦unigenesBrachypodium其中,只有1个SNP位点位于LDN和RSL65之间(BFR 1.9),被证实为单型。接下来,我们在小麦中开发了另外9个基因模型,用于绘制Illumina reads的参考集中没有相应的单基因。同样,这个搜索是不成功的,因为没有发现额外的亲本snp基于我们的映射标准。不管这些具体的结果如何,以迭代的方式检查数据的能力应该对其他基因目标有用。
覆盖对SNP呼叫和富集的影响
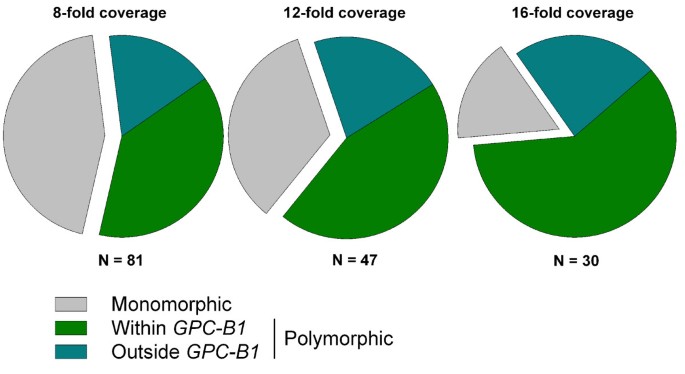
增加应用于整个SNP调用过程的深度阈值,可以增加来自每个同源语的读取被采样的概率。因此,我们检查了不同的最小深度阈值(8倍、12倍和16倍)对Maq-120分析中识别的snp假阳性率的影响。首先,BFR高于3.0的snp总数从8倍覆盖率时的212个减少到121个(12倍覆盖率),16倍覆盖率时的推定snp数量为84个。实验测试了Maq-120分析中82个snp的子集,并对其进行了进一步检查(图6).在8倍的覆盖率下,56.8%的预测snp(81个功能检测中的46个)在亲本系中得到验证,并可以在重组中进行映射。该值在12倍和16倍的覆盖率下显著增加,其中66%(47个中的31个)和83%(30个中的25个)的假定snp被证实是多态的。在三种覆盖情景下测定了相似数量的总假定snp(37-40%),这表明比较是有意义的。在所有三种情况下,67-72%的多态snp映射到GPC-B1目标区间。总而言之,将覆盖率从8倍增加到16倍,可将散装中确定的假定snp的总数减少60%,将验证率从57%-83%提高,但不影响已验证snp映射到目标区间的百分比(约67-72%)。
讨论
使用ngs支持的正向遗传学正在彻底改变模型二倍体生物中精细映射和克隆基因的速度和能力[17].为了了解这些方法如何在小麦等多倍体非测序物种中实施,我们研究了NGS在mRNA转录本上的使用,以发现SNP,并将其与BSA结合,以识别与致病基因密切相关的SNP。
ngs基因在小麦中的应用
我们的策略包括使用近等基因系(LDN和RSL65)将SNP发现集中在特定的染色体间隔上。我们假设大多数snp会映射到6B染色体上的~30 cM区间(包括短臂和长臂),在这些线之间分离。由于缺乏组装的小麦基因组序列,我们不可能对小麦的结果进行直接分析。因此,我们使用最接近的测序谷物进行了分析,Brachypodium distachyon[6],承认只有60-70%的基因具有真正的同位关系[31- - - - - -33].
三个Brachypodium区域被认为代表过多(附加文件1:图S4),包括3号染色体的两个不同的独立区域。这是意料之中的,基于麦类6组染色体之间建立良好的共线关系Brachypodium染色体3 [6,34].同步性横跨麦类第6组和第6组的短臂Brachypodium(至少到Bradi3g09080, 7.2 Mb),并继续在长臂小麦6和Brachypodium区间从大约47.4 Mb开始(Bradi3g45420),结束于染色体的末端(59.8 Mb;Bradi3g61020) [34].短臂共向区在整个段间富集,而长臂段仅在近段富集。这很可能反映了RSL65不是一个完整的染色体替代系,已知在染色体臂6BL的远端携带一个LDN片段[4].
我们还确定了一个区域Brachypodium第2号染色体(Bradi2g15900-Bradi2g20570)与小麦染色体臂1 L有整体共时关系,SNPs意外富集[6,32,34].在此区间内鉴定的66个snp中,只有一个(Ta#S13261135, Bradi2g18330)被映射到6BS区域,并且只有2个snp(3%)富集在大块(BFR≥3.0)。这与映射到Brachypodium在第3染色体上鉴定出351个SNPs,超过13%(351个中的46个)在大块中富集。这表明在RSL65中存在一个额外的野生二聚体染色体片段,这在以前没有被分子标记识别出来,或者在小麦6B中插入了一小部分基因。总的来说,该方法成功地识别了相应共线区域的snp,尽管所有区域的背景噪声水平不同Brachypodium染色体。在分析12倍和16倍的覆盖数据时,背景噪声下降,而之前确定的三个区域始终高于背景水平(附加文件)1:图S4)。
我们采样了健康的营养期叶片,其转录组在EST集合中应该有很好的体现,但我们最多只成功地将Illumina reads的57%映射到NCBI Unigene参考。尽管在Maq中测试了几个不同的参数,使用不同的库插入大小和不同的读取修剪阈值。在某种程度上,这是令人惊讶的,因为Unigene构建包括来自不同品系和发育阶段的100多万个小麦EST,尽管最近对小麦染色体1组基因的测序和分析表明,它们中只有不到65%在EST集合中得到了表达[32].Maq-default的结果与最近的研究一致芸苔属植物[28],该研究也使用了从80万ESTs中提取的单基因集作为参考,但有80个碱基reads。我们通过将最初未映射到NCBI unigenes上的基因与454个‘中国春小麦’基因组序列的5×组装(M. Bevan, JIC,个人交流)重新映射,进一步检查了我们的reads。我们发现57%的未映射LDN reads和65%的未映射RSL65 reads与这个新参考序列一致,这意味着至少80-85%的Illumina reads实际上是表达的小麦序列。这些结果也与花后12天旗叶的最近454 RNAseq数据一致[35].本研究共收集了1460个与NCBI小麦EST序列无序列相似性(BLASTN, E值,1E-10)的新转录本,突出了目前NCBI参考小麦Unigene集的不完整性。
这种抽样策略的一个结果是,我们消除了确定因果关系的可能性GPC-B1SNP。该基因仅在开花后表达[14],在用于RNAseq实验的营养组织中未检测到。这样做是有意识的,因为我们的目标是使用原始克隆项目中可用的信息来测试该方法的性能。衰老相关的表达模式仅在位置克隆的最后阶段被发现[36],因此抽样工作尽可能以最公正的方式进行。我们还考虑了最近在并行工作中证实的NCBI Unigene参考文献中衰老相关转录本的代表性不足[35]并且,我们预测,这将导致更少的映射读取和被检查方法的整体下降。
在小麦中进行ngs遗传学的任何尝试的一个关键特征是必须降低样本的复杂性。由于四倍体(~ 11000 Mb)和六倍体(~ 16000 Mb)小麦的基因组很大,即使有目前的测序能力,直接的基因组DNA测序对单个群体来说在经济上也是不可实现的。因此,必须使用替代方法。基因组捕获[37]目前有几个研究多倍体小麦的小组正在进行研究[38],尽管这种方法本质上仅限于捕获阵列上定义的基因。如上所述,目前的小麦单基因集可能还不够完整,无法达到作图项目所需的精度。另一种方法是RNAseq [39],代表了一个开放的平台,只要它们表达到与所实现的测序深度兼容的水平,就可以检测到新的转录本。
在本研究中,我们使用RNAseq作为降低四倍体小麦复杂性的策略。这种方法已经成功地应用于几个物种的SNP发现,并且只关注完整基因组的一小部分。例如,NCBI参考基因空间为31,671,110 bp /基因组当量,占完整四倍体基因组的不到1%。我们还有意识地使用非归一化RNA样本,因为下游BSA需要定量估计频率比,这将受到归一化程序的干扰。然而,这产生了一个主要的缺陷,因为50%的映射reads对应于非常高表达的unigenes的一小部分(LDN中< 1%,RSL65中< 2.5%)。在考虑到这些“丢失”的读取和那些没有映射到NCBI参考的读取后,生成的所有原始读取中不到30%(约1500万个读取)用于绝大多数SNP发现和BSA分析。尽管如此,这仍然为每个单基因提供了平均23倍的每个基因组覆盖率。
使用RNAseq进行基因组还原也会在RNA样本中的SNP调用和基因组DNA (gDNA)中的SNP验证之间产生差异。同源基因组之间的表达差异导致在RNAseq数据中识别出snp,但这些假定的snp随后在DNA样本中没有被识别出来。这混淆了我们的结果,并且在SNP检测中使用的低覆盖率进一步强调(下面将讨论,附加文件)1:表S1)。此外,与gDNA相比,RNA表达数据具有更大的可变性,这意味着BFR值与它们在整个地图上的位置并不完全相关GPC-B1(附加文件1:图S3)。尽管存在这些明显的低效率,但我们的结果表明,非标准化RNAseq对于四倍体小麦中的SNP发现和BSA分析都是一种成功的策略。
SNP的发现和验证
读取的初始映射使用Maq默认参数70进行,以获得不匹配碱基的最大质量总和分数。在多倍体物种中,高质量的错配不仅是由品种的SNPs造成的,而且还由基因组之间的同源SNPs造成。由于同源小麦转录本大约97%相同[27],我们将映射标准放宽到120,以便在整个reads中获得3个高质量的IHPs和一个额外的品种SNP (Maq-120分析)。这更好地反映了生物学和事实,即单基因参考是基于小麦的ESTs,它代表了所有三个同源体,并已被分解为一个一致的序列。
正如预期的那样,Maq-120参数增加了所有样本的映射读取百分比,并在22%的unigenes中多识别29%的假定snp,优于Maq-default。不同方法对假定snp的验证率非常相似(Maq-default和Maq-120的验证率分别为58.5%和56.1%)。Maq-120分析提供了检测接近IHPs(附加文件)的品种snp的能力1:图S5),这是在使用Maq-default参数时最初遗漏的。然而,通过放宽整体映射参数,从Maq-default分析中得到的几个已确认的品种SNPs被丢弃,因为相应的unigenes的SNP密度高于5 SNPs/kb。这突出了在映射参数中找到正确平衡的困难,并举例说明了每种方法的潜在缺陷。
检测罕见转录本的能力是SNP发现和BSA策略的一个重要方面,特别是在具有半SNP的多倍体物种中。兰德-沃特曼模型[40]提供了8倍覆盖率的初始估计,以99.97%的概率随机采样读取,但这并没有解释本研究中检测的序列的四倍体性质,并假设读取将是随机采样。温德尔和威尔逊[41]解决来自二倍体生物的杂合子样本的覆盖问题,这可以作为我们研究的近似值。根据他们的计算,在16倍覆盖率的情况下,两个亲本等位基因至少有两个独立读取的概率为99.39%,而在只有8倍覆盖率的情况下,这个概率下降到82.53%(对于一个读取,这些值分别为99.93%和96.37%)。我们分析表达序列的事实增加了一个额外的变异层,尽管亲本等位基因(R2= 0.85)表示线条之间的相对平衡。
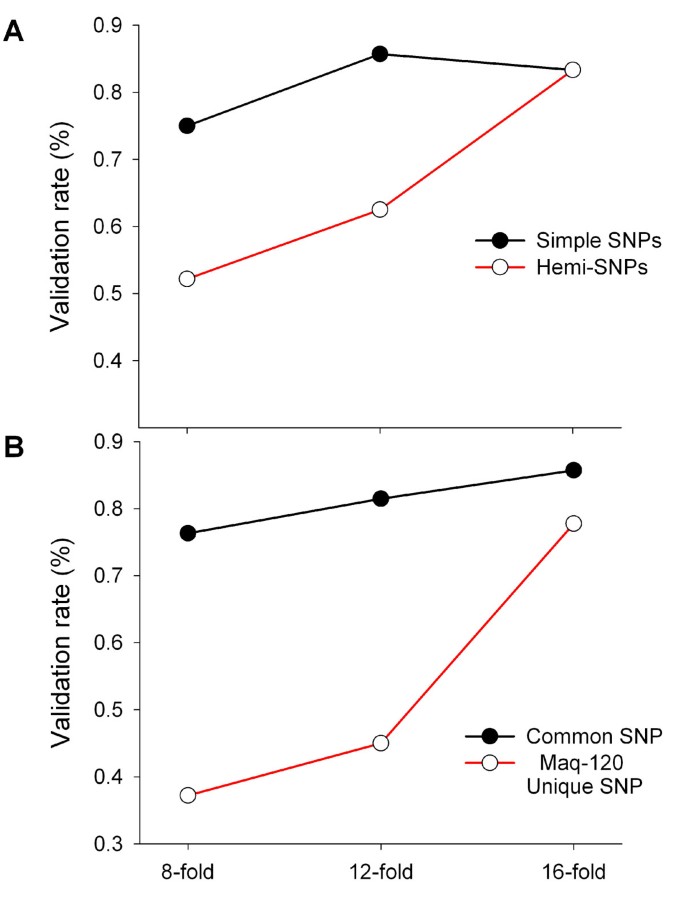
我们的结果证实,8倍的覆盖率不足以解释大样本内的抽样变化。所检查的Maq-120 snp的组成表明,在8倍覆盖率下,半snp的验证率低于简单snp。从上面的讨论中可以预料到这一点,因为简单的snp可以被认为是更符合兰德-沃特曼模型的单拷贝基因。大多数假定的半单态snp后来被证实是单态的,在大量比对中似乎只有一个同源转录本,这是由reads中缺乏相连的IHPs决定的。随着覆盖率的增加,验证半单核苷酸多态性的百分比从52%-83%增加,而简单单核苷酸多态性的百分比从75%-83%略微增加(图7一个).对Maq-120唯一snp的类似分析显示,当考虑到那些覆盖至少16倍的snp时,验证率增加到78%7 b).附加文件中提供了每个步骤中丢失的snp的详细分解,以及可能原因的摘要1:表S1。综上所述,这些结果表明,每个二倍体基因组最少覆盖8倍是通过减少采样误差来提高验证率的有效方法。
新型基因分型技术的发展极大地促进了SNP验证,这些技术灵活,可以以相对较低的成本进行快速分析设计。我们最初使用SSCP方法验证了snp,因为它可以从PCR反应中解析共扩增同源物的多态性。这避开了同源特异性分析的设计,这通常是大多数基因分型平台所需要的。尽管这种方法被证明是可靠的,但它有局限性,因为假定的SNP不能直接测定,而且它是一种过夜的基于凝胶的系统,不具有高通量(每个凝胶大约60个样品)。因此,对于Maq-120假定的snp,我们使用了KASPar平台,这是一种基于PCR的系统,只需要3分钟的终点荧光分析,比SSCP具有更高的通量。KASPar允许直接检测假定的SNP,因为两个替代碱基位于两个相互竞争的PCR引物的3'端(附加文件)1:图S5及S6)。这两种方法都需要对序列进行初步注释,因为内含子位置在RNAseq数据中是匿名的,必须考虑在内;但现在这只是公开的5×中国春小麦基因组的一小步。重要的是,KASPar已被证明在多倍体小麦中非常灵活和健壮,并迅速成为小麦研究中的重要标记系统[42和育种计划(S. Dreisigacker, CIMMYT,个人交流)。
BSA和映射
BSA的分辨率由标记密度和每个块内的组合重组给出。在小麦中,标记密度是可变的,取决于亲本系的亲缘关系、使用的标记系统、检测的个体数量和正在研究的基因组(D基因组的多态性最少)。撇开这些因素不谈,大多数小麦基因图谱的平均分辨率最多为5-10厘米。这包括使用SSR和DArT标记组合的硬粒小麦和二聚体小麦之间的图谱(标记之间平均7.5 cM [43]),以及最近公布的两种英国精英系的图谱,其中包括SSR、DArT和超过500个KASPar标记(标记之间平均4.7 cM) [42])。与测序基因组相比,这些相对较低的标记密度限制了BSA的定位分辨率。我们相信,随着NGS方法的出现,这种情况已经发生了改变,NGS方法允许在多倍体小麦中鉴定和评估前所未有的数量的snp。这表明小麦中BSA的分辨率将依赖于用于降低复杂性的方法(如上所述)和每个测序块中的重组。
第一个NGS研究在拟南芥从500华氏度的大体积中重新测序DNA2来绘制特定的EMS突变[18].最近的方法显示了50华氏度的一致结果2通过利用大量的SNPs来获取个体[19].这两种方法都利用了基因的物理图谱和已知的基因含量拟南芥,专注于特定的ems诱导的转变,导致蛋白质中的氨基酸变化,并受益于低突变密度拟南芥(每100-200 kb约1个EMS突变,或约1-2 cM) [17].这意味着拟南芥,将一个突变映射到300-400 kb以内就足以作为EMS突变对应的候选snp的数量而且导致氨基酸变化应该很小。在玉米中,使用BSA和定量基因分型(Sequenom)方法已经绘制了几种隐性突变表型[44].在这项研究中,使用至少20f的大块进行了全基因组扫描2个体,并实现了几个cM的映射间隔。
在我们的研究中,我们使用了由看似少量个体组成的群体;14和13纯合子F3.每批重组线。这些线条来自于~ 4500华氏度的大屏幕2根据12.2 cM区间内重组的存在来选择GPC-B1以前映射过。因此,虽然与其他研究相比,个体的绝对数量较低,但在重组方面的信息量是很高的。考虑到所有27个个体在12.2 cM区间内的重组概率相等,我们预计可以实现0.45 cM的映射分辨率。
在小麦中,每个位置克隆项目都需要一个定制的物理映射,使得快速达到亚cm间隔的能力对成功至关重要。根据以往的经验,一个BAC克隆大约相当于0.1 cM,因此在开始物理图之前通常需要较小的遗传距离(0.3-0.5 cM),以减少染色体行走步数。使用基于共线性的个体标记开发的传统映射方法,我们之前已经进行了映射GPC-B1到由水稻基因Os02g04520和Os02g04630定义的0.3 cM区间(附加文件)1:表S2) [11,14].在这项研究中,我们绘制了地图GPC-B1到Os02g04490和Os02g04650定义的遗传间隔为0.4 cM。虽然我们没有实现单bac分辨率,但考虑到近端侧翼标记Ta#S17984935与Os02g04650具有同源性,而Os02g04640距离为克隆而开发的物理图谱的末端(Os02g04640)只有一个基因GPC-B1远端标记Ta#S37941845与物理图谱远端Os02g04550仅5个基因的Os02g04490同源。
这种非常窄的映射间隔是在几个月内实现的,而不是几年,尽管重要的是要考虑到我们受益于高质量的表型数据和种质资源来组装批量。新的基因分型和测序技术将减少许多步骤所需的时间和精力,如筛选重组植物,识别多态标记,以及从散装中对个体进行基因分型。然而,植物的可靠表型仍然是任何BSA和测绘项目成功结果的主要决定因素。话虽如此,这些新技术现在允许一个人并行地针对几个基因,并允许在没有基因组序列信息的物种中进行BSA。
未来的发展方向
我们的研究结果表明,ngs基因在多倍体物种中是可行的,并强调了小麦基因组学有待进一步改进的几个领域。重要的第一步是为小麦生产一套更全面、更明确的单基因。有几项正在进行的努力应该协调起来,为所有小麦研究人员开发一个公开可用的基因集。我们希望能够获得这三个小麦基因组的IHPs目录,并根据二倍体祖细胞初步分配SNPs。这种策略以前已经被成功地使用过了。45]和公开的资料Ae。tauschii原始序列[46]是研究D基因组的一个很好的起点。综合基因集与IHPs的结合将通过最初掩盖IHPs,然后使用它们来分配基因组,从而使品种SNP检测管道更加有效。通过将IHPs与品种snp连接起来,应该有可能实现同源特异性引物的自动化设计。灵活的SNP检测平台,如KASPar,将使这种方法更加强大,并可用于大量研究小组。最后一个重要的因素是释放单个小麦染色体臂的序列。目前,第1组的序列是公开的[32]和第7组[47]染色体和其余的序列目前正在生成[48].结合上述信息将产生一组完整的unigenes,每个基因组都有单倍型信息,并映射到相应的染色体臂上。这将代表小麦基因组学的一个台阶变化,并将显著增强NGS-BSA方法。
结论
在这项研究中,我们概述了一种结合RNAseq发现SNP和BSA进行多倍体小麦基因精细定位的方法。生成了一组来自双亲的经过验证的平衡的snp,并在目标区间内映射~70%。这些SNPs分布良好,可以识别几乎所有的重组事件,成功地定位了一个基因(GPC-B1)至0.4厘米的间隔。大量的SNPs还在该地区产生了高密度单倍型,这在未来的项目中将用于育种目的。
方法
植物材料和生长条件
本研究使用的亲本系为四倍体小麦品种兰登(LDN)和LDN (dc - 6b) × LDN的重组替代系(RSL65) [4].用于大体和精细定位的纯合子重组系要么是RSL65的姐妹系(来自同一个组合,鉴定号小于100),要么是LDN × RSL65的组合(鉴定号大于100)。它们的基因型和籽粒蛋白浓度表型以前已被描述过[4,5,9,14].简单地说,使用了已知高蛋白表型的14个RSLs(8、28、50、54、58、65、117、121、147、152、158、241、259、300)和低蛋白表型的14个RSLs(14、19、59、77、78、129、135、148、209、289、290、293、215、3417)。这些细胞系在~ 12cm区间内以纯合子状态进行重组,其中包括GPC-B1并且由标记分隔Xwms508而且Xwms193.
为了尽量减少具有相反表型的植物之间的生长条件差异,对高蛋白和低蛋白RSLs一起生长在2 L花盆中,并适当标记。每对高低配对培养4个生物重复,但根据高低RSLs的目视比较,只选择一个盆栽进行采样。五人中的前三分之一th收集叶子用于DNA提取,而收集底部三分之一用于RNA提取(中间的三分之一保留作为备份)。
样品和RNA体的制备
个别样本的DNA已按先前所述的方法制备[49并分析了他们的基因型GPC-B1使用标记的间隔Xuhw89(远端),Xucw71(近端)和Xucw101(因果SNP为GPC-B1)使用已公布的条件[11,14].总RNA是通过研磨5个的底部三分之一来制备的th根据制造商的协议,使用TRIzol (Invitrogen)提取RNA。用NanoDrop ND-1000分光光度计测定RNA浓度,每个RNA样品取1 μL。RNA质量通过在Agilent RNA 6000 n LabChip (Agilent Technology 2100 Bioanalyzer)上运行1 μL来评估。根据Illumina mRNA-Seq协议,RNA完整性数(RIN)值大于8的样品被认为是可接受的。从先前被归类为高蛋白的14个个体中提取等量的RNA,混合产生高蛋白RNA。低蛋白RNA体使用上述RSLs构建,rsl135在DNA标记分析中被发现为杂合,因此被排除。为了在侧翼位点保持等位基因的平衡,我们将来自RSLs 77和78的RNA数量增加了一倍到低蛋白体中,因此其中包括来自15个RSLs(13种不同的基因型)的RNA。
Illumina库生产
Illumina mRNA-Seq 8-Sample试剂盒(RS-100-0801, Illumina Inc.)根据制造商的协议进行了以下修改。简而言之,使用聚t寡聚附着磁珠从5 ug总RNA中纯化出含有聚a的mRNA分子。通过添加5×片段缓冲液(Illumina, Hayward, CA)将纯化的mRNA片段化,并在94°C的热循环器中加热2个不同的时间(2 min和5 min)。5分钟的分片时间是协议中使用的标准时间,它产生~250 bp的片段。较短的破碎时间可以产生略大的350-400 bp的片段库。使用随机引物合成第一链cDNA,以消除转录本3'端的一般偏向。通过添加GEX第二链缓冲液(Illumina, Hayward, CA)、dNTPs、RNaseH和DNA聚合酶I,在16°C下孵育2.5 h,完成第二链cDNA合成。第二链cDNA进一步按照制造商提供的方案进行末端修复、a尾拖尾和适配器结扎。使用PE1.0和PE2.0引物和Phusion DNA聚合酶(Illumina, Hayward, CA),在98℃、65℃和72℃分别进行15次10 s、30 s PCR富集纯化的cDNA模板。使用QIAquick PCR纯化柱清洗样品,并按照制造商说明书(QIAGEN, CA)用30 μl EB(洗脱缓冲液)洗脱。 Purified cDNA libraries were quantified using Bioanalyzer DNA 100 Chip (Agilent Technology 2100 Bioanalyzer).
Illumina库聚类和测序条件
EB (Qiagen)的亲本文库归一化至7.5 nM。将样品用NaOH (4 μL 10 nM原液,1 μL 2 N NaOH和15 μL EB)稀释至1.5 nM,室温下静置2 min,然后将4 μL转移到496 μL HT1(高盐缓冲液,由群集套件成对端群集生成kit V4 PE-203-4001, Illumina提供)中,使最终浓度为12 pM。每个样品库在EB中归一化至10 nM, NaOH稀释至2 nM, 2.5 μL转入497.5 μL HT1中,最终浓度为10 pM。将120 μL的归一化文库转入200 μL的条形管中,放在冰上,然后装载到集群站,每个文库在单车道上运行。按照Illumina PE_amplification_Linearization_Blocking_PrimerHyb_v7配方,使用成对端聚簇生成试剂盒V4对流细胞进行聚簇。聚类过程完成后,按照制造商的说明将流细胞装载到Illumina基因组分析仪GAIIx仪器上。使用的测序化学试剂为v4 (FC-104-4001, Illumina),使用软件为SCS 2.6和RTA 1.6。每个父库在单个车道上运行120个循环,每个配对的端点运行80个循环。使用GERALD管道处理Illumina基调用文件,以产生成对的序列文件,其中包含Illumina FASTQ格式的每个样本的读取。
计算方法
在第一次将Illumina FASTQ文件转换为Sanger FASTQ格式后,使用Maq v0.7.1对来自单车道的配对读取进行初始对齐[50]对照包含40,349个单基因序列共31,671,110个碱基的小麦转录组参考(NCBI TA build 57) [25].在第一个实验中,使用了Maq默认参数。在随后的实验中(文中称为Maq-120),在工作流的每个步骤中,错配碱基的质量总和得分的最大值被设置为120(默认为70)。这使得发生在120个碱基读取内的SNP单倍型与更高的映射质量相匹配。还对未能映射到NCBI Unigene参考的reads的组成部分进行了Maq-120比对,对照由小麦中国春季基因组454 reads的5×组装构建的新参考(M. Bevan, JIC, personal communication)。对于大样本,分别由两个库大小分数构建的地图在进一步处理之前被合并。用以前为多倍体油菜开发的方法检测和评分SNPs芸苔属植物显著[21,28].至关重要的是,人们预期同源基因的读值将被映射到相同的单基因参考。简单地说,使用Maq分别调用关于每个父行引用的snp,然后调用SNP_parser.pl Perl脚本(附加文件5)来推导两个集合的对称差(A Δ B)。然后,通过访问由Maq比对生成的详细堆积文件,以编程方式比较和重新评估SNP位置上的基本调用和质量分数,从而在父节点之间产生筛选过的SNP集。这是在不同的最小深度阈值(8倍,12倍和16倍)下完成的。亲本和散装样品的Illumina reads保存在EMBL-EBI Sequence Read Archive (ERA050658)中。
一个新的Perl脚本bulk_frequencies.pl(附加文件6),用于分析两个体块之间亲本snp的等位基因频率。这使用了索引方法[28]用于快速访问堆积文件中的个别行,以提取每个SNP位置的基本呼叫和质量分数。对于每个半snp,例如来自亲本LDN的Y(即C/T)和来自亲本RSL65的C,计算每个批量的信息基的频率(在本例中为T),然后确定该频率在批量(BFR)之间的比率。我们的期望是,根据亲本来源的不同,与性状/基因相一致的半单核苷酸多态性(semi - snp)应该通过在两个批量中趋于0.5或0的信息性碱基频率来揭示。在排除除零误差后,以BFR≥3.0作为阈值对snp进行过滤。简单的snp以类似的方式处理。
显示明显SNP密度大于5 SNPs/kb的Unigenes被认为是人工的(或副同源的),并被排除在分析之外(LDN和RSL65之间的SNP密度经实验确定为2.2 SNPs/kb)。BFR结果以电子表格格式组织,以帮助进一步检查和排序。添加每个单基因的辅助同向性数据,包括预先计算的BLASTN分析对单基因的最佳命中Brachypodium水稻和高粱基因模型(e值截断1E-50),以及转录丰度测量,表示为每千碱基每百万映射reads (RPKM值)。
标记物设计和SNP检测
为了设计针对假定SNP的标记,从单基因中提取候选SNP左右两侧250 bp的区域。利用BLASTN分析,对中国春小麦品种的5×基因组序列(454个原始reads,未组装)进行外显子-内含子位置注释[30.].在1E-50位点上包含超过100个假定snp的序列被认为是重复的,不再进一步处理。基于PCR产物的单链构象多态性(single strand conformation polymorphism, SSCP),对Maq-default分析中识别的snp设计引物扩增~150-200 bp片段。Maq-120分析中的第二组snp使用类似的方法进行注释,但引物被设计用于扩增KASPar分析的产物[42,51)。PCR条件和SSCP分析使用已发表的协议进行[49].KASPar寡核苷酸从Sigma-Aldrich订购,引物带有标准的FAM或VIC兼容尾(FAM尾:5' GAAGGTGACCAAGTTCATGCT 3';VIC尾巴:5' GAAGGTCGGAGTCAACGGATT 3'),目标SNP在3'端。引物混合物按照Kbioscience推荐(46 μl dH2O,普通底漆30 μl (100 μM),尾底漆各12 μl (100 μM)) [51].采用384孔法,反应量为5 μl (2.5 μl模板[10-20 ng DNA], 2.43 μl V3 2xKaspar mix, 0.07 μl引物mix)。PCR在Peltier PTC-225 PCR四分体机上进行,该四分体机回顾性安装了384个片段,采用以下方案:在95°C下热启动15分钟,然后进行10个触地循环(95°C 20秒;触点65°C, -1°C每周期,25 s),然后随后进行26个周期扩增(95°C 10 s;57°C 60 s)。由于KASPar扩增子通常小于120 bp,因此在PCR方案中不需要扩展步骤。384孔样品板(猫。编号04729749001,Roche Diagnostics)在Roche Lightcycler上读取®i480qpcr仪。在环境温度(20-25°C;RAMP速度0.05°C / s),每°C有四个检测步骤。如果在初始扩增后尚未形成特征基因分型组,则应用额外的扩增循环(通常为5-10),并再次读取样本。使用内置的Roche Lightcycler手动进行数据分析®480软件(版本1.50.39)。提供了引物的完整列表(附加文件7).
加入代码
本文报告的短读序列数据已存放在序列读存档(SRA)中,登录代码为ERA050658。
缩写
- BAC:
-
细菌人工染色体
- BFR:
-
总体频率比
- BSA:
-
散装隔离分析
- cM:
-
centi-Morgan
- 省:
-
分集阵列技术
- DIC6B:
-
小麦属植物turgidumssp。dicoccoides6B染色体替代系
- 背景:
-
脱氧核糖核酸
- 美国东部时间:
-
表达序列标签
- gDNA:
-
基因组DNA
- GPC-B1:
-
籽粒蛋白质含量基因
- IHP:
-
Inter-homoeologue多态性
- 卡斯帕·:
-
竞争性等位基因特异性PCR基因分型系统
- LDN:
-
兰登
- NCBI:
-
国家生物技术信息中心
- 门店:
-
新一代测序
- 聚合酶链反应:
-
聚合酶链反应
- RNA:
-
核糖核酸
- RNAseq:
-
信使rna序列
- RPKM:
-
每千碱基每百万映射读取数
- RSL:
-
重组替代系
- SHOREmap:
-
短读映射器
- SNP:
-
单核苷酸多态性
- SSCP:
-
单链构象多态性
- 苏维埃社会主义共和国:
-
简单的序列重复。
参考文献
- 1.
粮农组织2005-2006年统计年鉴。[http://www.fao.org/es/ess/yearbook/)网络
- 2.
被子植物DNA c值数据库。[http://www.rbgkew.org.uk/cval/homepage.html]
- 3.
flaverl RB, Bennett MD, Smith JB, Smith DB:植物基因组大小和重复核苷酸序列DNA的比例。生物化学学报,1994,12(4):257-269。10.1007 / BF00485947。
- 4.
乔帕LR,杜C, Hart GE, Hareland GA:四倍体小麦籽粒蛋白定位基因(小麦属植物turgiduml)利用重组自交系染色体系的群体。作物科学,1997,37(5):1586-1589。10.2135 / cropsci1997.0011183X003700050030x。
- 5.
Olmos S, Distelfeld A, Chicaiza O, schlatar, Fahima T, Echenique V, Dubcovsky J:影响硬粒小麦籽粒蛋白质含量的基因座的精确定位。应用物理学报,2003,27(3):344 - 344。10.1007 / s00122 - 003 - 1377 - y。
- 6.
国际短肢动物计划:模式草的基因组测序和分析Brachypodium distachyon.自然科学学报,2010,38(4):344 - 344。10.1038 / nature08747。
- 7.
于俊,胡松,王娟,王刚,李松,刘波,邓勇,戴林,周勇,张旭,等:水稻基因组草图序列(栽培稻l . ssp。籼稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068037。
- 8.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A,等高粱二色的基因组与草的多样化。自然科学进展,2009,29(1):1-5。10.1038 / nature07723。
- 9.
Distelfeld A, Uauy C, Olmos S, schlate A, Dubcovsky J, Fahima T:围绕籽粒蛋白质含量位点的2 cm区域的微共线性Gpc-6B1在小麦第6B染色体和水稻第2染色体上有一个350kb的区域。中国生物医学工程学报,2004,4(1):59-66。10.1007 / s10142 - 003 - 0097 - 3。
- 10.
博索里尼E, Krattinger S, Keller B:简单序列重复标记的开发Lr34利用水稻和水稻的序列信息研究小麦的抗性区域山羊草属tauschi.应用理论,2006,26(6):1049-1062。10.1007 / s00122 - 006 - 0364 - 5。
- 11.
张晓东,王晓明,王晓明,等。小麦高粒蛋白基因的物理图谱分析Gpc-B1以及高通量分子标记的开发。植物学报,2006,39(4):353 - 353。10.1111 / j.1469-8137.2005.01627.x。
- 12.
Spielmeyer W, Singh RP, McFadden H, Wellings C, Huerta-Espino J, Kong X, Appels R, Lagudah E:利用间质缺失突变体进行精细尺度遗传和物理定位Lr34 / Yr18:对小麦多种病原体有效的抗病位点。应用物理学报,2008,44(4):481-490。10.1007 / s00122 - 007 - 0684 - 0。
- 13.
Cenci A, Chantret N, Kong X, Gu Y, Anderson OD, Fahima T, Distelfeld A, Dubcovsky J:硬粒小麦50万无性系BAC文库的构建与鉴定(小麦属植物turgidumssp。硬质).应用物理学报,2003,17(5):331 - 339。10.1007 / s00122 - 003 - 1331 - z。
- 14.
Uauy C, Distelfeld A, Fahima T, Blechl A, Dubcovsky J:一个调控衰老的NAC基因可以提高小麦籽粒蛋白质、锌和铁含量。科学通报,2006,29(3):344 - 344。10.1126 / science.1133649。
- 15.
Krattinger SG, Lagudah ES, Spielmeyer W, Singh RP, Huerta-Espino J, McFadden H, Bossolini E, Selter LL, Keller B:一种假定的ABC转运蛋白赋予小麦对多种真菌病原体的持久抗性。科学通报,2009,29(5):528 - 528。10.1126 / science.1166453。
- 16.
黄志刚,刘志刚,刘志刚。小麦和大麦基因克隆的研究进展。小麦科的遗传学与基因组学。编辑:Muehlbauer GJ, Feuillet C.纽约:施普林格;2009:337 - 357。
- 17.
Schneeberger K, Weigel D:新测序技术使遗传学快速发展。植物科学进展,2011,16(5):282-288。10.1016 / j.tplants.2011.02.006。
- 18.
Schneeberger K, Ossowski S, Lanz C, Juul T, Petersen AH, Nielsen KL, Jorgensen J-E, Weigel D, Andersen SU: SHOREmap:深度测序同时作图和突变鉴定。光子学报,2009,6(8):550-551。10.1038 / nmeth0809 - 550。
- 19.
张杰,冯鹏,龚颖,王培文,等:基于gis的新一代测绘方法拟南芥基因。植物学报,2011,27(4):715-725。10.1111 / j.1365 - 313 x.2011.04619.x。
- 20.
Mokry M, Nijman I, Van Dijken A, Benjamins R, Heidstra R, Scheres B, Cuppen E:分生组织功能因子的鉴定拟南芥使用一种新型的下一代测序快速遗传学方法。BMC基因组学杂志,2011,12(1):256-10.1186/1471-2164-12-256。
- 21.
崔克M,龙Y,孟J, Bancroft I:多倍体中单核苷酸多态性(SNP)的发现芸苔属植物显著使用Solexa转录组测序。植物学报,2009,7(4):334-346。10.1111 / j.1467-7652.2008.00396.x。
- 22.
Oliver R, Lazo G, Lutz J, Rubenfield M, Tinker N, Anderson J, Wisniewski-Morehead N, Adhikary D, Jellen E, Maughan PJ,等:基于高通量454测序技术的燕麦复杂基因组SNP构建模型。BMC基因组学杂志,2011,12(1):77-10.1186/1471-2164-12-77。
- 23.
Trebbi D, Maccaferri M, De Heer P, Sørensen A, Giuliani S, Salvi S, Sanguineti M, Massi A, Van der-Vossen E, Tuberosa R:硬粒小麦高通量SNP的发现与基因分型(小麦属植物硬质Desf)。应用物理学报,2011,23(4):555-569。10.1007 / s00122 - 011 - 1607 - 7。
- 24.
Michelmore R, Paran I, Kesseli R:通过散装分离分析鉴定与抗病基因相关的标记:一种通过分离群体检测特定基因组区域标记的快速方法。美国国家科学研究院。1991, 88(21): 9828-9832。10.1073 / pnas.88.21.9828。
- 25.
小麦: UniGene Build #57。[http://www.ncbi.nlm.nih.gov/UniGene/UGOrg.cgi?TAXID=4565]
- 26.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B:利用RNA-Seq绘制和量化哺乳动物转录组。光子学报,2008,5(7):621-628。10.1038 / nmeth.1226。
- 27.
Dvorak J, Akhunov ED, Akhunov AR, Deal KR, Luo M-C:驯化四倍体小麦诊断DNA标记的分子特征为野生四倍体小麦向六倍体小麦的基因流动提供了证据。分子生物学杂志,2006,23(7):1386-1396。10.1093 / molbev / msl004。
- 28.
Bancroft I, Morgan C, Fraser F, Higgins J, Wells R, Clissold L, Baker D, Long Y,孟J,王霞,等:多倍体油菜基因组转录组测序分析。生物技术学报,2011,29(8):762-766。10.1038 / nbt.1926。
- 29.
李红,阮娟,杜斌R:基于质量评分的DNA测序短序列测序。基因组学报,2008,18(11):1851-1858。10.1101 / gr.078212.108。
- 30.
中国春季序列。[http://www.cerealsdb.uk.net/]
- 31.
Rustenholz C, Hedley P, Morris J, Choulet F, Feuillet C, Waugh R, Paux E:利用大麦和小麦基因组资源组合揭示小麦基因空间组织的特定模式。BMC基因组学杂志,2010,11(1):714-10.1186/1471-2164-11-714。
- 32.
Wicker T, Mayer KFX, Gundlach H, Martis M, Steuernagel B, Scholz U, Šimková H, Kubaláková M, Choulet F, Taudien S,等:频繁的基因移动和假基因进化是小麦、大麦及其近缘种大而复杂的基因组的共同特征。植物细胞学报,2011,23(5):1706-1718。10.1105 / tpc.111.086629。
- 33.
masa AN, Wanjugi H, Deal KR, O'Brien K, You FM, Maiti R, Chan AP, Gu YQ, Luo MC, Anderson OD,等:植物进化过程中的基因空间动力学陶氏蛇,短柄草,水稻,高粱二色的基因组。分子生物学与进化,2011,28(9):2537-2547。10.1093 / molbev / msr080。
- 34.
Mayer KFX, Martis M, Hedley PE, Šimková H, Liu H, Morris JA, Steuernagel B, Taudien S, Roessner S, Gundlach H,等:通过染色体和比较基因组学解锁大麦基因组。植物细胞学报,2011,23(4):1249-1263。10.1105 / tpc.110.082537。
- 35.
Cantu D, Pearce S, Distelfeld A, Christiansen M, Uauy C, Akhunov E, Fahima T, Dubcovsky J:籽粒蛋白含量下调的效应(GPC)基因在小麦单果衰老过程中的转录组。BMC基因组学杂志,2011,12(1):492-10.1186/1471-2164-12-492。
- 36.
Uauy C, Brevis JC, Dubcovsky J:高粒蛋白含量基因Gpc-B1加速衰老,对小麦蛋白质含量有多效性影响。实验学报,2006,57(11):2785-2794。10.1093 / jxb / erl047。
- 37.
Hodges E, Xuan Z, Balija V, Kramer M, Molla MN, Smith SW, Middle CM, Rodesch MJ, Albert TJ, Hannon GJ,等:全基因组原位外显子捕获选择性重测序。植物学报,2007,39(12):1522-1527。10.1038 / ng.2007.42。
- 38.
王晓明,王晓明,王晓明,等。异体四倍体小麦基因组外显子捕获对核苷酸和拷贝数变异的影响。中国生物工程学报,2011,12:R88-10.1186/gb-2011-12-9-r88。
- 39.
王志强,王志强,王志强:RNA-Seq:转录组学的革命性工具。植物学报,2009,10(1):57-63。10.1038 / nrg2484。
- 40.
Lander ES, Waterman MS:通过指纹随机克隆的基因组定位:数学分析。基因组学杂志,1988,2(3):231-239。10.1016 / 0888 - 7543(88) 90007 - 9。
- 41.
温德尔M,威尔逊R:医学DNA测序的覆盖方面。生物医学工程学报,2008,9(1):239-10.1186/1471-2105-9-239。
- 42.
Allen AM, Barker GLA, Berry ST, Coghill JA, Gwilliam R, Kirby S, Robinson P, Brenchley RC, D'Amore R, McKenzie N,等:六倍体面包小麦转录特异性单核苷酸多态性的发现和连锁分析(小麦l .)。植物学报,2011,9(9):1086-1099。10.1111 / j.1467-7652.2011.00628.x。
- 43.
Peleg Z, Saranga Y, Suprunova T, Ronin Y, Röder M, Kilian A, Korol A, Fahima T:基于SSR和DArT标记的硬粒小麦×野生二聚体小麦高密度遗传图谱。应用理论,2008,17(1):103-115。10.1007 / s00122 - 008 - 0756 - 9。
- 44.
刘s,陈hd, Makarevitch I, Shirmer R, Emrich SJ, Dietrich CR, Barbazuk WB,施普林格NM, Schnable PS:基于定量单核苷酸多态性分型的高通量突变体遗传定位。中国生物医学工程学报,2010,34(1):344 - 344。10.1534 / genetics.109.107557。
- 45.
赵山,张伟,Akhunov E, Sherman J,马勇,罗明春,Dubcovsky J:美国小麦SNP基因来源标记多态性分析(小麦l .)品种。生物育种,2009,23(1):23-33。10.1007 / s11032 - 008 - 9210 - 6。
- 46.
山羊草属tauschii基因组测序计划。[http://www.cshl.edu/genome/wheat]
- 47.
Berkman PJ, Skarshewski A, Lorenc MT, Lai K, Duran C, Ling EYS, Stiller J, Smits L, Imelfort M, Manoli S,等:分离株低拷贝区和基因区测序和组装小麦染色体臂7DS。植物生态学报,2011,29(3):344 - 344。10.1111 / j.1467-7652.2010.00587.x。
- 48.
国际小麦基因组测序联盟。[http://www.wheatgenome.org/]
- 49.
Howard T, Rejab NA, Griffiths S, Leigh F, Leverington-Waite M, Simmonds J, Uauy C, Trafford K:水稻b型淀粉粒含量控制QTL的鉴定山羊草属.实验学报,2011,62(6):2217-2228。10.1093 / jxb / erq423。
- 50.
李红,阮娟,杜斌R:基于质量评分的DNA测序短序列测序。基因组学报,2008,18(11):1851-1858。10.1101 / gr.078212.108。
- 51.
KBiosciences。[http://www.kbioscience.co.uk/]
确认
我们感谢James Simmonds和Peter Sawdon在植物管理方面的帮助,Jonathan Jones和Simon Griffiths在这项工作中进行了有益的讨论,Jorge Dubcovsky和Francine Paraiso慷慨地提供了亲本和重组系的种子。我们也感谢基因组分析中心生成Illumina序列数据。本研究由英国生物技术和生物科学研究委员会(BBSRC) (BB/H018824/1)和John Innes基金会(NMA博士奖学金)资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
MT和CU构想并设计了实验;MF进行文库构建,NMA、SGM和CCJ进行SNP验证和作图实验;MT和CU进行数据分析;MT进行生物信息分析;MT、NMA、CCJ、CU共同起草并修改稿件。所有作者都阅读并批准了手稿的最终版本。
电子辅助材料
图S1:
附加文件1:概述NGS-BSA方法主要步骤的流程图。图S2:ta# S32574498的LDN和RSL65色谱图。图S3:经过验证和映射的snp的BFRGPC-B1时间间隔。图S4:具有推测SNP的小麦unigenes的图谱Brachypodium基因组。图S5:ta# S37941845的MAQ比对图显示,在582位置,LDN和RSL65包含了半snp (R = A/G)。图S6:两个半单核苷酸多态性的荧光输出可视化表S1:在验证和映射过程中丢失的snp的分解表S2:本研究中最密切识别的标记与以前用于物理地图构建的标记之间存在共线性。(pdf 683kb)
12870 _2011_980_moesm2_esm.xlsx
附加文件2:通过Maq-default分析在大量样品中识别的所有假定snp的特征。(xlsx504kb)
12870 _2011_980_moesm3_esm.xlsx
附加文件3:通过Maq-120分析在大量样品中鉴定的所有假定snp的特征。(xlsx926kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
特里克,M.,亚当斯基,n.m.,马格福德,S.G.et al。结合从下一代测序数据中发现的SNP与散装分离分析(BSA)来精细定位多倍体小麦中的基因。BMC植物生物学12日,14(2012)。https://doi.org/10.1186/1471-2229-12-14
收到了:
接受:
发表:
关键字
- 单核苷酸多态性
- 单核苷酸多态性标记
- 单链构象多态性
- 大宗分离分析
- 籽粒蛋白质含量