摘要
背景
组蛋白乙酰转移酶(Histone acetyltransferases, HATs)在真核生物转录中起着重要的作用。大米中的8种hat (OsHATs)可分为4个科,即CBP (OsHAC701、OsHAC703和OsHAC704)、TAF2250 (OsHAF701)、GNAT (OsHAG702、OsHAG703、OsHAG704)和MYST (oshag701)家族。由于HAT在水稻中的生物学功能尚不清楚,因此我们对HAT家族进行了全面的蛋白质序列分析,以研究其潜在的功能。此外,还分析了8种oshat的亚细胞定位和表达模式。
结果
在系统发育和结构域分析的基础上,单子叶CBP家族蛋白可细分为两类,即组I和组II。同样,双子叶植物CBP家族蛋白也可分为A组和b组,oshat及其同源物的蛋白序列、保守结构域和三维模型相似性较高拟南芥和玉米。亚细胞定位预测表明,所有OSHATS都可能定位在细胞核和细胞溶胶中。瞬态表达拟南芥原生质体证实了oshaac701、OsHAG702和OsHAG704的核和胞质定位。实时定量聚合酶链反应分析表明OsHATs在所有组织中均有表达,转录丰度差异显著,它们的表达受脱落酸、水杨酸以及盐、冷、热胁迫等非生物因素的调节。
结论
单子叶和双子叶CBP家族蛋白可分为两个不同的类群,这表明CBP家族蛋白存在功能多样化的可能性。oshat及其同源物的蛋白质序列、保守结构域和三维模型高度相似拟南芥和玉米表明职业健康辅助测试具有多种功能。在瞬时表达分析中,oshaac701、OsHAG702和OsHAG704均定位于细胞核和细胞质拟南芥原生质体。OsHATs在水稻中表达,它们的表达受到外源激素和非生物胁迫的调节,这表明OSHATS可能在植物防御反应中发挥重要作用。
背景
组蛋白修饰在基因表达调控中起着关键作用[1].组蛋白乙酰转移酶(HATs)的乙酰化通常与基因活性增加相关,而组蛋白去乙酰化酶(HDACs)的去乙酰化通常与基因抑制相关[2那3.].在真核生物中,组蛋白乙酰化由5个不同的HAT家族催化,包括p300/CREB (camp响应元件结合蛋白)-结合蛋白(CBP)家族,tata结合蛋白相关因子(TAF)2250家族,一般控制不可抑制的5相关N-末端乙酰转移酶(GNAT)家族,MOZ,YBF2 / SAS3,SAS2和TIP60(MYST)家族,以及核激素相关的帽子家庭[4.那5.].生物信息学分析表明目前有12个推定帽子拟南芥,这些蛋白质属于CBP系列(HAC1 / PCAT2,HAC2 / PCAT1,HAC4 / PCAT3,HAC5 / PCAT4和HAC12),TAF2与250相关的家庭(HAF1和HAF2 / TAF1),GNAT系列(HAG1 / GCN5,HAG2和HAG3 / ELP3)和MYST家族(HAM1 / HAG4和HAM2 / HAG5)[5.那6.].
在拟南芥,越来越多的证据显示净化海港有助植物多方面的生长和发育,包括根系的发育[6.那7.],花艺发展[6.那8.那9.],配子化学发育[10,以及器官生长过程中的细胞增殖[11].此外,hat的组蛋白乙酰化对植物适应环境变化(如光信号转导)也很重要[12-15,盐压力[16,冷压力[17-19]、热压力[20.]、脱落酸(ABA)信号传导[16那21那22和其他激素信号[23].
水稻是一种重要的经济作物,也是单子叶基因组学和分子生物学研究的模式植物。在水稻(oshat)中鉴定出8种hat蛋白,这些蛋白可分为4个主要家族,即CBP (OsHAC701、OsHAC703和OsHAC704)、TAF2250 (OsHAF701)、GNAT (OsHAG702、OsHAG703、OsHAG704)和MYST (oshag701)家族。GNAT家族又可细分为三个亚家族,分别是GCN5、ELP3和HAT1 [5.].OsHAG702、OsHAG703和OsHAG704分别属于GCN5、ELP3和HAT1亚家族[24].先前已对hat进行了一些系统发育分析[5.那10那24那25].然而,CBP和TAF的演化关系2250个家庭仍不清楚。此外,oshat在水稻中的生物学功能尚未被阐明。
为了研究oshat的潜在功能,我们进行了系统的生物信息学和表达分析。CBP和TAF的系统发育树2为了探讨单子叶植物(单子叶植物)、双子叶植物(双子叶植物)、苔藓植物、蕨类植物、动物和真菌代表物种之间的进化关系,共划分了250个科。通过多序列比对和结构域分析预测oshat与其他生物的hat的特异功能。我们还生成了hat与SWISS-MODEL的三维(3D)比较蛋白质结构模型[26-28].此外,通过蛋白序列分析预测了8种oshat的亚细胞定位。oshaac701、OsHAG702、OsHAG704在拟南芥利用原生质体进行亚细胞定位。最后,表达模式OsHATs采用实时定量聚合酶链反应(RT-qPCR)分析。研究结果将对阐明水稻中不同脱氢酶的功能有重要的贡献。
方法
HAT cDNA和蛋白序列的搜索
我们在NCBI (http://www.ncbi.nlm.nih.gov/),chromdb(http://www.chromdb.org/), UniProt (http://www.uniprot.org/)和kome(http://cdna01.dna.affrc.go.jp/cdna/)数据库。HAT cDNA序列和蛋白序列从ChromDB (http://www.chromdb.org/).序列表示单子叶植物奥雅萨苜蓿(japonica.品种组,OS),奥雅萨苜蓿(籼稻品种群,Osi),Zea Mays.Zm评选,高粱双色(某人),小麦(Ta);双子叶植物的拟南芥(在),杨树trichocarpa(Pt)大豆(GM);苔藓Physcomitrella金属盘(pp)和蕨类植物Selaginella Moellendorffii.(Sm);这些动物秀丽隐杆线虫(Ce),黑腹果蝇(Dm),智人(Hs);和真菌酿酒酵母(Sc)和Schizosaccharomyces Pombe(Sp)。所有HAT蛋白都遵循ChromDB命名法。利用计算机pI/Mw在线工具计算水稻HAT蛋白的理论等电点(pI)和分子量(Mw)http://web.expasy.org/compute_pi/)(表1).
系统发育分析
蛋白质序列与Clustalx 2.1对齐[29].通过使用系统发育推理包(Phylip)3.6版本3.6的引导分析,使用邻近加入方法产生邻近的径向树,与1000次重复的引导分析产生.3.6 [30.].蛋白质进化变化的Dayhoff PAM模型[31]用于使用PROTDIST程序度量树的分支长度(进化时间)。使用TreeView 1.6.6版本显示和编辑系统发生树[32].
序列分析和对齐
利用ClustalW2在线工具(http://www.ebi.ac.uk/tools/msa/clustalw2/).将来自水稻和其他生物的所有帽子蛋白的蛋白质结构域和功能位点分配给术语区域与Interprocan [33]使用瑞士模型工作区网站[26-28)(http://swissmodel.expasy.org/workspace/index.php?func=tools_sequencescan1).Uniprotkb / trembl(http://www.uniprot.org/blast/)用于进一步探索氨基酸序列中的保守结构域和组成偏差[34].使用DOG 1.0绘制蛋白质结构域[35.].Uniprot Blast(http://www.uniprot.org/blast/),用以确定来自其他生物体的职业健康安全及HAT蛋白的身份[34].HATs的三维比较蛋白结构模型采用SWISS-MODEL自动建模模式生成[26-28]已于SWISS-MODEL Workspace网站(http://swissmodel.expasy.org/).蛋白质结构用颜色编码。
亚细胞定位预测
SLP-Local (http://sunflower.kuicr.kyoto-u.ac.jp/~smatsuda/slplocal.html.)基于氨基酸序列的局部特征是一种亚细胞位置预测因子[36.那37.].TargetP版本1.1(http://www.cbs.dtu.dk/services/TargetP/)预测来自Chlorop和SignalP的输出的真核蛋白的亚细胞定位[38.].蛋白质亚细胞定位预测因子WoLF PSORT (http://wolfpsort.org/)[39.]基于psortii和ipsort。通过传统的Psortii预测歧视核定位信号(NLS)[40]来自WOLF Psort的详细结果。Netnes 1.1服务器(http://www.cbs.dtu.dk/services/NetNES/),结合神经网络和隐马尔可夫模型预测oshat中富亮氨酸核输出信号(NES) [41.].
植物材料和生长条件
拟南芥品种哥伦比亚-0(COL-0)用于原生质体分离和瞬时表达分析。植物在控制环境室中的土壤中生长,长光周周期(16小时光/ 8小时暗),在22±2℃下。
大米(奥雅萨苜蓿l .无性系种群。japonica.简历。日本Nipponbare)种子在37±1℃的黑暗中用水浸泡24小时,然后放在滤纸上(VWR International, Mississauga, on, Canada),在培养皿中23±1℃的黑暗中用水湿润。发芽后,水稻幼苗在盛有水的烧杯中生长,在培养室内进行光照9 h、黑暗15 h的日光周期培养。培养温度23±1℃。
对于ABA (Sigma, Oakville, ON, Canada),水杨酸(SA;在高盐处理下,将生长在烧杯中的二叶期幼苗转移到添加或不添加ABA (100 μM)、SA (100 μM)或NaCl (300 mM)的水中。冷处理时,将水稻幼苗置于4±1°C黑暗中培养3 h。热处理时,将幼苗置于42℃黑暗培养箱中培养3 h。幼苗在黑暗中保持在23±1°C的水中作为对照。双叶期水稻幼苗叶片处理后采收,液氮冷冻,−80℃保存。
原生质体的分离和瞬时表达
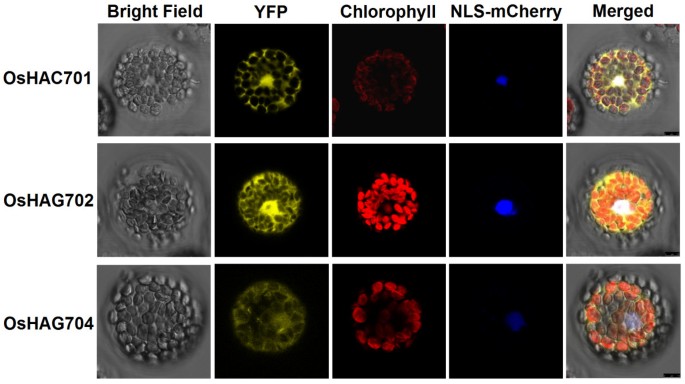
全长CDNAOsHAC701被亚克隆到p2ygw7.0载体中[42.]构建YFP-OsHAC701构建,而YFP-OsHAC701的全长cdnaOsHAG702和OsHAG704亚克隆到pSAT6-EYFP-N1载体[43.生成OsHAG702-YFP和OsHAG704-YFP结构。的分离和转染拟南芥叶肉原生质体的处理如前所述[44.那45.].简单地说,原生质体是从生长3周的叶片中分离出来的拟南芥植物。OSHAC701 / HAG702 / 704-YFP融合质粒和ViRd2NLS-MCHERRY作为核标记的体积(10-20μg)[46.]共转染150 μl原生质体(3 × 104.原生质体),使用peg -钙转染溶液。原生质体在22±2℃白光下孵育过夜,允许导入基因表达。使用Leica SP5共聚焦显微镜(Leica, Wetzlar, Germany)检测和拍摄YFP荧光。
实时荧光定量PCR分析
使用改性锂氯化物沉淀法从两叶阶段的水稻幼苗的叶片中萃取总RNA [47.].使用Thermo Scientific NanoDrop测量RNA的数量和质量TM1000分光光度计(威尔明顿,美国)。在cDNA合成之前,用DNase I(新英格兰Biolabs,Cailering,Canada,Canada)用DNase I(新英格兰生物Biolabs,Capering,Canada)进行处理。用热磷值从2μgRNA合成cDNATMRT-PCR系统(Invitrogen, Burlington, ON, Canada)。用SsoFast进行RT-qPCR分析TMEvaGreen®Supermix (Bio-Rad, Mississauga, ON, Canada)。使用CFX96收集数据TMReal-Time PCR Detection System (Bio-Rad)根据制造商的说明。RT-qPCR数据表达为达到阈值荧光值(Ct)所需的周期数,并用对比Ct方法(ΔΔCt)进行分析。报告值为3个生物重复的平均值,每个生物重复由3个技术重复组成。eEF-1α(真核生物延伸因子1- α [48.])和Ubq-1(AK059011.1泛素(49.)作为内参基因,使表达数据归一化。PR10A(致病相关基因),可以通过SA或ABA诱导[3.那50.,以确定SA和ABA处理在本研究中是否有效。RT-qPCR引物如表所示2.与三份无模板对照进行比较,通过熔融曲线分析证实了引物对的特异性。从CT的标准曲线与启动模板数量的标准曲线计算PCR扩增效率。每种测定进行优化,使扩增效率在90%至110%之间,并且测定系数(R.2)大于0.980(表 2).
所有RT-QPCR数据都表示为平均值±标准误差。每个表达的统计差异OsHAT采用单因素方差分析(ANOVA)评估水稻各组织间的差异,其次为最小显著差异(LSD)和SNK事后比较。采用SPSS 13.0软件(SPSS Inc., Chicago, IL, USA)进行分析。显著性阈值定义为P. < 0.05. Student’sT.-检验评估外源性处理与对照差异的显著性。重要性在P.< 0.05或P.< 0.01。
Western印迹分析
酸溶性蛋白的提取是根据Tariq等人的描述进行的[51.和Probst等[52.].新鲜大米叶(0.3 g)在液氮中粉碎,并在2.25 ml新鲜制备的提取缓冲液中均质,使用Fisher Scientific Model 100 Sonic崩解器。离心15 min, 20000 rcf, 4℃,2次,上清保存于−80℃。用Bio-Rad蛋白测定液进行微bradford法测定蛋白浓度。蛋白提取物加入18.5 mM二硫苏糖醇中,16%十二烷基硫酸钠聚丙烯酰胺电泳凝胶分离,使用Bio-Rad半干电泳转移池(15v, 15min)转移到聚偏氟乙烯膜上。使用以下抗体:抗组蛋白H3(1:7500稀释;Cell Signaling Technology, Inc., Danvers, MA, USA), anti-acetyl-Histone H3K18(1:10 000稀释;抗乙酰组蛋白H3K9(1:1000稀释;和抗乙酰组蛋白H4K5(1:20 000稀释;EMD Millipore, Billerica, MA, USA)。 Histone H3 was applied as an equal loading control. The bound immune-complexes were detected with ECL Plus western blot detection reagents (GE healthcare Life Sciences, Baie-d'Urfé, QC, Canada) and exposed to Classic Single-Emulsion Autoradiography Film (Mandel Scientific, Guelph, ON, Canada). The films were developed automatically with an AGFA CP1000 X-Ray Film Processor and scanned with an UMAX Powerlook 1120 scanner.
结果与讨论
海关和边境保护局的帽子家族
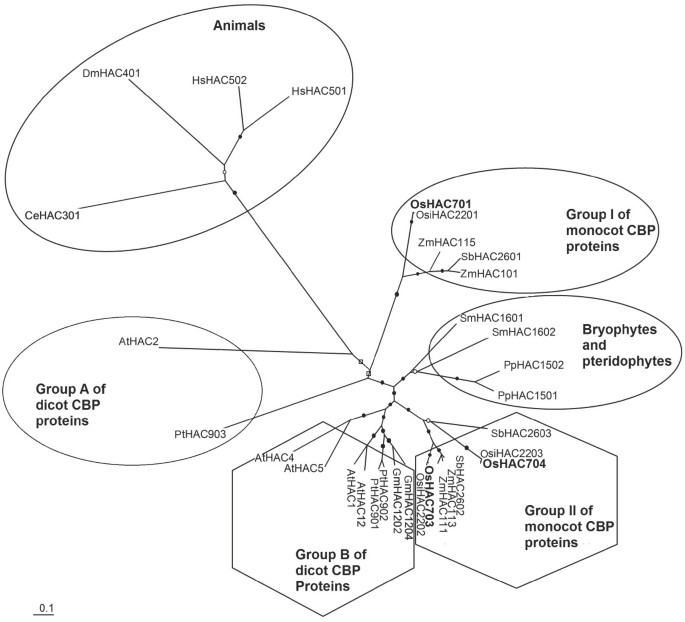
在水稻中鉴定出3个CBP家族蛋白,分别是OsHAC701、OsHAC703和OsHAC704。来自不同植物和动物的CBP家族蛋白的系统发育分析(图1结果表明,单子叶CBP蛋白可分为两个不同的组(组I和组II),组I包含OsHAC701,而组II包含OsHAC703和OsHAC704。同样,双子叶CBP蛋白被分为两个不同的组(A组和B组)1),表明OsHAC701与OsHAC703和OsHAC704的相似性分别仅为46.0%和42.0%。相比之下,OsHAC703和OsHAC704具有80.0%的序列一致性。此外,OsHAC701与I组单子叶CBP蛋白(SbHAC2601、ZmHAC101和ZmHAC115)的序列同源性平均为65.3%;见附加文件1),而与II组单子叶CBP蛋白(SbHAC2602、SbHAC2603、ZmHAC111和ZmHAC113)的序列平均同源性仅为43.0%。这些观察进一步证实了水稻CBP蛋白可分为两类的系统发育分析结果。此外,OsHAC701 OsHAC703, OsHAC704约41.0%,46.0%和45.5%一致,A组双子叶植物蛋白质,约42.9%,50.1%和50.0%一致,B组双子叶植物蛋白,这表明,第二组蛋白质更类似于蛋白质组A和B组比我蛋白质。水稻CBP与苔藓植物蛋白(PpHAC1501和PpHAC1502)和蕨类植物蛋白(SmHAC1601和SmHAC1602)序列同源性较低,与动物蛋白(HsHAC501、HsHAC502、DmHAC401和CeHAC301)序列同源性较低。综合起来,这些序列分析结果(参见附加文件1)与在系统发育树中获得的簇一致(图 1).
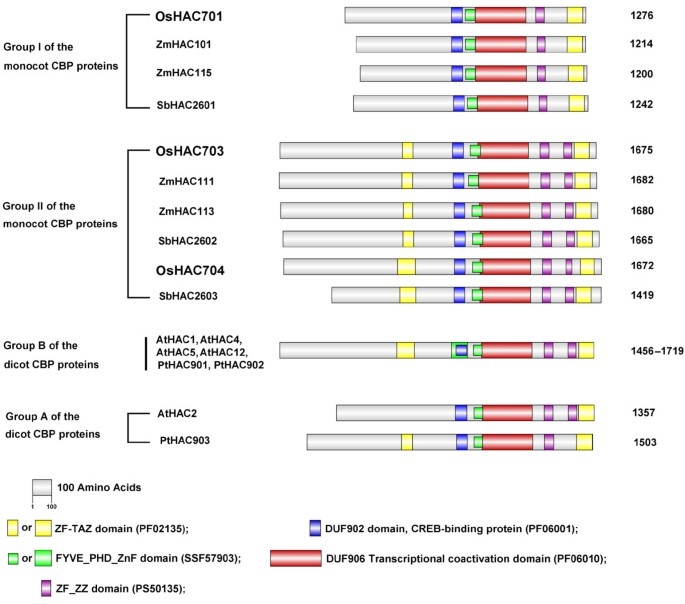
五个域(图2),包括duf902_creb结合蛋白结构域(PF06001,残余范围565-622)、FYVE/ phd型锌指(SSF57903, 639-690)、duf906_转录共激活结构域(PF06010, 691-958)、zz型锌指(PS50135, 1055-1177)和taz型锌指(PF02135, 1177-1260)。在OsHAC701和其他I组单子叶CBP蛋白(ZmHAC101, ZmHAC115和SbHAC2601)中使用InterProScan鉴定[33].相比之下,在OsHAC703和OsHAC704以及其他II组CBP蛋白中鉴定了两个额外的结构域;一个是位于z型锌指和taz型锌指之间c端附近的z型锌指,另一个是显示在duf902_creb结合蛋白结构域左侧的taz型结构域。与II组单子叶蛋白类似,B组双子叶蛋白都包含7个结构域,但有一个FYVE/ phd型锌指与duf902_creb结合蛋白结构域重叠。DUF902结构域存在于与转录激活和组蛋白乙酰化相关的几个转录共激活因子中[53.].参与转录共激活的DUF906结构域是cbp型HAT结构域的一部分[5.].taz型、zz型和FYVE/ phd型锌指在蛋白质识别和蛋白质相互作用中起重要作用[54.那55.].证据还表明,PHD手指与组蛋白和其他组蛋白识别蛋白相互作用[56.].这些分析表明OSHAC701,OSHAC703和OSHAC704可能具有帽子活动和转录辅助因子活动。DUF902和DUF906结构域在植物的CBP家族蛋白内高度保守。植物CBP家族蛋白的成员在锌手指的类型和数量方面不同。不同类型的锌手指可与不同的蛋白质或组蛋白结合[54.并被招募到染色质的不同靶区。
单子叶和双子叶中CBP家族蛋白的结构域。领域分析使用在SWISS-MODEL Workplace中实现的InterProScan进行。CBP蛋白质的名称和组显示在图的左边。蛋白质的长度显示在右边。在蛋白质序列中,从n端到c端,不同的结构域在其精确位置用不同的颜色和长度表示。括号中显示了域的Pfam登录号。此图使用了生物信息学工具DOG 1.0 [35.].有机体的缩写为:奥雅萨苜蓿无性系种群.japonica.(OS),Zea Mays.Zm评选,高粱双色(某人),拟南芥(在),杨树trichocarpa(PT)。
从氨基酸组成偏度来看,OsHAC701包含一个富含ser的区域(残基234-247)和一个富含cys的区域(1130-1157)。OsHAC703包含两个gln富区(111-135和402-625),而OsHAC704包含一个Poly-Ser延伸区(132-135)和一个gln富区(379-435)。OsHAC701中富含胱氨酸的区域表明该蛋白可能与其他蛋白相互作用[57.].OsHAC703和OsHAC704中富含gln的区域可能介导转录激活[58.那59.].与OsHAC703类似,AtHAC1、AtHAC4和AtHAC12均含有2个Gln-rich区,而AtHAC5含有1个Gln-rich区,与OsHAC704类似,AtHAC2含有1个sern -rich区,与OsHAC701类似。
AtHAC1显示乙酰转移酶活性[60.和原生质体系统中热休克诱导基因的转录共激活因子功能[20.].此外,AtHAC1、AtHAC5和AtHAC12具有广泛特异性的HAT活性,并可能共同作用使组蛋白H3赖氨酸9 (H3K9)乙酰化[61.].在活的有机体内,Athac1,Athac5和Athac12通过压制花卉阻遏物的表达,在促进开花中发挥冗余作用FLC.(开花轨迹C)[9.].同样,AtHAC1也被报道通过表观遗传修饰调控花期FLC.[6.].此外,AtHAC1与番茄热应激转录因子HsfB1相互作用在体外和在活的有机体内[20.].目前还不清楚大米是否会拟南芥CBP家族蛋白质的同源物具有相似的生物学功能。
TAF.2250个帽子家族
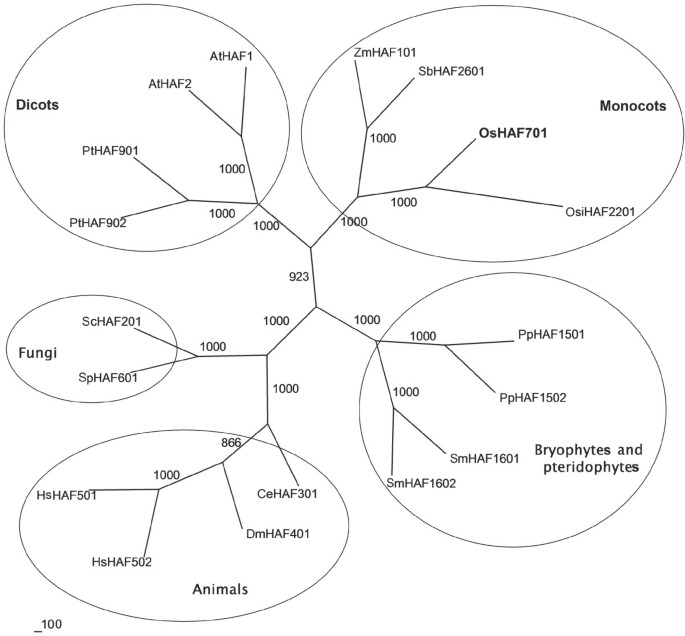
OSHAF701是唯一的TAF2250米基因组的家庭帽子。其他物种(图 3.)有一个或两个TAF2250 -类型的蛋白质。18 TAF的系统发育分析2250个家族蛋白显示,这些蛋白可以分为5个簇(图)3.).鉴于对树拓扑的高引导支持,我们推导出所有的TAF2250个家族蛋白可能有共同的起源,属于同一类。OsHAF701被放置在无根邻居连接树的单子叶簇中。在氨基酸水平,UniProt BLAST分析显示OsHAF701(见附加文件2)与Osihaf2201,其他单子叶同源物(ZM和Sb)的序列同一性约为97.0%,用Dicot同源物(AT和Pt),41.0%,含有薄膜和Pteridophyte同源物(PP和SM),29.8%,动物同源物(DM,CE和HS)和28.0%,真菌同源物(SC和SP)。
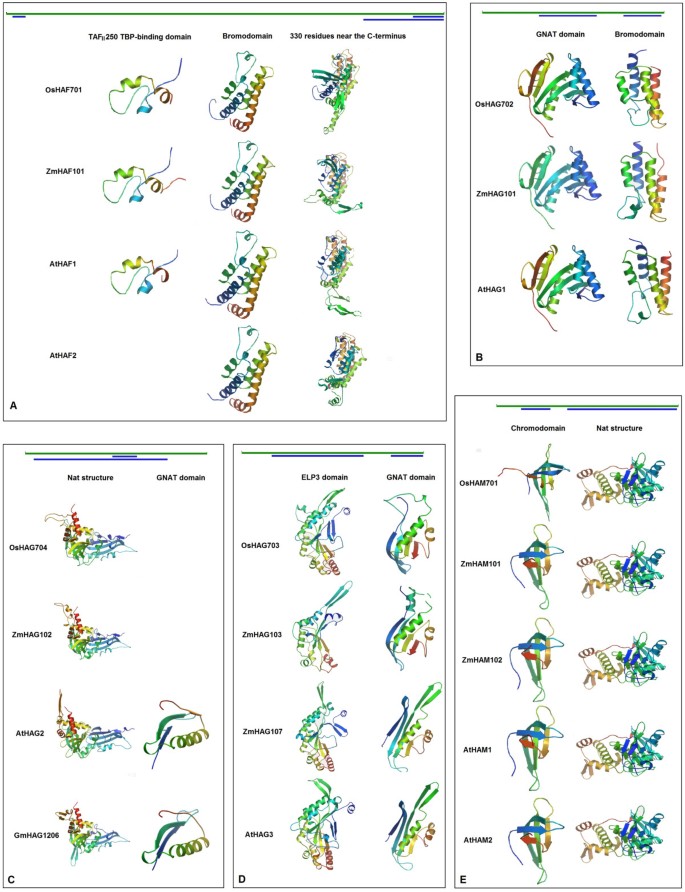
TAF的域分析2250家庭蛋白质在稻米稻草中表明OSHAF701中存在四个主要域:一个TAF2一个泛素结构域(PF00240, 641-710),一个C2HC锌指结构域(1370-1388)和一个溴结构域(PF00439, 1697-1779)。类似的结果也有报道拟南芥[5.]和玉米。3D模型与瑞士模型工作空间的比较[26-28发现OsHAF701、AtHAF1、AtHAF2和ZmHAF101的三维结构高度相似(特别是两个结合域)(图4.一个)。
在氨基酸组成偏倚方面,OsHAF701在n端有一个富含asp的区域(18-110),一个Poly-Ala延伸区(1004-1007)和一个Poly-Lys延伸区(1635-1645)。AtHAF1在n端附近有一个富含asp的区域(16-106),在c端附近有一个类似于OsHAF701的Poly-Lys延伸区(1289-1296)。AtHAF2包含两个Poly-Lys序列(999-1002和1195-1201)。
TAF.2250个家族基因编码塔塔结合蛋白(TBP)相关因子1 [62.].富含asp的区域,负责底物结合[63.与TAF重叠2OSHAF701和ATHAF1中的250 TBP绑定域。因此,富含ASP的区域可能参与酶活性的调节[64.那65.].已知与乙酰化的组蛋白赖氨酸残基结合的溴结构域[15那66.]在两个TAF的c端都有2250和GCN5家族蛋白[5.].序列比对显示,OsHAF701和OsHAG702的溴结构域序列是保守的(见附加文件)3.一个)。拟南芥,H3K9,H3K27和H4K12乙酰乙酰乙酰丙烯化所需的achaf2 / attaf1和AthAg1 / AtgcN5,参与生长和发育的光调节的光调节基因的乙酰化[12那15那62.].此外,AtHAF2调节了几个不受HAT活性影响的冷调节基因的表达[19].对于AtHAF1, rnai介导的基因沉默AtHAF1在拟南芥赋予抵抗力农杆菌属介导的转换(67.].OsHAF701、AtHAF1和AtHAF2的保守结构域和三维模型的相似性表明,OsHAF701可能与AtHAF1和AtHAF2具有相似的功能。
GNAT帽子
GNAT家族的GCN5亚家族
在水稻中鉴定的GCN5同源物是OsHAG702。OsHAG702存在两个结构域,即gcn5相关的n -乙酰基转移酶(GNAT)结构域(PS51186,残基范围168-315)和溴结构域(PF00439, 402-491)。利用SWISS-MODEL Workspace分析GNAT畴和溴畴的三维模型[26-28表明OsHAG702、AtHAG1和ZmHAG101之间存在高度相似性(图4.B).通过UniProtKB检测,OsHAG702在其n端区域有三种氨基酸组成偏差(见附加文件)3.B):一个富含sere的区域(残基8-67),一个Poly-Asp延伸(68-73),和一个Ala-rich区域(104-124)。在其他单子叶GCN5亚家族蛋白如ZmHAG101中也发现了类似的氨基酸组成偏差。AtHAG1在其n端还包含一个富含serf(3-66)的区域。
富含Ser的地区涉及酶活性的调节[57.].Ala-rich区是转录抑制和与TBP (TATA-box binding protein)相互作用的关键[68.].与3D模型分析一致,对GCN5亚家族蛋白的比对表明,种子植物GCN5亚家族蛋白的GNAT结构域和bromodomain高度保守[24].这些结果表明,OsHAG702可能具有与AtHAG1和ZmHAG101相似的功能。AtHAG1/AtGCN5显示HAT活性[17],乙酰化主要H3K14 [61.],并有助于冷调节基因表达[17-19].AtHAG1还参与通过调控的表达来控制花的分生组织活动WUSCHEL和钦am[8.].此外,光诱导基因表达需要AtaG1 [8.那12-15],并且对于根茎细胞的基础维护至关重要[7.]以及在转录和转录后水平上对miRNA积累的调控[69.].总之,这些数据表明AtaG1参与了染色质修饰的长期表观遗传调节和转录开关的短期控制。鉴于OSHAG702与ATHAG1显示高序列相似性,OSHAG702也可能在植物开发和压力反应的不同方面发挥重要作用。
蚋科的HAT1亚科
多序列比对显示,OsHAG704蛋白序列与单子叶和双子叶中其他HAT1亚家族蛋白具有高度的序列相似性(见附加文件)C3). InterProScan结构域分析和3D蛋白结构分析表明,水稻和其他5种被子植物hat1型蛋白都含有一个同源结构,称为乙酰辅酶a:氨基酸n -乙酰转移酶(Nat) (SSF55729),长度约为335个氨基酸残基。通过InterProScan结构域分析,在AtHAG2和GmHAG1204中发现了一个GNAT结构域(PF00583),但在其他HAT1亚家族蛋白中未发现。部分GmHAG1206序列(长度为61个残基)形成一个3D带状图,与AtHAG2的GNAT结构域非常相似(图)4.C)。在多序列对齐的基础上,GNAT域序列在GMHAG1204中显示出强的序列相似性与GMHAG1206序列的序列相似度(参见附加文件3.C),这表明GNAT结构域存在于GmHAG1206中。此外,由于6种被子植物的HAT1亚家族蛋白具有高度保守的序列(见附加文件)3.C),我们预测OsHAG704可能也包含GNAT结构域。
帽子1亚家族的单子叶和单萝卜同源物中的蛋白质序列和3D模型的高相似之处也表明它们可能具有类似的功能作用。Athag2(osshag704的同源物)乙酰化组蛋白H4K12 [61.和的表达ZMHAG102/HAT-BABA处理抑制玉米种子萌发[70].
GNAT家族的ELP3亚家族
在植物中发现ELP3亚家族蛋白具有高度的序列相似性[24,真菌和动物。利用SWISS-MODEL分析OsHAG703、ZmHAG103、ZmHAG107和AtHAG3中ELP3 (TIGR01211)和GNAT (PS51186)结构域的三维模型[26-28[这些蛋白质中存在3D结构的高相似性(图 4.D).序列对齐(参见附加文件3.D)和三维结构分析(图4.B,C,D)表明ELP3亚家族(OSHAG703)中的GNAT结构域不同于GCN5亚家族(OSHAG702)和HAT1亚家族(OSHAG704)中的域。
OsHAG703及其ELP3亚家族的同源物可能也具有类似的功能,因为它们在序列和3D结构上具有高度的相似性。AtHAG3/AtELP3是一种保守的拉长器HAT复合体的成员,在转录本伸长过程中与RNA Pol II相互作用,并在器官生长过程中的细胞增殖中发挥作用[11].此外,AtHAG3还可以调节植物对ABA的响应、抗氧化应激和花青素生物合成[21那22].拟南芥延长子也调控生长素信号基因SHY2 / IAA3,砧辊流入载体LAX2、乙烯信号转导、茉莉酸信号转导和非生物胁迫[23].此外,RNAi的谱线AtHAG3是耐农杆菌属介导的转换(67.].
MYST帽子家族
MYST家族被分为五个不相关的类。第I类由绿色谱系的蛋白质组成,绿色谱系包括MYST家族的蛋白质拟南芥和大米(10].OsHAM701是唯一在水稻中鉴定到的MYST家族蛋白。如图所示4.E,OSHAM701中染色体的3D结构类似于AthaM1,AthaM2,ZMHAM101和ZMHAM102的结构。此外,OSHAM701中的NAT结构的3D结构与AthaM1,AthaM2,ZMHAM101和ZMHAM102中的那些相似。此外,OSHAM701含有富含ALA的地区,并在其N-末端伸展(参见附加文件3.e)。
Myst型蛋白质的氨基酸序列在单焦点和Dicots内高度保守[24].Nat结构是乙酰转移酶活性的主要保守区域。据报道,hat的MYST家族中的染色体结构域与hat中的溴结构域相似,能够识别和结合特定的组蛋白残基[71].氨基酸序列和Myst型帽子的3D模型的高相似之处表明OSHAM701可能与其他神秘型蛋白质的功能相似拟南芥和其他被子植物。AtHAM1和AtHAM2优先乙酰化H4K5 [61.]并且参与男性和女性器官的配子形成拟南芥[10].
亚细胞定位预测
利用5个亚细胞定位预测程序确定水稻hat可能的定位位点(表1)3.),拟南芥和玉米(见附加文件4.).蛋白质的亚细胞定位与SLP-Local [36.那37.]产生的定位位点除OsHAF701可能定位于细胞核或胞质外,其余7个可靠性指数(RI)较低的oshat。athat和ZmHATs也得到了类似的SLP-Local结果。只有AtHAF1、AtHAF2和ZmHAF101对预测的亚细胞定位(细胞核或胞质)产生更高的RI。同样,TargetP [38.]检测到大多数可靠性较低的hat的可能本地化站点;然而,OsHAF701, AtHAF2, ZmHAC115和ZmHAF101被预测定位在细胞核或细胞质中,可信度最强(RC) 1。39.]还显示帽子的低频值;然而,OSHAC701对于核和细胞溶质本地化,OSHAC701评分相对较高的频率(数据集中的11个中的7.5分)。此外,Athac1和ZMHAC101为核和细胞源性本地化接受了相对高的RI分数。这些结果表明OSHAC701,ATHAC1和ZMHAC101可能是经历核细胞溶质转运的双重局部蛋白质[72].从狼Psort的详细结果,传统的psortii预测仪[40在所有OSHATS,ATHATS和ZMHAT中鉴定了NLS(图案4,模式7或二分钟)。在OSHATS中,OSHAF701具有最高的NLS评分,表明核定位的概率最高,而OSHAM701具有阴性NLS评分,为其细胞源定位提供了强烈的指示。针对所有OSHAT蛋白观察到NLSS。
ZmHAG101/ZmGCN5 (OsHAG702的同系物)在其n端含有一个NLS。进一步研究证实,n端负责ZmHAG101的核靶向[73].Earley等[61.[报告说,Athag1(OSHAG702的同源物),ATHAG2(奥什雅数的同源物)和AthaM1和Atham2(OSHAM701的两位同源物)往往被富集,以富集核仁的周边,而AthaM1和AthaM2重叠的含有含有的群体核仁组织区,这表明这些帽可能对核糖体RNA基因的激活是重要的。另一方面,ZMHAG102 / ZMHAT-B(OSHAG704和ATHAG2的同源物)主要位于胞质溶胶中,并且在核中存在显着比例的ZMHAG102 [74].NetNES [41.]在所有OSHATS和ZMHAG102中检测到富含亮氨酸的NES。所有OSHATS中存在NE的存在表明它们可能会出口核。
OSHAC701,OSHAG702和OSHAG704的核和细胞溶质定位
oshaac701 /OsHAG702/OsHAG704-YFP融合结构在拟南芥利用原生质体对oshaac701、OsHAG702和OsHAG704进行亚细胞定位。如图所示5.、oshaac701 - yfp、OsHAG702-YFP和OsHAG704-YFP均定位于细胞核和胞质。这些实验数据与亚细胞定位预测一致,OsHAC701、OsHAG702和OsHAG704均含有NLS和富含亮氨酸的NES。玉米中OsHAG704的同源基因ZmHAG102/ZmHAT-B也观察到细胞核和胞质定位[74].人体内的hat不仅催化组蛋白中特定赖氨酸残基的乙酰化,而且还催化非组蛋白底物中的乙酰化[75那76].在拟南芥,建议在细胞核外的不同蛋白质上可能发生赖氨酸乙酰化[77那78].OSHAC701,OSHAG702和OSHAG704的细胞溶质定位意味着这些OSHATS可能在组醇乙酰化之外发挥重要的催化作用。
的表达OsHATs通过RT-QPCR分析
oshat在不同组织中的表达
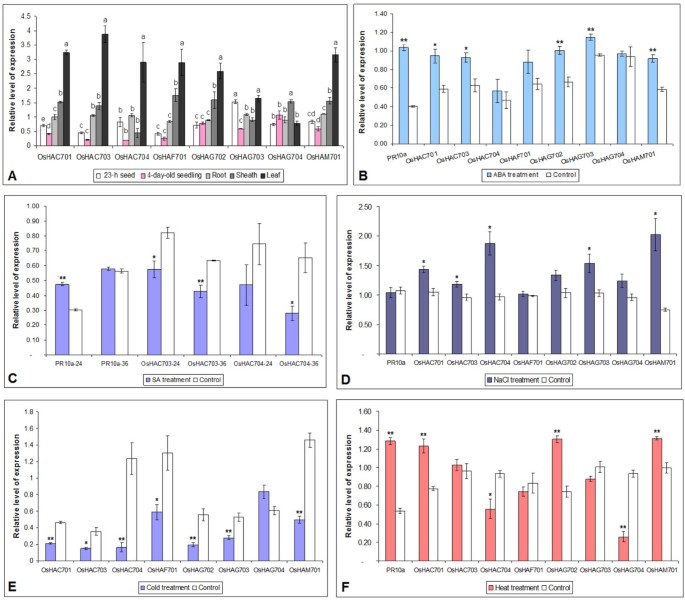
RT-qPCR检测表达OsHATs在五种类型的组织中,包括23-h吸入后的种子,4天龄幼苗和根,鞘和双叶阶段幼苗的叶子(图 6.)。8OsHATs在所有组织中表达,但在转录本丰度上有显著差异。最高的转录水平OsHATs除了OsHAG704在二叶期幼苗叶片中观察到,而O.S.帽子种子吸胀23小时后和4日龄幼苗的转录水平非常低。在所有组织中的持续表达表明hat在水稻中发挥重要作用,但这些蛋白质可能在发育和抵御不利条件方面具有不同的功能。
表达分析OsHATs在大米。(一种) RT-qPCR表达分析OsHATs在五个不同的组织中:种子23小时,种子吸水23小时;4日龄幼苗,4日龄幼苗全株;二叶期幼苗的根、鞘和叶组织。这八个分子的mRNA的相对数量OsHATs被测量。数据以达到阈值荧光值(Ct)所需的周期数表示,并用对比Ct方法(ΔΔCt)进行分析。表达式值归一化为eEF-1α和Ubq-1。结果为3个生物重复的平均值,每个生物重复由3个技术重复组成。误差棒表示标准误差。每条条形图上方不同的字母表示组织之间的显著差异(P.< 0.05,单因素方差分析和LSD/SNK事后检验)。(B.-F) RT-qPCR分析OsHATs激素或非生物胁迫处理后水稻叶片的表达。水稻基因PR10A(已知由ABA和SA诱导)作为阳性对照。表达相对于Ubq-1基因的表达。色条,处理过的植物;白条,未经处理的植物。根据学生的T.-test, *和**表明治疗组与对照组在P. < 0.05 andP.分别为< 0.01。(B.100 μM ABA处理二叶期幼苗24 h。幼苗在水里浸泡的时间与对照相同。(C)用100μmSA处理24小时或36小时。(D.300 mM NaCl处理12 h。(E.)在黑暗中用4±1℃处理3小时。(F)用42℃处理3小时。
ABA处理对oshat表达的响应
植物激素ABA在多种发育过程中发挥着关键作用,如调节种子萌发和幼苗建立[79].ABA还参与植物对各种胁迫的响应,如盐胁迫、渗透胁迫、冷胁迫、创伤胁迫和致病胁迫[16那80那81].确定是否表达了O.S.帽子在水稻幼苗生长期间由ABA调节,我们分析了表达模式OsHATs100 μM ABA处理24 h后(图46.B)。PR10A作为阳性对照。PR10AABA治疗后转录增加2.55倍,表明治疗是有效的。转录水平OsHAC701那OsHAC703那OsHAG702那OsHAG703,OsHAM701对外源ABA的响应显著升高。这些结果表明OsHAC701那OsHAC703那OsHAG702那OsHAG703,OsHAM701可以参与ABA信号传导途径以应对稻幼苗生长期间的环境胁迫。
用ABA处理短时间(0-120分钟)导致组蛋白H3和H4乙酰化和烟草中磷酸化的动态变化,拟南芥T87细胞和烟草全叶拟南芥[16].以前的研究表明,用100μmaba的水稻幼苗叶子24小时的治疗增加了表达OsHDA702,但压抑的表达OsSRT701和OsSRT702[3.].ABA治疗大麦幼苗也诱导表达HvGCN5HvELP3,HvMSYT[24].与此相反,ABA处理玉米种子48或72 h后,抑制了表达ZMHAG101(ZmGCN5),ZMHAG102(ZmHAT-B),以及几个ZmHDACs.ABA选择性地诱导组蛋白乙酰化VP1基因(胚胎发生相关的基因viviparous1.),并激活其转录,提示ZmHATs和ZmHDACs可能参与了种子萌发过程中的ABA信号通路[70].在拟南芥幼苗,ABA下调了表达AtHD2C,一个HD2-typeHDAC,而过度表达AtHD2C增强了对ABA的耐受性拟南芥,这表明ATHD2C可以调节ABA和压力响应[80].此外,AtHAG3 (ELP3)参与ABA的响应拟南芥据报道(21那22].最近,Chen等人[82报道,ABA治疗富集了基因活化标志物,组蛋白H3K9K14乙酰化,H3K4三甲基化,但降低了基因抑制标志物,H3K9二甲基化,其中一些ABA诱导基因拟南芥.这些观察结果表明,帽子和HDACs诱导的组蛋白乙酰化/脱乙酰化参与在包括各种植物中的ABA信号通路的调节中拟南芥烟草、玉米和大米。
卫生表达响应SA治疗
水杨酸在植物免疫应答等多种生理过程中起着重要的调节作用。此外,SA还可与其他植物激素如ABA、生长素和赤霉酸相互作用[83那84].利用两叶期幼苗叶片总RNA进行RT-qPCR分析OsHATs由SA管理(图6.C).成绩单水平PR10A与对照相比,SA处理24 h后,其产量增加了1.58倍。SA处理36 h后OsHAC703和OsHAC704分别为对照植株的67.43%和42.76%。其余6种蛋白表达无明显变化OsHATSA处理24小时或36小时后(数据未显示)。前人研究表明,100 μM SA处理水稻幼苗叶片12 h后,下调了水稻幼苗的表达OsHDA702那OsHDA704那OsHDA706那OsSRT701,OsSRT702而是上调表达OsHDT702[3.].这些观察表明帽子和HDAC基因家族可能参与了植物防御反应中的SA信号通路。
盐、冷、热处理对oshat表达的影响
通过盐处理,转录水平OsHAC701那OsHAC703那OsHAC704那OsHAG703,OsHAM701分别比对照组增加了36.02%、23.41%、94.11%、49.57%和170.32%(图6.D)。
表达八OsHATs也对暴露在低温下的幼苗进行了测量。除了OsHAG704,表达另一个OsHATs分别为45.84%、42.20%、13.32%、45.17%、34.81%、53.05%和34.16%(图6.e)。
响应热处理(图 6.F),PR10A在暴露于42℃后,转录物在42℃下叶片增加2.39倍。与对照相比,转录水平OsHAC701那OsHAG702,OsHAM701响应热处理,分别增加58.97%,75.77%和31.84%。相反,表达了OsHAC704和OsHAG704分别降低39.99%和71.92%。
以前的研究表明,盐压力诱导表达OsHDA702,而的表达OsHDA704那OsHDA712,OsSRT702减少了。冷治疗抑制了表达OsHDA704那OsHDA712,OsSRT701而是诱导的表达OsHDA702[3.].AtHAG1参与冷驯化过程中冷调节基因的表达[17-19].AtHAF2参与一些冷调节基因的调控[19].Athac1与番茄热应激转录因子HSFB1相互作用,含有组蛋白样图案[20.].此外,通过Genevistigator对微阵列数据的分析表明,热应力(38℃3小时)上调了表达AtHDA6那AtHDA7那AtHDA5那AtHDA8,AtHDA14[72].综上所述,hat和HDACs可被盐、冷或热胁迫调节,这表明这些蛋白可能在植物对非生物胁迫的响应中发挥重要的表观遗传调节作用。
关于用盐处理12小时的双叶液幼苗的叶片,Western印迹分析表明,增加了组蛋白H3K18乙酰化(参见附加文件5.B),但H3K9和H4K5的乙酰化没有变化(附加文件5.C和D)被观察到。以前的研究表明,在培养的细胞和叶拟南芥和烟草,组蛋白H3 Ser-10磷酸化,H3磷酸化乙酰化和组蛋白H4乙酰化被盐处理上调[16].我们观察到增加的转录水平OsHAC701那OsHAC703那OsHAC704那OsHAG703,OsHAM701这与组蛋白H3K18乙酰化的增加有关。因此,这些蛋白质可能在水稻盐胁迫反应中发挥作用。
结论
CBP和TAF的本文系统发育分析2250 HAT家族为这两个蛋白家族的进化关系提供了深刻的见解。单子叶和双子叶CBP家族蛋白可以细分为两个不同的组。不同职业安全卫生服务体系所识别的特定领域的多样性表明职业安全卫生服务体系经历了功能多样化。oshat及其同源物在蛋白质序列、保守结构域和三维模型等方面的高度相似性拟南芥玉米表明,欧沙在水稻生长和发展期间进行多种功能。亚细胞定位预测表明所有OSHATS可能在细胞核和细胞溶胶中定位。瞬态表达分析拟南芥原生质体证实了oshaac701、OsHAG702和OsHAG704的核和胞质定位。RT-qPCR分析表明OsHATs在水稻中表达。此外,它们的表达是通过与激素ABA和SA以及盐,冷和热应力的外源性处理来调节,这表明OSHATS可能在植物防御反应中发挥重要作用。
参考
- 1.
王志强,王志强,王志强。植物基因组可及性的组蛋白修饰和动态调控。植物生态学报。2007,10(6):645-652。10.1016 / j.pbi.2007.07.013。
- 2.
陈ZJ,田L:植物开发和多倍体中动态和可逆组蛋白乙酰化的作用。Biochim Biophys Acta。2007,1769(5-6):295-307。
- 3.
付伟,吴轲,段俊:水稻组蛋白去乙酰化酶的序列与表达分析。中国生物化学学报。2007,31(4):453 - 457。10.1016 / j.bbrc.2007.03.010。
- 4.
Neuwald AF, Landsman D: gcn5相关组蛋白n -乙酰转移酶属于包括酵母SPT10蛋白在内的一个不同的超家族。生物化学进展,1997,22(5):154-155。10.1016 / s0968 - 0004(97) 01034 - 7。
- 5。
Pandey R, Muller A, Napoli CA, Selinger DA, Pikaard CS, Richards EJ, Bender J, Mount DW, Jorgensen RA:拟南芥组蛋白乙酰转移酶和组蛋白去乙酰化酶家族的分析表明,多细胞真核生物的染色质修饰功能多样化。核酸学报2002,30(23):5036-5055。10.1093 / nar / gkf660。
- 6。
邓伟,刘超,裴颖,牛玲,曹晓霞:组蛋白乙酰转移酶AtHAC1通过抑制拟南芥开花位点C调控开花时间的作用。植物营养与肥料学报。2007,27(4):457 - 461。10.1104 / pp.107.095521。
- 7.
Kornet N, Scheres B:在拟南芥中,GCN5组蛋白乙酰转移酶复合物的成员调节plethora介导的根干细胞生态位维持和转运放大细胞增殖。植物学报,2009,21(4):1070-1079。10.1105 / tpc.108.065300。
- 8.
Bertrand C, Bergounioux C, Domenichini S, Delarue M, Zhou DX:拟南芥组蛋白乙酰转移酶AtGCN5通过WUSCHEL/AGAMOUS途径调控花的分生组织活性。中国生物医学工程学报。2003,27(3):427 - 434。10.1074 / jbc.M302787200。
- 9.
韩淑娟,宋丹东,卢载生,卢乙:CBP/p300类基因在植物开花时间调控中的作用。植物学报,2007,49(1):103-114。
- 10.
MYST组蛋白乙酰转移酶在拟南芥配子体发育中起重要作用。植物生态学报。2008,8:121-10.1186/1471-2229-8-121。
- 11.
Nelissen H, Fleury D, Bruno L, Robles P, De Veylder L, Traas J, Micol JL, Van Montagu M, Inze D, Van Lijsebettens M:延长体突变体在植物中发现了一种功能性延长体复合体,在器官生长过程中发挥细胞增殖的作用。美国国家科学院学报。[j] .环境科学学报,2003,21(4):457 - 461。10.1073 / pnas.0502600102。
- 12.
Benhamed M,Bertrand C,Servet C,Zhou DX:Arabidopsis GCN5,HD1和TAF1 / Haf2相互作用,以调节光响应基因表达所需的组蛋白乙酰化。植物细胞。2006,18(11):2893-2903。10.1105 / TPC.106.043489。
- 13.
郭玲,周娟,Elling AA, Charron JB,邓新文:拟南芥光调控基因的组蛋白修饰和表达受光照条件的影响。植物营养与肥料学报。2008,27(4):457 - 461。10.1104 / pp.108.122929。
- 14。
植物光信号的染色质重塑。植物生理学报,2011,142(4):305-313。10.1111 / j.1399-3054.2011.01476.x。
- 15.
组蛋白乙酰转移酶AtGCN5/HAG1在拟南芥发育和诱导基因表达中的作用。植物学报。2010,3(4):670-677。10.1093 / mp / ssq018。
- 16。
Sokol A,Kwiatkowska A,Jerzmanowski A,Prymakowska-Bosak M:烟草和拟南芥细胞中应激诱导基因的上调响应非生物应激和ABA处理与组蛋白H3和H4修饰的动态变化相关。Planta。2007,227(1):245-254。10.1007 / S00425-007-0612-1。
- 17。
Stockinger EJ,Mao Y,Regier Mk,Triezenberg SJ,Thomashow MF:转录适配器和组氨酸乙酰转移酶蛋白质拟南芥及其与CBF1的相互作用,一种参与冷调节基因表达的转录活化剂。核酸RES。2001,29(7):1524-1533。10.1093 / nar / 29.7.1524。
- 18.
Vlachonasios ke,Thomashow Mf,Triezenberg SJ:ADA2B的破坏突变和GCN5转录适配器基因显着影响拟南芥生长,发育和基因表达。植物细胞。2003,15(3):626-638。10.1105 / TPC.007922。
- 19.
组蛋白乙酰转移酶在拟南芥低温诱导基因调控中的作用acta botanica sinica(云南植物学报),2010,34(1):1 - 4。
- 20.
Bharti K, Von Koskull-Doring P, Bharti S, Kumar P, Tintschl-Korbitzer A, Treuter E, Nover L:番茄热胁迫转录因子HsfB1是一种新型的通用转录辅激活因子,它具有组蛋白样motif,与植物CREB结合蛋白同源物HAC1相互作用。植物学报,2004,16(6):1521-1535。10.1105 / tpc.019927。
- 21.
陈志刚,张海涛,张海涛,龚志:拟南芥ABO1/ELO2基因突变对干旱敏感性的影响。中国生物医学工程学报。2006,26(18):6902-6912。10.1128 / MCB.00433-06。
- 22.
Zhou X, Hua D, Chen Z, Zhou Z, Gong Z: longator介导拟南芥ABA响应、抗氧化应激和花青素合成。植物学报,2009,60(1):79-90。10.1111 / j.1365 - 313 x.2009.03931.x。
- 23.
Nelissen H, De Groeve S, Fleury D, Neyt P, Bruno L, Bitonti MB, Vandenbussche F, Van der Straeten D, Yamaguchi T, Tsukaya H, et al .:植物伸长器在RNA聚合酶II转录伸长过程中调控生长素相关基因。美国国家科学院学报。系统工程理论与实践,2016,35(4):527 - 534。10.1073 / pnas.0913559107。
- 24.
Papaefthimiou D, Likotrafiti E, Kapazoglou A, Bladenopoulos K, Tsaftaris A:大麦表观遗传染色质修饰物:III。大麦GNAT-MYST家族组蛋白乙酰转移酶的分离、鉴定及对外源ABA的响应植物营养与肥料学报。2010,48(2):98-107。
- 25.
水杨酸(aqua F, Timmermann T, Arce-Johnson P):葡萄组蛋白乙酰转移酶和去乙酰化酶家族的分析。植物生理学报。2010,48(2):194-199。
- 26.
Schwede T, Kopp J, Guex N, Peitsch MC: SWISS-MODEL:一个自动化的蛋白质同源建模服务器。中国生物医学工程学报,2003,31(13):3381-3385。10.1093 / nar / gkg520。
- 27.
Guex N, Peitsch MC: SWISS-MODEL和Swiss-PdbViewer:比较蛋白质建模的环境。电泳。1997,18(15):2714-2723。10.1002 / elps.1150181505。
- 28.
Arnold K, Bordoli L, Kopp J, Schwede T: SWISS-MODEL工作空间:基于网络的蛋白质结构同源建模环境。生物信息学。2006,22(2):195-201。10.1093 /生物信息学/ bti770。
- 29.
Larkin MA, Blackshields G, Brown NP, Chenna R, mcgetigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, et al: Clustal W and Clustal X version 2.0。生物信息学。2007,23(21):2947-2948。10.1093 /生物信息学/ btm404。
- 30.
Felsenstein J: phylip -系统发育推断包(3.2版)。演化论。1989,5:164-166。
- 31.
黄志强,王志强,王志强:蛋白质进化变化的一个模型。蛋白质序列和结构图谱。1972年,5:345 - 352。
- 32.
Page Rd:TreeView:应用程序在个人计算机上显示系统发育树的应用程序。计算Appl Biosci。1996,12(4):357-358。
- 33.
Zdobnov EM, Apweiler R: interproscan - InterPro签名识别方法的集成平台。生物信息学。2001,17(9):847-848。10.1093 /生物信息学/ 17.9.847。
- 34.
Jain E, Bairoch A, Duvaud S, Phan I, Redaschi N, Suzek BE, Martin MJ, McGarvey P, Gasteiger E:生命科学基础设施:UniProt网站的设计和实现。生物信息学。2009,10:136-10.1186/1471-2105-10-136。
- 35.
任建文,高旭,金超,薛勇,姚晓霞:DOG 1.0:蛋白质结构域图解。电池学报,2009,19(2):271-273。10.1038 / cr.2009.6。
- 36.
华胜,孙铮:基于支持向量机的蛋白质亚细胞定位预测。生物信息学。2001,17(8):721-728。10.1093 /生物信息学/ 17.8.721。
- 37.
Matsuda S, Vert JP, Saigo H, Ueda N, Toh H, Akutsu T:一种利用支持向量机预测亚细胞位置的蛋白质序列的新表示。中国生物医学工程学报。2005,14(11):2804-2813。10.1110 / ps.051597405。
- 38。
基于n端氨基酸序列预测蛋白质的亚细胞定位。中国生物医学工程学报,2000,30(4):457 - 461。10.1006 / jmbi.2000.3903。
- 39。
Horton P, Park KJ, Obayashi T, Fujita N, Harada H, Adams-Collier CJ, Nakai K: WoLF PSORT:蛋白质定位预测器。核酸学报2007,35 (Web服务器版):W585-W587。
- 40.
Horton P,Nakai K:用K最近邻居分类器更好地预测蛋白质细胞定位位点。Proc Int Conv Intel Syst Mol Biol。1997年,5:147-152。
- 41.
La Cour T,Kiemer L,Molgaard A,Gupta R,Skriver K,Brunak S:富含幼亮核导出信号的分析与预测。蛋白质ENG DES SEL。2004,17(6):527-536。10.1093 /蛋白/ gzh062。
- 42.
Karimi M,Depicker A,Hilson P:用植物网关向量重组克隆。植物理性。2007,145(4):1144-1154。10.1104 / pp.107.106989。
- 43.
Tzfira T,Tian GW,Lacroix B,Vyas S,Li J,Leitner-Dagan Y,Krichevsky A,Taylor T,Vainstein A,Citovsky V:PSAT载体:用于自发荧光蛋白标记的模块化组质粒,植物。植物mol biol。2005,57(4):503-516。10.1007 / s11103-005-0340-5。
- 44.
刘志强,赵永华,王志强:拟南芥叶肉原生质体的瞬时基因表达分析。光子学报。2007,2(7):1565-1572。10.1038 / nprot.2007.199。
- 45.
Alinsug MV,Chen FF,Luo M,Tai R,Jiang L,Wu K:拟南芥中II类HDA的亚细胞定位:HDA15的核细胞骨质穿梭由光驱动。Plos一个。2012,7(2):E30846-10.1371 / journal.pone.0030846。
- 46.
Lee Ly,Fang MJ,Kuang Ly,Gelvin SB:用于多色双分子荧光互补的载体,以研究生物植物细胞中蛋白质 - 蛋白质的相互作用。植物方法。2008,4:24-10.1186/1746-4811-4-24。
- 47.
At3g16150编码一个K +−依赖的L-天冬酰胺酶。植物学报。2006,34(3):668-679。10.1007 / s00425 - 006 - 0245 - 9。
- 48.
Jain M,Nijhawan A,Tyagi Ak,Khurana JP:通过定量实时PCR验证用于研究水稻基因表达的内部控制。Biochem Biophys Res Communce。2006,345(2):646-651。10.1016 / J.BBRC.2006.04.140。
- 49.
水稻长日照开花的基因网络激活了RFT1编码的移动开花信号。发展。2009,136(20):3443-3450。10.1242 / dev.040170。
- 50.
MIDOH N,IWATA M:水稻细胞内发病相关蛋白的探针诱导基因的克隆与表征。植物细胞生理。1996,37(1):9-18。10.1093 / oxfordjournals.pcp.a028918。
- 51.
拟南芥中CpG甲基化的消除改变了异染色质中组蛋白H3甲基化的模式。美国国家科学院学报。acta photonica sinica, 2011, 40(4): 427 - 434。10.1073 / pnas.1432939100。
- 52.
Probst AV, Fagard M, Proux F, Mourrain P, Boutet S, Earley K, Lawrence RJ, Pikaard CS, Murfett J, Furner I, et al .:拟南芥组蛋白去乙酰化酶HDA6是维持转录基因沉默所必需的,并决定rDNA重复序列的核组织。植物学报。2004,16(4):1021-1034。10.1105 / tpc.018754。
- 53.
Sachchidanand,Resnick-Silverman L,Yan S,Mutjaba S,Liu Wj,Zeng L,Manfredi JJ,周MM:基于目标结构的小分子发现,阻断人p53和Creb结合蛋白质结合。Chem Biol。2006,13(1):81-90。10.1016 / J.Chembiol.2005.10.014。
- 54.
黏性手指:锌手指作为蛋白质识别基元。生物化学进展,2007,32(2):63-70。10.1016 / j.tibs.2006.12.007。
- 55.
Bienz M: PHD手指,一个核蛋白相互作用域。生物化学进展,2006,31(1):35-40。10.1016 / j.tibs.2005.11.001。
- 56.
Lallous N, Legrand P, McEwen AG, Ramon-Maiques S, Samama JP, Birck C:人类UHRF1的PHD手指揭示了一个新的未甲基化组蛋白H3尾部阅读器亚群。公共科学图书馆。2011,6 (11):e27599-10.1371/journal. pon.0027599。
- 57.
植物蛋白激酶家族与信号转导。植物营养与肥料学报。2012,27(2):451-457。10.1104 / pp.108.2.451。
- 58.
拟南芥(Arabidopsis Thaliana) Histone Deacetylase 14 (Hda14)是一种α -微管蛋白去乙酰化酶,与Pp2a相关联,并在微管部分与推测的组蛋白乙酰转移酶Elp3富集。植物学报,2012,71(2):263-272。10.1111 / j.1365 - 313 x.2012.04984.x。
- 59.
在酵母GAL1启动子上哺乳动物转录激活域的功能差异。中国海洋大学学报(自然科学版),1994,13(3):641-645。
- 60。
研究表明,p300/CBP蛋白的同源性与p300/CBP蛋白的同源性有关。中国生物医学工程学报,2011,29(3):589-597。10.1093 / nar / 29.3.589。
- 61.
拟南芥共激活因子组蛋白乙酰转移酶的体外特异性:基因激活中组蛋白超乙酰化的意义。植物学报,2007,52(4):615-626。10.1111 / j.1365 - 313 x.2007.03264.x。
- 62.
Bertrand C, Benhamed M, Li YF, Ayadi M, Lemonnier G, Renou JP, Delarue M, Zhou DX:拟南芥HAF2基因编码塔塔结合蛋白(TBP)相关因子TAF1,需要整合光信号来调控基因表达和生长。中国生物医学工程学报。2005,27(2):1465-1473。
- 63.
梁pH,ko tp,王啊:戊基转移酶的结构,机制和功能。EUR J Biochem。2002,269(14):3339-3354。10.1046 / J.1432-1033.2002.03014.x。
- 64.
Kalmykova Ai,Dobritsa Aa,Gvozdev Va:苏(STE)在Y染色体和Betack2TES基因编码预测同种型同种蛋白激酶CK2调节β-亚基的同种型。费用。1997,416(2):164-166。10.1016 / s0014-5793(97)01195-2。
- 65.
酪蛋白激酶2的下调和多肽激活是由beta亚基55-64区域的酸性残基介导的。钙调蛋白作为磷酸化底物的研究。生物化学。1994,33(14):4336-4342。10.1021 / bi00180a030。
- 66.
染色质调控复合物中溴结构域的结构和功能。acta photonica sinica, 2001, 32(4): 441 - 446。
- 67.
rnai介导的基因沉默揭示了拟南芥染色质相关基因参与农杆菌介导的根转化。美国国家科学院学报。[j] .地理科学进展,2019,38(4):457 - 461。10.1073 / pnas.0706986104。
- 68.
Li C,Manley JL:通过结合Tata结合蛋白并阻断TFIID-TATA盒相互作用来跳过抑制转录。Mol细胞Biol。1998,18(7):3771-3781。
- 69.
张伟,周德新:组蛋白乙酰转移酶GCN5对拟南芥miRNA通路的干扰作用。电池学报,2009,19(7):899-909。10.1038 / cr.2009.59。
- 70.
张丽丽,邱志强,胡玉英,杨飞,严胜,赵磊,李波,何松,黄敏,李军,等:ABA处理对玉米萌发种子VP1基因表达和选择性启动子相关组蛋白乙酰化的影响。植物营养与肥料学报。2011,34(3):427 - 434。10.1111 / j.1399-3054.2011.01496.x。
- 71.
de la Cruz X, Lois S, Sanchez-Molina S, Martinez-Balbas MA:蛋白质基序读组蛋白代码吗?生物学报,2005,27(2):164-175。10.1002 / bies.20176。
- 72.
余春华,吴凯:植物RPD3/HDA1家族组蛋白去乙酰化酶的系统发育、亚细胞定位和表达模式。生物技术学报。2009,9:37-10.1186/1471-2229-9-37。
- 73.
Bhat RA, Riehl M, Santandrea G, Velasco R, Slocombe S, Donn G, Steinbiss HH, Thompson RD, Becker HA:玉米中GCN5水平的改变揭示了操纵组蛋白乙酰化的动态响应。植物学报,2003,33(3):455-469。10.1046 / j.1365 - 313 x.2003.01642.x。
- 74.
luser A, Eberharter A, Loidl A, Goralik-Schramel M, Horngacher M, Haas H, Loidl P:玉米胚胎组蛋白乙酰转移酶B复合物的分析。中国生物医学工程学报,1999,27(22):4427-4435。10.1093 / nar / 27.22.4427。
- 75.
GU W,Roeder RG:通过P53 C-末端结构域的乙酰化激活P53序列特异性DNA结合。细胞。1997,90(4):595-606。10.1016 / S0092-8674(00)80521-8。
- 76.
赖氨酸乙酰转移酶的不同超家族及其在白血病和其他疾病中的作用。核酸学报2004,32(3):959-976。10.1093 / nar / gkh252。
- 77.
在拟南芥中,不同功能和亚细胞位置的蛋白质赖氨酸乙酰化。植物营养与肥料学报。2011,32(4):429 - 434。10.1104 / pp.110.171595。
- 78.
在拟南芥中,赖氨酸乙酰化是一种广泛应用的蛋白质修饰方法。植物营养与肥料学报。2011,32(4):591 - 598。10.1104 / pp.110.165852。
- 79.
陈志强,陈志强,陈志强,等。种子和幼苗脱落酸信号转导的研究进展。植物学报。2002,14(增刊):S15-45。
- 80。
Sridha S,Wu K:鉴定ATHD2C作为拟南芥中脱落酸反应的新型调节因子。工厂J.2006,46(1):124-133。10.1111 / J.1365-313x.2006.02678.x。
- 81。
Fujita Y, Fujita M, Shinozaki K, Yamaguchi-Shinozaki K: aba介导的植物渗透胁迫的转录调控。植物营养学报,2011,24(4):539 - 543。10.1007 / s10265 - 011 - 0412 - 3。
- 82。
陈丽玲,罗敏,王艳艳,吴凯:拟南芥组蛋白去乙酰化酶HDA6在ABA和盐胁迫反应中的参与。中国生物医学工程学报。2010,61(12):3345-3353。10.1093 / jxb / erq154。
- 83。
植物激素在植物防御反应中的作用。acta botanica sinica(云南植物学报),2009,29(4):489 - 494。10.1007 / s11103 - 008 - 9435 - 0。
- 84。
安丙,牟忠:水杨酸及其在植物免疫中的作用。acta botanica sinica(云南植物学报),2011,53(6):412-428。10.1111 / j.1744-7909.2011.01043.x。
致谢
我们感谢Frédéric Marsolais提供的RT-qPCR和western blot设备。我们感谢Abdelali Hannoufa提供乙酰组蛋白H4K5抗体。我们感谢苏珊·西博尔德对手稿的英文编辑。我们感谢Sukhminder Sawhney, Xinhua Wang, Farida Meerja, Fuqiang Yin, Agnieszka Pajak, Ying Wang, Mimmie Lu, Fang Hui和Alex Molnar在加拿大农业和农业食品南部作物保护和食品研究中心的技术帮助和支持。感谢中国科学院华南植物园李国良、袁连宇、赵林茂和高燕的帮助。基金资助:广东省科技计划项目(no. 2011A020102008);国家自然科学基金项目(no. 30871465, no. 30971564, no. 90919038);2008AA10Z107),加拿大农业和农业食品研究基金,和台湾国家科学委员会(99-2321-B-002-027-MY3和101-2923-B-002-005-MY3)。XL项目由国家留学基金委资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
XL进行了实验,进行了生物信息学分析,并起草了手稿。ML进行原生质体转染和共聚焦显微镜观察。WZ和J Zhang参与了RT-qPCR分析。J Zhao参与了HAT-YFP融合载体的构建。LT、KW和JD构思了这个项目,并获得了资金。KW与LT和JD进行了讨论,并进行了设计。LT、KW、JD对手稿进行了修改。所有作者阅读并批准了最终的手稿。
电子辅料
12870 _2011_1127_moesm1_esm.doc
附加文件1:使用UniProt Blast对来自植物和动物的CBP家族蛋白进行序列识别分析。(DOC 58 KB)
OSHAF701的序列同一性与17 TAF
额外的文件2:2250型蛋白质使用UniProt BLAST。(DOC 37 KB)
使用Clustalw2的单焦点和Dicots在单焦点和Dicots中的氨基酸序列比对。
附加文件3:*,高度保守的位置;职位高度保守;.,低序列相似的位置;空白,可变位置。(a)摩擦染色体在osshag702和Oshaf701中的氨基酸序列比对。(b)GCN5亚家族蛋白的N-末端的多序列比对。富含Ser的地区由黑色遮蔽,聚架伸展为黄色阴影,富含绿色阴影的富有的地区,并通过红色的富型地区。(c)HAT1亚家族蛋白的多序列对准。上方序列的红线表示酰基-Coa n-酰基转移酶(NAT)结构。封闭在矩形中的序列是不同的或不确定的GNAT结构域。米中的聚衬弹伸展,用黄色遮荫突出显示。(d)HAG702,HAG703和HAG704中GNAT结构域的序列对准。 The predicted GNAT domain’s sequence in HAG704 has low similarity with the other two GNAT domains. (E) Amino acid sequence alignment of the N-terminus of the MYST-type proteins. Ala-rich regions in rice are shaded in gray, Poly-Gly stretches in rice are highlighted in red type, and the Pro-rich region in AtHAM2 is shaded blue. (PDF 4 MB)
预测hat的亚细胞定位
额外的文件4:拟南芥和玉米。(XLS 22 KB)
12870 _2011_1127_moesm5_esm.tiff
附加文件5:水稻叶片中组蛋白乙酰化反应的分析。分别用(盐)和(对照)300 mM NaCl处理两叶期水稻幼苗叶片12 h。在每幅图的底部,考马斯蓝染色显示了相同的蛋白质含量。组蛋白H3作为负载对照(a)。Western blot分析如下抗体:anti-Histone H3 (a), anti-acetyl-Histone H3K18 (B), anti-acetyl-Histone H3K9 (C)和anti-acetyl-Histone H4K5 (D)。数据代表三个独立实验。(TIFF 611 KB)
作者的原始提交的图像文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
刘,X.,罗,M.,张,W。等等。米中的组蛋白乙酰转移酶(奥雅萨苜蓿L.):系统发育分析,亚细胞定位和表达。BMC植物BIOL.12,145(2012)。https://doi.org/10.1186/1471-2229-12-145
收到了:
公认:
发表:
关键词
- 组蛋白乙酰转移酶
- 激素
- 种系发生树
- 亚细胞本地化
- 大米
- 压力