抽象的
背景
多倍体化是植物基因组进化的主要机制之一。同一基因的多副本的存在减少了选择压力,允许亚功能化和新功能化,从而导致植物多样化、适应和物种形成。在面包小麦中,多倍体化和转座因子的流行导致了大量的基因复制和移动。结果表明,小麦中与相关物种基因组非共线的基因数量明显增加。
结果
我们使用了新一代测序(NGS)来从小麦染色体臂3ds产生MB大小区域的序列。24个BAC克隆的序列组装导致两个支架为1,264,820和333,768个碱基。序列被注释并与小麦染色体3b上的同种型区域进行比较Broachypodium distachyon.和米饭。在3DS支架中的39个编码序列中,32在染色体3B上具有同种型。相比之下,在大米和稻米的相应区域中仅鉴定十五和十四天的原因毛花瓣, 分别。有趣的是,在3B基因座的非共线编码序列中鉴定了五个伪原,而在3DS基因座中没有发现。
结论
直接比较B面包小麦的B和D基因组的两种MB大小区域揭示了两种基因组中的非共聚物基因插入的类似速率,其中大多数基因重复发生在其发散之前。在非共线编码序列中鉴定了相对较低比例的假序。我们的数据表明,伪原不源于插入非功能性拷贝,而是在六倍体小麦的演变过程中稍后形成。沿B基因组基因座的基因侵蚀发现了一些证据。
背景
多倍体被认为是植物演化和形态的主要驱动力之一。全基因组复制(WGD)为植物基因组进化,多样化和适应提供了一种基材。相同基因的两种或更多种拷贝的存在降低了选择压力并实现了亚官能化和新功能化。全基因组序列的分析显示出在高等植物演变期间的频繁和经常反复发生的基因组倍增。甚至具有相对较小的基因组的植物,如拟南芥蒂利亚纳Broachypodium distachyon.和马吕斯有明显在他们的演变过程中经历过多倍化事件[1-3.].
一些全基因组复制事件发生在数千万甚至数亿年前(MYA)。这些古倍化事件不易用细胞遗传学方法检测,但可以在序列水平上进行鉴定。除了古倍性外,一些植物如面包小麦(Triticum Aestivum.最近经历了基因组复制。在这种情况下,除了一些染色体间重排外,完整的同源染色体仍然存在,没有明显的结构变化[4.].通过两个不同的杂交事件形成六倍体面包小麦(2n = 6x = 42,12,17gB)的基因组。第一次发生0.2 - 0.5 mya [5.]并涉及二倍体野生物种T. Urartu.(AA Genome)和与之相关的二倍体物种Aegilops Spottoides.(SS Genome)。此事件产生了同种异体一体化t . dicoccoides,栽培的硬质小麦的祖先t . turgidum用AABB基因组。只有大约10,000年前,D基因组的二倍体供体(AE。Tauschii.)与之杂交t . dicoccoides产生培养的祖先Triticum Aestivum.L.与AABBDD基因组[6.].
除了WGD,串联和分散的个体基因和基因簇的重复涉及基因组进化。这些事件破坏了相关物种的基因组之间的共同性,并限制了小型基因组的模型物种的潜力作为基因映射和克隆与复杂基因组的作物克隆。导致基因重复的机制仍然无法清楚地理解,但转换元素(TES),其占小麦基因组的85%以上可能发挥关键作用。TE Activity可以通过至少两个机制导致基因复制。首先,可以通过Te捕获基因并与可转换元件一起复制到新位置。TES对捕获基因和基因片段的能力在植物中有很好的记录[2那7.那8.].与TE活性偶联的基因复制的另一种机制基于通过合成依赖性链退火(SDSA)的双链断裂修复,其伴随TE插入[9.].在这种情况下,包含一个基因的序列片段被用作填充DNA来修复双链断裂,这是在填充目标位点复制过程中发生的。te介导的基因运动导致基因在非共线位置的插入。最近的研究表明,小麦染色体与两种近缘禾草的同线区域共线性毛花瓣而且大米的产量比之前估计的要低[10.].已经提出,越多的非共链基因来自TE活动的结果[10.那11.].但是,Choulet.等等。[10.]和柳条等等。[11.在估计的非共线基因插入率方面存在差异。虽然Choulet等等。[10.]建议在面包小麦和大麦的三种基因组中相同的非共聚物基因,柳条等等。[11.染色体1染色体中的基因插入率的显着差异。
基因损失是另一种机制,导致观察明显非共线基因。巨大的基因损失可以伴随着记录的染色体重新安排拟南芥[12.].损失可能是显着的,如[13.谁观察到多倍化后约500万年内从玉米两种祖细胞的50%的基因损失。在拟南芥中,在过去1000万年的进化期间,几乎一半的基因组被丢失了[12.].在更新的同型异丙醇中还描述了来自一种同源型段的基因的丧失,例如拟南芥suecica和Tragopogon Mirus.[14.那15.].因此,在毛花瓣在古聚百倍化后的米饭可能是小麦中非共线基因数量的表观增加,即使基因留在小麦的祖先位置。
虽然先前的报告确定了推动植物基因组的演变的主要机制,但需要额外的大规模基因组序列来研究非共链基因频率,重复机制和非共线基因功能。由于其最近的进化历史,六倍体小麦是研究这些过程的良好模型。由于小麦基因组的测序被多倍体和基因组大小(17GB)阻碍了,国际小麦基因组测序联盟(IWGSC)采用了一种基于染色体的方法,依赖于来自分离的染色体和染色体臂的物理图的构建[16.].在小麦中,染色体臂占整个基因组的1.3 - 3.4%,因此染色体基因组学大大降低了样品的复杂性。染色体分离和流式细胞分选的方法在小麦中已经建立[17.那18.]和BAC文库,由所有染色体武器构建[19.,被证明适合构建序列就绪的物理图[20.].
该研究的目的是检查六倍体小麦的同种型基因组的特征是否具有相同的非共线基因插入速率,并且如果正交基因保持相同的活性。我们使用了可用的染色体特定于面包小麦的特异性BAC库,并在染色体3B上以CONTIG CTG0954B进行染色体3D上的轨迹。CONTIG CTG0954B(〜3.1 MB)含有53个编码序列,并显示出高度的非相连性毛花瓣(62%的CDS是非线性共线的)[10.].由于其特征,轨迹出现了合适的候选者,用于比较六倍体小麦的同种型基因组中的非共线基因插入。我们在对应于ContiG CTG0954B的一部分的染色体臂3ds上识别并测序1.6 MB区域,并通过基因比较基因含量,基因。这首先比较了六倍体小麦的MB大小正交区域,揭示了B-和D-基因组基因组中的非共线基因插入的类似速率。在两个基因座之间共享大约三分之二的基因插入,而剩余的基因运动在普通祖先的分歧之后发生。此外,与D基因座相比,我们的结果表明沿B基因座的基因表达的减少。
方法
选择BAC克隆用于测序
使用PCR筛选小麦染色体臂3DS的物理图的最小平铺路径(MTP; 384-孔板中排序的克隆),以选择与3b contig ctg0954b同源的Bac克隆。准备了三维MTP池,使得十个板池代表单个板,16个行池由所有十个板的特定行组成,24个柱子池由来自十板的特定列的克隆组成。使用Genetac G转移每个池的BAC克隆3.机器人工作站(基因组溶液)在固体2YT培养基上并在37℃下孵育16小时。然后将细菌菌落洗涤至液体2YT培养基,并在250rpm和37℃下孵育8小时。通过标准碱性裂解分离细菌DNA,并进行微小的修饰。PCR反应含有1×PCR缓冲液,0.2mm DNTPS,0.5u Taq聚合酶,0.01%甲酚红色,1.5%蔗糖,特定池20ng DNA和1μm引物(用于引物列表,请参阅附加文件1:表S1)。PCR在C1000触摸热循环仪(BIO-RAD)中进行,如下:95℃的初始变性5分钟;35循环为95℃,30秒,60℃,30秒,72°C为30秒;最终延长在72°C 5分钟。将PCR产物在1.5%琼脂糖凝胶上分离,用溴化乙锭染色并在Genius Syngene Bio成像系统(Syngene)中可视化。阳性克隆的BAC地址从阳性水池使用大象软件 [21.].选择覆盖与CTG0954对应的区域的一组24个BAC克隆用于测序。
测序BAC克隆
使用适应的MIDI-PREP协议分离来自24个BAC克隆的DNA至3B CONTIG CTG0954B [22.].所有无性系分别培养和分离。在37℃下10ml 2YT培养基中孵育过夜,按标准碱性裂解程序分离[22.].平均700ng DNA的分离产生。为了准备配对结束的文库,在DNA分离后合并所有克隆的等分试样。合并DNA的量为每BAC克隆600和200ng,分别用于制备8kb和3-kb对末端文库。总共为单个克隆创建了24种未配对的文库,从代表所有24个BAC克隆的合并DNA中创建3 kB和8-KB成对末端(PE)文库。使用454 / Roche钛霰弹枪或标准配对端测序试剂盒单独处理每个图书馆,并根据制造商的规格在454 / Roche GS FLX测序仪上测序。原始序列数据以序列读取存档存放(研究登录号:ERP001545)。
序列组件
新手汇编程序(版本2.0)用于自动组件。首先,使用针对大型复杂基因组优化的算法和以下(默认)设置:最小读取长度 - 20bp;种子步骤 - 12;种子长度 - 16;种子计数 - 1;最小重叠长度 - 40;最小重叠身份 - 90%;对准身份得分 - 2;和对准差分分数 - 减去3.这些组件导致无序的Contigs。使用与在支架中排序的各个BAC克隆的单独的BAC克隆组装在一起组装在一起组装在一起的汇集克隆的3-KB和8-KB对末端读数。 Following this, the assembly was manually edited. Full sequencing reads (not clipped by assembler) at the end of individual scaffolds were compared to small repetitive contigs using爆炸(最小的身份:99%;最小对齐长度:100 bp)。当两个支架匹配同一contig的两端时,用contig序列将它们合并并闭合间隙。用类似的方法封闭支架上的缝隙。所有的手工编辑都是用Geneskipper(Embl,Heidelberg)。通过桑格生成的Bac-End序列的比对选自支架中的单个BAC克隆的序列(Bartoš等等。,未发表的脚手架使用爆炸。
序列注释
半自动管道TrianNot用于序列注释[23.]并诠释了诠释阿尔忒弥斯V.11 [24.].BAC克隆单独注释,然后合并以产生两个大支架的注释。使用的比较BAC克隆的序列重复克斯勒(http://repeatmasker.org)到由TREP建立的内部重复元素数据库(http://wheat.pw.usda.gov/itmi/repeats/)来自小麦染色体3b上的重复[10.].然后使用D策划基于转换元件的重复的转换元件注释獭[25.]在可能的情况下检测元素的确切边界。当识别目标站点重复(TSD)和Long终端重复(LTR)或终端反转重复(TIRS)时,该元素被认为是“完整”。最后,根据Choulet中描述的标准对所有重复元素进行分类等等。[10.]跟随柳条的命名等等。[26.]。
使用相似性搜索爆破[27.)对Broachypodium distachyon.和奥雅萨苜蓿注释的基因和蛋白质,ests和ungenes可供选择Triticum Aestivum.大麦芽和所有其他Poaceae,最后,针对小麦和大麦的全长CDNA。对于相似性搜索对成绩单,重要的爆炸然后恰好对齐GMAP.[28.]识别拼接网站。所有分析的结果都被写为embl格式的序列特征,并组合阿尔忒弥斯设计最可能的基因模型。在数据库中显示过早止损密码子,越来越突变或缺失的基因模型,可在数据库中的完全同源物的30%被视为假生素。
序列比较正交座位毛花瓣和米饭和3b染色体
我们使用NCBI爆破[27.]的默认设置,但E-value被设置为1e-10,对于所有同源性搜索3ds和3b基因套和米饭毛花瓣编码序列。首先,比较3DS基因座上的所有(伪)基因的编码序列爆炸从3b contig ctg0954b [genbank:fn564434]的基因[10.鉴定了3B的特异性同源物。然后,将3ds基因在3b基因中鉴定出没有同源物的基因与3b contig ctg0954b的完整序列进行比较,以解决两个基因座之间的注释中的不一致。以类似的方式,搜索3DS基因中没有同源物的3B基因被搜索到完整的3DS基因座中。然后将两个基因组与编码序列进行比较毛花瓣(BRADI1.0基因集;http://www.bachymodium.org/)和水稻(RGAP6.1基因集)[29.以确定在毛花瓣和水稻基因组。为了详细比较基因 - 假蛋白对,选自3ds基因座,跨越基因加1kb和下游序列的序列区域。含有伪基因的相应序列选自3B基因座。比较使用artemis比较工具[30.].
启动子和表达分析
我们在上游提取了1 kB序列以启动密码子,用于在3ds-and 3b特定位置处找到的每个编码序列。我们使用TSSP预测聚合酶II启动子,转录起始位点和塔塔箱位置[31.] 在http://linux1.softberry.com/berry.phtml.。1,071,335 ests在Genbank公开提供(http://www.ncbi.nlm.nih.gov/genbank/)用于CD的表达分析。独立爆炸程序用于执行具有以下参数的相似性搜索:核苷酸匹配2的奖励;期望值1E-10;和所有其他参数的默认设置。只考虑100%同一性和最小长度为100 bp的爆炸击中。
结果与讨论
选择BAC克隆用于测序
在染色体臂3ds的物理图建立期间建立了最小平铺路径(MTP)(Bartoš等等。,未发布)使用Taacsp3dsha 3ds特定的bac库[19.].构建了包含3827个克隆的MTP三维文库,并使用来自ESTs的12个标记进行筛选(见附加文件)1:以前映射到完全测序和注释的染色体3B的3 MB区域的表S1)(CTG0954B,[GENBANK:FN564434])[10.].4个标记分别鉴定出单克隆,其余标记分别鉴定出2个克隆。在5例病例中,2个阳性BAC克隆在3DS上属于不同的contig,表明contig重叠。在一个案例中,重叠得到了三个分子标记的支持。在3DS上,我们总共发现了三个重叠的部分。序列组装进一步证实了这些重叠。共鉴定到8个同源于3B contig ctg0954b (contig合并前)的contig。这些contigs的MTP由24个BAC克隆组成1)由454个GSFX钛技术测序。
霰弹枪BAC测序和装配
从24个BAC中的每一个产生了454个未配对的读数。这产生了3,418至46,129质量大肠杆菌通过BAC克隆过滤读数。总共产生了592,593读数,平均读取长度为328bp。将它们组装成13至155个大的折叠(> 500个碱基),平均每克隆平均为35个折叠(附加文件2:表S2)。获得最低数量的大角形,用于组装Bac克隆Taacsp3dsha_0065014,其具有最小的插入物(66.8kb,与所有24个bacs的平均插入尺寸为114.8kb;表2).经测序的BAC克隆的N50值为913 ~ 13617 bp。使用未配对序列读取获得的BAC序列组装的质量较低,多个组装导致50多个无序contigs和N50值较低,与克隆插入相比。显然,在像小麦这样富含TE的大基因组中,不配对短读测序无法提供高质量的组装。新手汇编器,用于在所有情况下在所有情况下产生组装,碎片序列,当重复元素太类似于被正确区分时。为了获得3ds Condigs的参考序列,我们决定使用配对末端读取将所有BAC克隆排序。
合并的BAC的配对组装导致两个脚手架
合并的BAC DNA的两次配对末端测序运行(3-KB和8-KB)总共产生686,372质量大肠杆菌过滤。组装与新手制作了282个大折叠和33个脚手架。最大的脚手架由548,429个碱基组成。在霰弹枪组件的情况下,TES表示装配的主要问题。然而,使用配对终端图书馆允许桥接有问题的区域并建造的脚手架明显长于霰弹枪组件的Contig。通过手动修订和差距关闭,组装中的折叠数量减少到两个支架中的62个折叠。由于支架末端的重复元素可能在手动合并期间导致犯散,我们将支架末端与Triticeae数据库进行比较,而是对核心重复的数据库(http://wheat.pw.usda.gov/itmi/repeats/).在66个支架末端中,八个具有超过95%的身份(数据未显示)的至少100bp击中。根据这些结果,我们为结束合并选择了严格的条件(参见方法)。基于组装从组件中提取24个单独的BAC的序列爆炸搜索可用的BAC结束序列(BES)。如预期的那样,许多BAC序列重叠,并且可以分别排序成2个脚手架A和B,分别与两个支架A和B相对应的BAC凸片,分别有18和5 bacs(表2).为了测试轨迹的覆盖范围,序列读被映射到组装序列。结果表明分布均匀(附加文件3.:图S1)。
其中一个BAC (TaaCsp3DShA_0061C24)保持单身,没有任何邻近的克隆。该克隆被从contig ctg801的物理映射中删除,并排除在进一步分析之外。此外,序列比较还发现了在物理图谱组装过程中没有发现的克隆重叠。利用这一信息,6个和2个物理地图contigs分别被合并到contigs ctg447和ctg1484中,从而得到该区域的2个物理地图。支架A [EMBL:HE774676]和B [EMBL:HE774675]的序列分别对应物理图谱contigs ctg447和ctg1484。
因此,使用对端读取导致contig的数量减少了13倍(从829减少到62),并显著改善了24个BACs的contig排序。经过共识构建,支架A和B的序列在40个contigs中有1,264,820个碱基,在22个contigs中有333,768个碱基,N50分别为48,251和22,881 bp。4个克隆组装在一个单独的contig中,另外4个克隆的序列只包含一个空白。大会所有空缺(40/60)的三分之二出现在两个特定的重复区域。26个缺失发生在逆转录转座子的ltr内,14个缺失发生在CACTA元件的蛋白编码部分。其余的缺口很可能包含GC岛的核心,在乳剂PCR中没有充分扩增。这些结果证实了配对末端阅读对小麦基因组序列的组装是绝对必要的,至少8kb的文库对跨越整个TEs是重要的。尽管在测序的contigs中有三分之二的序列是可移动的DNA元件,其中一些存在于10多个拷贝中,并且具有高度相同的ltr -反转录转座子长末端重复,但我们的组装质量与之相当Cucumis Melo.只有7.2%的重复内容[32.].在类似的实验中[33.[测序六个池,由物理图组成26-28个顺序BACoryza barthii.。最终组装o . barthii游泳池含有九点至31个无序脚手架。我们基于MATE对读取的方法导致组装类似数量的脚手架(33),其通过手动编辑进一步减少到两个。
遗迹组成反映了整体小麦D基因组含量
组装序列的注释鉴定了总共1,059,398bp(66.3%),作为重复景观的一部分。这个空间中的大部分(33.4%)被LTR逆机的吉普赛超家族占据了,这是识别所有超自府中最常见的常见(有关详情见表3.).无法将大约12%的重复空间分配给任何已知的TE超家族。另外,在两个支架中鉴定了覆盖57,167bp(组件的3.6%的组装)的39个编码序列(CDSS)。
3ds基因座(58.3%)中“已知重复”的估计分数与D型基因组祖细胞祖的BAC结束序列后观察到的56.8%完全一致AE。Tauschii.[34.].相比之下,李等等。[13.]报告了70.7%的已知重复AE。Tauschii.基因组通过质粒结肠测序。然而,目前尚不清楚,无论它们是否在核苷酸或读数方面都会计数重复元素,这可能会偏离结果。最近,你等等。[35.]测序的基因组AE。Tauschii.到1.35倍覆盖454,估计重复占基因组的59%。编码序列在我们的序列数据集中代表3.6%的碱基,这个值介于Li估计的2.5%和4.8%之间等等。[13.]和Akhunov.等等。[34.], 分别。因此,我们得出结论,HGA 3DS基因座的组成反映了面包小麦D基因组及其祖细胞的一般特征AE。Tauschii.。
Hga 3DS位点基因间的平均距离为25.3 kb(中位数为17.7 kb)。我们还估算了3B染色体同源区域的基因密度,以比较基因间区域的基因组扩展程度。假设三个小麦基因组(由亲缘关系密切的二倍体进化而来)的基因含量相同,那么B基因组的基因密度应该低于D基因组。因为B基因组比D基因组大大约27% [19.[该特定染色体3B基因座(21.8kb)的基因距离中位数与基因组大小相一致。然而,由于染色体3b的中值距离估计致两倍的基因组(42.5kb),基因组似乎富含基因,基因组似乎是基于175个基因的两倍较大(42.5kb)[10.]与小麦中的焦化区域相比,反映了端粒的基因密度略高于粒子[10.那36.].
在3DS和3B染色体同源位点的非共线基因插入率相似
在染色体3b染色体座位上发现总共38个CDS [Genbank:Fn564434] [10.].在3ds基因座的共线位置发现它们中的三十个(84%)。其他五个是3B基因座的特异性。最后一个CD是在3ds轨迹上的两个3ds contigs或缺失之间的间隙中。由于无法确定其存在或不存在,因此CD被排除在进一步的比较之外。相反,七个CD只能在3DS基因座(图1).然后,我们将特定于一个基因座的CD与其他轨迹的完整序列进行比较,以揭示可能的注释差异。爆炸对3B ctg0954b全序列在3DS上注释而不在3B上的CDS进行搜索,发现了一个与Taa3DS_ctg447.00270同源的额外基因片段(~500 bp),本研究命名为TAA_ctg0954b.0415。针对完整的3DS轨迹反向搜索3B特定的CDS并没有在3DS上发现任何额外的CDS。另外,两张CDS (TAA_ctg0954b.)00216和TAA_ctg0954b.0217)是Taa3DS_ctg447.00040同源的单个CDS片段,命名为TAA_ctg0954b。00216用于合并CDS。经过注释修改,在3B位点上共鉴定出38个CDS。
在3DS轨迹处发现的所有CDS也都进行了比较爆炸针对稻米的完整基因模型毛花瓣下载了ftp://ftp.plantbiology.msu.edu/pub/data/eukaryotic_projects/o_sativa/annotation_dbs/(RGAP 6.1)和http://ftp.brachymodium.org/(bradi_1.0)。每种物种中最好的匹配基因模型被认为是同源物。染色体与大米和小麦组3中的染色体毛花瓣是OS01和Bradi2,分别[2那37.].对应于小麦3B和3D测序基因座的区域是BRADI2G01380 - BRADI2G01590毛花瓣Os01g02900 - Os01g03110。在相应的区域分别发现了22和21个基因毛花瓣和米饭分别(图1).在所有四个同义区域(沸腾,大米和小麦B和D基因组)的共线位置中发现了十四个。在水稻和小麦B和D基因中发现了一种额外的基因,但不是犁囊,建议在稍后的物种中删除。共线基因占编码序列的63.6%和71.4%毛花瓣分别和大米。总的来说,这些结果证实了早期的观察毛花瓣水稻对比较基因组应用同样有用[38.].相反,这些共线基因在3DS和3B基因座的小麦中仅占38.5%和39.5%的编码序列。
小麦中的非共线基因分为两组。第一个组合的基因在两个小麦座位之间分享,但在同步位置缺失毛花瓣和米饭。非共线基因的四分之三(17)落入了这一类。第二组包含在3ds(7)或3b(5)基因座的基因。三个和两个基因分别从3DS和3B基因座的串联重复产生。序列相似度最高的基因副本(爆炸)到外科基因毛花瓣水稻被认为是共线的,而保守程度较低的基因被认为是非共线的。
我们的数据与观察到的非共线基因插入率在二倍体小麦祖先AE。Tauschii.[38.]和六倍体小麦[10.那11.]比非小麦科禾草品种要高。然而,两项对六倍体小麦的研究在同源基因组中估计的基因插入率存在差异。目前的结果支持Choulet的结论等等。[10.[指示小麦B和D基因组的相同数量的非共线基因插入。我们的结果还表明,当用完整序列而不是通过定义不完全进行的调查序列进行时,对基因组进化的研究更加稳健,并且在确定基因损失和其他结构基因组时可以引入偏差。
柳条在al.[11.]提出了基因运动/插入的两种机制:双链修复和TE介导的转移。乔莱也提出了后者等等。[10.他将最近TE的激活与六倍体小麦二倍体祖细胞中更多的重新定位基因联系起来。通过TEs获得的基因/基因片段也被记录在其他草的基因组中,如水稻、玉米、高粱和玉米毛花瓣[7.那8.那39.那40].具有获取基因片段的能力的特点包括包骡子[7.],Helitrons [8.], CACTA [10.那39.那40[],预兆40],DNA转座子和LTR回收转换[41.].TE活动的一个副产品是目标区域的扩展。然而,D基因组比B基因组小[19.,表明该基因组的TE活性较低,因此非共线基因较少。同源B和D基因组位点中非共线基因的数量相当,这表明要么是B位点上的TE降低了基因移动的效率(即每次TE插入的基因移动),要么是D位点上的其他机制在基因移动中的作用增加。然而,尽管mbp大小,本研究分析的位点相对较小,我们的观察需要在比较较大的同源小麦基因组区域后加以证实。
加速基因转移伴随多倍化吗?
虽然在没有捐赠者和受体序列的知识的情况下难以进行约会复制事件,但可以完成一些估计。Choulet.等等。[10.]发现,最重新安排的小麦染色体3B区域,包括CTG0954B,最近的TE活动(平均每年1.0-140万年),而最古老的TE活动(1.9 - 2.1)的地区最被保守。因此,大部分插入应该是最近的。
但是,我们的结果表明,三分之二的基因非共线毛花瓣和米饭在两颗小麦基因组之间共享。因此,不必在普通祖先的所有小麦基因组分散之前插入非共线基因约2.5-4.5 mya [42.]甚至在大麦谱系10 mya的分歧之前[5.由于部分基因在同源大麦Rph7位点保守[GenBank:AF521177] [10.那43.].因此,这些基因的重新定位不能与最近的TE活性联系起来。其余三分之一的非共线基因(分别有7个和5个在3DS和3B位点)必须在小麦B和D基因组从一个共同祖先分化后插入,大约在2.5 - 4.5 MYA [42.].
在30次进化后插入了三分之二的基因的事实毛花瓣和小麦谱系(35 mya [44.[]),并且有三分之一的基因是在过去的2.5 - 4.5 MY期间插入的,这表明小麦二倍体祖分化后的基因插入率提高了3 - 5倍。在此期间,唯一能刺激基因运动的重要事件是种间杂交。赖等等。[13.与其二倍体祖细胞相比,检测到多倍体玉米基因组中基因运动和损失的显着加速。使用祖先(1-200万岁),人为生成Gossypium.杂种,鞭毛等等。[45.]估计表达变化的¼与异源多倍体有关。在这里,如果不从小麦基因组的祖先以及小麦基因组本身获得额外的序列,就不可能了解伴随杂交和多倍体化的基因运动。
与3b相比,在3ds轨迹处发现少的伪原
柳条等等。[11.[认为,许多非共线基因是假生素或基因片段。在这里,我们发现在3DS基因座上的39个编码序列中仅有两个明显的伪原(参见定义方法)(TAA3DS_CTG1484.00030和TAA3DS_CTG1484.00050)中的两个明显的伪原(参见定义方法)。此外,其中一种假期引发物在3个基因组的所有四个区域中处于完美的共线位置,而第二个是串联重复(见图1).有趣的是,3B基因座仅携带这种特定假蛋白(TAA_CTG0954B.00150)的一种拷贝,从而表明在基因函数丧失和小麦基因组祖细胞之间的发散后发生重复。相反,在3b上相同数量的Cds鉴定六个伪原和基因片段。第一个是TAA3DS_CTG1484.00030和TAA3DS_CTG1484.00050的同源物,因此是线性的毛花瓣和米饭。剩下的四个具有完整兽人的3ds的同源物,而最后一个是特定于3b基因座的。因此,在3ds基因座的非共线基因中没有发现假基因,而在3b上发现的六个伪原因/基因片段中的五个是非共线位置的比较毛花瓣和米饭。伪原因/基因片段在3B基因座处表示小部分的非共线基因(5/22)。总而言之,这些结果表明,大多数如果不是存在于3B和3D正交基因座上的所有非共链基因,则不显示伪原的结构特征。
插入后的假基因形成
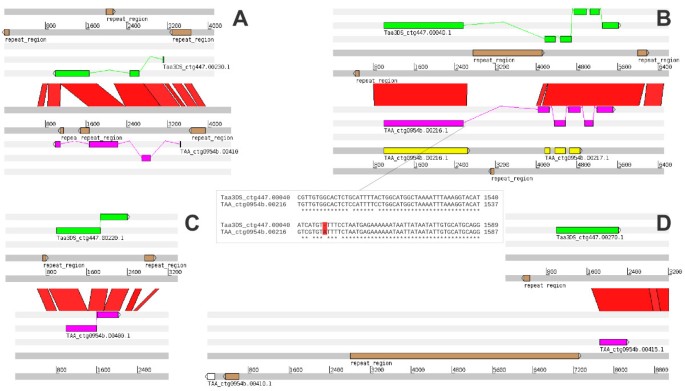
伪原的起源是什么?柳条等等。[11.[提出了许多小麦伪原从不完美序列的插入,通过双链断裂修复或TE捕获。然而,在3B基因座上发现的四个伪原在3DS基因座上具有完全ORF的同源物,从而仅在插入后构成假基因形成。四种基因(3DS) - 假基因(3B)对的详细比较(图2)通过在其中一个外显子(图中,通过插入Te插入形成一个假基因(Taa_ctG0954b.00410)(图2a)并且另一个(Taa_ctg0954b.00216)从由单个核苷酸插入引起的帧间移位突变产生(图2b)。两个剩余的CD(taa_ctg0954b.00400和taa_ctg0954b.00415)(图2在分次缺失编码序列后,C和D)被伪原因。
基因 - 假蛋白同源对的比较。完整的编码序列(来自3DS轨迹)的全部编码序列是在绿色中突出显示的上迹线。粉红色的较低迹线显示伪原(来自3b基因座)。棕色显示的序列功能代表重复元素。痕迹之间的红色连接表示爆炸序列之间的命中。一种) TAA_ctg0954b最后一个外显子插入重复元件。00410基因导致其假殖化。B.)伪影TaA_CTG0954B.00216原文为两种不同的基因片段(黄色特征)。与原始3DS基因的比较使其注释为一个假基因。序列比对显示出单核苷酸的插入导致框架移位(序列切出)。剩余的两个基因taa_ctg0954b.00400(C)和taa_ctg0954b.00415(D.)通过部分删除丢失了他们的功能。虽然内部删除破坏了基因TAA_CTG0954B.00400(CTaa_ctg0954B.00415的5'结束(D.) 去掉了。在最近基因的上游发现没有与3DS原始基因TAA3DS_CTG447.00270.1的同源性(白色的最左图)。
这些结果表明,在形成全多倍薄片之后,伪原子化主要发生。首先,基因在多倍化后不受强烈的选择。其次,在3b基因组中发现所有伪原,这可以通过与D基因组相比在多倍体上下文中花费的B基因组的较长时间来解释。伪果序列可以在延长的时间段内被侵蚀并丢失。在玉米和玉米中发现这种基因损失拟南芥在过去五年和一百万年后,多倍化事件分别[12.那13.].在我们的情况下,3bs伪原中的两种未删除,但在杂交之间的杂交〜50万年后在某种程度上降解小麦属植物urartu和Aegilops Spretoides.。
非共线基因携带启动子序列,很可能表达
启动子区域对驱动基因表达至关重要,许多伪基因实际上缺乏启动子或具有非功能序列[46.].我们搜索了同源型3ds的引擎样Cds上游的促进剂样序列1 kB,3bs基因座提供进一步支持的结论,即所研究的基因座的大多数非共线Cds不是假期原因。转录开始部位(TSS)分别在3DS和3B轨迹处鉴定54.2%和54.5%的非共线基因(附加文件4.:表S.3.).发现了一个TATA盒,其中大部分是TSS上游14-38 BP。在共线基因中,将TSSS鉴定为约73%。这些发现表明,并不多的非共链Cds缺乏启动子序列,因此可以表达其中大部分。迄今为止,只有大约16,000个小麦全长CDNA公开可用,从而限制了识别完全表达基因的可能性。结果,只有编码序列可以在本作工作中注释,而TSP预测的转录开始位点不能与FL-cDNA的5'末端进行比较。
然后利用GenBank中公开的小麦est对两个同源位点的基因表达进行评估。在3DS和3B基因座中,采用100%同源性的严格标准对匹配每个基因的ESTs数量进行评分。4个est与同源基因配对,从分析中删除。在3DS中41.7%的非共线基因在EST集合中至少有一次命中(附加文件5.:表S4)表示其表达。在共线位置发现的基因中表达基因的比例略高(53.3%)毛花瓣和米饭。类似地,3B基因座的共线基因更常见地表达(33.3%vs.2.7%)。有趣的是,3B基因座中的基因似乎比同源3DS基因座的基因较少。我们对表达不同的每对同种型基因进行了两次尾型二项式试验。不幸的是,3ds基因的表达水平明显高于两种情况下的3B对应物。在小麦组1染色体上的基因也发现了类似的非共线基因表达的减少(柳条等等。2011)。如在3B基因座在3B轨迹的较高份数的情况下,与D基因组相比,B基因组在多倍体小麦基因组中的较长共存可以解释表达基因的数量减少。
结论
充分的基因组覆盖和良好的序列组装是研究植物物种形成和驯化伴随的基因组进化变化的前提。我们对面包小麦B和D基因组的两个mb大小区域进行了直接比较,发现两个基因组的非共线基因插入率相似,大多数基因重复发生在它们发生分化之前。非共线编码序列中假基因的比例相对较低,说明假基因化并不伴随基因插入事件,而是在多倍体基因组进化过程中发生的较晚。全染色体测序正在IWGSC (wheatgenome.org)的保护下进行,完整的参考染色体序列的可用性将有助于证实这些发现,并进一步深入了解涉及形成多倍体小麦基因空间的机制。这些观察结果将得到RNASeq研究的支持,以评估同源拷贝的相对表达以及相关的结构和功能信息。
参考
- 1。
拟南芥基因组倡议:分析开花植物拟南芥的基因组序列。自然。2000,408:796-815。10.1038 / 35048692。
- 2。
国际毛细血管倡议:模型草原桥梁脱位的基因组测序和分析。自然。2010年,463:763-768。10.1038 / Nature08747。
- 3.
Velasco R,Zharkikh A,Comougtit J,Depouttit J,Destaro A,Kalyanaraman A,Fontana P,Bhatnagar Sk,Troggio M,Pruss D,Salvi S,Pindo M,Baldi P,Castelletti S,Cavaiuolo M,Coppola G,Coda F,Cova v,Ri Ad,Goremykin V,Komjanc M,Longhi S,Magnago P,Malacarne G,Malnoy M,Micheletti D,Moretto M,Perazzolli M,Si-Ammour A,Vezzulli S,等:驯化的基因组Apple(Malus x Domestica Borkh。)。NAT Genet。2010年,42:833-839。10.1038 / ng.654。
- 4.
Devos Km,Dubcovsky J,Dvorak J,Chinoy CN,Gale Md:小麦染色体4a,5a和7b的结构演变及其对重组的影响。Al Appl Genet。1995,91:282-288。10.1007 / BF00220890。
- 5。
DVORAK J,Akhunov ED:基因基因座缺失和重复及其与Aegilops-Triticum联盟中二倍体和多倍体演变期间的rRecombination率的重复及其关系。遗传学。2005,171:323-332。10.1534 / genetics.105.041632。
- 6。
Feldman M:栽培小麦的起源。世界小麦书:小麦养殖的历史。编辑:Bonjean AP,Angus WJ。Lavoisier Publishing,Paris,2001:3-56。
- 7。
Hanada K,Vallejo V,Nobuta K,Slotkin RK,Lisch D,Meyers BC,Shiu S-H,江N:净米在米净化选择和表达剖面中推断的功能作用。植物细胞。2009,21:25-38。10.1105 / TPC.108.063206。
- 8。
Yang L,Bennetzen JL:玉米基因组中陆螺胶的分布,多样性,进化和生存。pnas。2009,106:19922-19927。
- 9。
柳条T,Buchmann JP,Keller B:在植物基因组中修补间隙导致基因运动和侵蚀的热敏性。Genome Res。2010年,20:1229-1237。10.1101 / GR.107284.110。
- 10。
Choulet.F, Wicker T, Rustenholz C, Paux E, Salse J, Leroy P, Schlub S, Le Paslier M-C, Magdelenat G, Gonthier C, Couloux A, Budak H, Breen J, Pumphrey M, Liu S, Kong X, Jia J, Gut M, Brunel D, Anderson JA, Gill BS, Appels R, Keller B, Feuillet C: Megabase level sequencing reveals contrasted organization and evolution patterns of the wheat gene and transposable elements spaces. Plant Cell. 2010, 22: 1686-1701. 10.1105/tpc.110.074187.
- 11.
柳条T,Mayer Kfx,Gundlach H,Martis M,Steuernagel B,Scholz U,ŠimkováH,克巴拉沃哈米,Chooulet F,Tudien S,Platzer M,Feuillet C,Fahima T,Budak H,DoleželJ,Keller B,Stein N:频繁的基因运动和伪基因进化对于小麦,大麦及其亲属的大而复杂的基因组是常见的。植物细胞。2011,23:1706-1718。10.1105 / TPC.111.086629。
- 12.
prost S, Pattyn P, Gerats T, Van de Peer Y:穿越过去的旅程:1.5亿年的植物基因组进化。植物学报,2011,66:58-65。10.1111 / j.1365 - 313 x.2011.04521.x。
- 13。
莱吉,马杰,斯瓦戈拜诺瓦齐,兰卡里希纳W,林顿e,llaca v,tanyolac b,park y-j,jeong o-y,bennetzen jl,杂乱j:基因损失和玉米基因组的运动。Genome Res。2004年,14:1924-1931。10.1101 / gr.2701104。
- 14。
Chang PL,Dilkes BP,McMahon M,Comai L,Nuzhdin SV:同源物学特异性保留和在同源物中的保留和使用在同源物中拟南芥Suecica取决于原籍和网络合作伙伴的父母。基因组Biol。2010,11:R125-10.1186 / GB-2010-11-12-R125。
- 15.
Koh J,Soltis PS,Soltis de:最近和反复形成的异常形状的异丙龙米苏(菊科)的自然群体的自然群体变化。BMC基因组学。2010,11:97-10.1186 / 1471-2164-11-97。
- 16。
DoleëelJ,Kubalákovám,Paux E,BartošJ,Feuillet C:基于染色体的谷物基因组学。染色体res。2007,15:51-66。10.1007 / s10577-006-1106-x。
- 17。
Vrana J,Kubalákovám,Simkováh,CíhalíkováJ,LysákMa,Dolezel J:常见小麦的有丝分裂染色体的流动分类(Triticum Aestivum L.)。遗传学。2000,156:2033-2041。
- 18。
Kubalákovám,vránaj,číhalíkováj,Šimkováh,doleželJ:在面包小麦(Triticum Aestivum L)中的流量核型和染色体分类。Al Appl Genet。2002,104:1362-1372。10.1007 / s00122-002-0888-2。
- 19。
ŠafářJ,Šimkováh,克巴拉沃哈米,ČíhalíkováJ,如此,BartošJ,DoleželJ:用于面包小麦基因组的特定染色体的BAC资源开发。CytoOgenet基因组Res。2010,129:211-223。10.1159 / 000313072。
- 20。
Paux E, Sourdille P,泥火山J, Saintenac C, Choulet F, Leroy P,各地,旧事的M, Kianian年代,Spielmeyer W, Lagudah E,萨默斯D, Kilian,塞德cedreic Alaux M,魏特琳年代,berg H, Eversole K, appel R,回历2月J, Simkova H, Dolezel J,伯纳德·M Feuillet C:物理地图1-gigabase面包小麦染色体3 b。中国科学:地球科学。10.1126 / science.1161847。
- 21。
Paux E,Guilhot N,Adam-Blondon A-F,Alaux M,Salse J,Sourdille P,Leroy P,Feuillet C:大型基因组的物理映射:在硅分析中加速Bac Contig的锚固到遗传地图。Funct综合基因组学。2007,8:29-32。
- 22。
Sambrook J,Fritsch E,Maniatis T:分子克隆:实验室手册。1989年,冷泉港实验室出版社,纽约,2
- 23。
Leroy P,Guilhot N,Choulet F,Theil S,Flutre T,Seidel M,Alaux M,Tanaka T,Itoh T,Feuillet C:TrianNot:用于植物基因组的自动注释的多功能和高性能管道。正面。植物SCI。2012,3:5-
- 24。
Rutherford K,Parkhill J,Crook J,Horsnell T,Rice P,Rajandream M-A,Barrell B:Artemis:序列可视化和注释。生物信息学。2000,16:944-945。10.1093 / Bioinformatics / 16.10.944。
- 25。
Sonnhammer ell,Durbin R:具有适于基因组DNA和蛋白质序列分析的动态阈值控制的DOT矩阵程序。基因。1995,167:GC1-GC10。10.1016 / 0378-1119(95)00714-8。
- 26。
柳条T,Sabot F,Hua-Van A,Bennetzen JL,Capy P,Chalhoub B,Flavell A,Leroy P,Morgante M,Panaud O,Paux E,Sanmiguel P,Schulman AH:一种统一的真核转换元素的分类系统。NAT Rev Genet。2007,8:973-982。10.1038 / NRG2165。
- 27。
Altschul SF,Madden TL,SchäfferAA,张J,Zhang Z,Miller W,Lipman DJ:Papped Blast和Psi-Blast:新一代蛋白质数据库搜索程序。Nucl酸res。1997年,25:3389-3402。10.1093 / nar / 25.17.3389。
- 28。
Wu Td,Watanabe CK:GMAP:MRNA和EST序列的基因组映射和对准程序。生物信息学。2005,21:1859-1875。10.1093 / Bioinformatics / BTI310。
- 29。
欧阳S,朱武,汉密尔顿J,林H,Campbell M,Childs K,Thibaud-Nissen F,Malek RL,Lee Y,Zheng L,Orvis J,Haas B,Wortman J,Buell Cr:TIGR RES Genome注释资源:改进和新功能。核酸RES。2007,35:D883-D887。10.1093 / nar / gkl976。
- 30.
Carver TJ, Rutherford KM, Berriman M, Rajandream M- a, Barrell BG, Parkhill J: ACT: Artemis比较工具。生物信息学。2005,21:3422-3423。10.1093 /生物信息学/ bti553。
- 31。
Shahmuradov Ia,Solovyev VV,Gammerman AJ:施用置信度估计。Nucl。酸res。2005,33:1069-1076。10.1093 / nar / gki247。
- 32。
GonzálezVM,Benjak A,HénaffME,Mir G,Casacuberta JM,Garcia-Mas J,PuigdomènechP:使用BAC汇集策略测序6.7 MB的甜瓜基因组。BMC植物BIOL。2010,10:246-10.1186 / 1471-2229-10-246。
- 33。
rounsley s,marri pr,yu y,r,sisneros n,goicoechea jl,lee sj,angelova a,kudrna d,luo m,fucourtit j,desany b,骑士j,niazi f,egholm m,inggo:de nogo植物基因组的下一代测序。米。2009,2:35-43。10.1007 / s12284-009-9025-z。
- 34。
Akhunov E, Akhunova A, Dvorak J: Triticum urartu、Aegilops speltoides和Ae的BAC文库。多倍体小麦的二倍体祖先。中国科学(d辑),2005,31(4):417 - 422。10.1007 / s00122 - 005 - 0093 - 1。
- 35。
You FM, Huo N, Deal KR, Gu YQ, Luo M-C, McGuire PE, Dvorak J, Anderson OD: Annotation-based genome-wide SNP discovery in the large and complex Aegilops tauschii genome using next-generation sequencing without a reference genome sequence. BMC Genomics. 2011, 12: 59-10.1186/1471-2164-12-59.
- 36。
Rustenholz C,Choulet F,Laugier C,Safar J,Simkova H,Dolezel J,Magni F,Scalabrin S,Cattonaro F,Vautrin S,Bellec A,Berges H,Feuillet C,Paux E:A 3,000位染色体转录图3B揭开六倍体小麦基因岛的结构和功能特征。植物理性。2011,157:1596-1608。10.1104 / pp.111.183921。
- 37。
Bolot S,Abrouk M,Masood-Quraishi U,Stein N,Messing J,Feuillet C,Salse J:“内圈”的谷物基因组。CurrOp植物BIOL。2009,12:119-125。10.1016 / J.PBI.2008.10.011。
- 38。
Massa AN, Wanjugi H, Deal KR, O’Brien K, You FM, Maiti R, Chan AP, Gu YQ, Luo MC, Anderson OD, Rabinowicz PD, Dvorak J, Devos KM: Gene space dynamics during the evolution of Aegilops tauschii, Brachypodium distachyon, Oryza sativa, and Sorghum bicolor genomes. Mol Biol Evol. 2011, 28: 2537-2547. 10.1093/molbev/msr080.
- 39。
帕特森啊,鲍尔斯我,Bruggmann R, Dubchak我Grimwood J, Gundlach H,哈贝雷尔G, Hellsten U, Mitros T, Poliakov,污物J, Spannagl M,唐H,王X,柳条T, Bharti AK,查普曼J, Feltus FA, Gowik U,眼镜IV,里昂E,马赫CA, Martis M, Narechania, Otillar RP,彭宁BW, Salamov AA,王Y,张L, Carpita数控,等:高粱双色基因组与禾本科植物的多样性。《自然》,2009,457:551-556。10.1038 / nature07723。
- 40.
Vogel JP,Garvin DF,Mockler TC等:基因组测序和模型草BroChypodium distachyon的分析。自然。2010年,463:763-768。10.1038 / Nature08747。
- 41.
BAUCOM RS,ESTILL JC,CHAPARRO C,UPSHAW N,JOGI A,DERAGON J-M,WESTERMAN RP,SANNETZEN JL:卓越的多样性,非随机分布和B73玉米基因组中的retroelement的快速演变。Plos Genet。2009,5:E1000732-10.1371 / journal.pgen.1000732。
- 42.
Huang S,Sirikhachornkit A,Su X,Faris J,Gill B,Haselkorn R,Gornicki P:编码Plastid乙酰-CoA羧化酶和Triticum / Aegilops复合物的3-磷酸甘油激酶的基因和多倍体小麦的进化史。pnas。2002,99:8133-8138。10.1073 / PNAS.072223999。
- 43。
Brunner S, Keller B, Feuillet C:一个涉及基因和低拷贝DNA的大重排中断了水稻和大麦Rph7位点之间的微共线性。基因工程学报。2003,27(4):483 - 483。
- 44。
Kumar S,Mohan A,Balyan HS,Gupta PK:毛孢囊,小麦和米的基因组之间的正轨。BMC研究笔记。2009,2:93-10.1186 / 1756-0500-2-93。
- 45。
鞭毛L,UDALL J,Nettleton D,Wendel J:AllopolyPloid Gossypium中的重复基因表达揭示了表达演化的两个时间上不同的阶段。BMC BIOL。2008,6:16-10.1186 / 1741-7007-6-16。
- 46。
Yang L,Takuno S,Waters ER,Gaut BS:拟南芥的低表达基因Thaliana通过启动子降解来承担可能的假性化的签名。mol Biol Evol。2011,28:1193-1203。10.1093 / molbev / msq298。
承认
This work was supported by the European Community’s Seventh Framework Programme (FP7/2007-2013) under the grant agreement FP7-212019 (Triticeae Genome) grant No. ED0007/01/01 Centre of the Region Haná for Biotechnological and Agricultural Research and the 3BSEQ grant from ANR(09-GENM-025) - FranceAgriMer (201006-015-104). The work of Jan Šafář was supported by Czech Science Foundation grant No. P501/10/1778.
作者信息
隶属关系
通讯作者
额外的信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
JB,CV,PS,CF和JD设计了实验。JS和HSI构建了BAC库。JB,FCA和HB构建物理贴图和定义和重新排序的MTP。JS和KC用分子标记筛选了BAC库。CV和MD分离DNA和测序的BAC克隆。CV,JP和HST组装序列。jb和fch向序列注释。JB进行了比较分析。JB和JD起草了稿件和CF和PS编辑了手稿。所有作者都仔细阅读并批准了最终的手稿。
电子补充材料
12870_2012_1068_moesm1_esm.pdf.
附加文件1:表S1。用于BAC文库筛查和目标区域的Contigs识别的EST和PCR引物列表。(PDF 52 KB)
12870_2012_1068_moesm4_esm.pdf.
附加文件4:表S3。在染色体臂3ds和染色体3b阳性区域的HGA基因座上发现的基因的启动子预测。(PDF 84 KB)
12870_2012_1068_moesm5_esm.pdf.
附加文件5:表S4。染色体臂3DS和染色体3B同源区Hga位点的基因表达分析(PDF 78 KB)
作者的原始提交的图像文件
权利和权限
本文在BioMed Central Ltd.的许可下发布了这是一个开放的访问文章,根据Creative Commons归因许可(http://creativeCommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Bartoš,J.,Vlček,Č。,Choulet,F.等等。内部序列比较揭示了面包小麦的B和D基因组中的非共聚物基因插入的类似速率。BMC植物BIOL.12,155(2012)。https://doi.org/10.1186/1471-2229-12-155
已收到:
公认:
发表:
关键词
- 小麦
- BAC测序
- 同种型基因组
- 基因重复
- 非共线基因
- AllopolyPloidy.