抽象的
背景
钾(K+)是植物细胞中的重要营养离子,并在许多植物生理和发育过程中起重要作用。在自然环境中,k+缺乏是一种常见的非生物应激,抑制植物生长并降低作物生产力。在对各种应激的反应期间,已经对水稻的基因组基因表达谱进行了几项微阵列研究。但是,关于低k下水稻基因的转录变化很少+条件。
结果
我们分析了水稻根对低钾反应的转录组谱+压力。有或没有低k的米饭幼苗根+在6小时后收获处理,3和5d,并用于微阵列分析。微阵列数据显示,在低k期间,许多基因(2,896)上调或下调超过1.2倍+治疗。GO分析表明,出现转录变化的基因主要有以下几类:代谢过程、膜、阳离子结合、激酶活性、转运等。我们对两者的转录组变化进行了比较分析拟南芥低钾条件下的水稻+压力。通常,显示水稻和稻草转录变化的基因拟南芥响应低k+压力显示出类似的GO分配模式。然而,有更多的基因与压力反应和发育有关拟南芥而不是米饭。许多无辛相关的基因对k响应+而茉莉酸相关酶可能在水稻钾素中起重要作用+营养信号拟南芥.
结论
根据微阵列数据,较少的水稻基因显示出对K的转录变化+磷(P)或氮(N)的缺乏。因此,转录调控在应对低磷和-N胁迫时可能比低钾胁迫更重要+压力。然而,在某些类别的许多基因(蛋白激酶和离子转运蛋白家族)显著上调,表明它们在K+缺乏。转录组变化的比较分析拟南芥结果表明,单子叶植物和双子叶植物对钾的反应机制相似+缺陷,尽管有一些差异。需要进一步的研究来阐明单焦点和双斑点之间的转录调节的差异。
背景
钾(K+)植物细胞中最丰富的一价阳离子,占植物干重的2-10%[1].它在生活植物细胞中的许多生理过程中起着至关重要的作用,包括Osmoreculation,控制托尔戈尔压力,电中和和酶活化[1,2].K.+在土壤溶液中的浓度一般在1 ~ 200ppm(约1ppm)范围内。0.025 ~ 5 mM) [3.].在根际,K+浓度通常小于0.3 mM [4].因此,大多数植物都受到低钾的影响+压力在他们生命中的某个时刻。大多数植物都能在低钾环境下存活+条件,主要是因为高亲和力k+根部的摄取系统,尤其是高亲和力k+运输车[5].不同植物种和基因型具有不同的高亲和钾+带有多元化的运输车+吸收效率(6].
以前的研究报告说,许多高亲和力k+通过低k诱导运输基因+条件。例如,几个成员kup / hak / kt家庭包括ATKUP3.[7],阿纳克5.[8,9],Hvhak1.[10,11],OsHAK1[12]也是k+转运蛋白基因塔卡特1.[13] 和ATCHX17.[14],由K+饥饿。这些K.+转运体可能参与K+低钾胁迫下植物细胞的获取和稳态+条件。此外,转录组分析拟南芥下的性能+压力识别出许多与低钾相关的候选基因+感知,以及与K的反应相关的监管途径+不足(9,15].
微阵列技术是一种方便的工具,用于在各种环境条件下快速分析植物基因表达模式。在植物反应期间激活的信号转导途径的研究通常从微阵列数据分析开始[16].例如,水稻对干旱反应的全基因组基因表达谱[17,低氮[18],低磷[19- - - - - -21]低铁[22使用微阵列技术已经分析了应力。但是,在低k下的理解改变时已经取得了很少的进展+条件。
在本研究中,我们使用了Affymetrix微阵列来监测到短期和长期K期间稻根的转录组谱+缺乏。我们分析了差异表达基因的功能分类,并比较了低k+水稻和拟南芥.这些结果将进一步了解对植物养分缺乏的含量依赖性的分子机制。
结果与讨论
K+营养缺乏会抑制水稻幼苗的生长
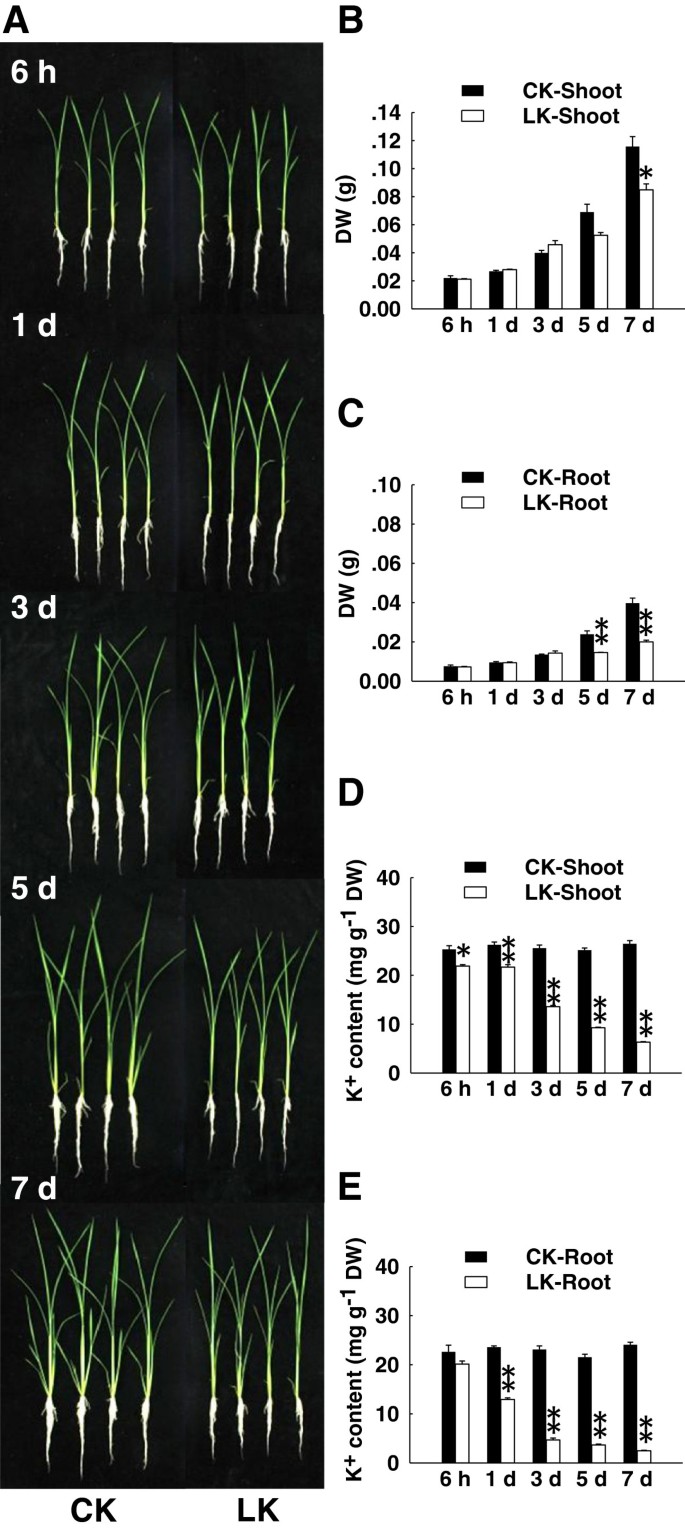
研究钾胁迫下水稻根的转录变化+缺钾时,我们首先确定低钾的适宜时期+治疗。水培水稻幼苗(2周龄)转移到K+-free solution(−K)和K+完全溶液(+K, 1mmk+)作为治疗(LK)和对照(CK)条件。使用的营养溶液是由国际水稻研究所描述的[23].治疗后的指示时间(6小时,1,3,5和7 d),K+- 评估水稻幼苗的缺血表型。干重和k+还在每个时间点测量内容。
K+-k后的幼苗和控制幼苗+缺乏治疗6小时,或1或3天(图1A). K处理5天后,幼苗表现出明显的表型差异+饥饿,当时k+- 菲力植物具有比对照的较小的根和芽(图1一种)。观察到的表型与生物质测量一致。与对照幼苗相比,根部的生物量和k的芽+5 d k后,幼苗较低+缺乏(图11 B, c)。幼苗K+内容仅在6小时后显示减少,并且在整个K中继续降低+缺乏治疗(图1d,e)。
根据这些结果,我们选择了三个时间点(6 h和3和5 d)来研究基因转录的变化。幼苗K+内容在6-H时间点已经减少。它此后进一步降低,但生物质不会受到影响,直到3-D时间点。在5-d时间点,k+含量和生物质均显着降低。因此,这三个时间点可以代表对K的不同阶段的水稻根响应+缺乏。
微阵列分析显示出显示对k响应转录变化的水稻基因+不足
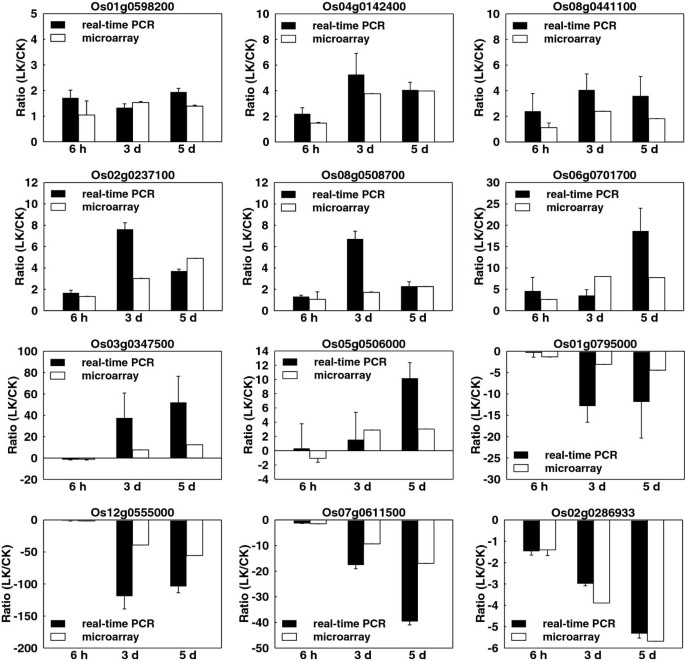
我们使用了一系列染色水稻基因组阵列,以揭示水稻响应期间的转录变化+缺乏。该阵列包含51,279个探针,代表两个水稻品种(48,564个)粳稻成绩单和1,260indic成绩单,http://www.affymetrix.com).根是在环境中首先感觉到营养缺乏的主要组织。因此,K的根+在三个时间点(6小时,3d和5d)收获自杀幼苗和对照。提取它们的总RNA并用于微阵列实验(详见“材料和方法”)。为确保微阵列数据的可重复性和可靠性,在每次点分析三种生物学重复,共18个基因组阵列。为了评估微阵列数据的再现性,使用Genespring GX 11软件计算生物重复的相关系数。所有相关系数大于0.9915(附加文件1).我们对随机选取的12个基因进行了实时PCR分析,以验证芯片数据的有效性。在实时荧光定量PCR分析中,12个基因中的大多数表达模式与水稻芯片数据预测的表达模式相似(图)2).原始数据集(CEL)和规范化表达数据集已储存在美国国家生物技术信息中心的基因表达集(GSE37161)中[24](http://www.ncbi.nlm.nih.gov/geo/).
实时PCR确认所选基因的转录组谱。12个基因的LK/CK比值与real-time PCR结果进行比较。用于PCR的基因特异性引物的序列列于附加文件中12.
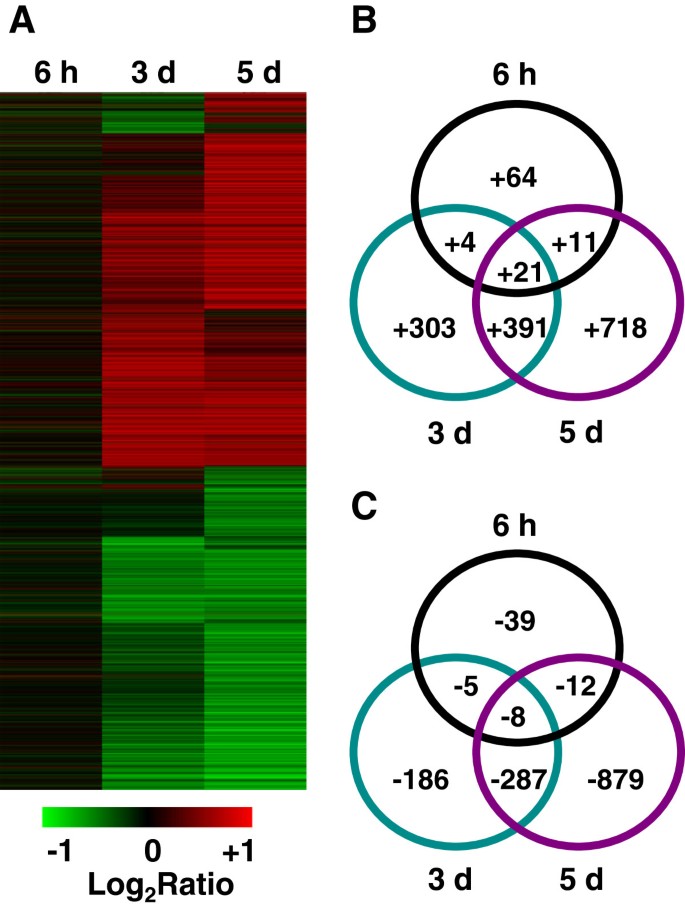
微阵列结果显示,在这组数据中约有25000个基因显示阳性信号。这代表了水稻基因组阵列上大约44%的探针集(附加文件2).在这些表达基因中,我们更感兴趣的是那些在低钾反应中表现出转录变化的基因+条件。我们使用DNA芯片分析仪(DCHIP)2007软件分析了这些差异表达的基因[25](http://www.dchip.org.).这揭示了在K+-饥饿幼苗上调或下调幅度分别为1.2、1.5、2.0倍以上(P-value < 0.05)与对照苗比较(详见“材料与方法”)。其中2896个、1166个和356个基因的表达变化分别超过1.2倍、1.5倍和2.0倍(附加文件)3.).由于在1.5倍或2.0倍截止阈值下上调或下调的基因较少,因此我们使用1.2倍截止阈值进行进一步分析,以便包含更多与水稻钾响应有关的候选基因+缺乏。
表达变化的基因数量随K值的增加而增加+饥饿时间从6 h增加到3 d,再增加到5 d1).在k的开头+-Tarvation时期(6小时和3d),比下调基因更高的基因(在6小时,719处为3d)(6小时,486,3d,3d)(表1).这一结果表明,许多K+相关基因被诱导,这些基因可能参与了对K的响应和适应+- 在此期间的缺点。但是,在k的后期阶段+-饥饿5 d时,下调基因(1186)多于上调基因(1141)(表2)1).这表明许多基因的表达可能被抑制以减缓水稻的生长速度,而另一些基因则被激活以适应长期钾胁迫+缺乏。
以前的转录组研究拟南芥表明只有少数基因被K+不足(9,15].相反,磷(P)和氮(N)的缺乏导致了大量基因的转录变化拟南芥[26,27].我们在水稻幼苗中观察到类似的趋势。与以往对受磷和氮饥饿的水稻幼苗的研究相比,目前的研究中显示转录变化的基因较少[18- - - - - -21].这些发现与以前的数据一起表明,翻译后调节可能比植物反应对低k的转录调节更重要+压力(9,16].
探讨在低k期间显示不同时间点转录变化的基因的相似性和差异+处理后,我们进行了层次聚类分析,并构建了维恩图(图3.).3 d和5 d表达变化的基因表达模式相似,与6 h表达变化的基因表达模式不同(图)3.一种)。这表明显示6小时的转录变化的基因可以代表参与短期反应的那些+缺乏,而那些涉及长期反应的可能在3天后开始被激活。此外,一些基因在所有时间点都显示出转录变化,这些可能在整个K期都有功能+饥饿(图3.B, C)。
水稻对钾响应差异表达基因的基因本体论分析+不足
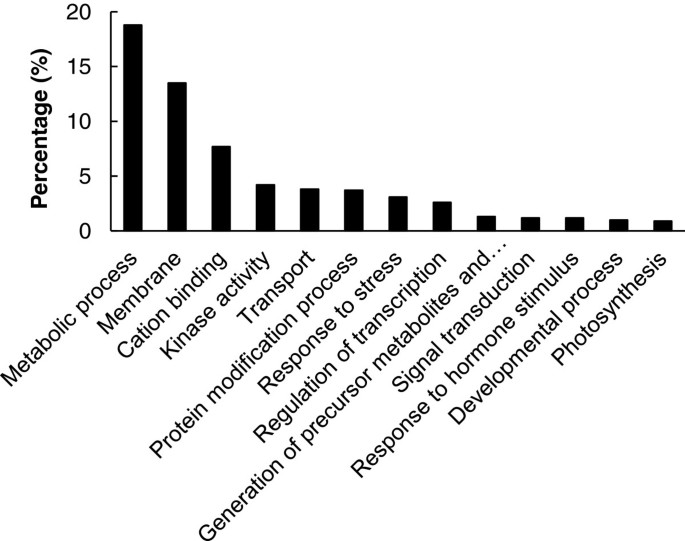
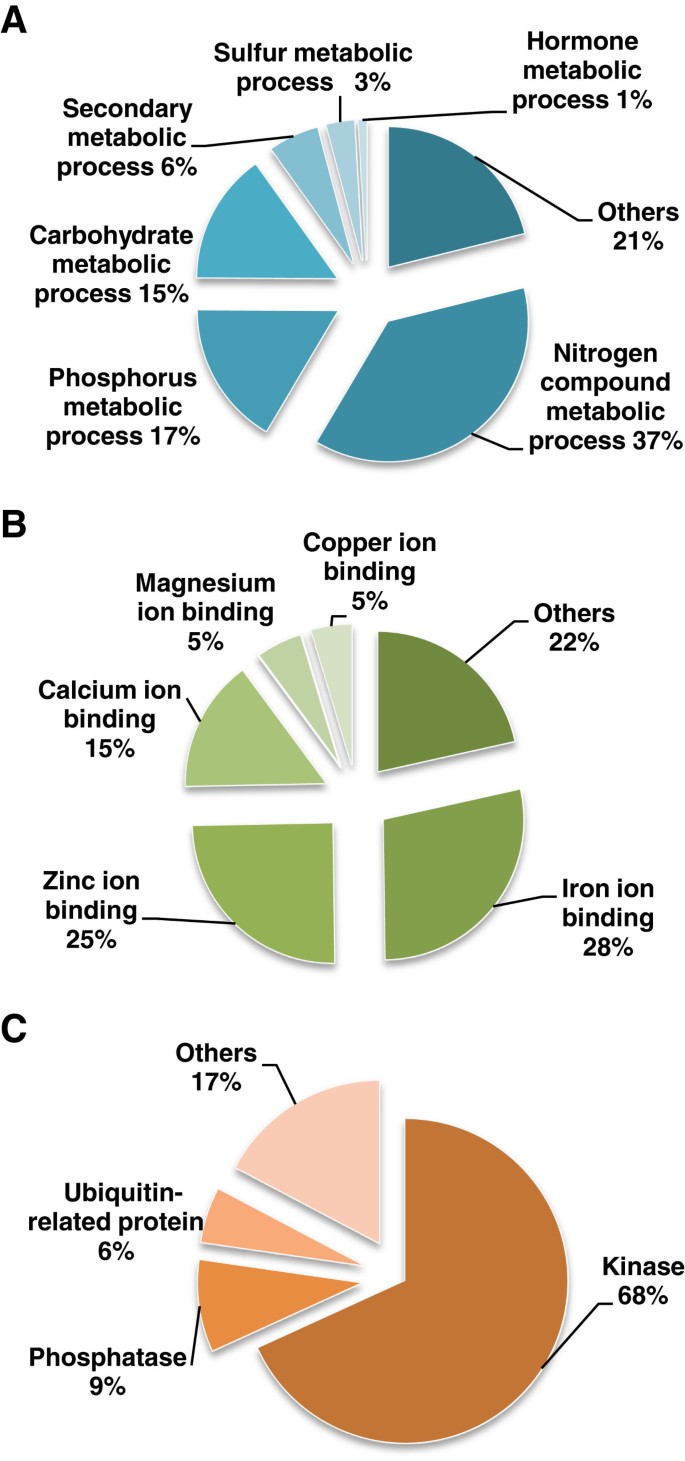
评估这些差异表达基因在应答K+缺陷,基因本体(GO)分析使用AgriGO在线服务[28](http://bioinfo.cau.edu.cn/agrego.).将2896个差异表达基因分为代谢过程(18.8%)、膜(13.5%)、阳离子结合(7.7%)、激酶活性(4.2%)、转运(3.8%)、蛋白修饰(3.7%)和应激反应(3.1%)13个功能类别,如图所示4.大多数这些过程被认为与K的响应密切相关+缺乏。进一步分析表明,在大多数GO类别中,在6-h和3-d时间点,上调的基因远远多于下调的基因(Additional file)4),但在5-D时间点(附加文件,比上调基因更下调4).氧化石墨烯分析的结果可能为了解水稻对钾的反应相关基因的转录组谱提供一些线索+缺乏。
代谢酶
进一步分析代谢过程类别中的差异表达基因表明,大约37%与氮代谢有关,磷代谢17%,碳水化合物代谢的15%,共计约70%的基因(图)5A).许多代谢酶需要K+离子作为辅影子[29];因此,胞质K降低+浓度可能影响代谢过程。以前的一项研究报道,代谢酶的转录调控可能在对K的适应中非常重要+不足拟南芥[30.].
在本研究中,两种硝酸还原酶(NR)基因(Os08g0468100和Os08g0468700)在K+缺乏。在磷相关代谢过程中发挥作用的一些蛋白激酶和磷脂酶也受到转录调控。两个PEPCK(磷酸胆管丙酸羧酮酶)基因(OS10G0204400和OS01G0208700)在5 d时间点显着下调。
许多代谢物拟南芥根系中丙酮酸、苹果酸和硝酸盐含量均显著降低+压力(30.].然而,再补钾24 h后+在生长培养基中,大多数这些代谢物的浓度已经恢复[30.].细胞代谢物浓度的变化可能是K+-诱导编码代谢酶的基因的转录变化。因此,K的变化+供给水平会影响它们的转录水平。综上所述,这些结果表明,长期K+缺乏可能影响代谢过程的调节,导致细胞活动下降和植物生长速度减慢,使植物能够在营养有限的条件下生存。
阳离子结合蛋白
在阳离子结合类别中差异表达的基因主要编码铁、锌和钙离子结合的蛋白质(图)5B).我们发现大约三分之一的铁结合蛋白基因编码过氧化物酶(POXs),这是植物细胞中含有血红素的蛋白质[31].Pox在细胞代谢和非生物应激过程中发挥清除反应性氧物种(ROS)的关键作用[32,33].前期研究表明,植物根系中ROS的积累是对K的早期响应+不足(34,35,许多与ROS信号传导相关的过氧化物酶基因在低钾条件下被诱导+条件(15,34].在本数据中,21个过氧化物酶基因显示低k下的转录变化+水稻(8个上调,13个下调)。这证实了过氧化物酶与低钾之间的关系+水稻的信号传感。这些转录调控过氧化物酶基因可分为六个亚组[36],并列入其他文件5.
我们还确定了九种钙传感器蛋白基因,显示出K的表达水平变化+饥饿;五个被上调(oscbl5.,OsCML1,OsCML18,OsCML20,OsCML31)和四次被下调(OsCML5,OsCML11,OsCPK9, OsCCaMK1)(额外的文件5).以前,加利福尼亚州2+- 困扰蛋白质是重要的k+反应的基因拟南芥[15].在这里,我们还确定了CA2+水稻中的传感器蛋白,表明这些蛋白质可以连接CA.2+植物中的信号和下游靶蛋白对k的反应+缺乏。
激酶和蛋白质修饰
在蛋白质修饰类中,大多数基因被鉴定为激酶(68%)和磷酸酶(9%)(图)5C)。该结果表明,磷酸化和去磷酸化可能是水稻反应中的非常重要的调节机制+缺乏。先前的研究表明,许多蛋白激酶通过在离子胁迫下调节相应的离子转运蛋白的活性来调节活细胞中的离子传输过程。这种调节机制在植物和哺乳动物细胞中发现[37- - - - - -39].本研究的GO分析结果表明,激酶和蛋白质修饰类中的许多基因都受到K的转录调控+饥饿(图4).这些基因编码的激酶可以参与调节k+吸收和K+钾胁迫下水稻的动态平衡+缺乏。
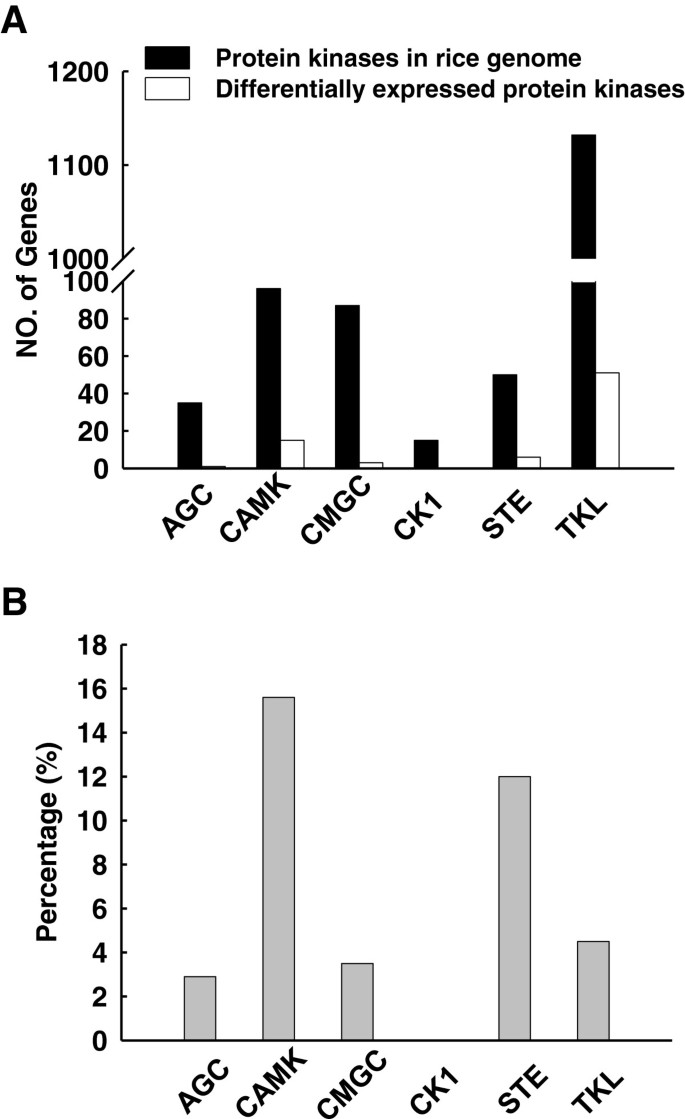
根据水稻激酶数据库[40](http://phylomics.ucdavis.edu/kinase/index.shtml.),存在1,467个激酶基因对应于水稻基因组中的1,934个转录物。这些蛋白激酶可分为六个主要的系统发育基团;AGC,CAMK,GMGC,CK1,STE和TKL [40].在本研究中,来自五组的123个激酶基因(除CK1之外的所有组)通过K转录调节+缺乏。这表示水稻中总激酶基因的8.4%。其中,最改变的激酶基因属于TKL组(图6一种)。TKL组主要由受体样蛋白激酶组成。据报道,许多人在植物响应各种外部信号中起着至关重要的作用[41].例如,富含亮氨酸的重复(LRR)受体样蛋白激酶Bri1和Bak1参与芸苔类固醇信号转导拟南芥[42,43].此外,RPK1还参与ABA信令路径[44].因此,我们推测TKL组中转录改变的基因可能也参与了水稻对低钾的响应+压力。这些蛋白质可能感觉到外部低k+信号并通过磷酸化将其转换为下游组分。
我们还分析了激酶基因的百分比,显示每个激酶组中的转录变化。与其他三组相比,凸起和STE基团中的激酶(分别为15.6%和12%),与其他三组相比,转录的变化(图6b)。该结果表明,这两组中的激酶在水稻反应中可能更为重要+缺乏。在CAMK组中,出现转录变化的基因有10个震动年代,两个OsSnRK1-type,二OsCPKs,和一个oscamk.(桌子2).STE组中,4个OsWNK蛋白基因和2个MAP3K激酶基因出现转录变化(表)2).在我们的微阵列数据中,许多激酶基因来自震动(CBL - 相互作用蛋白激酶)和oswnk.(没有K =赖氨酸)家族在经过K的水稻幼苗中进行转型调节+缺乏(附加文件6,7).
CIPKs是植物特异性丝氨酸/苏氨酸蛋白激酶,与钙传感器CBLs(钙调磷酸酶b样蛋白)形成蛋白复合物来执行功能[45,46].之前的调查拟南芥表明蛋白激酶CIPK23与CBL1 / 9一起激活AKT1介导的K.+在低钾条件下根系的吸收+条件(47,48].另一个研究发现CIPK6调制k的易位+通过与CBL4相互作用,使AKT2通道进入质膜,并增强质膜上AKT2的活性[49].水稻oscipks展现出高度相似之处拟南芥Cupks在氨基酸序列中;因此,水稻中也可能存在类似的调节机制。根据序列atcipks.在拟南芥, Kolukisaoglu等(2004)从水稻基因组中鉴定出至少30个OsCIPKs [50].在NCBI和RGAP数据库中,更新的注释中有33个OsCIPKs。在Affymetrix水稻微阵列中,这33oscipks.基因由31个探针组表示。大约三分之一oscipks.显示稻根的高表达水平(平均信号强度值≥500)。在目前的数据中,八震动基因(OsCIPK2, 6, 9, 10, 14/ 15,23, 26)上调和两个震动基因(OsCIPK29, 31岁)在低k下调节下调+压力(表2,附加文件6).这些激酶基因的表达增加可能调节K的活性+渠道,提高K+在K下的摄取+- 条件。
Wnk是另一种丝氨酸/苏氨酸蛋白激酶,首先在哺乳动物中研究[51].哺乳动物中的肾脏调节离子运输的态度,特别是在Na+和K+运输(39,52].在高等植物中,WNK同源物已在几种植物中鉴定出来拟南芥(10基因)和水稻(9基因)[53- - - - - -55].最近的一份报告显示,ATWNK8可以磷酸化真空H的亚基+-Atpase并调节离子运输拟南芥[56].在水稻中,奥斯威克1受非生物压力调节,并在内部昼夜节律中发挥作用[55].在我们的微阵列实验中,五个oswnks.在水稻根中强烈表达(平均信号强度值≥500)。此外,oswnk5,7,8上调和oswnk9.低于低k下调+条件(表2,附加文件7).
这两个家族的一些激酶磷酸化离子转运蛋白并调节植物细胞中的离子转运。在低钾条件下,这些激酶基因的表达水平可能增加+调节离子转运体的条件,从而提高转运体介导的K+在稻谷中的uptake。
膜蛋白和离子转运蛋白
K的吸收+在植物细胞中主要由K介导+运输车和K.+位于质膜上的通道[57- - - - - -59].K+运输扣是高亲和力K的主要组成部分+-在低外部K下工作的吸收机械+条件(5,60,61].许多的高亲和性K+低钾诱导不同植物的转运基因+压力,如阿纳克5.,Hvhak1., 和OsHAK1[8- - - - - -12].编码K的基因表达量增加+转运体可能是植物增钾的一种快速而直接的策略+- 缓和和克服k+缺乏。这似乎是不同植物物种中的共同调节机制。
在水稻基因组中,主要k+运输车来自OsHAK(27个基因)和奥克克(9个基因)家庭[62,63].在我们的微阵列数据中,有三个OsHAK基因(1,7和11)在低k下显着上调+压力(表2,附加文件8).这与先前的报告一致+饥饿诱导的表达OsHAK1[12].我们推测了这些k+转运体可能参与K+低k下的摄取+条件。
为奥克克基因,所有成员奥克克家族基因在水稻根中低水平表达(附加文件8).只有OsHKT2; 1在K+缺乏(表2).这一结果与相关报道一致OsHKT2; 1在小麦和大麦中,在低k下显着诱导+条件(64,65].有趣的是,该基因产物被报道为Na+运输车,不是k+在异种表达系统中发现的转运体[65,66].在K.+- 米扎根,oshkt2; 1增加了na的浓度+离子,让这些na+离子可作为营养离子,在低钾条件下促进水稻幼苗生长+条件(67].
除了K+结果表明,在钾胁迫下,水稻根系中某些磷酸盐和硝酸盐转运体基因的转录水平发生了变化+缺乏。在我们的微阵列数据中,两个磷酸盐转运蛋白基因(ospo1; 1和灰质1; 4)在5 d后明显上调+饥饿(表2).相比之下,两个硝酸盐转运体基因Os02g0112600和Os02g0689900同时下调(表)2).在。中也发现了类似的调节机制拟南芥.三种表达水平拟南芥硝酸盐转运基因(AtNRT2; 1,AtNRT2; 3,AtNRT2; 6)在低k下减少+, K+再补给[15].相反,缺乏P和n则增加了K的表达+转运蛋白等HAK5的,KUP10,KUP11在拟南芥[35,68].这些发现表明k,p和n营养上升性是相关的。一种类型的营养离子的缺乏可能影响植物中其他两种离子的吸收和倾斜。这种潜在的机制可以促进植物细胞中营养离子的稳态。但是,需要进一步研究来澄清该监管机制的细节。
低k的比较分析+米饭之间的反应拟南芥
米饭和拟南芥都是模范植物,前者是模范单子叶植物,后者是模范双子叶植物。比较这两个物种在低钾条件下的转录组变化具有重要意义+压力理解哪些机制是共享的,并且特定于每株植物,并深入了解共同的监管机制。先前的研究表明,许多基因在p-和n缺陷条件下转录调节;这两者都观察到这一点拟南芥[26,27,68]和米[18- - - - - -21].相比之下,k诱导较少的基因+缺乏缺乏拟南芥[9,15]和大米(目前数据;额外的文件9).这些结果表明,与低钾胁迫相比,植物对低磷、低氮胁迫的反应中,转录调控可能更为重要+压力。
分析水稻和拟南芥下的性能+压力,我们比较了微阵列数据拟南芥[9在本研究中。我们重新分析了他们的微阵列数据(四拟南芥ATH1微阵列,两个时间点,无生物重复)具有本研究中使用的相同数据处理方法(1.2倍的截止阈值)。然后,从米饭和米和水稻进行分析结果拟南芥比较(附加文件10).一般来说,在水稻和水稻中表现出转录变化的基因拟南芥在他们的回复中显示类似的Go分发模式+压力。这一结果表明,代谢过程、膜和阳离子结合等基因可能在低钾反应中发挥重要作用+大米和拟南芥.但是,也有一些差异。与压力反应有关的基因比例较大拟南芥而不是米饭(附加文件10),表明这些基因在对低k的反应中更重要+压力拟南芥.
在目前的数据中,我们发现了33个激素相关基因对K有反应+水稻根系缺乏。其中大部分与生长素有关(23个),其中10个与生长素有关OsIAAs,4个操作系统SUARs和2odsghs.等。这些基因被列在"附加文件"里11”。这些基因家族的几个成员的转录水平可以由生长素诱导[69].虽然,它们也在低k下调节或下调+这表明生长素与钾的关系+营养信号。据报道,生长素在植物的远端延伸区中心柱状细胞中积累拟南芥后根K+剥夺[70].因此,假设毒素也可能在k后产生稻根+钾胁迫对水稻根系形态的影响+- 条件。Armengaud等人,(2004)报道了许多与JA相关基因在K后诱导+剥夺,并强调了JA的新颖作用拟南芥K+营养信号。但值得注意的是,这些ja相关基因主要诱导于拟南芥芽,但根较少。因此,不同的植物激素可能在不同的器官中发挥作用,促进植物适应钾离子+- 压力。
这些转录组比较可能为理解水稻和拟南芥在他们对低k的反应中+压力。然而,单子叶和双子叶在营养缺乏条件下转录调控的差异还有待进一步研究。
结论
K+缺乏是自然环境中常见的非生物胁迫,抑制植物生长并减少作物生产。在本研究中,我们分析了Affymetrix微阵列,以研究对K的反应期间稻块的转录组谱+缺乏。微阵列数据显示,较少的基因参与了对K的响应+对磷和氮缺乏的反应。因此,在植物对低磷、低氮胁迫的响应中,转录调控可能比低钾胁迫更重要+压力。在低钾期,共有2896个基因的转录水平发生了显著变化+治疗。这些主要是在代谢过程的GO类别,膜,阳离子结合,激酶活性,运输等。蛋白激酶和离子转运蛋白家族中的许多基因都明显上调,表明它们可能在水稻反应中发挥重要作用+缺乏。我们还进行了转录组变化的比较分析拟南芥低钾条件下的水稻+压力。结果表明,单子叶植物和双子叶植物对K的反应机制相似+缺陷,尽管有一些差异。然而,单子叶和双子叶在转录调控方面的差异还需要进一步的研究。
方法
植物生长条件与低钾+压力治疗
水稻品种nipponbare.(奥雅萨苜蓿l . ssp。粳稻)在下列条件下,在生长室中培养植物:相对湿度,50-70%;12-H光/ 12-H黑色光周期;温度,30°C天,25°C。
营养液的成分如国际稻米研究所所述[23].该溶液含1.427 mM nhh4没有3.,0.323毫米2阿宝4h·22O, 0.512 mM K2所以4, 0.998 mM CaCl21.643 mM MgSO4·7小时2O, 9.474 μM MnCl2·4H.2O, 0.075 μm (nh .4)6莫7O24·4H.2O, 18.882 μm h3.薄3.,0.152μmznso4·7小时2o,0.155μmcuso4·5H.2o,0.031 mm feso4·7小时20.031 mM Na2EDTA·2 h2(pH = 5.5)。营养液每隔一天更换一次。首先,将水稻幼苗置于正常溶液中培养两周。然后将其中一半转入-K营养液(缺K营养液)2所以4)作为K+-缺陷处理(LK),另一半转入正常溶液(+K)作为对照(CK)。在指定的时间采集对照苗和处理苗的根和芽进行进一步测定。
生物量和K+内容测量
在不同时间采集对照和处理水稻幼苗的根和茎+-缺乏性处理(6 h, 1、3、5和7 d)。分别收获根和茎组织,80℃干燥48 h,称量(干重)。测量K+含量,干燥样品在马弗炉中575°C下焚烧5 h,残渣溶于0.1 N HCl中,K+含量采用原子吸收分光光度法(日立Z-5000)测定。利用3个生物重复进行表型、生物量和K+测量的内容。的t-测试(*P < 0.05, **P < 0.01) was used to analyze the statistical significance.
Affymetrix基因芯片的RNA分离、靶制备和杂交
在指定的时间采集对照和处理水稻幼苗的新鲜根+-缺失处理(6 h、3 d和5 d)。根系立即用液氮冷冻,在−80°C保存,直到进一步提取RNA。每个RNA样本都来自5株独立的幼苗。用Trizol试剂(Invitrogen, Carlsbad, CA, USA)从水稻根中提取总RNA。使用NanoDrop 2000荧光光谱仪和甲醛变性凝胶电泳确定RNA样品的质量特性。然后,用RNeasy Mini试剂盒(Qiagen, Valencia, CA, USA)纯化总RNA。将纯化的RNA在42°C 2 h和16°C 1 h的条件下,用含有One-Cycle Target Labeling and Control reagent (Affymetrix, Santa Clara, CA, USA)的反转录反应生成第一链cDNA。双链cdna通过体外转录反应生成cRNA。用生物素对cRNA进行标记,标记的cRNA在20 μg时呈碎片状。使用Eppendorf生物光度计和电泳检测cRNAs和片段化cRNAs的大小分布。将片段cRNA (15 μg)加入300 μL杂交液中,200 μL杂交于Affymetrix水稻基因组阵列,在45℃下杂交16 h。 The standard wash and double-stain protocols were applied using a fluidics station (Affymetrix GeneChip Fluidics Station 450). The arrays were scanned on a genechip scanner (Affymetrix GeneChip Scanner 3000).
微阵列数据处理与分析
首先,使用Affymetrix GCOS 1.0(MAS 5.0)软件来分析扫描阵列以生成RAW数据,该数据被保存为CEL文件。然后,DNA芯片分析器(DCHIP)2007软件[25](http://www.dchip.org.)用于标准化所有18个阵列组。当所有三个重复的检测到呼叫“P”时,认为当呼叫“P”时,何时进行检测P- 价值小于0.05。我们使用Genespring GX 11软件来分析每对阵列之间的相关系数。显示响应K的转录变化的基因+使用DCHIP 2007软件分析缺陷。当您满意的两个条件时,认为基因显示转录变化:(1)在k后的上调或下调超过1.2折+缺乏治疗;(2)叠化P -值小于0.05。负值或正值分别表示向下或向上调节。我们利用TIGR MeV 4.2软件对显示转录变化的基因进行了层次聚类分析[71.](http://www.tm4.org).分别收集至少在一个时间点上调或下调的基因,用维恩图分析。AgriGO网络服务(http://bioinfo.cau.edu.cn/agrigo/analysis.php.)用于分析显示转录变化的基因的功能类别。使用Microsoft Excel和Access提取和管理微阵列数据。
实时聚合酶链反应分析
微阵列实验的RNA样本(3个生物重复)也用于实时PCR检测,以确保结果的可靠性和重复性。为消除基因组DNA污染,用DNase I (RNase Free)处理总RNA(中国大连Takara)。然后,采用随机引物(Promega, Madison, WI, USA)逆转录合成cDNA。cDNA样品稀释至4 ng/μL。根据制造商的协议,使用Power SYBR Green PCR Master Mix (Applied Biosystems, Foster City, CA, USA)在7500 Real Time PCR System机器(Applied Biosystems)上进行3个生物重复。基因特异性引物(附加文件12)使用底漆3设计(http://frodo.wi.mit.edu/primer3/input.htm)和Dnaman软件。放大18 s rRNA用作内部控制以使数据标准化。
参考
- 1。
Leigh Ra,Wyn Jones RG:一个假设与植物细胞中该子离子的分布和功能相关的临理钾浓度。新植物。1984,97:1-13。10.1111 / J.1469-8137.1984.TB04103.x。
- 2.
高等植物的矿质营养。植物营养学报,1998,31(4):429 - 434。10.1146 / annurev.pp.31.060180.001323。
- 3.
土壤养分生物有效性的机制研究。钾。1995,Barber SA编辑,纽约:Wiley, 231-232。
- 4.
Schroeder Ji,Ward JM,Gassmann W:高植物中向内整流K +通道的生理学和结构的观点:K +摄取的生物物理意义。Annu Rev Biophys Biomol Surric。1994年,23:441-471。10.1146 / annurev.bb23.060194.002301。
- 5.
Epstein E,Rains DW,Elzam OE:通过大麦根的钾吸收双重机制的解决。《美国国家科学院学报》上。1963,49:684-692。10.1073 / pnas.49.5.684。
- 6.
黄志强,陈志强:植物对钾的吸收和利用。植物营养与肥料学报。10.1007 / BF02181962。
- 7。
Kim EJ,Kwak Jm,Uozumi N,Schroeder Ji:Atkup1:编码高亲和力钾运输活性的拟南芥基因。植物细胞。1998,190:51-62。
- 8。
Ahn SJ,Shin R,Schachtman DP:拟南芥Kt / kup基因的表达及根毛在K +摄取中的作用。植物理性。2004,134:1135-1145。10.1104 / pp.103.034660。
- 9。
Gierth M, Maser P, Schroeder JI:钾转运体AtHAK5在K+剥夺诱导的高亲和性钾离子吸收和AKT1钾离子通道对拟南芥钾离子吸收动力学的贡献。植物营养与肥料学报。2005,32(4):513 - 514。10.1104 / pp.104.057216。
- 10。
Santa-MaríaGe,Rubio F,Dubcovsky J,Rodriguez-Navarro A:大麦的Hak1基因是大型基因家族的成员,并编码高亲和力的钾转运蛋白。植物细胞。1997,9:2281-2289。
- 11.
Fulgenzi FR, Peralta ML, Mangano S, Danna CH, Vallejo AJ, Puigdomenech P, Santa-María GE:离子环境控制大麦HvHAK1转运体对钾获取的贡献。植物营养与肥料学报。2008,31(4):457 - 461。10.1104 / pp.107.114546。
- 12.
bañuelosma,garciadblas b,cubero b,rodríguez-navarro - 答:水稻钾钾运输司的库存和功能表征。植物理性。2002,130:784-795。10.1104 / pp.007781。
- 13.
Buschmann pH,Vairyanathan R,Gassmann W,Schroeder Ji:Na +摄取电流的增强,时间依赖于k +在小麦根细胞中的K +饥饿的K +通道转录物。植物理性。2000,122:1387-1397。10.1104 / pp.122.4.1387。
- 14.
Cellier F, Conejero G, Ricaud L, Luu DT, Lepetit M, Gosti F, Casse F: AtCHX17, CHX家族的阳离子/H+交换剂成员,拟南芥的特性表明其在K+稳态中的作用。植物学报,2004,39:834-846。10.1111 / j.1365 - 313 x.2004.02177.x。
- 15.
arengaud P, Breitling R, Amtmann A:拟南芥钾依赖转录组揭示茉莉酸在营养信号转导中的重要作用。植物营养与肥料学报。2004,32(4):457 - 461。10.1104 / pp.104.046482。
- 16.
Schachtman DP,Shin R:营养传感和信令:NPKS。Annu Rev植物Biol。2007,58:47-69。10.1146 / annurev.arplant.58.032806.103750。
- 17.
关键词:水稻,干旱响应,全基因组,时空基因表达谱基因组学杂志。2011,12:149-10.1186/1471-2164-12-149。
- 18。
连X,王某,张俊,冯Q,张L,风扇D,李X,袁D,汉B,张Q:使用CDNA微阵列测定水稻低氮胁迫早期10,422基因的表达谱。植物mol biol。2006,60:617-631。10.1007 / s11103-005-5441-7。
- 19。
Wasaki J,Yonetani R,Kuroda S,Shinano T,Yazaki J,Fujii F,Shimbo K,Yamamoto K,Sakata K,Sasaki T,Kishimoto N,Kikuchi S,Yamagishi M,Osaki M:转录磷胁迫分析代谢变化的转录组分分析在水稻植物根。植物细胞环境。2003年,26:1515-1523。10.1046 / J.1365-3040.2003.01074.x。
- 20.
Wasaki J,Shinano T,Onishi K,Yonetani R,Yazaki J,Fujii F,Shimbo K,Ishikawa M,Shimatani Z,Nagata Y,Hashimoto A,Ohta T,Sato Y,Miyamoto C,Honda,Kojima K,Sasaki T,Kishimoto N,Kikuchi S,Osaki M:转录组分析表明稻叶中磷可用性的操纵引起的推定代谢变化。J Exp Bot。2006,57:2049-2059。10.1093 / JXB / ERJ158。
- 21.
李丽,刘超,连新:低磷胁迫下水稻根的基因表达谱。植物生态学报。2009,32(4):453 - 457。
- 22.
郑L,黄F,NARSAI R,WU J,Giraud E,HE F,Cheng L,Wang F,Wu P,Whelan J,Shou H:水稻幼苗铁和磷互动的生理和转录体分析。植物理性。2009,151:262-274。10.1104 / pp.109.141051。
- 23.
Yoshida S, Forno DA, Cook JH, Gomez KA:水稻生理研究的实验室手册。1976年,马尼拉:国际水稻研究所,61-67。
- 24.
关键词:NCBI;基因表达;杂交阵列;核酸学报2002,30(4):497 - 503。10.1093 / nar / 30.1.207。
- 25.
Parmigiani G, Garret E, Irizarry R, Seger S,等:基因表达数据的分析:方法和软件。2003,纽约:施普林格:Giovanni Parmigiani编辑,120-139。
- 26.
吴P,马L,侯X,王男,吴Y,刘芳,邓XW:磷饥饿触发不同的拟南芥根和叶基因表达的改变。植物理性。2003,132:1260-1271。10.1104 / pp.103.021022。
- 27.
王R,奥马托托M,Xing X,Crawford NM:拟南芥根系硝酸盐反应的微阵列分析揭示了1,000多种迅速响应的基因和新的葡萄糖,海藻糖-6-磷酸铁和硫酸盐代谢的新联系。植物理性。2003,132:556-567。10.1104 / pp.103.021253。
- 28.
杜Z,周X,Ling Y,Zhang Z,Su Z:Agrigo:农业社区的GO分析工具包。核酸RES。2010,38:W64-W70。10.1093 / nar / gkq310。
- 29。
《无机植物营养》。蛋白质,酶和无机离子。1983,柏林:施普林格:Läuchli A和Bieleski RL编辑,528-562。
- 30.
Armengaud P,Sulpice R,Miller Aj,Stitt M,Amtmann A,Gibon Y:初级代谢的多级分析为钾营养的作用提供了新的见解和拟南芥中的氮氧化术。植物理性。2009,150:772-785。10.1104 / pp.108.133629。
- 31。
植物过氧化物酶基因家族的系统发育研究。中国科学:地球科学,2003,27(4):457 - 461。10.1007 / s00239 - 003 - 2489 - 3。
- 32。
李志刚,李志刚,李志刚:植物活性氧基因网络。植物科学进展。2004,9:490-498。10.1016 / j.tplants.2004.08.009。
- 33。
LüthjeS,Meisrimler CN,Hopff D,MöllerB:血管植物中膜结合III类过氧化物酶的系统发育,拓扑,结构和功能。植物化学。2011,72:1124-1135。10.1016 / J.Phytochem.2010.11.023。
- 34。
Shin R,Schachtman DP:过氧化氢介导植物根细胞对营养剥夺的反应。《美国国家科学院学报》上。2004,101:8827-8832。10.1073 / PNAS.0401707101。
- 35。
拟南芥根毛对氮、磷和钾缺乏的响应。植物营养与肥料学报。2005,32(4):451 - 457。10.1093 /卡式肺囊虫肺炎/ pci145。
- 36.
Passardi F,Longet D,Penel C,Dunand C:III类过氧化物酶多粒家庭在土壤中的稻米及其演变。植物化学。2004,65:1879-1893。10.1016 / J.Phytochem.2004.06.023。
- 37.
栾S:植物钙信号传导中的CBL-CIPK网络。趋势植物SCI。2009,14:37-42。10.1016 / J.Plants.2008.10.005。
- 38.
王志强,王志强。CBL-CIPK Ca2+解码信号网络的研究进展。新植物学报,2009,34(4):517-528。10.1111 / j.1469-8137.2009.02938.x。
- 39.
WNKs:一种典型的多效作用蛋白激酶。中国生物医学工程学报,2011,31(4):457 - 461。10.1152 / physrev.00017.2010。
- 40。
Dardick C,Chen J,Richter T,欧阳S,罗纳德P:水稻激酶数据库。水稻髓内的系统核发生组数据库。植物理性。2007,143:579-586。
- 41.
莫里斯尔,Walker JC:受体样蛋白激酶:回应的键。CurrOp植物BIOL。2003,6:339-342。10.1016 / s1369-5266(03)00055-4。
- 42.
Li J, Chory J:一种推测的富含亮氨酸重复受体激酶参与油菜素内酯信号转导。细胞。1997,90:929-938。10.1016 / s0092 - 8674(00) 80357 - 8。
- 43.
Li J,Wen J,Lease Ka,Doke JT,税务Fe,Walker JC:Bak1,拟南芥LRR受体样蛋白激酶,与Bri1相互作用并调节芸苔类固醇信号传导。细胞。2002,110:213-222。10.1016 / S0092-8674(02)00812-7。
- 44.
富含亮氨酸重复受体样激酶1 (leucine - repeat receptor-like kinase1)是拟南芥脱落酸早期信号转导的关键膜结合调控因子。acta botanica sinica(云南植物研究),2017,34(5):593 - 598。10.1105 / tpc.104.027474。
- 45.
Shi J,Kim Kn,Ritz O,Albrecht V,Gupta R,Harter K,Luan S,Kudla J:与拟南芥中的钙蛋白B样钙传感器相关的新型蛋白激酶。植物细胞。1999,11:2393-2405。
- 46.
Kim Kn,Cheong YH,Gupta R,Luan S:拟南芥钙素B样钙传感器及其靶激酶的相互作用特异性。植物理性。2000,124:1844-1853。10.1104 / pp.124.4.1844。
- 47.
Xu J, Li HD, Chen LQ, Wang Y, Liu LL, He L, Wu WH:一种与钙调磷酸酶b样蛋白相互作用的蛋白激酶对拟南芥K+转运蛋白AKT1的调控。细胞。2006,125:1347-1360。10.1016 / j.cell.2006.06.011。
- 48.
Li L, Kim BG, Cheong YH, Pandey GK, Luan S: Ca2+信号通路调节拟南芥低钾反应的K+通道。《美国国家科学院学报》上。2006年,103:12625 - 12630。10.1073 / pnas.0605129103。
- 49.
通过CBL4/CIPK6钙离子传感器/蛋白激酶复合物对AKT2钾离子通道的钙依赖调控和质膜靶向。生物医学工程学报,2011,31(4):531 - 534。10.1038 / cr.2011.50。
- 50.
Kolukisaoglu U,Weinl S,Blazevic D,Batističo,Kudla J:钙传感器及其相互作用的蛋白激酶:拟南芥和水稻CBL-CIPK信号传导网络的基因组学。植物理性。2004,134:43-58。10.1104 / pp.103.033068。
- 51.
徐B,英语JM,Wilsbacher JL,Stippec S,Goldsmith EJ,Cobb MH:Wnk1,一种新型哺乳动物丝氨酸/苏氨酸蛋白激酶缺少亚域II的催化赖氨酸。J Biol Chem。2000,275:16795-16801。10.1074 / JBC.275.22.16795。
- 52.
Kahle Kt,Ring Am,Lifton RP:Wnk激酶的分子生理学。Annu Rev Physiol。2008,70:329-355。10.1146 / annurev.physiol.70.113006.100651
- 53.
Nakamichi N,Murakami-Kojima M,Sato E,Kishi Y,Yamashino T,Mizuno T:参考昼夜节律,拟南芥拟南芥蛋白激酶的汇编和表征。Biosci Biotech Bioch。2002,66:2429-2436。10.1271 / BBB.66.2429。
- 54。
王Y,刘k,廖h,庄c,ma h,yan x:植物wnk基因家族和拟南芥开花时间的调节。植物BIOL。2008,10:548-562。10.1111 / J.1438-8677.2008.00072.x。
- 55。
水稻WNK1受非生物胁迫调节,并参与内部昼夜节律。植物学报,2011,33(3):316-320。
- 56。
A WNK激酶结合和磷酸化V-ATPase亚基C. FEBS Lett. 2006, 580: 932-939。10.1016 / j.febslet.2006.01.018。
- 57。
véryaa,sentenac h:高等植物中K +运输的分子机制和调节。Annu Rev植物Biol。2003,54:575-603。10.1146 / annurev.arplant.54.031902.134831。
- 58。
植物对缺钾的反应:钾转运蛋白的作用。中国生物医学工程学报,2006,27(4):434 -436。
- 59。
Ward JM,MäserP,Schroeder Ji:植物离子渠道:基因家族,生理学和功能基因组学分析。Annu Rev Physiol。2009,71:59-82。10.1146 / annurev.physiol.010908.163204。
- 60.
Maathuis FJ,ICHIDA AM,Sanders D,Schroeder Ji:高植物K +通道厂的角色。植物理性。1994年,114:1141-1149。
- 61。
植物钾离子转运的细胞机制。植物营养学报。2008,33(6):637- 643。10.1111 / j.1399-3054.2008.01067.x。
- 62。
水稻钾转运家族相关基因的全基因组分析与鉴定植物科学进展,2017,37(6):748 - 751。10.1016 / j.plantsci.2006.11.019。
- 63。
张春梅,邱雪梅,王玲,张春梅:水稻钾转运子家族KT/HAK/KUP基因表达谱分析。中国生物医学工程学报。2008,28(3):437-452。10.1007 / s00438 - 008 - 0377 - 7。
- 64。
Wang TB, Gassmann W, Rubio F, Schroeder JI, Glass ADM:高亲和钾转运基因HKT1在大麦和小麦根系中的快速上调。植物生理学报。1998,32(6):651-659。10.1104 / pp.118.2.651。
- 65.
Garciadeblásb,senn me,bañuelosma,Rodríguez-navarro - odium运输和HKT运输车:米模型。工厂J. 2003,44:788-801。10.1046 / J.1365-313x.2003.01764.x。
- 66.
Horie T,Yoshida K,Nakayama H,Yamada K,Oiki S,Shinmyo A:两种类型的HKT运输车,在奥里佐萨蒂扎族的Na +和K +运输的不同性质。工厂J. 2001,27:129-138。10.1046 / J.1365-313x.2001.01077.x。
- 67.
Horie T,Costa A,Kim Th,Han MJ,Horie R,Leung Hy,Miyao A,Hirochika H,G,Schroeder Ji:Rice Oshkt2; 1转运蛋白介导大型Na +流入组分进入K + -Starved Roots以进行生长。Embo J. 2007,26:3003-3014。10.1038 / sj.emboj.7601732。
- 68.
Misson J,Raghothama Kg,Jain A,Jouhet J,Block Ma,Bligny R,Ortet P,Creff A,Somerville S,Rolland N,Doumas P,Nacry P,Herrerra-Estrella L,Nussaume L,Thibaud MC:一个基因组 -使用Arabidopsis Thaliana Affymetrix基因芯片测定植物对磷酸盐剥夺的植物反应。《美国国家科学院学报》上。2005,102:11934-11939。10.1073 / pnas.0505266102。
- 69.
abel和theologis:早期基因和植物素作用。植物理性。1996,111:9-17。10.1104 / pp.111.1.9。
- 70.
Vicente-Agullo F, Rigas S, Desbrosses G, Dolan L, Hatzopoulos P, Grabov A:钾载体TRH1是植物生长素在拟南芥根中运输所必需的。植物学报,2004,40:523-535。10.1111 / j.1365 - 313 x.2004.02230.x。
- 71.
李赛义德AI, Sharov V,白色的J, J,梁W, Bhagabati N, Braisted J,卡拉帕米,制革匠T, Thiagarajan M, Sturn, Snuffin M, Rezantsev,波波夫D, Ryltsov, Kostukovich E, Borisovsky我,刘Z, Vinsavich, V,信赖Quackenbush J: TM4:免费、开源系统进行微阵列数据管理和分析。生物技术学报。2003,34:374-378。
确认
我们感谢甄苏(中国农业大学)博士关于数据分析的关键讨论。本工作得到了中国国家基础研究计划(973计划,Grant No.2011CB100300)和中国国家自然科学基金(Grat No.30900269至Wy)的支持。
作者信息
从属关系
通讯作者
附加信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
WY构思了这项研究。MTL进行了表型试验和生理分析。MTL进行RNA提取,实时RT-PCR分析,并起草手稿。WY和MTL设计了实验并分析了微阵列数据。WY和WWH监督了这个项目,并最终完成了论文。所有作者阅读并批准了最终的手稿。
电子补充材料
受控(CK)和低k的探头组数
额外的文件2:+(LK)条件。(PDF 48 KB)
所有基因显示在K期间转录变化
附加文件3:+缺乏。(XLS 5 MB)
在K的6 h (A)、3 d (B)和5 d (C)显示转录变化(上调和下调)的基因的功能分类
额外的文件4:+缺乏。利用AgriGO网络工具分析GO差异表达基因的类别。显示了13个主要生物类别中显示转录变化的基因数量。(PDF 75 KB)
钙传感器和过氧化物酶蛋白的基因在K
额外的文件5:+缺乏治疗。(XLS 60 KB)
表达水平
额外的文件6:震动基因在K+缺乏。(PDF 24 KB)
表达水平
额外的文件7:oswnk.基因在K+缺乏。(PDF 54 KB)
表达水平
额外的文件8:OsHAK(一)和奥克克(B)基因在K+缺乏。(PDF 189 KB)
稻米与差异表达基因的比较
额外的文件9:拟南芥对K的响应+缺乏。(PDF 91 KB)
稻米与稻米的GO分类比较
额外的文件10:拟南芥对K的响应+缺乏。(PDF 72 KB)
激素和生长素响应基因,显示在k期间转录变化
附加文件11:+缺乏。(XLS 80 KB)
作者为图像提交的原始文件
权利和权限
开放获取本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
马,TL。,吴,wh。&王,Y.水稻根系对钾缺乏的转录组分析。BMC植物杂志12,161(2012)。https://doi.org/10.1186/1471-2229-12-161
已收到:
接受:
发表:
关键词
- 大米
- K+不足
- 微阵列
- 转录组