摘要
背景
黑胡椒 (吹笛者nigrum.L.)是世界上最受欢迎的香料之一。它用于烹饪和保存食物,甚至具有药用特性。疾病生产的损失是这种作物文化的主要限制。主要疾病是根腐烂和脚腐,这是根系感染的结果Fusarium solani.和Phytophtora capsici,分别。了解病原菌与寄主根区之间的分子相互作用,对通过生物技术育种获得抗病品种具有重要意义。然而,这个物种的遗传和分子数据是有限的。本文首次采用RNA-Seq技术对黑胡椒的根转录组进行了描述。
结果
黑胡椒的根转录组由NGS固体平台测序并使用多k方法组装。Blast2Go和Orthomcl方法用于注释10338 unigenes。4472预测的蛋白质显示了约52%的同源性拟南芥蛋白质组。两个根蛋白质组鉴定出615种蛋白质,这些蛋白质似乎决定了植物的根模式。简单重复序列在遗传多样性研究中具有重要的应用价值,并可能在生物技术和生态学中得到应用。
结论
该数据集包含10338个unigenes,对黑胡椒的生物技术育种和木兰属植物(基部被子植物的主要类群)的生态基因组学研究具有重要意义。
背景
黑胡椒 (吹笛者nigrum.)是世界上最受欢迎,最古老的香料之一,烹饪用途,最近作为食品防腐剂[1].由于其药用特性,它用于传统医学中,用于其抗氧化剂,抗炎和抗癌性能[2那3.].该物种是世界上交易的第二次贸易香料,占2009年生产445900吨的价值9.29亿美元[4.].
P. nigrum.是一个多年生,攀登藤蔓土着印度西南部地区。其生产仅限于亚洲和南美洲发现的多雨热带地区。热带气候有利于疾病的发展,由于害虫和疾病导致的作物损失是黑胡椒生产中的主要限制[1].对两种主要病原体的抵抗力Phytophtora capsici和Fusarium solani.F。SP..Piperis.,尚未通过培养的黑胡椒种质的典型育种来实现,并且仍然是使用植物生物技术改善的主要挑战[1].尽管黑辣椒的农业和经济重要性,但目前对其遗传学的了解非常有限[5.].
对于黑胡椒等分子信息很少或没有的非模式植物,下一代测序(NGS)技术为快速获取遗传信息提供了巨大的机会[6.-9.].在植物生物学中,由于基因组尺寸和高水平的倍率,转录omes的表征是鉴定蛋白质的强大工具。RNA-SEQ可以以极高通量和定量方式序列,生物体或组织的转录组[9.].下一代测序技术产生大量高采样率的cDNA文库,提供更深入和更完整的转录组视图[8.].这种对RNA序列的转录组范围描述通过计数reads(一种被称为数字基因表达(DGE)的方法)识别编码序列、单核苷酸多态性(SNPs)、其他多态性、剪接变异和相对表达水平[9.那10].
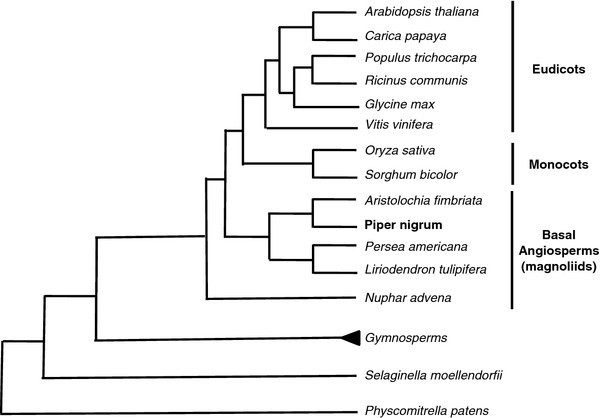
对黑胡椒转录组的RNA-seq分析可以极大地影响我们对这个物种的认识。黑胡椒属于木兰亚纲的胡椒科,被称为基部被子植物,这是一个大而重要的科,很少有基因组数据可用(图1).转录组数据可能会产生许多生态和生物技术方面的影响,从探索和描述这个家族的进化,到定义标记,如在育种计划中有用的微卫星,到建立与植物防御和代谢相关的表达基因目录。
在本研究中,我们使用RNA-seq技术分析黑胡椒的转录组。大约产生了7100万个读取和22363个德诺维组装的转录本从茎根组织中获得。转录本被功能性注释,代表了从转录组中得到的第一个序列P. nigrum..共发现257个新的简单重复序列(SSRs)。这些结果是木兰类群序列的第一个数据集,将为多样性和生态学的研究打开新的视角,并将有利于育种和基因组计划,旨在了解植物-病原体在根中的相互作用,这是感染过程的中心器官f .以上和P. Capsici.,是黑胡椒的两大病原菌。
结果
测序结果
原始reads数据集存储在NCBI数据库SRA047721登录号下,包含约71878419个reads 50 bp,总长度为3.5 Gbp1).预处理高度严格;在平均3bp的窗口中,质量值(QVs)低于20的读数和QVs低于18的基底被修剪。排除预处理后小于30 bp的Reads。在删除冗余读、过滤低质量读、修剪低质量端点和删除适配器后,数据集的大小为13300000万reads,约为665mbp。额外的文件1:图S1显示了经过过滤的读数据集在读取位置通过FASTQC监控时的qv [11].平均QV在29到22之间变化(基本呼叫精度为99.37-99.85%),并沿着读取长度缓慢下降。未发现QV在8以下的碱基(碱基呼叫正确率在85%以下),75%以上的碱基QV在12以上(碱基呼叫正确率在93.7%以上)。额外的文件2:图S2为读的长度分布;83%的读取经过修剪和过滤后长度超过45 bp。
新创转录组的组装
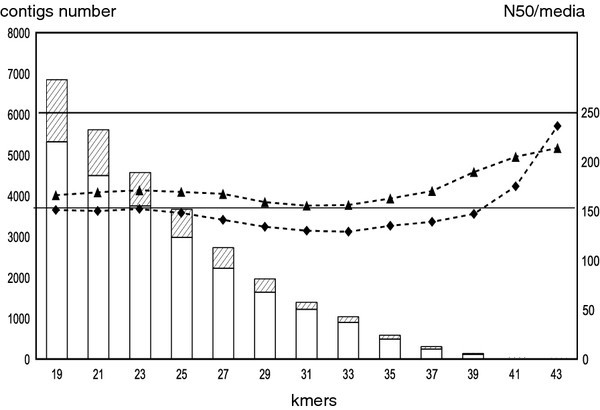
采用Surget-Groba所描述的添加倍数k法对黑胡椒茎根转录组进行组装等.[12].该方法的第一步是使用从19到43个碱基的单个k-mers组合读取。数字2介绍每个k-mer的装配结果。contigs的数量由6847个(k-mer = 19个碱基)减少到22个(k-mer = 43个碱基)。contigs的N50和平均长度分别由19 ~ 43个碱基的k-mers负向增加,分别从166 ~ 214 bp和151 ~ 236 bp(图2)2).因此,k-mers越大,contigs的平均长度就越大,但生成的contigs的数量就越少。加性多k法的第二步是使用不同的k-mers合并组装体,去除多余的contigs。该方法在检测低表达基因和提高长contigs的组装方面具有优势。为了识别可能的转录本异构体,使用oasis脚本进行组装,该脚本将类似的contigs聚集到小组中,以获得转录本的异构体。STM的-方法的使用,考虑马兜铃fimbriata预测蛋白质组作为引用产生支架。
获得了22363个Contig的最终数据集,对应于3.8 MBP。在Contigs组装之后,使用SEQCLean工具过滤CONTIGS数据集,并用带有线粒体的RDNA序列,1640个,用线粒体,539种,154种植物DNA重复序列(http://seqclean.sourceforge.net/).排除了长度低于100bp的斑点。
在清理步骤之后,IASSEMBLER工具用于群集和组装成果以获得UNIGENE序列[13].最后,从contigs数据集中鉴定出10338个unigenes。最大的unigene长度为1314 bp, N50长度为168 bp1,图3.).
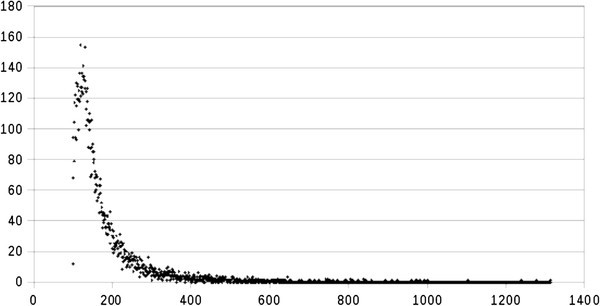
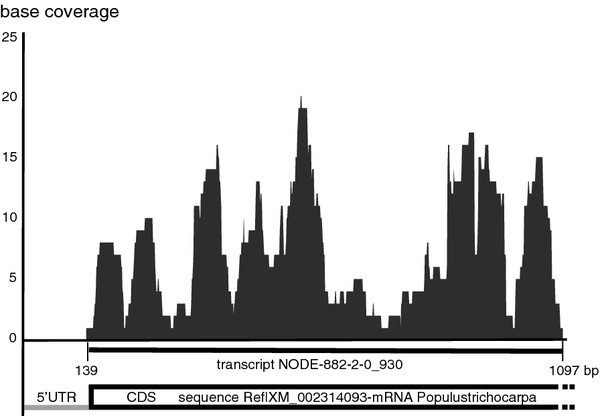
平均读覆盖率为每个contig 211读,最高的覆盖率为每个contig 46239读,对应的rpkm单位为68250。每个碱基的平均覆盖率为62倍,这表明,平均而言,contigs的每个碱基都被测序了62倍。例如,绘制了最长的数据集之一的contig NODE-882-2-0_930的基底覆盖率,每个基底的覆盖范围沿959 bp从1到22不等(图5)4.).
转录组功能注释
预测部分或完全编码序列(CD)的数量为4472,表示总转录物的约44%。NCRNA搜索Nocode Databank [14]表明,只有32个reads与非编码rna具有显著的同源性,并具有推测的调节功能。约52%的预测蛋白具有显著的同源性(1e-05),拟南芥蒂利亚纳NCBI数据库中的蛋白质序列(表 2).死亡率最高的物种是Populus Trichocarpa.54.38%,马兜铃fimbriata54.02%和葡萄点击率为53.93%。数据集中显示同源性的序列的百分比(1e-5)和5种模式植物的蛋白质组2.同源性从50%到54%不等,单子叶植物的同源性低于双子叶植物。在图4., contig NODE-882-2-0_930与推测的pre-mRNA剪接因子RBM22/SLT11的mRNA序列进行比对Populus Trichocarpa..这P. nigrum.contig和p . trichocarpamRNA在假定的CDS序列的核苷酸中显示77%的同源性。相应的蛋白序列同源性为87%,相似性为96%,覆盖率为94%,无缝隙。
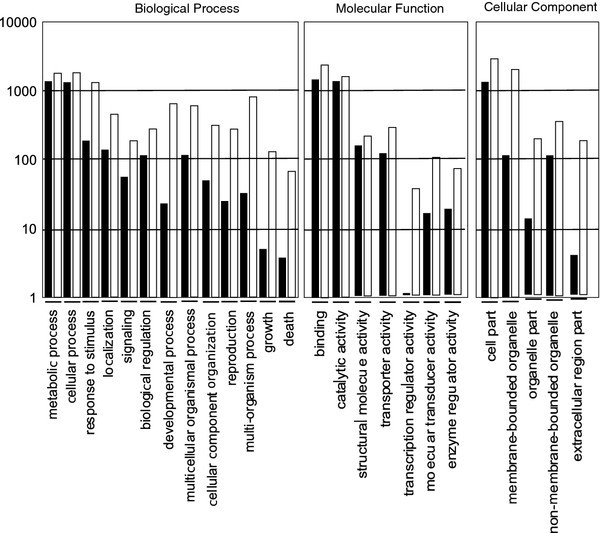
使用BLAST2GO结果进行功能注释[15],其中3055个(占总数的29.5%)在基因本体论(GO)数据库中进行了功能注释P. nigrum.一个根图书馆和9664 unigenes(73.3%)答:芥用于比较的根库(ATGI-5336)。黑辣椒根和一个的Go-Slim注释拟南芥蒂利亚纳根库如图所示5..转录本数量较多的生物过程属于“细胞过程”和“代谢过程”两类,分别有1499个和1328个unigenes。“分子功能”分类显示,“结合”功能与“催化活性”功能相似,分别有1712和1772个unigenes。“单元格部分”注释,如图所示5.,这表明高比例(约39%的预测CDS)应定位于细胞器。在表3.是否报道了答:芥和P. nigrum.根。利用生物信息学工具从cDNA序列中预测出8004个蛋白序列答:芥Root和4472 forP. nigrum.(表3.).利用orthoMCL工具,共识别出3408个正群和37个拟对数答:芥1017和159P. nigrum..各株系的正群系/蛋白数所占比例不同,为42.6%答:芥22.7%,P. nigrum.,应该用这样的事实来解释答:芥是二倍元P. nigrum.是四倍体。
检测微卫星
使用MISA脚本识别微卫星(http://pgrc.ipk-gatersleben.de/misa/).分析22363个ContIgs,168种,三核苷酸重复,分别表示总量的43%,56%和1%(表 4.).最常检测到的SSR是三核苷酸AGC,ACT和ACG最常见。
讨论
在植物育种中成功使用生物技术方法[16例如,增强对病原体的抵抗力。然而,在育种计划中使用生物技术,需要预先的遗传和基因组知识来分离基因和描述基因型和表现型[16].植物基因组学,特别是非模式植物的基因组学,一直是一个挑战,因为高倍数、大基因组、低基因组复杂性和大比例的重复序列。除了对整个基因组进行测序外,对转录组进行测序是快速有效地访问表达基因和表征表型的一种很好的替代方法[17].
转录组的特性可以使用杂交或基于序列的技术,但所有基于杂交的技术都需要转录组的先验知识。在植物生物学中,基于cDNA文库测序的表达序列标签(ESTs)技术是一项重大进展,为植物基因组学的研究做出了重要贡献。然而,cDNA文库的测序有许多局限性,如克隆和Sanger测序所涉及的时间和金钱成本。基因表达序列分析(SAGE)和改进的变种superpersage已被用于增加吞吐量和取样,但转录本鉴定的可靠性是其局限性之一[18].第二代测序(2-GS)技术克服了这些限制,与Sanger测序相比,能够在更短的时间内生成大量的reads或短reads数据集,而且每个碱基的试剂成本更低。2-GS技术为非模式植物的遗传研究开辟了广阔的前景[17].
黑胡椒的基因组特征非常差,在Genbank中仅沉积了184个序列(Access 3/11/2011)。以前的遗传学研究侧重于栽培黑胡椒和野生亲属的多样性,系统发育和分类[19-22].尽管这种香料具有农业和经济上的重要性,但在分子数据库中只有系统发育标记的DNA序列可用(见图)1).黑胡椒基因组研究的另一个困难是缺少木兰类群(被描述为基生被子植物类群)的基因组序列[23那24)(图1).数据库中所有的全序列基因组都来自单子叶或双子叶植物,与黑胡椒的系统发育距离较远,如图所示1[23那24].
大约20gb的数据可以被映射,对应7100万个50 bp的短读。新创合成了22363个contigs, 10338个unigenes, N50为168 bp。N50略小于Illumina 75 bp数据集获得的202 bp番薯甘薯转录组和208bp在75bp单个和配对的数据集中获得山茶花Sinensis.[25那26].然而,contigs的数量低于这些其他研究:56516个和127094个I. Batatas.和C. Sinensis,分别。尽管在我们的研究中阅读数字更高,(7100万vs 5900万和3500万I. Batatas.和C. Sinensis)时,连列数较低。这一结果可以解释为读取长度的不同(50 bp vs 75 pb)和SOLiD数据集的质量较低,只有50 - 60%的读取映射到转录组参考[27那28].
由于缺乏黑胡椒的遗传数据,估计转录组的覆盖率是很复杂的。黑胡椒是四倍体,2n = 52。据细胞遗传学研究估计,其基因组的大小约为1220 Mbp (1C = 1.25 pg),约为其基因组的10倍拟南芥蒂利亚纳[29].在答:芥,约6%的基因组被转录,代表41671转录物[18].在里面答:芥然而,TGI数据库(TGI数据库)在很大程度上被测序并已鉴定了5609和5884转录物(11.3和9.3 Mbp),该转录物似乎表明只有14%的预测基因在根中表达[30.].假设有类似的转录比例P. nigrum.,整个转录组估计为73.2mbp(6%),根部转录组为10.3mbp(0.84%)。
测序结果表明,cDNA文库的平均覆盖率为62x,总长度为3.8 Mbp。如图所示4.,该转录本NODE-882-2-0_930具有良好的阅读覆盖率,最高可达20倍,并与来自mRNA序列具有较高的同源性和良好的共线性p . trichocarpa,表示良好的测序和装配质量。预测CDS与其他植物蛋白质蛋白酶的比较表明,具有显着同源性的序列百分比高于单子叶(15-23.4%)的双斑点(51-53%)。这种低位的同源性可能是由于magnoliId和其他组(南息和所述弯曲)之间的系统发育距离以及不同的平均转录尺寸(211pb),如在测序期间所述I. Batatas.转录组(25].k-mer加性组装的方法能有效地最大化转录本的数量,有趣的是,每个k-mer (19-43 bp)具有显著同源性的转录本比例(22-35%)、N50 (129 - 236 bp)和平均转录本长度(156-214 bp)相对保守(图)2).与植物转录om的其他研究的比较很困难,因为与Illumina平台使用的读取长度更长(75bp)并且图书馆是配对的,这显然有助于组装过程,如图所示I. Batatas.[25].最后,2-GS平台的局限产生了大量的短折叠,但我们的数据集长度超过200bp的未成年人,大约72%的读取具有重要同源性的读数,这代表了我们对黑胡椒生物学知识的显着进展。
转录本数据集的功能注释为黑胡椒茎和根的生理学提供了信息。来自植物根组织的NGS数据集仅用于人参和甘薯。对“刺激反应”和“本土化”的分类图5.在甘薯和黑胡椒根的转录组中都有很高的代表性[25].这两种根植物的分子功能是保守的。两者的功能“转运体活性”都相当高。这一特征可能是由于根在吸收微量元素方面非常重要。对黑胡椒转录组的初步注释是非常重要的,应该有助于鉴定新的基因编码的转运蛋白、根和茎组织特异性转录因子或用于防御的蛋白质。
结论
在模式植物中,NGS提高了产生和分析大多数转录本的表达和检测、识别基因及其可变剪接形式以及在基因组规模上分析DNA甲基化和组蛋白修饰的能力[6.那8.].基于Sanger测序的技术已经产生了从模型物种的大量数据,并且具有序列基因组和转录om的植物提供了一种反映基因表达的鲁棒数据库。然而,非模型植物的转录组资源,特别是与NGS有限,特别是对于分析表达序列,替代剪接和SNP。
测序P. nigrum.利用SOLiD平台(short-reads)生成了属于Magnoliid组和Piperales目的非模式物种的第一个序列数据集。没有参考基因组或转录组的组装被认为是困难的,但非模式物种的部分转录组P. nigrum.是组装的。这项工作得到了一些人的帮助德诺维使用非模式植物(如鹰嘴豆)的NGS数据进行组装Cicer Arietinum.L. [31],桉树祖母X桉树urophylla混合(32)、橡胶橡胶树取代巴西橡胶树[33],甘娃番薯甘薯[25]和荞麦Fagopyrum[34].
SSRs的分析在植物遗传多样性研究、育种和繁殖等方面具有很大的应用潜力。本研究所获得的黑胡椒序列具有很强的相关性,因为在生物数据库中有关黑胡椒的信息很少,所获得的数据可用于生态学和生物技术研究。
方法
植物材料
组织样本取自8个70日龄植株的基部根部。本研究所用的植株均来自巴西品种P. nigrum.,布拉加蒂娜(附加文件3.:图S3)。Bragantina品种是亚洲Panniyur-1品种的生态型,这是Uthirankotta和Cheriyakaniyakadan品种的混合动力。从黑胡椒植物获得的赌注植根于含有沙子的托盘中,并在含有先前高压灭菌的蛭石的罐中生长两次。本研究中使用的植物在温室中生长并适应约两个月大约。
cDNA制备和测序
总RNA从35 mg的组织中提取P. nigrum.使用Illustra Rnaspin Mini Kit(GE Healthcare,USA)。为了去除任何DNA污染,用寡替氏菌直接MRNA迷你套件(USA)用oligotex Direct MRNA迷你套件清洁样品。使用RNA-SEQ和RNA-SEQ(Invitrogen,USA)的Ribominus真核试剂盒耗尽RRNA。使用Qubit分析仪(Invitrogen,USA)测定mRNA的浓度。
将耗尽的RNA片段(126.5 ng/μL)制成cDNA文库,用于高通量测序。使用SOLiD Total RNA-Seq Kit (Invitrogen公司,美国)获得片段库。该试剂盒的整体程序是基于带有简并末端的适配器杂交,然后进行逆转录和PCR文库扩增[35].在文库扩增之前和之后,通过在聚丙烯酰胺凝胶上和之后选择CDNA。在流动细胞的孔中进行cDNA文库的测序。
预处理测序数据
使用CutAdapt(0.9.5)从读取中裁剪SOLiD适配器序列[36],读取序列由SOLiD编码颜色空间转换为FASTQ格式的双编码颜色。PRINSEQ (lite 0.14.4) [37]用于删除冗余读,修剪低质量读(3 nt窗口的平均QV < 20, QV < 18),并删除小的预处理读(读长度< 30 bp) [37].使用FASTQC软件(V.0.9.2)获得读取数据集的统计信息(http://www.bioinformatics.bbsrc.ac.uk/projects/fastqc/).
新创转录组组装和功能注释
转录组组装使用Velvet和oasis [38那39].为优化德诺维加法多k法[12]被用来结合使用不同k-mers的多个组装物的性质(19-43)。
利用ESTScan程序检测组装得到的转录本序列中的潜在编码区域[40].为了获得蛋白质序列,使用框架工具[41].计划BWA [42用于对准由天鹅绒组件获得的所有轮廓的原始读取,用于计算测序的覆盖范围。
本地BLASTX (E-value 1e-05年)从PlantGDB (ftp://ftp.plangdb.org), nr-viridiplantae (NCBI),V. Vinifera那答:芥那p . trichocarpa那G. Max.和o .漂白亚麻纤维卷.
功能注释基于BLASTX结果与NR-VIRIDIPLANTAE数据库,并且程序BLAST2GO用于将生物学功能,细胞组分和细胞过程分配给转录物[43].使用orthoMCL工具预测同源和副同源蛋白组[44].
检测微卫星
MISA Perl脚本用于标识序列数据集中的SSRS的位置(http://pgrc.ipk-gatersleben.de/misa/misa.html).在我们的转录组中,通过检测双、三和四核苷重复序列来寻找SSRs,这些重复序列超过5倍,最小距离为100 bp。
参考
- 1.
黑胡椒和小豆蔻的农学和经济,香料的“国王”和“女王”。爱思唯尔,纽约2011。
- 2.
Nishimura Y, Kitagishi Y, Yoshida H, Okumura N, Matsuda S:黑胡椒或姜黄乙醇提取物下调了Daudi培养细胞中SIRT1蛋白的表达。中华医学杂志。2011,4:727-730。
- 3.
刘y,yadev Vr,Aggarwal Bb,Nair Mg:黑胡椒(吹笛者Nigrum)提取物和化合物对人肿瘤细胞增殖,环氧化酶酶,脂质过氧化和核转录因子-kappa-b的抑制作用。Nat Prod Communce。2010年,5:1253-1257。
- 4.
联合国粮食及农业组织:农业生产统计。粮农组织,罗马2011年。
- 5.
DiaS JS,Ortiz R:植物育种评论。编辑:Janick J.2011,Wiley Blackwell,纽约,151-224。Janick J.(系列编辑),转基因蔬菜作物:进度,潜力和前景,植物育种评论,
- 6.
Hawkins Rd,Hon Gc,Ren B:下一代基因组学:一种综合方法。NAT Rev Genet。2010,11(7):476-486。
- 7.
Lister R, Gregory BD, Ecker JR:下一个是现在:基因组测序的新技术,转录组等等。植物学报,2009,12(2):107-118。10.1016 / j.pbi.2008.11.004。
- 8.
Morozova O, Hirst M, Marra MA:新测序技术在转录组分析中的应用。基因组学杂志。2009,10:135-151。10.1146 / annurev -染色体组082908 - 145957。
- 9.
Wang Z, Gerstein M, Snyder M: RNA-Seq:转录组学的革命性工具。中国生物医学工程学报。2009,10(1):57-63。10.1038 / nrg2484。
- 10.
RNA-Seq:一种全面的转录组分析方法。《中华医学杂志》2010年第1期。89: 4.11.1-4.11.13。
- 11.
Babraham生物信息学:FASTQC,高吞吐量序列数据的质量控制工具。2012年,http://www.bioinformatics.bbsrc.ac.uk/projects/fastqc/那
- 12.
surgo - groba Y, Montoya-Burgos JI:从下一代测序数据中优化从头转录组组装。基因组杂志,2010,20:1432-1440。10.1101 / gr.103846.109。
- 13.
郑艳艳,赵丽玲,高军,费志:iAssembler:一种用于重组Roche-454/Sanger转录组序列的软件包。生物信息学。2011,12:453-10.1186/1471-2105-12-453。
- 14.
他年代,刘C, SkogerbøG,赵H,王J,刘T,白B,赵Y,陈总:NONCODE v2.0:非编码解码。核酸学报2008,36:D170-D172。10.1093 / nar / gkn294。
- 15.
Gotz S,Garcia-Gomez JM,Terol J,Williams Td,Nagaraj Sh,Nueda MJ,Robles M,Talon M,Dopazo J,Conesa A:高吞吐功能注释和与Blast2Go套件的数据挖掘。核酸RES。2008,36(10):3420-3435。10.1093 / nar / gkn176。
- 16.
Langridge P, Fleury D:利用“组学”进行作物育种。生物技术进展,2011,29(1):33-40。10.1016 / j.tibtech.2010.09.006。
- 17.
Wang L,Li P,Brutnell TP:使用超高通量测序探索工厂转录om。简短的Funct基因组蛋白质组学。2010,9(2):118-128。
- 18.
龚文,沉益,马lg,潘y,杜yl,王dh,杨杰,hu ld,刘Xf,dong cx等:拟南芥克隆和拟南芥转录因子基因的分析。植物杂志(Rockv)。2004,135(2):773-782。10.1104 / pp.104.042176。
- 19.
Jaramillo MA,Manos PS:Phylocy和Pliper(Piperaceae)的花卉多样性模式。我是J机器人。2001,88(4):706-716。10.2307 / 2657072。
- 20.
黑胡椒(Piper nigrum L.)微卫星位点的分离与鉴定。植物保护学报。2009,1(1):209-212。10.1007 / s12686 - 009 - 9051 - 6。
- 21.
基于RAPD标记的胡椒属植物遗传多样性分析。生物技术学报,2010,46(1):72-79。10.1007 / s12033 - 010 - 9281 - 6。
- 22.
Joy N,Abraham Z,Soniya EV:黑胡椒的农艺学重要品种遗传关系初步评价。BMC Genet。2007,8:42-
- 23.
Paterson AH:浏览我们主要农作物的基因组:获取独特信息的策略。中国生物医学工程学报。2006,27(3):174-184。
- 24.
Jiao Y Y, Wickett NJ, Ayyampalayam S, Chanderbali AS, Landherr L, Ralph PE, Tomsho LP, Hu Y, Liang H, Soltis PS, et al .:种子植物和被子植物的先祖多倍体。自然。2011,473(7345):97-100。10.1038 / nature09916。
- 25.
王Z,方B,陈继,张X,罗Z,黄l,陈X,李y:De Novo组装和根部转录组的表征使用Illumina成对序列测序和CSSR标记的CSSR标记(Ipomoea Batatas).BMC基因组学。2010,11:726-10.1186 / 1471-2164-11-726。
- 26.
Shi Cy,Yang H,Wei Cl,Yu O,张Zz,江CJ,Sun J,Li Yy,Chen Q,夏T等:山茶叶中的深度测序转录组显示茶叶主要代谢途径的候选基因 -具体化合物。BMC基因组学。2011,12:131-10.1186 / 1471-2164-12-131。
- 27.
GOFF L,DAVILA J,SWERDEL M,MOORE J,COHEN R,WU H,SUN Y,HART R:AIGO2免疫沉淀识别人胚胎干细胞和神经前体的预测微大研。Plos一个。2009,4(9):E7192-10.1371 / journal.pone.0007192。
- 28.
Ribeiro-dos-Santos A, Khayat AS, Silva A, Alencar DO, Lobato J, Luz L, Pinheiro DG, Varuzza L, assume cao M, assume cao P,等:超深测序揭示了人胃的microRNA表达模式。公共科学图书馆。2010,5 (10):e13205-10.1371/journal. pon.0013205。
- 29.
被子植物基因组大小的进化。中国海洋大学学报(自然科学版)。10.3732 / ajb.90.11.1596。
- 30.
关键词:TIGR基因,基因转录序列,真核生物abstract:核酸学报2000,29(1):159-164。10.1093 / nar / 29.1.159。
- 31。
Garg R, Patel RK, Jhanwar S, Priya P, Bhattacharjee A, Yadav G, Bhatia S, Chattopadhyay D, Tyagi AK, Jain M:鹰嘴豆基因发现和组织特异性转录组分析与大规模并行测序和Web资源开发。植物杂志(Rockv)。2011年,156:1661 - 1678。10.1104 / pp.111.178616。
- 32.
Mizrachi E, Hefer CA, Ranik M, Joubert F, Myburg AA:由Illumina mRNA-Seq产生的一种快速生长桉树的从头组装表达基因目录。基因组学杂志。2010,11:681-10.1186/1471-2164-11-681。
- 33.
夏Z,徐H,翟j,李d,罗h,他c,huang x:Rna-seq分析和veogo转录组合组装Hevea brasiliensis。植物mol biol。2011,77(3):299-308。10.1007 / s11103-011-9811-z。
- 34.
Logacheva MD, Kasianov AS, Vinogradov DV, Samigullin TH, Gelfand MS, Makeev VJ, Penin AA:两种荞麦(Fagopyrum)花转录组的De novo测序和特性分析。基因组学杂志。2011,12:30-10.1186/1471-2164-12-30。
- 35.
applied biosystems: SOLiD™Total RNA-Seq Kit. 2010
- 36.
Martins M:Cutadapt从高吞吐量测序读取中删除适配器序列。embnet.journal。2011,17(1):10-12。
- 37.
宏基因组数据集的质量控制和预处理。生物信息学。2011,27:863-864。10.1093 /生物信息学/ btr026。
- 38.
舒尔兹MH, Zerbino DR, Vingron MA, Birney EC: oasis: Robust de novo RNA-seq assembly across dynamic range of expression levels。生物信息学。2012,28(8):1086-1092。10.1093 /生物信息学/ bts094。
- 39.
天鹅绒:基于de Bruijn图的从头短读汇编算法。中国生物医学工程学报。2008,18:821-829。10.1101 / gr.074492.107。
- 40。
Iseli C,Jongeneel CV,Bucher P:Essscan:用于检测,评估和重建EST序列中的潜在编码区的程序。Proc Int Conv Intel Syst Mol Biol。1999年,ISMB99:138-148。
- 41。
Schiex T,Gouzy J,Moisan A,De Oliveira Y:框架:一种灵活的质量检查和基因预测中原核基因组和嘈杂的成熟真核序列的基因预测。核酸RES。2003,31(13):3738-3741。10.1093 / nar / gkg610。
- 42。
李H,Durbin R:快速准确的短读对齐与挖洞轮转器变换。生物信息学。2009,25(14):1754-1760。10.1093 / Bioinformatics / BTP324。
- 43。
Blast2GO:植物基因组学中功能分析的综合套件。植物基因组学杂志,2008,29 (4):434 - 438
- 44.
利用OrthoMCL将蛋白质组划分到OrthoMCL- db组或聚类到新的OrthoMCL- db组。生物信息学。2011,6:6.12.11-6.12.19。
致谢
作者感谢MSR。巴博萨寻求技术帮助。这项工作得到了Rede Paraense de Genômica e Proteômica(西班牙国家政府Pará/SEDECT/FAPESPA)、PROPESP/UFPA、FADESP、FINEP和CAPES (Coordenação de Aperfeicoamento de Pessoal de Nivel Superior)的支持。
作者信息
从属关系
相应的作者
附加信息
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
构思设计实验:SMCG, ECOM, SMR, AS, HS, WASJr, MICS, SD。进行SMCG、DGP、ECOM、SMR、MCP、ORFL、IT、RTJR、SD实验。提供试剂/材料/分析工具:SMR, AS, HS, WASJr, MICS。撰写论文:SMCG, AS, HS, WASJr, MICS, SD。所有作者阅读并批准了最终的手稿。
电子辅料
12870 _2012_1095_moesm1_esm.pdf
附加文件1:图S1。测序数据集质量值(QV)的统计分析。为读数数据集的每个位置绘制了四分位数,中值和平均值。(PDF 67 KB)
12870_2012_1095_moesm2_esm.pdf.
附加文件2:图S2。从黑胡椒根的2-GS测序中筛选和修剪的reads的大小分布。(PDF 41 KB)
12870 _2012_1095_moesm3_esm.png
附加文件3:图S3。用于cdna图书馆的黑胡椒根的图片。a)70天老植物的全球视图。红色框表示用于提取RNA的根区域。b)用于提取RNA的区域的黑胡椒根的剖视图。(PNG 1 MB)
作者为图像提交的原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Gordo,S.M.,Pinheiro,D.G.,Moreira,E.C。et al。黑胡椒根转录组的高通量测序。BMC植物杂志12,168(2012)。https://doi.org/10.1186/1471-2229-12-168
收到了:
公认:
发表:
关键字
- cDNA图书馆
- 黑胡椒
- 数字基因表达
- 四核苷酸重复
- 基部被子植物