摘要
背景
小的非编码rna (smRNAs)在真核生物的基因调控中起着重要作用。在植物中,关于包括microRNAs (miRNAs)、短干扰rna (siRNAs)和反式作用siRNAs (tasiRNAs)在内的smRNA类的生物发生和作用机制的知识主要是通过对植物的研究获得的拟南芥.近年来,smRNA群体的高通量测序使知识从模型系统扩展到具有更大、更复杂基因组的植物。大豆(大豆)现在有许多基因组学资源,包括完整的基因组序列和预测的基因模型。然而,关于其内源性smRNAs种群和沉默基因的全部补体,我们所知相对较少。
结果
利用Illumina测序和计算分析,我们从大豆的多个组织和器官(包括发育中的种子和营养组织)中鉴定了8个smRNA群体。总共4100万个原始序列reads被分解成135055个独特reads被映射到大豆基因组及其预测的cDNA基因模型中。生物信息学分析用于区分miRNAs和siRNAs,并确定其基因组起源和潜在的靶基因。此外,我们还鉴定出两种大豆TAS3基因同源,推测引导其转录物裂解的mirna,以及可以靶向大豆基因的衍生tasirna,这些基因被注释为生长素响应因子。基于8个smRNA文库中归一化miRNA和siRNA丰度通量的组织差异表达是明显的,其中一些是通过smRNA印迹证实的。我们对这些smRNA群体的整体观察还揭示了smRNA的大小类别在不同组织之间有所不同,与发芽幼苗的营养组织相比,发育中的种子和种皮具有更多数量的24-nt类独特的smRNA。24-nt类是由包括转座子在内的重复元素衍生而来的。与核糖体rna和转座因子家族相关的尺寸类的详细分析显示,22-和24-nt尺寸类的smrna具有更大的多样性。
结论
在种子发育的多个阶段和组织中,内源性smRNAs的通量与大豆的营养组织进行了对比,大豆是世界市场上蛋白质和油脂的主要来源之一。这些smrna在大小、种类、起源的复杂性和可能的目标上有所不同。测序显示某些smrna的组织优先表达以及密切相关的miRNA家族成员之间的表达差异。
背景
高通量测序技术已成为全球小RNA (smRNA)群体分析的最强大方法,揭示了植物和其他真核生物的复杂性水平[1- - - - - -5].有证据表明,通过多种生物遗传途径产生的几种内源性smrna类别需要不同的分子复合物和网络,包括DICER-LIKE (DCL)、ARGONAUTE (AGO)和RNA依赖的RNA聚合酶(RDR)保守蛋白家族[6].在模型植物中拟南芥,其基因组编码4个dcl, 10个AGOs和6个rdr,迄今为止,至少有4个内源性smrna生成途径被发现(由[7- - - - - -9]): microRNAs (miRNAs)、短干扰rna (siRNAs)、事务性siRNAs (tasiRNAs)和天然反义siRNA (natsiRNA)。
microRNAs (miRNAs)来源于由倒置重复伪基因序列组成的miRNA基因,在转录时产生双链RNA (dsRNA)底物,被DCL1/AGO1复合物切割成21个核苷酸(nt)片段。在大多数情况下microrna的基因会产生单个miRNA的多个副本,这些miRNA指导多种基因的转录后调控。虽然mirna在进化上是古老的,但基因组中最近的重复和倒置区域可能是dsRNA的来源,这也引发了21-和22-nt短干扰(siRNAs)的产生,这也主要在转录后水平上起作用。
24-nt类小RNA也由植物特异性RNA聚合酶IVa (Pol IVa)合成的dsRNA加工而成,随后RNA依赖RNA聚合酶2 (RDR2)逆转录,DCL3/AGO4(AGO6)切割成24-nt siRNAs。这些siRNAs产生于高度重复的序列、转座子和逆转录元件,也可以通过与AGO4和甲基转移酶HEN1一起指导DNA甲基化而在转录水平上沉默基因[10- - - - - -12].与mirna不同,siRNA位点产生多个重叠的小rna簇[6,13]通常在两条链上。
事务性siRNAs (tasiRNAs)是miRNA引导的DLC4/AGO1(AGO7)/RDR6复合物转换的产物助教从特定的助教基因转化为dsRNAs,被切割成连续的21-nt片段。tasiRNAs以mrna为目标反式以类似mirna的方式[14- - - - - -16].最后,自然反义siRNA (nat-siRNA)是由DCL2/AGO/RDR6作用于非生物或生物胁迫诱导的dsRNA正义和反义转录对而产生的21个nt片段。它们已被证明是参与植物病原体防御和胁迫反应的基因的转录后调节因子[17,18].
来自不同植物的高通量测序结果揭示了这些smRNA内源群体的丰富性和复杂性。重点是那些高度重复的序列,大小在21到24 nt之间。反过来,smRNA序列长度已经成为DCL酶和产生它们的途径的一个指标。miRNAs大多为21nt,是含有DCL1的复合物的结果。另一方面,大多数代表高重复元件的siRNAs是由植物特异性RNA聚合酶IVa (Pol IVa)合成的双链RNA (dsRNA)经过RNA依赖的RNA聚合酶2 (RDR2)逆转录,再经DCL3切成24 nt的siRNAs。其他长度为21 nt的siRNAs来源于具有反向重复序列或反向重复基因的基因组区域。这些siRNAs很可能是由单链发夹RNA前体在类似miRNA生物发生的过程中加工而成。这些siRNAs中的一些可能代表“进化的miRNAs”,以序列相似的内源性基因为目标。最近,人们发现DCL1/DCL2的低丰度22-nt miRNA/交易siRNA产物与特定的AGO/DCL/RDR2配合物结合,产生dsrna和第二波阶段性的次级(或传递性)siRNAs,这些siRNAs来自其靶点周围的区域[19].21-nt tasiRNAs是mirna定向断裂的产物助教通过涉及DCL4/RDR6复合物的途径转录[16].
六个中的三个拟南芥rdr (RDR1、RDR2和RDR6)在许多植物物种中都有同源物,这表明这些蛋白质是保守的[20.].rdr的功能取决于它们招募的DCL (1-4)/AGO(1-10)复合物。人们可能会认为,植物基因组越大、越复杂,效应蛋白的多样性可能就越高。作为证据,该水稻植株有5个DCLs和19个AGO家族成员,属于7个家族[21,22].这两种植物物种之间的这种变异可能表明,具有更大重复基因组(>60%)的植物,如大豆和玉米,可能在基因沉默复合体的成分、它们产生的smRNA类别以及被调节和沉默的基因方面进化出了额外的变异。初步的计算估计已经确定了大豆中的7个DCL同源物,包括4个典型DCL的高度保守同源副本,这可能是过去4500万年中两到三轮基因组复制的结果[23].迄今为止在大豆中发现的增加的smRNA复杂性的一个例子是大量的siRNAs(21和22 nt),它们沉默了查尔酮合成酶的9个成员(CHS)大豆种皮的基因家族[24].另一方面,在玉米中,在根和茎组织中发现了大量的22-nt siRNAs,根据其起源和靶向基因的不同,它们的功能与21-和24-nt siRNAs不同[4].相比之下,这类22-nt siRNA在其他单子叶植物如小麦、大麦和水稻中缺乏[25].
在一些植物物种中进行的全球smRNA测序研究表明,不同组织/器官中不同种类的smRNA库发生了变化。一般来说,植物细胞具有少量独特且高度丰富的21-nt miRNAs和大量多样的sirna,主要大小为24-nt [1].在拟南芥除了发现smRNA在组织表达上的差异外,siRNA在花序中的数量整体上比在幼苗中的数量大,这表明PolIVa可能受到发育调控[9,26].
在玉米中,85%的基因组由转座元件组成[27],在根中读取数最多的是21nt,而在茎中读取数最多的是24nt。22-nt大小类在两个组织中都有大量的重复,根据基因的起源和靶向区域,有人提出22-nt siRNAs与21-和24-nt siRNAs功能不同[4].
迄今为止,已经报道了四项主要集中在大豆miRNAs的小RNA测序研究(大豆) [28- - - - - -31].在这里,我们记录了通过Illumina短读SBS(合成测序)方法获得的小RNA序列群[32]在黄种大豆品种的多个组织(早期发育的全籽、种皮、子叶、发芽子叶、芽和叶阶段)和黑色种子品种的种皮中。根据现有数据库的注释、序列长度、丰度和组织特异性表达,将每个组织的smRNA群体分为不同的smRNA组(miRNA, siRNA)。通过映射到大豆基因组和预测的cDNA基因模型,确定了许多mirna的基因组起源和可能的靶点。对这些种子和营养组织中存在的所有mirna的家族成员进行了全面分析。8个smRNA文库中基于归一化miRNA通量和siRNA丰度的组织差异表达普遍存在,其中一些通过小RNA印迹证实。我们还关注了siRNAs及其相关基因,以及那些来自包含基因组序列的大量重复26S核糖体DNA (rDNA)和匹配的转座子序列的siRNAs。证据也提供了两个G. max TAS3基因和衍生的tasirna。种子和非种子组织的比较表明,不同组织的小rna的大小类型不同,与发芽幼苗的营养组织相比,发育中的种子和种皮具有更多的24-nt类独特的小rna。
结果
来自多个大豆组织和器官系统的小RNA群体的测序和计算分析
鉴于内源性非编码smRNA在基因调控、发育以及对生物和非生物胁迫的响应中发挥的日益多样化和重要作用,我们确定了大豆植物多个器官中smRNA池的大小和组成。所选择的组织/器官包括非常年轻(开花后12-14天)的完整种子,从中期成熟胚胎中剥离的种皮和子叶,以及从发芽或成体组织中提取的子叶、茎和叶(表2)1以及材料和方法)。低分子量(LMW)或总RNA提取物在Illumina公司按照其制备方案进行进一步分离,在Illumina基因组分析仪上进行SBS测序(见材料和方法)。
如表所示1,共获得近4,100万份原始资料。来自所有库的唯一读取数总计为244,994。在研究的8个库中减去重叠序列后,这个数字被分解为135,055个unireads。每个smRNA群体由6,855 - 38,961个独特reads组成1).这些数字可能代表了在选择的特定发育阶段和最佳环境条件下所研究的大豆组织中大豆小RNA群体复杂性的总体程度。额外的文件1显示每个库中所有135,055个单读、它们的注释和规范化的读计数。在Phytozome数据库中,大多数(107278)或平均80%的独特smRNA签名映射到当前的8X大豆基因组序列(Williams 82, Williams的一条等值线)[33]使用Novalign [34],如附加文件所示1而与Sanger miRNA数据库(miRBase)匹配的相对较少(1804个)[35].许多序列在miRBase或nt(核苷酸)-NCBI数据库中都没有表示[36].
smRNA群体的大小分布揭示了种子组织和营养组织的差异
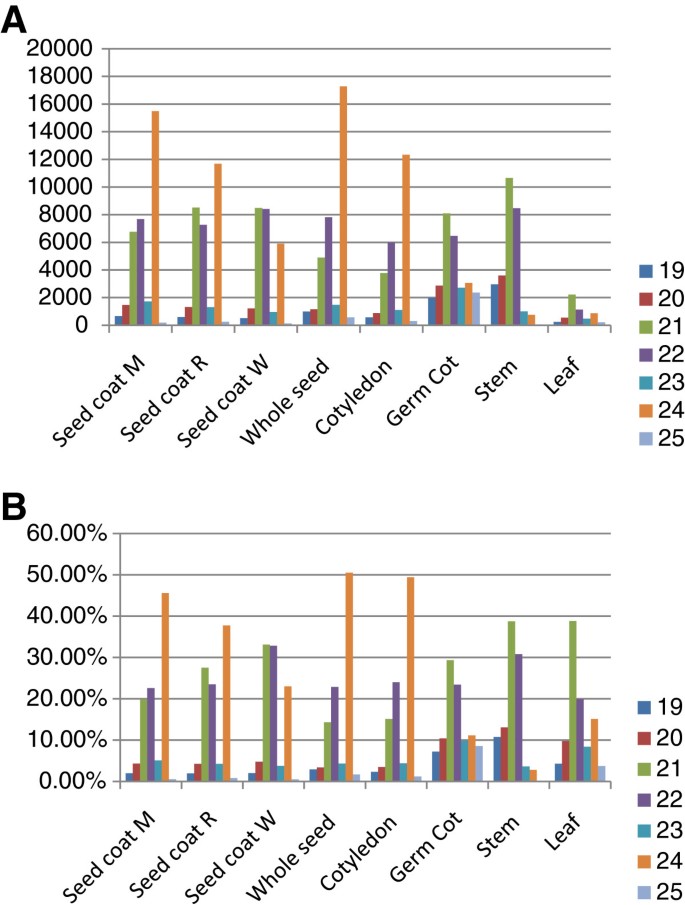
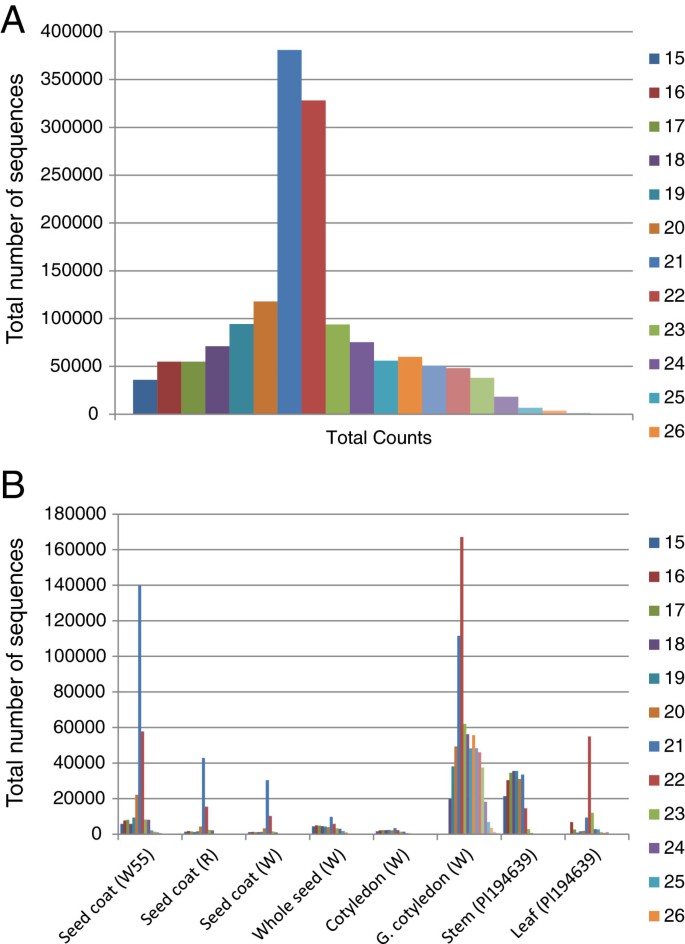
小RNA的序列长度是其生物遗传途径和与其相应smRNA类别相一致的功能的预测因子,因此,检查每个群体样本中单读体的大小范围是有信息的。长度在19 - 25 nt之间的smRNA签名被考虑到图中所示的每个组织样本总体的示意图大小分布1A及B.24-nt smRNAs在种子(未成熟的外层和子叶)文库中是最突出的一类,而在发芽或成体植物的器官(茎、叶和发芽子叶)中,21-nt smRNAs具有最多的唯一reads(图1A及B).种子smRNA文库中数量第二多的是21-nt smRNA,紧随其后的是22-nt smRNA。有趣的是,在非常年轻的整个种子和子叶smRNA文库中,22-nt签名比21-nt签名更丰富。这些结果的意义将在讨论部分讨论。
大豆各组织器官文库中smRNA序列特征的大小分布长度为19-25个核苷酸的独特签名如图所示的颜色代码所示。(一个)唯一签名的实际数目的大小。(B唯一签名总数的百分比。见表1用于库描述。
mirna的差异表达和假定靶标的鉴定
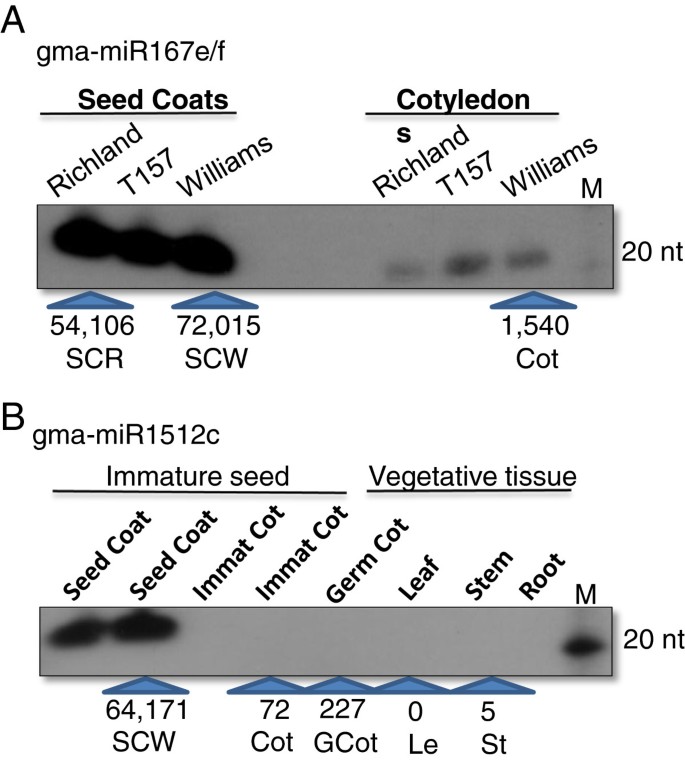
仔细观察不同smRNA组织文库中最能代表每个miRNA的签名的出现次数,揭示了一些被选作进一步研究的差异表达模式。表格2给出了8个smRNA文库中23个大豆miRNAs表达的归一化reads数(计数)/ million total reads。很明显,在代表个别家庭成员的读数数量上存在很大差异。更有趣的是,一些miRNAs在未成熟的种皮或子叶中含量丰富,而在营养组织中含量不高,如gma-miR167e/f(#1)和gma-miR1512c(#4)。通过小RNA印迹,我们证实了gma-miR167e/f(#1)和gma-miR1512c miRNA(#4)在未成熟种皮中的高度优先表达(图)2).
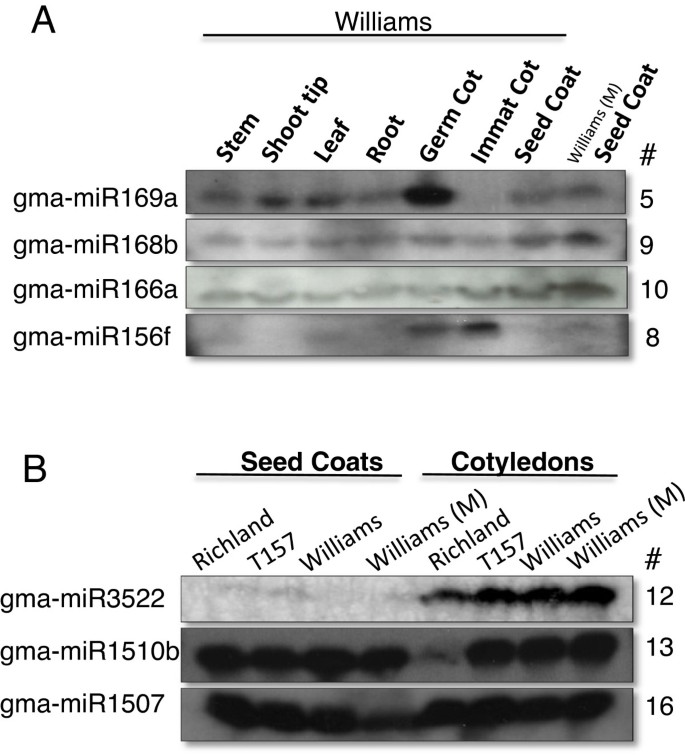
一个smRNA(表中#52与种子或其他营养组织相比,与gamma - mir169a匹配的基因在发芽子叶中含量最高,这一点由图中所示的RNA印迹证实3.另一方面,gma-miRNA168b(#9)和gma-miR166a-3p(#10)存在于测序和印迹实验检测的所有组织中(表10)2和图3).有趣的是,另一个miRNA166序列,gma-miR166a-5p(#11),位于前体RNA茎环的5 '端,在所有组织中积累量非常低,而来自同一RNA前体的3 '端的gma-miR166a-3p则大量积累(表中#10)2).
表格2显示了大豆中几个著名的miR156家族成员,其中一个(gamma - mir156a)在所有组织中积累到相对较高的水平(表中序列#6)2).但最令人惊讶的结果是同一miR156家族的两个miRNAs的组织表达差异,它们与gma-miR156c/d/e/g和gma-miR156b/f相同,其序列仅在T - > g的一个核苷酸跃迁中存在差异(表2)2序列#7 vs #8)。gamma - mir156c /d/e/g序列(含T核苷酸)在未成熟子叶中只有25个reads,而gamma - mir156b /f家族成员(g)在同一组织中有98,674个reads。在两个家庭成员的茎库中观察到相反的表达(13327对9个reads)。如图所示3,一个21 mer探针,反映了gamma - mir156b /f家族成员(表序列#8)2)在未成熟子叶中显示出较高的水平。带有单核苷酸差异的7号序列的探针也显示了相同的模式,因为只有一个碱基对差异的家族成员不能通过杂交来区分(数据未显示)。这些结果清楚地证明了在凝胶-印迹放射自显像中,通过序列读取数而不是信号强度来分析特定smrna丰度的优势。测序数据揭示了两组miR156家族成员在大豆基因组中不同位置的差异表达(表2)2).
表格2还说明了序列#12,它类似于最近发现的伽马- mir3522 [37],在未成熟子叶、萌发子叶和叶片中含量较高,但在未成熟种皮中含量不高。事实上,miR3522在叶片样本610万次总阅读量中占了惊人的65%,在未成熟子叶样本中占了11%。数字3 b证实了其在两个不同品种的未成熟子叶和种皮之间的差异表达。另一方面,最近发现的大豆家族gma-miR1510和gma-miR1507 [28- - - - - -31有特定的家庭成员(表中的#13和#162),在大多数组织中以中高水平表达。最后,其他几个miRNAs(序列#s 5、18、19、20、21、22、23)在发芽子叶、茎或叶的营养组织中相对于未成熟种子组织表现出最高的差异表达,尽管即使是读取数最高的家族成员的序列读取数总体上也较低。
表格2仅代表miRNA家族成员序列的一小部分选择及其在不同组织库中的丰度。图中显示了来自117个家族的约213个不同mirna的4031个序列的完整补充附加文件2它还显示了每个序列在每个组织中发现的归一化读数。该数据集显示了所有相关的miRNA家族和小序列或核苷酸变体。一般来说,每个家族中reads丰度较高的序列很可能是真实的miRNAs。其他长度不同或具有核苷酸差异的序列可以在mirna生成的处理步骤中或在高通量格式的测序错误中从生物学上产生。
通过对大豆基因组的比对和预测发夹重复序列来区分mirna的来源和靶向基因
miRNA基因不断进化,往往源于转录产物折叠成稳定的二级结构时转录的重复序列或伪基因。大多数miRNAs大小为21 nt,由DCL1/AGO1复合物从发夹的茎部剪切。一般来说,单个miRNA的许多相同副本从给定的pre-miRNA转录本的同一链位置被剪切下来。在大多数情况下,miRNA表现出链偏倚,发夹的一侧产生与AGO复合物相关的miRNA,而代表相反链的miRNA*不稳定。然而,在某些情况下,相反链的miRNA*已被恢复,并被指定为5p和3p。为了确定miRNA的起源,通常在200 nt范围内的前体区域必须形成一个能量稳定的发夹环。在表2,我们通过产生稳定的发夹RNA二级结构的基因组位置,提出了大豆中23个miRNA基因的假定起源。gma-miR1512c在种皮中高度表达(序列#4),映射到8X大豆基因组模型的基因组位置Gm02:8609718 [33]如图所示的发夹结构所示4.该位点与Phytozome中任何计算预测的大豆基因模型(称为Glyma模型)无关。该miRNA(序列#4)唯一的其他匹配物和可能的靶标是Glyma08g17790.1,它被预测编码一个富半胱氨酸受体样激酶和Glyma10g21350.1具有绒毛蛋白注释。
差异表达miRNAs的茎环二级RNA结构。表中列出了8种差异表达的mirna2在折叠的前体RNA序列上进行映射和高亮显示。黄色突出显示的是5p miRNAs(#4, #12, #11, #14, #13, #18),在水中是3p miRNAs(#10, #15和gma-miR396a-3p)。通过Phytozome显示了与miRNA基因相关的大豆基因组中的位置(Gm数)和任何低置信Glyma基因模型数(Glyma数)。
与仅在种皮中丰富的gma-miR1512c相反,miR156家族成员序列(表中的#6,#7,#8)2)映射到大豆基因组中至少15个位置,这些位置不编码蛋白质,但能够形成真正的发夹结构,并且可能是这些mirna的起源。miR156家族的靶标是许多编码鳞状启动子结合样蛋白(SBP)的基因模型,这些模型从其他研究充分的系统中已知[38].其他miRNA靶向基因包括注释为生长素反应因子、HD-zip转录因子和ap2样转录因子的cDNA模型。一些mirna的基因组起源落在Phytozome大豆基因组组装的区域,这些区域已被注释为低置信基因模型(表中有星号标记的区域)2).由于所有这些基因都含有能够形成发夹的重复序列,它们很可能是miRNA基因,而不是蛋白质编码基因。附加文件1而且附加文件2包含213个miRNAs序列的完整信息,它们的基因组位置,以及它们可能靶向的Glyma模型。数字4表中显示了一些mirna的预测发夹结构2.正如miRNA #10和#11发夹对所描述的,通常其中一个与之相关的计数明显多于另一端,在大多数情况下,来自环两侧的miRNA的处理错开了2-nt(图4).
序列比对区分miRNAs和siRNAs
在Phytozome的8X大豆基因组数据库中发现的107,279个smRNA序列中[33],绝大多数人在miRBase中没有匹配。这些可能代表新的大豆miRNAs或siRNAs,可以沉默大豆基因。使用八个库中所有唯一smrna的列表(附加文件1),我们首先根据它们的注释对这些序列进行分组,并对包含最多签名的组进行检查,以确定它们的起源(miRNAs或siRNAs)并预测它们的目标。尽管最近已经报道了一些大豆mirna的分析[28- - - - - -31],这是关于在多种大豆组织中发现的小rna的siRNA部分的全球观点的第一份报告。这些siRNAs可能来自于miRNAs靶向的转录本的二次扩增。或者,siRNAs可以从基因组中的反向序列重复中以类似于自然发生的方式产生CHS(查尔酮合成酶)大豆产生的siRNAs我大豆种皮颜色的控制位点[24].我们之前对黄色种皮的种皮文库(文库SCR和SCW)进行了检查,它们积累了大量的种皮CHSsiRNAs与突变的有色种皮(SCM)和子叶库(Cot)相比没有CHSsiRNAs,提供了数据来证明基因型和组织生产CHSsiRNAs。下面我们将描述从8个文库中收集到的可能下调互补基因的其他siRNAs的几个例子。
含有Pentatricopeptide repeat (PPR)的蛋白质
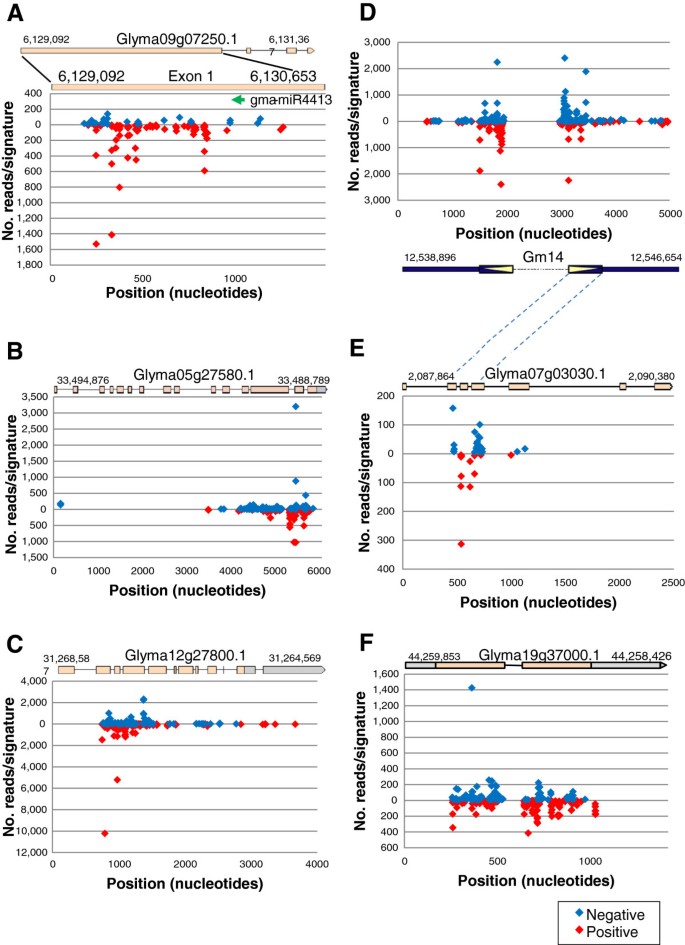
一组标记为pentatricopeptide repeat-containing protein (PPR)的标记有不同的来源,一些标记来自miRNAs,另一些标记来自siRNAs。最丰富的签名(表中#22)2)与gma-miR1508a和gma-miR1508b相似,在所有组织中均有表达,在萌发子叶中表达量最高(7892次)。该miRNA映射到两个起始位点[染色体16和19 (Gm16, Gm19)],并可能靶向四种不同的Glyma cdna,注释为含有PPR重复蛋白。在这个注释组中还有一个smRNA(#23,表2),与Gm19上的反向重复区相匹配,能够形成dG = -61.60的二级结构,与gamma - mir4413相似。它在所有组织中表达水平相对较低,并可能靶向六种具有PPR基序的不同Glyma基因模型。使用领结作进一步检查[39)(或MultAlin[40])发现了一系列115个独特的sirna,它们与Glyma09g07250 PPR基因的第一个外显子的义链或反义链相匹配5).
与大豆基因组序列一致的siRNAs。将具有相同注释的smrna集合与Bowtie的特定基因组序列进行比对。(一个Pentatricopeptide repeat-containing protein (PPR) siRNAs to Glyma09g07250.1。(B生长素反应因子(ARF)对Glyma05g27580.1的siRNAs。(C) TIR结构域耐药基因(TIR- nbs - lrr)对glyma12g278001的siRNAs。(D(F)与D和E中不相关的水通道蛋白sirna与另一个水通道蛋白转运体Glyma19g37000.1相连接,与水通道蛋白基因NIP6-1相似的水通道蛋白sirna。蓝色菱形表示排列在负链上的sirna,红色菱形表示排列在正链上的sirna。(A)中的绿色箭头标记了gma-miR4413的结合位点。虚线连接Glyma07g03030.1 (E)到水通道蛋白反向重复(D)。
总之,这些结果揭示了三个miRNA基因,miR1508a, miR1508b和miR4413的存在,它们可能是一组包含PPR基序的基因的调节因子。PPR蛋白是参与细胞器中RNA加工的许多方面的序列特异性RNA结合蛋白。高度丰富的伽马- mir1508a /b(#22,表2)靶向PPR基因的一个子集,包括glyma09g305001、glyma16g278001、Glyma09g39260.1和Glyma09g39250.1。另一方面,我们的研究结果表明,gamma - mir4413(#23,表2)靶向PPR基因的另一个子集,包括Glyma07g11410, Glyma07g11290, Glyma16g31950和Glyma09g07250,并可能是产生siRNAs的诱导剂,显示与Glyma09g07250外显子1的更广泛区域一致(图5).区分siRNAs与miRNAs的特征之一是它们与靶基因的一个大区域对齐,而miRNAs通常只与与miRNA匹配的21-或22-nt序列对齐。
生长素反应因子
Bowtie分析发现超过400个独特的smRNAs映射到Glyma05g27580,被注释为生长素反应因子(ARF)。这些siRNAs主要排列在6 kb的Glyma05g27580基因3 '端2 kb的区域5 b).在这种情况下,在沉默的情况下CHS基因家族[24],没有一个生长素响应因子的siRNAs与预测的内含子对齐,这表明这些siRNAs是由剪接的mRNA加工而来的。
此外,由95个独特的smrna组成的第二个亚群被标记为生长素反应因子6,并映射到具有生长素反应因子motif的第二个基因Glyma08g10550。在四个种皮文库中发现了该亚群中读数最多的smRNA签名。这两个基因模型Glyma08g10550和Glyma05g27580代表了一个基因家族的成员,因为它们的序列具有高度的相似性。有趣的是,这两个基因的3 '端外显子是两个siRNA亚群的来源。一个g·马克斯在Phytozome中BLAST搜索与siRNAs对齐的2 kb区域,发现Gm13 (Glyma13g29320)和Gm15 (Glyma15g09750)中有另外两个具有生长素响应基序的位点。然而,生长素反应因子亚组中只有9个siRNAs与Glyma13g29320和1个与Glyma15g09750匹配。所有四个相关基因都映射到四个不同的染色体上,生长素反应因子siRNAs的主要诱导因子起源于何处以及如何尚不明显。不管起源在哪里,产生它们的途径都是组织特异性的,因为所有这些siRNAs大部分都是在种皮文库中发现的。总之,这两个siRNA集合可能是由于在种皮中以组织特异性的方式沉默了至少两个生长素反应因子基因。
水通道蛋白
另一大组约400个独特的smrna被标记为水通道蛋白NIP6-1,映射在Gm14中5 kb基因组区域内或附近,包含一个倒置重复序列(621 bp),被大约1,000 nt的未知序列分开(图4)5 d).在含有621-nt序列重复序列的大豆基因组5 kb区域中,发现了两个具有水通道蛋白基序(MIP超家族)的低置信Glyma基因模型(Glyma14g13250和Glyma14g13260)。此外,在Phytozome中对621-nt序列进行BLAST搜索,发现在染色体7、8和15中与几个水通道蛋白Glyma基因模型相似。将这些重复区域与水通道蛋白注释的smrna进行比对发现,Glyma07g03030中外显子2、3和4的区域也与Gm14上的反向重复区域相匹配,与40个唯一的sirna相匹配,不匹配(图4)5 e).在检测的所有组织中都发现了代表该水通道蛋白siRNA组的siRNAs,尽管大多数reads在非常年轻的发育中的种子中发现,其中倒置重复序列的表达可能最强,这可能与年轻种子中靶向水通道蛋白转录本的下调有关。
相比之下,另一组被注释为“可能的水通道蛋白tip型”的smrna似乎只存在于种皮中。近300个独特的smrna映射到19号染色体上的一个区域,该区域被鉴定为水通道蛋白转运体(Glyma19g37000),而其中只有25个标记也映射到编码另一个水通道蛋白推定基因(Glyma03g34310)的3号染色体序列上。然而,这些水通道蛋白基因与在Gm14中发现的反向重复序列没有相似性,这些重复序列创建了上述二级siRNA组的初级siRNA诱导物,因此,这300个与Glyma19g37000和Glyma03g34310相似的种皮组织特异性siRNAs的生物成因仍然未知。将siRNAs与Glyma19g37000基因组序列进行比对,发现它们与该基因的两个外显子相匹配5 f).
其他假定的siRNAs及其潜在靶标
许多匹配siRNAs的其他Glyma模型的注释都预测了它们参与转录或生理过程,例如Glyma13g32810.1,注释为MADS-box转录因子27,它可能翻译参与dna依赖转录调控的转录因子。此外,Glyma09g02920.1被注释为“dsrna特异性核酸酶Dicer和相关核糖核酸酶”,与37个独特的siRNAs对齐。一组334个独特的siRNAs映射到glyma12g278001,被注释为具有TIR结构域的信号转导跨膜受体。MultAlin[40]清楚地揭示了它们是分散的sirna,其中一些数量很高,与这个Glyma模型一致,这可能会沉默这个假定的TIR抗病基因(图5度).
与核糖体RNA基因匹配的小RNA
我们分析了大豆基因组中超过25个不同位置的小rna。其中一类siRNAs与大豆基因组中含有26S和5.8S核糖体rna重复序列的染色体13、15和16的区域相匹配。我们的研究结果表明,这些区域是大小范围很广的siRNAs的来源。这些可能由不同或多种机制产生。由26S核糖体重复序列产生的最多的smrna大小为21nts和22nts(图6).我们注意到,目前的Phytozome大豆基因组没有正确地去除或注释核糖体DNA区域,但显示它们具有具有不同注释的Glyma模型号,可以预测肽域(见讨论)。该核糖体siRNA类的总体百分比从每个文库中的百分之几到萌发子叶文库中的最高约10%不等。
来自转座子和逆转录因子的小rna
植物中使用最多的小RNA途径似乎是沉默重复序列的途径,包括逆转录元件和转座子,并依赖于RNA依赖性RNA聚合酶(RDR2)和dicer样蛋白(DCL3)的活性,以产生大量高度多样化的24-nt sirna (Xie et al., 2004) [10].这些24-nt siRNA序列与重复序列相关,并与argonaute蛋白(AGO4)和甲基转移酶HEN1一起作用,通过帮助DNA和组蛋白甲基化来介导此类重复序列的转录沉默[10- - - - - -12].目前的估计表明,高达60%的大豆基因组由重复序列组成[41].为了识别和分类来自各种重复元件的小rna的来源,使用Bowtie软件对Phytozome的大豆转座元件数据库[33].BLAST分析了41377个24nt长的smRNA标记,发现5187个(12.5%)与95%保真度的重复序列匹配。有趣的是,在所有107279个与大豆基因组数据库匹配的smrna中,41897个(39%)被映射到多个(11 - 103)基因组位置。在所有与转座子元件数据库(5187)匹配的24-nt smrna中,只有762个与不同的基因组位置有超过11个匹配(从11 - 51),这表明至少有6767个在大豆基因组中有超过50个(50 - 103)匹配的额外24-nt smrna可能参与调控在目前可用的大豆转座子元件数据库中没有显示的重复序列[33].或者,许多24-nt smrna可能是沉默非转座子相关重复序列和非重复序列的结果。
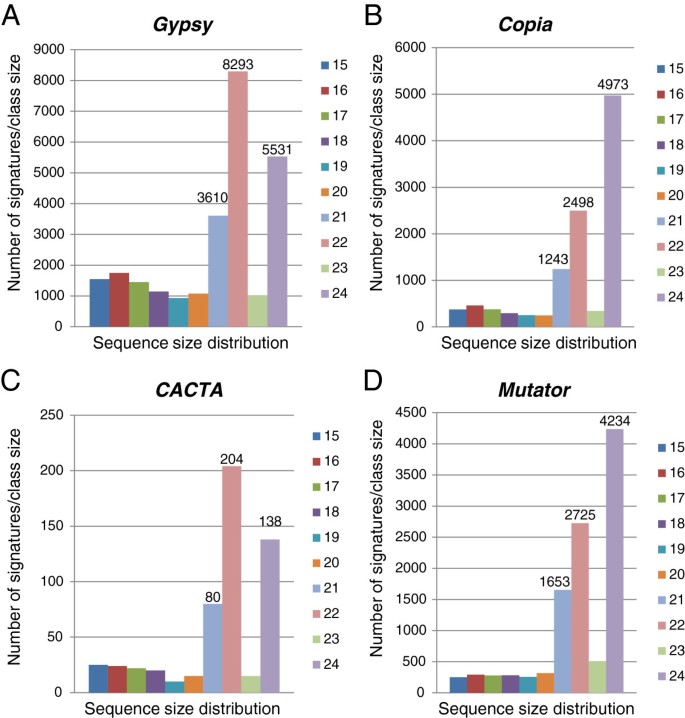
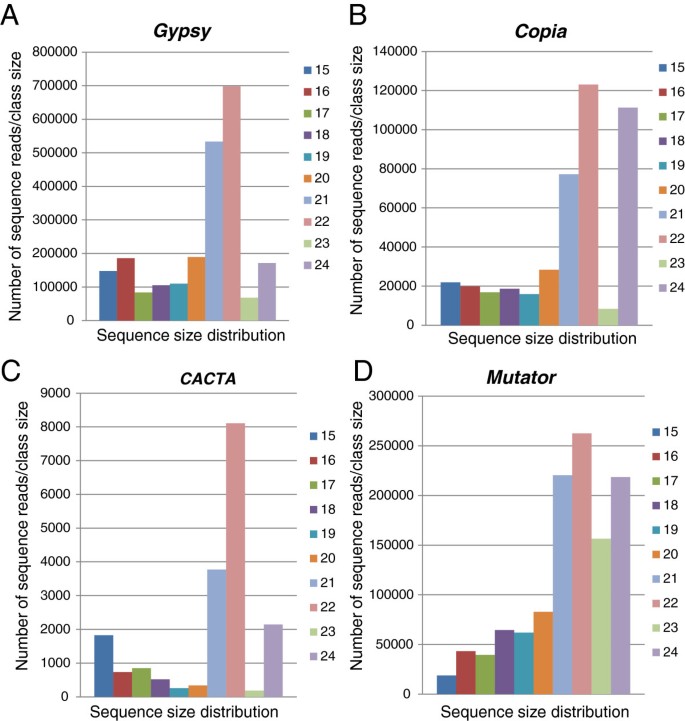
一个额外的搜索使用不同的g·马克斯(Williams 82)转座元素数据库[42]进行了研究,以从整个集合中识别可以派生或链接到重复元素和特定TE类的smrna。在te数据库中,共有54,281个独特的smRNA签名与序列匹配,其中27,731个是与基因序列匹配的吉普赛元素族,11397突变, 11165年Copia, 1384年PIF-Harbinger, 1377 l1, 553CACTA, 311个未知家庭,170个螺丝管, 76年帽子, 75年Tc1-Mariner和42发出难闻的气味.虽然与重复元素相关联的唯一签名的大小范围从15到35 nt不等,但24 nt大小的类别是最多的(17,206),其次是长度为22 nt的类别(14,024)(图7一个).当考虑到每个签名的序列总数时,22-nt大小类是最丰富的(1,107,944),其次是21-nt(849,187)和24-nt类(597,566)(图7 b)表明24 nt的签名比22 nt和21 nt的签名更多样化。在分析过程中,观察到极为丰富的大豆gma-miR3522 miRNA,之前提到在叶片文库中占390万个reads,在所有八个文库中占480万个reads,不精确匹配两个ltr两侧的单个位置(Gm02:21389789.21394849),与a吉普赛元素。这个21-nt靶向位点,在Bowtie比对中允许2-nt错配,它不会在基因组的其他地方完整地重复,而是在其他10条染色体上以退化的形式重复。在这些位置,不完美的21-nt靶区被截断,将5 '端的16-nt与3 '端的5-nt分开。如果吉普赛结果表明,尽管miR3522 21-nt序列中存在2-nt错配,但该基因仍可能被miR3522沉默。然而,我们没有发现与Gm02互补的小rna吉普赛在对所有8个小RNA文库的蝴蝶结搜索中发现,该元素不太可能在所检查的组织中表达和沉默。不管miR3522对这个逆转录因子家族的潜在作用如何,miR3522的高丰度与逆转录因子LTR末端的重复性质无关,它的序列在其他位置甚至更加退化,因此,图中所示的与转座子匹配的小rna的大小分布分析中删除了这个极其丰富的miR3522序列7.
一个有趣的观察是附属于每个TE家族的独特siRNAs数量的明显大小偏好,如图所示8.这就提出了失活机制的可能性Copia(图8 b),突变(图8 d)相关序列主要生成24nt smrna,而失活机制吉普赛(图8),CACTA(图8 c)元素主要产生22 nt smrna。然而,当考虑到每个签名的总读取次数时,在所有四个TE家族中,22-nt序列的数量都超过了24-nt序列9).因此,22-nt smRNA类似乎是大豆中大多数TE家族沉默的相关参与者。
大豆假定的反式作用sirna和TAS3相关基因
在拟南芥miR173, miR390和miR828促进一类特定的小rna的加工,称为反式作用小干扰rna (tasiRNAs),它们反过来针对其他mrna进行转录后下调。四个家庭助教基因已经在拟南芥miR173触发tasirna的处理TAS1而且TAS2基因,miR390来自TAS3的miR828TAS4[14- - - - - -16,43].tasiRNAs处理的一个特征是助教从miRNA结合位点开始,mrna以21 nt为间隔进行分期[16].
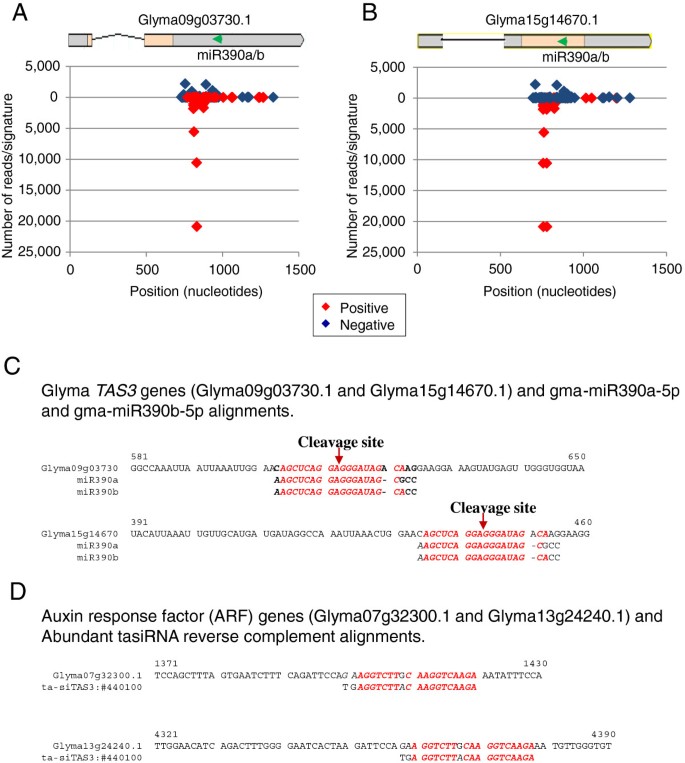
在大豆中,miRBase中至今未输入miR173或miR828序列。对我们整个smRNA种群的搜索发现没有与miR173相关的序列,这似乎是唯一的拟南芥.然而,我们发现了两个推测的miR828基因座(Gm12:3213774。3213795和Gm11:9033043。9033064,图10)以及一个假定TAS4基因(Glyma19g44660)的序列相似性拟南芥同行。miR828的低表达仅在种皮smRNA文库中发现3.)和没有tasirna派生自假定TAS4基因被发现。相比之下,miRBase中已经输入了两个miR390基因,分别映射到Gm3 (miR390a)和Gm14 (miR390b)。此外,我们还发现了另外5个能够使用RNA-形成稳定二级结构的位点mfold,其中四种可能编码mir390a样miRNAs,另一种编码mir390b样序列(图10).表格3.显示在许多组织库中,gamma - mirna390a和gamma - mirna390b的序列计数水平为数百。
至少两个具有数百个reads的miR390家族成员基因的表达水平表明这些miRNAs靶向该基因同源的可能性拟南芥TAS3基因。在Phytozome中使用核苷酸- blast搜索AthTAS3基因组序列(NR_022742 = gb|CP002686.1: 5861491-5862437)鉴定出两个位点大豆9年级和15年级。这两个基因都与两个预测的低可信度基因模型Glyma09g03730和Glyma15g14670一致,同源性为87%,表明至少存在两个假定的TAS3基因在大豆.miR390a-5p和gma-miR390b-5p均与上述两种特征吻合TAS3基因发现它们在两个基因的同一区域分别有17个和18个相同的匹配(图11 c).这些结果表明,这两种miRNAs都可以靶向这两种基因的转录本TAS3基因和调解破裂。断裂的mRNA产物将被RNA依赖的RNA聚合酶6 (RDR6)的活性转化为双链RNA,随后是dicer样4 (DCL4)介导的切割,以加工阶段性的tasiRNAs。
为了确定tasirna的存在和性质,从这些假定中推导出TAS3转录本中,Glyma09g03730.1基因组序列被用于大豆smrna的Bowtie搜索。收集了218个与Glyma09g03730.1基因组序列分期、21-nt间隔一致且没有错配的tasiRNA唯一签名(图11个而且11 b而且附加文件3).在茎中发现了读取最多的tasiRNA签名,分别为2999个和1593个标准化读取。所有tasirna均排列在低置信度Glyma09g03730.1基因模型的3'UTR上。两个假定的TAS3基因具有相似的序列,但不同的预测结构。这些tasiRNAs是从Glyma09g03730.1的3'UTR区域生成的11个),但它们可能来源于Glyma15g14670.1中的一个外显子和3'UTR(图11 b).
在拟南芥,TAS3tasiRNAs通过抑制生长素反应因子(ARF- 2, - 3和-4)调节叶片形态、发育时间和侧根生长速率(Marin et al.,2010).识别假定的大豆以ARF靶基因最为丰富TAS3tasiRNAs,对Phytozome中标注为生长素响应因子的12个Glyma模型序列进行比对(ARF)进行检查。在tasiRNA靶位点核苷酸匹配最多(18个)的转录本是Glyma07g32300.1和Glyma13g24240.2(图11 d).这些结果反过来预测了这些假定的大豆ARF基因在发生时可能受到复杂机制的调控拟南芥。
讨论
大豆miRNA基因变异及其在种子和营养组织中的表达
从代表大豆植物多个组织/器官的8个文库中提取了4100万个smRNA序列,对其中存在的miRNAs进行了全面分析。至少有4031个独特的签名,对应于213个mirna,代表117个家族,每个家族中的每个序列都被详细描述(附加文件2).对每个签名的读取数的检查揭示了在所有miRNA家族成员的高通量群体中发现的所有长度或序列变化。因此,在代表种子和幼苗营养组织的8个文库中每个独特签名的表达模式的通量被详细描述。小RNA印迹验证了某些家族成员在种皮或未成熟种子子叶中的优先表达(表2)2,数据2而且3.).一些miRNAs在某些组织中非常丰富,如miR167家族成员和gma-1512c,它们在多个样本的种皮中非常普遍。其他几种在发芽子叶、茎或叶中表现出优先表达。一个miRNA, gma-miR3522,在未成熟子叶中占总reads的11%,在1个月大的扩展叶库中占总reads的65%。对编码miR3522的基因Glyma15g06080的启动子分析发现,该基因含有组织特异性和增强基因表达的顺式作用调控元件(附加文件4).在叶片样本中,与所有其他文库相比,独特的小rna的多样性减少了4到5倍(表2)1).这是否源于测序反应中聚类形成过程中高度富集的gamma - mir3522的优先扩增,尚不清楚。然而,所有其他文库样本都产生了27-38,000个不同的序列,但一些单个miRNAs签名重复占多个种皮文库总数的1-5%。因此,高数量的mirna在本质上可能是生物的。
特别令人感兴趣的是同一miR156家族的两个亚群的不同组织表达。尽管这两个亚群的序列仅在一个核苷酸上存在差异,但在发育中的子叶中,gamma - mir156c /d/e/g成员的表达水平非常低(25次计数),而gamma - mir156b /f成员的表达水平很高,达到98,674次归一化计数,接近子叶样本中相同smRNA群体的10%。茎和叶的表达则相反2).发现的gamma - mir156c /d/e/g签名与6条染色体中至少7个具有miR156基因编码潜能的位点相匹配。考虑到发育中的子叶中reads的数量较少(25),一种可能性是这7个miRNA基因中的许多以组织特异性的方式被抑制。分散在至少六条染色体上的这么多miRNA基因的协调调控可能涉及到一种转录因子[44].总的来说,大豆中的miR156基因家族由至少18个假定的差异表达的成员基因组成。成熟的miR156家族成员已被证明靶向和沉默SBP转录因子家族的基因拟南芥[38].我们的研究结果表明,高通量测序在定量区分成熟miRNA序列中具有单核苷酸多态性的miRNA家族成员的差异表达方面具有强大的能力,这是用印迹法无法做到的,用定量RT-PCR也难以实现。
综上所述,从表中大豆miRNA序列的选择2而且附加文件2,很明显,归一化读取计数的组织差异在许多已知的广泛保守于植物系统的miRNAs中是明显的,以及一些最近被发现是大豆特异性的miRNAs [28- - - - - -31].此外,通过将我们样本中的大豆miRNAs的起源映射到能够形成稳定的二级RNA发夹结构的基因组位置,我们确定了31个不同大豆miRNAs的新家族成员。基于序列相似性,我们还确定了大豆miRNAs的许多可能靶点。
miRNA前体5 '端和3 '端的差异表达
一些miRNA基因被观察到在RNA发夹前体的5 '和3 '端编码两个miRNA。我们记录了3个来自3 '端(3p)的miRNA签名的丰度高于或低于来自5 '端(5p)的miRNA签名的丰度。
表中给出了三个这样的例子2和图4: miR166a (#10, #11), miR1510a(#14, #15)和miR1510b(#13, #15)。茎环两端明显不同的序列在所有这些病例中重叠2nt,并在非常不同的水平上积累。有时3p-miRNA的积累水平高于5p-miRNA (miR166a),但也观察到相反的情况,如miR1510a/b。在发夹3p侧加工的miRNA是否在生物胁迫下被诱导,正如在研究中发现的一些miRNA拟南芥[45]是未知的,但这两种gma-1510a/b-3p miRNAs都可能靶向参与抗病途径的蛋白质,正如序列匹配所预测的那样(表2)2).表中没有显示的另一个例子2但表达方式相似的是γ - mir482 -5p和它的-3p对应物。
基于恢复的siRNAs鉴定可能被沉默的基因
尽管miRNA基因是由基因组中的一个小发夹结构定义的,但siRNAs的起源可能不那么明显,但通常与较大的倒置复制相关,这可能刺激dsRNA或反义RNA的产生。或者,它们可能源于miRNA/siRNA定向转录本裂解,随后从断裂的dsRNA和包含更广泛转录本区域的siRNA片段中RDRP合成更长的dsRNA。有助于区分mirna和sirna的关键区别之一是smrna的排列分布与它们来源的基因序列。miRNAs通常排列在基因序列的~ 21-nt区域,并在代表一条链的拷贝数上优先偏向于相反链,因此,它导致相对较少的不同签名。在sirna的情况下,序列中有一个扩展区域与许多不同的签名完美或接近完美匹配,这些签名覆盖了一个假定沉默基因的两条链,尽管只有与mRNA链互补的smrna在基因的持续下调中有效。
尽管人们对植物中的miRNAs了解很多,但对siRNAs的了解却相对较少。sirna改变植物表型的一个明显例子是大豆CHSsiRNAs已经被证明可以下调CHS显性大豆种皮色素减少的基因家族我或我我等位基因在我轨迹(24].它的起源CHSsiRNAs是一个倒置的重复簇CHS基因而不是miRNA。染色种子的突变被证明是重复簇区域的缺失,否定了dsRNA和的产生CHS导致CHS酶活性和色素种皮的siRNAs。
smrna与潜在Glyma模型转录本的排列模式是区分siRNAs和miRNAs的一种方法。以CHSsiRNAs [24],我们寻找与典型miRNA相比,与更广泛的Glyma模型区域一致的smrna。通过注释组对smrna进行分类,可以发现假定沉默的基因,在某些情况下,还可以发现可能触发沉默的miRNA或siRNA起源。其中包括标记为PPR重复序列的标记,分析显示了3个靶向多个PPR重复序列基因家族的miRNA基因的表达。序列miR1508a和miR1508b(表中的#22)2)在所有组织中均有表达,但以发芽子叶为主,归一化计数为7892 reads / million。他们的目标是一组PPR基因,包括Glyma09g30500、Glyma16g27800、Glyma09g39260和Glyma09g39250。另一方面,miR4413(表中的#232)有可能靶向至少6个PPR基因的不同家族,包括Glyma09g07250。此外,我们发现miR4413是一系列siRNAs的诱导子,这些siRNAs起源于Glyma09g07250.1的外显子1(图5).我们从Multalin图中为Glyma基因和衍生siRNAs的序列比对5方法获得的分布模式相同G.最大TAS基因和gma-tasiRNAs。然后我们可以推断,来自PPR基因(Glyma09g07250.1)的siRNAs的加工也是分阶段的。
另外两个可能沉默的基因Glyma05g27580(图5 b)和Glyma08g10550(未显示),都编码假定的生长素反应因子,由两组独立的smRNA揭示。由于额外的相关基因组序列位于不同的染色体上,因此很难破译能够诱导产生与这两种生长素反应因子基因匹配的sirna的驱动机制。然而,它似乎是组织特异性的,因为大多数siRNAs是在种皮文库中发现的。这可能是种皮特有的沉默的另一个例子,正如丰种所表现的那样CHSsiRNAs [24].Glyma12g27800编码一种含有TIR-motif的抗病蛋白,被确定为334个独特siRNAs集合的来源(图5度).其他基因Glyma13g32810和Glyma15g06490被标记为“Mads-box转录因子27”,被发现可能是77个独特siRNAs集合的来源。
在一个案例中,smRNA的起源和潜在的沉默基因都被鉴定为一个特定的水通道蛋白基因家族。Glyma07g03030基因被标记为水通道蛋白NIP6-1(图5 e)可以被至少41个来自Gm14上发现的倒置重复序列的主sirna靶向(图5 d).从倒置重复序列和Glyma07g03030衍生的水通道蛋白sirna在所有组织中都被发现,这表明它们是组成型生成的,并且能够靶向该水通道蛋白家族成员基因。相比之下,第二组水通道蛋白转运体siRNAs来自Glyma19g37000(图5 f)和Glyma03g34310基因,它们不是由Gm14中倒置重复序列中产生的主要siRNAs诱导的(图5 d),因为水通道蛋白重复序列与Glyma19g3700和Glyma03g34310水通道蛋白转运体基因的序列相似性很小。诱导产生这第二种水通道蛋白siRNA组的机制仍未确定,但基于与Glyma19g37000.1排列的siRNAs的组织特异性发生,它似乎仅限于种皮(图5 f).
包括核糖体基因和转座子在内的高重复基因组位点的潜在沉默
在研究与大豆基因组中10多个位置匹配的smrna的性质时,我们发现其中许多是来源于核糖体RNA基因(rDNA),代表26S rRNA序列(AK286061.1)和18S rRNA基因(X02623.1),尽管这些基因组区域的注释在Phytozome中没有显示为rDNA基因。相反,Phytozome将Glyma模型号和肽基序分配到这些编码区域,包括一个只与26S rRNA序列(AK286061.1)相似的丝氨酸-苏氨酸蛋白激酶基因(Glyma16g29220)和几个预测基因,如调抑素(Glyma13g11820)、聚合酶Rpb1 c端重复序列(Glyma13g11940)和铜结合八肽(Glyma13g11980)。它们出现在多达14个重复序列中,包含26S rRNA (AK286061.1)、18S rRNA (X02623.1)或5.8S rRNA (FJ980442.1)基因。最近,一项研究[46]的研究表明,在许多原核生物和真核生物的测序研究中,rDNA基因的错误注释会产生假阳性匹配,因为这些错误注释已经在GenBank和SwissProt等数据库中传播。Gm13中超过15个含有多个rRNA重复序列的Glyma模型可能构成核组织者,可保护核糖体转录本不被沉默。补充这些核糖体基因序列的siRNAs可能来源于其他染色体重复的类似序列,如上述Glyma16g29220(一种丝氨酸-苏氨酸蛋白激酶基因)中的26S rRNA插入,也在Gm05、Gm15、Gm14和其他几个染色体中发现。
在8个文库中发现的核糖体siRNA序列的数量在发芽子叶文库中从百分之几到大约10%不等。这些siRNAs丰度的波动是否反映了核糖体相关siRNAs最高的组织中某些核糖体基因的生理性沉默尚不清楚。然而,尺寸分布表明这些是真正的小rna,而不是rRNA降解产物,因为smRNA尺寸在21 - 22 nt范围内达到峰值,如图所示6.同样的事情也可以对图中显示的假定沉默基因对齐的siRNAs说5.
对与重复序列匹配的smrna进行了全面分析,包括可用的大豆转座因子数据库。来自不同TE家族的smRNAs具有明显的大小偏好,提示可能存在不同的相互作用机制Copia(图8 b),突变(图8 d),其中24nt签名占主导地位,而吉普赛(图8),CACTA(图8 c)家族中22-nt特征最为丰富。当考虑到每个签名的总读数时,这种差异就模糊了,因为所有TE家族的22-nt签名都是最丰富的(图9).因此,22-nt smrna似乎是大豆中大多数TE家族和重复序列沉默的主要组成部分。
有趣的是,在一项全球smRNA大小分布分析中,我们发现24-nt smRNA是种子(未成熟的被毛和子叶)文库中最突出的一类,而在发芽或成体植物的器官(茎、叶和发芽子叶)中,21-nt smRNA具有最多的唯一reads(图1A及B).种子smRNA文库中数量第二多的是21和22 nt smRNA。然而,对于非常年轻的完整种子和子叶smRNA文库,22-nt的独特签名比21-nt的更丰富1A及B).有人提出,22-nt smRNAs需要作为AGO/DCL/RDR2复合物的向导,以驱动相相次级siRNAs的第二RNA链合成底物[19].但是,这个大小类可以是其他过程的结果,并提供其他功能。在玉米中,在根和茎组织中发现了大量22-nt siRNAs,根据它们的起源和基因的靶向区域,它们的功能似乎与21-和24-nt siRNAs不同[4].在大豆种皮smRNA群体中,22-nt smRNA与21-nt一样丰富,这表明22-nt smRNA在种皮中也有一定的作用。
与发芽子叶和营养茎叶组织相比,为什么24-nt smRNAs是种子组织中数量最多的独特序列,这很有趣。这可能表明,重复序列如转座元件序列在发育中的种子组织中被积极转录,作为siRNA生成的底物,而siRNA生成反过来又通过DNA/组蛋白甲基化介导重复序列的基因沉默。在拟南芥,通过重新激活种子胚乳中的转座因子,产生大量可移动的24nt- sirna,从而增强了胚胎中转座因子的沉默[47- - - - - -49].请注意,大豆文库中不同的24-nt smRNAs数量最多(占独特序列总数的50%)是在非常年轻的种子(12-14 DAF)和未成熟的子叶中发现的(图1A及B).
在大豆种子组织中,24-nt类含量最高拟南芥在野生型(Col-0)花序组织中,24-nt siRNAs的数量是21-nt大小类的3倍和10倍[1,10].同样,在玉米未成熟穗中,24-nt smrna的数量是21-nt大小类的2.5倍[25].与大豆茎和叶的结果相反,21-nt签名比24-nt smrna更丰富,在玉米芽和拟南芥叶片中,24 nt smRNAs数量超过21 nt smRNAs [4].然而,在拟南芥幼苗中,21-nt类smRNAs的丰度略高于24-nt类,类似于大豆茎中的大小分布[1].
的识别大豆反式作用sirna和助教基因
寻找可以指导假定反式作用siRNAs的生物发生的mirna助教大豆基因同源物鉴定出两个miR390家族成员。这些gamma - mir3390也可能是tasirna的诱导物G. max TAS3基因Glyma09g03730和Glyma15g14670的同源性为87%AthTAS3.这一过程在大豆中发生的证据是存在397个与两种假定大豆一致的tasiRNA标记TAS3基因。此外,至少有两个大豆ARF基因Glyma07g32300和Glyma16g02650被选为最丰富的可能靶点TAS3-衍生的tasirna(图11 d).这些结果预测了几种假定的大豆ARF基因可能受miR390/调控TAS3-衍生的tasiRNAs机制,因为它发生在拟南芥在哪里TAS3tasiRNAs通过抑制ARF2、3和-4基因调控叶片形态、发育时间和侧根生长速率。生长素信号因子对miR390的正负反馈调控维持ARF在一定浓度下的表达,该浓度将决定各种器官形成的发育时机[50].因为在拟南芥劈理的TAS3其独特之处在于它需要miR390/ARGONAUTE7 (AGO7)复合物的特定作用才能产生tasiRNA [51],我们的结果可能预示着大豆中存在ago7样功能。
结论
通过高通量Illumina测序对8个smRNA序列群体进行分析,揭示了在最佳条件下生长的大豆植株中存在的非编码smRNA的全球分布。代表大豆5个组织/器官的8个smRNA序列群,总共有4100万个原始序列,分解成135055个独特reads。所有独特的序列reads均根据其在起源组织/器官、基因组起源和可能的靶基因中的丰度进行分类和特征描述。全面分析从4013个独特的miRNA序列中发现了117个大豆miRNA和213个家族成员。一些mirna的丰度非常高。使用寡核苷酸探针对smRNA序列进行反补体的印迹,证实了不同的未成熟种子或营养组织中某些mirna的表达差异。分析了mirna的预测靶基因,包括家族成员表现出高度差异表达的miR156家族成员。一些被加工成sirna的序列被确定为生长素反应因子和水通道蛋白,它们可能在种子发育过程中起重要作用。基于同源的拟南芥TAS3基因上,两颗大豆相当TAS3经鉴定,gamma - mirna390a /b家族成员具有靶向性。miRNA引导大豆裂解后TAS3转录本,21个核苷酸的tasiRNAs被处理,反过来,可以靶向具有生长素反应因子基序的基因。
未成熟种子与营养组织的smRNA类别之间的差异被注意到,种子组织可能具有显著更高的24-nt大小类别的百分比,代表来自沉默重复区域的sirna。在与重复序列相关的包括rnas和转座子的smRNAs中,22-nt smRNAs也是一个高度丰富的类别,表明其在大豆序列重复沉默中的相关作用。
方法
植物材料和遗传命名法
的大豆本研究使用的品种来自美国农业部大豆种质资源收藏(美国农业部作物科学系/伊利诺伊大学农业研究所;乌尔班纳,IL)。各株系的基因型见表1.所示位点的所有系均为纯合子,为简洁起见,仅显示其中一个等位基因。
植物在温室中生长,每一株系至少采集四株植物的组织。叶子和茎是从两到四周大的植物中采集的。茎样本由一厘米大小的碎片组成,直接位于单叶叶的底部。叶片样品由第一个三叶叶片在一周内取样制成。发芽子叶采自2周龄的幼苗。根据整个种子的鲜重,从不同发育阶段的种子中解剖出种皮和子叶。本研究使用了50-75 mg重量范围内种子的解剖种皮和子叶。开花后12-14天收集幼小的完整种子。花朵在完全开放的第一天(第0天)被标记,并被追踪到所需的发育阶段。年轻的豆荚在12-14天后收获,整个种子在奥林巴斯SZ61显微镜下解剖(梅尔维尔,纽约)。 Whole young seeds were placed in microfuge tubes kept on ice during harvest and dissection. The collected very young whole seeds were fast frozen in liquid nitrogen upon collection and stored at -80°C until further use.
总RNA和小RNA提取
从所选发育阶段(开花后12-14天)的非常年轻的完整种子中提取RNA,使用改良版的McCarty [52)程序。使用FastPrep FP120 (Savant Instruments, Holbrook, NY)将快速冷冻的种子细磨20-30秒。取350 μl完全RNA提取缓冲液(0.1 M Tris-HCl;0.2 M NaCl;20mm EDTA;10mm二硫苏糖醇;16mm巯基苯并噻唑;Sarkosyl 2%;在每个样品中加入15 mM巯基乙醇),用苯酚-氯仿和氯化锂沉淀法提取总RNA [53,54].
对种皮、子叶、叶和茎样品采用标准苯酚氯仿法提取总核酸[54]或TRIzol试剂(Invitrogen, Carlsbad, CA, USA)方法[55].采用Wang等对Williams 55系种皮和子叶进行预处理[53总核酸提取前的程序。对于种皮和子叶样品,如前所述分离出低分子量(LMW) rna [56]稍加修改[24].
RNA提取用于凝胶印迹分析
对于本文报道的小RNA凝胶印迹,使用我们的标准方案分离全部RNA [54]除了LiCl沉淀步骤被取消。值得注意的是,该方案使用了储存在-20°C的冻干组织,结果显示小rna被很好地保存了下来。
总RNA (50 μg)在20 μl 25%甲酰胺中浓缩,70℃变性10分钟。变性的rna在15%聚丙烯酰胺7 M尿素变性凝胶上进行分离,然后使用Bio-Rad Trans-Blot仪器(Bio-Rad, Hercules, CA)在100 V下转移到hybone - nx(或N)膜(Amersham, Piscataway, NJ)。膜经过干燥和紫外交联(Stratalinker, Stratagene, La Jolla, CA)。预杂交用50%甲酰胺、7% SDS、0.05 M NaHPO进行4/不2阿宝4、pH 7.0、0.3 M NaCl、5X Denhardt 's溶液和100 μg/ml剪切的变性三文鱼精子DNA,在40℃下孵育至少2小时。在同一溶液中加入变性的[γ-]进行杂交32P] ATP标记的低聚探针在40°C浸泡15-20小时。过滤器在2X SSC和0.2% SDS中在40°C下洗涤15分钟,并将其暴露在带有增强屏的Hyperfilm (Amersham, Piscataway, NJ)中过夜或24小时。
小RNA反补体DNA寡聚探针为T4 Polynucleotide Kinase (PNK) 5 '末端标记[γ-32P] ATP使用Promega 5 '端标记试剂盒(猫。#UD2010)。标记的寡聚探针在70°C下变性10分钟,然后进行杂交。在杂交之前,过滤器在40°C预杂交至少2小时。
为了准确测定siRNA的种类,使用了一个RNA阶梯(10 - 150 nt),它已经用[γ-进行了放射标记32P] ATP遵循Ambion, Inc. (Austin, TX)的Decade™标记试剂盒提供的协议。
小RNA文库测序及数据分析处理
在Illumina公司(San Diego, CA)使用SBS(合成测序)技术对多个组织样本中的小rna进行凝胶纯化、克隆和测序。简单地说,每个样品的纯化LMW RNA部分(或总RNA) 2.5 - 5 μg提供给Illumina,用于后续质量检查,在TBE缓冲液(45 mM三硼酸盐,pH 8.0, 1.0 mM EDTA)中,用含有7 M尿素的15%聚丙烯酰胺凝胶分离。一个含有15到35个核苷酸的rna的凝胶切片被切除并洗脱。凝胶纯化的小RNA被连接到3 '适配器(TCGTATGCCGTCTTCTGCTTG)上,得到的小RNA文库使用Illumina遗传分析仪进行测序。序列信息通过Illumina firerest和Bustard应用程序从图像文件中提取。
从上述文库深度测序中共获得了40,814,540条长度为35个碱基的reads。在去除人工产物(多个接头连接或空结构的产物)后,仍有244,994个序列有待分析。使用子字符串TCG作为适配器开头的唯一标识符(TCGTATGCCGTCTTCTGCTTG)执行适配器修整。修剪后的小rna的大小在15-35-nt之间,其中20%的小rna的大小为21- nt。
然后将适配器修剪过的序列置于脚本count.pl*中,该脚本将相同的序列相加,生成一个合并的fasta文件,其中包含给定库中每个惟一序列的原始计数。计数小于的序列k=5(或10或16)作为可能的工件被删除,以减少后续步骤的计算成本。k在较早的总读数< = 300万(M)的序列库中设置为5,在读数为6 M的库中设置为10,在最新的>=12 M的库中设置为16。在以这种方式处理每个库之后,序列以非冗余的方式导入到数据库中,即,一个序列在数据库中只出现一次,而与拥有它的库的数量无关。该数据库分别存储每个库中每个序列的读取数。数据库中存在任何序列的阈值将由最小值表示k至少在一个图书馆里阅读。本研究共从8个文库中鉴定出135,055个独特的小RNA序列。
注释
我们数据库中的每个序列都用于对NCBI核苷酸(nt)的BLAST搜索[36]和miRBase [35)数据库。在这两种情况下,BLAST输出都被解析,以确定匹配长度为18个或更少的匹配中没有错配的对齐,以及在较长的对齐中有一个错配的对齐。然后将注释(如果有的话)上传到数据库。此外,每个序列都与大豆基因组(大豆: Glyma1),使用Novoalign [34].对齐并没有限制允许的不匹配的数量,但最多的是4个,这表明映射质量对于任何更高的不匹配来说都太低了。
在此阶段,从数据库导出一个列分隔的文本文件,列出内部数据库ID、序列及其长度、所有唯一小rna的注释以及从每个库读取的数量。在此阶段还附加了该序列图的基因组位置列。在将序列映射到多个位置的情况下,只输入映射到小于10个区域的序列。与此同时,一个名为addModel.pl*的脚本创建了一个单独的文件,其中包含序列ID、基因组位置和锚定在这些位置的任何基因模型(Glyma模型)。使用Phytozome的GFF文件在映射位置检索预测的基因模型(如果有的话)。基因模型信息被反馈到数据库中,以便将来更容易检索。pl*在文件中添加了基因模型的注释信息。最后一个脚本teratogen.pl*合并了这两个文件:以列分隔的文件和基因组位置以及注释文件,使用序列ID生成AllSoyExpanded Excel电子表格。
所得Excel文件的内容用于所有后续smRNA数据比较。在Phytozome中使用BLAST将这些策划的smRNA序列与一个或多个基因组DNA序列进行比对[33]和领结[39].这些分析的结果与Excel表格工具和家庭脚本进行了进一步的描述、交叉比较和审查。在少数情况下,详细的对准执行MultAlin[40,57].
Novoalign
Novocraft开发的Novoalign [34]被用来鉴定大豆基因组中小RNA的起源。使用整个基因组作为参考,而不是较小的CDS序列,可以识别小RNA生成位点,而无需应用计算识别的基因和/或重复位置的稍微任意限制。以大豆基因组Glyma 1版本为参考。
小RNA数据集的接入数量
研究中使用的小RNA数据集(Illumina SBS)可通过Gene Expression Omnibus (GEO)数据库获得[58]:全籽W (12-14 DAF) (GSM899820);种皮,Richland, (GSM543393);种皮W55 (GSM543396);种皮W (GSM543394);W子叶(GSM543395);G. W子叶,7天苗,(GSM899821);茎,2周株,(GSM899822);叶,1个月植株,(GSM899823)。
附加文件
参考文献
- 1.
Kasschau KD, Fahlgren N, Chapman EJ, Sullivan CM, Cumbie JS, Givan SA, Carrington JC:拟南芥sirna的全基因组分析。公共科学图书馆,2007,5 (3):e57。10.1371 / journal.pbio.0050057。
- 2.
Nobuta K, Venu RC, Lu C, Belo A, Vemaraju K, Kulkarni K, Wang W, Pillay M, Green PJ, Wang G, Meyers BC:水稻mrna和小rna的表达图谱。中国生物工程学报,2007,25:473-477。10.1038 / nbt1291。
- 3.
Lister R, O 'Malley RC, Tonti-Filippini J, Gregory BD, Berry CC, Millar AH, Ecker JR:拟南芥表观基因组高集成单碱基分辨率图谱。细胞学报,2008,33(4):523-536。10.1016 / j.cell.2008.03.029。
- 4.
王X,我和我的朋友AA,李X,李N,彭Z, G,太阳H,气X,刘年代,邓XW:全基因组和表观遗传修饰瀑特异性景观及其关系在玉米mRNA和小RNA转录组。植物学报,2009,21:1053-1069。10.1105 / tpc.109.065714。
- 5.
Fahlgren N, Sullivan CM, Kasschau KD, Chapman EJ, Cumbie JS, Montgomery TA, Gilbert SD, Dasenko M, Givan SA, Carrington JC:基于高通量测序的小RNA分析计算框架。RNA。2009, 15: 992-1002。10.1261 / rna.1473809。
- 6.
Baulcombe D:植物中的RNA沉默。自然科学进展,2004,29(3):349 - 349。10.1038 / nature02874。
- 7.
植物转录后小RNA通路:机制和调控。基因开发,2006,20:759-771。10.1101 / gad.1410506。
- 8.
李文强,李志强:内源性小RNA通路的特化和进化。中国农业科学,2007,30(4):344 - 344。10.1038 / nrg2179。
- 9.
张欣:植物的表观遗传景观。科学通报,2008,32(4):489-492。10.1126 / science.1153996。
- 10.
谢z, Johansen LK, Gustafson AM, Kasschau KD, Lellis AD, Zilberman D, Jacobsen SE, Carrington JC:植物小RNA通路的遗传和功能多样化。公共科学学报,2004,2:642-652。
- 11.
Zilberman D, Cao X, Jacobsen SE: ARGONAUTE4对位点特异性siRNA积累和DNA和组蛋白甲基化的控制。科学通报,2003,29(4):344 - 344。10.1126 / science.1079695。
- 12.
刘娟,何勇,Amasino R,陈旭:拟南芥天然开花行为调控中以内含子转座子为靶点的siRNAs。基因工程学报,2004,18:2873-2878。10.1101 / gad.1217304。
- 13.
男仆H:植物arggonautes。植物科学进展,2008,13:350-358。10.1016 / j.tplants.2008.04.007。
- 14.
谢泽,谢志强,王晓明,王晓明:植物反式作用siRNA生物发生过程中microrna定向的相位调控。光子学报,2005,29(3):344 - 344。10.1016 / j.cell.2005.04.004。
- 15.
谢忠,杨晓明,王晓明,王晓明:拟南芥DICER-LIKE4基因在植物生长发育中的作用。《美国国家科学院学报》上。2005, 30(4): 344 - 344。10.1073 / pnas.0506426102。
- 16.
Yoshikawa M, Peragine A, Park MY, Poethig S:拟南芥反式作用sirna的生物发生途径。基因与发育,2005,19:2164-2175。10.1101 / gad.1352605。
- 17.
Borsani O,朱娟,Verslues PE, Sunkar R,朱景江:拟南芥耐盐性的内源性sirna调控。电子学报,2005,23(3):344 - 344。10.1016 / j.cell.2005.11.035。
- 18.
张晓东,张晓东,张晓东,张晓东,张晓东,等。植物免疫系统中siRNA的研究进展。《美国国家科学院学报》上。2006, 30(3): 374 - 374。10.1073 / pnas.0608258103。
- 19.
陈宏宏,陈lt, Patel K,李永华,Baulcombe C,吴双生:22-核苷酸rna触发植物siRNA次生生物发生。《美国国家科学院学报》上。2010, 27(4): 457 - 457。10.1073 / pnas.1001738107。
- 20.
王志刚,王志刚,王志刚:RNA定向RNA聚合酶的命名与功能。植物科学进展,2006,11:142-151。10.1016 / j.tplants.2006.01.003。
- 21.
刘强,冯勇,朱智:植物中的dicer样蛋白。中国生物医学工程学报,2009,29(3):379 - 379。10.1007 / s10142 - 009 - 0111 - 5。
- 22.
吴玲,张强,周华,倪峰,吴霞,祁勇:水稻microRNA效应复合物及靶标。植物学报,2009,21:3421-3435。10.1105 / tpc.109.070938。
- 23.
Curtin SJ, Stupar RM:植物中RNA沉默机制,Keystone研讨会。大豆dicer样同源重复体和串联重复体在生物和非生物胁迫下表现出转录亚功能化。2010年,69年。
- 24.
Tuteja JH, Zabala G, Varala K, Hudson M, Vodkin LO:内源性组织特异性短干扰rna沉默了甘氨酸种子皮查尔酮合成酶基因家族。植物科学学报,2009,29(3):344 - 344。10.1105 / tpc.109.069856。
- 25.
Nobuta K, Lu C, Shrivastava R, Pillay M, De Paoli E, Accerbi M, Arteaga M, Sidorenko L, Jeon DH, Yen Y, Green PJ, Chandler VL, Meyers BC:玉米内源sirna大小分布的差异:来自mop1-1突变体深度测序的证据。《美国国家科学院学报》上。2008, 30(4): 344 - 344。10.1073 / pnas.0808066105。
- 26.
陆超,Tej SS, Luo S, Handenschild CD, Meyers BC, Green PJ:转录组中小RNA成分的解析。科学通报,2005,29(4):457 - 457。10.1126 / science.1114112。
- 27.
Schnable PS等:B73玉米基因组:复杂性、多样性和动态。科学通报,2009,29(4):344 - 344。10.1126 / science.1178534。
- 28.
苏巴曼尼亚·S,付毅,孙卡尔·R,朱建科,于奥:大豆根系中新型和结节调控的microrna。中国生物医学工程学报,2008,29(3):344 - 344。10.1186 / 1471-2164-9-160。
- 29.
王艳,李鹏,曹霞,王霞,张安,李霞:大豆固氮根瘤中mirna的鉴定及表达分析。生物化学学报,2009,39(3):344 - 344。10.1016 / j.bbrc.2008.11.140。
- 30.
Joshi T, Yan Z, Libault M, Jeong DH, Park S, Green PJ, Sherrier DJ, Farmer A, May G, Meyers BC, Xu D, Stacey G: Glycine max新型miRNAs及其靶基因的预测。生物信息学杂志,2010,11 (S1): S14。
- 31.
宋庆霞,刘玉峰,胡晓霞,张伟科,马斌,陈淑云,张建军:大豆种子发育过程中mirna及其靶基因的深度测序鉴定。中国生物医学工程学报,2011,29(4):529。10.1186 / 1471-2229-11-5。
- 32.
Bentley DR:全基因组重测序。现代科技与发展,2006,16:545-552。10.1016 / j.gde.2006.10.009。
- 33.
Phytozome数据库。[http://www.phytozome.net/soybean].
- 34.
Novoalign。[http://www.novocraft.com].
- 35.
miRBase。[http://www.mirbase.org/].
- 36.
NCBI核苷酸(nt)。[http://www.ncbi.nlm.nih.gov/blast/Blast.cgi].
- 37.
Kulcheski FR, de Oliveira LF, Molina LG, Almerao MP, Rodrigues FA, Marcolino J, Barbosa JF, Stolf-Moreira R, Nepomuceno AL, Marcelino-Guimaraes FC, Abdelnoor RV, Nascimento LC, Carazzolle MF, Pereira GA, Margis R:涉及非生物和生物胁迫的新型大豆microrna的鉴定。中国生物医学工程学报,2011,29(3):357。10.1186 / 1471-2164-12-307。
- 38.
Fornara F, Coupland G:植物相变引起轰动。细胞学报,2009,38(3):344 - 344。10.1016 / j.cell.2009.08.011。
- 39.
Langmead B, Trapnell C, Pop M, Salzberg SL:人类基因组短DNA序列的超快和内存高效对齐。中国生物医学工程学报,2009,10:R25。10.1186 / gb - 2009 - 10 - 3 - r25。
- 40.
MultAlin。[http://bioinfo.genotoul.fr/multalin/multalin.html].
- 41.
许mutz J, Cannon SB, Schlueter J, Ma J, Mitros T,等:古多倍体大豆基因组序列分析。自然科学进展,2010,38(4):344 - 344。10.1038 / nature08670。
- 42.
转座元素数据库。[http://www.soytedb.org/].
- 43.
Rajagopalan R, Vaucheret H, Trejo J, Bartel DP:拟南芥microrna的多样性和进化流体集。基因与发育。2006,20:3407-3425。10.1101 / gad.1476406。
- 44.
Levesque MP, Vernoux T, Busch W, Cui H, Wang JY, Ikram Blilou I, Hassan H, Nakajima K, Matsumoto N, Lohmann JU, Scheres B, Benfey PN:拟南芥SHORT-ROOT发育通路的全基因组分析。公共科学图书馆,2006,4:0739-0752。
- 45.
张伟,高晟,周旭,夏娟,Chellappan P,周旭,张霞,金华:多个不同的小rna来源于相同的microRNA前体。中国生物医学工程学报,2010,30(4):344 - 344。10.1186 / gb - 2010 - 11 - 8 r81。
- 46.
Tripp HJ, Hewson I, Boyarsky S, Stuart JM, Zehr JP:在元转录组学研究中,rRNA的错误注释现在可以产生90%的假阳性蛋白匹配。NAR。2011年,10.1093 / nar / gkr576。
- 47.
Mosher RA, Melnyk CW, Kelly KA, Dunn RM, Studholme DJ, Baulcombe DC:拟南芥发育胚乳中poliv依赖性siRNAs的单亲本表达。自然科学学报,2009,29(3):344 - 344。10.1038 / nature08084。
- 48.
王晓明,王晓明,王晓明,等:种子发育过程中重复元件的去甲基化是基因印迹的基础。科学通报,2009,34(4):344 - 344。10.1126 / science.1171609。
- 49.
Ex FV, Jacob Y, Martienssen RA:小rna在植物繁殖中的多重作用。植物生物学,2011,14:1-6。10.1016 / j.pbi.2010.11.003。
- 50.
Marin E, Jouannet V, Herz A, Lokerse AS, Weijers D, Vaucheret H, Nussaume L, Crespi MD, Maizel A: miR390拟南芥TAS3 tasiRNAs及其生长素响应因子靶点定义了一个定量调节侧根生长的自动调节网络。中国生物医学工程学报,2010,30(4):344 - 344。10.1105 / tpc.109.072553。
- 51.
Montgomery TA, Howell MD, Cuperus JT, Li D, Hansen JE, Alexander AL, Chapman EJ, Fahlgren N, Allen E, Carrington JC: ARGONAUTE7-miR390相互作用的特异性和TAS3反式作用siRNA形成的双重功能。细胞科学,2008,33(4):344 - 344。10.1016 / j.cell.2008.02.033。
- 52.
一种从玉米组织中提取RNA的简单方法。中国农业科学,1986,16(3):344 - 344。
- 53.
王超,陶杰,吴晓明,吴晓明,等:显性I等位基因降低了黄豆种皮查尔酮合成酶的mRNA表达和活性。中国农业科学,2004,27(4):349 - 349。10.1104 / pp.105.2.739。
- 54.
Todd JJ, Vodkin LO:从查尔酮合成酶多基因家族中抑制表达的重复和恢复表达的缺失。植物科学学报,1996,8:687-699。
- 55.
邹娟,Rodriguez-Zas S, Aldea M, Li M, Zhu J, Gonzalez D, Vodkin LO, DeLucia E, Clough SJ:大豆对丁香假单胞菌反应的表达分析揭示了新的防御相关基因和光合作用的快速hrspecific下调。植物与微生物的相互作用,2005,18:1161-1174。10.1094 / mpmi - 18 - 1161。
- 56.
Hamilton AJ, Baulcombe DC:植物转录后基因沉默中的一种小反义RNA。科学通报,1999,29(4):457 - 457。10.1126 / science.286.5441.950。
- 57.
Corpet F:具有层次聚类的多序列比对。自然科学进展,1998,16(22):10881-10890。10.1093 / nar / 16.22.10881。
- 58.
基因表达集合,基因组库数据库。[http://www.ncbi.nlm.nih.gov/geo/].
- 59.
植物顺式作用调控DNA元件的数据库。[http://www.dna.affrc.go.jp/PLACE/signalscan.html].
致谢
我们非常感谢Illumina测序设备人员提供的小RNA测序专业知识。我们也感谢本科生助理Martin Blistrabas, Kevin Nee, Leenanshu Kapur, Achira Kulasekara和Cameron Lowe在数据分析方面的帮助。这项工作得到了UI关键研究倡议计划、联合大豆委员会、美国农业部和伊利诺斯州大豆协会的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
GZ分析数据并起草手稿。EC进行凝胶印迹实验。KKV、SB和HW对Illumina小RNA原始序列文件进行了处理,并对多个数据库进行了BLAST和BLAT搜索。JT、SJ、BC和SC制备组织和RNA样本进行测序。MH提供生物信息学专业知识。LOV获得资金,协调和领导研究,起草章节,并编辑手稿。所有作者都阅读并批准了最终的手稿。
电子辅助材料
Excel电子表格,包含135,055个独特的序列签名及其在本研究中检测的8个大豆组织群体(WS, SCR, SCM, SCW, Cot, GCot ST和LE)中的计数。
附加文件1:见表1用于组织描述和注释描述的材料和方法。这些文件包含总非规范化读计数。(xlsx14mb)
独特的小RNA序列的Excel文件-reads与本研究中检测的八个大豆组织群体(WS、SCR、SCM、SCW、Cot、GCot ST和LE)获得的注释mirna具有高度相似性。
附加文件2:见表1用于组织描述和注释描述的材料和方法。这些文件包含总非规范化读计数。(xlsx397 kb)
12870 _2012_1166_moesm4_esm.doc
附加文件4:在编码miR3522的Glyma15g06080基因启动子上发现的顺式作用调控基序,由植物顺式作用调控DNA元件数据库PLACE确定(http://www.dna.affrc.go.jp/PLACE/signalscan.html) [59].(doc 114kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用授权协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
扎巴拉,G.,坎波斯,E.,瓦拉拉,K.K.et al。种子和营养组织内源性小RNA群体的分化模式大豆.BMC植物生物学12,177(2012)。https://doi.org/10.1186/1471-2229-12-177
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-12-177
关键字
- 种皮
- microrna基因
- 植物组织
- 生长素反应因子
- 大豆基因组