摘要
背景
植物采用不同的繁殖策略来适应在不同气候下的生长。在拟南芥FRIGIDA(星期五)通过增加MADS盒转录抑制因子的表达,赋予了春化需求,从而使冬季一年生习性开花位点c(方法).变化在星期五起着重要作用答:芥生活史策略,作为独立的功能丧失等位基因,在不同的资源中导致了快速循环的习惯,似乎已经进化了很多次。本研究的目的是识别和描述的直系同源星期五在芸苔属植物oleracea.
结果
我们描述的特征星期五从芸苔属植物oleracea然后区分两者B.甘蓝orthologues (BolC.FRI.a而且BolC.FRI.b).这些表明了广泛的氨基酸保存在中央和c端区域的FRI从其他芸苔科,包括答:芥,但有一个发散的n端。基因映射到三个区域中的两个b . oleracea染色体同线到的部分答:芥第5号染色体表明星期五自从古代的三次复制事件形成了b . oleracea基因组。这一基因组位置与星期五在答:芥并通过比较分析,发现了一种重组事件A. thaliana启动子。这个搬迁A. thaliana到第4染色体,非常靠近核仁组织者区,留下一个片段星期五在同步位置上答:芥5号染色体。我们的数据显示,这种重排发生在从答:lyrata.我们探索了等位基因变异BolC.FRI.a在培养b . oleracea并鉴定出两个主要的等位基因,它们对彼此和A. thaliana,当表示为融合在答:芥.
结论
我们将两者区分开来甘蓝FRI基因,我们展示了其中之一答:芥互补实验是功能性的,并表明它们的基因组位置不同位A. thaliana由于一个古老的重组事件。这使得之前的关联分析变得复杂星期五随着生命历史的变化策略芸苔属植物属。
背景
向生殖发育的转变是植物生命周期中的一个基本过程。这一发育转变的分子机制已被广泛研究拟南芥.一个环境响应性遗传途径的综合网络聚集在一组共同的目标上,以定量调节将顶端分生组织从植物状态转换为花态所需的基因[1- - - - - -3.].一个重要的环境线索是持续的寒冷,它加速了开花,这一过程被称为春化,并使授粉和结实率与春季的有利条件保持一致。许多植物物种对春化的需求存在差异,这影响了生活史策略,与在第一个生长季节开花的夏季一年生植物相比,需要春化的植物采用多年生、两年生或冬季一年生的习惯。这与其他更依赖光周期信号或内源性线索的物种形成对比,例如水稻[4].开花时间变化的显著适应度结果,在年[5,6]和多年生植物[7],很可能对花期控制的广泛变异性的进化做出了贡献。开花还影响整个季节的生长模式,并影响许多农艺性状,包括作物生产的数量和质量。这在栽培芸苔属植物中尤其明显,在开花过程中的变化已被选择,以产生多种经济上重要的形态形式。
春化需求变化的主要决定因素答:芥等位基因变异在FRIGIDA(星期五) [8- - - - - -11].FRI通过促进花抑制因子的表达来抑制开花开花位点c(方法) [12,13].春化对FRI具有拮抗作用,并通过下调来加速开花方法.一些快速循环的变种答:芥那些不需要春化的植物被发现是由于其功能的丧失而产生的星期五,这一进化步骤已经发生过多次[8,9,11,14].在其他生物中也发现了通过等位基因变异在一个共同目标上的平行进化[15].因此,有趣的是,在其他植物物种中是否也发生了类似的进化步骤。许多其他物种的春化需求也有变化,这是许多主要作物的重要农艺性状。例如,在b . oleracea(园艺芸苔属)需要春化的两年生植物以卷心菜和球芽甘蓝为代表,夏季一年生作物包括一些甘蓝和花椰菜品种。Orthologues的星期五已在答:lyrata[16),危害物种(17和盐生植物Thellungiella halophila[18芸苔科内,更广泛地在紫花苜蓿,日本莲花,葡萄[19),杨树balsamifera[20.),栽培稻[21].到目前为止,春化需求的自然变化与星期五多态性在答:lyrata[8]和等位基因变异在一个同源芸苔属植物显著(BnaA.FRI.a)与开花时间的变化有关[22].
我们有兴趣了解园艺芸苔属植物开花时间和春化需求变化的分子基础。遗传信息答:芥一般可以应用于芸苔属物种,因为它们的进化亲缘关系。的拟南芥而且芸苔属植物属属于同一科(芸苔科)b . oleracea被认为是由一个祖先基因组的三倍相似的拟南芥[23- - - - - -26].植物开花控制的遗传信息拟南芥可以应用于芸苔属植物,因为拟南芥而且芸苔属植物基因组(23,27,28].这已被用于推断可能解释开花时间和其他变异的QTL的候选基因[22,29,30.];然而,在某些情况下,它可能会误导[31].在这里,我们确定了两者星期五基因b . oleracea并绘制它们的基因组位置。我们还研究了其中一个基因的变异星期五已培养的基因座b . oleracea种质。这些新数据将提供必要的信息,以阐明一种作用有多普遍星期五在芸苔科的生活史变异中起作用。
结果与讨论
两个星期五基因存在于芸苔属植物oleracea基因组
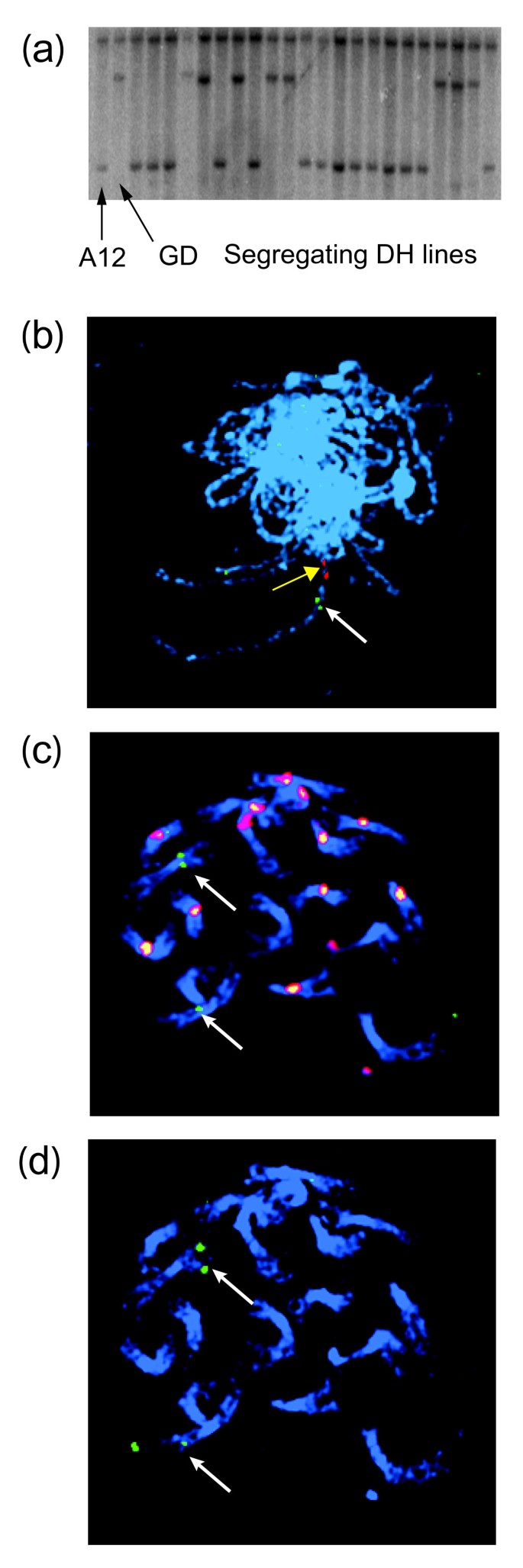
的BoFRI从该菌株JBo BAC文库中分离得到基因b . oleracea芥蓝基因型A12DHd [32通过与的杂交A. thaliana基因克隆。七个BAC阳性克隆中有两个表现出明显的差异星期五杂交模式(JBo72I23和JBo88G16,图1 a, b)进行亚克隆。这些分析证实他们携带不同的芸苔属植物paralogues指定BolC.FRI.a而且BolC.FRI.b[33]并在以后称为BoFRIa而且BoFRIb[GenBank JN191450和JN191449]。和其他物种一样,BoFRIa而且BoFRIb含有3个外显子,分别编码594和585残基的预测开放阅读框(orf)(图1 c).BoFRIa包含两个线圈结构域,通常参与蛋白质低聚(正如线圈预测的那样)http://www.ch.embnet.org/software/COILS_form.html[34]),与预测的结构非常相似答:芥农贸总厂(农贸总厂)[8,35].相比之下,BoFRIb被预测在c端区域只包含一个卷曲线圈域,因为发现在四个FRI中有两个是这样显著[22].
克隆BoFRIa而且BoFRIb.(一个) A12 BACs菌落杂交鉴定的Southern分析AtFRI.第1、4、5、8、11、12和13列的克隆体与FRIGIDA.第2、3、6、7和10车道的BACs不交叉杂交(b)的6个BACs的HindIII摘要从外显子2和3的保守区探测BoFRIa.Lane 5包含其中的JBo72I23BoFRIa测序。7巷包含JBo88G16。第1、2、3和6道包含另外4个BACs,表现出与JBo88G16相同的杂交模式。注意杂交的强度表明了BoFRIa和BoFRIb之间的序列分歧(见1c) (cBoFRIa和BoFRIb蛋白序列与FRI亚家族其他成员的比较。从上到下都是芸苔属植物oleraceaBoFRIa,芸苔属植物oleraceaBoFRIb,Thelliungiella halophilaThFRI,拟南芥芥AtFRI拟南芥lyratassplyrataAllFRI和拟南芥lyratassp佩特拉-AlpFRI。n端结构域包含37个氨基酸的保守区(用实心条表示),定义了frii超家族其他成员的FRIGIDA副本[17].卷曲线圈域由红线表示。
AtFRI是7种蛋白质家族的原始成员答:芥除了两个预测的线圈结构域外,它与任何其他蛋白质都没有同源性,其功能尚未确定。FRI蛋白家族的最新分析[19]发现了一个保守的核心中心区域。在这个域之外,观察到显著的变化,使FRI家族被细分为五个不同的组。AtFRI及其在其他物种中的同源物由该蛋白n端37个氨基酸组成的保守区定义。我们在这里描述的BoFRI蛋白包含这个保守的37个氨基酸区域,这加强了它们是FRI同源物的观点;然而,该区域两侧的氨基酸与AtFRI的同源性较低(图1 c).这一区域包括了BoFRIa中第一个预测的卷曲线圈的大部分。BoFRIb中该结构域的变异导致预测的卷曲线圈的丢失,强调了该区域氨基酸多态性的可能功能意义。从盐生植物中分离出的FRI同源物的n端区域也发现了与AtFRI相似程度的分化t . halophila和四种FRI的同源体显著[18,22].相比之下,BoFRIa、BoFRIb和AtFRI之间在中央和c端区域有广泛的氨基酸守恒(图1 c).植物AtFRI功能域的转基因分析答:芥N或C端被删除表明N端区域对功能的重要性降低[19],也许可以解释观察到的高度分歧。
的BoFRI基因映射到与非同程的区域A. thaliana
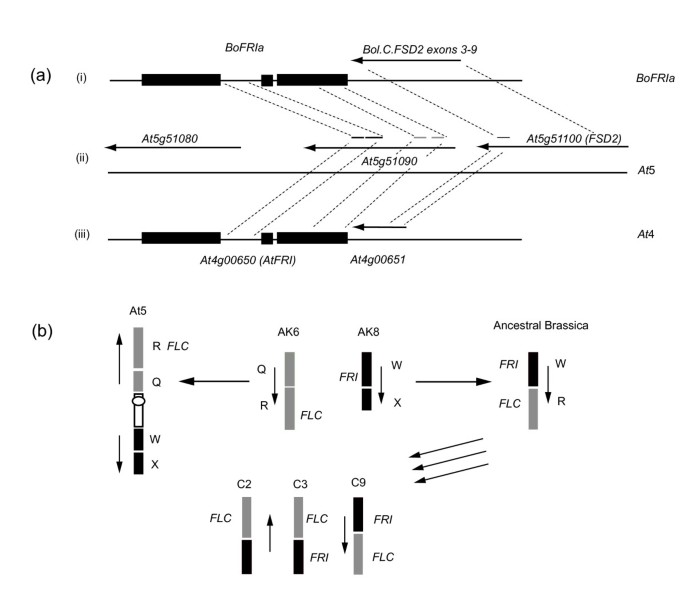
包括外显子2、内含子2和外显子3的基因组片段BoFRIa(并显示出高度的保护BoFRIb而且AtFRI)杂交到两个映射滤波器b . oleracea定位群体:芥蓝×甘蓝(var。alboglabrax var。italica;A12DHdxGDDH33, (36];数字2)和花椰菜×抱子甘蓝(var。葡萄孢属x var。gemmifera;N × g [37])。两者之一的rflpBoFRI在A12DHdxGDDH33定位群体中分离出的一个位点,使该位点可以定位到该基因的C3连锁群的39.5 cM上b . oleracea基因组。C3位点定位为BoFRIa通过荧光原位杂交(FISH)与BAC JBo72I23进行杂交,从中得到BoFRIa最初的序列(图2 b).经FISH定位,JBo88G16位于C9染色体的短臂(图2 c, d).因此,第二个轨迹,BoFRIb,在链接组C9上。另外两个BACs显示了与JBo88G16相同的限制模式(图1 b)杂交到C9上的相同位置(数据未显示)。这些结果证实了b . oleracea基因组包含两个同源体。
比较分析芸苔属植物而且答:芥基因组研究表明,C3和C9的染色体区域与这两种基因相关联BoFRI已映射的位点均同向于某区域答:芥5号染色体,而不是在上面答:芥染色体4,其中AtFRI(At4g00650)位于[23].第5号染色体的这个区域包括一些已知参与控制开花的基因,包括方法,财政年度而且君士坦斯(有限公司).一些QTL研究发现,开花时间变化的位点映射到这个基因组区域在许多芸苔属植物人群包括b . oleracea[38- - - - - -41),b·拉伯[42- - - - - -46];b .黑质[47,48),显著[29,49].我们绘制的地图显示了BoFRI不仅是BoFLC,但也BoFY而且杯蟹;之前绘制的其他开花时间基因。的序列BoFRIa而且BoFRIb进一步让我们确定四个正交体中的哪一个AtFRI最近在显著[22]是两个C基因组副本。四份星期五被指定的BnaA.FRI.a,BnaX。星期五b-d.与BoFRIa和BoFRIb的氨基酸序列比较表明,BnaX.FRI.d与BoFRIa同源,与BnaA.FRI.a C基因组同源。这一结论进一步得到事实的支持BnaA.FRI.a被映射到A3的一个区域,与我们所映射的C3的区域同源BoFRIa.经氨基酸序列比较,BnaX.FRI.c与BoFRIb完全相同。BnaX.FRI.c似乎与BnaX.FRI.b最相似,因此很可能是BoFRIb的A基因组同源物显著.
对象特定的重组事件答:芥天堂已搬迁至FRIGIDA4号染色体顶部的基因
在答:芥,AtFRI该位点位于第4染色体的顶部。然而,此前有报道称星期五在答:lyrata映射到链接组8 [50,51].这个连杆群与的下臂是同源的答:芥第5号染色体[50,52,53].有趣的是,在这个区域有一个注释的基因模型答:芥第5染色体(At5g51090)显示出高度的同源AtFRI,包含内含子1和外显子3的部分,但缺乏编码区其他部分,因此可能是假基因[50].基因研究者的数据表明At5g51090表达水平非常低,支持了这一假设[54].下游,方向相反,是At5g51100,编码铁超氧化物歧化酶和BoFRIaBAC克隆含有3'序列,与其中的3-9外显子同源答:芥第5染色体基因(图3).
BoFRIa而且BoFRIb的区域地图b . oleracea与拟南芥非共线基因组.(一个)比较BoFRIa的注释区域的基因组克隆答:芥染色体4和5以5'到3'的方向排列。纯黑色矩形对应三个外显子组成星期五.箭头表示其他基因的方向。(我)BoFRIa,基因组克隆。(ii)区域答:芥第5号染色体含有铁超氧化物歧化酶(FSD2, At5g51100),At51090显示同源性星期五内含子1和外显子3。(iii)区域答:芥含有4号染色体的AtFRI而且At4g400651与外显子9同源FSD2。(b)建议推导答:芥而且b . oleracea祖先核型(AK6和AK8)的连锁群,如[53显示…位置的星期五而且方法.祖先块W和R的重排导致的正交星期五而且方法在芸苔属祖先的基因组中被拉近了距离。该区域存在于染色体C2, C3和C9上b . oleracea并位于各地区b . oleracea显示同向的基因组答:芥5号染色体。BoFRIa位于C3和BoFRIb在制备过程。副本星期五在C2上丢失了。
由于其在基因鉴定和标记开发方面的潜力,共时性在芸苔科基因组中得到了广泛的研究。拟南芥而且芸苔属植物被认为在大约43 Mya与祖先基因组的三倍(类似于拟南芥)大约在2300万年前发生,并产生了现代二倍体芸苔属基因组[55].答:芥而且答:lyrata被认为与祖先n = 8的核型(如在答:lyrata)的派生状态答:芥n = 5 [24,55,56].芸苔科的祖先核型被认为是由24个保守染色体块组成的8条染色体[57].这些基因块可以被重新排列来模拟基因的基因组结构A. thaliana, A. lyrata还有现代的二倍体芸苔属植物[24].因此,9条染色体(C1-C9)的基因组组成b . oleracea和10条染色体(A1-A10)b·拉伯可以与祖先的核型和答:芥基因组。
祖先基因组分别从祖先核型的第6和第8染色体上阻断QR和WX,今天由答:lyrata[24],都在祖传中重新组合芸苔属植物基因组之前的三倍,导致块WR被代表三次b·拉伯A2, A3和A10的基因组[25,26].的平行区域b . oleracea在C2, C3和C9(图3 b).这种重新排列带来了方法(R区)和星期五(W区)在这些染色体上。因此在答:lyrata而且B.甘蓝,FRI地图非常接近VIN3(也在W区,用于春化[58],也是它的主要目标方法(块R;数字3 b).BoFRIa映射到C3和BoFRIb到C9因此代表三个共向区域中的两个。的第三段星期五似乎是从C2丢失的b . oleracea进化;这种基因损失并不少见[59].这与目前的位置形成对比星期五在染色体4的顶端答:芥显示与祖先核型的6号染色体上的O区同源。
我们在这里提出的数据表明,染色体重排发生在祖先的十字花科基因组进化成答:芥包括一个重组/重排事件,该事件重新定位包含AtFRI到第4染色体短臂远端附近的一个位置,靠近核仁组织区,在第5染色体的基因组位置上留下一个与星期五在其他芸苔科(龙尾草,马齿苋;[25,52])。
存在两个共同的等位基因BoFRIa在不同的基因型b . oleracea
原始序列BoFRI一个和BoFRIb的亲本之一A12DHd。38,39].这些研究在我们绘制的区域绘制了C3上开花时间的QTLBoFRIa.因此,我们对其外显子1处650bp进行了测序BoFRIa这个群体的另一个亲本GDDH33(数据未显示)。与A12DHd相比,GDDH33序列有两个氨基酸取代(A118V和Q125E)。因此这个群体的父母携带了不同的等位基因BoFRIa有可能在BoFRIa导致了这个种群开花时间的变化。的等位基因中也发现了单氨基酸的取代BnaA.FRI.a从Tapidor × Ningyou7 (TN)亲本测居群中获得显著并将其映射到开花时间变化的QTL基础区域[22].
我们测序BoFRIa而且BoFRIb的另外两种基因型B.甘蓝.A12DHd参考序列来源于芥蓝的BAC克隆,甘蓝,八周后开花[38],可认为是快速循环类型,不需要春化。因此,我们选择了另外两种西兰花基因型(甘蓝);E1具有兼性春化反应,在寒冷期后提早开花,但在种植当年的10月/ 11月(秋季)成熟;E8具有专性春化要求,于次年4月/ 5月(春季)成熟。BoFRIb在三个基因型之间高度保守,仅发生5个氨基酸取代(E1中的D6G、K20Q、Q372K和R532W, E8中的N105T)。排序BoFRIa在这些基因型中发现了外显子1的多态性区域,其中包括E8相对于E1的7个氨基酸和3个氨基酸的两次缺失,即定义FRI蛋白的37个氨基酸保守块的任何一侧1 c).13个非同义替换和12个同义替换将A12DHd和E1 BoFRIa等位基因与E8等位基因区分开来。因此,我们指定了E1和E8BoFRIa等位基因的BoFRIa-1而且BoFRIa-4分别为[GenBank JN191393, JN191392]。
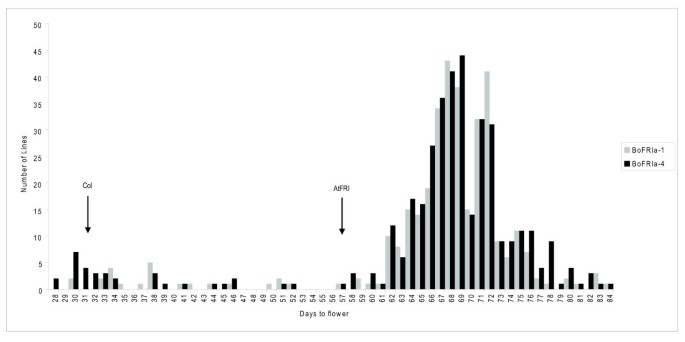
我们将后续的分析重点放在BoFRIa由于这显示了大多数的多态性,并扩展了我们的分析,包括55个来自栽培的基因型b . oleracea由华威大学(BolDFS, King等人)开发的多样性基金会集。http://www.brassica.info/resource/plants/diversity_sets.php;[60)表1).一个650 bp的区域BoFRIa覆盖包含这两种缺失的外显子1多态区进行测序(表1, GenBank JN191394-191448)。我们确定了6个BoFRIa来自BolDFS的55个基因型子集中的等位基因。这些可以分为两组;BoFRIa 1 - 3而且BoFRIa 4 - 6其中第二组的等位基因包括7个氨基酸和3个氨基酸缺失。的BoFRIa-1而且BoFRIa-4在研究的55种基因型中,等位基因是最常见的。此外,在两个删除确定BoFRIa-4总同时发生,并被发现在高频率与少量非同义核苷酸多态性。在花期晚的花椰菜中发现的这两种缺失在b . oleracea蔬菜类型,如抱子甘蓝和甘蓝,冬季一年生或两年生的习惯,通常是为了食用它们的营养形式而不是花的形式(表1,图4).有趣的是BnaX.FRI.d从显著冬季品种快线[22],我们已经在这里确定为C基因组同源BoFRIa在显著这两处删除是否都在BoFRI 4 - 6在具有冬季一年生或两年生习性的芸苔属蔬菜类型中过度代表的等位基因类。经进一步研究发现,BnaX.FRI.d与BoFRIa-5具有相同的氨基酸序列,在TN定位群体的欧洲冬季型和中国半冬季型亲本系中也存在[22].
功能分析BoFRIa等位基因的答:芥
以确定这两种最常见的BoFRIa等位基因赋予任何功能差异,我们进行转化实验。的编码和3'UTR序列BoFRIa-1而且BoFRIa-4等位基因被用来取代AtFRI编码和3' UTR序列答:芥基因克隆。通过保留5'区域的共同调控序列AtFRI我们希望使基因表达正常化,从而关注两者之间的结构差异芸苔属植物蛋白质。这些结构被转化为快速循环答:芥哥伦比亚(Col-0)。Col-0内部携带功能丧失突变AtFRI,但具有功能性方法所以这些实验将决定是否BoFRIa可以补充星期五导致Col-0突变,导致花期晚。这两个BoFRIa等位基因补充功能丧失突变与> 100初级(T1)转化植物包含每一个BoFRIa与Col-0型植物相比,等位基因开花很晚,令人惊讶的是,也比Col-0型植物晚AtFRI(图5).
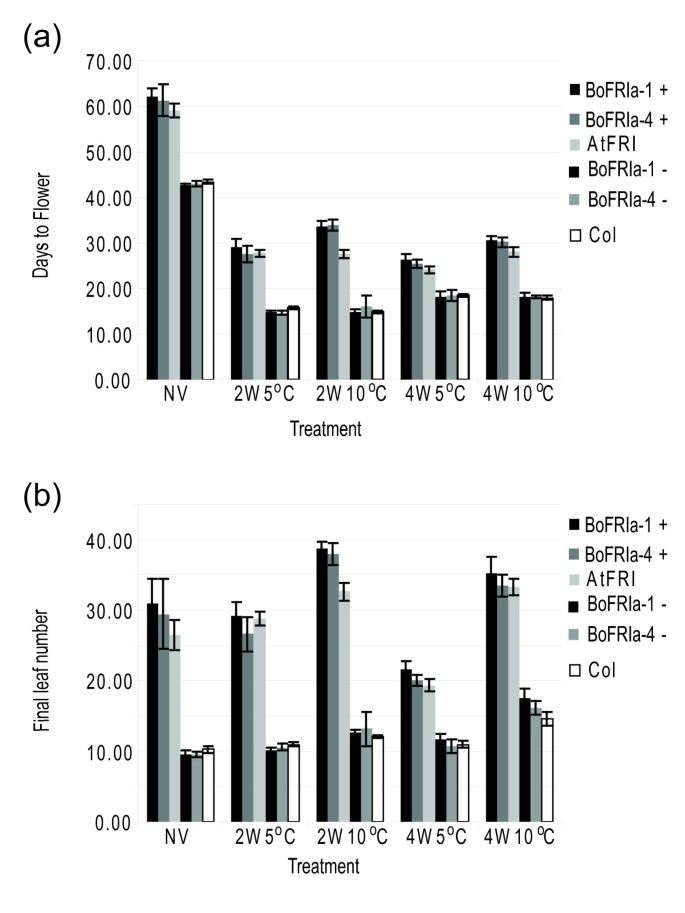
研究…的功能BoFRIa-1而且BoFRIa-4在不同环境条件下,对每个等位基因的5个转化子进行下一代(T2)分析。在5°C或10°C下,对未春化或春化2周或4周的植物的开花时间和总叶片数进行分析(图6).在所有处理中,除了在10°C (2W10°C)下的两周外,种植任何一种BoFRIa等位基因开花的时间和携带等位基因的时间一样晚AtFRI.在2W10°C时,植物携带任何一种BoFRIa等位基因开花时间晚于AtFRI.数字6还表明,与5°C相比,在10°C进行春化处理的植物继续生长并以更快的速度长出叶子。因此,当考虑总叶数作为开花时间的衡量标准时,似乎只有4W5°C是有效的春化处理(图6).
两种编码序列的表达式BoFRIa等位基因在AtFRI5’调控序列表明,这两个等位基因都能产生功能相当的蛋白质,在某些环境条件下,它们对花期的影响甚至比内源的更强答:芥蛋白质(图6).相比之下,两个lyrata FRI通过关联研究和转基因研究,等位基因在开花时间上存在数量差异[16].两者的维护答:lyrata在自然种群中,中间频率的等位基因表明它们在不同的环境中有不同的选择。如果BoFRIa等位基因确实是开花时间QTL的基础,那么两个基因之间一定存在表达差异,以解释开花时间的差异。这两种基因都可能以一种非常不同的模式表达AtFRI由于重排将其移至4号染色体导致转录起始位点上游不到1 kb的完全不同的5'序列,并将其置于非常不同的染色质环境中,因为它现在位于异色核仁组织区NOR4下游200-300 kb [61].
结论
的知识B.甘蓝基因数量,功能和图谱位置现在使我们处于一个强有力的地位,以进行广泛的研究,对等位基因变异的贡献星期五开花、春化和生活史行为。在生活史上的差异答:lyrata而且答:芥例如异交和自交,多年生和一年生的习惯可能会导致对某种程度的要求星期五功能答:lyrata这是可选的答:芥[16].b . oleracea,就像答:lyrata这是一个很大程度上的异种和一些野生物种b . oleracea,被认为是现代作物植物的祖先,据报道花期长达20年[62].我们的分析BoFRIa说明只有少量功能BoFRIa等位基因在培育的品种中被捕获b . oleracea种质。到目前为止,我们还没有发现证据表明功能丧失突变是在AtFRI.的5'和3'调控区进一步分析BoFRI正在进行中。接近BoFRI来BoFLC, BoFY而且杯蟹提出了这如何影响开花行为的新问题。在育种计划中选择特定的等位基因,使我们能够增强对日益增加的气候变化的稳健性,这将是特别重要的。
方法
克隆BoFRI基因
JBo BAC库与AtFRI基因组克隆,原产于斯德哥尔摩[8]和七种BACs,其中六种具有相同的限制模式,一种不同。从这些BACs中的两个(72I23和88G16)制备纯化的基因组DNA (Qiagen Maxi Prep Kit),并用于在pCR中生成1-2 kb片段的shotgun文库(TOPOr shotgun Kit)R4 blunt-topoR矢量,6倍的覆盖范围。这些文库中的菌落被网格化到尼龙膜(HyBond-N+)上,并杂交到由AtFRI(5'区域,外显子1,外显子3和3' UTR的3'端)。这些探针分别鉴定了BAC亚克隆。序列分析证实两种BACs携带差异芸苔属植物paralogues。
映射BoFRI位点在b . oleracea映射的数量
基因映射
生成A12DHdxGDDH33映射群体的映射滤波器,并与保守分子杂交BoFRI探针如[36,63].外显子2和3的900 bp保守区BoFRIb使用引物J2NG_F1 (5' AAGTATCAAGCGTGGAAAGCA 3')和J2NG_R1 (5' GTTACGAGGAGACCTGTGATT 3')从A12DHd基因组DNA中扩增,并用于探测A12DHdxGDDH33和NxG映射过滤器(由Graham Teakle, WHRI提供)。联动分析图BoFRIa使用Joinmap 3.0 [64]与BrassicaDB提供的地图数据http://brassica.bbsrc.ac.uk/BrassicaDB/.
荧光原位杂交(FISH)
对A12DHd基因型的染色体进行FISH扩增b . oleracea使用[65].染色体现在根据它们相应的连锁基团命名。JBo72I23与JBo62M08一起应用于减数分裂粗线系,JBo62M08的BAC与42 cM处C3上的RFLP标记pN22相关,先前被FISH分配到C3染色体上。JBo88G16 (BoFRIb)与与六对染色体周围异染色质杂交的BAC BoB061G14和来自克隆pTa71的45S rDNA探针一起应用于有丝分裂中期扩散[66), EMBLX07841。JBo88G16与之杂交的染色体缺乏来自其他探针的信号,其形态与C9相似。因此,用JBo32J18重新检测玻片,这是一种与BoFLC1该区域位于C9上pN47E4NM (87 cM)和pN3E1 (103 cM)之间[31,67并被FISH确认在C9上(未发表)。另外两个与JBo88G16显示相同限制模式的BACs与JBo88G16分别应用于粗线传播。
测序BoFRIa在BolDFS中
的b . oleracea多样性基础集(diversity foundation set, BolDFS)是代表该物种形态多样性作物遗传变异的核心系集合http://www.brassica.info/resource/plants/diversity_sets.php.DNA使用DNeasy 96 Plant Kit (Qiagen)进行分离,并使用GenomiPhi全基因组扩增试剂盒(GE healthcare)进行扩增。一个650 bp的片段BoFRIa使用AMPLITAQ GOLD TAQ DNA聚合酶(Life Technologies Ltd (Invitrogen Division)),用引物YWFRI_F (5'CGCACATCGTCCATCAACAAG 3')和FRIJ1_R2 (5'ATCCTTCACCCACCAGCCT 3') PCR扩增出55种基因型BolDFS的基因组DNA。使用AlignX在Vector NTI (Invitrogen)中进行序列分析。
功能分析BoFRIa等位基因
质粒pFRIg(在pbluscript - ks +中,Stratagene)被诱变以引入aBamHI位点即刻5′的ATG(质粒pFRIg-B)。pfrr - b的消化Bam你好,加班我允许移除AtFRI编码序列,留下5'区域AtFRI.一个4.3 kb的片段包含BoFRIa引物为BoFRI1_Bam_ATG (5'CTTCCGCGGATCCCATGGCCGTCCGTAAC3')和BoFRI1_R2_ClaI (5'CAGAGATCGATCTCGAGAAAGGTAGCTGTTT 3'),分别从菌株E1和E8的基因组DNA中分离得到PfuUltra融合HS DNA聚合酶(安捷伦科技)并测序。PCR产物用Bam你好,加班我和提纯的碎片结扎成Bam你好,加班我对pFRIg-B进行了消化,得到了包含of的5'UTR的最终结构AtFRI的编码和3' UTR序列BoFRIa-1(在BoFRIa-1中)或BoFRIa-4(在BoFRIa-4)。pfri - b被用作A. thaliana控制。最后的结构被连接到二进制向量pSLJ755I6(来自乔纳森·琼斯教授的礼物,http://www.tsl.ac.uk/research/jonathan-jones/plasmids.htm),在生态国际扶轮+XhoIfragment (pffr - b)或生态国际扶轮+班I片段(来自BoFRIa-1和BoFRIa-4)。构念被转移到农杆菌属通过三亲本交配[68]并转化为答:芥通过花浸法获得Col-0(由[69])。通过对Basta基因的筛选分离得到T1转化子™阻力。收集T2种子,花期以开花天数确定,不包括春化处理时间和开花时终叶数。
植物的生长
T2答:芥种子播种在“拟南芥混合物”(Scotts®Levington F2 8.75 l袋子,100 l沙砾,200 g Imidasect®5克),在塑料盆(7厘米× 7厘米)中,在5°C、8小时光周期和恒定湿度的春化室中分层,放置3天。花盆在2010年5月被转移到一个自然光照的长日温室7天,以允许发芽和预生长。没有接受春化处理的幼苗留在温室里;待春化处理的幼苗转移回春化室或受控环境柜(Snijder Economic Deluxe),在5°C或10°C下处理2周或4周。春化后,每株系20株移栽到有40个2 cm × 2 cm细胞的托盘中,放回温室。托盘定期移动到随机位置,以防止任何位置对植物生长的影响。花期记录为总叶数(开花时莲座叶加茎叶)或抽苔时间;以花序茎高3 cm时离花期天数为抽苔时间。
缩写
- QTL:
-
数量性状位点
- BAC:
-
细菌人工染色体
- 子:
-
开式阅读架
- 鱼:
-
荧光原位杂交。
参考文献
- 1.
辛普森GG,迪恩C:开花-拟南芥,开花时间的罗塞塔石碑?科学通报,2002,29(6):528 - 528。10.1126 / science.296.5566.285。
- 2.
Baurle I, Dean C:植物发育转变的时间。细胞学报,2006,25(4):655-664。10.1016 / j.cell.2006.05.005。
- 3.
Amasino R:开花季节和发育时间。植物学报,2010,29(3):344 - 344。10.1111 / j.1365 - 313 x.2010.04148.x。
- 4.
叶山,杨秀珍,田木生,王志刚,王志刚:水稻光周期调控途径对短日开花的影响。自然科学,2003,29(3):344 - 344。10.1038 / nature01549。
- 5.
刘志刚,王志刚,王志刚,等。植物开花时间对气候变化的响应。国立科学院(美国)。2007, 30(4): 344 - 344。10.1073 / pnas.0608379104。
- 6.
Wilczek AM, Roe JL, Knapp MC, Cooper MD, Lopez-Gallego C, Martin LJ, Muir CD, Sim S, Walker A, Anderson J, Egan JF, Moyers BT, Petipas R, Giakountis A, Charbit E, Coupland G, Welch SM, Schmitt J:遗传扰动对季节生活史可塑性的影响。科学通报,2009,34(3):344 - 344。10.1126 / science.1165826。
- 7.
杨春华,杨春华,杨春华,杨春华,杨春华。高寒和低海拔柽柳种群开花时间和花期的选择拟南芥lyrata.中国生物工程学报,2007,20:558-567。10.1111 / j.1420-9101.2006.01260.x。
- 8.
李志刚,李志刚,李志刚,李志刚,李志刚FRIGIDA这是拟南芥开花时间自然变异的主要决定因素。科学通报,2000,29(4):344-347。10.1126 / science.290.5490.344。
- 9.
加扎尼S, Gendall AR, Lister C, Dean C:拟南芥开花时间变异的分子基础分析。植物科学学报,2003,27(3):357 - 357。10.1104 / pp.103.021212。
- 10.
兰佩J,巴拉苏bramanian S, Sureshkumar S, Singh A, Schmid M, Weigel D:野生植物开花响应的多样性拟南芥菌株。PLoS Genetics, 2005, 1 (1): e6-10.1371/journal.pgen.0010006。
- 11.
Shindo C, Aranzana MJ, Lister C, Baxter C, Nicholls C, Nordborg M, Dean C:角色FRIGIDA而且开花位点c在确定拟南芥开花时间变异方面。中国生物医学工程学报,2005,29(3):344 - 344。10.1104 / pp.105.061309。
- 12.
谢尔登·CC,罗思·DT,芬尼根·EJ,孔雀·WJ,丹尼斯·ES:春化的分子基础:春化的核心作用开花位点c(方法).国立科学院(美国)。2000, 37(3): 357 - 357。10.1073 / pnas.060023597。
- 13.
Michaels SD, Amasino RM:损失开花位点c活动消除了晚开花表型FRIGIDA和自主途径突变,但不响应春化。植物学报,2001,13:935-941。
- 14.
Le Corre V, Roux F, Reboud X: DNA多态性在FRIGIDA基因拟南芥:广泛的非同义变异与开花时间的局部选择一致。中华生物医学杂志,2002,19:1261-1271。10.1093 / oxfordjournals.molbev.a004187。
- 15.
colsimo PF, Hosemann KE, Balabhadra S, Villarreal G, Dickson M, Grimwood J, Schmutz J, Myres RM, Schluter D, Kingsley DM:通过反复固定异质蛋白等位基因,刺鱼广泛平行进化。科学通报,2005,30(4):344 - 344。10.1126 / science.1107239。
- 16.
Kuittinen H, Niittyvuopio A, Rinne P, Savolainen O:自然变异拟南芥lyrata春化要求授予FRIGIDAindel多态性。分子生物学杂志,2008,25:319-329。10.1093 / molbev / msm257。
- 17.
黄红,黄红,马尾虫,马尾虫A:多倍体物种形成并没有立即产生生殖隔离危害(十字花科).中华生物医学杂志,2008,25:1472-1481。10.1093 / molbev / msn092。
- 18.
方强,刘娟,徐震,宋锐:植物开花时间基因的克隆与鉴定Thellungiella halophila.生物学报,2008,40:747-753。
- 19.
Risk JM, Laurie RE, Macknight RC, Day CL: FRIGIDA及相关蛋白具有保守的中心结构域和家族特异性的N-和C-末端区域,这些区域具有重要的功能。中国生物医学工程学报,2010,29(3):344 - 344。10.1007 / s11103 - 010 - 9635 - 2。
- 20.
Keller SR, Levsen N, Ingvarsson PK, Olson MS, Tiffin P:基于纬度梯度的局部选择对苦瓜杨树核苷酸多样性的影响,杨树balsamifera中国生物医学工程学报,2011,29(3):344 - 344。10.1534 / genetics.111.128041。
- 21.
栽培稻l . ssp粳稻).科学通报,2002,29(3):344 - 344。10.1126 / science.1068275。
- 22.
王娜,钱伟,Suppanz I,李伟,毛波,龙勇,孟J, Müller AE,荣昌:甘蓝型油菜花期变异与FRIGIDA同源基因bnaa . fra .a.等位基因变异的关系。应用物理学报,2011,32(3):344 - 344。10.1093 / jxb / err249。
- 23.
Parkin IAP, Gulden SM, Sharpe AG, Lukens L, Trick M, Osborn TC, Lydiate DJ:部分结构芸苔属植物显著基于基因组的比较分析拟南芥.中国生物医学工程学报,2005,27(3):344 - 344。10.1534 / genetics.105.042093。
- 24.
Schranz ME, Lysak MA, Mitchell-Olds T:比较基因组学的基础知识十字花科:十字花科植物基因组的组成部分。植物科学进展,2006,11:535-542。10.1016 / j.tplants.2006.09.002。
- 25.
崔M,权世杰,崔sr, Fraser F, Soumpourou E, Drou N,王忠,李世勇,杨铁军,文俊华:基于片段重排的基因组进化复杂性研究芸苔属植物拉伯序列级分析揭示。中国生物医学工程学报,2009,10:539-10.1186/1471-2164- 10539。
- 26.
Mun JH, Kwon SJ, Kim JA, Jin M, Kim JS, Lim MH, Lee SI, Hong JK, Park TH, Lee SC, Kim BJ, Seo MS, Baek S, Lee MJ, Shin JY, Hahn JH, Hwang YJ, Lim KB, Park JY, Lee J, Yang TJ, Yu HJ, Choi IY, Choi BS, Choi SR, Ramchairy N, Lim YP, Fraser F, Drou N, Soumpourou E,等芸苔属植物拉伯染色体A3。中国生物医学工程学报,2010,11:R94-10.1186/gb-2010-11-9-r94。
- 27.
Suwabe K, Tsukazaki H, Iketani H, Hatakeyama K, Kondo M, Fujimura M, Nunome T, fuoka H, Hirai M, Matsumoto S:基于简单序列重复的比较基因组学芸苔属植物拉伯而且拟南芥:棍棒病抗性的遗传起源。中国生物医学工程学报,2006,29(3):344 - 344。10.1534 / genetics.104.038968。
- 28.
李高米,G,杨B,邱D,萨利米,奎洛斯C:高密度芸苔属植物oleracea连杆图:识别有用的新连杆。应用理论与实践,2007,29(1):1 - 7。10.1007 / s00122 - 007 - 0568 - 3。
- 29.
龙艳,石娟,邱东,李瑞,张超,王娟,侯娟,赵娟,石玲,朴斌:芸苔属油籽多环境开花时间数量性状位点分析及与拟南芥的全基因组比对。遗传学报,2007,37(3):349。
- 30.
Smooker AM, Wells R, Morgan C, Beaudoin F, Cho K, Fraser F, Bancroft I:水稻脂肪酸去饱和途径候选基因和QTL的鉴定和定位芸苔属植物显著.应用物理学报,2011,29(3):344 - 344。10.1007 / s00122 - 010 - 1512 - 5。
- 31.
李志刚,李志刚,李志刚,李志刚:植物开花时间QTL与FLC序列多态性有关芸苔属植物oleracea?。理论与实践,2008,29(1):1 - 4。10.1007 / s00122 - 007 - 0657 - 3。
- 32.
O'Neill CM, Bancroft I:基因组片段的比较物理映射芸苔属植物oleraceavar。alboglabra与第4和5号染色体的序列区域同源拟南芥.植物学报,2000,29(3):344 - 344。10.1046 / j.1365 - 313 x.2000.00781.x。
- 33.
Østergaard L, King GK:芸苔属的标准化基因命名法。植物学报,2008,4:10-13。10.1186 / 1746-4811-4-10。
- 34.
李志强,李志强,李志强。基于蛋白质序列的螺旋结构预测。科学通报,2001,29(3):344 - 344。10.1126 / science.252.5009.1162。
- 35.
Michaels SD, Bezerra IC, Amasino RM:FRIGIDA拟南芥冬生习性需要相关基因。美国国家科学研究院。2004, 21(3): 388 - 388。10.1073 / pnas.0306778101。
- 36.
Bohuon EJR, Keith DJ, Parkin IAP, Sharpe AG, Lydiate DJ:保守C基因组的排列芸苔属植物oleracea而且芸苔属植物显著.应用理论与实践,1996,29(3):344 - 344。10.1007 / BF00224083。
- 37.
杨晓明,王志军,王志军:水稻数量性状基因座的研究进展芸苔属植物oleracea[j] .理论与实践,2002,(4):457 - 457。10.1007 / s001220100743。
- 38.
Bohuon EJR, Ramsay LD, Craft JA, Arthur AE, Marshall DF, Lydiate DJ, Kearsey MJ:开花时间数量性状位点与重复区域和候选位点的关系芸苔属植物oleracea.遗传学杂志,1998,15:393-401。
- 39.
Rae AM, Howell EC, Kearsey MJ:植物开花时间的QTL分析芸苔属植物oleracea.遗传与遗传学报,2003,23(4):344 - 344。10.1038 / sj.hdy.6886050。
- 40.
冈崎K,坂本K,菊池R,斋藤A, Togashi E, Kuginuki Y, Matsumoto S, Hirai M:映射和表征方法开花时间的同源性及QTL分析芸苔属植物oleracea.应用理论与实践,2007,29(1):1 - 5。10.1007 / s00122 - 006 - 0460 - 6。
- 41.
Uptmoor R, Schrag T, Stützel H, Esch E:基于作物模型的跨环境QTL分析和基于QTL的花诱导和开花时间估计芸苔属植物oleracea.昆虫学报,2008,21:205-216。10.1007 / s11032 - 007 - 9121 - y。
- 42.
Schranz ME, Quijada P, Sung SB, Lukens L, Amasino R, Osborn TC:开花时间复制基因的特征及其影响方法在芸苔属植物拉伯.中国生物医学工程学报,2002,29(3):344 - 344。
- 43.
Nishioka M, Tamura K, Hayashi M, Fujimori Y, Ohkawa Y, Kuginuki Y, Harada K:螺栓插入时间qtl的映射芸苔属植物拉伯(syn。定)在不同的环境条件下。中国畜牧兽医学报,2005,29(3):344 - 344。10.1270 / jsbbs.55.127。
- 44.
Lou P,赵俊杰,Kim JS, Shen SX, Del Carpio DP, Song XF, Jin MN, Vreugdenhil D, Wang XW, Koornneef M, Bonnema G:菊科植物多居群开花时间和形态性状的数量性状位点芸苔属植物拉伯.应用物理学报,2007,38(4):344 - 344。10.1093 / jxb / erm255。
- 45.
杨霞,于玉军,张福龙,邹志荣,赵晓霞,张德生,徐建斌:基于双单倍体群体的油菜抽采连锁图谱构建及数量性状位点分析芸苔属植物拉伯.中国植物学报,2007,49:664-671。10.1111 / j.1744-7909.2007.00447.x。
- 46.
赵j, Kulkarni V,刘n, Del Carpio DP, Bucher J, Bonnema G:BrFLC2(开花位点c)作为春化响应QTL的候选基因芸苔属植物拉伯.应用物理学报,2010,31(2):344 - 344。10.1093 / jxb / erq048。
- 47.
Axelsson T, Shavorskaya O, Lagercrantz U:芸苔属植物中多个开花时间qtl可能是一个祖先基因重复拷贝的结果。基因组学报,2001,44:856-864。
- 48.
Osterberg MK, Shavorskaya O, Lascoux M, Lagercrantz U:自然发生的内因变异黑芸苔菌COL1基因与开花时间的变化有关。中国生物医学工程学报,2002,26(3):349 - 349。
- 49.
皮雷,赵建文,Schranz ME, Leon EJ, Quijada PA, Lukens LN, Osborn TC:重组芸苔多倍体开花时间差异与基因组重排(十字花科).中国生物医学工程学报,2004,29(3):344 - 344。10.1111 / j.1095-8312.2004.00350.x。
- 50.
Kuittinen H, de Haan AA, Vogl C, Oikarinen S, Leppala J, Koch M, Mitchell-Olds T, Langley CH, Savolainen O:比较近亲缘关系的连锁图谱拟南芥lyrata而且答:芥.中国生物医学工程学报,2004,29(3):344 - 344。10.1534 / genetics.103.022343。
- 51.
杨晓明,王晓明,王晓明,等:拟南芥亚种的遗传连锁图谱。lyrata和拟南芥subsp。结合匿名和拟南芥来源的标记。中国生物工程学报,2007,30(4):344 - 344。10.1139 / g06 - 144。
- 52.
Yogeeswaran K, Frary A, York TL, Amenta A, Lesser AH, Nasrallah JB, Tanksley SD, Nasrallah ME:拟南芥进化历史中染色体重排事件的比较基因组分析答:芥.中国生物医学工程学报,2005,30(4):457 - 457。10.1101 / gr.3436305。
- 53.
胡TT, Pattyn P, Bakker EG,曹j,程jf, Clark RM, Fahlgren N, Fawcett JA, Grimwood J, Gundlach H, Haberer G, Hollister JD, Ossowski S, Ottilar RP, Salamov AA, Schneeberger K, Spannagl M,王x, Yang L, Nasrallah ME, Bergelson J, Carrington JC, Gaut BS, Schmutz J, Mayer KFX, Van De Peer Y, Grigoriev IV, Nordborg M, Weigel D, Guo YL拟南芥lyrata基因组序列的基础和基因组大小的快速变化。王志强,2011,30(4):344 - 344。10.1038 / ng.807。
- 54.
Hruz T, Laule O, Szabo G, Wessendorp F, Bleuler S, Oertle L, Wdimayer P, Gruissem W, Zimmermann P: genevsigator V3:转录组元分析参考表达数据库。生物信息学进展。2008,420747.q。
- 55.
Beilstein MA, Nagalingum NS, Clements MD, Manchester SR, Mathews S:古分子系统发育表明中新世的起源拟南芥.国立科学院(美国)。2010, 37(4): 457 - 457。10.1073 / pnas.0909766107。
- 56.
科赫马,马特辛格M:进化和遗传分化的亲缘关系拟南芥.国立科学院(美国)。2007, 34(4): 457 - 457。10.1073 / pnas.0701338104。
- 57.
李萨克MA, Berr A, Pecinka A, Schmidt R, McBreen K, Schubert I:染色体数目减少的机制拟南芥和相关的十字花科物种。国立科学院(美国)。2006, 30(4): 344 - 344。10.1073 / pnas.0510791103。
- 58.
宋SB, Amasino RM:春化拟南芥由PHD手指蛋白VIN3介导。自然科学学报,2004,27(4):439 - 439。10.1038 / nature02195。
- 59.
Town CD,张F, Maiti R, Crabtree J, Haas BJ, Wortman JR, Hine EE, Althoff R, Arbogast TS, Tallon LJ, Vigouroux M, Trick M, Bancroft I:比较基因组学芸苔属植物oleracea而且拟南芥揭示多倍体后基因丢失、破碎和扩散。植物学报,2006,18:1348-1359。10.1105 / tpc.106.041665。
- 60.
Allender CJ, Allainguillaume J, Lynn J, King GJ:简单序列重复揭示了植物叶绿体基因组遗传多样性的不均匀分布芸苔属植物oleraceaL.和(n = 9)野生亲缘。应用理论与实践,2007,29(1):1 -6。10.1007 / s00122 - 006 - 0461 - 5。
- 61.
Schmidt R, West J, Cnops G, Love K, Balestrazzi A, Dean C: 4个YAC contigs的详细描述,代表17 Mb的第4染色体拟南芥生态型哥伦比亚。植物学报,1996,9:755- 755。10.1046 / j.1365 - 313 x.1996.9050755.x。
- 62.
Raybould AF, Mogg RJ, Clarke RT, Gliddon CJ, Gray AJ:野生白菜微卫星和同工酶位点的变异与群体结构(芸苔属植物oleraceaL.)在多塞特(英国)。作物学报,1999,29(4):359 - 366。10.1023 /: 1008658630440。
- 63.
Sharpe AG, Parkin IAP, Keith DJ, Lydiate DJ:油菜双二倍体基因组的频繁非互易易位(芸苔属植物显著).中国生物工程学报,1995,38:1112-1121。10.1139 / g95 - 148。
- 64.
van Ooijen JW, Voorrips RE: JoinMap版本3.0:遗传连锁图谱计算软件。瓦赫宁根:国际植物研究中心;2001.
- 65.
Howell EC, Barker GC, Jones GH, Kearsey MJ, King GJ, Kop EP, Ryder CD, Teakle GR, Vicente JG, Armstrong SA:细胞遗传学和遗传连锁图谱的整合芸苔属植物oleracea.中国生物医学工程学报,2002,26(3):344 - 344。
- 66.
Gerlach WL, Bedbrook JR:小麦和大麦核糖体基因的克隆和特征。中国生物医学工程学报,1997,27(3):357 - 357。10.1093 / nar / 7.7.1869。
- 67.
甘蓝和拟南芥的生物钟调控。华威大学:博士学位论文;2003.
- 68.
figfigski DH, Helinski DR:质粒RK2含起源衍生物的复制依赖于trans中提供的质粒功能。国立科学院(美国)。1979, 76: 1648-1652。10.1073 / pnas.76.4.1648。
- 69.
Bechtold N, Ellis J, Pelleteir G:植物农杆菌属成虫内流介导的基因转移拟南芥植物。中国生物医学工程学报,2003,26(3):344 - 344。
确认
我们感谢David Laurie, Lars Østergaard和Martin Trick对手稿的批判性阅读,Theresa Townsend对初始BAC识别的帮助,David Turner (JIC基因组实验室)对BAC文库的构建和初始测序BoFRIa而且BoFRIb约翰·沃尔肖(John Walshaw)在线圈分析方面提供建议,Elsoms Seeds Ltd的苏·肯尼迪(Sue Kennedy)提供E1和E8种子。我们也感谢Andreas Mueller提前访问b .矮麝香鹿电子出版后的FRI序列。这项工作由Defra拨款HH3708SFV, Defra可行性园艺LINK拨款HL0186和BBSRC战略拨款John Innes中心资助。的b . oleracea《多样性基金会集》由英国沃里克大学制作,由Defra项目IF0157和HH3723XS资助。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
JI和CD构思并设计实验,监督工作并撰写论文。CL、YZ和JI对BoFRIa等位基因进行分析。ES和JI分析了BolDFS, GT从BolDFS中贡献了DNA。EH进行了FISH实验。所有作者都同意并阅读了最终的手稿。
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
欧文,j.a.,李斯特,C.,索姆普鲁,E.。et al。开花时间调节因子的功能等位基因FRIGIDA在芸苔属植物oleracea基因组。BMC植物生物学12日,21(2012)。https://doi.org/10.1186/1471-2229-12-21
收到了:
接受:
发表:
关键字
- FRIGIDA
- 开花的时间
- 春化处理
- 同线性
- 芸苔属植物oleracea
- 拟南芥