抽象的
背景
男性繁殖是植物生命周期中的基本生物事件,分离二倍体孢子体和单倍体配对聚合物代理,涉及约20,000个基因的表达。男性繁殖的控制也是植物育种和杂种种子生产的经济意义。随着前进和逆向遗传和基因组技术的出现,已经确定了大量的雄性生殖相关基因。因此,个别研究人员来系统地收集和不断更新的所有可用信息,对植物男性繁殖有关的所有可用信息是极为挑战。本研究的目的是手动愈合这些基因和突变信息,并提供网络可访问的资源,以促进植物男性繁殖的有效研究。
描述
工厂男性再现数据库(PMRD)是浏览和检索与植物男性繁殖相关的基因和突变体知识的综合资源。它基于文献和生物数据库,包括506个雄性无菌基因和484个突变体,具有来自各种植物物种的男性繁殖的缺陷。基于基因本体论(GO)注释和文献,系统地收集和包括与另外的3697名雄性再现相关基因有关的信息,并将在文献中,在文献中捕获基因表达和表型信息。PMRD提供了一个Web界面,允许用户轻松访问策划的注释和基因组信息,包括全名,符号,位置,序列,表达模式,基因的功能,突变表型,雄性无菌类别和相应的出版物。PMRD还提供迷你工具来搜索和浏览微阵列数据集中的基因的表达式模式,运行BLAST搜索,转换基因ID并生成基因网络。此外,MediaWiki引擎和论坛已集成在数据库中,允许用户分享他们的知识,发表评论并讨论主题。
结论
PMRD提供遗传研究与快速增长的基因组信息之间的综合联系。因此,该数据库提供了植物男性繁殖的全球视野,因此有助于这个重要领域。
背景
男性繁殖是一种复杂且高度协调的生物过程,包括雄性生殖器官,雄蕊的雄性孢子/花粉的发展,以及随后的花粉释放,授粉,花粉管生长,指导,接待,配子迁移和最后受精[1- - - - - -6].雄蕊包括花药,其具有多种专门的细胞/组织,用于生产可行的花粉和支持花药的长丝。微孔/花粉发育需要减少菌和随后的有丝分裂的,并且在花药内的配子育和孢子体组织之间的许多合作功能相互作用。花粉发育需要精确的时尚表达基因,策划活动和酶局部控制,细胞对细胞通信,细胞开发和分化[2,6].此外,通过环境影响或基因突变的基因表达破坏,经常导致生育率降低,或完全雄性不育,导致农业收益率丧失。对植物生育的控制也具有经济意义与农业农业用于作物改善的一些男性无菌线,例如在繁殖超级杂交水稻中[7].
由于雄性生殖的重要性,人们对植物雄性生殖的分子调控进行了大量的研究。转录组分析表明,水稻中有20,000多个基因表达(奥雅萨苜蓿)产生花药,大约18000个拟南芥(拟南芥蒂利亚纳)花粉[8- - - - - -10.];提示在花药开发和花粉形成过程中发生广泛的基因表达变化[11.].此外,最近的前向和逆向遗传研究鉴定了大量的雄性无菌突变体和相关基因[1,12.,13.].然而,单个研究人员获取植物雄性生殖的准确信息既费时又低效。这与物种间的比较分析特别相关。
为了弥合遗传研究与植物男性繁殖中的基因组信息之间的差距,我们通过手动策策系统地收集了雄性不育突变体和基因信息,并创建了PMRD(植物男性再生数据库)数据库。该数据库提供了从遗传研究的快速增长的基因组数据和知识的双向整合,无疑将改善我们对植物男性繁殖机制的理解。PMRD不仅可以作为一种高质量的策划数据库,用于浏览和检索植物男性繁殖中基因和突变体的知识,而且作为具有用于访问基因组信息的生物信息工具的动态网站。此外,PMRD设计有知识共享功能,包括维基和论坛工具,以促进社区注释,信息共享和教育。
结构和内容
植物男性繁殖相关基因的集合
PMRD包含的基因分为两类:雄性不育基因(MS)和雄性生殖相关基因(MR)。MS基因和MR基因之间的差异在于MS基因的功能是通过对男性生殖能力或传播效率降低的突变体进行分析来证明的,而MR基因则是在没有遗传学证据的情况下,MR基因在男性生殖中具有假定的功能。MS基因是从文献和生物数据库检索中鉴定出来的。MR基因根据GO注释、TAIR种质表型及文献表达信息鉴定[14.].为了建立一个文献库,我们通过Pubmed和期刊数据库检索广泛收集了植物雄性生殖的遗传和分子研究的文献。全文检索共370篇论文,其中水稻相关论文143篇(奥雅萨苜蓿),187篇论文拟南芥蒂利亚纳和40篇植物物种的纸张。从该本地文献343 MS基因储存和321毫秒突变体。接下来,我们收集了来自两种稻米数据库的163毫秒突变体:奥比扎萨斯和中国稻米数据中心[15.,16.].为了识别先生基因,我们收集了与GO联盟的植物男性复制相关的41条术语[14.].随后,我们将41术语映射到RAP-DB,TAIR和PLAZA网站的注释[17.- - - - - -19.)(见附加文件1).关于文献中鉴定的先生,我们收集了3697个先生基因。因此,当与MS基因组合时,我们在33种含有涉及植物男性繁殖中的33种中鉴定了4203个基因和484个突变体(表1).
数据输入和策策
将出版物中的信息整理成结构良好的可搜索知识库是生物数据库建设的关键步骤。这包括论文的手工审查、生物实体的鉴定、使用的实验方法的定义、将实验结果和表型观察转化为标准格式,以及总结基因功能数据。在PMRD筛选过程中,首先检查论文是否适合作为PMRD中的MS/MR基因。纳入MS基因的标准是该基因的突变必然导致男性生殖缺陷。一旦确定了全名,基因符号和基因的简要描述就得到了。对基因产物表达模式、分子生物学功能等相关信息进行整理。然后将水稻和拟南芥的基因定位到RAP-DB和TAIR位点上,并纳入PMRD。对于其他物种,使用了论文中提到的基因名称。在水稻和拟南芥中,利用对照解剖学和阶段词汇表进行了详细的基因表达分析。如果这些论文包含突变体的遗传或转基因研究,馆长会收集以下信息:突变体名称、突变基因、突变方法、显性、突变表型和男性不育类别。 All curated information was checked and confirmed by senior experts in this field.
数据库设计实现
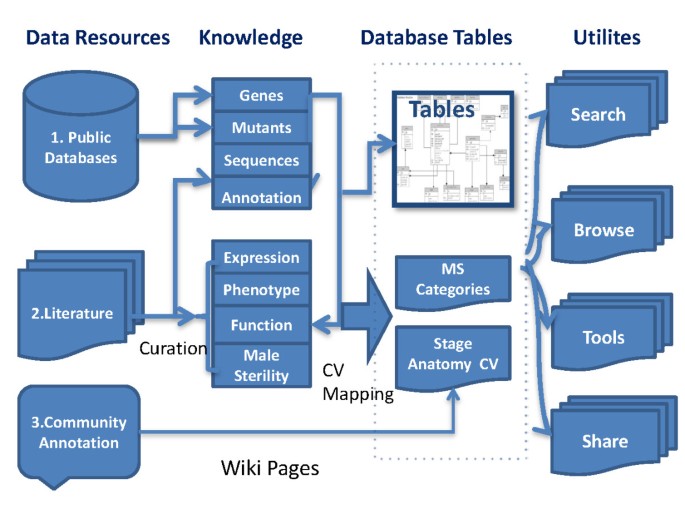
PMRD用作数据库系统,汇集了三个主要知识来源:1)来自公共数据库的一般基因组信息;2)文献中的遗传学研究的详细策委;3)研究界的公开注释(图1).在基因组注释部分中,显示染色体位置,序列,GO条款,KEGG路径信息和Interproito注释[14.,15.,17.,18.,20.,21.].从Geo下载植物男性再现相关的微阵列数据集[22.].为了提供关于突变表型和基因表达的详细解剖信息,我们首先设计了标签和受控词汇(CV),然后用于在策施过程期间归一化信息。根据公开接受的标准,设定了发展阶段和解剖学的控制词汇[1,23.].PMRD中的愈合信息包括:基因函数,基因表达模式,突变体背景,诱变方法,突变体表型和雄性无菌类型定义的描述。对花药开发和花粉形成的基因进行了整理,并且在网页上显示的二维模块中组织的信息将基因和突变体与阶段和组织联系起来,允许多种方式浏览感兴趣的基因和突变体。对于我们资本化社区注释的其他男性再现流程,并创建了4个在线数据收集表,包括“授粉”,“花粉萌发和管生长”,“指导和感知”,“迁移和融合”。我们还将MediaWiki发动机集成到PMRD中,从而允许用户贡献他们对突变体,发展阶段,解剖学的知识,并创建他们对其有兴趣的其他主题。最后,还设置了一个论坛以便于讨论讨论。
PMRD使用MySQL作为存储引擎运行在Nginx服务器上。使用PHP和JavaScript库实现Web界面[24.].MediaWiki引擎作为社区注释工具集成到PMRD中[25.].PRMD中的不同界面由Joomla内容管理系统进行网站维护[26.].这个网页在所有主流浏览器中都运行良好。
实用与讨论
数据库Web界面
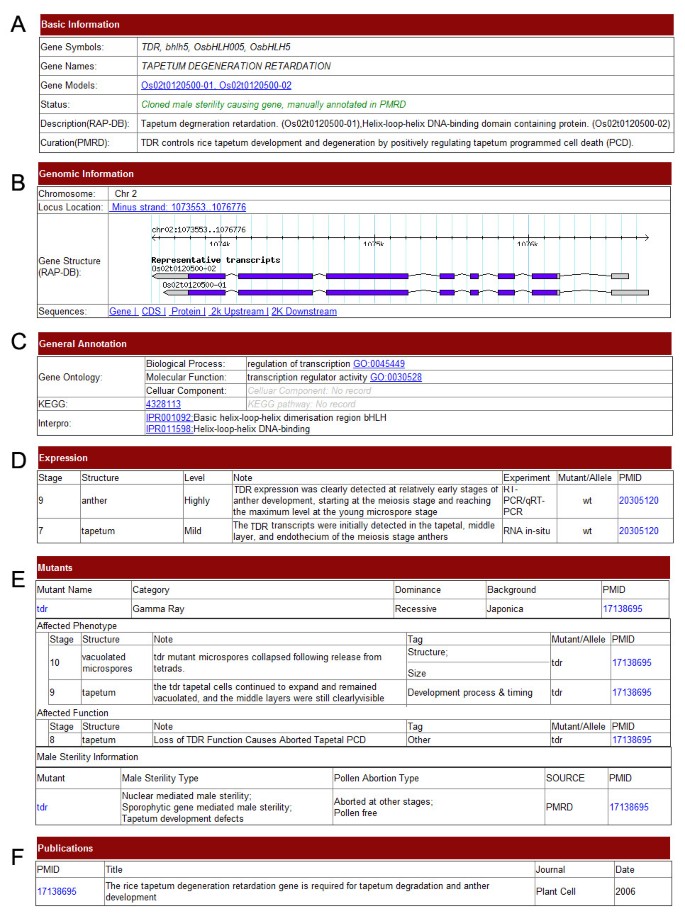
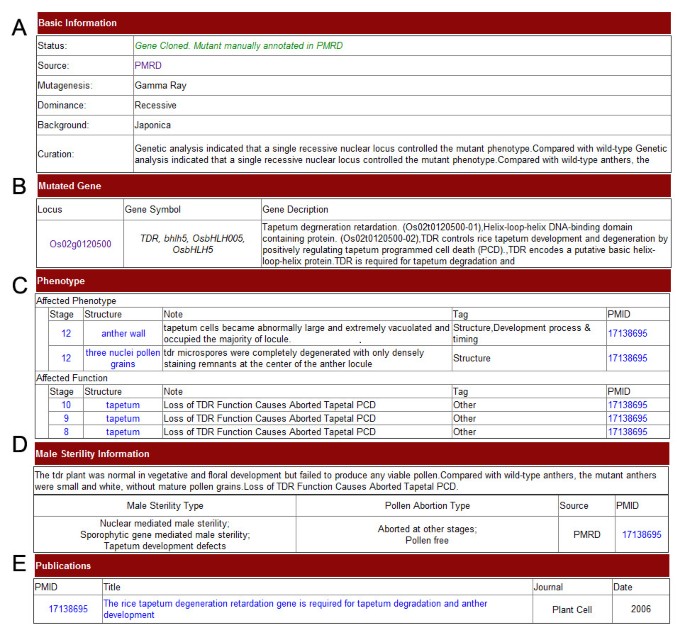
我们开发了一个用户友好的Web界面,用于搜索和浏览PMRD中的信息。用户可以通过名称,标识符,序列,表达,表型和相对突变体的雄性无菌类别来轻松搜索基因。由于不同物种的数据结构不相同,因此搜索和浏览的网页被分组为主要PMRD网站菜单中的水稻,拟南芥和其他物种。要使信息检索方便精确,搜索页面旨在包括简单和高级选项。显示关于MS基因信息的网页包含六个部分(图2).第一部分显示基因的“基本信息”,包括基因符号,基因名称,来自外部数据库的基因的描述,并用作PMRD工作人员的策委。第二部分包含“基因组信息”,例如位置,基因结构和序列。第三部分显示从外部数据库中检索的“一般注释”,包括GO条款,Kegg Pathway信息和Interpro蛋白质签名[14.,20.,21.].第四部分显示基因的“表达模式”。大米和拟南芥的表达信息从文献策策和TAIR注释中获得[18.].第五部分总结了该基因的“突变体”信息,包括突变体、表型和雄性不育类别。雄性不育分类是根据植物本体(PO)和水稻知识库[27.,28.].一个突变体可以被分配到多个类别。如果无法从数据源中获得突变体的详细雄性不育信息,则将其赋值为“未定义”。第六部分为与基因相关的“出版物”。显示突变信息的Web页面被组织成五个部分(图3.).基本信息包括诱变方法,优势,背景以及突变体的简短描述。以下部分包括突变基因的信息和链接。第三部分显示突变表型观察的策析。最后两部分显示雄性不育信息和相关的出版物。如果是一个很长的页面,用户可以折叠/展开每个部分的面板,但由于异构数据源,并非所有这些都包含上述所有部分的完整数据集。
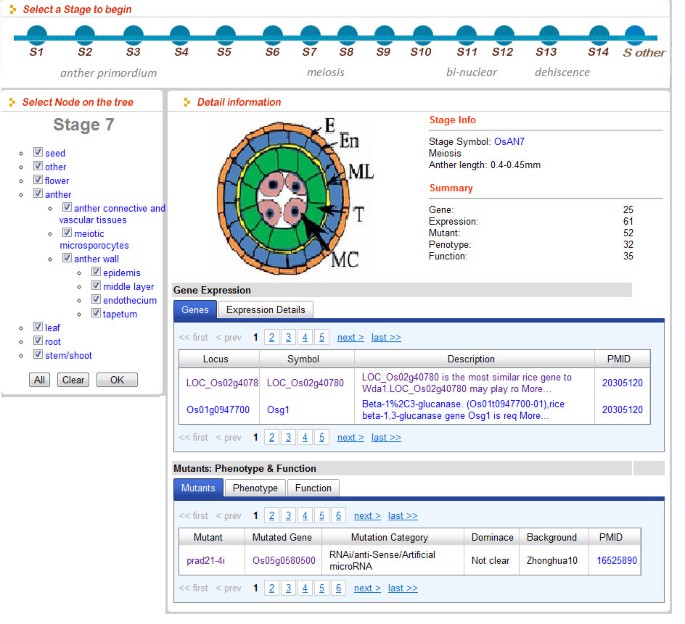
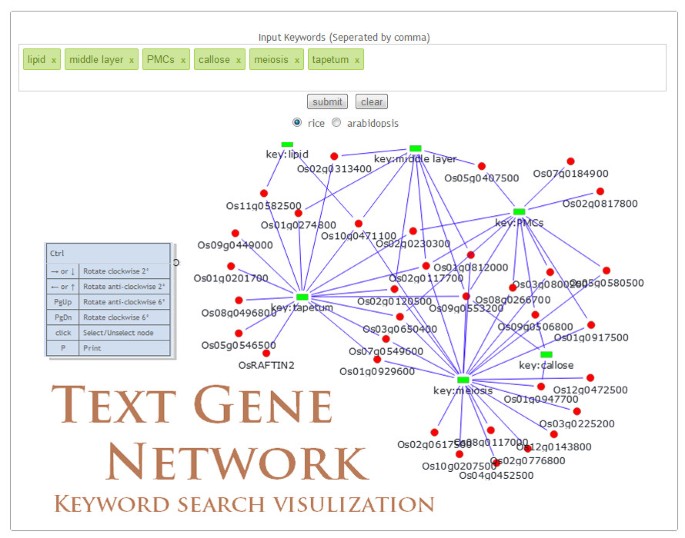
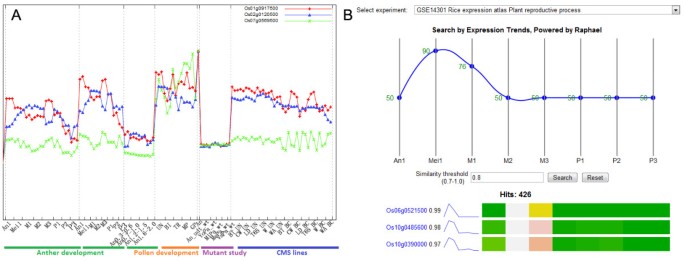
PMRD还提供了各种工具,用于信息检索和显示。创建了“浏览页面”(图4)作为集线器,根据不同雄性再现过程的阶段和组织,将关于基因,突变体,表达和表型的信息集成到单个界面中。使用Ajax技术来浏览阶段和组织而无需刷新。为了使更直观和信息丰富的多个关键字搜索,我们开发了一种使用Canvasxpress和CytoscapeWeb来可视化关键字 - 基因关系的工具(图5)[24.,29.].这在与基因相关的数据条目中出现关键字,包括基因描述,表达,相关突变表型和GO注释,这是在MS基因和关键字之间的连接。对于微阵列数据,用户可以在微阵列可视化页面和微阵列搜索页面上浏览和搜索表达信息(图6).我们还提供了一种用于爆炸搜索米和拟南芥的工具,以帮助用户通过序列和ID转换器来搜索基因,用于不同的数据库[18.,30.- - - - - -33.].最后,一个具有易于使用的编辑器插件的Wiki页面已经设置为推广社区信息贡献;我们鼓励用户在Wiki页面中贡献他们的知识并向我们推荐文献。
未来发展方向
比较功能基因组学研究是一种新兴的研究方法,它依赖于将模式物种积累的大量知识应用于较少特征物种。最近,一些植物比较或功能基因组学网站已经开发出来,如PLAZA、Phytozome、the flora Genome Project、MoccaDB、SolRgene和BRAD [19.,34.- - - - - -38.].随着更多植物基因组序列可用,延长并应用PMRD的当前知识将是有趣的,以进行比较研究。PMRD的未来版本将提供用于比较和开采男性再现相关基因的跨物种工具。最后有迫切需要自动文献策划,因为手动文本策策是为注册者提供有挑战性的工作,这需要更有专业知识和奉献。用于模型物种的许多网关数据库采用了文本挖掘工具。鼠标基因组信息学已启动基于字典的文本挖掘工具,以帮助生物趋势[39.].Flybase开发了自然语言处理和自动实验信息分类工具,以帮助策策[40,41.].目前PMRD的数据来源多为遗传学和分子研究的文献。在这些论文中,信息通常被组织成可识别的部分,如基因的初始特征描述、基因表达分析和形态表型观察等。目前有两种文本挖掘工具可用于拟南芥[42.,43.];希望这样的文本处理软件将用于数据库的未来更新和维护中。
最后,植物男性繁殖涵盖了广泛的生物过程,PMRD的改善需要持续的努力和社区贡献。PMRD的第一个版本基于主要来自花粉和花粉发育的数据。对于未来的更新,我们已打开在线数据收集表以扩展相关主题的详细覆盖范围。
结论
植物雄性生殖数据库(PMRD)是一个综合性的资源,用于浏览和检索与植物雄性生殖有关的基因和突变体。目前,PMRD保存了33种植物雄性生殖相关的4203个基因和484个突变体的信息。两种主要模式植物,水稻和拟南芥,有最多的条目和最详细的管理。该数据库的最终目标是进一步扩展这一功能,为植物雄性生殖研究提供一个动态的、全面的信息资源和相关的数据挖掘工具。
可用性和要求
PMRD数据库可在[44.].
缩写
- 简历:
-
受控词汇表
- 地理:
-
基因表达综合
- 走:
-
基因本体论
- Kegg:
-
Kyoto基因和基因组的百科全书
- 基因先生:
-
男性繁殖相关基因
- 基因女士:
-
雄性不育基因
- PO:
-
植物本体
- RAP-DB:
-
稻米注释项目数据库
- TAIR:
-
拟南芥信息资源。
参考
- 1。
张D,罗X,朱L:水稻撒疹发展的细胞学分析和遗传控制。j遗传学基因组学。2011,38(9):379-390。10.1016 / J.JGG.2011.08.001。
- 2。
张达,威尔逊Za:米饭规范和稻米花药开发。Chin Sci Bull。2009,54(14):2342-2353。10.1007 / s11434-009-0348-3。
- 3.
常王,王Y,王S,MA H:拟南芥中孢子发生的分子控制。CurrOp植物BIOL。2011,11(1):66-73。10.1016 / J.PBI.2010.11.001。
- 4。
Berger F, Hamamura Y, Ingouff M, Higashiyama T:双受精-被捕获的行为。植物科学进展,2008,13(8):437-443。10.1016 / j.tplants.2008.05.011。
- 5。
张艾,吴亨:花粉管中的结构和功能性分区化。J Exp Bot。2007,58(1):75-82。
- 6。
开花的浪漫:开花植物中的配子相互作用。细胞生物学杂志。2006,8(1):14-16。10.1038 / ncb0106-14。
- 7。
程世,庄杰,范益,杜杰,曹莉:杂交稻研究与开发研究:中国超级驯化。Ann Bot。2007,100(5):959-966。10.1093 / AOB / MCM121。
- 8。
Fujita M,Horiuchi Y,Ueda Y,Mizuta Y,Kubo T,Yano K,Yamaki S,Tsuda K,Nagata T,Niihama M等:乳房生殖发展中的米表达式地图集。植物细胞生理。2010,51(12):2060-2081。10.1093 / PCP / PCQ165。
- 9。
Pina C, Pinto F, Feijo JA, Becker JD:拟南芥花粉转录组基因家族分析揭示了细胞生长、分裂控制和基因表达调控的生物学意义。植物营养与肥料学报。2005,32(2):441 - 446。10.1104 / pp.104.057935。
- 10。
Deveshwar P,Bovill Wd,Sharma R,能够Ja,Kapoor S:鉴定水稻中有含量分裂和雄性配子瘤发育的特征om的分析。BMC植物BIOL。2011,11:78-10.1186 / 1471-2229-11-78。
- 11.
Chen C, Farmer AD, Langley RJ, Mudge J, Crow JA, May GD, Huntley J, Smith AG, Retzel EF:植物减数分裂特异性基因的发现:RNA-Seq在拟南芥雄性减数分裂细胞中的应用。植物生态学报。2010,30(4):593 - 598。
- 12.
铃木G:植物繁殖研究中的最新进展:雄性配子体通过施肥的故事。植物细胞生理。2009,50(11):1857-1864。10.1093 / PCP / PCP142。
- 13。
MA X,FEENG B,MA H:AMS依赖性和对花药转录组的独立调节,并与受其他Arabidopsis制药基因影响的人的比较。BMC植物BIOL。2012,12(1):23-10.1186 / 1471-2229-12-23。
- 14。
Ashburner M,Ball Ca,Blake Ja,Botstein D,Butler H,Cherry JM,Davis AP,Dolinski K,Dwight SS,EPPIG JT,等:基因本体:统一生物学的工具。基因本体组织。NAT Genet。2000,25(1):25-29。10.1038 / 75556。
- 15.
库拉塔n,yamazaki y:oryzabase。米饭的综合生物学和基因组信息数据库。植物理性。2006,140(1):12-17。
- 16。
中国稻米数据C: [http://www.ricedata.cn.].
- 17。
Ohyanagi H,Tanaka T,Sakai H,Shigemoto Y,Yamaguchi K,Habara T,Fujii Y,Antonio Ba,Nagamura Y,Imanishi T,等:Real Annotation项目数据库(RAP-DB):oryza sativa ssp的集线器。粳稻基因组信息。核酸RES。2006,34:741-744。10.1093 / nar / gkj094。
- 18。
王志强,王志强,王志强,等:拟南芥信息资源(TAIR)的基因结构和功能注释。核酸学报2008,36(4):593 - 598。
- 19。
Van Bel M, prost S, Wischnitzki E, Movahedi S, Scheerlinck C, Vande Peer Y, Vandepoele K:利用PLAZA比较基因组学平台解剖植物基因组。植物营养与肥料学报。2012,29(2):429 - 434。10.1104 / pp.111.189514。
- 20。
亨特S,Apweiler R,阿特伍德TK,贝罗奇A,贝特曼A,Binns的d,博克P,达斯U,多尔蒂L,Duquenne L,等人:的InterPro:一体的蛋白质签名数据库。核酸RES。2009,37:211-215。10.1093 / nar / gkn785。
- 21。
Ogata H,Goto S,Sato K,Fujibuchi W,Bono H,Kanehisa M:Kegg:基因和基因组的百科全书。核酸RES。1999,27(1):29-34。10.1093 / nar / 27.1.29。
- 22。
Barrett T, Troup DB, Wilhite SE, Ledoux P, Evangelista C, Kim IF, Tomashevsky M, Marshall KA, phillip KH, Sherman PM, et: NCBI GEO:功能基因组学数据集存档——10年。中国生物医学工程学报。2011,39(4):491 - 497。10.1093 / nar / gkq1184。
- 23。
桑德斯PM,Bui AQ,Weterings K,McIntire Kn,Hsu YC,Lee Py,Truong Mt,Beals TP,Goldberg RB:拟南芥拟南芥的植物发育缺陷。性植物团体。1999,11(6):297-322。10.1007 / S004970050158。
- 24。
YUI图书馆:[http://developer.yahoo.com/yui/].
- 25。
canvasxpress javascript libarary:[http://www.canvasxpress.org/].
- 26。
拉斐尔javascript libarary:[http://raphaeljs.com/].
- 27。
植物本体和植物本体。Comp Funct基因组学。2002,3(2):137-142。10.1002 / CFG.154。
- 28。
- 29。
Lopes CT,Franz M,Kazi F,Donaldson SL,Morris Q,Bader Gd:Cytoscape Web:基于交互式Web的网络浏览器。生物信息学。2010,26(18):2347-2348。10.1093 / Bioinformatics / BTQ430。
- 30.
Altschul SF,Gish W,Miller W,Myers EW,Lipman DJ:基本的本地对齐搜索工具。J Mol Biol。1990,215(3):403-410。
- 31。
欧阳松,朱伟,Hamilton J, Lin H, Campbell M, Childs K, Thibaud-Nissen F, Malek RL, Lee Y, Zheng L, et al: TIGR水稻基因组注释资源:改进和新特性。核酸学报2007,35(5):593 - 598。10.1093 / nar / gkl976。
- 32。
Pruitt KD,Tatusova T,Brown Gr,Maglott Dr:NCBI参考序列(Refseq):当前状态,新功能和基因组注释策略。核酸RES。2012,40:135-10.1093 / NAR / GKS395。
- 33。
Boutet E,Lieberherr D,Tognolli M,Schneider M,Bairoch A:Uniprotkb / Swiss-prot。方法Mol Biol。2007,406:89-112。
- 34。
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J,Mitros T,Dirks W,Hellsten U,Putnod N等:Phytozome:绿色植物基因组学的比较平台。核酸RES。2012年,40:1178-1186。10.1093 / NAR / GKR944。
- 35。
Albert VA,Soltis de,Carlson Je,Farmerie Wg,Wall PK,ILUT DC,Solow TM,Mueller La,Landerr LL,Hu Y等:来自基础上的Grene资源,对比较基因组学研究。BMC植物BIOL。2005,5:5-10.1186 / 1471-2229-5-5。
- 36。
Plechakova O,Tranchant-Dubreuil C,Benedet F,Couderc M,Tinaut A,Viender V,De Block P,Hamon P,Campa C,De Kochko A等Al:Moccadb - 一种综合数据库,用于功能,比较和多样性研究Rubiaceae家族。BMC植物BIOL。2009,9:123-10.1186 / 1471-2229-9-123。
- 37。
vleeshouwers vg,Finkers R,Budding D,Visser M,Jacobs MM,Van Berloo R,PEL M,Chipcroit N,Bakker E,Krienek P等人:Solrgene:探索核心溶朗物种中疾病抗病基因的在线数据库.BMC植物BIOL。2011,11:116-10.1186 / 1471-2229-11-116。
- 38。
程F,刘某,吴俊,方L,Sun S,Liu B,Li P,Hua W,Wang X:Brad,甘蓝型植物的遗传学和基因组学数据库。BMC植物BIOL。2011,11:136-10.1186/1471-2229-11-136。
- 39。
Dowell KG,McAndrews-Hill Ms,Hill DP,Drabkin HJ,Blake JA:将文本挖掘整合到MGI酶的工作流程中。2009年,数据库(牛津),19。
- 40。
Karamanis N,Seal R,Lewin I,McQuilton P,Vlachos A,Gasperin C,Drysdale R,Briscoe T:vicebase策展人的自然语言处理。BMC生物信息学。2008,9:193-10.1186 / 1471-2105-9-193。
- 41。
方岚,帆列师G,van Auken K,Fernandes J,Chen W,Wang X,Davis P,Tuli Ma,Marygold S,Millburn G等:在生物科学文献中的多样化实验信息自动分类。BMC生物信息学。2012,13(1):16-10.1186 / 1471-2105-13-16。
- 42。
Bajic VB, Veronika M, Veladandi PS, Meka A, Heng MW, Rajaraman K, Pan H, Swarup S: Dragon Plant Biology Explorer。一种文本挖掘工具,用于集成与基因组注释和生化术语列表的遗传和生化实体之间的关联。植物营养与肥料学报。2005,32(4):457 - 461。10.1104 / pp.105.060863。
- 43。
Krallinger M,Rodriguez-Penagos C,Tendulkar A,瓦伦西亚A:Plan2L:用于集成文本挖掘和基于文学的生物性关系提取的网络工具。核酸RES。2009,37:160-165。10.1093 / nar / gkp484。
- 44。
植物男性再现数据库:[http://www.pmrd.org].
承认
我们感谢Hugh Dickinson教授、杨伟才教授、Chris Franklin教授、张兵博士、瞿丽佳教授、David Twell教授、李耀光教授、叶德教授和徐杰博士的有益讨论。我们也要感谢BBSRC中国伙伴计划提供的机会,将在英国和中国从事植物繁殖工作的同事联系在一起,并发起建立繁殖数据库的想法。
来自中国国家自然科学基金的资金支持这项工作[31110103915和30830014至DB.Z.];来自中国国家基础研究计划[2013CB129602至DB.Z.];中国转基因项目[2011ZX08012-002至DB.Z];国家863高科技项目[2011AA10A101和2012AA10A302至DB.Z];并由江苏高等教育机构优先学术方案开发资助的项目。
作者信息
隶属关系
通讯作者
额外的信息
相互竞争的利益
提交人声明他们没有竞争利益。
作者的贡献
XC,DW和Hy设计并实施了数据库,网站页面和在线工具。XC,DW,Zaw,DZ,WY负责数据收集,手动策划和质量控制。PS,CW参与了数据库模式的设计。Zaw和Dz构思了这项研究。XC,Zaw,DZ起草了手稿。所有作者均阅读并批准了手稿。
作者的原始提交的图像文件
权利和权限
开放访问本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
崔,X.,王,Q.,Yin,W。et al。PMRD:涉及植物雄性生殖的基因和突变体的策划数据库。BMC植物BIOL.12,215(2012)。https://doi.org/10.1186/1471-2229-12-215
已收到:
接受:
发表:
关键字
- 植物男性繁殖
- 数据库
- 基因
- 突变体
- 花粉
- 花药