抽象的
背景
异源多倍体植物的遗传研究具有挑战性,因为存在相似的亚基因组,导致多个等位基因和复杂的分离比。在这项研究中,我们描述了一种新的方法来建立任何异源多倍体植物物种的微卫星等位基因的精确剂量和配置。该方法被命名为微卫星等位基因剂量和配置建立(MADCE),可用于异源多倍体群体和系谱(育种)种质的定位。
结果
通过两个实例验证了该方法的有效性和鲁棒性。在绘图方面,分析了五颗微型卫星。这些微型卫星根据大小放大了35个不同的等位基因。利用MADCE,我们发现了30个信息丰富的分离等位基因。传统方法只能产生19个完全信息性等位基因和6个部分信息性等位基因。在所有子代中存在的10个等位基因中(因此在使用常规方法时被忽略或认为是纯合的),有6个在MADCE分析时被发现按剂量分离。此外,还可以建立定位亲本的完整等位基因构型,包括空等位基因、纯合位点和存在于多个同源位点上的等位基因。在第二种情况下,利用MADCE分析了21个品种的系谱,建立了所有21个品种的全等位基因配置,并在多个世代中追踪等位基因流动。
结论
本研究所描述的程序(MADCE)提高了异源多倍体定位研究的效率和信息量。更重要的是,该技术首次实现了异源多倍体植物系谱种质全等位基因配置的测定。这使得基于系谱的标记-性状关联研究能够使用二倍体作物开发的算法,并可能提高基于ld的关联研究的有效性。因此,MADCE方法使研究人员能够解决在异源多倍体进行定位、系谱和关联研究时出现的许多基因分型问题。我们讨论了MADCE与其他多倍体标记系统(包括SNPs)相比的优点,以及MADCE如何在异源多倍体SNP标记的发展中发挥作用。
背景
多倍体是所有植物物种演变的一部分[1].有几个重要的作物品种是多倍体,包括面包小麦(Triticum Aestivum.,六倍体),棉花(Gossypium spp。,allotetraploid),马铃薯(茄属植物tuberosum,同源四倍体)和非常复杂的甘蔗(蔗糖spp自动异源多倍体)。多倍体的成功可归因于多种因素,包括它们保留有益等位基因的能力,同时允许在重复等位基因中产生新的变异,并通过永久杂交增强活力[2].
AutopolyPLoidy是多种基因组的组合或重复的结果,其足够类似于允许随机二价配对和在减数分裂期间形成多元化。这种随机染色体配对使遗传学研究结合,特别是映射研究,并限制映射研究,以耦合阶段的大多数单纯性等位基因。相比之下,从一个Zygote内的分化基因组的合并来源于衍生所有多元素[1]或通过同源多倍体的逐渐二倍化[1那3.那4.].这些分化的基因组本质上表现为减数分裂独立的亚基因组(同源基因),几乎完全在同一亚基因组的染色体之间形成二价体,导致本质上的二体遗传。异源多倍体的遗传研究是复杂的,因为等位基因可以从多个同源基因中扩增,而且这些等位基因在同源基因之间是相同的(共享的)。然而,异源多倍体表达的二体遗传使它们易于进行二倍体定位。在大多数基因图谱研究中,仅通过评估亲本一方或双方为杂合子的等位基因来规避共享等位基因的陷阱[5.].一旦初始单个亲地图已经基于1中创建:1分离的等位基因作为桥标记[:1分离等位基因,母本和父本地图可以使用3被集成6.].虽然这种方法使多倍体中的基因分型可靠且相对简单,但它是高度限制性的,并且丢失了大量信息。此外,该方法限制了在存在共享等位基因时可以用各个微卫星引物对的同优素基因座的数量限制。使用微卫星标志物在各种多多合物中的遗传学研究有几个好处。因为这些标记是多个等位基因的,所以它们可以显示出多种不同的等位基因作为个体植物的倍性水平。这允许同时映射几种同源染色体和随后对其宏观联合的评估。随着PCR产物的荧光检测技术的出现,可以可靠地量化PCR反应中的扩增子的丰度。这种等位基因剂量信息的可用性是在多倍体中建立等位基因配置的第一步。几项研究研究了使用定量数据来估算自身传递物中的等位基因剂量[7.- - - - - -12.].MAC-PR方法由Esselink等人开发[8.,使用一个附加位点中单个位点的等位基因之间的比率,并将这些比率与同一等位基因一起出现的其他附加位点的比率进行比较。不同比例的存在表明,对于正在调查的两种等位基因中至少有一种,剂量存在差异。本文提出了一种基于改进MAC-PR方法的MADCE剂量估算方法。在MADCE中,我们也使用二体分离模式和虚拟参考等位基因来改进剂量估计。本文采用MADCE方法测定了异源八倍体草莓(Fragaria×ananassa.)并展示其实用性。这种作物物种最近已经广泛研究了创建联动地图[13.- - - - - -16.].提出了与MADCE分析的五次微卫星引物对的结果,以证明其对遗传联系地图构建的有效性以及测定父母的等位基因配置。提出了延长的方法,用于养殖群品种和繁殖线中的Madce。最后,我们展示了这种方法,用于通过使用FlexQTL™通过逐个逐个概念来检查多代百分比的等位基因流程[17.]及Pedimap [18.]软件包。
结果
mapstudies的MADCE程序
微卫星等位基因剂量和构型建立(MADCE)方法由五个连续阶段组成。开始(我)微卫星数据的定性解释,紧随其后的是(2)等位基因剂量的定量评估,(3)评估最初的等位基因的配置映射的父母,(IV)分子标记连锁图谱的一代,和(V)的最后描述父母的单,包括纯合子基因座。下面通过对五对微卫星引物对的分步分析,对这些步骤进行了阐述和举例(表)1)用于一个由八倍体草莓品种'Holiday' (H)和'Korona' (K)杂交衍生的作图群体,其原始数据在附加文件中给出1通过5..这些例子两本文件中给出,并在其他文件中提供的其他三个6.那7.那8..保持表的打印输出可能很有用2和3.经过这样的例子。
微数据的一定性解释
作为第一个步骤中,我们确定所有等位基因偏析以定性的方式,即,存在/不存在。通过对这些等位基因的存在和不存在过滤,可以鉴定同源(斥力)的等位基因,由此形成等位对。
例如:微1 - UFFxa16H07:引物对UFFxa16H07扩增9个不同的等位基因(见表)2和3.附加文件2),而这些偏析定性六。因此,有可能以建立这六个等位基因的5同源:等位基因273 K是在排斥等位基因287 K,和269HK是在排斥298H和306 K.定性地确定因此等位基因对包括273 K-287 K,269H-298H和269 K-306 K.没有斥力等位基因尚未发现268 K.
2微卫星数据的定量解释
参考等位基因的鉴定
理想参考等位基因符合以下标准:在剂量无变化,存在于所有的子代,而不是通过从其它等位基因口吃的影响。对于所有后代稳定引用来自等位基因是在父母一方或双方(即,AA×AA或AA×AA,AA不×AA)纯合地存在。1为存在和不存在:一般,这些等位基因只能通过使用其他初始,非最佳参考等位基因,如分离1单纯形的等位基因区分。
例如:微1 - UFFxa16H07:等位基因262HK在所有后代和双方都存在。与几个1:1存在/缺席的比例簇显示出等位基因262HK不会被剂量隔离,因此父母双方都是纯合的。因此,该等位基因可以用作参考等位基因。
b.比率计算,聚类识别
等位基因剂量是通过检查等位基因相对于参考等位基因的峰面积的峰面积来估计的。得到的比率值(rv)绘制在频率分布中(参见附加文件)1那2那3.那4.那5.).当参考等位基因执行良好时,RV显示出围绕某种平均RV的明显正常频率分布。此比率值称为比率 - 簇(RC)。在后代鉴定多于一个透明的RC意味着存在剂量变化,这反过来表示偏析。RCS周围的窄,非重叠频率分布表示参考等位基因表现良好。每个等位基因的获得的RVS乘以经验所获得的因子,以使每个等位基因类似的RVS的范围,并促进频率分布的产生。
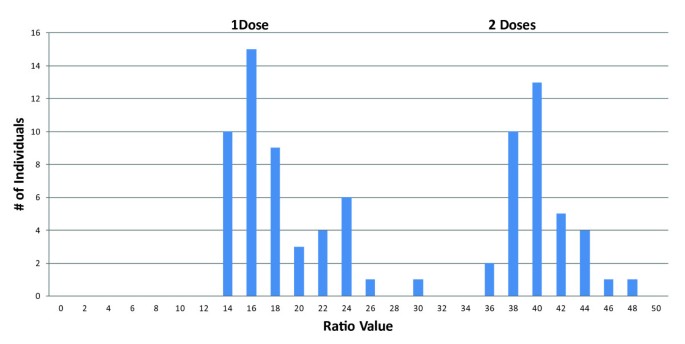
例如:微1 - UFFxa16H07:等位基因266HK和279h总是存在于所有后代。266hk有两个rcs,平均值为17和39(表3.)和允许剂量量化的簇分布(图1),加强262小时的合适性作为参考。RCS显示规则步骤,均比下平均值大约两倍。279h还显示两个rcs,平均值为十和15(表3.)因此,也以剂量分离。
c.比率团簇的分离
通过将特定RC中的个体数量与其他RCS中的数字进行比较来建立比例簇(RCS)的分离模式。存在/缺失等位基因的常见RC分离比例是:1:1(AA×AA),1:2:1(AA×AA)和1:3:3:1(AABB×AABB,其中A和B是同源性的).这些比率也发生在至少一个父母中始终存在的等位基因,但仍然在剂量方面仍然被隔离。例如,对于总是存在的等位基因的rc分离比为1:2:1(并且因此必须存在于多个同种型上)可能是由几个父母的等位基因配置引起的,例如AABB×AABB,AABB×AABB,aabb×aabb和aabb×aabb。这些父母的等位基因剂量分别为3×1,2×2,3×1和3×3。
例如:微1 - UFFxa16H07:两个266k的两个簇(图1)和279H以1:1的比例分离(表3.).
天。鉴定同源等位基因
以类似于定性方法的方式鉴定同源等位基因对,但是认为等位基因剂量代替存在和不存在。如果等位基因在其最高剂量中存在,这将自动导致剂量或不存在同源等位基因的降低,反之亦然。如果没有发现这样的等位基因,则拒绝等位基因可能是零等位基因。
例如:微1 - UFFxa16H07:最高剂量的266HK导致268k等位基因缺失,定性分析未发现排斥等位基因。这使我们确定266 K- 268k为等位基因对。由于266HK存在于所有的后代中,我们对之前的两个同源‘Korona’等位基因的分析表明‘Holiday’是266纯合的。我们现在已经完成了所有四个Korona同源基因和四个Holiday同源基因中的三个的等位基因对。剩下的唯一要分析的等位基因是279H,这将在下一节中讨论。
当父母一方或双方有多个剂量的等位基因时,有必要确定每个等位基因的剂量,以适当的基因型个体和识别等位基因对。等位基因剂量是通过检测同一PCR扩增中一个等位基因相对于其他等位基因的峰值面积来估计的,类似于Esselink等人发表的程序[8.].在两个不同的等位基因的峰面积之间的比率大的,一致的变化指示等位基因剂量的一个或两个等位基因的分离。定量解释方法被分为四个阶段,这在下面的章节中描述。
3确定亲代等位基因配置并交叉核对
该步骤的目的是通过它们的后代,以确定所述映射父母的等位基因配置。这些配置使用所估计的等位基因剂量的父母和反之亦然的随后验证。首先,母源等位基因对与使用父母之间共享的等位基因其父系同源物匹配。例如,如果父母一方(P1)具有尺寸(223223)和其他亲本(P2)的一个纯合的等位基因223和225之间的杂合配对,(223225),则等位基因对P1(223223)和P2(223225)被认为是同源的。类似地,如果一个等位基因是在父母双方纯合(例如,P1(229229),P2(229229)),这些对也被认为是同源的。当没有等位基因父母之间共享,匹配有时可以基于其他标准,如在等位基因大小的范围在同源染色体或差之间的扩增效率的差异来实现。此外,当已经确定用于同种异体八倍体三套匹配亲本等位基因对,所述第四组自动由剩余的等位基因对的最后两个的。继续匹配亲本等位基因对所述过程,直到所有组是否相符;这些集合代表同源染色体。接着,所获得的等位基因集合的数目是基于所述倍性水平预期等位基因组的数量进行比较。 If fewer sets are obtained, one of the homozygous alleles may actually be present in two homoeologues (A & B), e.g., P1(229A,229A)P2(229A,229A) + P1(229B,229B)P2(229B,229B). Alternatively, there may be a homoeologue that contains only null alleles, such as P1(229A,229A)P2(229A,229A) and P1(0B, 0B)P2(0B, 0B). Establishing whether a homoeologue is homozygous null or homozygous for a shared allele can be difficult. Examining the consistency of the allele doses inferred between the parents and progeny may be helpful, as long as the alleles have similar amplification efficiencies. For example, in an AAxAa cross, the RV of the mother should be approximately twice that of the father (2:1), and these RVs should be in agreement with the two RCs of the progeny (AA, Aa). If the RVs have a 4:3 relative value, this could indicate the presence of an additional homozygous set of allele pairs that had not been initially identified (i.e., AABB.XAABB.).在某些情况下,等位基因的数量可能超过倍性水平。在这种情况下,应检查重复或放大效率差异的指示。
例如:微1 - UFFxa16H07:基于共享等位基因(262HK,266HK,269HK),我们可以将等位基因对的“假期”和Korona分为三组同源性(表3.).第四组同源染色体取决于279H的分析。该等位基因显示了两个的RC,因此通过偏析剂量。然而,高剂量的279的存在不与不存在任何其它等位基因的关联。因此,逻辑假设是279同源等位基因是无效等位基因和279是在另一个homoeologue纯合子。然而,这一结论是有问题的,因为它导致在九个等位基因剂量是在单亲(“假日”) - 六等位基因剂量从三个等位基因对和三个剂量的279,但只有八剂量的同种八倍体是可能的(当等位基因是不重复)。另一种方法是假定扩增效率(279A和279B)不同的两个同源等位基因的存在。在扩增效率导致低的平均RC值(10),用于一个等位基因和另一个等位基因一个高的平均RC值(15)的降低(表3.).因为它们是同源的,所以所有后代中都有一个。进一步证实这一假设的证据是,“假日”品种279的RV远高于后代的最高RC平均值;如果第一个假设是正确的,“假日”RV值应与子代的最高RC平均值相似。在另一种解释中,“Holiday”和“Korona”中的总剂量为每位家长产生8剂。因此,279等位基因对279aH-279bH与最后剩下的“Korona”等位基因对273K-287K结合。
虚拟参考等位基因:增加吞吐量和功率
原则
上面例子中的参考等位基因是基于一个单一的等位基因。使用基于单等位基因的文献可能是一个费力的过程,特别是如果没有发现不被结巴带混淆的单形、纯合子位点。对于大多数微卫星来说,通过使用虚拟参考等位基因可以大大提高通量,虚拟参考等位基因是基于单个引物对的全部或部分等位基因峰值面积的平均值。自动计算平均值和比率值,使快速剂量评估成为可能。因为它基于多个等位基因,虚拟参考等位基因减少了单个等位基因面积估计不准确的风险,并减少了口吃对单个峰的影响。矛盾的是,随着倍性水平的增加,这使得剂量估计更好,因为平均值是基于大量的观察。
检查性能
检查虚拟参考的性能最好是通过检查已知剂量等位基因产生的RC分布的窄性来实现的。如果它们的宽度很窄,设计的虚拟参考是足够的。如果它们的范围很广,对数据的解释可能会受到阻碍。在这种情况下,进一步优化虚拟参考通常可以通过考虑假定的干扰因素来实现,如分离空等位基因和等位基因之间的巨大放大差异。干扰等位基因可以通过从平均值中排除可疑等位基因,然后检查RC分布的窄性来识别。
等位基因扩增效率差异的影响
PCR扩增效率随等位基因大小的增加而降低[10.].此外,引物位点中的突变的存在可以对扩增效率具有很大的影响。因此,同源单纯克等位基因可能在峰面积(这里,两个至三倍的峰值差异有多倍的差异,如表所示3.).这可能对剂量估计的可靠性产生重大影响。举例来说,假设一对引物在一个四倍体定位群体上扩增了四个不同的等位基因。八个亲代等位基因中有七个扩增的效率相同,而第八个等位基因扩增的效率要高出三倍。如果这种差异放大不占,一半的四倍体的平均后代代表六个“放大单元”((3×1)+(1×3)),而另一半代表四个单元(4×1)。average-based参考个人携带有效等位基因将是50%高于个人缺乏这种等位基因。参考文献中的这种可变性将极大地影响比率簇的宽度,并可能导致错误的解释。在太宽的RCs的情况下,使用具有相似扩增效率的等位基因子集通常会大大提高参考等位基因的宽度。如果这种方法仍然不够,如果所涉及的标记是非常有趣的,可以考虑一种更复杂但也更费力的方法:引入等位基因特异性倍增器。这种乘数是根据它们与已知剂量的初始参考等位基因的比率计算的。然后,在计算虚拟参考时,可以使用这个比率的平均值的倒数作为乘数。
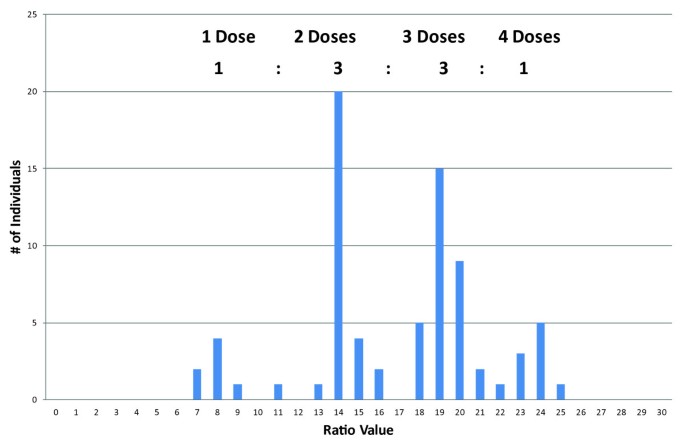
例如:微型卫星2 - UAFv7500:引物对UAFv7500扩增六种不同的等位基因,其中四种按存在/不存在进行分离(表2和3.附加文件3.).定性分析揭示了三种:1分离等位基因(330 k,333 k,336h)和一个3:1分离等位基因(345hk)。345HK是330 k和336h的等位基因,表明它们属于同一同源套(336h-345h,330 k-345 k)。没有等位基因对333 K.使用单纯克等位基因作为参考,所有的等位基因总是通过剂量分离。这使得不可能使用单峰作为所有样本的参考。因此,必须构建基于平均的虚拟参考等位基因。检查单纯克等位基因的RCS显示窄分布(附加文件3.),因此不需要进一步优化参考等位基因。接下来,345HK显示了1:2:1的RC偏析,证实了3:1偏析的定性分析。342HK和348HK始终存在,表明每个位点至少存在一个纯合位点。342HK显示了4个有规律增加的RCs(约为2×、3×和4× 6的比例)和1:3:3:1的分离(表1)3., 数字2).在异源多倍体中,这种分离模式表明三杂合性,至少有三对杂合等位基因参与至少两个同源基因。等位基因348HK在RCs中分离为1:2:1,显示双杂合度。当样品被过滤为342HK的最高RC值时,单剂量333 K等位基因就消失了,而348HK处于其最低RC值。相比之下,当样品被过滤到最低RC值342HK时,333 K始终存在,348HK具有最高RC值。342HK与333 K (333 K-342 K)完全互补,另外两个分离的342HK等位基因与两个分离的348HK等位基因都互补。接下来,纯合子等位基因必须分配给他们的父母。为此,本文探讨了父母的比例值。对于342HK,这些比率表明在“假日”中有三剂,在“Korona”中有两剂3.).因为两个等位基因342“科罗纳”中的一个是已知的分离由于其互补性以333 K,第二342ķ等位基因也有偏析。纯合基因座342和第三偏析342等位基因因此必须从“假日”母体来源。两个所涉及的同源染色体的推导等位基因组成是这样(342H-342H,333 K-342 K),(342H-348H,342 K-348 K)。这样就完成了342HK分析。
在这一点上,纯合子348等位基因尚未被分配,并假定存在于4thhomoeologue。因为没有其他未分配的等位基因可用,双亲可能是348纯合子,或者有一个可能是纯合子零。亲代比率值有助于区分这两种选择。它们表明在每个父母中存在三种剂量(表3.),而仅基于后代的分离模式分配每个父母的一个隔离等位基因。这表明两个父母在第四个同源中的348个纯合。
IV。测绘和验证
具有分配等位基因同源套,我们现在就可以开始映射。所述映射步骤用于5个用途:验证并完成等位基因对的分配,以从多个基因座组的等位基因对一起为不同的部分同源子的基因组,以确定标记顺序和遗传距离,以建立一个集成的地图,并确定父母的单倍型。映射步骤被划分成四个阶段类似于由Barrett等人描述的地图整合过程。[19.]。MADCE衍生等位基因得分和同源基因分配的最终验证通过生成连锁图来完成。
a.建立先天整合位点
在之前的等位基因配置步骤(III)中鉴定的单亲本等位基因集被合并成双亲本等位基因集,然后翻译成整合位点。在我们的研究中,这些位点在JoinMap (Kyazma B.V., Wageningen, NL)软件中被定义为交叉授粉者(CP)型位点。由于两个原因,整合位点在映射之前被构建。首先,这可以作为评分错误的额外检查(例如,一个位点有三个等位基因的个体)。第二,方便数据管理;首先生成集成的位点,然后在需要的时候使用JoinMap 4.1将它们转换回单亲位点,这比在后面的阶段集成位点要高效得多。
湾创建单独的父母地图:额外的错误检查
为了创建单独的亲本地图,集成的位点在JoinMap 4.1中被自动转换为单亲位点。生成映射后,将执行一些标准错误检查,例如与参考映射进行比较(对于strawberry FvxFb [20.])检查扭曲的基因座和基因座,产生高张力。最后,标记基因型在Excel中有条件地格式化,使用JoinMap的相位信息,这使得能够容易地识别发现双交叉事件。
创建单独的亲本图:验证整合和同源赋值
接下来,计算一个新的分组并绘制地图。单个亲本图谱根据等位基因共享相互匹配,可以用来验证整合位点,并从以前的未整合位点创建新的位点,如Barrett等人所描述的[19.].然后将匹配的父母映射对在八倍体的情况下随机分配同源学信(A,B,C或D)。
天。创建集成贴图:最终错误检查
从上一步的升级数据加载到JoinMap中,并生成最终集成贴图。由JoinMap生成的地图和链接相位信息与来自步骤III的等位基因信息组合以建立父母单倍型。
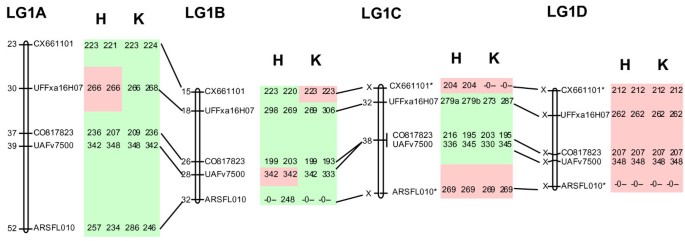
例子:5对引物样本的最终标记分数(见表)1)以供在JoinMap中使用,请参阅附加文件1那2那3.那4.那5..映射结果和最终的单倍型如图所示3..对于所有同种主因来说,标记顺序是相同的,尽管一些包含较少或没有隔离基因座。标记数据的图形分析显示,在短距离上没有任何双重重组事件,表明微卫星数据的定量解释成功,因为它导致明确的数据。因此,Madce导致高度稳健的标记基因型。
五,为纯合基因座单倍型父母完成
如果在第三步中发现纯合子位点,则手动将位点添加到单倍型信息中,使最终的单倍型完整。它们的遗传位置是根据同源基因组标记之间的距离推断或插入的。这些位置在生物学上不应该被认为是“正确的”,但它们可以作为一个指导方针,使纯合子延伸的可视化。
例子:在目前的研究中,其中一个纯合子(1D)显示为完全纯合子(图1)3.).
系谱品种的MADCE程序
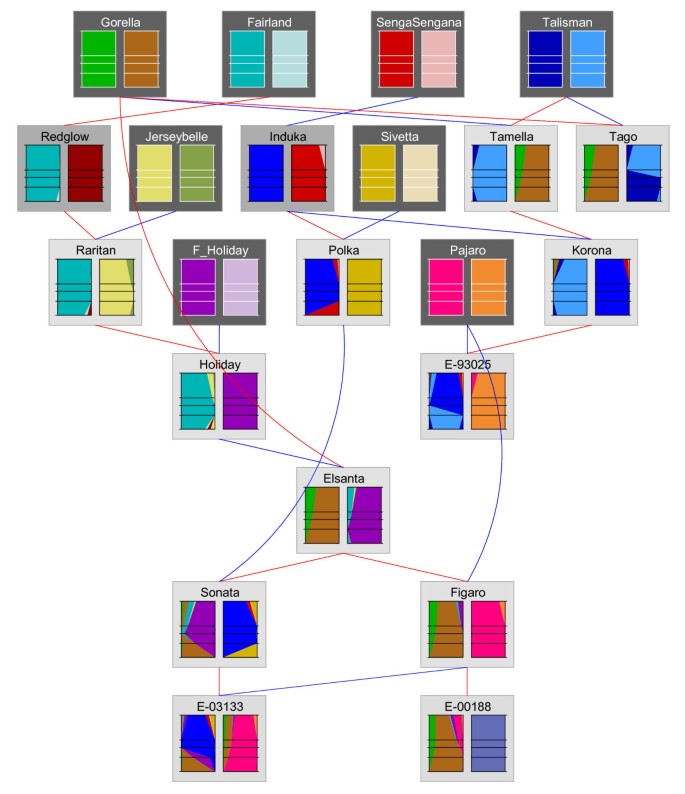
MADCE作图研究程序不直接适用于纯种品种,因为它们具有更高的遗传复杂性(例如,每个位点的等位基因数量更多)和分离数据的可用性非常有限(每个个体的后代很少)在此,我们将描述适用于纯种品种的MADCE程序,并说明微卫星CX661101的该程序(表1)4.)使用一组21个纯种栽培种(图4.).
一、等位基因剂量估算需要实际参考
我们推荐使用虚拟的、基于平均的参考等位基因,因为符合参考要求的单等位基因在大型育种种质中非常罕见。比率值(RV)的计算、频率分布和剂量分配与绘制人口图相同。当RV落在两个不同的簇之间时,可以临时分配最可能的剂量,并在第二步进行验证。空等位基因的出现更有可能在广泛的种质中发生。无效等位基因剂量是通过从总剂量中减去估计的总剂量来确定的(即,与倍性水平相同)。一个单独的列为空等位基因剂量添加到电子表格(表4.).这些无效等位基因可被分配给同源染色体以类似于常规等位基因分配的方式,通过将在下一节中描述的方法。对于纯种品种,无效等位基因不能对先验补偿。等位基因效率的差异只能用乘数时,他们很清楚现在和可量化的补偿。这些缺点使用量估计有些在这些缺陷都存在的情况下不太可靠,但也不是没有可能。
例子:对于微卫星CX661101,创建了一个基于平均值的参考。不同的rv簇的存在表明存在不同的剂量水平和虚拟参考的良好性能。RV簇间的规则距离表明等位基因之间的扩增效率没有很大的差异(表)4.).对于等位基因204,我们观察了1到2个剂量的RCs,分别在RVs 5和10附近。等位基因212始终存在,并显示两种不同的RCs(~8和~16),再次提示单剂量和双剂量。223等位基因的房车数量从4到23不等,说明情况更为复杂。RCs似乎在大约5-6个步骤中增加和减少(尽管每个集群的观测数量相当低)。因此,这个步长很可能代表单一剂量值。由此我们推断出每个品种的剂量水平,从1-4剂量不等。因为我们在“假日”和“科罗娜”中绘制了这颗微卫星的地图3.),我们可以用这些品种作为检验,以正确估计剂量,我们发现结果是一致的。现在,我们可以推断无效等位基因剂量,这是我们从地图研究中知道的。扩增等位基因的总剂量从6到8个不等(见表)4.因此,因此,无效等位基因的总剂量范围从两个('korona')到零。毫不奇怪,212 k和224 k的比率值是所有品种和选择中的比率值(考虑到等位基因剂量后)。这是由于存在两个无效等位基因,这导致了相对较低的虚拟参考值,因此是高比率值。
2将等位基因分配给同种质
a.程序:通过同源等位基因总剂量分配
二体遗传物种的一个非常简单的规则是,在任何给定的个体中,属于同一同源基因的所有等位基因的总剂量需要恰好为2(在这个计算中也使用了零等位基因剂量)。如果一个相同大小的等位基因被多个同源基因共享,那么总剂量需要是涉及的同源基因数量的两倍。这些规则是一个很好的起点,在这种情况下,从以前的地图研究没有事先的等位基因分配可用。它们对于大数据集和频率为> 5%的等位基因特别有用。我们通过选择第一个等位基因开始等位基因分配过程,然后消除不可能在同一同源基因上的等位基因,因为它们的联合剂量之和超过两个。最容易从剂量从0到2不等的高频等位基因开始。合计剂量从不超过2的等位基因可能彼此是等位基因。从另一个未分配的等位基因开始重复分析,以确定其他同源序列的组成。
例子:我们假设除了同系物的命名外,从映射群体中没有先验的等位基因分配信息。我们从204等位基因开始分析。该等位基因因存在/不存在而分离,当存在时,单剂量或双剂量发生(见表)4.).假设204发生在单个同种型上,我们可以针对每个基因型,将其剂量与每个其他假设的特异性等位基因中的每一个的那些。导致任何人中高于两个的等位基因不属于相同的同源疗法。除了210和NULL等位基因外,所有等位基因都是这种情况。总结这三个等位基因的剂量导致所有基因型恰好两种剂量,表明这种同源性的等位基因分配是完整的。这组等位基因根据映射研究的结果分配了同种字母C.
我们继续对等位基因212进行类似的分析。总剂量超过两个结合所有等位基因,除了218。这种等位基因只在212单次使用时才存在,所以212和218很可能是同源的。212和218的总剂量总和正好是所有品种的2,因此,对这个同源物的分配是完成的。这一组等位基因被指定为同源字母D基于测绘研究的结果。
我们观察到223等位基因总是存在,并且在四次剂量下发生。因此,它必须至少存在于两个同系词上。因为已经分配了两个同系词,另外两个必须有223个同系词(A&B)。因此,其余的等位基因也必然存在于这两个同源基因上。在这些病例中,只有225例和224例发生在双重剂量下。双剂量(最大)等位基因只存在于一个同源基因上的假设消除了221和225在同一个同源基因上的可能性,因为在选择E-00188中,它们加起来是3个剂量。在“Holiday”中,等位基因221存在于同源基因A上,所以我们将225分配给同源基因b。对于双剂量等位基因224(“波尔卡”),不能消除,因为“波尔卡”只携带共享的等位基因223。其余的等位基因(220,224,229)不能通过剂量分配。
b.程序:通过传输进行转让
为了帮助完成或验证任务,我们可以考虑从父母到后代的等位基因传播。对于在同一同源性上的等位基因,应该仅传播一个等位基因。同时(非)等位基因的传输表明它们属于不同的同源疗法。通过跟踪大型血统的等位基因的传播,可以建立哪个等位基因绝对不属于相同的同源性,并且哪些等位基因可能属于同一同源性。以上原则不适用于同源性之间共享的等位基因。
例子:使用图中所示的谱系4.结合表中的数据4.,我们观察到等位基因224和225都存在于“戈罗拉”中,并且这些等位基因都不是从“戈罗拉”到'tamella'中的。这表明这两种等位基因存在于不同的同源性上。从“戈罗拉”到'elsanta'的传输是证实的,其中两者都经过传播,也可以来自'假期',这是'elsanta'的另一个父母。因为225被分配给同种型B,我们现在可以将224分配给同种型A.同样,我们发现221和220都存在于“假期”中,但也没有传输到'Elsanta'。这意味着220对221不是同源的,因此必须在同种型B上。这只留下仅229个未分配;该等位基因在“Sivetta”以及225个同源型B中存在。因为这两个等位基因中都没有传递给“圆头”,229必须存在于同种型A上。我们获得了两种等位基因组:221-223-224-229用于同种型A和220-223-225的同种型B.
一旦所有的等位基因已被分配,我们可以基因型的223等位基因A和B同源染色体。例如,“波尔卡”具有以双倍剂量223和224。因为只对A基因组发生224,“波尔卡”必须是纯合的224上的A homoeologue并因此必须是纯合的223在B homoeologue。“Elsanta”具有223以双倍剂量和224和225在单次剂量。等位基因224和225属于不同的同源染色体。因此,223的双倍剂量必须来自两个不同的同源染色体(223A-224A,223B-225B)。最后,我们确定的任务是否与来自定位群体的“假日”和“科罗纳”的一致。
其他四颗微型卫星的结果:对另外五颗微型卫星进行了类似的分析(见表)1).对于每个,表格中所产生的等位基因集的摘要5..其中一个微卫星(CO817823)在少数品种的剂量估算中遇到困难,无法完成对该微卫星的分析。12个等位基因在多个剂量下出现(7个2×, 4个3×, 1个4×),所有这些都可以分配到特定的同源基因。
剂量估算完成后,我们可以继续识别属于同一同源等位基因的等位基因对。这最好通过结合两种方法来实现,一种基于等位基因剂量,另一种基于等位基因传播。
3使用FlexQTL™进行阶段确定和分配验证
现在,数据被用来产生连锁相和身份通过血统使用FlexQTL™软件[(IBD)估计17.].FlexQTL™还监控连续标记之间的观察和预期单,双重组事件的数量,使这个软件一个简单的工具,使用它可以快速有效分配和检查方案(错误地分配等位基因导致整个表观重组事件的数量增加谱系)。显然,正确的相位估计只能在创始人有足够的后代情况下,作出。
例子:数字4.介绍了21个研究品种的单倍型。数字5.展示了这些品种的子集对血统的个体等位基因的流动。'korona'的谱系衍生的单倍型与由绘图人口决定的人一致(图3.和5.).这也为“假日”品种的情况下(未示出数据),从而提供MADCE是否适合纯种种质在异源多倍体作物基因分型的概念证明。
讨论
MADCE的方法论方面
过去十年已经研究了各种等位基因剂量估算技术。所采用的方法包括对强度的视觉评估[9.那21.那22.],峰值高度[10.]及峰面积比(MAC-PR) [8.那11.].Mac-Pr [8.]很可能是最准确的这些由于其使用内部参考,其允许样品之间PCR效率变化补偿的。MADCE改进了这种方法具有虚拟(平均为基础)参考等位基因的新的概念和与用于异源多倍体基因型的全部等位基因构造的评估数据分析管道延伸它。MADCE的鲁棒性是有保证的感谢的监测的剂量信息的准确性和一致性几个内部反馈控制的存在。
虚拟参考等位基因
剂量估计的产量和可靠性严重依赖于参考等位基因的种类和质量和微卫星的“整体”质量(陷阱,峰形,等位基因尺寸接近)。建议使用“虚拟”平均峰面积的参考资料,因为它们是强大的映射研究和绑架品种,是广泛适用和增加吞吐量。虚拟引用更加强劲,因为它们对单个峰和陷阱的质量不太敏感。虚拟引用增加吞吐量,因为可以使用单个完全信息的参考而不是一系列部分信息的参考,这需要更多的数据处理。通过测量RC簇的宽度,可以容易地监控虚拟参考的性能,并且可以容易地检测到另外的性能提高措施。Madce对虚拟参考的使用提供了半自动分析的基础,包括用于剂量估计的专用软件的开发[12.,从而使未来的吞吐量进一步增加。
同种型扩增
在所有聚吡喃啉中,引物对可以潜在地扩增来自一个或全部同源疗法的产物,因此可以产生大量具有复杂条带和分离模式的等位基因。这可以被视为缺点,因为它妨碍了快速且易于数据解释,特别是在对特定同源性的等位基因分配中。或者,它可以看作是一个优势,因为它允许检查宏观同步。另外,相对于使用具有同种型引物的方法需要更少的引物组合。
同源扩增的主要缺点是多个亚基因组之间可能出现完全相同大小的等位基因。这些共享等位基因导致了异源多倍体中大多数复杂的分离模式。共享等位基因可能表明同源基因之间的微卫星重复数守恒,但也可能是通过不同事件独立产生的(同源性)[23.- - - - - -25.].在我们的研究中,其中一个共享等位基因显示了扩增效率的差异,这一事实证实了同源性3.),表明在引物退火位点存在indele或SNPs等多态性,这些类型的多态性的突变率低于重复位点。利用MADCE,可以通过将所有等位基因分解成简单的孟德尔分离模式,并将这些等位基因分配到各自的亚基因组中,尽管它们的大小相似。
MADCE提高映射效率
在我们的八倍体草莓为例,5个微引物组合扩增的35点不同的等位基因基于大小,和这些展出存在/不存在的偏析25。使用MADCE,我们发现30个非常翔实的分离式等位基因,而传统的方法将已经取得了19只完全信息和六个部分信息等位基因。是存在于所有的后代,从而忽视或使用传统方法时,会考虑纯合子十个等位基因,六人发现剂量分离。另外,这六个3的信息内容:1分离标记物增加,因为MADCE允许纯合和杂后代的辨别,从而信息减数分裂的比例从25%增加一倍至50%。通过识别匹配斥力等位基因,所有四个基因型类可以区分开来,因此增加了减数分裂信息内容100%。此外,一个3:这些微卫星中的一个的1偏析等位基因可以被正确地分配给不同的同源染色体。如果该等位基因已被用作桥梁标记为亲本的地图整合,根据由Ripol等人定义的协议。[6.,这将导致错误的整合,将同源染色体而不是同源染色体连接在一起。
Madce改进映射质量
同源(排斥)等位基因的识别和在定位之前为两个等位基因创建一个单一的位点以前已经被使用[14.].这减少了冗余基因座的数量,其中一些可能看起来像由于得分或缺少分数的不一致而在略微不同的地图位置时看起来不同的基因座。Madce的错误删除步骤改善了最终地图质量。由于与Barrett等人所述的地图集成方法,通过适量的等位基因分配来防止由于共享等位基因而导致的父母级映射。[19.].MADCE的测绘父母的完全等位基因配置允许容易地鉴定完全纯合的基因组中的区域,如连锁组1D所示。关于大型纯合段存在的信息可以在包围植物种类中制作联系地图时揭示间隙和过量的部分连锁群体的发生。因此,该信息可以通过测试许多额外的底漆组合来防止徒劳的努力来填补这些间隙或完成这些联动组。
使pedigree-based分析
在Allopolyploids中,尽管有许多经济上重要的作物,如小麦和棉花,但协会研究仍处于初期的婴儿。尽管一些这些植物具有良好分歧的子基因组[26.- - - - - -28.[通过近亲繁殖,传播,使它们变得遗传更复杂。在过去十年中,一直从双亲子QTL映射研究转变为优选地通过LD映射研究了非结构化植物种质的研究[29.].此外,战略已经通过基于谱系分析开发的基因结构养殖种质(PBA)的方法[17.那30.].这两种方法都提供了异源多倍体的研究视角。
PBA是一种多家系、多品种、多选择的QTL定位方法。它允许利用已知的系谱关系,允许相对较低的标记密度[30.].每5cm有一个信息丰富的微卫星标记就足以用于PBA。PBA提供了对育种种质遗传结构的理解,并通过不需要熟悉相关性状的情况下精确定位处于高选择压力下的区域,发现育种家的特征。该方法的概念证明和统计分析已经通过EU-HiDRAS项目在苹果进行[31].从那时起,PBA已经接受了在基因研究上二倍体作物蔷薇科[32].PBA需要先进的统计软件包,如FlexQTL™[17.]及Pedimap [18.].MADCE能够提供该软件所需的数据,因为它可以用来推断给定植物中给定微卫星的所有同源基因的完整等位基因配置,包括空等位基因和纯合子区域。因此,MADCE首次使PBA在复杂的异源多倍体中的表现成为可能。通过对21个系谱品种的测试,我们能够跟踪系谱上标记等位基因的流动,从而为PBA的产生提供了一个前体。目前,MADCE已被广泛应用于草莓育种种质的基因分型。
能力区分异常的多态继承
关于多倍体的特定植物种类的知识是至关重要的,因为继承的模式决定什么类型的方法和软件都可以在基因研究中使用。传统的方法来评估套数的类型是基于减数分裂过程中染色体配对行为的细胞遗传学研究[33以及种族隔离问题。细胞遗传学方法不能提供绝对的确定性,因为在异源多倍体减数分裂早期也可以观察到多价[34].在偏析的研究中,倍性类型是基于我区别)分离比率为双工等位基因,ⅱ)后代基因型的只能因双减速出现,以及iii)通过连锁的基因座的该标记物的比例发生在地耦合和排斥阶段[5.].这些方法都不能为倍性类型提供绝对的确定性。除去比例不能确定,因为除非使用非常大的人群,否则难以区分彼此大于3:1的分离比。另外,偏析失真的发生可能会干扰这些分析。双重减少不可靠,因为由于表型错误,基因分型误差,屈服和DNA混合物,可以评分假双减少。这些类型的错误通常以低频发生,但对于实际双重减少事件也是如此[35].最后,通过联动比率的适应性导致了混合多膜的引人不正确的栽培草莓和肤浅的疾病[13.那36].而且,关于遗传模式的结论只能在连锁群建立之后才能得出,而二倍体方法学已经为时过晚。最后,该方法使用位点之间的耦合和排斥相连接,而不是简单地使用位点内的排斥等位基因。因此,随着距离的增加,确定减数分裂分离模式的可靠性降低。相比之下,MADCE为建立型多倍体提供了一种非常有效的方法,即通过检测单个位点内等位基因对的存在,在定位之前先确认二体遗传。如果在每一个不同的染色体或一组足够大的随机位点上都发现了这样的配对,那么就可以认为是完全功能的二体分裂,也就是异源多倍体。
MADCE和新的高吞吐基因组工具
下一代测序技术的负担能力和吞吐量的快速进展使高吞吐量标记系统的开发产生了发展。MicroTaWellites以外的标记平台,包括基于阵列的SNP检测技术[37],荧光单核苷酸多态性探针[12.那38]和实时定量PCR [39],也可用于剂量估计。这些技术良好,准确且经常高吞吐量,而微卫星的使用相对昂贵,劳动密集型。因此,这些平台可能会在不久的将来超越SSR方法。但是,由于他们的发展是非常复杂的,因此不经常可用的SNP阵列并不经常可用。因为亚基因组高度分化,因此所有多元素中的大多数SNP可能在同源素之间具有多态性,但不在同源层内。另外,对于在同种质中的相对较少的SNP,因此可以在测定中使用,大多数将具有来自其他亚基因组的干扰。为了说明这一点,想象一下,试验需要如何执行,以将来自SNP测定的信号簇分离在Allo-Octoplod中(Aattttttttttttttttttttttttttttttttttttttttttttttttttt。例如,必须通过使用相邻的亚基因组特异性SNP来处理这种干扰以使分析细胞组织组特异性。因此,SNP开发所需的投资比二倍体的多级多倍体更陡峭。继续使用微卫星的其他论据是它们更适合于由于其高转移性和多态性水平而在遗传上不同的种质中的应用。 This high level of polymorphism also makes them more likely to tag a haplotype (or trait) than random SNPs. Polyploidy hampers discovery of SNP haplotypes. MADCE could help in the discovery of haplotype tagging SNPs through their association with well-defined microsatellite alleles. Furthermore, information on homozygosity (for cross-pollinators) generated by MADCE enables the selection of the best lines for SNP development by complementing regions that are homozygous and heterozygous between these lines. Based on these perspectives, MADCE currently supports the efforts of the international RosBREED-Illumina consortium in testing SNP development strategies by helping to identify the most appropriate germplasm for use in SNP discovery.
结论
本文提出的微卫星数据定量解释的MADCE方法为复杂异源多倍体植物的遗传分析提供了一种新的工具。MADCE通过处理亚基因组、空等位基因和纯合位点之间的共享等位基因,增强了异源多倍体的基因分型。这可以用来建立任何异源多倍体植株的完整等位基因配置。MADCE通过提高分子标记连锁图的生成效率和系谱育种种质的充分信息的基因分型,促进了异源多倍体的遗传研究。因此,MADCE使得为二倍体系统设计的统计和遗传软件能够用于异源多倍体。MADCE还可用于复杂多倍体的SNP检测和SNP阵列开发。
方法
植物材料
为了建造分子标记键的地图,使用来自草莓品种“假期”(H)和“Korona”(K)之间的交叉之间的82个幼苗。对于血统分析,使用了一组21种品种和繁殖线,包括“假期”和“Korona”(图4.).叶片取材于美国俄勒冈州科瓦利斯的国家克隆种质资源库。
DNA孤立
根据Fulton等人的修饰版本提取基因组DNA。[40] mini-prep协议。短暂地,年轻的、折叠的叶子被收割了。叶子被冻干并在2ml试管中磨成粉末。在该管中加入700 μL温(65℃)2% CTAB缓冲液,旋涡混合孵育10 min,然后加入700 μL氯仿异戊醇(24:1)。将混合物在10,000 × g室温下离心2 min,然后将600 μL的上相转移到新鲜管中。加入480 μL异丙醇,混匀后室温10000 × g离心2 min。弃上清,用500 μL 70%乙醇洗涤2 min, 10000 × g离心2 min。移液弃上清,400 μL TE重悬。加入LiCl (135 μL, 8 M)去除RNA和多糖,-20℃孵育30 min。孵育后,室温10000 × g离心2 min,上清转移到新鲜管中。加入320 μL异丙醇,-20℃孵育30 min, 10000 × g离心5 min,弃上清。 The pellet was then washed and centrifuged twice with 500 μL of ethanol (70%), and then the dried pellet was dissolved in 50 μL of TE.
微卫星
起源
6个已知位于单个连锁群(LG1)上的微卫星引物组合取自各种来源(见表1)1).5个标记同时用于作图和家系分析,而标记PScontig944仅用于家系分析。
PCR.
在正向引物上用间接荧光标记与间接荧光标记进行PCR,其在正向引物上与通用荧光标记的引物(6-FAM,Hex或ROX)匹配的前底底底尾序列(AacaggtatgactgactgaTGA)进行。该方法从Schuelke中描述的协议调整了[41].根据Brownstein等人的研究方案,反向引物添加了GTTT尾部以减少口吃[42].PCR混合物由1X Goldstar PCR缓冲液、0.5 μM无标记带尾正向引物、2 μM无标记反向引物、2 μM有尾通用引物、0.3 U GoldstarTaq聚合酶(Eurogentec, Netherlands)和10 ng DNA组成,总反应体积为20 μL。PCR条件为94°C 3 min, 94°C 30 s, 50°C 30 s, 72°C 2 min, 35个循环,72°C 10 min。
微卫星数据准备
根据琼脂糖凝胶中观察到的扩增子强度,PCR产物被稀释(平均大约300倍),以防止荧光强度水平超过(或低于)检测水平,从而妨碍剂量估计。使用ABI毛细管自动测序平台(ABI 3730, Perkin Elmer Biosystems, Foster City, California)分离和检测荧光标记的扩增子。使用Genemapper 4.0软件分析ABI平台的输出。确定了与等位基因对应的峰值,并定义了它们的bin range,即等位基因大小的窗口,这些窗口被认为代表一个等位基因。接下来,对于每个样本,软件自动识别等位基因(峰值)的存在,它们的高度和峰值下的面积。人工检查正确的等位基因检测,必要时进行调整。每个个体的等位基因数据(大小,面积)被转移到一个Excel表格(见附加文件)1那2那3.那4.那5.).Excel表格的格式为每行显示每个个体的面积数据,每列代表一个不同的等位基因。这些表格随后用于进一步的定性和定量分析,如结果部分所述。
施工联系地图
使用JoinMap分别为每个父级创建链接图®4.1(新西兰瓦赫宁根Kyazma B.V.)和Kosambi作图函数。使用最小LOD阈值1、重组分数阈值0.45、波动值1和跳跃阈值5确定标记位置。使用单独亲本连锁图的比较来创建和验证整合基因座并消除错误。在此数据准备之后定量步骤/升级,使用与单独亲本图谱相同的JoinMap设置创建整合图谱。使用分离纯合位点图谱距离的内插和外推估计纯合位点的位置。首先使用软件包Mapchart创建连锁图谱[43]后来在Microsoft PowerPoint完成。
谱系分析
从图中选出21个品种4.使用六个微卫星在表格中进行基因分型1.根据本文提出的,根据微卫星等位基因剂量和配置建立(MADCE)方法,建立了等位基因的剂量和构型。接下来,使用IBD估计和根据软件包FlexQTL™的等位基因的最可能链接阶段分析了这些品种的遗传。17.].为了图形化地表示谱系中等位基因的流动,IBD估计被载入Pedimap [18.].
参考文献
- 1。
被子植物基因组大小的进化。acta botanica sinica(云南植物学报). 2003,30(4):593 - 598。10.3732 / ajb.90.11.1596。
- 2。
开花植物的多倍体和新颖性。王志强,王志强,王志强,等。10.1086/284115。
- 3.
Gaut BS,Doebley JF:玉米片段异源四倍体起源的DNA序列证据。美国国家科学院学报,1997,94(13):6809-6814.10.1073/pnas.94.13.6809。
- 4.
Soltis DE,Soltis PS:分子数据和多倍体的动态性质。Crit Rev工厂Sci。1993, 12 (3): 243-273.
- 5。
吴熊,烧焦W,鼻子Me,Tew T1,Moore pH,坦克斯利SD:使用单剂量限制碎片检测和估计多倍体中的联动。Al Appl Genet。1992,83(3):294-300。
- 6。
Ripol MI, Churchill GA, da Silva JAG, Sorrells M:同源多倍体遗传图谱的统计方面。基因。1999,235(1-2):31-41。10.1016 / s0378 - 1119(99) 00218 - 8。
- 7.
D'Hoop BB,Paulo MJ,Mank Ra,Van Eeck HJ,Van Eeuwijk Fa:土豆(Solanum Tuberosum L.)质量性状的关联映射。Euphytica。2008,161(1-2):47-60。10.1007 / S10681-007-9565-5。
- 8.
利用微卫星DNA等位基因计数峰比(MAC-PR)方法确定多倍体的等位基因配置。应用计算机学报,2004,109(2):402-408。
- 9.
利用SSR标记研究紫花苜蓿(Medicago sativa)品种间的遗传多样性。应用计算机学报。2005,111(7):1420-1429。10.1007 / s00122 - 005 - 0074 - 4。
- 10。
Landergott U,Naciri Y,Schneller JJ,Holderegger R:四倍体牙科胸腺术中高度可变微卫星的等位基因配置和多元组遗传。Al Appl Genet。2006,113(3):453-465。10.1007 / S00122-006-0310-6。
- 11.
Nybom H,Esselink GD,Werlemark G,Vosman B:微卫星DNA标记遗传表明多倍体和半性犬玫瑰中两个高度同源的基因组之间优先配对,Rosa L.Sect.Caninae DC.遗传.2004,92(3):139-150.10.1038/sj.hdy.6800332。
- 12.
voorrips Re,Gort G,Vosman B:使用混合模型从双位标记数据中呼出四倍体物种的基因型。BMC生物信息学。2011,12(1):172-10.1186 / 1471-2105-12-172。
- 13。
Barrot Rousseau-Gueutin M, Lerceteau-Kohler E, L,萨金特DJ, Monfort涉嫌,辛普森D,阿鲁P, Guerin G, Denoyes-Rothan B:比较遗传octoploid和二倍体草莓属物种之间的映射显示高水平的同线性基因与栽培octoploid草莓的二体的行为。中国生物医学工程学报。2008,29(4):457 - 461。10.1534 / genetics.107.083840。
- 14.
Sargent DJ, Fernandez-Fernandez F, ruizr - roja JJ, Sutherland BG, Passey A, Whitehouse AB, Simpson DW:栽培草莓(Fragaria × ananassa)的遗传连锁图谱及其与Fragaria二倍体参考图谱的比较。分子育种,2009,24(3):293-303。10.1007 / s11032 - 009 - 9292 - 9。
- 15.
八倍体草莓病毒MA的SSR和RFLP连锁图谱Fragaria×ananassa.).植物、动物和微生物基因组×会议:2002年;加利福尼亚州圣地亚哥,2002:660。
- 16。
Sargent D,Passey T,Šurmanovskin,洛佩斯·赫罗纳e,kuchta p,davik j,哈里森r,passey a,whitehouse a,Simpson d:栽培草莓的微卫星连锁地图Fragaria×ananassa.建议在可能受到繁殖和选择的基因组中大量纯合子区域。标记理论和应用的遗传学。2012,DOI 10.1007 / S00122-011-1782-6
- 17。
Bink Mcam,Boer MP,Ter Braak CJF,Jansen J,Voorrips Re,De Weg WEV:鸽舍种群中复杂性状的贝叶斯分析。Euphytica。2008,161(1-2):85-96。10.1007 / S10681-007-9516-1。
- 18.
Pedimap:用于可视化遗传和表型数据的软件。[http://www.plantbreeding.wur.nl/uk/software_pedimap.html.].
- 19.
Barrett B, Griffiths A, Schreiber M, Ellison N, Mercer C, Bouton J, Ong B, Forster J, Sawbridge T, Spangenberg G,等:白色三叶草的微卫星地图。应用计算机学报,2004,109(3):596-608。
- 20。
Sargent DJ,Clarke J,Simpson DW,Tobutt KR,Arus P,Monfort A,Vilanova S,Denoyes-Rothan B,Rousseau M,Folta Km,等:一个增强的二倍体Fragaria微卫星地图。Al Appl Genet。2006,112(7):1349-1359。10.1007 / s00122-006-0237-y。
- 21。
Truong C、Palme AE、Felber F、Naciri Graven Y:四倍体桦树(Betula pubescens ssp tortuosa)微卫星标记的分离和鉴定。分子生态学笔记。2005, 5 (1): 96-98. 10.1111/j.1471-8286.2004.00848.x。
- 22。
Wickramasinghe Ham,Miura h:小麦Wx等位基因的基因剂量效应及其对胚乳淀粉糖合成的相互作用。Euphytica。2003,132(3):303-310。10.1023 /答:1025098707390。
- 23。
Ellegren H:微卫星:具有复杂进化的简单序列。NAT Rev Genet。2004,5(6):435-445。
- 24。
Estoup A,Jarne P,Cornuet JM:趋同性,并在微卫星位点突变模型及其在群体遗传学分析的结果。Mol Ecol。2002年,11(9):1591年至1604年。10.1046 / j.1365-294X.2002.01576.x。
- 25。
dna序列进化的一种主要机制。分子生物学与进化。1987,4(3):203-221。
- 26.
郭WZ,蔡CP,王CB,韩ZG,宋XL,王K,牛XW,王C,鲁KY,石B,等人:微卫星为基础,丰富的基因图谱揭示了基因组的结构,功能和进化棉属。遗传学。2007年,176(1):527-541。10.1534 / genetics.107.070375。
- 27。
罗德华,李玉华,李玉华:小麦微卫星图谱的构建。遗传学报。1998,149(4):2007-2023。
- 28。
acta physica sinica, 2017, 36(3): 461 - 467 .(通讯作者)王志强,王志强,王志强,等。10.1007 / BF00288605。
- 29。
朱聪,Gore M,Buckler ES,于杰:植物关联图谱的现状与展望.植物遗传学,2008,1(1):5-20.10.3835/plantgenome2008.02.0089。
- 30.
范得WEG WE,Voorrips RE,Finkers R,Kodde LP,Meulenbroek EJ,詹森Ĵ,宾克MCAM:良种基因分型:的QTL识别一个新的基于系谱的方法,并通过利用繁殖材料等位基因挖掘。Acta Hort。2006年,708:483-488。
- 31。
Patocchi, Fernandez-Fernandez F,埃文斯K, Silfverberg-Dilworth E, Matasci CL, Gobbin D, F Rezzonico, Boudichevskaia, Dunemann F, Stankiewicz-Kosyl M, et al:开发一套苹果ssrs标记生成苹果基因组pcr HiDRAS植物材料和基因型数据的验证。Acta photonica sinica, 2009, 40(4): 441 - 446。
- 32。
引用本文:张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚。基于分子标记辅助育种的蔷薇科植物育种研究进展。Acta physica sinica, 2010, 32(6): 643 - 646。
- 33。
Grant V:植物物种形成。美国纽约:哥伦比亚大学出版社;1981.
- 34。
李志刚,李志刚:异源多倍体植物二价形成机制的研究。王志强,王志强,王志强,等。10.1098 / rspb.1991.0033。
- 35。
Haynes KG,Douches DS:栽培四倍体马铃薯双减量系数的估算。应用基因理论。1993, 85 (6-7): 857-862.
- 36。
Lerceteau-Kohler E,Guerin G,Laigret F,Denoyes-Rothan B:使用AFLP映射的八薄草莓(Fragaria×Ananassa)中的混合性和多乐遗传的表征。Al Appl Genet。2003,107(4):619-628。10.1007 / S00122-003-1300-6。
- 37。
Ishikawa S, Komura D, Tsuji S, Nishimura K, Yamamoto S, Panda B, Huang J, Fukayama M, Jones KW, Aburatani H:基因分型微阵列的等位基因剂量分析。中国生物医学工程学报,2003,23(4):429 - 434。10.1016 / j.bbrc.2005.06.040。
- 38.
De Jong WS, De Jong DM, Bodis M:一种荧光5’核酸酶(TaqMan)测定方法,用于评估同源四倍体马铃薯中与红色皮肤颜色紧密相关的标记的剂量。应用计算机学报,2003,14(8):1384-1390。10.1007 / s00122 - 003 - 1420 - z。
- 39.
Hosono N, Kato M, Kiyotani K, Mushiroda T, Takata S, Sato H, Amitani H, Tsuchiya Y, Yamazaki K, Tsunoda T, et al .: CYP2D6基因分型在功能基因剂量分析中的应用。临床化学。2009,55(8):1546-1554。10.1373 / clinchem.2009.123620。
- 40
Fulton TM,Chunwongse J,Tanksley SD:MicroPrep用于从番茄和其他草本植物中提取DNA。植物mol biol rep。1995,13(3):207-209。10.1007 / BF02670897。
- 41.
Schuelke L:用于PCR片段的荧光标记的经济的方法。NAT BIOTECHNOL。2000年,18(2):233-234。10.1038 / 72708。
- 42.
Brownstein MJ,Carpten JD,Smith JR:通过标记DNA聚合酶调节非模板核苷酸添加:促进基因分型的引物修饰。生物技术,1996,20(6):1004。
- 43。
vooorrips Re:MapChart:用于链接地图和QTL的图形演示软件。j hered。2002,93(1):77-78。10.1093 / jhered / 93.1.77。
致谢
我们非常感谢俄勒冈州Coregalis国家克隆种子储存库的Nahla Bassil,用于提供“Fairland','Jerseybelle','Raritan'和'Redlow'的叶片。我们承认Wageningen大学植物育种部的Rene Smulders和Chris Maliepaard,用于批评这份手稿。通过技术顶级研究所绿色遗传学与新鲜前锋繁殖B.V合作提供了这项研究的资金。
作者信息
隶属关系
通讯作者
补充资料
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
TvD进行了实验工作,分析了数据,为方法的发展做出了贡献,并起草了手稿。TD对方法开发有贡献。YN对实验工作有贡献。MB对数据分析有贡献。BM对实验工作做出了贡献。RV对手稿有贡献。构思项目,参与方法开发,并起草文稿。所有作者阅读并批准了最终的手稿。
电子补充材料
12870 _2011_998_moesm1_esm.xls
附加文件1:ARSFL010分析Excel文件。包含峰值区域数据和ARSFL010分析的Excel文件。(XLS 342 KB)
12870_2011_998_MOESM2_ESM.XLS
附加文件2:UFFXA16H07分析Excel文件。包含峰值区域数据的Excel文件和UFFXA16H07的分析。(XLS 298 KB)
12870 _2011_998_moesm3_esm.xls
附加文件3:UAFv7500分析Excel文件。UAFv7500的峰面积数据分析Excel文件。(XLS 223 KB)
12870_2011_998_moesm4_esm.xls.
附加文件4:CO817823分析Excel文件。包含峰面积数据和CO817823分析的Excel文件。(XLS 222 KB)
12870_2011_998_MOESM5_ESM.XLS
附加文件5:CX661101分析Excel文件。CX661101的Excel文件包含峰值区域数据和分析。(XLS 170 KB)
12870 _2011_998_moesm6_esm.docx
附加文件6:CO817823分析MS Word文件。CO817823分析过程的文字描述。(多克斯24 KB)
12870_2011_998_moesm7_esm.docx.
附加文件7:CX661101分析MS Word文件。本文描述了CX661101的分析过程。(多克斯16 KB)
12870 _2011_998_moesm8_esm.docx
附加文件8:ARSFL010分析MS Word文件。本文描述了ARSFL010的分析过程。(多克斯14 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Van dijk,T.,Noordijk,Y.,Dubos,T.等等。微卫星等位基因剂量与配置建立(MADCE):各种聚氯吡啶素遗传研究的综合方法。BMC植物BIOL.12,25(2012)。https://doi.org/10.1186/1471-2229-12-25
已收到:
公认:
发表:
关键词
- null等位基因
- 共享等位基因
- 参考等位基因
- 等位基因剂量
- 虚拟参考咨询