- 研究文章
- 开放访问
- 发表:
木薯对轴突黄单胞菌(Xanthomonas axonopodis pv)感染差异表达mirna的生物信息学鉴定Manihotis.
BMC植物生物学体积12., 文章编号:29.(2012)
抽象的
背景
MicroRNAs(miRNA)是通过沉积互补mRNA来控制基因表达的短RNA分子。它们在植物中的压力反应中发挥着至关重要的作用,包括生物应激。已知一些miRNA响应细菌感染拟南芥蒂利亚纳但目前未知这些反应是否在其他植物中保存,以及新型特异性米兰国家是否可以在防御中发挥作用。
结果
这项工作解决了MiRNA在的作用Manihot Esculenta.(木薯) -黄axonopodispv。Manihotis.(XAM)交互。下一代测序技术用于分析感染和未感染木薯组织的小RNA库Xam.一个完整的Cassava Mirnas的曲目被特征,其中包括56个保守的家庭和12个新的木薯特异性家庭。对于许多miRNA家庭的木薯基因组预测内源性靶标。一些MiRNA家庭的表达响应于细菌感染,包括通过靶向生长素响应因素以及一些木薯特异性miRNA来介导防御的miRNA。一些细菌压抑的mirnas包括参与铜调节的家庭以及靶向抗病基因的家庭。鉴定了推定的转录因子结合位点(TFBs)miRNA.基因启动子区域并与miRNA靶基因和蛋白质编码基因中的启动子区相比,揭示了差异miRNA.基因转录调控等基因。
结论
这些结果表明,木薯的MiRNA在防守方面发挥作用Xam,并且该机制类似于所知道的机制拟南芥并涉及一些同一个家庭。
背景
简单地说,植物-细菌的相互作用可以被认为是在分子水平上主要由三种蛋白质控制的:植物PRRs(病原体识别受体),细菌效应剂和植物抗性蛋白。PRRs是识别微生物MAMP(微生物相关分子模式)中高度保守结构和分子的蛋白质,并介导MAMP触发免疫(MTI),对非适应病原体有效。病原体已经产生了抑制MTI的效应蛋白。反过来,植物可以通过抗性蛋白介导的效应子特异性识别来抵消效应子的作用,这将触发一种被称为ETI(效应触发免疫)的强大防御反应[1].
在过去十年中,也发现小RNA是调解植物病原体相互作用以及许多其他生物过程中的关键球员。microRNA(miRNA)是真核基因表达的重要调节因子。它们是从核转录的miRNA.RNA聚合酶II(RNA POL II)的基因为原发性miRNA(PRI-miRNA)。然后通过Dicer样蛋白(DCL)将PRI-MIRNA在植物中加工成前体miRNA(预米兰),其形成特征的发夹状结构[2那3.].DCL切割预先生的后续处理以形成miRNA:miRNA *双链体(21-22nt)。然后将双链体甲基化并从细胞核输出到细胞质,在菌毒素(前)蛋白质识别并掺入RNA诱导的沉默复合物(RISC)中。只有成熟的miRNA链(通常是具有更稳定的5'末端配对)的成熟miRNA链,在该复合物中保留,而乘客(miRNA *)链变得降解[3.].然而,在某些情况下,miRNA*已被检测到表达水平与前导链相同甚至更高,并可能具有沉默活性[4.].RISC复合物将引导互补mRNA(靶标)沉默,通常通过10之间的裂解th11.th配对miRNA的NT [3.].
早期的miRNA通路控制机制来源于miRNA.基因转录法规独联体监管元素和trans行为因素。最近的工作试图确定参与miRNA调控的关键元素[5.-9.];然而,在不同条件下,迄今为止对miRNA共调节甚至知之甚少。
大多数已知的植物mirna靶向的转录因子在调控植物发育中发挥重要作用。现在有越来越多的证据表明miRNA在植物应对生物和非生物胁迫中的重要性[2那10.].重编程mirna介导的基因表达在植物免疫应答过程中尚未得到深入研究,但它可能是控制病原菌入侵的一个重要因素。已有研究表明,细菌诱导的miR393介导了抗细菌防御A. Thaliana.对假单胞菌含油pv。番茄(PST)通过靶向TIR1,植物蛋白受体的F箱系列,因此压制植物素信令[11.].反过来,细菌使用效应蛋白来破坏miRNA积累[12.].已知的细菌应答mirna已经增加,包括几个已知调节激素信号的家族,如参与生长素信号的miR160, miR167和miR390,参与ABA信号的miR159和参与茉莉酸信号的miR319 [13.-15.].
木薯(将木薯耐糖)木薯是一种主要作物,其根部储存了大量的淀粉。这些根是世界上超过5亿人的主要热量来源,主要分布在热带地区[16.].淀粉在工业中也具有重要用途,包括生物乙醇生产[16.-18.].木薯对非生物胁迫具有显著的耐受性,可在低肥力酸性土壤中种植,耐旱能力强[16.].由革兰氏阴性菌引起的木薯细菌性枯萎病(CBB)会严重影响木薯的产量黄axonopodispv。Manihotis.(XAM).这种疾病存在于所有种植木薯的地区,产量损失可高达80%或100% [19.].随着许多植物疾病的CBB发病率预计将大大增加气候变化[20.].随着人类数量的不断增加,了解植物抗菌防御的潜在机制,旨在为作物的遗传改良提供生物技术策略。
Cassava Mirna Repertoire大多是未知的。最多20个保守的miRNA家庭通过使用生物信息学方法在ESTS系列中盲目化[21.-23.[Cassava和其他大戟属23种成熟MiRNA家族的表达[24.].然而,目前没有来自木薯的mirna存储在miRBase(已验证mirna的共识数据库)中[25.].Cassava Genome的初稿于2009年10月发布,2010年10月提供了新版本[26.].这是发现和预测新的和特定的Cassava MiRNA的重要工具。
本研究利用小RNA库的表达数据对木薯miRNA库进行了特征分析,并鉴定了木薯基因组中的前miRNA。mirna介导的反应Xam也分析了木薯的感染以及MiRNA调节的可能转录因子。
结果
Cassava SRNA库的深度测序
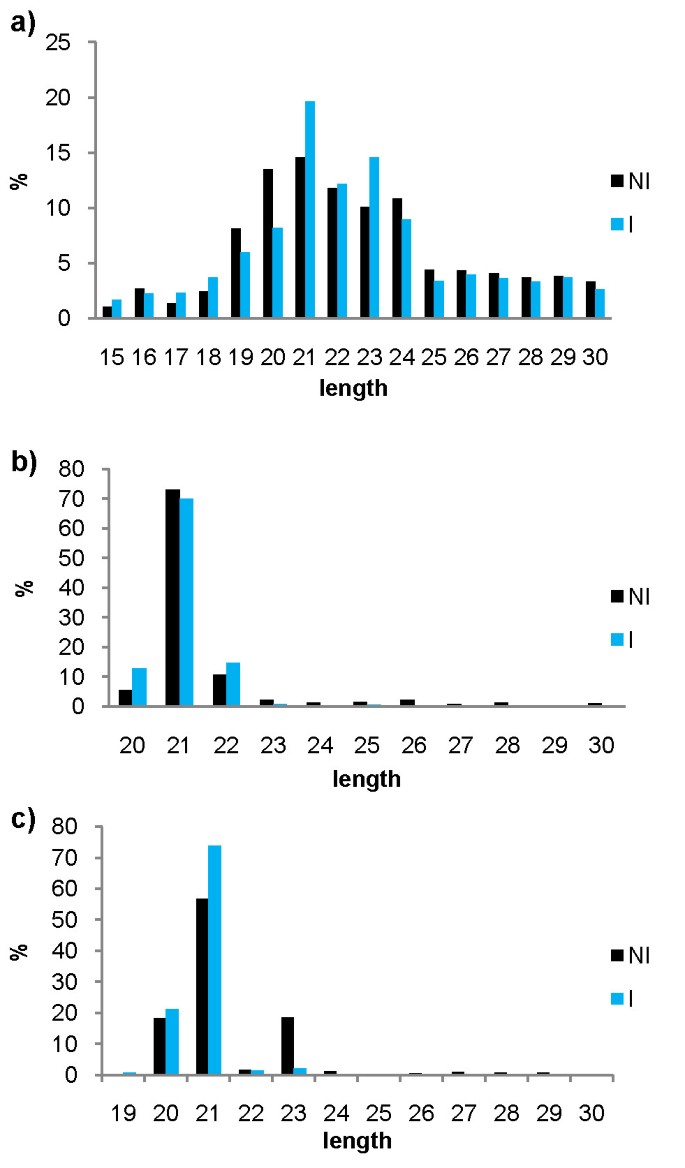
利用Illumina SBS技术测序的小RNA图谱库研究了木薯mirna在响应玉米生长中的作用Xam.利用从叶和茎中提取的RNA构建了两个小的RNA库Xam-Resistant Cassava品种MBRA685。其中一个没有接种(倪)另一个则接种了疫苗(一世)具有高度攻击性的Xam应变CIO151。接种文库由不同接种后时间的RNA库构建,因此只能检测到稳健和一致的反应。从NI和I库中分别获得了15和1100万个读修剪后的读(删除了适配器)1).两个库中的大多数修剪读数都在20-24NT范围内(最小的RNA尺寸)(图1).这些文库的处理和原始文件可在NCBI基因表达综合数据库中访问,登录号为GSE29379。
从两个文库的读取被映射到木薯基因组(表1),导致800万(NI)和700万(I)完全映射到基因组。然后删除映射到已知的snoRNAs、tRNAs和rnas的Reads,分别从NI和I库中获得5,655,383和5,187,027个Reads,用于进一步分析。
这两个图书馆也映射了Xam基因组以验证来自I但不在NI文库中的细菌的核酸存在(表1).正如预期的那样,NI库的读取较少映射了Xam基因组(0.8%)与I库(5.2%)相比。大多数ni读匹配Xam基因组也匹配木薯基因组(91%),因此在两个基因组之间共享短区域。我读取的数据中只有33%映射到Xam基因组对应这些共享区域;其余67%可能为非编码RNA或降解RNAXam.
miRNA和pre-miRNA鉴定
为了识别保守的miRNA,将来自两个文库的读数映射到来自miRBase的一组已知的植物miRNA [25.]及PMRD [27.];在木薯鉴定了56个保守的miRNA系列。读取映射到已知的miRNA主要是21nt长(图1 b).这些mirna也使用miRProf筛选[28.]所有56个家庭都用这种方法确认。与miRBase不同,PMRD包括没有实验验证的MIRNA。仅针对小型RNA木薯库映射的七个PMRD-独家miRNA家庭;考虑到前miRNA鉴定,但不能在木薯基因组中鉴定出前体,并且被排除在以后的分析中。
读取56个保守的miRNA家族被映射到基因组,并分析了485个相邻地区,发现116符合被视为真正的麦克马斯(结构特征和二级结构统计测试)的主要标准。这些前miRNA代表了24家MiRNA系列(附加文件1).一些家族未发现pre- mirna,这可能是由于木薯基因组测序不完整或存在非典型pre- mirna所致。
为了识别新的MIRNA,读取从两个文库的20到24次的读数被映射到木薯基太基地区(表1)和相邻的±150NT区域被提取(总共64,876个区域)。mirchek [29.], miPred [30.]和mireap [31]用于初始过滤可能的前miRNA。迎接上述主要标准的候选人(结构特征和二级结构统计测试)以及从Mircheck,Mireap或Mipref的支持证据被认为是真正的梅尔纳斯。通过Cass_Mirl(临时名称)确定来自12个miRNA家庭的12个新的miRNA前体,并命名为Cass_mira(临时名称)(附加文件1).
与保守mirna一样,预测的新mirna的Reads大多为21nt长(图)1 c).一个爆炸[32[Genbank的核苷酸NR数据库在其他植物基因组中寻找高预浸型NR数据库,揭示了在12个预先生的12个前10名中的10个不同的植物核苷酸序列的相似性(E值<0.001,> 80%覆盖率),虽然两个前miRNA,CASS-MIRK和CASS_MIRL,类似于非编码序列Populus Thricocarpa.和Helianthus petiolari.分别。
目标预测
使用Miranda软件的修改版本预测靶向Cassava基因组中鉴定的所有miRNA的靶标[33];277鉴定了43个保守的miRNA系列的可能目标,以及120个新的miRNA家庭的70个目标(附加档案2).利用psRNAtarget搜索木薯基因组中的所有mirnahttp://plantgrn.noble.org/psRNATarget/.在米兰达确定的347个目标中,有162个获得了实际目标的额外支持(附加文件)2).鉴定的可能靶标是转录因子(24%),对于许多已知的植物miRNA是常见的[2].其他常用的预测靶点是激酶(7%)、DNA结合蛋白(3%)和抗病蛋白(3%)。4个家族(miR167、miR397、miR894和Cass_miRJ)与KEGG通路(map00500)基因的相似性表明,它们可能参与淀粉的生物合成或代谢,这可能是生物乙醇生物技术策略的重要焦点。
miRNA差异表达
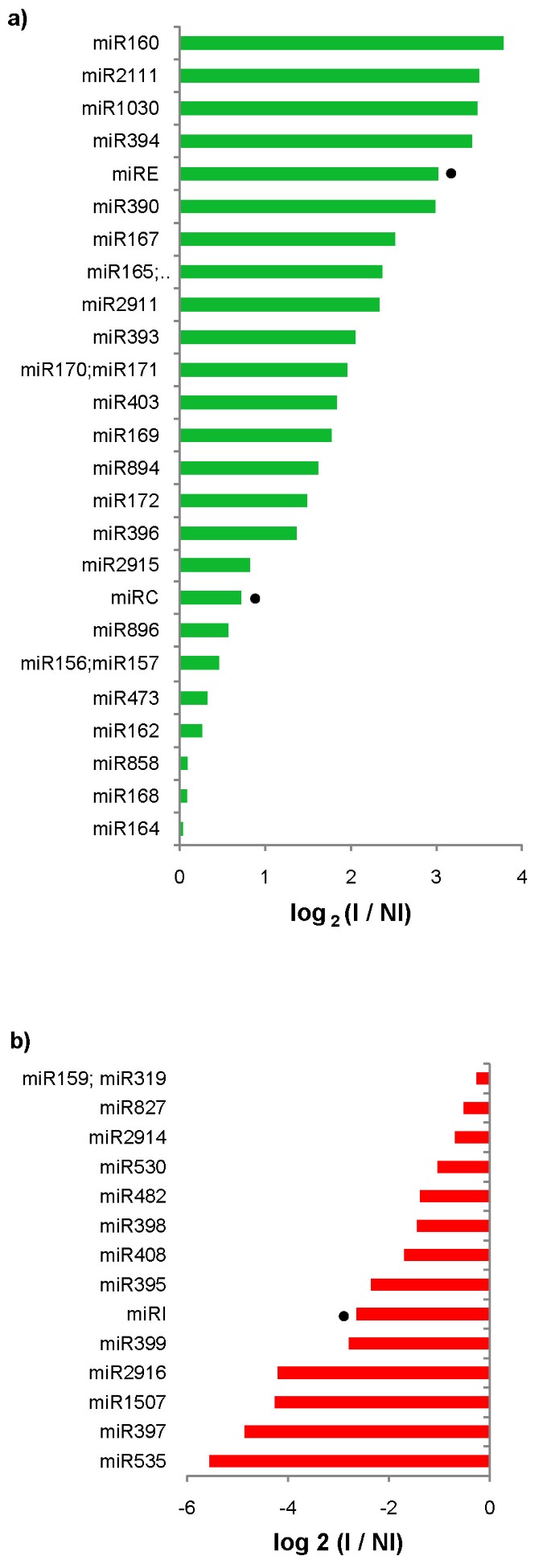
木薯的miRNA表达水平被量化为两个文库中每个成熟miRNA的标准化reads映射。10个保守的miRNA家族表达显著增加2折叠> 2)响应Xam感染(图2),包括MIR160和MIR167,其既已知靶向生长素响应因子(ARFS)[13.和miR393和miR390家族,这些家族也被认为调节生长素信号[13.].Cassava中MiR160的预测靶标是ARF样基因,而预测的miR167的靶标包括磷酸酶和肽酶。通过半定量RT-PCR确认这些目标响应细菌的下调(图3.).可以确认其他诱导的保守miRNA家族的预测目标下调,包括靶向F盒家族蛋白,ATPB转录因子和稻草人样转录因子的miR394,miR165和miR171(图3.).在其中一些情况下(MiR171,MiR171,MiR394,MiR197b)靶表达在接种后4天下降(DPI),然后在8 dpi下增加,但从未达到比没有接种的表达比那些更高的表达值。这可以预期,因为文库由从接种后的不同时间汇集的RNA构建。尽管这些数据的重要性,仍然需要对成熟miRNA的特定量化进行准确地与目标表达相关。
另一方面,七个miRNA家庭的表达响应而变化Xam感染(日志2折叠<-2)(图2 b).这些抑制家族包括MIR535,MIR395和MIR482,其预测靶向各种候选NB-LRR和LRR疾病抗性蛋白质。还压抑MIR397,MIR398和MIR408系列(日志2褶皱< -1.4);它们分别通过靶向漆酶、铜超氧化物歧化酶和植物青素参与铜调控[34].可以确认细菌诱导的抗性样蛋白的表达预测,预测由miR535靶向(在4 dpi诱导),对于预测的植物素,预测由mir408靶向(在8 dpi诱导)(图3.).
新的miRNA具有整体低表达水平。只有三个新颖的miRNA(CASS_MIRC,CASS_MIRE和CASS_MIRI)的表达值高于至少一个库中的10个标准化读数。因此,虽然许多响应于细菌的差异表达,但由于它们的低表达值,它们未被考虑。已知物种特异性miRNA与保守的miRNA相比具有低表达[35].Cass_miRE对细菌(log2fold = 3)(图2)和下调其预测的目标,一个与之相关的激酶答:芥FERONIA,通过RT-PCR确定(图3.).已知该基因参与病原体相互作用和花粉发育[36那37].
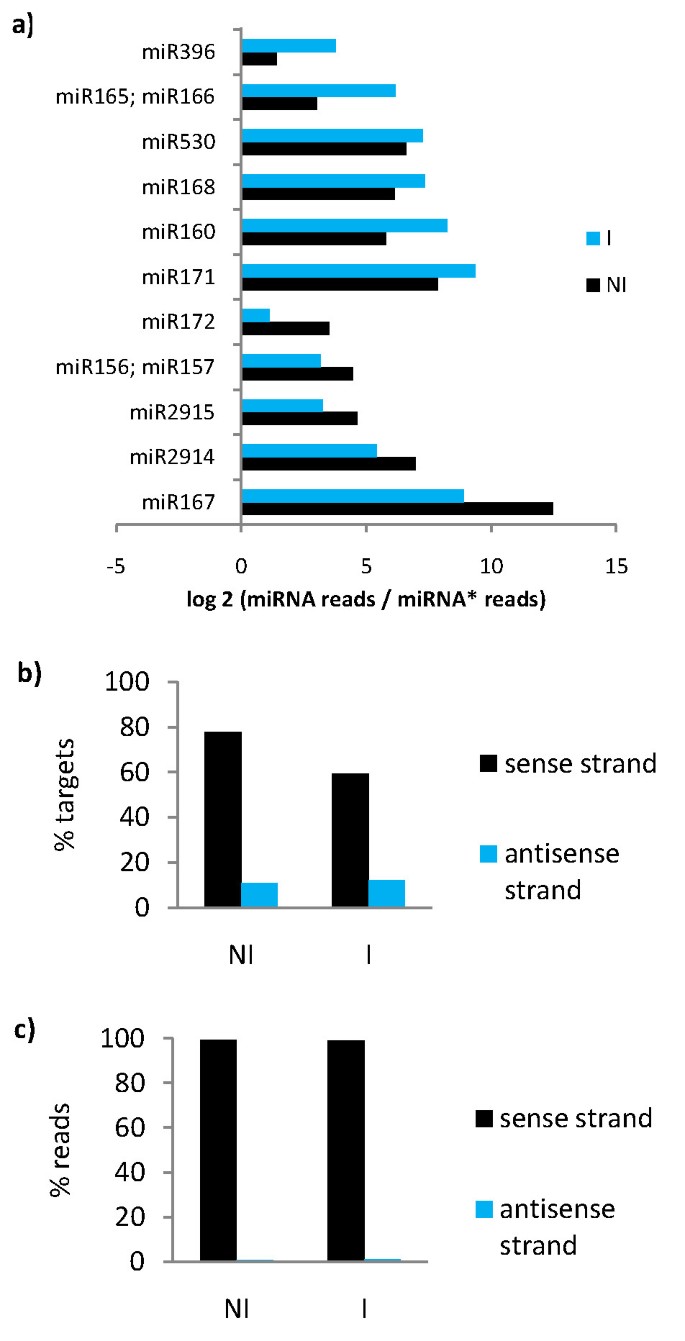
乘客链(miRNA *)在miRNA:miRNA *双面破坏和领导链中的速度迅速降解到RISC中[3.].然而,它的表达可以在小RNA文库中检测到,并且认为它被认为是miRNA验证的支持标准[38].木薯miRNA*的表达量化为每个文库链中从pre-miRNA预测到的木薯miRNA*的reads数量。检测11个保守miRNA家族的miRNA*表达。在NI和I文库中,所有家族的miRNA/miRNA*比值均高于1,这意味着如预期的那样,leader链总是比passenger链高表达。在NI和I文库中,每个miRNA家族的miRNA/miRNA*比值都发生了变化4A).miRNA / miRNA *比例的变化可以在不同条件下表明miRNA *链的差异降解;然而,由于检测到降解分子,它很可能是随机效应。
认为可以通过衍生自切割靶MRNA的瞬时siRNA来扩增miRNA沉默信号[39].两个文库的读数被特异性映射到预测的Cassava miRNA靶标'感觉或反义链,以评估在木薯中发生过渡或miRNA沉默信号放大的程度。读取到反义链的映射将指示RDRP识别之后的DSRNA衍生的传递siRNA的存在,而读取映射到义子链的映射可能主要裂解MiRNA靶产物以及可能的siRNA。预测miRNA靶标具有读取到两种文库中的反义链(产生传递siRNA)的反义链(靶标)的百分比约为10%(图4B.).读取到miRNA目标的反义链(107在Ni中的107读数,I中的235读数)约为预测目标的读取映射的1%(图4C.),即可能的传递分泌siRNA表达。这表明只有小百分比的靶向基因产生传递siRNA,与miRNA靶裂解率相比,它们的产生非常少量。这些结果表明,通过靶分发性的miRNA信号传导扩增不是Cassava中的一种普遍的机制。
启动子分析
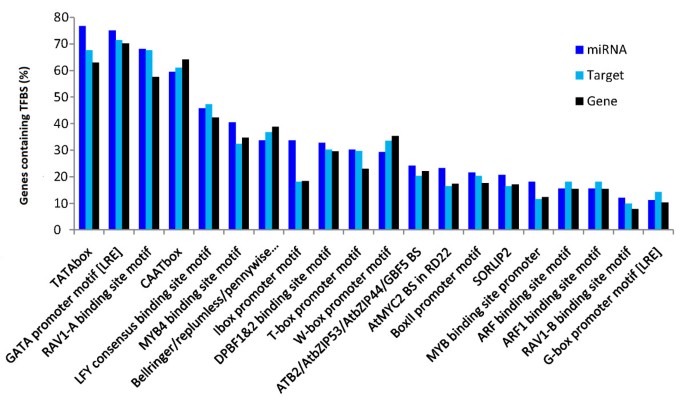
miRNA.在转录因子识别后,基因被RNA POL-II转录;但是,关于miRNA转录调节的知识毫无疑问[5.].转录因子结合位点(Transcription factor binding sites, TFBS)位于木薯pre- mirna上游的1000nt启动子区域,以寻找可能的模式miRNA.基因调节响应细菌。在116 miRNA启动子区中鉴定了59种转录因子的结合位点。常见的监管主题包括塔塔和gata盒和Rav(Abi3 / VP1相关)和LFY(叶茂)TFBS(图5.).
研究发现,ARF结合位点在生长素相关的miRNA家族如miR160和miR167中过度代表,从而形成一个调控环路[40].在我们的结果中,MIR160也是具有鉴定的最多ARF结合位点的家庭(在MiR160启动子区中发现了44个ARF结合位点中的10个)。
然后对木薯miRNA靶基因的启动子区域和一组随机选择的1000个木薯基因进行了同样的预测,以确定miRNA是否与其他基因组转录不同。配对wilcoxon检验比较各组的频率分布,发现各组间有显著差异(α = 0.1)miRNA.基因及随机选择的基因(p-value = 0.001248)之间miRNA.基因和miRNA靶标(p-value = 0.08904),而靶基因与随机选择基因之间无差异(p-Value = 0.5462)。然而,在响应细菌和非差异表达的miRNA家庭的频率分布之间没有发现频率分布或压抑的miRNA系列之间的显着差异(p-Value> 0.05)。
讨论
在此基础上,提出了一个通用模型Arabidopsis-Pseudomonas描述miRNA介导对细菌感染的反应的相互作用结果。在该模型中,诱导靶向负防御响应调节剂的MIRNA,并在细菌感染时抑制靶向阳性调节剂(例如抗性基因)的miRNA [41那42].在这项工作中,我们报告了一些木薯miRNA家庭的差异表达以响应黄axonopodispv。曼尼霍提斯,大体上同意这个模式。mirna诱导被发现参与调控生长素信号:miR160, miR167, miR390和miR393 [11.那13.].这些miRNA家族表达模式的保存拟南芥和木薯指向植物保守的防御机制,可能与PTI有关,因为这些家庭已被证明是响应旗杆的诱导[14.].因此,助长信号传导中断似乎是损害植物细菌生长的重要策略。最近的工作表明,毒素信号传导的酶促破坏是广谱抗性的重要机制,并且病原体在感染期间分泌毒素,使植物易受松动细胞壁易受攻击的植物[43那44].一些保守的miRNA家族不存在A. Thaliana.被诱导表达回应Xam感染,包括到目前为止的mir2911和mir1030家庭,只有迄今为止已被识别Populus Euphratica.和Physcomithrella金属盘, 分别 [45].它们在调节植物-细菌相互作用中可能发挥的作用仍然未知。
在这项工作中显示的MiRNA下调也同意了一般模型,因为预计三个压抑的家族(MiR535,MiR395和MiR482)靶向木薯中的Nb-LRR和LRR抗性等基因。这些基因随后的增加的表达可能在Cassava特异性ETI反应中具有作用;但是,这仍然需要证明。其他压抑的家庭是miR397,mir398和mir408,已知参与铜调节,并响应生物应激差异表达[34那46那47].铜因其抗菌作用,在农业中被广泛用作农药[48].已经表明了一些Xanthomonas oryzae.菌株使用转录活化剂样(tal)效应蛋白改性水稻中的铜分布,以使植物易感[49].mirna介导的铜稳态调节也可能是细菌防御机制的关键。
传递性通过量化可能靶标切割衍生的瞬时sinas进行分析。本文对该机理进行了详细的研究A. Thaliana.[50.-52.在苔藓里Physcomitrella patens.[53.].然而,最近的一项研究表明,这种机制可能比以前认为的更广泛;分析小RNA库的下一代测序,同样的比例发现了小21nt reads的图谱拟南芥在感觉和反义链中的miRNA靶向[39].相反,它在这项工作中发现了读取映射O木薯靶的反义链,在木薯文库(<1%)中大大面积,并且只有约10%的预测目标产生反义读,同意传递态度是一种不常见的机制。
最后,预测TFBS在启动子区miRNA.基因的发现miRNA.基因的总体频率分布似乎与包括miRNA靶点在内的蛋白质编码基因的频率分布不同,这表明存在特异性作用的调控元件miRNA.基因。它以前报告过miRNA.与蛋白质编码基因相比,基因在塔塔盒,Atmyc,Arf,Sorlep3和Lfy结合位点中丰富[40].在我们的结果中,这些基因组中TFBS的频率分布显示,即塔塔盒,MYB4和L1箱图案更加丰富miRNA.基因。然而,在细菌诱导或细菌抑制的miRNA家庭和家庭之间没有发现TFBS频率的差异,这表明MiRNA转录调节响应细菌可能是家庭特异性或基因特异性的。
结论
这项工作表明了Cassava MiRNA的深入表征,响应于Xam这些分子在植物与病原体相互作用中的重要性得到了新的认识。这些数据是令人鼓舞的,但仍然是初步的,还需要进一步的实验验证,以充分了解miRNA途径在木薯-的影响Xam交互。了解miRNA的调控和/或其靶点的调控可以(给予miRNA通路灵活性)导致开发更好的生物技术策略,旨在生产增强对病原体抵抗力的木薯植物。
方法
植物材料和植物接种
木薯植物从成熟的茎切屑生长,并在26-30℃下保持温室,具有12小时光照的光周期和80%相对湿度。木薯品种MBRA685(耐用Xam-CiO151)用于小RNA文库构建和品种MCOL1522(易受影响XAM-CiO151)用于RT-PCR实验。
将6周龄的植株与36 h龄的进攻性培养物接种黄axonopodispv。Sanihotis菌株CiO151在叶子和茎中。通过在叶片中刺穿六个孔并放置5μL液体来接种叶子Xam-MgCl2文化在OD校准600 nm.= 0.002(1×108.cfu / ml)。接种每种叶子的两只小叶和每株植物的三片叶子。茎被穿刺接种。每个实验接种每个收集时间的至少三种植物。
小RNA图书馆建设和测序
对于接种的文库叶子和茎被接种植物收集(0h接种后-HPI,6hPI,24hpi,2天接种后-DPI,5 dpi,7 dpi和15dpi)。使用苯甲酸酚:氯仿方法。将来自接种植物的RNA提取(每个时间点至少六种植物)以相等的量合并在一起。对于非接种的文库,使用来自来自六种未处理植物的组织的单个RNA提取。
在总RNA (150 μg/mL)中加入适配器构建文库,并使用上标双链cDNA Synthesis kit (Invitrogen)合成cDNA。通过PCR富集cDNA,然后在变性聚丙烯酰胺凝胶上分离小RNA片段(10-50 nt)。不列颠哥伦比亚省癌症机构的迈克尔·史密斯基因组科学中心建立了图书馆http://www.bcgsc.ca/使用Illumina Genome Analyzer IIX的Illumina SBS深度测序技术测序http://www.illumina.com/.
小RNA测序数据分析
将各种质量过滤器应用于RAW读取数据。除去在前25个碱基中具有小于0.6浓度值(通过测序软件测量)的序列。然后用内部C ++程序除去适配器序列;该程序从匹配库中使用的适配器序列(5'-atctcgtatgccgtcttcttctg-3')和序列片段的原始序列中删除了大于10nt的任何序列片段,并且只有在15后开始短于10nt的序列片段thnt在原始序列中。该程序使用了浮雕WordFinder工具[54.]查找适配器片段(参数:最小匹配分数= 70,对齐宽度= 6,缺口打开0.0 +差距延伸10年或缺口打开500 +差距延伸10)。小于15nt的序列以及低复杂度和低质量的序列然后使用UNIX命令删除。
读取被映射到从RFAM数据库获得的已知的Snornas,TRNA和RRNA进行映射[55.]然后删除。这是通过使用BLASTN,v.2.21(参数:电子价值<0.0001,ungapped、文字大小= 4) [56.].除非表明,否则这些是用于所有映射分析的标准参数。
然后将读数映射到木薯基因组v.4(Cassava Genome项目2010,http://www.phytozome.net/cassava.)(参数:电子价值< 1e-5, 100% identity)和初步版本黄axonopodispv。manihotis基因组(Xam基因组项目,未发表的(参数;电子价值< 1e-5, 100% identity)。
miRNA和pre-miRNA鉴定
为了识别系统遗传学上保守的mirna,我们将两个小RNA库的reads定位到所有成熟的Viridiplantae mirna集合上,这些mirna来自于2010年9月16日发布的miRBase [45]以及从植物miRNA数据库PMRD,v的完整植物miRNA设置。2010年9月[22.].读取少于两种与已知miRNA不匹配的读错被认为是保守的miRNA [38].使用UEA sRNA工具包中的miRProf工具(默认参数)也鉴定了保守mirna(及其表达谱)[28.].从24NT尺寸范围内的两个库的读数都映射到Cassava Genome(Cassava Genome项目2010,http://www.phytozome.net/cassava.)识别可能的新的木薯特异性miRNA。
前miRNA分析使用相邻区域(-150,+150nt)从读取的读取位置,使用Fastacmd从基因组中提取[56.].然后将这些被映射到注释的木薯基因上,并从进一步的分析中除去具有基因的大重叠(> 25%)的区域。
考虑了两个主要标准,用于真实的预先miRNA:从预测的折叠和二级结构统计测试中识别的结构特征。候选前miRNA与维也纳RNA包的rnafold折叠[57.]和mfold v。3.5 [58.].使用sRNA工具包中的RNAfolding工具获得了其他更容易可视化的二级结构[28.].用内部珍珠脚本分析了结构。如果在结构中的预测成熟miRNA和miRNA *之间有少于六个不匹配,则考虑真实的预先匹配,很少(最多三个)和结构中的短(小于3nt)不对称凸起[38那59.].采用统计检验计算二次结构最小折叠能(MFE)显著性;每个可能的前体产生1000个随机序列,保持相同的碱基组成和二核苷酸频率(k-let = 2)使用ushuffle [60.].然后使用rnafold折叠随机结构[57.], p值计算为MFE等于或低于原始前驱体的随机结构的百分比[61.].真正的pre- mirna的p值必须< 0.05。
使用miRcheck(默认参数)对候选的新pre- mirna进行分析和过滤[29.] mireap(参数,b = 55,a = 19,b = 24,u = 1,000,e = -10 kcal / mol,d = 200,p= 7,v = 10,s = 100,f = 10)[31]https://sourceforge.net/projects/mireap/和米镜(无随机森林预测模块未使用)[30.]获得额外的预测支持。
microrna的目标预测
使用修改后的Miranda版本v.2.0 2008年9月[33],如前所述,以满足植物miRNA:目标配对标准[62.].使用PSRNATarget,v。2010年12月(默认参数)也搜索目标(默认参数)http://plantgrn.noble.org/psRNATarget/),是对miRU软件的更新[63.].在Cassava编码序列中搜索目标(Cassava Genome项目2010,http://www.phytozome.net/cassava.).
miRNA,miRNA *和瞬态sirnas在网上量化
来自两个文库的读数被映射链 - 特别是使用blastn量化miRNA和miRNA *表达(电子价值<0.0001,100%的身份,s = 1microrna的和s = 2.对于miRNA *)。由于成熟miRNA序列中的高相似性,为每个系列而不是每个基因座分配表达值。使用此公式归一化表达式值:匹配读/总读取×1,000,000。
从两个sRNA文库中从20到24nt的Reads被特异性地针对所有已识别的所有木薯mirna的可能靶标进行链图,以量化可能的瞬时sirna和由mirna介导的切割生成的mRNA片段(电子价值<0.0001,100%的身份,s = 1microrna的和s = 2.对于miRNA)。仅用于预测的miRNA靶基因裂解位点的-100nt,+ 100nt区域用于映射以避免映射到未通过miRNA裂解产生的随机RNA片段。表达式值被标准化为读取目标/总读取的读取映射的数量。
启动子分析
提取1000 nt上游区域鉴定的木薯pre-miRNA、预测miRNA靶标和随机选择的木薯基因进行启动子鉴定;只提取完整、高质量的序列。与基因有重叠的区域被划去排除基因,除非重叠超过600 nt;在这种情况下,启动子区域没有被使用。TFBS在这些区域被识别,如[6.].
Wilcoxon配对测试(双尾)由R版本v.2.12.0(R开发团队)进行比较TFBS频率为每个所需的基因组获得:具有给定TFB的基因数/评价的基因总量。
半定量RT-PCR分析
从木薯MCOL1522未接种和接种(4dpi和8dpi)的叶片和茎组织中提取RNAXamCIO151,如上所述,用于量化miRNA靶表达。经DNAse I (Fermentas)处理后,用oligo dTs (Fermentas)合成cDNA第一链。cDNA浓度用微管引物(F = 5'-GATCCTACTGGGAAGTACATTGG-3', R = 5'-GATCATTCTCCACCAACTGA-3')进行PCR后归一化。设计用于预测miRNA靶点的基因特异性引物,覆盖预测的剪切位点。pcr共32个周期,94℃30 s, 54-60℃30 s, 72℃30 s。每个目标所使用的引物在附加文件中列出3..
参考文献
- 1。
寄主-微生物的相互作用:塑造植物免疫应答的进化。细胞。2006,124:803-814。10.1016 / j.cell.2006.02.008。
- 2。
Jones-Rhoades MW,Bartel DP,Bartel B:MicroRNAS及其在植物中的监管角色。Annu Rev植物Biol。2006,57:19-53。10.1146 / annurev.arplant.57.032905.105218。
- 3.
植物microrna的研究进展。植物RNA技术中的非编码RNA。编辑:Erdmann and Barciszewski。柏林:施普林格;2011:251 - 268。
- 4.
德国MA, Pillay M, Jeong DH,等:通过RNA末端的平行分析来识别microrna靶RNA对。生物工程学报。2008,26:941-946。10.1038 / nbt1417。
- 5。
谢Z,Khanna K,Ruan S:MicroRNA的表达及其在植物中的调节。细胞和发育生物学研讨会。2010,21(8):790-7。10.1016 / J.SEMCDB.2010.03.012。
- 6。
MicroRNA启动子分析。方法:中国生物医学工程学报。2010,592:149-161。10.1007 / 978 - 1 - 60327 - 005 - 2 _11。
- 7。
孟Y,黄F,Shi Q等:新型抗悬雍垂抗突变体的根系中水稻microRNA和MicroRNA-target对的基因组调查。Planta。2009,230:883-898。10.1007 / S00425-009-0994-3。
- 8。
孔卫文,杨志明:拟南芥缺铁反应microRNA基因和顺式元件的鉴定。植物营养与肥料学报。2010,31(4):489 - 494。10.1016 / j.plaphy.2009.12.008。
- 9。
崔旭,徐思明,穆德胜,杨志明:水稻microRNA启动子及群体的基因组分析。基因。2009,431:61-66。10.1016 / j.gene.2008.11.016。
- 10。
Shukla Li,Chinnusamy V,Sunkar R:MicroRNA和其他内源性小RNA在植物应激反应中的作用。BBA - 基因调节机制。2008,1779:743-748。
- 11.
Navarro L,Dunoyer P,Jay F,等:植物miRNA通过抑制养肝信号传导来促进抗菌性。科学。2006,312:436-439。10.1126 / Science.1126088。
- 12.
作者认为,细菌效应蛋白对microRNA通路的抑制作用是一种有效的途径。中国科学:地球科学。10.1126 / science.1159505。
- 13.
张伟,高胜,周昕,等:细菌应答microrna通过调节植物激素网络调控植物天然免疫。植物生态学报,2010,36(4):591 - 598。
- 14.
李勇,张玉琴,张军,等:植物天然免疫中与病原体相关分子模式相关的microrna的鉴定。植物营养与肥料学报。2010,22(2):491 - 498。
- 15.
Fahlgren N,Howell Md,Kasschau KD等:拟南芥MicroRNA的高通量测序:MiRNA基因频繁出生和死亡的证据。Plos一个。2007,2:e219-10.1371 / journal.pone.0000219。
- 16。
El-Sharkawy MA:木薯生物学和生理学。植物生态学报。2004,32(6):591 - 598。10.1007 / s11103 - 005 - 2270 - 7。
- 17。
Ziska LH, Runion GB, Tomecek M,等:阿拉巴马州和马里兰州对木薯、甘薯和玉米作为生物乙醇生产潜在碳水化合物来源的评估。生物质能源。2009,33:1503-1508。10.1016 / j.biombioe.2009.07.014。
- 18。
木薯:植物化学及其生物技术前景的评价。植物化学学报。2010,31(4):489 - 494。10.1016 / j.phytochem.2010.09.001。
- 19。
Verdier V:血管血管(OAñubumbacteriano)de la yuca gearada por xanthomonas axonopodis pv。Manihotis。EN CIAT EDS La Yuca en El Tercer Milenio Sistemas Modernos deProducciónCrocesamientoMustizaciónyCercialización。2002,148-159。
- 20。
Gregory PJ,Ingram JSI,Brklacich M:气候变化和粮食安全。伦敦皇家社会的哲学交易。生物科学。2005,360:2139-2148。10.1098 / RSTB.2005.1745。
- 21。
Mhuantong W, Wichadakul D: MicroPC (MicroPC):植物microrna预测和比较的综合资源。中国生物医学工程学报。2009,10:366-10.1186/1471-2164-10-366。
- 22。
张Z,yu J,Li D等人:PMRD:Plant MicroRNA数据库。核酸RES。2010,38:D806-D813。10.1093 / nar / gkp818。
- 23。
Amiteye S,Corral JM,Sharbel TF:MicroRNA的潜力及其对木薯(Manihot Esculenta)改进的靶基因检测的概述。j biotechnol。2011,110:2562-2573。
- 24。
曾C,王W,郑Y等,MicroRNA的保护和分歧及其在大戟属植物中的功能。数据库。2009,38:981-995。
- 25.
Griffiths-Jones S:MiRBase:MicroRNA序列和注释。Wiley在线图书馆。2010年,单位12.9。1-10。第12章
- 26.
木薯基因组计划2010:木薯基因组计划2010,[http://www.phytozome.net/cassava.].
- 27.
张Z,yu J,Li D等人:PMRD:Plant MicroRNA数据库。核酸RES。2010,38:D806-D813。10.1093 / nar / gkp818。
- 28。
Moxon S, Schwach F, Dalmay T,等:一个分析大规模植物小RNA数据集的工具包。生物信息学。2008,24:2252-2253。10.1093 /生物信息学/ btn428。
- 29。
Jones-Rhoades MW,Bartel DP:植物微小RNA的计算鉴定及其靶标,包括应激诱导的miRNA。Mol细胞。2004年,14:787-799。10.1016 / j.molcel.2004.05.027。
- 30.
姜鹏,吴洪波,王伟,等:基于混合特征的随机森林预测模型对真实和伪microRNA前体的分类。核酸学报2007,35(4):437 - 444。10.1093 / nar / gkm368。
- 31。
陈X,Li Q,Wang J等:SOLEXA测序的新型Amphioxus microRNA的鉴定与表征。基因组Biol。2009,10:R78-10.1186 / GB-2009-10-7-R78。
- 32。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中华生物医学杂志,1996,15(4):427 - 434。
- 33。
Enright AJ, John B, Gaul U,等:果蝇中的MicroRNA靶标。生物谷bioon . 2003, 5: R1-10.1186/gb-2003-5-1-r1。
- 34。
微rna介导的拟南芥铜蛋白表达的系统性下调。中国生物医学工程学报。2008,29(3):457 - 461。10.1074 / jbc.M801406200。
- 35。
Cuperus JT, Fahlgren N, Carrington JC: MIRNA基因的进化和功能多样化。acta botanica sinica(云南植物学报),2011,23(4):431-442。10.1105 / tpc.110.082784。
- 36。
Kessler SA,Shimosato-Asano H,Keinath NF等:Pollen管接收和真菌入侵的保守分子组分。科学。2010,330:968-971。10.1126 / Science.1195211。
- 37.
Escobar-Restrepo J-M,哈克N,Kessler S等人:在花粉管接收过程中介导男性雌性相互作用的Feronia受体样激酶。科学。2007,317:656-660。10.1126 / Science.1143562。
- 38.
Meyers Bc,Axtell MJ,Bartel B等人:植物MicroRNA的注释标准。植物细胞。2008,20:3186-3190。10.1105 / TPC.108.064311。
- 39.
罗启杰,Samanta MP, Köksal F,等:拟南芥microRNA靶标mrna的反义转录证据。公共科学图书馆。2009,5:e1000457-10.1371/journal.pgen.1000457。
- 40.
Megraw M,Baev V,Rusinov V,等:MicroRNA启动子元素在拟南芥中发现。RNA。2006,12(9):1612-1619。10.1261 / RNA.130506。
- 41.
植物小rna在生物胁迫反应中的作用。植物生态学报。2009,60:485-510。10.1146 / annurev.arplant.043008.092111。
- 42.
陈志刚,陈志刚,陈志刚:小rna在宿主-微生物相互作用中的作用。植物病理学杂志。2010,48:225-10.1146/annurev-phyto-073009-114457。
- 43。
傅军,刘洪,李勇,等:通过抑制病原菌诱导的水稻生长素积累调控水稻广谱抗病。植物营养与肥料学报。2011,32(4):591 - 598。10.1104 / pp.110.163774。
- 44。
丁X,CaO Y,Huang L等:吲哚-3-乙酸-Amido合成酶GH3-8的活化抑制了扩张素表达并促进水稻中的水杨酸盐和淫秽的基础免疫力。植物细胞。2008,20:228-240。10.1105 / TPC.107.055657。
- 45。
Griffiths-Jones S,Saini HK,Van Dongen S,Engright AJ:Mirbase:MicroRNA基因组学的工具。核酸RES。2008,36:D154-D158。10.1093 / nar / gkn221。
- 46。
刘H,田X,李Y,吴C:拟南芥压力监管微稻草的基于微阵列的分析。RNA。2008年,14:836-843。10.1261 / RNA.895308。
- 47。
Sunkar R,朱家·朱茹:来自拟南芥的新型和压力调节的微小RNA和其他小RNA。sci stke。2004,16:2001-2019。
- 48。
Borkow G, Gabbay J:将铜付诸行动:具有强杀生物活性的铜浸渍产品。美国实验生物学学会联合会的官方刊物。2004,18:1728-1730。
- 49.
袁敏,褚志强,李晓霞,徐超,王树生:水稻黄单胞菌通过调节寄主铜的再分配来克服水稻防御。植物学报。2010,22(1):1-14。10.1105 / tpc.109.220110。
- 50.
植物siRNA生物发生的双重触发。细胞。2006,127:565-577。10.1016 / j.cell.2006.09.032。
- 51.
拟南芥RNA- dependent RNA POLYMERASE6/DICER-LIKE4通路的全基因组分析揭示了miRNA-和tasirna定向靶向的依赖性[W][OA]。acta botanica sinica(云南植物研究),2007,19(4):427 - 434。10.1105 / tpc.107.050062。
- 52.
Ronemus M,Vaughn MW,Martienssen Ra:MicroRNA靶向和小干扰RNA介导的mRNA降解由拟南芥中的Argonaute,Dicer和RNA依赖性RNA聚合酶调节。植物细胞。2006,18:1559-1574。10.1105 / TPC.106.042127。
- 53.
Khraiwesh B,ARIF MA,Seumel Gi,等:MicroRNA的基因表达转录控制。细胞。2010,140:111-122。10.1016 / J.Cell.2009.12.023。
- 54.
EMBOSS:欧洲分子生物学开放软件套件。《趋势通讯》,2000,16:276-277。10.1016 / s0168 - 9525(00) 02024 - 2。
- 55。
Rfam:更新RNA家族数据库。核酸学报2009,37(4):497 - 503。10.1093 / nar / gkn766。
- 56。
alschul SF, Madden TL, Schaffer AA, et al: gap BLAST和pci -BLAST:新一代蛋白质数据库搜索程序。核酸学报1997,25:3389-3402。10.1093 / nar / 25.17.3389。
- 57。
Hofacker IL,Fontana W,Stadler PF等:快速折叠和RNA二级结构的比较。Monatsheftefürchemie月刊。1994年,125:167-188。10.1007 / bf00818163。
- 58。
Zuker M:用于核酸折叠和杂交预测的MFOLD Web服务器。核酸RES。2003,31:3406-3415。10.1093 / nar / gkg595。
- 59。
Ambros V, Bartel B, Bartel DP等:microRNA注释的统一系统。RNA。2003年,9:277 - 10.1261 / rna.2183803。
- 60.
uShuffle:一种有用的工具,用于在保留k-let计数的同时对生物序列进行洗牌。生物信息学。2008,9:192-10.1186/1471-2105-9-192。
- 61.
Freyhult E,Gardner PP,Moulton V:RNA折叠措施的比较。BMC Bioinforma。2005,6:241-10.1186 / 1471-2105-6-241。
- 62.
Pérez-Quintero Al,Neme R,Zapata A,LópezC:植物microRNA和它们在防御病毒中的作用:生物信息学方法。BMC植物BIOL。2010,10:138-10.1186 / 1471-2229-10-138。
- 63.
张Y:MIRU:自动化植物miRNA目标预测服务器。基因。2005,33:701-704。
确认
该项目得到了DIB、quipu No . 201010011438和Colciencias(合同号221-2008)的部分资助。我们要感谢Adriana Bernal和LAMFU研究小组允许我们进入Xam他对脚本开发和数据分析的建议,以及西蒙Cortés对木薯淀粉生物合成的见解。
作者信息
隶属关系
通讯作者
额外的信息
作者的贡献
ALPQ:制备的植物材料,处理SRNA库数据并起草了手稿。AQ:参与miRNA预测和数据分析。OU:参与miRNA预测和数据分析。PV:进行半定量RT-PCR分析。CL:设想研究,参与其协调并帮助起草稿件。所有作者阅读并认可的终稿。
电子补充材料
Cassava Pre-MiRNAS描述
附加文件1:。基因组中的位置是基于木薯基因组计划2010提供的Cassava4版本的基因组,http://www.phytozome.net/cassava..用Vienna RNAFold计算了折叠能。(XLS 44 KB)
木薯microrna的目标
附加文件2:。米兰达预测在Cassava中确定的每种miRNA系列,预测来自来自Cassava 4版本的Cassava4版本的CASAVA 4版本2010年,http://www.phytozome.net/cassava..描述基于最相似的可用注释拟南芥或水稻基因。(XLS 78 KB)
作者的原始提交的图像文件
权利和权限
开放访问本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0.),允许在任何媒介上无限制地使用、分发和复制,但必须正确引用原作。
关于这篇文章
引用这篇文章
Pérez-Quintero,Á.L.,Quintero,A.,Urrogo,O.et al。木薯对轴突黄单胞菌(Xanthomonas axonopodis pv)感染差异表达mirna的生物信息学鉴定Manihotis。BMC植物杂志12,29日(2012年)。https://doi.org/10.1186/1471-2229-12-29
已收到:
公认:
发表:
关键字
- 转录因子结合位点
- miRNA目标
- microrna的家庭
- miRNA基因
- 反义链