摘要
背景
很少有人知道的潜力Brachypodium distachyon作为低温胁迫响应的模型。冰再结晶抑制蛋白(IRIP)基因、果糖基转移酶(FST)基因和许多C-repeat结合因子(CBF)基因在低温响应中具有特异性和重要意义。本文采用比较分析的方法研究了这些基因家族在植物中的保存和进化b . distachyon为了更好地了解其作为农业上重要的温带草的模式种的潜力。
结果
Brachypodium distachyon含有对寒冷敏感的IRIP基因Brachypodium特定基因家族扩展。在大肠杆菌中发现了一个大的冷应答CBF3亚家族b . distachyon而CBF4同源体在基因组中不存在。没有b . distachyonFST基因同源体编码典型的蒲竹科核心FST基序,低温诱导果聚糖积累差异显著b . distachyon与核心的蒲竹科物种相比。
结论
我们的结论是b . distachyon可以作为一个有趣的模型,用于研究在低温响应中涉及的特定分子机制。然而,与低温反应相关的关键基因的进化历史却不尽相同Brachypodium和核心种。这些差异限制了的使用b . distachyon作为农业核心种的整体研究模式。
背景
Brachypodium distachyon成为首个基因组完全测序的蒲草科草种[1].的Brachypodium属是小麦科(Triticeae)和禾草科(Poeae)的系统发育姐妹群,这为测序提供了令人信服的理论依据b . distachyon基因组来开发一个比水稻更适合温带草的模型(栽培稻)。b . distachyon具有典型模式植物的特征[2];它的高度较短(15-20厘米),有自交系,生活期一年一次(8 - 12周),是二倍体草基因组中最小的之一(约300 Mbp) [1],并可通过农杆菌属,介导转化[3.].不同的生态类型表现出对环境的一系列适应,这也是农业生产系统面临的重要挑战,例如开花时间的差异,春化要求[4],以及抗病能力[5].总之,这些特征使b . distachyon是研究蒲草科牧草农艺性状的适宜模式植物。
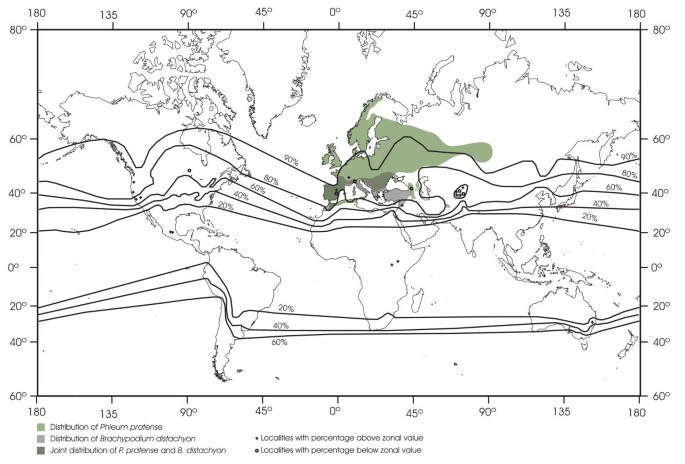
Pooideae亚科物种的一个特征是它们对温带生态系统的适应,这反映在Pooideae草的全球分布上[6)(图1)。Pooideae草的最近的共同祖先适应了热带或亚热带气候[7,8].Pooideae随后向较冷环境的辐射被认为与低温胁迫机制的进化有关[9].因此,Pooideae对较冷气候的适应使该类群成为研究植物适应性进化的理想模型系统[10].然而,在对寒冷和冰冻胁迫的耐受性方面,蒲竹科存在较大的种内变异。一些蒲葵科的种(例如:Phleum pratense)可以忍受极端的冬季气候,其物种分布范围包括亚北极地区(图1),而b . distachyon另一方面,不适应极端冬季气候,这反映在中东和地中海的地理分布(图1)。
系统发育研究表明b . distachyon大约在3500万年前从核心的Pooideae中分离出来[2]而pooideae对寒冷气候的关键适应是在始新世-渐新世冷却期(34-26亿年)进化的b . distachyon-core Pooideae split [9].如果这是正确的,那么在寒冷和冰冻压力中共享的祖先分子机制可能是不同的b . distachyon小麦科和波伊族的重要农业物种(以后称为核心波伊科)。
草属植物在世界范围内的分布,占总草区系的百分比。的原生分布Brachypodium distachyon和典型的凉爽季节的禾本科牧草,Phleum pratense,以两种不同的绿色阴影表示。分布数据来自Hultèn和Fries [11]p . pratense,及欧陆[12], Filiz等。[13]和来自全球生物多样性数据门户网站(data.gbif.org, 2011-11-08)的数据b . distachyon.此数字(不含b . distachyon而且p . pratense分布)改编自Hartley [6]经CSIRO出版许可。Pooideae在Hartley中被引用为Festucoideae [6]原始论文。
在低温胁迫耐受性方面,已经很好地描述了三个pooideae特有的遗传特征;(1)冰再结晶抑制蛋白(IRIPs),(2)果糖基转移酶基因(FSTs), (3) c重复序列结合因子(CBF)家族的扩增。IRIPs被认为可以通过限制外质体空间中的冰晶生长来减少冰形成过程中的细胞损伤[14- - - - - -16],这一过程被称为冰晶再结晶。Pooideae的祖先IRIP基因被认为是从一种llr蛋白激酶进化而来的。17]通过获得冰结合域[16]通过重复主题的扩展(NxVxG/NxVxxG) [18].在核心种中,广泛的基因重复产生了较大的IRIP基因家族[18,19].fst可将蔗糖分子转化为果聚糖[20.,21]而低温胁迫诱导桃科核心种FST基因表达和果聚糖积累[22- - - - - -24].将FSTs引入缺乏内源FST途径的植物,以及在Pooideae核心种中过表达内源FSTs均已显示出提高抗冻性[25,26].CBFs是在干旱和低温胁迫下调节基因组的转录因子[27- - - - - -29].在Pooideae中,有两个CBF亚家族CBF3和CBF4经历了谱系特异性复制[30.这些cbf3 /4亚家族的成员被认为在Pooideae的冻结胁迫中起作用[31- - - - - -33].
尽管IRIP、FST和Pooideae的CBF3/4基因已经在核心的Pooideae物种中进行了非常详细的研究,但对这些基因的同源性进行系统的研究b . distachyon是缺乏的。在这项研究中,我们问了一个问题;IRIP、FST和Pooideae特定CBF基因之间的保守程度如何b . distachyon以及重要的农业核心种?我们通过采用一套包括比较基因组学、基因表达分析和碳水化合物代谢表征的方法来回答这个问题。我们的目的是(1)评估的使用b . distachyon(2)提高我们对冷胁迫响应演化的认识。
方法
用于基因表达研究的植物材料、生长条件和组织取样
四个二倍体近交系b . distachyon采用实时定量聚合酶链式反应(qRT-PCR)对冷诱导IRIP基因的表达进行表征,以Bd1-1、Bd21-1、Bd1-1和Bd29-1为对照。种子由美国明尼苏达大学的David Garvin博士提供。Bd3-1和Bd21-1来自伊拉克,是春季基因型,不需要春化来诱导开花。Bd1-1和Bd29-1分别来自土耳其北部和乌克兰,属冬季基因型,需要较长的春化期(分别为6周和12周)才能开花(http://www.ars.usda.gov/SP2UserFiles/person/1931/GarvinLabCoreBrachypodiumdistachyonLineSet (2) . pdf) [34,35].微阵列基因表达实验只使用Bd21-1。
短句来源qRT-PCR实验以种子为材料,在昼夜温度20/16℃、光周期16 h、光子通量密度150 μmol m的条件下,培养7周龄植株-2年代-1.一半的植物按照Alm等人概述的程序进行冷驯化(CA)。36]除预驯化在光周期12 h,昼夜温度12/6℃下进行1周外。在恒温1°C、光周期14 h的CA条件下保存。在预驯化期开始时采集对照植物(未驯化的NA)的叶片组织,在冷驯化期开始后4 h、1 d和10 d采集CA植物的叶片组织。为了避免基因表达的昼夜时钟调节带来的实验偏差,所有样本都在每个时间点的同一天(上午)收集。在每个时间点从每个基因型的不同植物中取样用于RNA提取的组织。所有取样的叶片组织立即在液氮中冷冻,并保存在−80°C。
在微阵列实验中,植物在受控生长室中在16小时光周期下生长。温度为23℃,光子通量密度为200 μmol m-2年代-1.冷实验以3周龄植株为研究对象,在4℃、光子通量密度为200 μmol m的步入式冷室中进行-2年代-1.对照植物在23°C的环境控制生长室中。实验治疗开始于黎明后两小时(上午10点)。在实验开始后1、2、5、10和24小时,从单株植物中收集叶片和茎(地上组织总数)。
的识别Brachypodium distachyonirip同源物和para同源特异性引物设计
Brachypodium distachyonIRIP同源物通过基于网络的爆炸搜索(http://www.Brachypodium.org)的IRIP基因多年生黑麦草(AY968588;EU680848;EU680850;EU680851)作为查询。在MAFFT web服务器上使用默认设置对翻译的IRIP基因进行多次比对[37来验证b . distachyonIRIP基因包含典型的NxVxG/NxVxxG-repeat冰结合域。使用primer3设计IRIP-paralog特异性引物[38]使得每个引物对中至少有一个引物5 '端irip -谬误序列不匹配。通过TOPO TA克隆试剂盒(Invitrogen)克隆paralog特异性引物对的pcr产物,随后对5 ~ 10个克隆进行测序,验证了IRIP的paralog特异性。使用primer Express Software (Applied Biosystems)设计用于IRIP基因定量逆转录酶PCR (qRT-PCR)的最终TaqMan MGB探针和引物集。
qRT-PCR基因表达分析
总RNA用RNeasy植物微型试剂盒(Qiagen)分离,RNA提取按照制造商的方案进行,使用100mg冷冻组织粉(用液氮砂浆研磨)。DNA酶消化用于消除基因组DNA污染。RNA质量由Agilent 2100生物分析仪(Aglilent Technologies)控制,RNA数量由Nanodrop ND-1000紫外可见分光光度计(Nanodrop Technologies)测量。2.5 μg总RNA采用SuperScriptVilo cDNA合成试剂盒(Invitrogen)反转录。在qRT-PCR中,我们使用了EXPRESS两步qRT-PCR通用试剂盒(Invitrogen)和上标VILO cDNA合成试剂盒(Invitrogen)。
每次qRT-PCR反应使用2 μl cDNA,总反应量为20 μl。引物浓度为0.5 μM, TaqMan探针浓度为0.2 μM。最终的ROX参比染料为0.05 μM。转录水平分析使用ABI7500实时PCR仪(应用生物系统公司)与快速循环程序;95°C持续20秒,40个循环,95°C持续3秒,60°C持续30秒。以甘油醛3-磷酸脱氢酶(GAPDH)为内参基因。制作标准曲线以控制引物和探针对的效率接近100%。b . distachyon使用比较阈值循环法(ΔCt法)计算IRIP基因相对于GADPH基因的转录水平。使用三个生物重复(来自三种不同植物的叶片样本)来估计表达水平。2的均值和标准差——ΔΔCt计算CA与NA样品的相对表达量。一个t-test用于检测CA样本ΔCt值与NA样本是否存在显著差异(即冷诱导基因表达)。对每个基因的qRT-PCR板进行三次无模板对照,以控制引物二聚体的形成和DNA污染。
微阵列表达分析
叶片组织在液氮中研磨,使用RNA植物试剂(Invitrogen)和RNeasy试剂盒(Qiagen)提取细胞总RNA,并如前所述使用无rnase - DNase处理[39].RNA完整性使用安捷伦生物分析仪进行评估。使用NuGen掌声WT-Amp PlusST RNA扩增系统Kit协议(Cat# 5510-24)和Encore Biotin模块V2 (Cat# 4200-12)从125 ng总RNA样本中制备标记目标cDNA。每个样本约4.55 μg cDNA片段杂交18小时,得到一个AffymetrixBrachypodium基因组阵列(BradiAR1b520742)。使用genchip®fluics Station 450进行杂交。使用带自动装药器的GeneChip®扫描仪3000在570 nm处扫描阵列,并根据标准Affymetrix协议进行质量控制(Affymetrix genchip®表达分析技术手册,701021 Rev. 5;http://www.affymetrix.com)在俄勒冈州立大学基因组研究和生物信息学中心,中央服务实验室(详细协议可在http://www.cgrb.oregonstate.edu/)。采用AGCC 3.0版本软件进行图像处理和数据提取。采用Affymetrix真核杂交控制试剂盒和Poly-A RNA控制试剂盒,确保杂交和cDNA扩增的效率。同时合成冷胁迫处理和对照样品的所有cdna,同时进行微阵列杂交。每个阵列图像都经过视觉筛选,以剔除信号伪影、划痕或碎片。
探针水平归一化是利用Affymetrix Power Tools (APT)软件包(http://www.affymetrix.com/partners_programs/programs/developer/tools/powertools.affx;[40])。使用Affymetrix的apt-probeset- summary(1.14.3)程序对每个基因进行探针集总结和表达估计。数据操作使用Perl脚本来计算规范化处理和控制探针集值之间的折叠变化。
系统发育分析
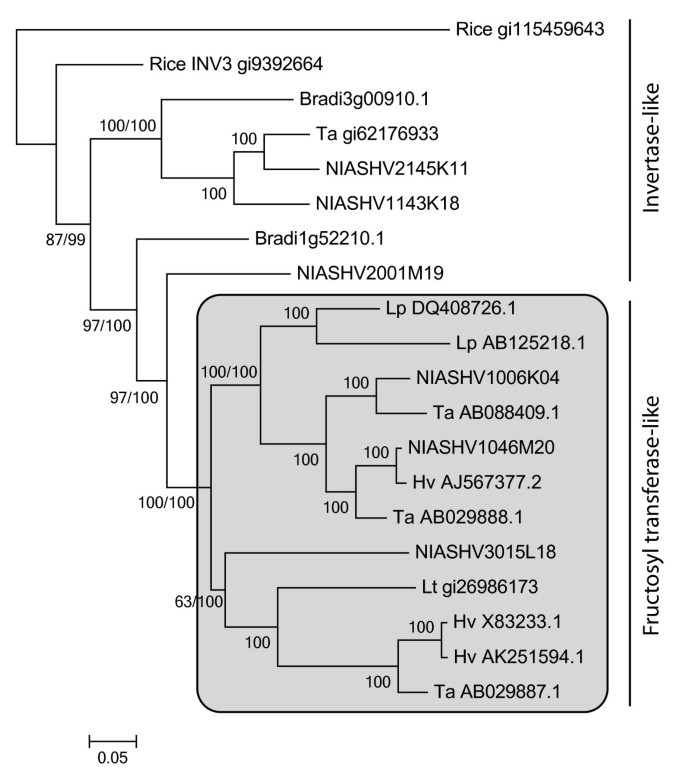
IRIP、FST和CBF基因家族包含新近衍生的同源同源(即很少有取代),因此所有的系统发育分析都是在核苷酸水平上进行的。从不同来源收集了蒲竹科核心种IRIP、FST和CBF3/CBF4基因的编码序列(CDS)。对于IRIP系统发育,从NCBI下载了来自Triticeae和Poeae tribe物种的具有代表性的cd集,并与已识别的cd集合并b . distachyonIRIP同系物(见上文)。鉴定CBF3/4和FST同源物b . distachyon, CDS (v1.2)注释从(http://ftp.brachypodium.org/files/Annotation/),并使用Skinner等人鉴定的CBF3/4基因进行局部爆炸搜索。[41]作为查询。利用小麦科CBF3/4基因构建了CBF3/4系统发育[41, Poaea [42]和米[30.].从大量的全长(fl) cDNA中收集了额外的大麦CDS序列,并用于FST系统发育的分析。从fl-cDNA序列用orfpredictor (http://proteomics.ysu.edu/tools/OrfPredictor.html)使用blastx搜索的同源信息与水稻、玉米、高粱和b . distachyon下载地址:ftp.plantbiology.msu.edu、ftp.brachypodium.org、ftp.maizesequence.orgftp://ftpmips.helmholtz-muenchen.de/plants/sorghum/,分别。只有爆炸值<1e的目标序列-10纳入进一步分析。利用MAFFT对IRIPs和cbf进行多序列比对[37]使用密码子模型在指南web服务器(http://guidance.tau.ac.il) [43].每个引导比对计算100个自举重复,序列分数低于0.6和列分数低于0.8的序列自动从比对中删除。FST对齐是由prankster [44]这些氨基酸残基被翻译回密码子进行系统发育分析。所有对齐都在BioEdit中手动检查和编辑[45].
在Treefinder中根据赤池信息准则(Akaike Information Criterion, AIC)选择最佳进化模型进行最大似然(Maximum Likelihood, ML)系统发育重建[46].ML系统发育重建树与MrBayes估计的替代贝叶斯系统发育重建树的一致性进行了检查[47].MrBayes分析采用的模型为GTR + G + I (lset nst = 6 rates = invgamma),模拟时间为1 × 105或1 × 106每100或1000代进行一次树采样。在所有分析中,分裂频率之间的平均偏差<0.01,并且使用排除一半样本树的“burn in”来生成共识树。系统发育树在MEGA v5中可视化[48].
噬菌体文库的构建与筛选
利用自交系二倍体Bd3-1的DNA构建噬菌体lambda基因组文库。Bd3-1基因组DNA是美国明尼苏达大学David Garvin博士的一份厚礼。用编码C-Repeat结合因子(CBF)/脱水响应元件结合蛋白(Os-DREB1A)的水稻cDNA探针和编码Os-DREB1B的探针对该文库进行筛选[49].DREB1A是CBF3亚家族CBF,而DREB1B是CBF4亚家族CBF。与Os-DREB1A杂交的克隆与Os-DREB1B杂交,未发现额外的Os-DREB1B杂交克隆。根据限制性内切酶的模式,所有的克隆都可以分为两类。对2个代表性克隆进行测序,并将这些序列存入GenBank(株系JQ180470和JQ180471)。
果聚糖测量
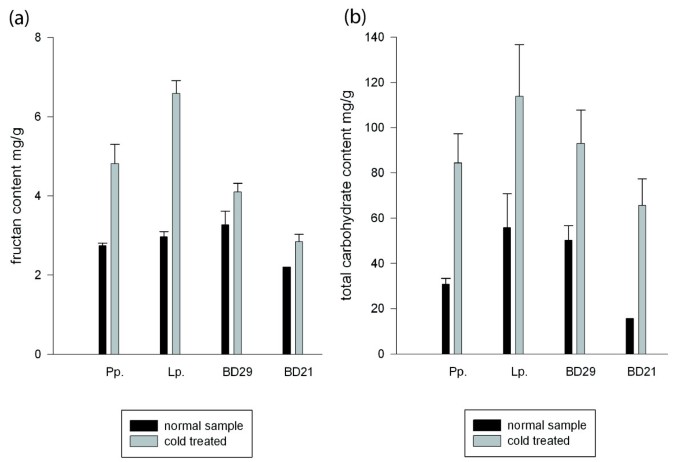
我们测量了其中的总碳水化合物和果聚糖含量b . distachyon春季型(21-1)和冬季型(29-1),在核心种黑麦草.(多年生黑麦草)和白杨;(蒂莫西)在冷驯化前后。在光周期为16 h的条件下,在温室内各种植8株。8周后,一半的植物被放置在2℃的冷室中,另一半则被保存在温室中作为对照。冷处理和对照植物的叶片组织在四天后收获,并在−80°C保存。总碳水化合物和果聚糖的提取如Thorsteinsson等所述。[50].对于比色定量,我们对波洛克[Pollock]中描述的方法作了轻微修改。51];因为levan是草类中主要的单子果聚糖[52,53我们选用了果聚糖标准品,而不是菊粉。不同浓度(0.2,0.4,0.6,0.8,1,2,3,4,和5毫克毫升-1)分别作标准曲线。
果聚糖样品采用高效阴离子交换色谱法(HPAEC)在Dionex ICS3000系统(Dionex Corp.)上进行分析。每种滤液各2 μl注入CarboPac PA1色谱柱(2x250 mm分析柱,2x50 mm保护柱),30℃,0.25 ml min-1分析用脉冲安培检测(PAD)进行检测。分析物分离得到运用梯度洗脱液的氢氧化钠(100毫米)和B (1.0 NaOAc 0.1 M氢氧化钠)从100%洗脱液开始,紧随其后的是一个两分钟的线性梯度洗脱液5% B,然后增加到25%洗脱液B在23分钟,最后增加到50%洗脱液B达到45分钟保持三分钟。回到初始条件获得的列整理在一分钟保持10分钟。采用以下外部标准进行峰值识别;levan (Sigma-Aldrich),果糖(Sigma-Aldrich),葡萄糖(Sigma-Aldrich),蔗糖(Sigma-Aldrich),以及一组低聚果糖(1-Kestose, Nystose和1-Fructofuranosylnystose) (Wako Chemicals)。
结果与讨论
冷反应的IRIP基因在鹅科谱系中很早就进化了
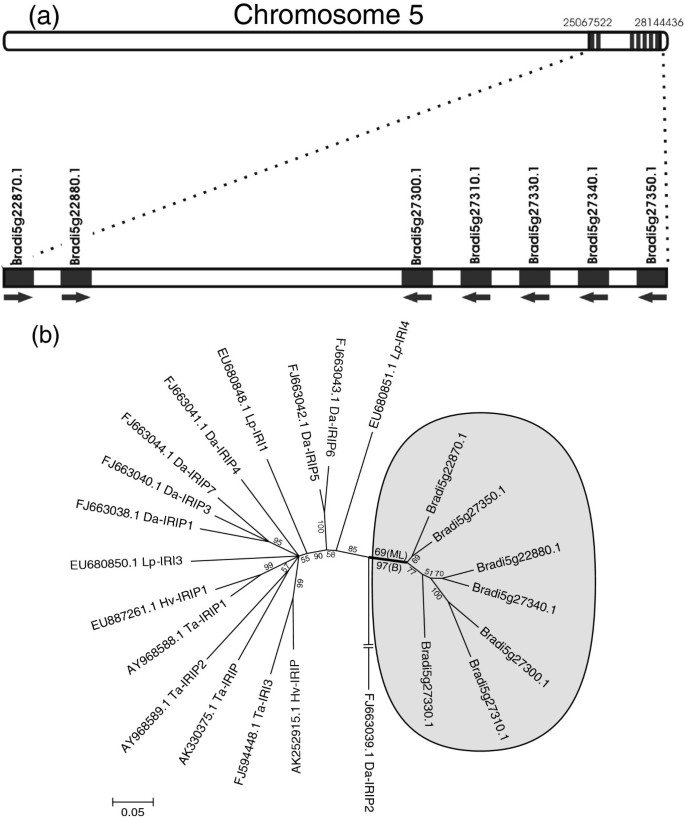
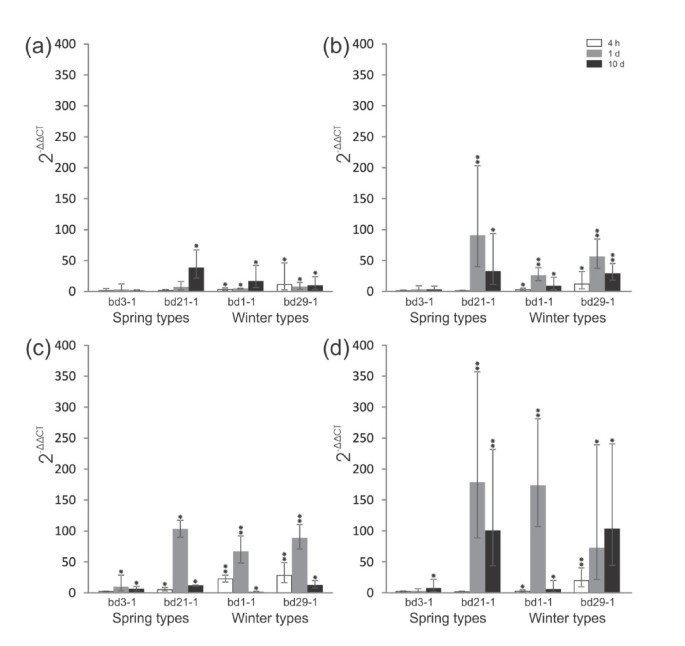
七个Brachypodium distachyon在第5号染色体上鉴定出两个包含2个和5个基因的基因簇中的irip样基因(图2a).近端聚类中的Bradi5g22870.1和Bradi5g22880.1都有截断的冰结合域(附加文件1)。测试是否b . distachyonIRIPs是低温诱导的,为4个具有非截断冰结合结构域的IRIP序列设计了特异qRT-PCR引物(Bradi5g27300.1, Bradi5g27310.1, Bradi5g27330.1, Bradi5g27350.1)1)。在两种冬季类型和21-1系冷驯化1天后,除bradi5g273001外,所有基因均表现出较强的冷诱导3.)。bradi5g273001在3个基因型中均被冷诱导,但表达水平普遍低于其他IRIP基因(图3.,附加文件2)。芯片实验的表达数据证实了qRT-PCR实验中观察到的IRIP基因冷诱导的一般模式;bradi5g273001很少或没有冷诱导,其他IRIP基因在24小时内冷诱导中至强(2.5-25倍)2)。
7个IRIP基因的定位和分布Brachypodium distachyon在第5染色体上b . distachyonIRIP基因系统发育。(一)七的位置Brachypodium distachyon5号染色体上的IRIP基因。(b)poideae核心种的一个IRIP序列子集和全部7个IRIP序列的50%一致性ML系统发育(模型J2 + G)b . distachyon基于100个自举重复的IRIP同源物。来自替代贝叶斯系统发育(1.000.000代)的支系支持与ML引导支持一起显示在定义b . distachyonIRIP基因为单系。种的缩写如下:Lp,多年生黑麦草;哒,Deschampsia南极洲;高压,大麦芽;助教,小麦。
qRT-PCR表达值的大标准差掩盖了表达差异的详细分析,然而IRIP表达数据中一个有趣的模式是明显的。在我们有限的数据集中,冬季类型比春季类型表达IRIPs更快。对于四种IRIP基因中的三种,只有冬季型在4小时具有显著的p值(图3.a-b,d),而第四个IRIP基因(图3.c)与Bd21-1相比,在冬季型中4小时表达量更高。在微阵列实验中,Bd21-1在CA处理早期(<5小时)也观察到低IRIP基因诱导的模式(表2)2)。因此,一种假设是冬季型比春季型有更快的IRIP基因冷诱导。必须指出的是,需要更多不同开花习性的系的表达数据来验证这一假设。
发生的善意的IRIP基因b . distachyon,包含保守的冰结合域,具有冷响应性,IRIP的进化早于Pooideae的进化Brachypodium-核心Pooideae发散。此外,系统发育分析支持单系起源b . distachyonIRIP基因(图2b)这意味着独立的IRIP基因家族发生了扩张Brachypodium从核心蒲竹科物种分化后的谱系。
Bd3-1的低IRIP诱导可由极端的弹簧型习惯解释
与其他品系相比,Bd3-1弹簧型的IRIP冷诱导显著降低,只有3个qRT-PCR检测结果显著高于NA样品(图3.)。由于弹簧型Bd21-1具有较强的低温诱导IRIP基因表达,Bd3-1的IRIP表达表型与弹簧型生命策略本身不相关。最近的春化响应研究表明,Bd3-1是一种快速开花的春季类型,在生命周期早期大量表达促花基因VRN1和VRN3。事实上,与Bd21相比,未春化的Bd3-1幼苗的两个VRN1旁系中的一个和VRN3的表达水平分别约高4倍和6倍[4].谷物从营养生长阶段向生殖生长阶段的生理转变与VRN1的诱导、低温诱导基因表达的抑制和抗冻性的丧失有关[54].因此,在Bd3-1中观察到的非常低的IRIP基因表达可能与该系从营养生长形式到开花的非常快速的转变有关[4].
Brachypodium distachyon有一个大的冷反应CBF3家族,但缺乏CBF4基因
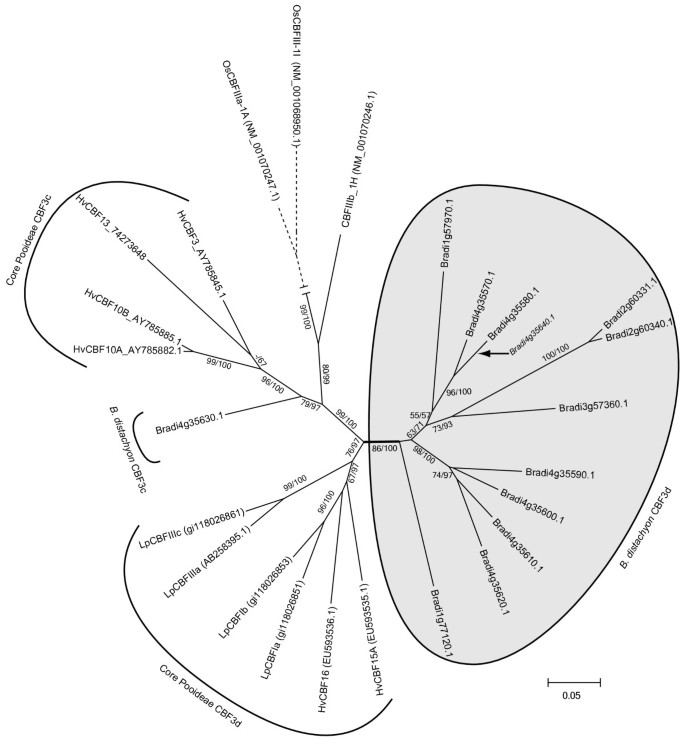
18人中的14人b . distachyon根据对所有CBF同源物的系统发育分析,在blast搜索中鉴定出的CBF3/4基因同源物可归类为CBF3成员(附加文件3.)。这些基因中有13个属于CBF3c/d支。图4中CBF3c/d基因的系统发育b . distachyon和两个核心种。一个b . distachyonBradi4g35630.1基因属于CBF3c亚群,其他12个基因属于CBF3d亚群。CBF3c (Bradi4g35630.1)基因成员和大多数CBF3d基因b . distachyon属于4号染色体上的一个基因簇。该染色体在很大程度上与麦类第5染色体同线[1],其中含有低温诱导小麦和大麦CBF3c/d基因簇[55,56].微阵列表达数据显示,在冷暴露24小时内,除了两个CBF3d基因(Bradi1g57970.1和Bradi3g57360.1)外,所有基因都被冷诱导(>2倍)(表2)2)。结合系统发育分析,该表达数据表明,在此之前,冷反应CBF3c和CBF3d基因都存在于Pooideae的祖先中Brachypodium核心Pooideae散度。
万物的单系起源b . distachyonML和贝叶斯系统发育重建都支持CBF3d平行谱(图4)。此外,在2号染色体和3号染色体上发现了几个CBF3d成员(图4),这并不符合麦类5的共统关系[1].综合来看,这些数据显示了广泛的影响b . distachyonCBF3d基因的特定重复,包括串联的和与其他染色体的,即使其他场景可以解释观察到的CBF3d拓扑结构。首先,看起来是一个b . distachyon-特异性CBF3d进化枝可能在Brachypodium散,后来迷失在核心Pooideae。其次,基因转换可能使基因序列同质化,并造成一种进化亲缘关系的“幻觉”[57].的所有序列正交b . distachyonCBF3d基因可能尚未在Pooideae核心种中被发现。
令人惊讶的是,13个都没有b . distachyon在爆炸分析中鉴定的CBF同源物属于CBF4组基因(附加文件3.)。筛选噬菌体lambda基因组文库b . distachyonCBF基因含量也未能恢复CBF4同源体b . distachyon.2个噬菌体克隆λBd1C (JQ180470)和λBd5D (JQ180471)分别含有4个和3个CBF3.与Bd21-1基因组比较,λBd1C和λBd5D分别对应于Bd21-1 4号染色体上的两个区域。两个克隆所包含的序列与bd21 -1序列在其整个长度上共线。由于水稻中含有一个CBF4基因(OsCBF4 AY785894),最简约的模型解释了CBF4在水稻中的缺失b . distachyon是谱系特异性基因丢失。
Brachypodium distachyon缺乏FST基因,冷胁迫下果聚糖积累与核心种不同
共鉴定出15个与Pooideae核心FSTs有一定同源性的基因。其中13个基因与水稻中最接近的FST同源基因(一种类似反转酶的基因)的亲缘关系更远,因此在分析中没有考虑到。鹅科FST核心基因编码诊断性h(a /G)Y/F基序[58]和由低温引起的[59,60].两个类似fst的同系物都不是b . distachyon编码fst基序,但只是转化酶基序(图5)。与FSTs最遥远的同源物(Bradi3g00910.1)是Bd21中冷诱导的2),属于一个单系的倒置类基因簇,包含来自核心的Pooideae种的基因成员(图5)。Bradi1g52210.1在系统发育中被放置在离FSTs最近的位置,但Bd21-1在低温处理24小时内没有被冷诱导(表2)2)。已知一些fst基因仅在CA的几天后才被寒冷诱导[60],但不幸的是,我们没有实验数据b . distachyon超过24小时的冷敷。因此,Bradi1g52210.1也有可能在CA过程的后期被诱导。
种系发生分析Brachypodium distachyonFST和转化酶基因与Pooideae核心FST和转化酶基因比较。poideae核心种FST基因50%的ML系统发育(GTR + G + I模式)一致,b . distachyon基于Lasseur等人的诊断性氨基酸基序W(A/S/G)W和wmnndpng, FST同源物和两个水稻倒置酶样基因被分组为倒置酶样和果糖转移酶样。[58].物种的缩写是:Os,栽培稻;Lp,多年生黑麦草;Lt,Lolium temulentum高压,大麦芽;助教,小麦.名为“NIASHv”的基因来自大麦全长cdna数据库。ML引导和贝叶斯支持值表示为ML/贝叶斯。如果相同,则只显示一个值。“-”表示支持值低于50。
FST的增加可能是通过液泡转化酶基因底物结合位点的突变而进化的[61].低温诱导水稻液泡转化酶[62],在本研究中,液泡转化酶同源物的表达b . distachyon(Bradi3g00910.1)被证明是低温诱导的。因此,祖先的FST基因很可能在FST功能进化之前就对寒冷做出了反应。观察到FST分支外最接近的同源基因是大麦基因(NIASHV2001M19),这支持了在Pooideae核心群从分裂后FST功能获得的进化模型Brachypodium.然而,一个不太吝啬的FST基因(s)丢失的情况Brachypodium也可以解释系统发育结果,因此不能排除。与核心Pooideae的FST增加相比,在Brachypodium’场景将需要更多的进化变化(即2个变化)来与图中的拓扑保持一致5;除了FST缺失外,在Pooideae谱系中还必须发生多次FST增加,或者NIASHV2001M19大麦基因必须从FST基因恢复到反转类基因。
测试是否b . distachyon测定冷驯化前后累积果聚糖对低温的响应,测定总碳水化合物和果聚糖水平。两种核心种和b . distachyon在冷胁迫下碳水化合物的积累有明显的反应(图6),正如许多植物物种所显示的那样[63- - - - - -67].有趣的是,冷诱导的果聚糖含量在桃心科中增加了0.8 ~ 1.2倍b . distachyon(0.2 - -0.3折)。HPAEC定性分析证实两者均有效b . distachyon而且l为诱导果糖、葡萄糖和蔗糖在低温下大量增加。但是,短寡糖的剖面堆积在b . distachyon与核心Pooideae不同(附加文件4)。例如,在冷驯化后,酮糖,日乳糖和其他不明寡糖的显著水平l为但不能在寒冷中适应b . distachyon(附加文件4a).此外,在经过冷处理的核心Pooideae中,具有更高聚合度(DP)的糖的存在程度也要大得多b . distachyon(附加文件4b)冷诱导的果聚糖含量变化与赋予果聚糖转移酶活性的氨基酸基序存在/不存在的系统发育聚类的相关性支持这一观点b . distachyon不具有Pooideae核心FST酶的同源物(图5)。
果聚糖合成植物的分布倾向于以间歇性干旱和低温胁迫为特征的生态系统[68].此外,两者都相关[69- - - - - -72]和转基因研究[73为果聚糖在蒲草科核心草抗旱性和抗寒性中的重要作用提供了有力证据。与冷胁迫相关的果聚糖积累在历史上被认为与植物为冬季准备时容易获得的能量储备储存有关[74].然而,功能研究的结果为果聚糖在非生物应激保护中的更直接作用提供了深入的认识,作为冻结应激期间细胞膜脂质双层稳定性增强复合物的一部分[75- - - - - -77].因此,在Pooideae的核心祖先中,FST功能的进化,以及随后这种酶家族的增加和多样化,对于适应非生物压力水平增加的环境(如更冷的生态系统)很重要。
结论
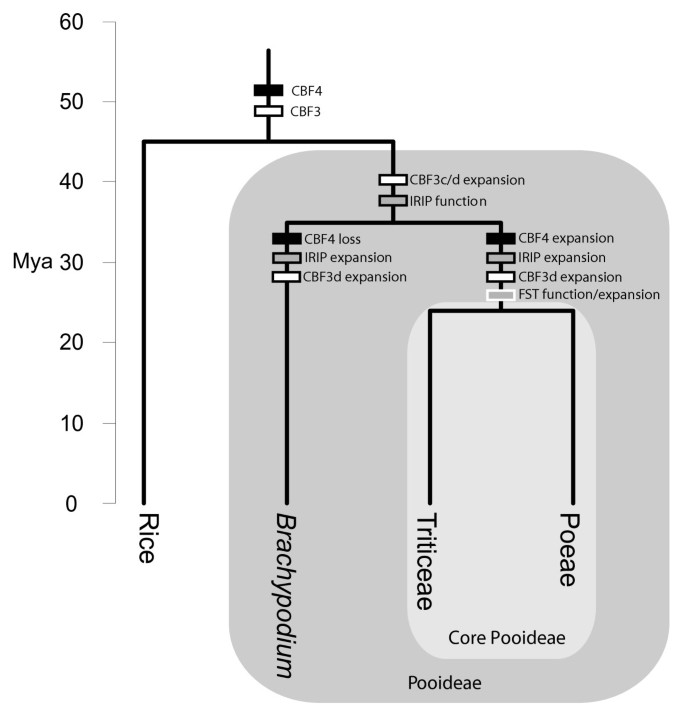
从我们的比较分析中可以明显看出b . distachyon和核心Pooideae在关键冷胁迫途径上存在差异(图7)。尽管这种差异限制了的使用b . distachyon作为Pooideae核心物种低温胁迫分子生物学的整体模型,b . distachyon将有助于研究特定的基因和通路,如CBF3或IRIP基因。例如,IRIP RNAi敲除/敲除系可用于测试IRIP基因功能对Pooideae抗寒和抗冻性的重要性,IRIP启动子缺失构建将有助于阐明IRIP的转录调控。b . distachyon这也有助于了解不同cbf基因家族(如CBF3d和CBF3c组基因)之间的功能差异,从而增加我们对蒲竹科冷胁迫应答的转录控制的总体理解。由于CBF3c基因存在于单个拷贝中b . distachyon这可能有助于功能分析和该基因在机械水平上影响的途径。
总结了蒲竹科与水稻分化后CBF3/4、IRIP和FST基因的进化关键步骤。进化步骤分为三类,“功能”(即基因功能的改变),“扩展”(即基因家族的扩展),或“损失”。绝对时间刻度基于[18].
之间的差异b . distachyon和核心部分也揭示了有趣的,可能具有生物学意义的线索,以了解Poaceae的冷驯化和耐寒性的进化和功能。在果聚糖生物合成中编码酶的基因的缺失提供了一个独特的机会来进行研究,使用转基因技术,来检验关于果聚糖代谢进化与草类适应非生物胁迫的关系的假设。
了解控制物种内气候适应的潜在遗传因素对于培育作物非生物胁迫抗逆性具有重要意义。我们的研究表明,不同植物对冷胁迫的转录反应存在很大差异b . distachyon行。在冷驯化过程中,Bd3-1弹簧型在所有时间点的IRIP表达水平均显著降低(图3-1)3.),与研究中的其他bd系相比。这种“冷无反应性”可能与开花途径基因bd1 -1的早期表达有关[4].因此,Bd3-1可能是一个有趣的模型,用于研究向开花(生殖期)的过渡如何与CA途径相互作用。此外,冬季型和春季型IRIP基因的转录反应时间不同。这些差异可能与当地对气候条件的适应有关,因此提供了一个有趣的模型系统来研究一般群体的差异和对冷胁迫反应的适应。由于主要的转录调节途径在高度不同的物种中是保守的,关于局部适应机制的知识b . distachyon种群具有可转移性和农业价值,是重要的蒲草科作物。
参考文献
- 1.
国际短肢草计划:模式草短肢草的基因组测序和分析。自然科学学报,2010,38(4):344 - 344。10.1038 / nature08747。
- 2.
Bevan MW, Garvin DF, Vogel JP:短柄藻基因组学的可持续粮食和燃料生产。生物技术学报,2010,21(2):211-217。10.1016 / j.copbio.2010.03.006。
- 3.
刘志刚,刘志刚,刘志刚,等。农杆菌介导短柄藻自交系Bd21-3的高效转化。植物细胞学报,2008,27(3):471-478。10.1007 / s00299 - 007 - 0472 - y。
- 4.
Schwartz CJ, Doyle MR, Manzaneda AJ, Rey PJ, Mitchell-Olds T, Amasino RM:短柄草开花时间和春化反应的自然变异。生物能源学报,2010,3(1):38-46。10.1007 / s12155 - 009 - 9069 - 3。
- 5.
Draper J, Mur LAJ, Jenkins G, Ghosh-Biswas GC, Bablak P, Hasterok R, Routledge APM:草本植物功能基因组学的新模式系统。中国植物学报,2001,27(4):379 - 379。10.1104 / pp.010196。
- 6.
Hartley W:禾本科的起源、进化和分布研究。v .羊毛草亚科。地球物理学报,1997,21(2):201-234。10.1071 / BT9730201。
- 7.
Barker NP, Clark LG, Davis JI, Duvall MR, Guala GF, Hsiao C, Kellogg EA, Linder HP,禾草系统发育工作组G:禾草(禾草科)的系统发育和亚科分类。中国机械工程学报,2001,32(3):373-457。10.2307 / 3298585。
- 8.
Stromberg CAE:草和草地生态系统的进化。地球科学,2011,39:517-544。10.1146 / annurev -地球- 040809 - 152402。
- 9.
Sandve SR, Fjellheim S:始新世-渐新世边界气候变冷期间基因家族的扩张是否在pooideae适应寒冷气候中发挥了作用?分子生态学报,2010,19(10):2075-2088。10.1111 / j.1365 - 294 x.2010.04629.x。
- 10.
杨春华,李志刚,王志刚,王志刚,王志刚。多年生草本植物抗冻性的分子机制研究。中国植物科学,2011,18(1):69-77。10.1016 / j.plantsci.2010.07.011。
- 11.
胡腾E, Fries M:北欧维管植物图集-北回归线以北,卷3。Königstein,德意志联邦共和国:KoeltzScientific Books;1986.
- 12.
Tutin TG: CXCIII禾本科。在欧洲植物科。编辑Tutin DHH TG,Burges NA, Moore DM, Valentine DH, Walters SM, Webb DA。剑桥:剑桥大学出版社;1980:118 - 267。
- 13.
菲利兹E, Ozdemir BS,布达克F, Vogel JP,金枪鱼M,布达克H:短柄藻不同自交系的分子、形态和细胞学分析。中国生物医学工程学报,2009,52(10):876-890。10.1139 / g09 - 062。
- 14.
张超,费淑珍,Arora R, Hannapel D, Hannapel D:多年生黑麦草冰再结晶抑制蛋白增强抗冻性。植物生态学报,2010,32(1):1 - 5。10.1007 / s00425 - 010 - 1163 - 4。
- 15.
John UP, Polotnianka RM, Sivakumaran KA, Chew O, Mackin L, Kuiper MJ, Talbot JP, Nugent GD, Mautord J, Schrauf GE,等:南极耐冻毛草的冰再结晶抑制蛋白(IRIPs)及其抗冻性。植物细胞环境,2009,32(4):336-348。10.1111 / j.1365-3040.2009.01925.x。
- 16.
Sidebottom C, Buckley S, Pudney P, Twigg S, Jarman C, Holt C, Telford J, McArthur A, Worrall D, Hubbard R,等:植物化学:草的热稳定抗冻蛋白。自然科学,2000,40(6):357 - 357。10.1038 / 35018639。
- 17.
Tremblay K, Ouellet F, Fournier J, Danyluk J, Sarhan F:谷物冷调节冰再结晶抑制蛋白的分子特征及其来源。植物细胞物理学报,2005,46(6):884-891。10.1093 /卡式肺囊虫肺炎/ pci093。
- 18.
杨晓东,王晓明,王晓明,王晓明,等:耐寒草低温胁迫相关基因家族的进化。中国生物医学工程学报,2008,29(4):344 - 344。
- 19.
Kumble KD, Demmer J, Fish S, Hall C, Corrales S, DeAth A, Elton C, Prestidge R, Luxmanan S, Marshall CJ,等:黑麦草冰活性蛋白家族的特征分析。低温生物学,2008,57(3):263-268。10.1016 / j.cryobiol.2008.09.005。
- 20.
Sprenger N, Bortlik K, Brandt A, Boller T, Wiemken A:大麦中合成果聚糖的关键酶蔗糖-果聚糖6-果糖转移酶的纯化、克隆及功能表达。美国国家科学院。1995, 92(25): 11652-11656。10.1073 / pnas.92.25.11652。
- 21.
Smeekens S, Vijn I:果糖:不仅仅是一种储备碳水化合物?植物营养学报,2004,27(2):359 -359。10.1104 / pp.120.2.351。
- 22.
Livingston DP, Knievel DP, Gildow FE:燕麦中果聚糖的合成:1。低聚物在茎冷硬化过程中的积累及其在粗酶提取物中的体外合成。植物学报,1994,27(1):27-36。10.1111 / j.1469-8137.1994.tb04256.x。
- 23.
饶rsp, Andersen JR, Dionisio G, Boelt B: 3个草甸早熟禾品种冷驯化过程中果聚糖积累及候选基因转录。植物营养学报,2011,26(4):344-351。10.1016 / j.jplph.2010.07.019。
- 24.
Kawakami A, Yoshida M:冬小麦冷硬过程中蔗糖1-果糖基转移酶和蔗糖6-果糖基转移酶的分子特征。中国生物工程学报,2002,26(4):457 - 457。10.1271 / bbb.66.2297。
- 25.
川上A,佐藤Y,吉田M:合成果聚糖和增强耐冷性水稻的基因工程研究。应用实验学报,2008,59(4):793-802。10.1093 / jxb / erm367。
- 26.
Yamada T, Hisano H, Kanazawa A, Kawakami A, Yoshida M, Shimamoto Y:表达小麦果糖基转移酶基因的多年生黑麦草积累了大量的果聚糖,并在细胞水平上获得了对冷冻的耐受性。中国植物科学,2004,27(4):366 - 366。10.1016 / j.plantsci.2004.05.037。
- 27.
Thomashow MF, Gilmour SJ, Zarka DG, Stockinger EJ, Salazar MP, Houghton JM:低温调控拟南芥CBF家族AP2转录激活因子在低温诱导COR基因表达中的早期作用。植物学报,1998,16(4):433-442。10.1046 / j.1365 - 313 x.1998.00310.x。
- 28.
Hayes PM, Skinner JS, von Zitzewitz J, schzucs P, Marquez-Cedillo L, Filichkin T, Amundsen K, Stockinger EJ, Thomashow MF, Chen THH:大麦CBF基因家族的结构、功能和系统发育特征。植物分子生物学,2005,59(4):533-551。10.1007 / s11103 - 005 - 2498 - 2。
- 29.
Minami A, Nagao M, Ikegami K, Koshiba T, Arakawa K, Fujikawa S, Takezawa D:苔藓植物的冷驯化:小立穗(Physcomitrella patens)低温诱导的抗冻性与胁迫相关基因表达水平升高有关,而与内源脱落酸水平升高无关。植物学报,2005,22(3):414-423。10.1007 / s00425 - 004 - 1361 - z。
- 30.
Badawi M, Danyluk J, Boucho B, Houde M, Sarhan F:六倍体小麦CBF基因家族及其与谷类CBF系统发育复杂性的关系。中国生物医学工程学报,2007,27(5):533-554。10.1007 / s00438 - 006 - 0206 - 9。
- 31.
Choi D-W, Rodriguez EM, Close TJ:大麦Cbf3基因鉴定、表达模式及图谱定位。植物学报,2002,29(4):379 - 379。10.1104 / pp.003046。
- 32.
Vágújfalvi A, Galiba G, Cattivelli L, Dubcovsky J:低温调控转录激活子Cbf3与小麦5A染色体抗冻位点Fr-A2相关。中国生物医学工程学报,2003,26(1):344 - 344。
- 33.
Knox AK, Dhillon T, Cheng HM, Tondelli A, Pecchioni N, Stockinger EJ:温带气候谷物抗冻性与CBF基因拷贝数变异相关。应用理论,2010,21(1):21-35。10.1007 / s00122 - 010 - 1288 - 7。
- 34.
Vogel J, Bragg J:小麦科的一种新模式。小麦科的遗传学和基因组学。第7版。muehlbauer GJ, Feuillet C.编辑纽约:施普林格;2009:427 - 449。
- 35.
Febrer M, Goicoechea JL, Wright J, McKenzie N, Song X, Lin J, Collura K, Wissotski M, Yu Y, Ammiraju JSS,等:短枝草物理、遗传和细胞遗传学综合图谱。PLoS ONE。2010, 5 (10): e13461-10.1371/journal.pone.0013461。
- 36.
Alm V, Busso CS, Ergon A, Rudi H, Larsen A, Humphreys MW, Rognli OA:草甸羊茅(Festuca pratensis Huds.)抗冻性、冬活性和耐旱性的QTL分析及比较遗传定位。应用物理学报,2011,23(3):369-382。10.1007 / s00122 - 011 - 1590 - z。
- 37.
Katoh K, Kuma K-i, Toh H, Miyata T: MAFFT版本5:提高多序列对齐精度。中国生物医学工程学报,2005,33(2):511-518。10.1093 / nar / gki198。
- 38.
Rozen S, Skaletsky H:面向普通用户和生物学家程序员的WWW上的Primer3。生物信息学方法与协议:分子生物学方法。第132版。编辑:Misener S, Krawetz SA: HumanaPress;1999:365 - 386。
- 39.
Fox S, Filichkin S, Mockler TC:超高通量测序的应用。方法分子生物学,2009,553:79-108。10.1007 / 978 - 1 - 60327 - 563 - 7 - _5。
- 40.
Bolstad BM, Irizarry RA, Astrand M, Speed TP:基于方差和偏差的高密度寡核苷酸阵列数据归一化方法的比较。生物信息学,2003,19(2):185-193。10.1093 /生物信息学/ 19.2.185。
- 41.
Skinner J, Zitzewitz J, szundcs P, Marquez-Cedillo L, Filichkin T, Amundsen K, Stockinger E, Thomashow M, Chen T, Hayes P:大麦CBF基因家族的结构、功能和系统发育特征。植物分子生物学,2005,59(4):533-551。10.1007 / s11103 - 005 - 2498 - 2。
- 42.
田村K,山田T:多年生黑麦草CBF基因簇位于禾本科种间保守共时性预测的区域。应用理论与实践,2007,34(2):379 - 379。
- 43.
Penn O, Privman E, Landan G, Graur D, Pupko T:一种捕获鲁棒性的对齐置信度评分,以引导树不确定性。分子生物学杂志,2010,27(8):1759-1767。10.1093 / molbev / msq066。
- 44.
惠兰S,戈德曼N:一个一般的经验模型的蛋白质进化源于多个蛋白质家族使用最大似然方法。分子生物学杂志,2001,18(5):691-699。10.1093 / oxfordjournals.molbev.a003851。
- 45.
大厅TA: BioEdit:一个用户友好的生物序列比对编辑器和分析程序,适用于Windows 95/98/NT。化学学报,1999,41:95-98。
- 46.
Jobb G, von Haeseler A, Strimmer K: TREEFINDER:一个强大的分子系统发育图形分析环境。中国生物医学工程学报,2004,4(1):1-9。10.1186 / 1471-2148-4-1。
- 47.
Ronquist F, Huelsenbeck JP: MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学,2003,19(12):1572-1574。10.1093 /生物信息学/ btg180。
- 48.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然,进化距离和最大简约方法进行分子进化遗传学分析。分子生物学杂志,2011,28(10):2731-2739。10.1093 / molbev / msr121。
- 49.
Dubouzet JG, Sakuma Y, Ito Y, Kasuga M, Dubouzet EG, Miura S, Seki M, Shinozaki K, Yamaguchi-Shinozaki K:水稻,Oryza sativa L. OsDREB基因编码转录激活子,在干旱、高盐和寒冷响应基因表达中起作用。植物学报,2003,33(4):751-763。10.1046 / j.1365 - 313 x.2003.01661.x。
- 50.
李志刚,李志刚,李志刚,等:温度对蒂莫西(Phleum pratense Vega和Climax)叶片果聚糖和总碳水化合物积累的影响。植物营养学报,2002,29(3):379 - 379。10.1078 / 0176-1617-00657。
- 51.
中国植物学报,2002,19(4):379 - 379。10.1111 / j.1469-8137.1982.tb03273.x。
- 52.
瓦卢鲁,范登恩德W:逆境环境中的植物果聚糖:新概念和未来展望。实验学报,2008,59(11):2905-2916。10.1093 / jxb / ern164。
- 53.
李士敦,辛查,Heyer A:果聚糖及其与植物非生物胁迫耐受性的关系。细胞生物学与生物工程学报,2009,26(3):344 - 344。10.1007 / s00018 - 009 - 0002 - x。
- 54.
Dhillon T, Pearce SP, Stockinger EJ, Distelfeld A, Li C, Knox AK, Vashegyi I, Vágújfalvi A, Galiba G, Dubcovsky J:温带谷物抗冻性和开花的调控:VRN-1连接。中国生物医学工程学报,2010,29(4):344 - 344。10.1104 / pp.110.159079。
- 55.
Miller A, Galiba G, Dubcovsky J:单粒小麦抗冻性基因座Fr-Am2上有11个CBF转录因子簇。中国生物医学工程学报,2006,27(2):344 - 344。10.1007 / s00438 - 005 - 0076 - 6。
- 56.
Skinner J, szponcs P, von Zitzewitz J, Marquez-Cedillo L, Filichkin T, Stockinger E, Thomashow M, Chen T, Hayes P:拟南芥低温耐受性基因的同源基因定位。应用物理学报,2006,32(5):832-842。10.1007 / s00122 - 005 - 0185 - y。
- 57.
黄志刚,张志刚,张志刚:黑腹果蝇基因组的复制和基因转换。公共科学学报,2008,4 (12):e1000305-10.1371/journal.pgen.1000305。
- 58.
Lasseur B, Schroeven L, Lammens W, Le Roy K, Spangenberg G, Manduzio H, Vergauwen R, Lothier J, Prud’homme M-P, Van den Ende W:多年黑麦草果实聚糖:果聚糖6g -果糖基转移酶转化为蔗糖:蔗糖1-果糖基转移酶。植物学报,2009,29(1):327-339。10.1104 / pp.108.125559。
- 59.
del Viso F, Puebla AF, Fusari CM, Casabuono AC, Couto AS, Pontis HG, Hopp HE, Heinz RA:巴塔哥尼亚耐寒草Bromus pictus的一种蔗糖:果聚糖6-果糖基转移酶(6-SFT)在低温下与果聚糖积累相关的分子特征。植物生理学报,2009,30(3):489-503。10.1093 /卡式肺囊虫肺炎/ pcp008。
- 60.
Hisano H, Kanazawa A, Yoshida M, Humphreys MO, Iizuka M, Kitamura K, Yamada T:多年生黑麦草果糖转移酶基因协调表达与低温胁迫下果聚糖积累相关。环境科学学报,2008,29(3):344 - 344。10.1111 / j.1469-8137.2008.02409.x。
- 61.
Francki MG, Walker E, Forster JW, Spangenberg G, Appels R:通过基因复制和重排进化的果糖转移酶和转化酶基因:水稻,多年生黑麦草和小麦基因家族。基因组学报,2006,49(9):1081-1091。10.1139 / g06 - 066。
- 62.
Oliver SN, Van Dongen JT, Alfred SC, Mamun EA, Zhao X, Saini HS, Fernandes SF, Blanchard CL, Sutton BG, Geigenberger P,等:低温诱导下水稻花药特异细胞壁转化酶基因OSINV4的抑制与蔗糖积累和花粉不育相关。植物细胞环境,2005,28(12):1534-1551。10.1111 / j.1365-3040.2005.01390.x。
- 63.
Vágújfalvi A, Kerepesi I, Galiba G, Tischner T, Sutka J:小麦低温驯化过程中碳水化合物变化对抗冻性的影响。植物科学,1999,44(2):85-92。10.1016 / s0168 - 9452(99) 00058 - 8。
- 64.
杨晓明,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明:绿型一年生蓝草低温驯化过程中碳水化合物的变化及其抗冻性。作物科学,2001,41(2):443-451。10.2135 / cropsci2001.412443x。
- 65.
Livingston DP:冬季燕麦冠在冷硬前后非结构性碳水化合物的积累。作物科学,1991,31(3):751-755。10.2135 / cropsci1991.0011183X003100030043x。
- 66.
利文顿DP, Premakumar R, Tallury SP:燕麦和黑麦在冷驯化和冷冻过程中冠上和下部碳水化合物分配。低温生物学,2006,52(2):200-208。10.1016 / j.cryobiol.2005.11.001。
- 67.
Tronsmo AM, Kobro G, Morgenlie S, Flengsrud R:两种草种冷硬化后碳水化合物含量和糖苷酶活性。植物营养学报,2003,26(4):489 - 495。10.1111 / j.1399-3054.1993.tb01390.x。
- 68.
Hendry GAF:果聚糖的进化起源和自然功能——气候、生物地理学和机械评价。植物化学学报,1993,23(1):1 -14。
- 69.
Puebla AF, Salerno GL, Pontis HG:两种雀麦在低温和水分胁迫下的果聚糖代谢。环境科学学报,1997,36(1):1 - 3。
- 70.
Livingston DP, Henson CA:在冬燕麦中的异形糖、果聚糖、果聚糖外水解酶和转化酶:对第二阶段冷硬化的响应。植物营养学报,2004,16(1):344 - 344。10.1104 / pp.116.1.403。
- 71.
杨晓明,刘志刚,刘志刚,刘志刚,王志刚,王志刚,王志刚:绿型一年生蓝草42个生态型抗冻性的变异分析。作物科学,2010,50(1):321-336。10.2135 / cropsci2008.12.0712。
- 72.
Amiard V, Morvan-Bertrand A, Billard JP, Huault C, Keller F, Prud'homme MP:干旱胁迫对多年生黑麦草果实聚糖,而不是蔗糖-半乳糖苷、棉子糖和松糖的影响。植物科学进展,2003,26(4):339 - 339。10.1104 / pp.103.022335。
- 73.
Hisano H, Kanazawa A, Kawakami A, Yoshida M, Shimamoto Y, Yamada T:表达小麦果糖基转移酶基因的多年生黑麦草积累了大量的果聚糖,并在细胞水平上获得了对冷冻的耐受性。中国植物科学,2004,27(4):366 - 366。10.1016 / j.plantsci.2004.05.037。
- 74.
Vijn I, Smeekens S:果糖:不仅仅是一种储备碳水化合物?植物营养学报,2004,27(2):359 - 366。10.1104 / pp.120.2.351。
- 75.
Demel RA, Dorrepaal E, Ebskamp MJM, Smeekens JCM, de Kruijff B:果聚糖与模型膜相互作用强。BBA-Biomembranes。1998,(1): 1 -4。
- 76.
Hincha DK, Hellwege EM, Heyer AG, Crowe JH:植物果聚糖在冷冻干燥过程中稳定磷脂酰胆碱脂质体。中国生物医学工程学报,2000,26(2):535- 538。10.1046 / j.1432-1327.2000.01028.x。
- 77.
Vereyken IJ, Chupin V, Demel RA, Smeekens SCM, De Kruijff B:果糖插入磷脂头基之间。BBA-Biomembranes。2001, 40(1): 1 -3。
确认
感谢Øyvind Jørgensen的技术支持和Dr. David Garvin的善意提供Brachypodium distachyon种质资源。这项工作得到了挪威生命科学大学植物与环境科学系和美国能源部植物原料基因组学生物能源赠款DE-FG02-08ER64630给TCM的支持。
作者信息
从属关系
相应的作者
额外的信息
作者的贡献
CL进行了qRT-PCR基因表达和总糖/果聚糖水平分析的实验工作,并参与了稿件的起草。HR帮助设计了这项研究,进行了基因表达的实验工作。EJS和HC对b . distachyon基因组库。EJS还参与了文稿的起草。MC参与了数据的采集和稿件的准备。SF收集和分析了物种分布,并帮助起草了手稿。SEF和TCM进行了基于微阵列的基因表达分析,并参与了稿件的起草。BW负责所有的糖含量分析,并进行HPAEC分析。OAR参与了研究的设计,并帮助起草了手稿。SRS参与了研究设计,进行了系统发育分析,并负责最终的手稿准备。
电子辅助材料
氨基酸序列比对分析
附加文件1:Brachypodium distachyonIRI蛋白多年生黑麦草IRI蛋白序列。带黑色阴影的位点高度保守(>70%的序列)。Bradi5g27350.1和Bradi5g22870.1具有截断的冰结合域。(pdf 2mb)
qRT-PCR表达水平的折叠变化和p值
附加文件2:Brachypodium distachyonIRIP基因。(pdf 58kb)
最小演化CBF3
附加文件3:/4基因系统发育包括全部Brachypodium distachyon同源染色体。采用带有gamma分布率变异和成对缺失的Tamura 3参数法计算进化相关性。按系统发育分类的所有非cbf3c /d和CBF4基因分别为绿色和红色,其他cbf同源基因为黑色。物种缩写:Os,栽培稻;高压,大麦芽;Lp,多年生黑麦草.(pdf 188kb)
低温处理前后碳水化合物含量的HPAEC结果。
附加文件4:以纳米库仑(nC)表示的探测器强度。黑色曲线和蓝色曲线分别为冷处理和未处理多年生黑麦草植物碳水化合物分别提取洗脱。紫色曲线和棕色曲线分别为冷处理和未处理Brachypodium distachyon植物碳水化合物萃取洗脱。(a)检测到低聚合度低聚糖(DP 2 - 5)(葡萄糖、果糖、蔗糖、酮糖、乳糖和其他不明低聚糖)l为而且b . distachyon使用HPAEC。(b)检测到聚合度较高的寡糖l为比在b . distachyon使用HPAEC。(pdf 222kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
李,C.,鲁迪,H.,斯托金格,E.J.et al。比较分析揭示了潜在的用途Brachypodium distachyon作为温带草类冷胁迫反应的模型。BMC植物生物学12日,65(2012)。https://doi.org/10.1186/1471-2229-12-65
收到了:
接受:
发表:
关键字
- Brachypodium distachyon
- 适应寒冷气候
- 冰再结晶抑制蛋白
- 基因表达
- Fructosyltransferase
- c -重复序列结合因子
- 基因家族进化