- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
MIKC的AGAMOUS LIKE 6亚家族的新成员gydF4y2BaCgydF4y2Ba型大豆MADS-box基因gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba13gydF4y2Ba文章编号:gydF4y2Ba105gydF4y2Ba(gydF4y2Ba2013gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
经典的(gydF4y2BaCgydF4y2Ba) mikc型MADS-box转录因子由一个基因家族组成,在开花过程中起着不同的作用,从花的起始到花器官的发育。尽管它们在调节影响作物产量的发育过程中具有重要作用,但在主要的豆科油料作物大豆中,它们在很大程度上仍未被发现。gydF4y2Ba
结果gydF4y2Ba
我们发现了57个MIKCgydF4y2BacgydF4y2Ba型转录因子,并测定gydF4y2Ba在网上gydF4y2Ba大豆MIKC基因表达谱gydF4y2BacgydF4y2Ba型基因存在于不同的组织中。我们的研究涉及三个MIKCgydF4y2BacgydF4y2Ba型转录因子的新成员gydF4y2Ba无性生殖的6gydF4y2Ba(AGL6)亚家族的MIKCgydF4y2BaCgydF4y2Ba类型的MADS-box基因,我们将其命名为姐妹分支PsMADS3。虽然在其他豆科植物如杨树和葡萄中发现了类似的基因,但在豆科植物中没有发现这种基因gydF4y2Ba拟南芥gydF4y2Ba或大米。RT-PCR分析了这3个大豆PsMADS3基因在早期花启动过程中的时间表达gydF4y2BaAPETALA1gydF4y2Ba该基因被认为是一种花分生组织识别基因。然而,RNAgydF4y2Ba原位gydF4y2Ba杂交结果表明,它们的空间表达模式明显不同于其他基因gydF4y2BaAPETALA1。gydF4y2Ba
结论gydF4y2Ba
豆科植物的花发育系统与模式植物拟南芥不同。大豆不同花轮的起始具有重叠性,花序分生组织可根据环境条件恢复成叶。MIKCgydF4y2BaCgydF4y2Ba型MADS-box基因已被证明在花发育的不同阶段发挥关键调控作用。我们在豆科植物中发现了PsMADS3子分支的成员,它们在花起始过程中表现出不同的空间表达,表明它们在花起始过程中可能发挥新的作用。本研究的结果将有助于更好地理解豆科植物特有的花发育过程。gydF4y2Ba
背景gydF4y2Ba
植物花的发育包括从花的萌发到花的形成的严格控制的过程。由于花的发育是决定作物产量的重要农艺性状,因此其潜在过程已被广泛研究。各种转录因子在调节这些发育过程中是必不可少的,包括MADS-box转录因子家族。gydF4y2Ba
MADS-box转录因子,尤其是植物特异性的经典(gydF4y2BaCgydF4y2Ba) mikc型MADS-box基因,在花发育的不同阶段起着关键的调节作用。它们在协调花发育过程中的作用已被主要在模式植物中进行的功能研究所揭示,gydF4y2Ba拟南芥gydF4y2Ba.的MIKCgydF4y2BaCgydF4y2Ba型基因的特征是保守的结构组织gydF4y2Ba米gydF4y2BaADS-box,gydF4y2Ba我gydF4y2Bantervening,gydF4y2BaKgydF4y2Baeratin-like - - -gydF4y2BaCgydF4y2Ba域。DNA结合需要高度保守的mads -结构域和弱保守的i -结构域,而强保守的k -结构域和可变的c -结构域调节蛋白质相互作用[gydF4y2Ba1gydF4y2Ba].gydF4y2Ba
MIKC的全基因组分析gydF4y2BaCgydF4y2Ba型基因已在拟南芥中进行了研究[gydF4y2Ba2gydF4y2Ba)、大米(gydF4y2Ba3.gydF4y2Ba和poplar [gydF4y2Ba4gydF4y2Ba].而拟南芥和水稻基因组中MIKC的数量相近gydF4y2BaCgydF4y2Ba型基因(39对38),杨树有55个这样的基因,这表明与拟南芥或水稻相比,杨树的出生率更高。这些MIKCgydF4y2BacgydF4y2Ba型基因可分为15个不同的基因分支,每个分支以第一个被识别的成员命名[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba5gydF4y2Ba].除两种(TM8和OsMADS32)外,其余均在拟南芥中发现[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba5gydF4y2Ba],并且FLC分支可能在水稻基因组中缺失[gydF4y2Ba3.gydF4y2Ba].目前尚不清楚是否所有的进化支都存在于杨树基因组中,因为在系统发育分析中没有使用TM8基因[gydF4y2Ba4gydF4y2Ba].gydF4y2Ba
SQUA亚科分支包括4个拟南芥成员,APETALA1 (AP1)、CAULIFLOWER (CAL)、FRUITFULL (FUL)和AGAMOUS-LIKE 79 (AGL79) [gydF4y2Ba2gydF4y2Ba].AP1、CAL和FUL的功能已被鉴定,表明它们在决定花分生组织的特性中起部分冗余作用[gydF4y2Ba6gydF4y2Ba].SEPALLATA (SEP)家族属于AGL2分支,在拟南芥中记录了4个成员[gydF4y2Ba7gydF4y2Ba].SEP1、SEP2、SEP3和SEP4这4个成员在决定花器官的同源性和分生组织的确定性方面都起着冗余作用。AP1已被证明可以直接与SEP3启动子结合,从而增加gydF4y2BaSEP3gydF4y2Ba迅速(gydF4y2Ba8gydF4y2Ba].AGL6亚家族在拟南芥中具有相对较小的代表性(只有两个基因,AGL6及其相似基因AGL13)。虽然在拟南芥中还没有描述这些基因的敲除表型,但在水稻、玉米和gydF4y2Ba佩妮矮牵牛gydF4y2Ba已经在很大程度上证明了AGL6亚家族在调节花器官识别和花分生组织确定性方面的作用,表明与密切相关的基因包括gydF4y2Ba9月gydF4y2Ba[gydF4y2Ba9gydF4y2Ba- - - - - -gydF4y2Ba11gydF4y2Ba].系统发育研究表明,SQUA、SEP和AGL6的亚家族总是根植于一个超枝中,这可能与它们在调控花发育中的作用重叠有关。gydF4y2Ba
在大豆最新基因组序列中预测了212个MADS-box基因。gydF4y2Ba12gydF4y2Ba].之前我们报道了豆科植物SAM中一些基因表达和microRNAs的多样性[gydF4y2Ba13gydF4y2Ba- - - - - -gydF4y2Ba16gydF4y2Ba].然而,关于这些基因还有很多需要了解,特别是考虑到它们对作物生产的潜在影响。大豆是世界上最大的豆类作物,占全球油籽产量的50%以上。在本研究中,我们对所有大豆MIKC进行了鉴定gydF4y2BacgydF4y2Ba型MADS-box基因使用目前的Glyma1.0基因集,并确定了其与拟南芥、水稻和杨树的潜在系统发育关系。我们检测了整个家族不同大豆组织的表达模式。有趣的是,结果发现了一个新的AGL6姐妹分支MIKCgydF4y2BacgydF4y2Ba型基因,我们将后续的分析重点放在这个新亚分支的成员上。gydF4y2Ba
结果与讨论gydF4y2Ba
大豆MIKC的分子进化分析gydF4y2BacgydF4y2Ba-type MADS-box转录因子gydF4y2Ba
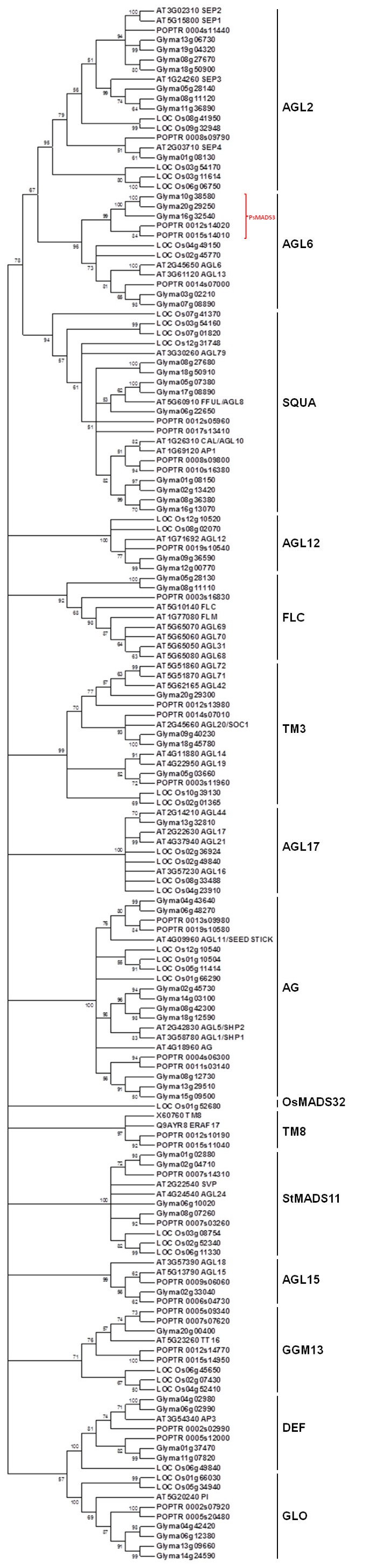
当我们在Phytozome上搜索大豆预测基因集[gydF4y2Ba12gydF4y2Ba对于同时包含MADS-box和k域的序列,我们总共识别了57个序列。随后的检查发现其中三个序列是不完整的。我们试图使用基因预测软件在这些部分序列周围的基因组序列上获得这些基因的完整序列,但没有产生任何结果。因此,我们在进一步的分析中省略了这些序列。研究其与MIKC的系统发育关系gydF4y2BacgydF4y2BaMIKC报道,拟南芥、水稻和杨树的型基因gydF4y2BacgydF4y2Ba组蛋白序列[gydF4y2Ba3.gydF4y2Ba- - - - - -gydF4y2Ba5gydF4y2Ba,从各自的数据库中检索。共159个概念翻译蛋白序列用于系统发育分析。gydF4y2Ba
从生成的系统发育树中鉴定出15个进化支:AGL2、AGL6、SQUA、AGL12、FLC、TM3、AGL17、AG、OsMADS32、TM8、STMADS11、AGL15、GGM13、DEF和GLO(图1)gydF4y2Ba1gydF4y2Ba).不同分支之间的关系与其他报告相似[gydF4y2Ba2gydF4y2Ba- - - - - -gydF4y2Ba5gydF4y2Ba].除了OsMADS32和TM8外,大豆基因存在于所有的进化支中,每个进化支中的大豆序列数量从1个(AGL15和AGL17)到9个(SQUA1)不等,大多数基因都是重复出现的。与前一份报告一致[gydF4y2Ba5gydF4y2Ba, AGL2, SQUA和AGL6中的基因一起形成一个超枝(图gydF4y2Ba1gydF4y2Ba).在AGL6分支中,一个强有力的内部分支似乎与现有的AGL6成员分离,这表明它是一个新的姐妹分支,在拟南芥或水稻中没有表现出来。这个新的亚分支由三个大豆基因Glyma16g32540和一对同源基因Glyma10g38580 (GmMADS3a)和Glyma20g29250 (GmMADS3b)组成。这些基因的顶部BLASTX匹配是花园豌豆的MADS-box转录因子,注释为PsMADS3 [gydF4y2Ba17gydF4y2Ba](数据未显示)。因此,我们将其命名为PsMADS3子分支。gydF4y2Ba
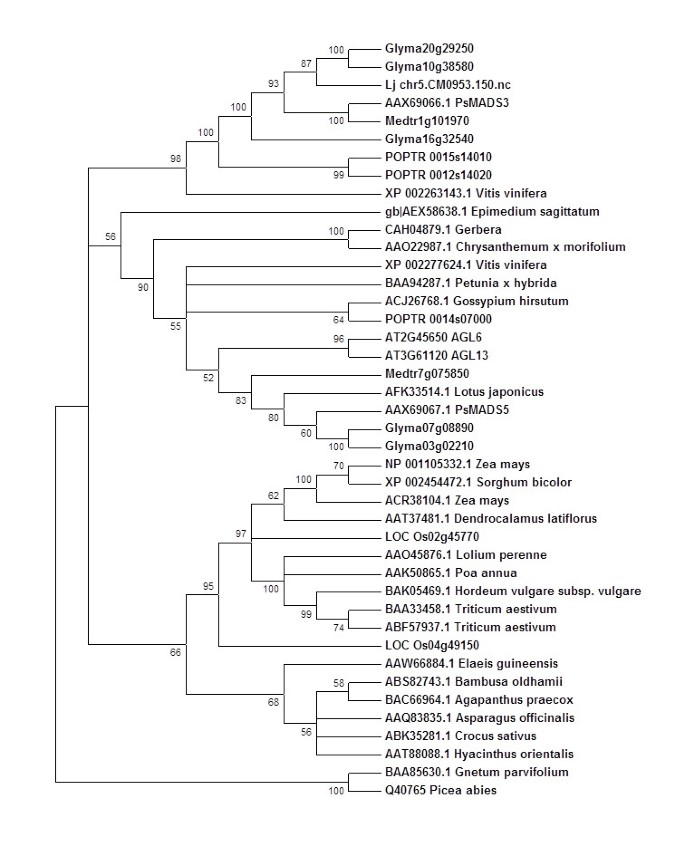
我们接着研究了PsMADS3的类似同源成员是否存在于大豆、豌豆和杨树以外的物种中。在NCBI公共数据库的BLASTP搜索中发现了其他物种的潜在PsMADS3序列(数据未显示)。利用包括姐妹枝AGL6序列在内的顶部匹配的40个同源序列进行系统发育分析,在以裸子植物AGL6为外群的树中确定了三个不同的类群(图gydF4y2Ba2gydF4y2Ba).其中一个分支包含所有的单子叶序列,而另外两个分支包含AGL6和PsMADS3序列gydF4y2Ba2gydF4y2Ba).由于我们只使用了公共数据库中可用的序列,很可能类似的PsMADS3序列存在于其他物种中,而不是被研究的物种。未来的研究增加类群采样将有助于阐明该节点。PsMADS3亚枝的成员不仅存在于豆类植物中,还存在于杨树和葡萄中gydF4y2Ba2gydF4y2Ba).由于拟南芥和水稻的基因组序列已经获得并得到了很好的注释,我们相信PsMADS3亚分支的成员在这两个物种中是不存在的。此外,观察到在包括小麦和玉米在内的其他单性状的顶级匹配序列中没有发现同源PsMADS3基因(图1)gydF4y2Ba2gydF4y2Ba)意味着这些基因可能在单子叶植物中缺失。PsMADS3分支可能是在拟南芥单株-双株分化后和拟南芥种出现之后进化而来的。这些基因可能因为在拟南芥和水稻中缺失而被忽视。事实上,最近一项关于eudiicot AGL6的研究没有分析这些豆科基因[gydF4y2Ba18gydF4y2Ba].gydF4y2Ba
的发展史gydF4y2BaAGL6gydF4y2Ba基因。gydF4y2Ba树的生成如图所示gydF4y2Ba1gydF4y2Ba而是从完整的蛋白质序列。GmMADS3a (Glyma10g38560)的预测肽序列被用于NCBI的BLASTP搜索。从NCBI下载前40个匹配项(E值< 1e-50),过滤重复或短序列,然后进行系统发育分析。以裸子植物AGL6为外群生根。用于分析的植物:gydF4y2Ba真毛芥,拟南芥,芦笋,大竹,野菊花,番红花,大叶竹,山茱萸,山楂,小Gnetum,毛棉,大麦,东方风信子,黑麦草,日本莲花,截叶苜蓿,水稻,矮牵牛,冷杉,Pisum sativum, Poa annua, trichocarpa,双色高粱,小麦,葡萄,玉米。gydF4y2Ba
大豆MIKC的硅分析gydF4y2BacgydF4y2Ba基因表达模式gydF4y2Ba
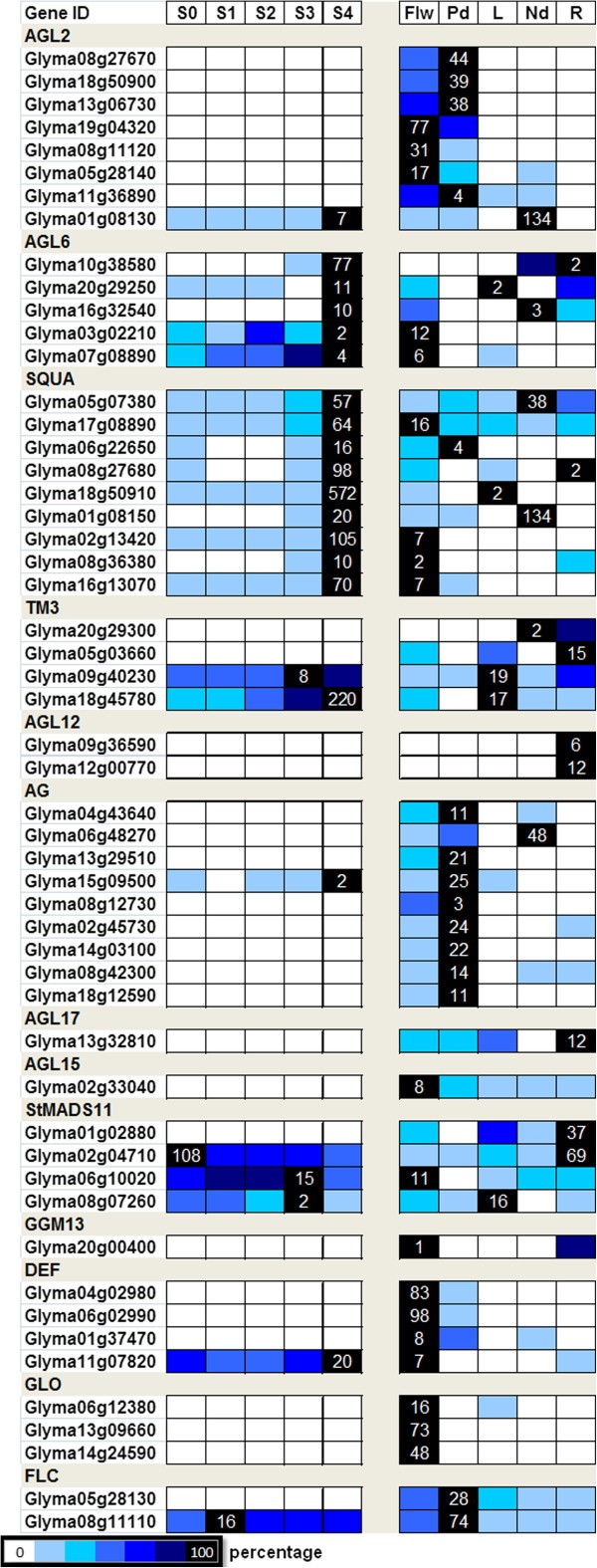
我们之前对经历早期花起始过程的微解剖芽尖分生组织(SAMs)进行了高通量rna测序[gydF4y2Ba29gydF4y2Ba].样品来自10日龄的大豆植株,经过不同长度的短日(0SD, 1SD, 2SD, 3SD和4SD)处理,并诱导花分生组织识别基因包括gydF4y2BaGmAP1gydF4y2Ba发生在4 sd。由于参与开花后期过程(如花器官发育)或只在其他组织(如根)中表达的基因可能无法被我们的数据集捕获,我们还使用了Libault和同事报告的转录组数据[gydF4y2Ba19gydF4y2Ba]包括各种不同的组织,包括花、种荚、根、根瘤和根尖gydF4y2Ba在网上gydF4y2Ba分析(图gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
大豆MIKC的表达谱gydF4y2BacgydF4y2Ba类型的转录因子。gydF4y2Ba在两组不同的样本中,每种基因的最高表达水平作为RPKM值给出(参见方法)。基因的表达水平表示为最大表达水平的百分比,颜色编码从0%(白色)到100%(黑色)。S0-S4:将10日龄的大豆植株转移到方法中所述的短日生长条件后的SAM样品,间隔为0短日(S0)、1短日(S1)、2短日(S2)、3短日(S3)或4短日(S4)。Flw花;Pd,豆荚;L,叶;Nd,结节;R,根。agamamous, agl2/6/12/15/17 - agamous-like2/6/12/15/17, squa-squamosa, flc开花位点c, ggm13 -gydF4y2BaGnetum gnemongydF4y2BaMADS box转录因子13,StMADS11-gydF4y2Ba茄属植物tuberosumgydF4y2BaTM3-番茄MADS盒子转录因子3,DEF: deficiency ens, GLO:GLOBOSA。gydF4y2Ba
均鉴定出大豆MIKCgydF4y2BacgydF4y2Ba-基因至少在三个生殖组织(生殖的SAM,花和荚果)中的一个中表达,除了AGL12分支的成员,它们只在根中表达(图gydF4y2Ba3.gydF4y2Ba).拟南芥gydF4y2BaAGL12gydF4y2Ba在根中优先表达,最近的功能丢失分析表明,它不仅在调节根分生组织细胞增殖,而且在花期转变中发挥作用[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba].基于大豆gydF4y2BaAGL12-LIKEgydF4y2Ba表达谱,很容易让人推测它们在植物调控中的功能可能已经丧失。在大多数MIKC中观察到类似的表达模式gydF4y2BacgydF4y2Ba-基因聚集在一个分支中。所有重复的基因都被转录并具有相似的表达谱,特别是在生殖性SAM中(图gydF4y2Ba3.gydF4y2Ba),说明它们的功能意义。gydF4y2Ba
至于由AGL2、SQUA和AGL6组成的超进化支,三个进化支之间的基因表达谱存在显著差异(图gydF4y2Ba3.gydF4y2Ba).例如,所有gydF4y2BaAGL2-LIKEgydF4y2Ba除了一个基因(Glyma01g08130)在花的早期转变过程中不在SAM中,但在花和荚果的花发育过程中表达。这种模式是预期的,因为这些基因是已知的,随后被激活gydF4y2BaAP1gydF4y2Ba拟南芥的诱导[gydF4y2Ba22gydF4y2Ba].系统进化树分析表明,Glyma01g08130是拟南芥的对应基因gydF4y2BaSEP4gydF4y2Ba.除了被发现在花和豆荚一样其余的gydF4y2BaAGL2-LIKEgydF4y2Ba在花起始过程中,它也在SAMs中表达,在小结中表达非常高。的一种可能的发散功能gydF4y2BaGmSEP4gydF4y2Ba在早期花的起始过程和结的形成中有额外的作用。Glyma01g08150是拟南芥AP1的四个大豆对应物之一,也可能在结节形成中发挥作用。虽然Glyma01g08150在SAM中的表达在4SD上被强烈诱导(20 RPKM),但其表达水平是结节中的6倍(134 RPKM;数字gydF4y2Ba3.gydF4y2Ba).有趣的是,其同源物Glyma02g13420在结节中不表达,但在繁殖SAM中表达最高(105 RPKM;数字gydF4y2Ba1gydF4y2Ba&gydF4y2Ba3.gydF4y2Ba),表明这对同源异构体之间存在功能上的分歧。gydF4y2Ba
虽然大豆AGL6基因的成员在繁殖性SAMs中表达,但其转录水平的变化与大豆AGL6基因的转录水平的变化没有可比性gydF4y2BaPsMADS3-LIKEgydF4y2Ba而且gydF4y2BaSQUA-LIKEgydF4y2Ba表明后两个进化支可能在发育转变过程中发挥更显著的作用。因为没有可用的信息gydF4y2BaPsMADS3-LIKEgydF4y2Ba基因,我们的研究集中在这个新姐妹分支的成员上。gydF4y2Ba
GmMADS3在花起始过程中的表达gydF4y2Ba
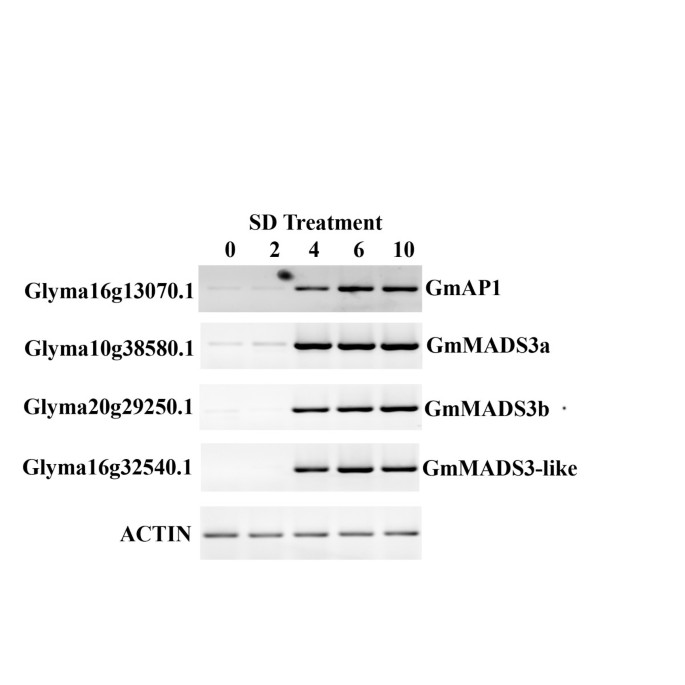
我们进行了RT-PCR分析,以验证该新家族在大豆SAM中早期花启动过程中的表达(图gydF4y2Ba4gydF4y2Ba).从经过0、2、4、6和10 SD处理的植物的解剖SAMs中提取RNA。RT-PCR对PsMADS3分支的每个大豆成员(Glyma16g32540、Glyma10g38580和Glyma20g29250)进行了特异性引物gydF4y2BaGmAP1gydF4y2Ba(Glyma16g13070)作为诱导花过程的对照。与我们之前的研究一致[gydF4y2Ba23gydF4y2Ba的归纳gydF4y2BaGmAP1gydF4y2Ba在4个短天的治疗后发生。PsMADS3分支中的3个大豆基因均表现出与大豆相似的表达模式gydF4y2BaGmAP1gydF4y2Ba,符合gydF4y2Ba在网上gydF4y2Ba分析。gydF4y2Ba
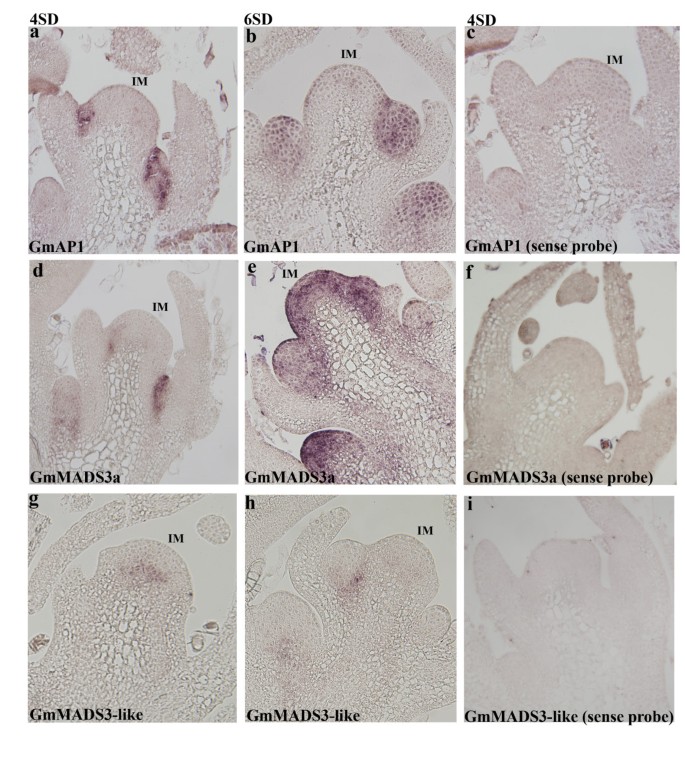
为了研究这些基因在花起始过程中的空间表达模式,gydF4y2Ba原位gydF4y2Ba进行杂交分析。sd 4日,gydF4y2BaGmAP1gydF4y2Ba在花序分生组织的早期花原基中检测到表达gydF4y2Ba5gydF4y2Baa).在6SD时,新形成的花分生组织更加突出gydF4y2BaGmAP1gydF4y2Ba在这些分生组织中检测到表达gydF4y2Ba5gydF4y2Bab)。gydF4y2BaGmMADS3agydF4y2Ba(Glyma10g38580)表达模式与gydF4y2BaGmAP1gydF4y2Basd(图。4日gydF4y2Ba5gydF4y2Bad),但其表达随后扩散到整个花序分生组织以及6SD上新建立的花分生组织(图gydF4y2Ba5gydF4y2Bae)。gydF4y2BaGmMADS3gydF4y2Ba同源对在包括UTR区域在内的核苷酸序列上几乎完全相同(数据未显示),无法进行基因特异性探针,因此观察到的信号可能对应于两个基因的表达。然而,表达gydF4y2BaGmMADS3gydF4y2Ba贯穿整个花序和花分生组织表明它可以作为花序和花分生组织的身份基因。因为表达式gydF4y2BaGmMADS3gydF4y2Ba最初与的重叠gydF4y2BaGmAP1gydF4y2Ba,它的功能可能与gydF4y2BaGmAP1gydF4y2Ba.它随后在花序分生组织中广泛表达,可能确保SAM上的所有营养活动都被分生组织侧翼的花分生组织所取代。gydF4y2Ba
大豆MIKC基因的空间表达谱gydF4y2BacgydF4y2Ba类型的转录因子。一个gydF4y2Ba&gydF4y2BabgydF4y2Ba.gydF4y2BaGmAP1gydF4y2Ba(Glyma16g13070)首先在4 SD上的花序分生组织的早期花原基中表达,然后在6SD上的新建立的花分生组织中表达。gydF4y2BadgydF4y2Ba&gydF4y2BaegydF4y2Ba.gydF4y2BaGmMADS3gydF4y2Ba(Glyma10g38580, Glyma20g29250)在花序分生组织的起始花原基中表达,随后在整个花序分生组织和6SD上新建立的花分生组织中表达。gydF4y2BaggydF4y2Ba&gydF4y2BahgydF4y2Ba.gydF4y2BaGmMADS3-likegydF4y2Ba在花序和花分生组织中心检测到Glyma16g32540基因。gydF4y2Bac、fgydF4y2Ba&gydF4y2Ba我。gydF4y2Ba切片与相应基因的感觉探针杂交,如所示。即时通讯:花序分生组织。gydF4y2Ba
Glyma16g32540的表达与gydF4y2BaGmAP1gydF4y2Ba而且gydF4y2BaGmMADS3gydF4y2Ba.在花序分生组织中心检测到与其表达相关的微弱信号(图gydF4y2Ba5gydF4y2Bag);在6SD上,它也在新出现的花分生组织中心表达(图gydF4y2Ba5gydF4y2Bah). Glyma16g32540在分生组织中心的表达表明其在花序分生组织中组织事件的潜在调控作用。大豆的空间表达模式gydF4y2BaPsMADS3-LIKEgydF4y2Ba基因支持这些基因是新的,因为它们的表达明显不同于密切相关的家庭成员的空间表达,如gydF4y2BaGmAP1gydF4y2Ba(本研究)或拟南芥gydF4y2BaAGL6gydF4y2Ba[gydF4y2Ba24gydF4y2Ba].gydF4y2Ba
结论gydF4y2Ba
与拟南芥花轮的起始时间不重叠不同,豆科大豆的花发育系统具有重叠的花轮[gydF4y2Ba25gydF4y2Ba].此外,与通常无法经历开花逆转的拟南芥不同,[gydF4y2Ba26gydF4y2Ba],当环境生长条件由SD转换为LD时,大豆花序分生组织可恢复为叶片生长[gydF4y2Ba27gydF4y2Ba].因为MIKCgydF4y2BaCgydF4y2Ba类型的MADS-box基因在花发育的不同阶段发挥关键调控作用,可以想象,本研究发现的PsMADS3子分支的成员可能与关键的花调控因子如GmAP1或GmFLC合作,促进发育可塑性。未来的研究旨在确定这些基因的相互作用伙伴将有助于我们理解花的转变过程。gydF4y2Ba
方法gydF4y2Ba
序列和系统发育分析gydF4y2Ba
概念性翻译蛋白序列从公共数据库(Phytozome,水稻基因组注释项目,TAIR和LjGDB)中检索。用于大豆MIKC的初步鉴定gydF4y2BaCgydF4y2Ba型MADS-box转录因子,所有注释基因都进行了MADS-box结构域(PFAM00319)和k结构域(PFAM01486)的筛选。然后对结果进行人工检查和筛选,以筛选出截断的蛋白质序列,最终得到54个序列(附加文件gydF4y2Ba1gydF4y2Ba:表S1)。将序列导入MEGA version 5软件,用于后续的系统发育和分子进化分析[gydF4y2Ba28gydF4y2Ba].使用MEGA中的默认设置对跨越MADS-、I-和k -结构域的蛋白质序列进行MUSCLE校准。对齐后,使用基于Mega 5中实现的基于JTT矩阵的模型的最大似然方法估计进化历史,bootstrap分析设置为200个重复。gydF4y2Ba
表达谱分析使用了两个单独的转录组测序数据[gydF4y2Ba19gydF4y2Ba,gydF4y2Ba29gydF4y2Ba].每个基因的丰度在每个数据集中被归一化,并以每百万kb读数(RPKM)表示,并在附加文件中提供gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
植物生长和RNA提取gydF4y2Ba
大豆植物;(左)稳定。简历。是在澳大利亚维多利亚州墨尔本大学的一个温室里生长的。为了诱导开花,将10天大的植物转移到温度为25°C、每天10小时(150 μmol m)的生长室中gydF4y2Ba-2gydF4y2Ba年代gydF4y2Ba-1gydF4y2Ba)和14小时的夜晚(白天较短)。如前所述,茎尖分生组织(SAMs)被显微解剖[gydF4y2Ba24gydF4y2Ba].使用Qiagen RNeasy Mini Kit (Qiagen, Victoria, Australia)进行dna酶柱上消化,从解剖的SAM中提取总RNA(每次提取约80个SAM)。gydF4y2Ba
rt - pcr分析gydF4y2Ba
在所有的RT-PCR分析中,按照制造商的说明使用Qiagen一步RT-PCR试剂盒。从10日龄大豆幼苗(0 SD)和经过不同短日处理(2、4或6 SD)的植株的SAMs中分离的总RNA (20 ng)作为模板,反应体积为10-μl,扩增25个循环。引物使用的是:gydF4y2Ba
Glyma10g38580F CAACTGGATAGAACGCTTGCACAAGgydF4y2Ba
Glyma10g38580R CATCAATGGACGCTTAACGTACTATATAGCgydF4y2Ba
Glyma16g32540F CTTGAGCTGACACAAAGGCAgydF4y2Ba
Glyma16g32540R GCTTTGACTACCGTCTGTCTTGgydF4y2Ba
Glyma20g29250F AGCTCGGAAGCACCTAACGgydF4y2Ba
Glyma20g29250R CATCAATGGACCCTCAACTATAGCgydF4y2Ba
Glyma16g13070F GCCTCAAAGAGCTTCAGAGTCTGGAGCgydF4y2Ba
Glyma16g13070R AGAAAGCCTAGCCTTGTGACCAgydF4y2Ba
ACTINF ATCATGTTTGAGACCTTCAATGTGgydF4y2Ba
ACTINR CTCGAGTTCTTGCTCATAATCTAGG。gydF4y2Ba
大豆肌动蛋白基因作为内控基因。PCR反应在含0.1 μg/μl溴化乙锭的1%琼脂糖凝胶上分离,在紫外线下观察。gydF4y2Ba
核糖核酸gydF4y2Ba原位gydF4y2Ba杂交gydF4y2Ba
将大豆茎尖切开,真空浸润后,用4%多聚甲醛(Sigma, Victoria, Australia)在磷酸盐缓冲盐水中固定,4℃过夜。后续的固定和杂交步骤如前所述[gydF4y2Ba13gydF4y2Ba].gydF4y2Ba
确认gydF4y2Ba
我们要感谢Bernie Carroll教授提供的大豆种子,感谢Sheh May Tam博士和Lim Chee Liew博士在系统发育分析方面的帮助。澳大利亚研究委员会以综合豆科植物研究卓越中心(CE0348212)和ARC DP0988972的形式提供的财政支持也得到了感谢。gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
Kaufmann K, Melzer R, Theißen G: mikc型mds结构域蛋白:陆地植物的结构模块化、蛋白相互作用和网络进化中国生物医学工程学报,2005,32(2):359 - 359。10.1016 / j.gene.2004.12.014。gydF4y2Ba
- 2.gydF4y2Ba
Parenicova L, de Folter S, Kieffer M, Horner DS, Favalli C, Busscher J, Cook HE, Ingram RM, Kater MM, Davies B,等:拟南芥中完整MADS-box转录因子家族的分子和系统发育分析:MADS世界的新打开。植物科学学报,2003,15(7):1538-1551。10.1105 / tpc.011544。gydF4y2Ba
- 3.gydF4y2Ba
Arora R, Agarwal P, Ray S, Singh A, Singh V, Tyagi A, Kapoor S:水稻MADS-box基因家族:生殖发育和胁迫过程中的全基因组识别、组织和表达谱分析。中国生物医学工程学报,2007,29(1):366 - 366。gydF4y2Ba
- 4.gydF4y2Ba
基因学报,2006,32(4):344 - 344。gydF4y2Ba
- 5.gydF4y2Ba
Becker A, Theissen G: MADS-box基因的主要分支及其在开花植物发育和进化中的作用。分子系统发育学报,2003,29(3):464-489。10.1016 / s1055 - 7903(03) 00207 - 0。gydF4y2Ba
- 6.gydF4y2Ba
张丽娟,顾青,张丽娟。果瓣和花菜对分生组织特性和植物结构的冗余调控。科学通报,2000,29(4):729 -734。gydF4y2Ba
- 7.gydF4y2Ba
Ditta G、Pinyopich A、Robles P、Pelaz S、Yanofsky MF:拟南芥SEP4基因在花器官和分生组织鉴定中的作用植物生物学杂志,2004,14(21):1935-1940。10.1016 / j.cub.2004.10.028。gydF4y2Ba
- 8.gydF4y2Ba
Kaufmann K, Wellmer F, Muino JM, Ferrier T, Wuest SE, Kumar V, Serrano-Mislata, Madueno F, Krajewski P, Meyerowitz EM,等:花瓣起始的配器作用。科学通报,2010,28(5):559 - 561。10.1126 / science.1185244。gydF4y2Ba
- 9.gydF4y2Ba
Ohmori S、Kimizu M、Sugita M、Miyao A、Hirochika H、Uchida E、Nagato Y、Yoshida H: MOSAIC flower organans1是一个AGL6-like MADS box基因,调控水稻花器官识别和分生组织命运。植物学报,2009,21(10):3008-3025。10.1105 / tpc.109.068742。gydF4y2Ba
- 10.gydF4y2Ba
Rijpkema AS, Zethof J, Gerats T, Vandenbussche M:矮牵牛AGL6基因在花模式中具有类似sepallata的功能。植物学报,2009,30(1):1-9。10.1111 / j.1365 - 313 x.2009.03917.x。gydF4y2Ba
- 11.gydF4y2Ba
Thompson BE, Bartling L, Whipple C, Hall DH, Sakai H, Schmidt R, Hake S:须穗编码一个对玉米花发育至关重要的MADS盒转录因子。植物学报,2009,21(9):2578-2590。10.1105 / tpc.109.067751。gydF4y2Ba
- 12.gydF4y2Ba
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL, Song Q, Thelen JJ, Cheng J,等:古多倍体大豆基因组序列。自然学报,2010,463(7278):178-183。10.1038 / nature08670。gydF4y2Ba
- 13.gydF4y2Ba
黄志明,黄志明,黄志明,黄志明:大豆茎尖分生组织基因表达的全基因组分析。植物生物学杂志,2009,39(6):711-727。10.1007 / s11103 - 008 - 9450 - 1。gydF4y2Ba
- 14.gydF4y2Ba
Liang D, Wong CE, Singh MB, Beveridge CA, Phipson B, Smyth GK, Bhalla PL:豌豆茎尖分生组织的分子解剖。中国生物医学工程学报,2009,32(4):369 - 369。10.1093 / jxb / erp254。gydF4y2Ba
- 15.gydF4y2Ba
黄超,Khor SY, Bhalla PL, Singh MB:大豆初花原基中WUSCHEL的空间表达。植物学报,2011,33:553-560。10.1007 / s00425 - 010 - 1320 - 9。gydF4y2Ba
- 16.gydF4y2Ba
王采,赵玉涛,王晓杰,Croft L,王志华,Haerizadeh F, Mattick JS, Singh MB, Carroll BJ, Bhalla PL:大豆茎尖分生组织中的microrna。中国生物医学工程学报,2011,32(5):359 - 361。10.1093 / jxb / erq437。gydF4y2Ba
- 17.gydF4y2Ba
Hecht V, Foucher F, Ferrandiz C, Macknight R, Navarro C, Morin J, Vardy ME, Ellis N, Beltran JP, Rameau C,等:模型豆科植物拟南芥开花基因的保存。植物生理学报,2004,25(4):369 - 369。10.1104 / pp.104.057018。gydF4y2Ba
- 18.gydF4y2Ba
Viaene T, Vekemans D, Becker A, Melzer S, Geuten K:核心eudicot复制后AGL6 MADS域转录因子谱系的表达差异提示功能多样化。植物生物学杂志,2010,10(1):148-10.1186/1471-2229-10-148。gydF4y2Ba
- 19.gydF4y2Ba
Libault M, Farmer A, Joshi T, Takahashi K, Langley RJ, Franklin LD, He J, Xu D, May G, Stacey G:作物模型的转录组整合图谱gydF4y2Ba大豆gydF4y2Ba及其在植物比较分析中的应用。植物生态学报,2010,32(1):1 - 6。gydF4y2Ba
- 20.gydF4y2Ba
Tapia-Lopez R、Garcia-Ponce B、Dubrovsky JG、Garay-Arroyo A、Perez-Ruiz RV、Kim SH、Acevedo F、Pelaz S、Alvarez-Buylla ER:与agammus相关的MADS-box基因XAL1 (AGL12)调控拟南芥根分生组织细胞增殖和开花过渡。植物生理学报,2008,26(3):366 - 366。10.1104 / pp.107.108647。gydF4y2Ba
- 21.gydF4y2Ba
rantsley SD, Ditta GS, Yanofsky MF: MADS Box基因在拟南芥发育中的不同作用。植物生态学报,2004,22(4):369 - 369。gydF4y2Ba
- 22.gydF4y2Ba
Schmid M, Uhlenhaut NH, Godard F, Demar M, Bressan R, Weigel D, Lohmann JU:利用全局表达分析分离花诱导通路。工程物理学报,2004,24(3):361 - 361。10.1242 / dev.00842。gydF4y2Ba
- 23.gydF4y2Ba
黄采,Singh MB, Bhalla PL:大豆茎尖分生组织花转变的分子过程。植物生态学报,2009,29(5):369 - 369。10.1111 / j.1365 - 313 x.2008.03730.x。gydF4y2Ba
- 24.gydF4y2Ba
Koo SC, Bracko O, Park MS, Schwab R, Chun HJ, Park KM, Seo JS, Grbic V, Balasubramanian S, Schmid M,等gydF4y2Ba拟南芥gydF4y2BaAGAMOUS-LIKE6 MADS-box基因。植物学报,2010,32(5):371 - 371。10.1111 / j.1365 - 313 x.2010.04192.x。gydF4y2Ba
- 25.gydF4y2Ba
豆科植物的花发育。植物生理学报,2003,31(3):369 - 369。10.1104 / pp.102.017459。gydF4y2Ba
- 26.gydF4y2Ba
Tooke F, Ordidge M, Chiurugwi T, Battey N:花和花序反转的机制和功能。中国生物医学工程学报,2005,32(4):359 - 359。10.1093 / jxb / eri254。gydF4y2Ba
- 27.gydF4y2Ba
沃什伯恩CF,托马斯JF:恢复开花gydF4y2Ba大豆(豆科)gydF4y2Ba.中国生物工程学报,2000,29(3):369 - 369。10.2307 / 2656869。gydF4y2Ba
- 28.gydF4y2Ba
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S: MEGA5:利用最大似然、进化距离和最大简约方法进行分子进化遗传学分析。分子生物学与病原学杂志,2011,28(10):2731-2739。10.1093 / molbev / msr121。gydF4y2Ba
- 29.gydF4y2Ba
Wong CE, Singh MB, Bhalla PL:大豆叶和茎尖分生组织在花起始过程中的转录组动态。科学通报,2013,8 (6):e65319-10.1371/journal.pone.0065319。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明他们没有竞争利益。gydF4y2Ba
作者的贡献gydF4y2Ba
CEW进行了所有的实验和数据分析,并撰写了手稿。PB和MS设计并指导了研究并编辑了手稿。所有作者阅读并批准了最终稿件。gydF4y2Ba
电子辅料gydF4y2Ba
12870 _2012_1327_moesm1_esm.xlsxgydF4y2Ba
附加文件1:表S1:大豆MIKC的表达水平gydF4y2BaCgydF4y2Ba型转录因子在不同组织中的表达。(XLSX 15 KB)gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文由BioMed Central Ltd.授权发布。这是一篇开放获取文章,根据创作共用授权协议(gydF4y2Bahttps://creativecommons.org/licenses/by/2.0gydF4y2Ba),它允许在任何媒体上不受限制地使用、分发和复制,只要原著被恰当地引用。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
Wong, c.e., Singh, M.B. & Bhalla, P.L. MIKC的AGAMOUS LIKE 6亚家族的小说成员gydF4y2BaCgydF4y2Ba型大豆MADS-box基因。gydF4y2BaBMC植物杂志gydF4y2Ba13日,gydF4y2Ba105(2013)。https://doi.org/10.1186/1471-2229-13-105gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/1471-2229-13-105gydF4y2Ba
关键字gydF4y2Ba
- 发展gydF4y2Ba
- 植物分生组织gydF4y2Ba
- MADS-box转录因子gydF4y2Ba
- 大豆gydF4y2Ba