摘要
背景
生菜(摘要以L.)是叶类蔬菜中的主要作物。开发了几种有效应用于生菜育种和遗传研究的分子标记。然而,只有数量非常有限的基于微卫星的标记是公开的。采用富集微卫星文库的方法,开发了97个基因组SSR标记。
结果
对36个新开发的标记进行测试摘要以加入(33l.漂白亚麻纤维卷,各一个l.serriolall.salignaL。,l.virosaL.)的研究结果表明,过程中= 0.56),每个SSR的位点数(Na= 5.50),基因组SSR标记显著高于先前开发的基于est的SSR标记(过程中= 0.32,Na= 3.56)。在莴苣分子连锁图谱上放置了54个基因组SSR标记。标记在基因组中的分布似乎是随机的,除了可能在连锁组6上的簇。32个基因组ssr的任意组合都能区分所有36个材料的基因型。新开发的14个SSR标记来源于与抗性候选基因(RGCs)和RGC假基因序列相似性较高的片段。分子方差分析(AMOVA)l.漂白亚麻纤维卷遗传多样性约3%发生在种质内,79%发生在种质间,18%发生在园艺类型间。
结论
新开发的基因组SSR标记被添加到先前开发的EST-SSRs标记库中。这两种ssr标记为莴苣品种指纹图谱、综合分子连锁图谱的开发和基因定位提供了有用的工具。
背景
栽培生菜(摘要以菊科(菊科)自交受精的二倍体植物n= 2x= 18条染色体。世界各地种植了几种供人类食用的园艺莴苣。将生菜品种划分为园艺类型通常是基于头和叶的形状、大小、结构和茎的长度。这七种类型包括脆头莴苣(冰山莴苣和巴达维亚莴苣的组合)、长叶莴苣、butterhead莴苣、拉丁莴苣、叶莴苣、茎莴苣和油莴苣。
几种类型的生化和分子标记已应用于生菜基因分型,如同工酶[1]、限制性片段长度多态性(RFLP) [2]、随机扩增多态性DNA (RAPD) [3.],扩增片段长度多态性(AFLP) [4],简单序列重复序列(简写为SSR) [5],靶区扩增多态性(简写为TRAP) [6,7],基于表达序列标签的SSR - EST-SSR [8]、单核苷酸多态性(简写为SNP) [9]和单位置多态性(SPP) [10]。利用分子标记进行基因分型可用于品种指纹识别、遗传多样性检测、群体结构评估、定位感兴趣的基因,以及在育种计划中选择理想的基因型。由于SSR标记(微卫星)具有共显性、多等位基因的特点,比显性型标记具有更高的信息量,因此常用SSR标记(微卫星)进行品种指纹鉴定。然而,SSR标记的开发是昂贵和耗时的,因此只有非常有限数量的SSR标记可公开用于生菜[5]。此前,我们开发了一套EST-SSR标记[8从大约2万个基因中提取l.漂白亚麻纤维卷以及它的野生近亲多刺莴苣(l.serriolal .)。本文描述了从基因组DNA中提取SSR标记用于莴苣品种指纹图谱的开发。为了开发这组新的SSR标记,我们采用了富集微卫星文库的方法[11- - - - - -13]。
本工作的目的是:1)建立一套基因组SSR标记;2)不同生菜品种间的试验标记多态性;3)将SSR标记整合到生菜分子连锁图谱中。
方法
基因组SSR标记的开发
基因组SSR标记是从l.漂白亚麻纤维卷简历。根据Glenn和Schable的协议13]和Farias等人。[12],并做了一些修改。该过程包括DNA提取、DNA酶切、连接DNA片段、含微卫星片段的pcr富集、与微卫星特异性探针杂交、含微卫星片段的回收以及产物的克隆和测序。
从一个月大的温室植物的嫩叶中收集大约100毫克的组织,并立即进行冻干。在使用dnasy Plant Mini Kit(均来自Qiagen, Valencia, CA)提取DNA之前,使用TissueLyser磨粉机将样品磨成细粉。用ND-1000光谱仪(NanoDrop Technologies, Wilmington, DE)分析DNA浓度和质量。3 μg基因组DNA用BfuCI的同分体分3AI(新英格兰生物实验室伊普斯维奇,马萨诸塞州)根据制造商的说明。酶在80℃下失活20 min,在0.8%琼脂糖凝胶上用5 μl液相色谱验证酶解。连接体是由两个寡核苷酸:Er1BhGATCSticky 5杂交而成”- CGG - CGG - CGG - CGG - CGG”和Er1Bh1Blunt”-gcg at tca GTG at CCT gcc-3”.然后使用T4 DNA连接酶(New England Biolabs, Ipswich, MA)按照制造商的说明将这些连接物连接到分离的DNA上。
建立PCR以增加含有SSRs的片段的数量。以结扎产物为模板,Er1Bh1Blunt为引物进行pcr富集。反应设置如下:1 × PCR混合液(Promega, Fitchburg, WI), 0.25 mM Er1BhBlunt引物,模板1 μl,蒸馏水至终体积25 μl(表1)1)。未整合的核苷酸和引物用核酸外切酶I和南极磷酸酶(New England Biolabs, Ipswich, MA)清理。按照制造商的说明,使用末端转移酶(New England Biolabs, Ipswich, MA)将寡核苷酸探针生物素化。为了每寡核苷酸产生1 - 3个生物素,比例为1 pmol / 3”终点为0.01 mmol生物素-14- datp (Invitrogen, Grand Island, NY)。寡核苷酸按照Glenn和Schable的建议进行混合[13]:混合2 ((AG)12(TG)12(AAC)6(亚美大陆煤层气有限公司)8(AAT)12(行为)12(ATC)8);混合3 ((AAAC))6(AAAG)6(AATC)6(AATG)6(ACAG)6(ACCT)6(ACTC)6(代理)6);混合4 ((AAAT))8(AACT)8(AAGT)8(ACAT)8(阿加特设计局)8)。37℃独立生物素化30 min,加热至70℃失活10 min。用3 M乙酸钠和无水乙醇沉淀去除多余的生物素,探针在100 μl的蒸馏水中重悬。为了分离含有ssr的片段,根据制造商的说明,将探针附着在Streptavidin磁珠(New England Biolabs, Ipswitch, MA)上。富集- pcr产物在95°C下变性5分钟,并在冰上快速冷藏。将产物与探针在55℃的烤炉中杂交3小时,用2 × SSC缓冲液和0.1% SDS缓冲液洗涤2次,然后用1 × TE缓冲液-50 mM NaCl洗涤2次,再用200 μl的1 × TE缓冲液重悬。为了恢复含有ssr的片段,探针- ssr复合物在95°C下变性5分钟,并用磁铁快速去除珠子。以Er1Bh1Blunt寡核苷酸为引物,杂交产物为模板,建立PCR检测片段的恢复情况(表1)1)。PCR产物随后在1.2%琼脂糖凝胶上运行。
一旦片段恢复被验证,第二次pcr富集被设置为准备克隆序列。以0.8 mM dNTPs、1× PCR缓冲液、0.4 μM Er1Bh1Blunt引物、2.5 U Taq Polymerase和1 μl杂交产物进行4次反应(表1)1)。PCR产物汇集,用QiaQuick色谱柱(Qiagen, Valencia, CA)清洗,使用Topo TA克隆试剂盒进行测序和克隆E.杆菌Mach1-T1R细胞(Invitrogen, Grand Island, NY),根据制造商的说明。将转化后的细胞移入含有50 mg/ml氨苄西林的溶菌肉汤(LB)的96孔板中,在37℃下培养至少4小时。用标准的M13正、反向引物和2-3 μl细菌生长的LB培养基为模板进行PCR验证。加入浓度为25 μg/ml的牛血清白蛋白;所有其他试剂均按上述浓度使用。E.杆菌将含有预期大小产物的菌落转移到添加氨西林的Wu Broth中,并将其提交给美国农业部-农业部基因组学和生物信息学研究部门进行测序。测序数据清除载体污染,使用CLC DNA workbench 5.0 (CLCBio Aarhus, Denmark)组装成contigs。最小长度为14 bp的ssr使用WebSat进行了识别[14]。用Primer3软件设计SSR扩增引物[15集成到WebSat。引物质量分析使用OligoAnalizer 3.1 (Integrated DNA Technologies Inc ., Coralville, IA)。当序列包含多个SSR时,为每个SSR设计不同的引物对。如果用Primer 3设计的引物扩增不能产生预期的产物,则使用CLC DNA工作台设计第二对引物。2012年1月将含ssr片段的序列与GenBank数据库(http://www.ncbi.nlm.nih.gov),使用CLC DNA工作台5.0。BLAST算法的“blastn”选项[16]应用低复杂度滤波器对viridiplantae数据库的核苷酸集合(nr)进行搜索,以避免仅基于微卫星序列的虚假命中。报告相似性的显著性阈值设为1e-4。
标记多态性检测
利用36份材料对新开发的SSR标记进行多态性分析。这套有33个l.漂白亚麻纤维卷栽培品种加上与栽培莴苣有性相容的三种野生品种中的每一种的单一加入;带刺生菜(l.serriola)、柳叶莴苣(l.saligna)和苦生菜(l.virosal .)。基因型栽培品种属于7个园艺类型:脆头莴苣、叶莴苣、长叶莴苣、butterhead莴苣、茎莴苣、拉丁莴苣和油莴苣2)。
SSR标记分型:优化每对引物的SSR扩增条件。最佳PCR条件在附加文件中描述1.一般情况下,每个引物0.2 μM, DNA模板5 ng, Taq PCR主混合物1倍(New England Biolabs, Ipswich, MA),终体积为10 μl(表2)1)。PCR产物使用eGene HDA-GT12 DNA分析仪(目前称为QIAxcel System,来自Qiagen, Valencia, CA)进行分离,并使用Biocalculator软件(eGene, Irvine, CA)进行评分。
遗传杂合度分析:SSR数据采用GenAlEx 6.1进行统计分析[17]共显性标记和gendive v.2.0b20 [18]。缺失数据和无效等位基因被排除在分析之外。遗传杂合性的无偏估计过程中[19],并观察到不同等位基因的数量Na测量标记信息值(GenAlEx 6.1)。遗传距离(F圣) [20.]在至少有两个品种的所有园艺品种对之间,分子变异分析(AMOVA) [21],主成分分析(PCA)采用gendive v.2.0b20进行计算。基于est的[22]和基因组ssr用Student 's进行检测t以及。
分子标记数据集的一致性:使用van Hintum编写的程序,使用数据分辨率(DR)统计来评估SSRs数据集的内部一致性[23]。DR取值范围为0 ~ 1;其中值越高表示数据的内部一致性越高。复制数设置为10,000。
基因型鉴定:软件MultiLocus ver。1.3 b (24]用于估计在一组36份材料中可以鉴定出的不同基因型的数量,标记数量逐渐增加。这一分析表明,标记越多是否会导致鉴定的基因型数量增加。从1到。随机抽取1000个标记米1,米是标记的总数。用鉴定的基因型数除以种质总数计算鉴定的相对基因型数。
将SSR标记整合到生菜分子连锁图谱中
新开发的基因组ssr被整合到l.漂白亚麻纤维卷(简历。萨利纳斯)×l.serriola(accession UC96US23)分子连锁图谱[25]。框架连锁图谱由SNP和AFLP标记组成,间隔约5-10 cM,覆盖所有9个生菜连锁群。这些框架标记是从整合的SNP/AFLP连锁图谱中选择的,标记信息于2010年4月从Compositae Genome Project网站(compgenomics.ucdavis.edu/compositae_LettMap.php)下载。亲本基因型和96f8重组种间自交系(RILs)l.漂白亚麻纤维卷×l.serriola用SSRs对作图群体进行基因分型。用SSRs进行DNA分离和基因分型。使用MapManager QTX版本0.30将SSR标记整合到框架连锁图中[26]。程序设置包括用于联动评价的SelfRI、Kosambi映射功能、缺失数据推断、标记分布命令p-value≤0.001。除了基因组ssr,我们实验室之前开发的EST-SSR标记[22]也被整合到这个分子连锁图谱中。
标记分布的建模与分析
为了分析SSR标记在分子连锁图谱上的分布,我们将观察到的标记分布与假设标记随机分布的模型进行了比较。该模型是通过在分子连锁图谱的9个连锁基团上随机放置标记来建立的。每个包含基因组ssr和est - ssr的连锁组生成1000个模型。标记分布的分析基于两个连续标记之间的间隔长度和标记的聚类。根据链接图(或建模数据)计算两个连续标记之间的间隔长度(以cM为单位)。随后,将间隔分组到含有相同大小间隔(增加10 cM)的箱中。通过将9个连锁组中的每一个划分为20 cM长的片段来进行聚类评价。对真实数据和模型数据计算每20厘米长段的标记数量。
观察到的标记分布与模型之间的拟合优度用K-S检验和皮尔逊卡方(χ2)测试。采用Microsoft Excel v.14.1.4 (Microsoft, Redmond, WA)和JMP 6.0.3 (SAS Institute, Cary, NC, USA)进行建模和统计分析。
结果与讨论
标记的发展
在选择性培养基(含氨苄西林LB)上培养的548个菌落共扩增出217个产物。其中154种产品来自混合物2,24种来自混合物3,39种来自混合物4。在217个产品中,对192个产品进行了测序,得到117个包含微卫星的独特序列。测序显示,一些碎片含有不止一颗微卫星。在这种情况下,试图设计引物,使每个微卫星单独放大。含微卫星序列是根据它们的起源(lactuca年代ativa简历。年代alinas),后面跟着车牌代码(a或B)和连续数字(lssa# #或lssb# #)。
79%的测序产物含有二核苷酸重复序列;14%的产物含有三核苷酸重复序列;3%的产物含有四核苷酸重复序列,4%的产物含有五个或更多核苷酸的重复序列。在24%的产物中检测到两个独立的重复序列,在22%的产物中发现不完全重复序列。
序列同源性检索结果
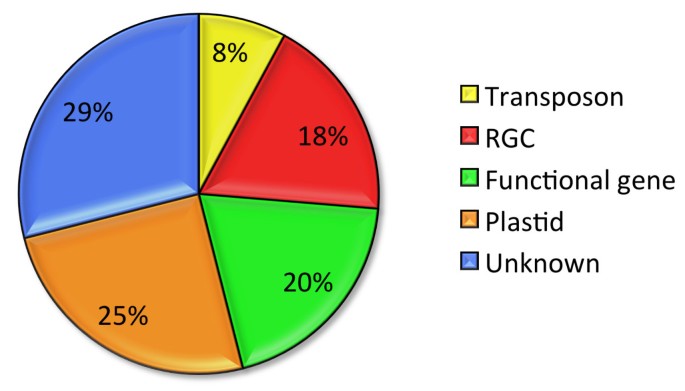
76个含有ssr的片段与viridiplantae GenBank数据库的核苷酸序列高度相似(<1e-4) (http://www.ncbi.nlm.nih.gov)。有19个序列与叶绿体DNA (cpDNA)或线粒体DNA (mtDNA)相似。然而,其中一些测序片段在定位群体中显示分离,表明它们可能起源于与cpDNA或mtDNA序列相似的核DNA。在植物物种中,有很大比例(61.4% ~ 94.3%)的mtDNA序列与核DNA序列高度相似[27]。一组14个测序片段似乎与抗性候选基因(RGCs)和RGC假基因高度相似[28]。从这些片段中开发的SSR标记可以评估与抗病的关联,并且(如果检测到关联)可能用于标记抗性表型。其中6个序列片段与转座子或反转录转座子相似。15个序列与一些假定的基因相匹配,包括几丁质酶、过氧化物酶、胰蛋白酶抑制剂、丁香烯氧化酶或大刍草分枝的基因。其余22个片段与基因或已知功能的推定基因没有明显的相似性(图2)1)。
文献标记信息价值及分析
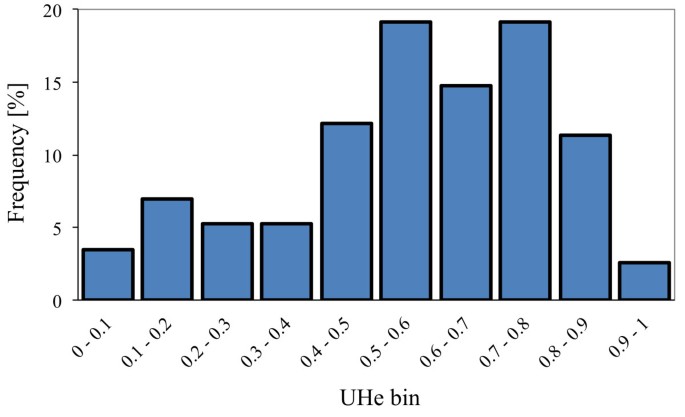
当开发的ssr标记用于36个材料的基因分型时,一些标记的大小比预期的要长得多,每个纯合材料显示几个位点,或者仅在极少数材料中扩增。这些标记被排除在进一步的分析之外,将优质标记的数量减少到97个(附加文件)1)。通过无偏估计来测量的遗传杂合性过程中,范围从0(单态标记)到0.92,平均值为0.56(图2)2)。平均过程中基因组SSR标记的比例显著高于(t以及,p= 5.9 × 10-11年)比过程中EST-SSR (0.32) [22]。同样,每个SSR的位点数量(Na)明显高于(t以及,p= 7.3 × 10-6),而非EST-SSRs。每个基因组SSR的位点数为1 ~ 19个,平均值为5.50个;EST-SSRs的平均值为3.56 [22]。虽然之前用EST-SSRs进行基因分型的一组材料[22与目前用基因组SSRs进行基因分型的一组资料不相同,这两组资料重叠。两套包含材料从相同的园艺类型莴苣允许有限的比较。与基因组ssr相比,EST-SSR标记的多态性较低,在其他几种植物物种中也有报道,如葡萄[29],大米[30.],小麦[31]和向日葵[32]。EST-SSRs的多态性较低可能是由于基因组编码区域的保守性[33]。
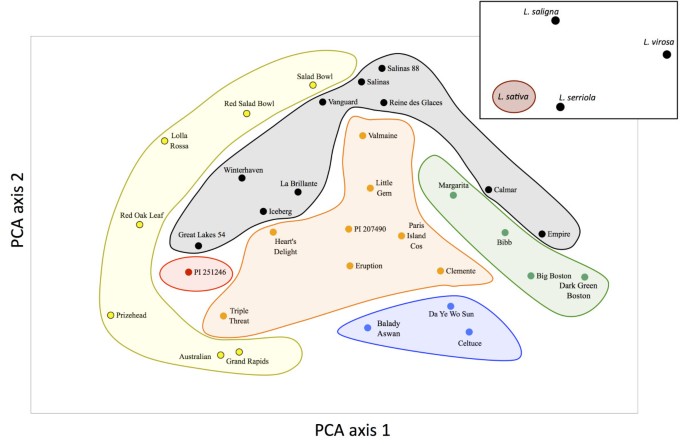
AMOVA分析结果显示,约3.2%的遗传多样性存在于种质内,78.9% (p< 0.001),其余17.9% (p< 0.005)3.)。这些结果与使用SNP标记对5种园艺莴苣进行基因分型的结果相似。Kwon等。[9]发现23%的遗传变异存在于园艺类型中,68.2%的遗传变异存在于园艺类型内。我们还计算了两两微分(F圣),适用于所有园艺品种对,每个品种至少有两个种属(表4)。这种变化F圣数值范围从0.038(脆型和长叶型之间)到0.202 (butterhead型和leaf型之间)。这些结果与我们之前使用TRAP [7]和EST-SSR标记[22],将脆薯和长叶薯分成了各自的亚群[7]或群集[22]。主成分分析结果显示,某些园艺类型的材料聚集在一起(如茎生生菜),与其他类型的材料分离良好,而其他类型的材料聚类不佳(图2)3.)。例如,脆生菜似乎形成了三个独立的子集群,一个包含四个成员(Great Lakes, Winterhaven, La Brillante和Iceberg),第二个包括Vanguard, Salinas, Salinas 88和Batavia Reine des Glaces,第三个包括两个成员(Calmar和Empire)。由于这种分布,F圣脆型的值通常较低,范围从只有0.038(长叶莴苣)到0.145(茎型莴苣)。尽管PCA明确地将所有野生种与栽培莴苣区分开来,l.serriola离的簇最近吗l.漂白亚麻纤维卷登记入册,l.virosa是离这个星团最远的(图3.,插入右上角)。观察到的野生物种与l.漂白亚麻纤维卷这三个品种与栽培莴苣的性亲和性相对应。同样,标记转移率最高的是l.serriola(83%),是栽培莴苣的近亲,其次是l.saligna(66%)和l.virosa(63%)。这些结果与先前使用基因组SSRs和EST-SSRs获得的观察结果一致[22]。
数据集的一致性和基因型多样性
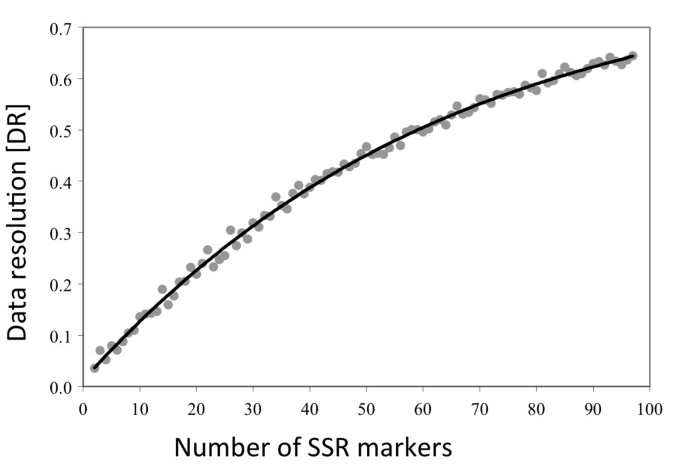
DR分析显示了曲线的预期形状,2个标记的初始值较低,为0.036,97个标记逐渐增加到0.644(图1)4)。DR值越高,说明分析标记越多,SSR数据集内部一致性越高。范·欣图姆[23观察到类似的结果l.serriola用AFLP标记分型。在我们的分析中,需要61个SSR标记才能达到0.5的DR;Van Hintum的估计[23]表示大约需要70个AFLP标记才能达到相同的DR值。先前观察到,对于相同数量的标记,SSR数据集的一致性通常高于显性标记数据集的一致性[23,34]。
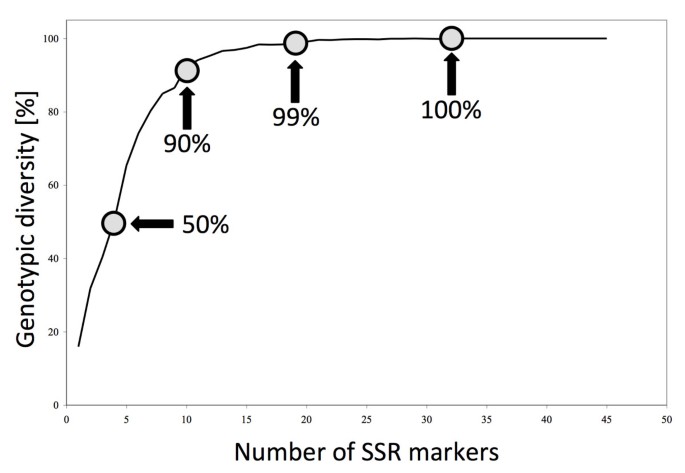
为了分析获得更多SSR标记是否增加了区分更多基因型的可能性,对36份材料进行了基因型多样性分析。平均而言,鉴定50%的基因型只需要4个标记,鉴定90%的基因型需要10个标记,鉴定99%的基因型需要19个标记(图2)5)。我们的分析表明,任意32个SSR标记都能明确区分所有36份材料的基因型。这是基因分型所需的相对较高数量的标记。例如,鉴定54个甜菜杂交种平均只需要17个SSR标记[34]。然而,某些园艺生菜品种之间的变异率一般很低,而同一品种品种之间的相似性很高[7,22]。因此,需要更多的分子标记来区分遗传相似性高的近缘物质。此外,本研究中测试的一些SSRs来自同一基因组区域,从而限制了它们区分基因型的能力。
SSR标记在种间分子连锁图谱上的分布与聚类
基于在盐碱地(Salinas)观察到的等位基因分离,我们在莴苣分子连锁图谱上定位了54个基因组SSR标记。l.漂白亚麻纤维卷(x) x (x)l.serriola)映射种群(图6)。由于亲本之间的纯合性或与框架图上的标记的弱连锁,其余的ssr未被定位。SSR标记分布在9个连锁群(LG)上。标记物最少的是LG 9(2个),最多的是LG 8(12个)。拟合优度检验表明,标记在连锁组上的分布与标记的均匀分布无显著差异(p= 0.115)。除了基因组SSRs,我们还绘制了52个先前开发的EST-SSRs [22],使已映射的ssr总数达到106个。有趣的是,最少的EST-SSRs位于lg8(两个标记)上,而lg8拥有最多的基因组SSRs(12个标记)。然而,基因组SSRs和EST-SSRs在所有LGs中的分布差异不显著(p= 0.056)。与基因组SSRs相似,EST-SSRs以及基因组和EST-SSR组合标记的分布与所有LGs上标记的均匀分布没有显著差异(p= 0.422p= 0.770)。Truco等。[25]在同一定位群体中报道了729个AFLP和18个SSR标记。LG 4上标记最多(142个),LG 9上标记最少(51个)。
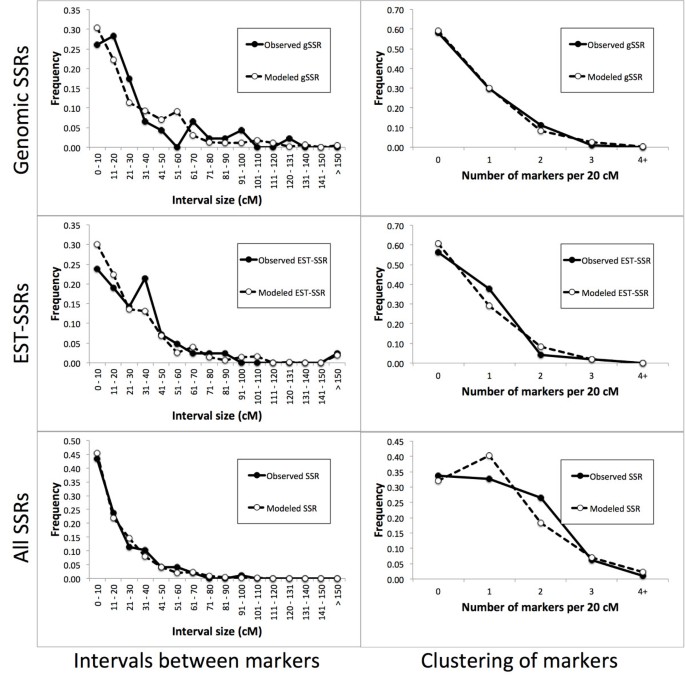
两个连续标记之间的平均间隔长度对于基因组SSRs为29 cM,对于EST-SSRs为30 cM,当考虑所有已定位的SSRs时为18 cM。区间长度的全基因组分布与模型分布吻合较好(图2)7,左栏)。无论是K-S测试还是χ2拟合优度检验发现,模型和观测到的区间分布(p取值范围为0.502 ~ 0.823)。同样,每20 cm长片段中观察到的标记数量与基于标记随机分布的建模数据吻合良好(图2)7(右列)。的pK-S检验的拟合优度检验值为0.586 ~ 0.960,K-S检验的拟合优度检验值为0.203 ~ 0.738χ2测试。我们观察到,标记的模型聚类与基于泊松分布(相关性)的理论聚类非常接近r= 0.999;rEST-SSRs = 0.995r= 0.993)。因此,泊松分布可用于识别连锁图中标记聚类高于预期的部分[35]。用泊松分布公式,在那里k(k= 0,1,2,3,…)是计算概率的每段标记的数量,λ是每段标记的平均数量,我们计算出可疑的标记聚类(at)p< 0.01),如果一个20厘米长的片段包含5个或更多的标记(当仅单独考虑基因组ssr或est - ssr时,每个片段有4个或更多的标记表明可能存在聚类)。分子连锁图的检查显示一个单一的区域与可能的聚类标记。该集群位于LG 6,其中9个标记(6个基因组SSRs和3个EST-SSRs)位于标记LSSB3B和SML-023之间约44 cm长的间隔内。我们的结果与之前的研究一致,表明ssr的分布通常是均匀的;虽然一些标记的聚类(主要围绕着着丝粒区域)是可能的[36,37]。在生菜中,先前报道了aflp在多个基因组区域的分子标记聚类,而只有少数区域显示了RFLP和RAPD标记的聚类[25]。
结论
我们开发了一套97个基因组ssr,并将其中54个定位在生菜种间分子连锁图谱上。SSR标记在基因组中大多随机分布,在LG 6上可能存在单个区域的标记群。根据基因分型结果样本,每个样本的最大估计基因分型误差可达8%。当分析等位基因的大小差异小于3bp时,错误率最高。这一误差率与玉米的误差率相似[38],尽管比其他一些报告中的数字要高[39,40]。一般来说,用基因组ssr对生菜进行基因分型比用EST-SSR对生菜进行基因分型产生更高的错误率[22]。增加错误率的因素包括存在口吃带,每个位点的等位基因数量多,以及产品尺寸大[39]。在我们的基因分型系统中观察到的相对较高错误率的另一种可能性是,eGene DNA分析仪的分辨率低于其他一些用于SSR分析的仪器[41]。新开发的基因组SSRs集与先前开发的EST-SSRs相结合,将有助于品种指纹识别、构建综合分子连锁图谱和定位感兴趣的基因[42]。
数据访问
所描述的序列已提交到GenBank数据库,注册号为JX474909至JX474987。
缩写
- AMOVA:
-
分子方差分析
- 英国石油公司:
-
碱基对
- cM:
-
厘摩
- 博士:
-
数据解析
- 美国东部时间:
-
表达序列标签
- 钴测试:
-
Kolmogorov-Smirnov测试
- 磅:
-
溶原性肉汤
- 格林:
-
连锁群
- 拿拿淋:
-
每个SSR标记的座数
- 主成分分析:
-
主成分分析
- 聚合酶链反应:
-
聚合酶链反应
- RGC:
-
抗性候选基因
- 苏维埃社会主义共和国:
-
简单序列重复
- 过程中:
-
遗传杂合性的无偏估计。
参考文献
[4]王晓明,王晓明,王晓明,等。中国水稻属植物同工酶标记的遗传变异及系统发育分析摘要以.中国生物医学工程学报,2004,31(2):326 -331。
王晓明,王晓明,王晓明,等。植物RFLP基因多态性的研究进展。栽培莴苣(L. sativa)的起源。中国生物医学工程学报,2001,34(4):433 -436。10.1139 / g91 - 065。
Kesseli RV, Paran I, Michelmore RW:详细的遗传连锁图谱分析摘要以(莴苣)由RFLP和RAPD标记构建。遗传学报,1994,26(3):344 - 344。
李建军,李建军,李建军,李建军,等。基于aflp的pcr指纹图谱研究摘要以[j] .理论与应用,1996,(3):1202-1210。10.1007 / BF00223451。
范德威,陈晓明,王晓明,等。微卫星遥感技术在我国莴苣研究中的应用(摘要以l .)。基因工程学报。1999,42(2):139-149。
胡杰,Ochoa OE, Truco MJ, Vick BA:诱捕技术在生菜上的应用(摘要以l .)基因分型。植物生态学报,2009,29(4):557 - 557。10.1007 / s10681 - 005 - 6431 - 1。
胡军,胡军。栽培莴苣种群结构及其对关联图谱的影响。中国生物医学工程学报,2008,33(3):661 - 668。
Simko I, Pechenick DA, McHale LK, Truco MJ, Ochoa OE, Michelmore RW, Scheffler BE:莴苣抗枯死基因的关联定位和标记辅助选择Tvr1.植物生态学报,2009,29(9):1359 - 1368。
Kwon SJ, Truco MJ, Hu J: LSGermOPA, 384个est衍生的高通量生菜snp的定制OPA (摘要以L.种质指纹图谱;中国生物医学工程学报,2012,29(4):887- 991。10.1007 / s11032 - 011 - 9623 - 5。
Stoffel K, van Leeuwen H, Kozik A, Caldwell D, Ashrafi H, Cui X, Tan X, Hill T, Reyes-Chin-Wo S, Truco MJ, Michelmore RW, van Deynze A:莴苣单位置多态性的650万特征affytrix基因芯片的开发与应用[j]。摘要以spp)。中国生物医学工程学报,2012,31(3):559 - 564。
Edwards KJ, Barker JHA, Daly A, Jones C, Karp A:植物微卫星序列库的富集。生物技术学报,1996,20:758-760。
王晓明,王晓明,王晓明,等。DNA微卫星引物的分离与鉴定巨滑舌鱼牡蛎这是亚马逊流域一种经济上重要但被严重过度开发的鱼类。生物工程学报,2003,32(3):1280 - 1290。10.1046 / j.1471-8286.2003.00375.x。
Glenn TC, Schable NA:分离微卫星DNA位点。方法:中国生物医学工程学报,2005,35(5):522 - 522。
李建军,李建军,李建军,等。WebSat -一种用于微卫星标记开发的网络软件。生物信息学报,2009,32(3):282-283。10.6026 / 97320630003282。
Rozen S, Skaletsky H:为一般用户和生物学家程序员提供的入门级WWW。分子生物学方法,第132卷:生物信息学方法和协议。编辑:Misener S, Krawetz SA人类出版社,托托瓦NJ, 2000, 365-386。
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ:基本的局部对齐搜索工具。中国生物医学工程学报,1997,26(2):444 - 444。
Peakall R, Smouse PE: GENALEX 6: Excel基因分析。用于教学和研究的种群遗传软件。生物工程学报,2006,26(6):387 - 398。10.1111 / j.1471-8286.2005.01155.x。
Meirmans PG, Van Tienderen PH: GENOTYPE和GENODIVE:两个分析无性生物遗传多样性的程序。生物化学学报,2004,4(4):792-794。10.1111 / j.1471-8286.2004.00770.x。
对少数个体的平均杂合度和遗传距离的估计。遗传学报,2008,29(2):583- 589。
Michalakis Y, Excoffier L:基于微卫星位点特殊参考的等位基因间距离估计种群细分。遗传学报,1996,42(2):1061-1064。
Excoffier L, Smouse PE, Quattro JM: DNA单倍型间距离的分子变异分析:在人类线粒体DNA限制性数据中的应用。遗传学报,1993,31(1):479-491。
用于莴苣群体结构研究的EST-SSR标记的开发(摘要以l .)。[J]中国生物医学工程学报,2009,31(1):369 - 369。
van Hintum TJL:数据分辨率:确定分子标记数据集一致性的折刀程序。计算机工程学报,2007,31(1):343-349。10.1007 / s00122 - 007 - 0566 - 5。
李春华,李春华。多基因座连锁不平衡指数。生物通报,2001,31(1):101-102。10.1046 / j.1471-8278.2000.00014.x。
Truco MJ, Antonise R, Lavelle D, Ochoa O, Kozik A, Witsenboer H, Fort SB, Jeuken MJW, Kesseli RV, Lindhout P, Michelmore RW, Peleman J:莴苣高密度综合遗传连锁图(摘要以spp)。理论与应用,2007,15:735-746。10.1007 / s00122 - 007 - 0599 - 9。
Manly KF, Cudmore RH,Jr Meer JM: Map Manager QTX,跨平台基因定位软件。中国生物医学工程学报,2001,12:930-932。10.1007 / s00335 - 001 - 1016 - 3。
高瑞敏,李建平,李建平,等:线粒体基因组研究马吕斯有明显以及种子植物线粒体基因组扩增的进口驱动假说。植物科学学报,2012,31(1):615-626。10.1111 / j.1365 - 313 x.2012.05014.x。
michamore RW, Meyers BC:植物抗性基因簇通过分化选择和生灭过程进化。中国生物医学工程学报,1998,8(3):559 - 559。
Scott KD, Eggler P, Seaton G, Rossetto M, Ablett EM, Lee LS, Henry RJ:葡萄est的ssr分析。理论与应用,2000,100:723-726。10.1007 / s001220051344。
Cho YG, Ishii T, Temnykh S, Chen X, Lipovich L, mcouch SR, Park WD, Ayres N, Cartinhour S:水稻基因组库和GenBank序列微卫星多样性研究(ei)栽培稻l .)。理论与应用,2000,100:713-722。10.1007 / s001220051343。
李建军,李建军,李建军,等。小麦est微卫星标记的分离与分析。理论与应用学报,2002,(4):394 - 397。10.1007 / s001220100738。
Pashley CH, Ellis JR, McCauley DE, Burke JM: EST数据库作为分子标记的来源:经验教训向日葵.[J]中国生物医学工程学报,2006,27(3):381-388。
李建军,李建军,李建军,等。火炬松est单拷贝微卫星标记的研究。理论与应用,2004,(10):361-369。
Simko I, Eujayl I, van Hintum TJL:甜菜杂交品种基因分型、聚类和分配的DArT、SNP和SSR标记系统的实证评价。植物科学,2012,18(4):554 - 562。
杨华平,Schupp JM, Keim P:大豆基因组DNA甲基化和AFLP标记分布。理论与应用,1999,(9):785-792。10.1007 / s001220051297。
Röder MS, Korzun V, Wendehake K, Plaschke J, Tixier MH, Leroy P, Ganal MW:小麦微卫星图。遗传学报,1998,39(4):779 - 779。
王晓明,王晓明,王晓明,王晓明,王晓明。小麦双单倍体六倍体群体遗传连锁图谱中DArT、AFLP和SSR标记的分布。基因工程学报。2006,49:545-555。10.1139 / g06 - 002。
Jones ES, Sullivan H, Bhattramakki D, Smith JSC:用于玉米基因型分析的简单序列重复和单核苷酸多态性标记技术的比较(英文)玉米l .)。计算机工程学报,2007,31(1):361-371。10.1007 / s00122 - 007 - 0570 - 9。
王晓明,王晓明,王晓明,等。微卫星基因分型错误:检测方法、常见来源及后果。中国生物医学工程学报,2005,35(4):599-612。
[3]王志强,王志强。微卫星标记物的应用与评价。生态学报,2006,(9):615-629。10.1111 / j.1461-0248.2006.00889.x。
王晓,Rinehart TA, Wadl PA, Spiers JM, Hadziabdic D, Windham MT, Trigiano RN:微卫星等位基因电泳分离新技术。生物工程学报,2009,28(8):2432-2436。
黄春华,李春华,李春华,等。小麦白粉病抗性的QTL定位。植物病理学杂志,2009,19 (3):591 - 591
致谢
作者要感谢R. Michelmore、M. Truco、O. Ochoa、L. McHale和R. Hayes提供种子和/或标记信息,B. Scheffler提供测序服务,A. Atallah、A. Folck和M. Estrada提供技术支持。这项工作得到了加州绿叶蔬菜研究项目的部分支持。本出版物中提及的商品名称或商业产品仅用于提供特定信息,并不意味着美国农业部的推荐或认可。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
GR和IS设计了研究,GR进行了实验室实验和序列分析,IS进行了建模和标记数据分析,GR和IS编写了稿件。两位作者都阅读并批准了最终的手稿。
电子补充资料
12870 _2012_1207_moesm1_esm.xlsx
附加文件1:引物组合及扩增基因组SSR标记的PCR条件。请注意,一些引物对扩增来自同一测序产物的两个不同的微卫星。不完全重复序列用星号(*)表示。(xlsx54kb)
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
Rauscher, G., Simko, I.莴苣指纹基因组SSR标记的开发(摘要以L.)品种和定位基因。BMC Plant Biol13,11(2013)。https://doi.org/10.1186/1471-2229-13-11
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-11
关键字
- 数据解析统计
- 基因分型
- 摘要以
- 连锁图
- 标记分布
- 微卫星