摘要
背景
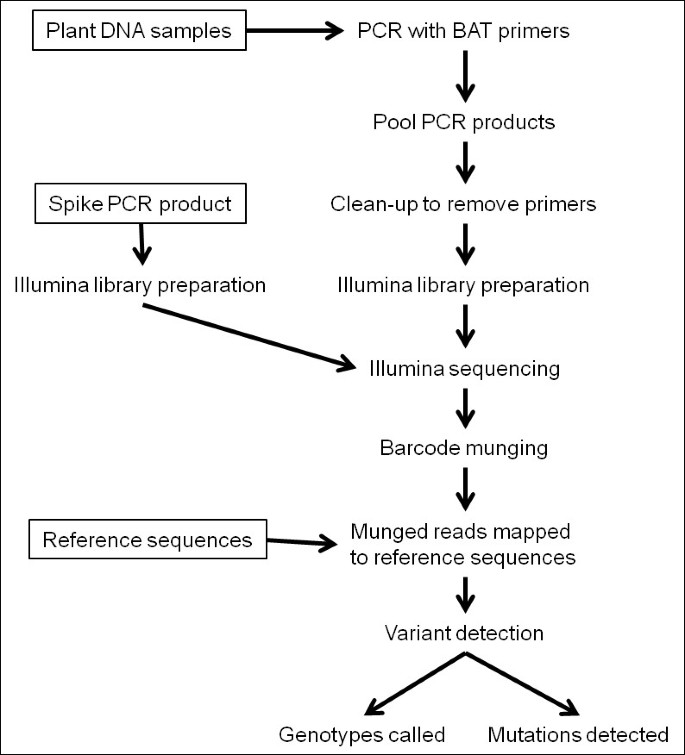
遗传变异的检测和利用是作物改良的基础。然而,我们许多最重要的作物基因组的多倍体特性代表了一个障碍,特别是对基因变异的分析。为了克服这一问题,我们的目标是开发基于扩增子测序的方法,包括将条形码扩增标签(BATs)纳入PCR产品。
结果
开发了一种方案,在使用标准Illumina适配器进行测序文库生产之前,用5 ' 6碱基寡核苷酸条形码扩展标记PCR产物。提出了一种计算方法,用于产品的去卷积和序列变异的鲁棒检测和评分。使用这种方法,针对基因序列的扩增子被筛选显著绘制种群和由此产生的24个标记的等位基因评分字符串,映射到基因组的预期区域。此外,使用一维8倍池化,4608行a显著对突变人群进行基因座特异性扩增子诱导突变的筛选GL2.b)和三个共扩增位点(同源基因)的混合产物FAD2),分别鉴定出10个和41个突变体。
结论
通过Illumina测序,利用条形码标签在多重聚合聚合酶链反应产物中去旋,为SNP基因分型和突变检测提供了一种经济有效的方法,甚至在多倍体物种中,也可能为致病变化提供标记。将这种方法与现有的Illumina多路复用工作流程相结合,可以在一次测序运行中以最低的库生产成本廉价高效地分析数千行。
背景
到2050年,世界人口预计将达到91亿,比今天增加34%,而要满足全球需求,粮食产量将增加70% [1].扩大种植面积是一种越来越不可能和昂贵的解决办法[2]虽然可以通过改进农艺管理来获得收益[3.],目前遗传学和基因组学的进展不仅为作物遗传研究提供了许多机会,而且为技术辅助的品种开发提供了许多机会。
“模式”植物物种的基础研究,如拟南芥,是第一个获得高质量基因组序列的植物[4]的研究,导致了对植物发育、代谢和环境相互作用的许多方面的控制基因的鉴定。然而,我们最重要的许多作物,包括面包小麦、马铃薯、棉花和甘蔗都是多倍体,作物物种中的许多农艺性状通常由多个基因或数量性状位点(QTL)控制。因此,低成本、高效、高密度的标记平台对于通过QTL分析、关联研究或突变筛选促进比较遗传作图和候选基因和有利等位基因的鉴定至关重要。最终,这些平台还将有助于将这种遗传变异成功地纳入育种计划和优良品种的开发,特别是在遗传力低的性状逐渐渗入时(即环境影响相对较大的地方)。
常用的分子标记,如扩增片段长度多态性(AFLP) [5],限制长度多态性(RFLP) [6]和简单序列重复(SSR) [7]通常用低通量琼脂糖或丙烯酰胺凝胶电泳检测大小或构象多态性。利用毛细管测序技术通过将荧光标记加入到PCR产物中实现了多路复用。Schuelke(2000)开发了一种通用的M13尾部三引物标记系统,允许氟标记与许多不同的特定引物组合使用,并以最有效的排列进行多路复用,降低了基因分型成本[8],然而,作为一种分析,它们仍然是昂贵的每个数据点。
阵列技术的最新发展允许在单个杂交试验中筛选个体上的数千个标记。多样性阵列技术(DArT)标记可提供成本相对较低的基因分型,无需事先提供序列信息,已广泛应用于作物的基因分型[9]、[10].最近,单核苷酸多态性(SNP)已成为作物改良的主要标记类型。Illumina的金门[11]是目前最流行的SNP标记分析技术之一,该技术基于BeadArray/Chips,在并行检测大量标记时,提供每数据点低成本的基因分型,但与DArT不同的是,它需要先前的序列知识。基因组DNA与顺磁粒子结合,并添加一个寡核苷酸池,其中包含具有通用引物位点的等位基因特异性引物和携带珠地址序列的snp链接的位点特异性引物。在扩展和结扎之后,进行等位基因特异性氟标记PCR,所得到的产物与载玻片上排列的携带位点地址序列的珠子杂交。然后通过读取每个地址的荧光进行基因分型。相当大的样本集合(最多384个)可以为定制的标记库进行基因分型,同时最多可达1536个SNPs [9].在这项技术的基础上,Illuminas Infinium阵列也使用珠子芯片,但基于整个基因组片段的随机扩增[12].每个芯片分为4至24个部分,每个部分包含100万至90,000个珠点[13],既可以通过加载单个样品的不同珠池/切片对数十万个snp进行基因分型,也可以通过在每个切片中加载不同的样品对数千个snp进行基因分型。这些高通量SNP基因分型技术可以有效地对相对较少的个体进行大量标记的评分。然而,对于标记引导育种,更典型的要求是对数千个个体的少数标记进行分析。随着我们识别等位基因变异的精确分子基础的能力不断提高,重点将从关联匿名标记的发展转移到基因内的致病变异,在多倍体物种中出现特殊问题,其中将包含多个非常密切相关的基因,这会干扰分析。
高通量第二代测序方法,即使是其台式平台,每次运行也可产生高达1.6 Gb的序列[14],已经彻底改变了我们进行基因分析的方法,其中基因分型测序(GBS)越来越受欢迎。使用Illumina测序技术已对SNP发现、连锁映射和关联遗传学进行了阶跃变化,甚至对基因组序列不可用的多倍体物种也是如此[15- - - - - -17].尽管这些测序技术对于从单个样本中产生大量的序列数据是非常划算的,但它们并不适合从大量样本中产生适量的序列数据。这是因为测序库的制备是昂贵的。迄今为止,绕过这一问题的尝试集中在基于变体适配器的索引或条形码的各种策略上,使每次仪器运行的多个样本多路复用,并已用于植物中适度数量的扩增子分析以及人类遗传研究[18- - - - - -20.].然而,测序适配器的长度和设计限制使得这种方法过于昂贵,无法在作物物种的大量目标位点的数千个PCR产物中进行序列变异鉴定。
目前检测突变筛选中简单序列变异的方法依赖于靶向诱导的局部病变基因组(TILLING)等方法[21],该方法识别来自聚合植物的位点特异性PCR产物中的序列变异,代表基因的编码区域,通常使用一种内切酶(Cel1),在异双工DNA的错配处裂解[22,23]或对目标扩增子进行常规Sanger测序。这些技术虽然已经很成熟,但依赖于基因中位点特异性扩增子的发展,这在多倍体作物中经常是有问题的。正如Wang所演示的,TILLING已经在具有非常高保守水平的混合扩增子上进行了et al。(2008),他们在两者的混合扩增子中鉴定出突变体BnFAE1基因,其序列相似性为98.6% [24].然而,通过传统的TILLING方法筛选这种混合扩增子几乎是不可行的,因为在整个扩增子的每个错配位置都会产生异双工裂解产物,从而掩盖了群体中诱导突变的更罕见(和更弱)的信号。
在本研究中,我们的目标是开发一种快速、可靠和低成本的技术,即使是在基因内进行SNP基因分型,因此有可能部署“完美的”标记,代表等位基因变异的致病变化。我们以全球重要的油料作物油菜为代表的多倍体,在Illumina NGS平台上建立了方法学;物种芸苔属植物显著.油菜是最近形成的多倍体,据信是在不到10,000年前,在二倍体之间的杂交事件中出现的芸苔属植物物种b·拉伯(基因组)和b . oleracea基因组(C)。与二倍体的比较映射答:芥建议芸苔属植物基因组由祖先基因组的三种重新排列的变体组成[25,使情况更加复杂显著基因组结构。由于这种复杂性,位点特异性PCR扩增子并不总是能够获得(特别是对于同源位点,即A和C基因组中的对应位点),这限制了通过常规方法筛选基因组中序列变异的位点数量。基于一种完善的荧光标记方法,我们开发了一种将寡核苷酸条形码纳入PCR产品的方法[26].在测序文库生产之前,可以从许多不同的反应中汇集条形码扩增子,并使用标准的Illumina工作流程进行测序。基于多倍体中SNP发现所开发的变异的计算分析[16]用于混合扩增子(包括同源位点对)的SNP检测。在这项研究中,我们选择了接近两个重要脂肪酸生物合成基因家族成员的位点:BnaFAD2而且BnaSAD3.BnaFAD2有四份显著位于A1 (BnaA.FAD2.b-like), a5 (BnaA.FAD2.a), c1 (BnaC.FAD2.b)及C5 (BnaC.FAD2.a),BnaSAD3有六份副本,位于A1 (BnaA.SAD3.a), c1 (BnaC.SAD3.a), a3 (BnaA.SAD3.b), c3 (BnaC.SAD3.b)及A5 (BnaA.SAD3.c)及C5 (BnaC.SAD3.c).通过将标记物转换为传统的GoldenGate分析来验证映射结果。此外,该方法还适用于在ems治疗人群中识别靶基因家族中的病变。这些靶标由一个特定的扩增子组成BnaC.GL2.b(拟南芥的同源植物GLABROUS2,GL2)和三个共扩增副本的混合产物Bna。FAD2.
结果与讨论
PCR产物条形码
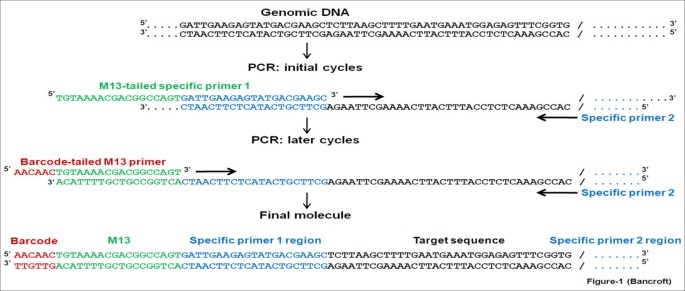
一种向PCR产物添加荧光标签的现有方法[8]被用作我们将寡核苷酸条形码添加到PCR产品的方法的基础。在PCR反应中使用了三种寡核苷酸:(1)在其5 '端具有M13(−21)尾的序列特异性正向引物,(2)在其5 '端具有寡核苷酸条形码尾的通用M13(−21)引物(以下称为条形码扩增标记;BAT)和(3)序列特异性反向引物。通过限制带有M13(−21)尾的序列特异性正向引物的反应量,可以在单级反应中进行PCR,其中前几轮扩增包含通用正向引物,后几轮扩增包含BAT引物。条形码过程如图所示1, BAT实验设计示意图如图所示2.
328个条形码进行了放大测试,192个,显示在附加文件中1当用7个测试扩增子放大时,它产生了单一的、干净的条带,被选择用于进一步的实验。
SNP基因分型
通过设计包含半snp标记的PCR扩增子,即从两个基因组中扩增出的位点,对BAT尾迹进行多重基因分型进行了研究显著,其中只有一个品种间存在等位变异,是多倍体中数量最多的标记类型[16].总共有35个表达基因转录区域的扩增子,见附加文件2,旨在包含先前检测到的半snp多态性[16].其中,通过PCR产物的毛细管测序显示,24个位点包含预期的多态性(其中一些位点包含额外的snp),但其余11个位点的多态性无法验证,这些位点被认为是单态的。然而,为了将序列复杂性提高到更现实的水平,所有35个都被用于评估。所有35个扩增子的PCR扩增成功地进行了46个系的子集显著TNDH定位群体(加上亲本品种Tapidor和宁优7号),PCR产物在多个单独的试管中扩增(具有不同的条形码),并结合用于Illumina测序库的生产。通过添加“spike”测序库来补充条形码和读取的M13(−21)区域上的碱基组合,避免了在Illumina运行的一个周期中,在大量簇中合并相同碱基的风险,从而触发错误并终止运行。在簇生成之前,以约1:10的最终库摩尔浓度的比例将约280碱基长度的尖刺添加到样品库中。
在GAII平台上,Illumina测序的一个lane(76碱基单端reads)产生了8,002,602个单端reads用于基因分型。对这些成分进行了分析,结果见表1.在序列读取中,1.1%(89,517)对应于“spike”库,22%(1,725,988)包含M13(−21)序列。在后者中,只有2.3%的人携带完整的6碱基条码,18.4%的人携带5碱基条码,42.2%的人携带4碱基条码,37%的人携带不到4个碱基条码。这种条形码的截断很可能是由于外切酶在连接到Illumina适配器之前降解了分子的末端。然而,由于在实验中使用的地址空间只需要条形码的最后四个碱基,63%携带M13(−21)标签的读取可用于基因分型。截断为4或5个碱基的序列通过添加2个碱基或1个碱基(视情况而定)进行修复(计算),以恢复完整的6个碱基条形码。
我们开发了一种计算方法,允许序列变异的鲁棒检测,基于先前报道的方法,涉及Illumina reads映射到参考序列[16,17].具有完整/修复的6碱基条形码的序列读取通过一种称为“munging”的过程将其条形码复制四次来处理。这种篡改确保了读取到正确参考序列的特定映射,因为四个不匹配(篡改条形码之间的最小差异)将取消读取的映射。然后使用Maq v0.7.1 [27]以引用包含特定放大序列加上M13(−21)和所占用地址空间的四倍munged条形码的序列。这样,Maq处理的64.4%(699,449)读序列被映射到一个参考序列,并通过munged BAT与单个行相关联。使用Perl脚本识别相对于祖先参考序列的扩增序列中的变体,并根据SNP位置读取的碱基计数调用基因型(至少读取20个碱基)。生成常规标记评分字符串(基于A等位基因用于种群的母本,Tapidor, B等位基因用于父本,Ningyou7和U(例如由于读取深度不足而无法调用等位基因)。
由此得到的24个标记(来自17个扩增子)的评分字符串被成功链接映射,如图所示3.并在附加文件中列出3.分别在A3、A5、C1、C5连锁群上有14个标记,在A1、A3、A5连锁群上有10个标记。其余的snp由于缺乏多态性或读取深度过低而无法进行稳健的等位基因调用而无法被映射。为验证,Illumina GoldenGate平台[11]标记被开发了包含由BAT方法绘制的SNPs的8个unigenes,并在整个种群中进行了评分。此外,来自已发表的油菜SNP图谱的评分字符串[17],并将两种类型标记的评分字符串与BAT方法获得的评分字符串进行比较,见附加文件4.BAT标记的得分准确率较高,483个等位基因中仅有5个误判得分(1.0%)。
基因突变检测
利用BAT尾迹进行突变检测,通过在两个位点特异性的176 bp扩增子中搜索ems诱导的突变进行了研究GL2;BnaC.GL2.b)和一个276 bp的扩增子,代表三个共扩增位点FAD2;BnaA.FAD2.a,BnaA.FAD2.b而且BnaC.FAD2.a)在EMS诱变人群JBnCAB_E(来源于显著各种配合)。对于三组单独的集合,每一组集合用于Illumina测序文库的构建,使用192种不同的bat进行PCR扩增,筛选通过一维8倍池法组合的1536个种群。使用三个Illumina GAII测序通道(76个碱基,配对末端reads)筛选4608个扩增子突变系。平均每个车道获得56,898,937个读取。表中显示了读取和映射分析的摘要2.同样按照SNP基因分型所描述的最终文库摩尔浓度的1:10比例添加的Spike序列,平均占读数的4.9%(2,791,863)。在30.5%(17,329,509)的reads中检测到M13(−21)序列。与初始实验相比,在改进方案后,外切酶活性大大降低,平均89.5%(15,505,456)包含完整的条形码,3.7%(638,532)携带5 bp代码,1.7%(290,934)携带4 bp代码。只要有可能,这些都被修复了。只有3.8%的条形码小于4 bp,因此无法修复以允许行识别。由于可以识别序列末端对,通过计算将相邻条形码的M13(−21)序列添加到配对端(最初对应于序列特定的反向引物),导致平均56.8%(32,317,892)用于读映射和突变检测。对扩增子参考序列的映射导致32,317,892个“munged”reads中平均有62.5%(20,195,287)被成功映射,与SNP基因分型分析中的映射reads比例相似。然而,在扩增子中,读值并不是均匀分布的FAD2orthologuesBnaA.FAD2.a,BnaA.FAD2.b而且BnaC.FAD2.a分别以平均8.5%,32%和4.1%的读数表示。这可能是由于共扩增位点的PCR效率存在偏差。以3:1的摩尔比从库中生成集群BnaFAD2图书馆BnaGL2两个扩增子的库。然而,平均55.5%(11,202018)的映射reads来自于BnaC.GL2.b扩增子。
在读映射之后,Maq被用于识别相对于野生型参考序列的扩增序列中的变体,基于一些参数调用突变。研究发现,在特定的8行中调用突变的一组适当的标准是:(1)至少10个读取携带序列变体,(2)至少1%的调用显示混合扩增子目标的变体。FAD2);至少0.2%的位点特异性目标(即。GL2),(3)来自参考碱基的主要变体比在该位置调用的任何其他变体碱基多出至少10倍,(4)该位置不应对应于一个para - inter(或inter- homoologue)多态性。在此标准下,我们检测到14、19、8和10个突变系BnaA.FAD2.a,BnaA.FAD2.b,BnaC.FAD2.a而且BnaC.GL2.b,分别。使用位点特异性引物从原始(非汇总)DNA样本中PCR扩增这些已识别突变的子集。2、4、2和7都测试了假定的突变BnaA.FAD2.a,BnaA.FAD2.b,BnaC.FAD2.a而且BnaC.GL2.b,分别被确认存在,见表3..种群中可能存在其他突变,但由于对某些品系的序列读取过少而未被检测到;这是每行用于PCR扩增的DNA池数量和/或用于文库构建的DNA池数量不平衡的结果,后者在本实验中未归一化。
结论
我们描述了一种稳健且具有成本效益的寡核苷酸条形码方法,通过一步纳入PCR产品,适用于Illumina测序的标准工作流程。然后,计算方法允许对序列进行反褶积,并对已知多态性进行评分和检测突变。我们已经演示了使用4个碱基的寡核苷酸条形码地址空间,但是使用这种方法,如果使用更多的尾部地址空间(可能超过6个碱基),则可以聚合和分析更多的PCR产物,尽管包括一些未使用的碱基有助于减轻任何外切酶污染的影响。合成和验证的192个BAT寡核苷酸面板可以与许多不同的m13尾特异性引物重复使用,使得该方法比合成位点特异性PCR引物或结合条形码的位点特异性文库适配器更具成本效益。
当前下一代测序平台上的多路复用系统依赖于文库生产适配器的条形码。在图书馆建设之前对产品进行条形码编码,提供了一种以最低的图书馆生产成本实现多路复用的系统。由于它不涉及用于测序文库生产的适配器的修改,聚合PCR产品可以用于标准的Illumina工作流程,包括当前的索引技术,因此可以在测序仪的每个通道内实现更高阶的多路复用,并(潜在地)在单个通道中分析数千个聚合样本,只涉及几十个测序文库的生产。即使使用了相同的bat,也可以对多个不同的靶标位点进行反卷积(因为大多数测序的扩增子在不同的靶标之间是不同的)。
我们对GoldenGate SNP平台和bat -tail的结果进行了比较,结果表明该方法为SNP标记的基因分型提供了一个强大而经济的系统,即使在多倍体物种中也是如此。它特别适用于作物育种项目中的标记辅助选择等应用,因为少量的标记可以在数千个个体中筛选。事实上,该技术可用于定位和开发基因内致病序列变化的标记,这些标记是更可取的(在可以识别的地方),因为它们是“完美的”标记,不受通常与基于仅仅遗传关联的多态性的标记相关的破坏的影响。虽然目前使用低成本平台(如Illumina)可以测序的扩增子长度比传统TILLING可以评估突变的长度(~1 kb)短,但技术的进步继续增加读取长度,现在使用Ion Torrent平台可以达到400 bp,例如[28].然而,扩增多个较小的扩增子用于bat -tail分析的额外成本已经被该过程的效率所抵消,该过程能够检测多个共扩增pcr产物中的突变。利用该方法检测混合扩增子内突变的能力确立了bat -tail作为多倍体突变育种方法的一种有价值的技术。
方法
材料与方法
BAT PCR扩增
这种基于Scheulke描述的M13氟标记方案的条形码扩增标记策略的PCR [8]用5 μl 20 ng/μl DNA, 2 μl 10 × PCR缓冲液(500 mM KCl, 100 mM Tris-HCl (ph9.0), 1% Triton X-100 15 mM MgCl进行测定20.5 μl正向引物(0.5 mM), 2.3 μl反向引物(2mm), 2.3 μl条形码M13(−21)引物(2mm), 1.3 μl dNTPs (2 μM Invitrogen Cat。0.2 μl Amplitaq Gold 5 u/μl (Applied Biosystems Cat. 10297-018);No. 4311820), 6.4 μl ddH2O对以下循环:94°C保存5分钟,40 ×(94°C保存30秒,49°C保存30秒,72°C保存30秒),72°C保存7分钟,在8°C保存。
SNP标记分析设计和文库制作
用于测试的SNP标记被设计用于检测代表基因模型的同源基因的unigenes中的半SNP多态性答:芥与脂质生物合成相关基因接近的基因组(FAD2At3g12120;SAD3串联三份,At3g02610/20/30)。使用BLAST对齐芸苔属植物unigenes反对答:芥在CDS模型中,假定的外显子被识别出来(基于内含子通常会在相关物种的同源体中处于相同位置的假设,如答:芥而且显著).在这些假定的外显子序列中设计PCR引物,目的是设计大约130个或280个碱基的扩增子。通过对栽培品种Tapidor和宁优7的扩增、毛细管测序和微量文件的检测,验证PCR产物的多态性。用于Illumina序列reads映射的参考序列被设计用于表示祖先碱基位于半snp位置的扩增序列(即已解析的碱基等位基因)(见附加文件)2,作为参考序列)。
46个要进行基因分型的TNDH系(加上Tapidor和Ningyou7)中的每一个都被分配了一个特定的条形码(见附加文件)5用于条形码分配)。对35次检测中的每一次PCR产物进行聚合,按照制造商的说明,在Illumina mRNA-seq单端文库从协议的适配器连接反应阶段生产之前,使用PAGE清洗和浓缩样品。库的准备方法如Bancroft所述et al。2011年(17].
穗样制备
通常情况下,Illumina实时分析软件会在所有簇报告在给定周期内合并相同核苷酸的情况下出错,但这种情况会发生在PCR扩增子读取的通用引物部分。为了克服这一问题,实验样品以1:10的比例加标,基于文库摩尔浓度(文库被归一化到大约10 nM), DNA组成的序列应该与M13和条形码区域的序列互补。制备了两组穗样,一组长度约为130个碱基,另一组长度约为280个碱基,每组由两个不同的扩增子组成。对~130碱基的穗,用来自显著品种Tapidor和引物对R00005和D00002(分别为bbnnnnvhvbbbbbbdhbdhhddbhvcagagcacgtggagcag和tgcatttctgttggtttcacc)和R00006和D00006(分别为BBNNNNVHVBBBBDHBDHHDDBHVCGTACATTTGAGAGACGTTCC和GAGCAGCGAGCAATGCAGC),扩增产物被池化。对于~280个碱基的刺突,用来自显著品种Tapidor和引物对R00008和D00032(分别为bbnnnnvhvbbbbbbdhbdhhddbhvtgacagagagacctcctc和gatctccgctccaagatatgg)和R00009和D00041(分别为BBNNNNVHVBBBBDHBDHHDDBHVGATGTCTTCAAGAGCCCAGC和GATAGCAGTGACAATATCTCC),扩增产物被池化。按照与汇总条形码实验样本相同的方法,生成了一个用于较小尺寸范围的扩增子(~130个碱基)测序的库,以及一个用于较大尺寸范围的扩增子(~280个碱基)测序的库。质量检查及加工工作按上文所述进行[17].
Illumina公司测序
Illumina在GAII平台测序,质量检查和处理如前所述[17].使用GERALD处理Illumina碱基调用文件,生成成对序列文件,每个样本包含80个碱基读取,采用FASTQ格式。在进一步处理之前,Illumina FASTQ格式被转换为Sanger FASTQ格式。
数据处理
为了处理条形码的读取,从而处理单个PCR井或池,我们开发了一种基于先前报道的技术的新方法[16,17].我们使用Maq v0.7.1 [27]对包含目标扩增子的参考序列进行比对,并识别错配(snp或突变)。然而,条形码区域(编码样本起源)中读取序列和参考序列之间的不匹配必须从目标区域(这将标志snp或突变)中分割出来。在非常高水平的多路复用(如本实验)中,由6个基本条形码提供的4 K地址空间的大部分被占用,单个条形码可能仅相差一个基址。通常,这可能导致交叉对齐,即从给定条形码派生的野类型读取可能会映射到地址空间中紧密相邻的不正确条形码。我们通过替换1 - 1来解决这个问题,在网上,真正的六基条形码,其人工构造源自更大的地址空间,因此能够携带更多的识别信息。我们的简单实现是将条形码复制四倍(“munging”),这样一个碱基的差异就被放大到四个,从而击败了使用默认参数的Maq的交叉对齐。这是通过简单的Perl脚本mung_barcoded_reads.pl(附加文件6)和mung_refseqs.pl(附加文件7)分别对读和引用操作。在我们的分析过程中,我们发现有一部分读取的条形码是不完整的,但这些条形码可以被挽救,因此只有那些与M13引物完全匹配且至少有四个条形码碱基(并且可以被重建回唯一的完整条形码)的条形码才会被使用。
鉴定并去除与PCR穗扩增子相对应的Reads,并将单个系与BAT中的序列相关联。根据在SNP位置读取的碱基计数,使用Maq(默认参数)将munged reads与每个标记的参考序列和所调用的基因型进行对齐,但前提是SNP位置至少被20个高质量的reads覆盖(否则等位基因被定义为未分类,U),使用Perl脚本BAT_SNPs.pl(附加文件8).
SNP标记映射
使用Joinmap 3.0版本执行链接映射[29] [30.]在TNDH群体测序的46个株系中的42个株系的数据上(其余4个株系产生的映射序列相对较少,因此很大比例的等位基因仍然未分类)。BAT SNP标记被添加到基于该人群的现有SNP图谱的最新版本[17],之前映射的标记被分配到固定的顺序。映射标准如下:默认重组(REC)阈值(0.4)和差异概率(LOD)值的对数(2.0)[30.].平均chi平方贡献,即标记对拟合优度的平均贡献,小于2。
突变筛选扩增子设计与DNA池化策略
设计引物以产生一个276 bp的三种混合扩增子BnaFAD2副本请于显著栽培品种cabriollet和一个176 bp的扩增子BnaC.GL2.b.(见附加文件)9每个扩增子的正向引物都有一个5 ' M13(−21)尾巴(附加文件)5:表S5)。
DNA浓度的测定显著Cabriolet EMS人群,JBnCAB_E,使用PicoGreen (Molecular Probes, Invitrogen Corporation, Carlsbad, California, USA)比对Tecan Genios平板阅读器上的通用DNA浓度标准,4608个0.6和0.8% EMS品系的DNA归一化至1ng / ul。然后使用一维八倍池化策略将DNA进行池化。
192个单独的M13引物标有一个独特的6 bp“条形码”,其序列可用于识别产品起源的池,用于两个池板,其中每个孔都有一个唯一标记的产品,并按Illumina车道进行组合和运行。不同的目标基因扩增和标记相同的条形码可以组合在同一车道内运行。
序列库建设
分别处理两个扩增子,PCR产物来自每对96孔板,标记有192个BAT序列,分别分配到各个孔,并将30 ul通过Min-Elute反应柱(Qiagen cat。编号28204)生产3 × 10 ul清洁,浓缩产品。在Agilent DNA 1000生物分析仪上运行每个柱的2 ul洗脱液,以检查质量并在混合剩余洗脱液之前量化DNA浓度。浓度调整为20-25 ng/ul, 23 ul用于Illumina mRNA-seq配对端文库的生产,从协议的适配器连接反应阶段开始,按照制造商的说明进行。在聚类之前,每个扩增子的单个文库归一化到~10 nM,摩尔比为3:1。SNP标记检测增加Spike库。
突变数据处理
突变检测使用用于SNP检测的程序的改编。通过Maq(带有默认参数)对munged引用进行竞争对齐。然后使用maq pileup -v命令生成封装这些对齐和相关基本质量信息的文本文件。Perl脚本TILLING_parse.pl(附加文件10)被开发用于在目标扩增子内的每个ems可变基上提取所谓的基频率组成,使用基质量,通过访问预索引版本的堆积文件。
基于一些启发式方法,将池识别为可能包含突变系。首先,携带EMS突变候选基因的绝对读数必须超过10,然后,对于特定扩增子(基于多路复用深度的预期),该候选基因与参考(野生型)碱基的调用信号之比必须大于0.002,对于混合同源扩增子的产物必须大于0.01,最后,信噪比(计算为候选变量的调用数与其他两个可能的基数变化的调用数之比)必须大于10倍。
突变的确认
使用原bacc . gl2对来自阳性识别池的个体进行包含候选突变的特异性扩增子的PCR。B引物和BnaFAD2引物和组合,详见附加文件11,12而且13如前所述,在制备、测序和突变检测之前进行57°C标准PCR循环。
参考文献
- 1.
粮农组织:粮食安全世界首脑会议。如何在2050年养活全世界。2009年,罗马:粮农组织,2009年
- 2.
戈弗雷HCJ,贝丁顿JR,克鲁特IR,哈达德L,劳伦斯D,缪尔JF, Pretty J,罗宾逊S,托马斯SM,图尔明C:粮食安全:养活90亿人的挑战。科学通报,2010,27(3):344 - 344。10.1126 / science.1185383。
- 3.
测试者M, Langridge P:在不断变化的世界中增加作物产量的育种技术。科学通报,2010,27(3):344 - 344。10.1126 / science.1183700。
- 4.
拟南芥基因组计划:分析开花植物拟南芥的基因组。中国生物工程学报,2000,29(4):489 - 497。
- 5.
Vos P, Hogers R, Bleeker M, Reijans M, Lee T, Hornes M, frites A, Pot J, Paleman J, Kuiper M, Zabeau M: AFLP:一种新的DNA指纹识别技术。核科学,1995,23:4407-4414。10.1093 / nar / 23.21.4407。
- 6.
孙文杰,王晓明,王晓明。限制性片段长度多态性在遗传改良中的应用。应用理论,1983,67:35-43。10.1007 / BF00303919。
- 7.
Levison G, Gutman GA:滑移链错配:DNA序列进化的主要机制。中国生物医学工程学报,1997,4:439 - 439。
- 8.
Schuelke M: PCR片段荧光标记的经济方法。中国生物工程学报,2000,18:334 - 334。10.1038/72708。
- 9.
陈晓明,王晓明,王晓明,等。基于DNA标记的作物改良研究。遗传,2008,101:5-18。10.1038 / hdy.2008.35。
- 10.
杨丽娟,杨晓明,王晓明,王晓明。基于基因分型技术的基因分型研究。核酸工程,2001,29:e25-10.1093/nar/29.4.e25。
- 11.
Deulvot C, Charrel H, Marty A, Jacquin F, Donnadieu C, lejeun - henaut I, Burstin J, Aubert G:豌豆高多重SNP基因分型的遗传定位和种质多样性研究。中国生物医学工程学报,2010,26(4):468-10.1186/1471-2164-11-468。
- 12.
张伟,李国强,张志强,张志强,李志强。基于单碱基延伸法的全基因组基因分型研究。化学学报,2006,3:31-33。10.1038 / nmeth842。
- 13.
Infinium HD化验。2012年,Illumina公司,http://www.illumina.com/technology/infinium_hd_assay.ilmn,
- 14.
Loman NJ, Misra RV, Dallman TJ, Constantinidou C, Gharbia SE, Wain J, Pallen MJ:台式高通量测序平台的性能比较。生物技术学报,2012,30:434-439。10.1038 / nbt.2198。
- 15.
李志刚,李志刚,李志刚,李志刚。甘蓝型甘蓝型油菜的转录组学研究。生物技术学报,2012,30:798-802。10.1038 / nbt.2302。
- 16.
杨晓明,王晓明,王晓明,王晓明,等。基于Solexa转录组测序的甘蓝型油菜(Brassica napus)单核苷酸多态性研究中国生物工程学报,2009,7:334-346。10.1111 / j.1467-7652.2008.00396.x。
- 17.
Bancroft I, Morgan C, Fraser F, Higgins J, Wells R, Clissold L, Baker D, Long Y,孟J,王霞,等:多倍体油菜基因组转录组测序分析。生物技术学报,2011,29:762-766。10.1038 / nbt.1926。
- 18.
De Leeneer K, Hellemans J, De Schrijver J, Baetens M, Poppe B, Van Criekinge W, De Paepe A, Coucke P, Claes K:乳腺癌基因BRCA1和BRCA2的大规模平行扩增子测序:机遇、挑战和限制。胡木,2011,32:335-344。10.1002 / humu.21428。
- 19.
Homolka A, Eder T, Kopecky D, Berenyi M, Burg K, Fluch S:基于454个扩增子测序的奥地利橡木10个候选干旱响应基因的等位基因发现。中国生物医学工程学报,2012,29(5):457 - 457。
- 20.
范德伟,李志强,李志强,等:油菜茎秆抗锈病QTL的快速鉴定多年生黑麦草.物理学报,2011,29(3):344 - 344。10.1007 / s00122 - 011 - 1546 - 3。
- 21.
Stemple DL: TILLING -功能基因组的高通量收获。自然科学评论,2004,5:5-
- 22.
麦凯伦,李志刚,刘志刚,刘志刚。植物功能基因组学的研究进展。中国农业科学,2003,23(4):439- 439。10.1104 / pp.123.2.439。
- 23.
Oleykowski CA, Bronson Mullins CR, Godwin AK, yang AT:利用一种新型植物核酸内切酶进行突变检测。中国生物医学工程学报,1998,26(4):497 -4602。10.1093 / nar / 26.20.4597。
- 24.
王王N, Y,田F,国王GJ,张C,长Y, L,孟J:芸苔属植物的功能基因组学资源显著:EMS诱变处理人口发展和发现FAE1点突变的耕作。中国生物工程学报,2008,30(3):344 - 344。10.1111 / j.1469-8137.2008.02619.x。
- 25.
林永平,林永平,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚,王志刚芸苔属植物显著以及它的二倍体亲戚。植物学报,2004,30(4):344 - 344。10.1111 / j.1365 - 313 x.2004.02244.x。
- 26.
杨强,范超,郭震,秦娟,吴娟,李强,付涛,周勇:识别FAD2而且FAD3基因在芸苔属植物显著高油酸和低亚麻酸含量等位基因特异性标记的基因组和开发。清华大学学报(自然科学版),2012,29(3):344 - 344。10.1007 / s00122 - 012 - 1863 - 1。
- 27.
李红,阮娟,杜斌R:基于质量评分的DNA测序短序列测序。基因组学报,2008,18:1851-1858。10.1101 / gr.078212.108。
- 28.
Johnson J:离子激流达到400 bp阅读长度标记…为什么我们兴奋。2012年,http://www.edgebio.com/ion-torrent-hits-400bp-read-length-markwhy-were-excited,
- 29.
Stam P:利用一种新的计算机包:连接图构建综合遗传连锁图。植物学报,1993,3:339 - 344。10.1111 / j.1365 - 313 x.1993.00739.x。
- 30.
Van Oijen JW, Voorrips RE: Joinmap 3.0,用于计算遗传连锁图的软件。荷兰:瓦赫宁根:2001年。
确认
我们感谢Sainsbury实验室生成Illumina序列数据。这项工作得到了英国生物技术和生物科学研究委员会(BBSRC;LK0843)和HGCA (RD-2007-3356)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明没有相互竞争的经济利益。
作者的贡献
IB和RW构思并策划了该项目。RW, FF, ES和LC进行了实验。IB、RW、MT、FF、CLM进行数据分析。JP开发并进行了GoldenGate分析并验证了标记物,IB和RW撰写了手稿,所有作者都对其进行了审查。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2013_1320_moesm1_esm.docx
附加文件1:用于扩增子BAT的192个条形码M13引物,用于生产干净的单带PCR产品。条形码底漆细节字表。(docx16kb)
12870 _2013_1320_moesm2_esm.docx
附加文件2:使用BAT检测转录组SNP标记。引物序列Word表,SNP细节和检测标记的单基因位置。(docx19kb)
12870 _2013_1320_moesm3_esm.docx
附加文件3:BAT法评分SNP标记的连锁映射。GoldenGate和BAT评分确定的基因分型SNP标记的映射位置Word表和BAT SNP评分的读取深度细节。(docx13kb)
12870 _2013_1320_moesm4_esm.xlsx
附加文件4:在不同平台上生成的等位基因得分的比较。包含SNP标记的等位基因评分字符串的MS Excel文件。(xlsx21 kb)
12870 _2013_1320_moesm5_esm.docx
附加文件5表S5:使用BAT进行SNP转录组标记基因分型的条形码分配。字表包含个别人口线条形码分配。(docx12kb)
12870 _2013_1320_moesm7_esm.pl
附加文件7:Perl脚本将6nt条形码附加到扩增子和endoreduplicate以创建伪引用序列。(pl 2 kb)
BAT突变检测扩增子。
附加文件9:放大序列显示Bna。FAD2家庭(面板a)和BnaC.GL2.b(面板b).扩增子区域用颜色编码:条形码(蓝色),M13(红色)和特定引物(绿色)位置。在实验中,可以用76个碱基重测序的扩增子区域是没有颜色的。未被76 bp读数覆盖的区域为灰色。描述:BAT突变屏幕中覆盖的序列区域说明。(docx18kb)
12870 _2013_1320_moesm10_esm.pl
附加文件10:Perl脚本用于从Illumina读取的EMS填充与参考序列中解析Maq堆积文件。(pl 5kb)
coamplification和
附加文件11:Bna。FAD2coamplification和BnaC.GL2.b -用于产生BAT突变筛选扩增子的特定引物。包含筛选扩增子引物细节的单词表。(docx11kb)
用于扩增的引物
附加文件12:Bna。FAD2位点。用于扩增的引物的字表Bna.FAD2。(docx12kb)
位点特异性扩增所需的引物组合
附加文件13:FAD2orthologues。包含引物组合的单词表。(docx12kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
威尔斯,R,特里克,M,弗雷泽,F。et al。基于序列的多倍体油菜变异检测。BMC植物生物学13日,111(2013)。https://doi.org/10.1186/1471-2229-13-111
收到了:
接受:
发表:
关键字
- 单核苷酸多态性
- 突变
- 多倍体
- 作物