摘要
背景
在人类和细菌系统中,核苷磷酸化酶(NPs)在代谢核苷酸修复和肿瘤发生中的作用已被广泛研究。在植物中,np样蛋白尚未得到全面的研究,可能是因为没有证据表明其在核苷回收中具有代谢功能。然而,在森林树木属杨树一个np样蛋白家族在季节间和季节内的氮储存和循环中起着重要的生态生理适应作用。
结果
我们进行了系统发育分析,以确定np样蛋白在植物中的分布和进化。这些分析揭示了植物中两个主要的np样蛋白簇。I组蛋白由广泛的植物分类群的基因编码,而II组基因编码的蛋白则由属于该目的物种主导Malpighiales包括杨树树皮储存蛋白(BSP)和win4样蛋白。此外,我们对NP-like基因在杨树通过检测13NP-like基因发现于杨树暴露于长日(LD)和短日(SD)光周期的植物的不同组织的基因组。我们发现所有13个杨树NP-like属于第一类或第二类的基因在LD和SD的各种组织中都有表达。自然选择试验和表达进化分析杨树基因表明,基因表达的分化可能发生在最近的进化过程中杨树,支持自适应维护模型。最后,在网上分析独联体- 13个发起人中的监管因素NP-like基因在杨树揭示了已知参与光调节、应激/发病机制和植物激素反应的共同调控元件。
结论
在杨树在美国,np样蛋白和基因家族的进化是由复制事件和自然选择形成的。表达数据表明,以前未被表征的np样蛋白可能在营养感知和/或信号传导中起作用。这些蛋白属于一类np样蛋白,广泛分布于许多植物类群中。我们得出结论,np样蛋白可能在植物中起作用,尽管这种功能尚不明确。
背景
核苷酸、核苷酸前体和衍生物是生命的基本组成部分。它们组成核酸,充当信号分子、细胞间能量转运体,并可转化为必需的酶辅因子。因此,核苷酸代谢是一种必要的细胞功能[1,2]。在哺乳动物和细菌系统中,核苷磷酸化酶(NPs)通过在无机磷酸盐(Pi)存在下切割(脱氧)核糖核苷的糖苷键来挽救核苷,从而产生(脱氧)核糖-1-磷酸和核碱基[1,3.]。游离核碱基可合成有机分子或被降解,(脱氧-)核糖-1-磷酸可被戊糖磷酸途径和糖酵解利用[3.]。研究最广泛的NPs是嘌呤核苷磷酸化酶(PNPs),因其在突变相关免疫缺陷疾病、前列腺癌、白血病和牙周病中的作用而成为临床和癌症研究的焦点[4- - - - - -6]。
在保留和再动员氮(N)的情况下,回收嘌呤的能力对氮有限的植物尤为重要[7,8]。提出的嘌呤降解在植物中的生理作用是通过从源到汇的动员来提高氮利用效率[8]。然而,没有证据表明NPs参与植物嘌呤核苷的回收;相反,核苷的水解发生在核苷酶(EC 3.2.2x) [9- - - - - -11]。值得注意的是,嘌呤回收和降解途径中的基因可由损伤、干旱、脱落酸(ABA)、黑暗条件和黑暗诱导的衰老诱导,这与NUE中的作用一致[12- - - - - -14]。虽然嘌呤回收似乎是NUE的一个组成部分,但np样蛋白在植物中与核苷回收和NUE有关的作用尚不清楚。
有大量的研究是关于一个与PNPs序列相似的蛋白质家族杨树(由[15])。在这个属中,这些蛋白质在季节性和短期氮储存中具有生态生理作用。这些蛋白质的一个亚家族被称为树皮储存蛋白(BSPs),因为它们在秋季积聚在树皮薄壁和木质部射线中,而在春季恢复生长时丰度下降[16- - - - - -18]。BSP的秋季积累和相关基因表达是光敏色素介导的光周期反应[19,20.]。此外,BSPs和相关的营养储存蛋白(VSPs)基因也在伤害、高氮和干旱胁迫下表达,表明其在短期储存中起作用[j]。21- - - - - -23]。BSPs促进的季节性储存可能是一种重要的进化适应,促进了低营养森林系统的多年生生长,而短期储存有助于保护营养物质以应对压力。这种季节性适应将生长对氮的需求与吸收和同化分开,并在低氮供应条件下提供竞争优势[24,25]。杨树为研究np样蛋白和基因提供了一个模型系统,这不仅是因为它们在季节性和短期储存中的重要性,而且因为它们的存在杨树比任何已知的植物属拥有更多的np样蛋白质。
np样蛋白参与季节性氮循环可能是由于蛋白质和基因家族的扩展和功能进化,这为适应和物种形成提供了创新[26,27]。基因复制是这种创新的一种机制,它通过全基因组复制(WGD)或小范围基因组复制(如串联复制(TD))发生。[28]。在重复之后,基因可以有多种命运:重复的基因可以放大或缓冲原始功能[29- - - - - -31],获得新的功能(新功能)[32,33],积累使原始功能细分的突变(亚功能化)[34,35]或变得无功能(伪化)[32]。由于季节性氮循环可能是一种适应性性状,因此我们考虑了适应性维持和基因扩增的主要模型,包括正剂量、新功能化、亚功能化、多功能基因多样化和剂量平衡模型。36])。正剂量描述了通过缓冲或功能冗余来增加适应度的重复基因的保留[29,30.,37]。新功能化是指在保留亲本拷贝的同时,通过中性突变和正选择,在重复基因中获得新的功能[32,33]。亚功能化模型表明,重复基因中的中性突变削弱或改变了原始功能,从而维持两个副本执行原始功能[35,36]。在多功能基因多样化模型下,一个多功能亲本基因在基因副本之间是解耦的[38]。剂量平衡假说有助于解释重复事件类型后基因的保留:重复维持蛋白质复合物的化学计量平衡,有利于具有许多相互作用的基因和蛋白质的高保留率[39,40]。
摘要本研究的目的是探讨植物中np样蛋白的系统发育和进化关系杨树以及植物界,以进一步了解它们的功能进化,以及它们可能在多大程度上参与养分回收,特别是氮循环和氮肥利用。我们首先构建了13个物种之间的进化关系NP-like基因在杨树在长日(LD)和短日(SD)条件下,研究了四种组织类型的转录本丰度,并比较了这些基因的启动子区域结构,以深入了解基因的表达和调控。我们进一步研究了基因的功能差异,通过测试是否有基因在不同的自然选择下,以及基因表达何时出现分歧。最后,我们对整个植物界的np样蛋白进行了系统发育分析。我们对自然选择和基因表达模式进化的分析,然后在这个更广泛的系统发育背景下进行考虑,以得出关于进化模式和可能的基因命运的结论NP-like体内的基因和蛋白质杨树.
结果
蛋白质系统发育与染色体定位NP-like基因家族杨树
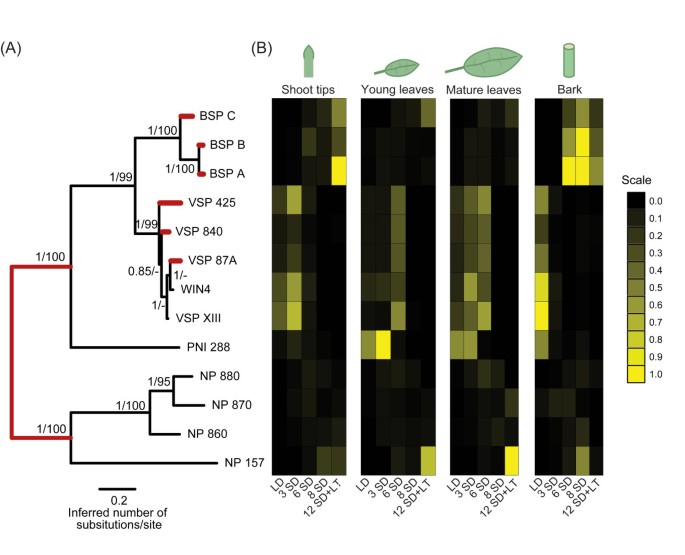
为了更好地理解其中的类np蛋白之间的关系杨树我们构建了一个进化树的基础上,从全长蛋白质序列检索杨树trichocarpa通过Phytozome的基因组(表1)。这棵树表示具有强支持度、后验概率为1.0、bootstrap支持度超过99%的聚类蛋白的三个亚家族(图1)1第一个亚族由BSP A、BSP B和BSP C组成,称为BSP亚族(图2)2)。第二组蛋白包括WIN4和WIN4样蛋白,被称为WIN4样亚家族。第三个亚家族包括四个未表征的np样蛋白,我们将其命名为np样亚家族。PNI 288聚集在由WIN4和BSP亚家族组成的簇中,但基于该分析,该蛋白与WIN4和BSP亚家族不同(图2)1A)初级基因结构NP-like转录本揭示了在每个基因亚家族中发现的共同的内含子-外显子边界(图2)2)。
的内含子-外显子结构NP-like基因在杨树trichocarpa.利用原代构建了基因结构的比较NP-likePhytozome (http://www.phytozome.net)和在PIECE数据库(http://wheat.pw.usda.gov/piece/index.php)。NP-like基因亚家族用括号表示。
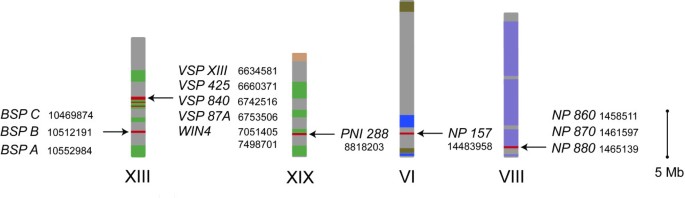
基因的染色体位置显示杨树NP-like由三个亚家族组成的基因位于四条染色体上(图2)3.)。有两组NP-like在13号染色体上的基因,包括所有染色体的一个簇BSP亚家族成员和第二个集群,包括WIN4-like亚家族基因成员。的NP-like亚家族基因聚集在染色体VIII上。只有288句和NP 157被发现没有与其他成员聚集在一起NP-like基因与288句位于第19号染色体上NP 157位于六号染色体上。
染色体定位NP-like基因在杨树trichocarpa.箭头表示基因的大致位置。罗马数字表示染色体名称,数字反映起始位点位置(http://www.phytozome.net)。彩色块表示已知的同工区杨树trichocarpa基因组v2.0和这些坐标的链接可以在方法中找到。
的表达模式杨树NP-like基因家族
基因表达分析NP-like基因家族p . trichocarpa采用qPCR对LD、SD和SD联合低温处理植物的4种组织类型(茎尖、幼叶、成熟叶和树皮)进行评估(表1和图1B).总体而言,每个亚家族内的基因表达与特定类型的组织和环境处理有关。三者的表达BSP在SD处理期间,以及SD与茎尖LT联合处理后,树皮中的s增加(图2)1B)尽管这三种表达制订与SD相关,观察到一些表达差异,特别是诱导BSP C在SD处理的最后4周与低温相结合时,在SD处理12周后的幼叶和成熟叶中的表达式BSP的和BSP B是非常相似的,这两个基因彼此关系更密切,而BSP C更遥远。
与BSP表达主要与SD和多年生组织(茎尖和树皮)相关的亚家族WIN4-like亚科成员仅在LD处理植株的树皮和SD处理初期(3周和6周)的茎尖、幼叶和成熟叶中较多。尽管如此WIN-4-like持续暴露于SD导致mRNA的稳态丰度下降。表达的下降WIN4-like经SD处理的植株中,树皮基因的表达最为显著。这些基因表达模式表明BSP在茎尖和树皮等多年生组织中,亚科成员与SD密切相关WIN4 -亚家族在这些多年生组织中与LD相关。此外,与BSP亚科的WIN4 -亚家族成员在幼叶和成熟叶中也有更大程度的表达。288句表达式类似于成员的WIN4-like在SD处理期间表达下降的所有组织中均检测到。
除了NP 157,表达NP-like基因(NP 880,NP 870,NP 860)在芽尖、幼叶、成熟叶和树皮中发生的水平较低BSP和WIN4-like亚科。的表达式NP-like亚家族基因在所有组织中也倾向于与SD相关。NP 157被表达到比其他成员更高的水平NP-like亚家族和表达在所有组织中都与SD相关。表达的最高水平NP 157在SD处理12周后(SD +低温处理后4周),在幼叶和成熟叶中均观察到
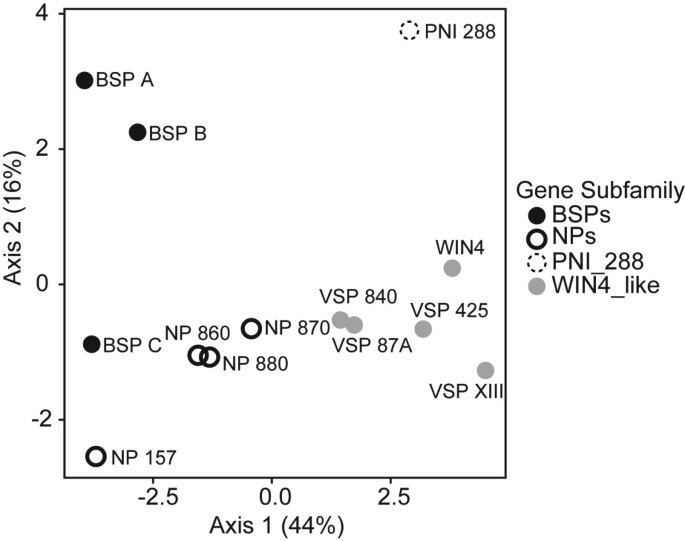
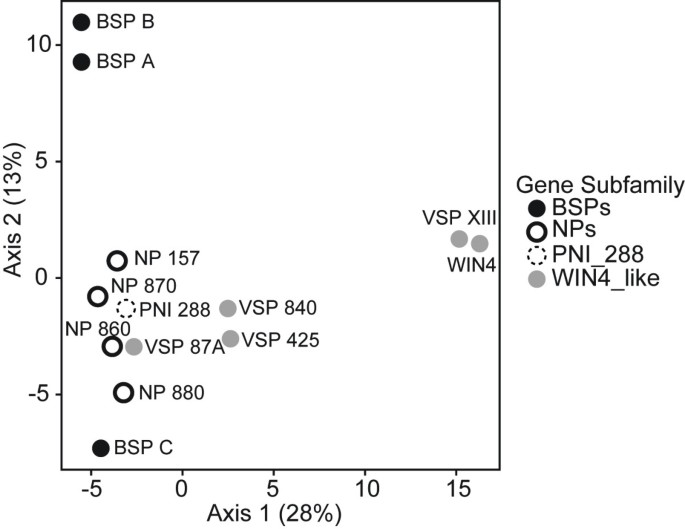
总而言之,这三者的成员NP-like亚科(BSP,WIN4-like和NP-like)被观察到具有与每个亚家族相关的相似表达模式。委员会成员BSP亚家族在SD中表达,并倾向于多年生组织。委员会成员WIN4-like亚家族存在于多年生和落叶组织中,但受SD抑制。的NP-like亚科存在于多年生和落叶组织中,但只有一个成员;NP 157,在不同组织中似乎显示出一致的SD反应。结合系统发育分析,我们的基因表达分析证明了系统发育定义的相关性NP-like亚家族和基因表达。基于表达式数据的PCA也支持这种关系(图5)4),这表明同一基因亚家族的成员比其他基因亚家族的成员更接近彼此。在这个分析中,PCA轴1和轴2分别解释了44%和16%的方差。为了确定通过PCA产生的非平凡成分的数量,我们将观察到的特征值与通过对观察数据集的10000个随机矩阵进行分析所产生的分布进行了比较(例如,λk)。基于这种方法,我们发现只有第一个轴的特征值显著大于(P< 0.05),比随机预期值高。
的主成分分析NP-like基因表达杨树trichocarpa.的关系的PCANP-like基因在杨树trichocarpa根据图中所示的表达式数据1B.每个轴解释的变化百分比在每个轴旁边的括号中给出。
自然选择和持续的性格进化
基于分支站点REL方法,我们发现了具有统计学意义的情景多样化选择的证据垂直地震剖面425,垂直地震剖面840,垂直地震剖面87,BSP的,BSP B和BSP C(Holm-Bonferroni纠正p-value < 0.05)(图1A和表2)。我们还在基因树的基础上发现了多样化选择的证据,这些选择与最初的基因复制相对应,产生了系统发育集群,称为第I组和第II组np样蛋白(图2)1A和表2)。
对基因表达模式是否有系统发育信号λ的测试与没有信号的替代(即性状进化的布朗运动模型)有显著不同(表2)3.)。此外,我们测试的进化差异之间的np样蛋白杨树说明在进化过程中,表达差异集中在较晚的阶段。具体来说,δ大于1,这表明最近发生了进化变化,并且将所有分支的δ次方提高的模型比布朗运动模型更适合(表1)3.)。
在网上启动子序列分析NP-liKe基因家族杨树
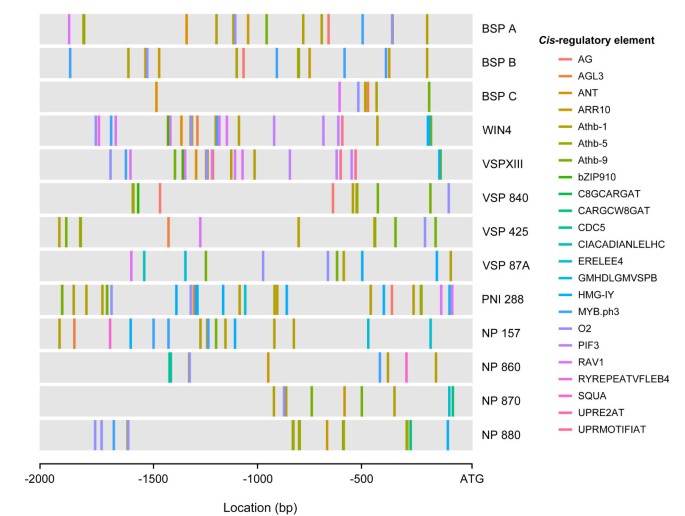
我们在上游的2kb区域中发现了过度代表性的基序杨树NP-like表现的基因在网上分析使用两个预测程序,MEME和MotifClick [41,42]。我们确定了与已知的相对应的基序独联体-转录因子(TF)数据库整合程序PlantPAN中的调控元件(CREs) [43]。在由MEME和MotifClick识别的200个基序中,有23个基序与已知的cre相对应(图2)5、表4、附加文件1表6)。这些区域已被证明与TFs结合,TFs可能调节一系列组织中的转录,并对植物激素、应激和光照做出反应(表1)4)。CREs在杨树NP-like可能促进组织特异性表达的基因启动子(AG, AGL3, Athb-1, -5, -9, CDC5, MYB)。ph3, O2 and SQUA), phytohormone responses (ARR10, ERELEE4, RAV1 and RYREPEATVFLEB4), stress responses (RAV1, UPRE2AT and UPRMOTIFIAT) and light responses (CIACADIANLELHC and PIF3). Both motif prediction programs identified motifs corresponding to the known CREs AG, AGL3, Athb-9, O2 and RAV1 (Table4)。
基于各基因预测基序数的主成分分析结果与系统发育关系基本一致。沿着PCA轴1,它解释了数据中28%的方差,启动子区域的基序垂直地震剖面十三世和WIN4基因主要是从细胞分离出来的BSP和NP基因(图6)。PCA轴2解释了数据中13%的方差,主要分离了基因的启动子区域的基序BSP来自另一方的基因NP-like基因。然而,沿着轴2的启动子区域的基序BSP的和BSP B与启动子的基序比较,它们是紧密相连的吗BSP C它在轴的负端是孤立的。基于λk采用上述方法来评估非平凡成分的数量,我们发现轴1和轴2的特征值显著大于(P< 0.05)。
植物界的系统发育分布
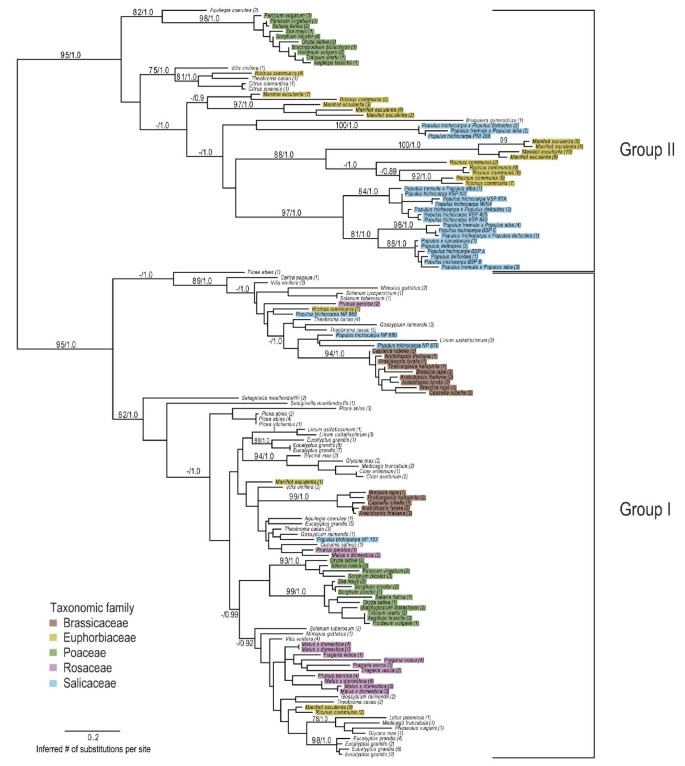
为了研究植物np样蛋白的多样性,我们检索了植物界142个完整和非冗余的氨基酸序列(附加文件)2:表S5),基于BLASTP搜索,具有NP序列同源性。基于38个属预测的NP-like区域比对的系统发育分析揭示了2类一般的NP-like蛋白(图2)7)。组I和组II有很强的支持度,后验概率为1.0,bootstrap支持度为95%。第一类包括来自所有代表属的np样蛋白,它们具有完整的基因组序列,其亚类通常根据Rosids和单子叶谱系聚集[44,45]。第一类还包括杨树不知道参与储存的蛋白质(np样亚家族成员)。包括细菌外群的系统发育分析可以在附加文件中找到3.:图S1外群的加入不会改变植物NP群的拓扑结构。
植物界np样蛋白的系统发育分析。采用贝叶斯和最大似然方法构建系统发育关系。分支上的数字分别表示基于1000次重复的后验概率和自举百分比。括号中的数字对应于Phytozome或NCBI序列标识符,可以在附加文件中找到7表S4。五个主要的分类科由高亮的颜色表示。
II族主要由蛋白质组成杨树,木薯耐和萝藦。有一个较小的蛋白质簇来自单子叶属和两个柑橘类蛋白质。的杨树第二组蛋白包括BSPs、win4样蛋白和PNI 288(图2)7)。
讨论
蛋白质系统发育与染色体分布NP-like基因家族杨树
先前的研究确定了三种BSP基因,三WIN4-like基因和288句[15]。在这项研究中,我们检查了杨树基因组测序并鉴定了另外6个基因,将该基因家族扩展到13个成员。根据我们的系统发育分析,我们建议将这个基因家族命名为NP-like基因家族有三个亚家族,包括BSP亚家族(三个基因成员)WIN4-like亚家族(5个基因成员)和NP-like亚家族(四个基因成员)。此外,288句也是np样基因家族的成员,但不属于三个亚家族中的任何一个。
植物界内np样蛋白的进化关系及染色体分布的蛋白质系统发育表明NP-like基因家族杨树通过WGD和TD活动进行扩展。这与之前的研究是一致的杨树基因组(46- - - - - -48]。发生了WGD事件杨树大约6500万年前(数百万年前)或更早,随后是基因组广泛的重组,在其他变化中,导致了类似的染色体组,其中XIII和XIX是一对(其中288句和BSP/WIN4-like亚科分别居住)[46- - - - - -48]。染色体VI和VIII(其中NP 157和NP-like子家族,分别居住)不是一个平行的集合[46]。每个基因亚家族的成员位于一个100kb的区域内,可能是TD的结果(图2)2)。这种基因之间的接近通常是TD的良好迹象[49,50]。重复事件的类型(即TD或WGD)对基因功能有影响杨树通过TD扩展的细胞具有防御反应、细胞凋亡和蛋白激酶等功能[49]。
在网上的启动子分析NP-like基因家族杨树
启动子分析是研究基因功能进化的重要组成部分,特别是在基因由TD产生的情况下。这是因为TD主要是通过不平等重组发生的,如果调控区域没有复制,这可能导致亚功能化[51]。这些分析还提供了关于可能的调控和组织特异性表达的信息,这对未来研究确定功能至关重要。
尽管有可能从中获得有益的结论在网上从生物信息学的角度来看,启动子分析、基序发现是困难的[52]。仅使用已知cre数据库来定位启动子区域的cre会导致高假阳性率,并且不能证明这些区域是TF结合位点。为了提高可信度,我们使用了2个motif预测程序(MEME和MotifClick)来识别在2 kb的启动子区域中过度代表的8 bp motifNP-like基因在杨树.从结果基序中确定已知的cre并进行映射(表2)4,图5)。这两个程序都预测了与TFs AG、AGL3、Athb-9、O2和RAV1基因激活相关的cre对应的基序。这些cre参与了广泛的发育和代谢过程。AG已被证明与AGAMOUS TF结合,该基因在花干细胞的时间发育中起作用[53]而相关的TF AGL3可能参与地上器官的发育,并与AGL3 CRE区域结合[54]。Athb-9似乎调节叶片的模式,而O2或Opaque-2是一个TF,负责表达一个突出的储存蛋白玉米并促进胚乳代谢[55,56]。最后,RAV1是一种促进并可能调控叶片衰老的TF [57]。总的来说,我们发现了与光线、压力、防御和激素反应有关的cre。这些潜在的CRE在启动子中的存在杨树NP-like基因为这些基因的调控提供了可能的线索。的潜在调控NP-like基因在杨树通过激素信号传导途径的研究将会特别有趣。
预测启动子基序的主成分分析与相似树表达数据一致(图2)1A)和系统发育分析(图6)。这并不意外,因为基因表达受tf及其与启动子区域cre的相互作用调控。两者之间的距离BSP的,BSP B和BSP C,表示启动子区域中基序的差异,这可能是不平等重组和多样化选择的结果(图2)6, 1a) [51]。这也可以解释基因启动子区域中基序之间的距离WIN4-like基因(图61)。
表达差异与自然选择分析
我们对选择压力的评估表明,在最初的复制事件中存在多样化选择,而在最近的复制事件中存在分化选择BSP和WIN4-like亚科的成员。这种重复事件的连续发生与植物的系统发育研究是一致的。58- - - - - -60])。杨树特定的复制事件也已被评估[46,48,60]。在杨树有报道将串联复制的基因与植物防御和胁迫基因家族的扩展联系起来,这与基因剂量模型一致[49,50,61]。观察到在WGD或TD事件后保留的相反功能基因群之间的强模式p . trichocarpa[49,58]。这与BSPs和VSPs在受伤和干旱胁迫后积累的观察结果非常吻合[22,23]。
我们的结果表明BSP和WIN4-like亚家族基因表达模式的进化相对较晚,这些亚家族成员也面临着选择压力;这表明它们正在经历多功能基因的亚功能化或多样化。这得到了表达数据的支持BSP和WIN4-like基因亚家族(图21B).也许数据反映了放大、缓冲或“近”亚功能化,所有这些都可以促进功能冗余,使生物体能够对更大范围的细胞、环境和/或遗传扰动作出反应[62,63]。这种正剂量模型特别适用于参与应激和环境反应的基因[29,30.,37]。这种策略将在功能上促进最佳的营养循环和储存途径[24]。扩增模型描述了不稳定(即串联)复制中基因的潜在功能进化和固定[31]。核酸之间的高度相似BSP基因(BSP的vs。BSP B: 98.2%;BSPA+Bvs。BSP C~94%)也支持扩增模型,因为序列发散与表达发散是松散相关的[49]。
我们调整了对功能基因命运的预测,承认分类仅限于目前的基因复制模型,而这些模型由于对种群规模、选择压力和复制前后的固定作用的理解不足而受到限制[26,64,65]。研究趋向于发展一种更通用的适应性维持复制模型[64- - - - - -66]。如果没有这些信息,就特别难以区分亚功能化模型和多功能基因的多样化[66]。另一种复杂性涉及在进化过程中发生的多种功能化[67]。
NP-like基因家族扩张秩序内杨树trichocarpa
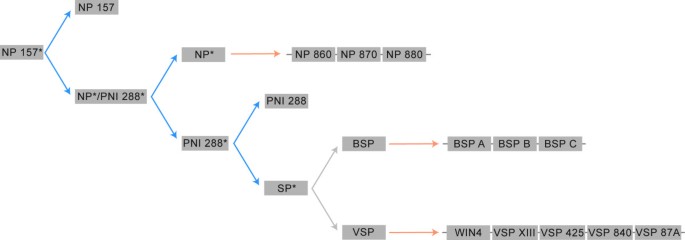
从这里提出的结果,我们提出一个假设的起源NP-like基因在杨树trichocarpa,如图所示8.np样亲本蛋白在古WGD事件后被保留,np样亲本蛋白和副本分别成为I组和II组np样蛋白的祖蛋白。如果这些基因在信号网络或通路(剂量平衡模型)中有功能参与,它们就可能被保留。II类np样蛋白的祖先可能经历了一个功能化过程,赋予适应性或在稳定的选择压力之后发生变化(即环境)后变得有利。发生了另一个WGD事件,特定于杨树随后是串联重复,在此功能的扩增将导致立即稳定的选择压力,正剂量模型。
我们的选择和系统发育分析表明NP 157可能是祖先基因。的表达模式NP 157明显不同于BSP和WIN4-like基因亚科。资源似乎不太可能直接用于高水平的转录NP 157衰老叶片中无功能基因或假基因的转录本,可能是蛋白质的翻译。两种解释是NP 157在衰老之前清除核苷,或者保留了与嘌呤回收和降解相关的其他基因相似的调控元件。在黑暗诱导的衰老条件下,其他参与嘌呤回收和降解的基因表达增加[14]。此外,敲低嘌呤降解的关键瓶颈酶黄嘌呤脱氢酶会导致生长减慢、早期衰老和不育[12,68]。然而,另一种解释可能是NP 157作为瞬时储存蛋白或以其他方式参与营养信号传导。至少,NP 157以及BSP和WIN4-like基因可能对SD光周期和/或衰老开始引起的营养信号变化作出反应。相比之下,这似乎是合理的NP在其他系统中的表达式。在细菌中枯草芽孢杆菌,PNP型转录由生长介质中的核苷诱导,葡萄糖抑制,指向调节PNP型按碳和能源供应[69,70]。在人类中,PNP型S在发生代谢变化的病变和癌变组织中表达上调[4- - - - - -6]。虽然需要更多的研究来确定NP 157的功能,但我们认为NP样蛋白可能仍然参与植物的营养信号传导。
植物界的系统发育分布
我们的数据表明,np样蛋白在所有高等植物的全基因组中都是保守的(图2)7)。有趣的是,np样蛋白在Physcomitrella还有绿藻基因组。这些分类群可能失去了类似np的基因。在细菌中,有多个门没有PNPs或相关的尿苷磷酸化酶,假设这可能是由于细菌中不同门对资源的需求不同[71]。非维管植物卷柏moellendorffii有两个np样的代表,聚集在第一类np样蛋白的底部,这表明这些蛋白是最古老的(图7)。细菌外群的包含支持了np样蛋白是单系的并且是从np进化而来的假设3.:图S1)。
整个植物界的np样蛋白的系统发育揭示了p . trichocarpa亚家族的指定与在13个np样蛋白树中发现的一致杨树(数据1和5)。BSP和WIN4-like亚家族以及PNI 288属于第二类,NP-like亚家族属于第一类。如果NP-like蛋白在所有植物中都起储存蛋白的作用,那么我们预计其他多年生木本植物的NP-like蛋白也会聚集在附近杨树储存蛋白质李属,云杉和马吕斯np样蛋白不会聚集在一起杨树存储蛋白质。相反,来自这些属的蛋白质聚集在I组,靠近来自np样亚家族成员的蛋白质杨树.这表明np样蛋白并不是整个植物界的储存蛋白。
除了基于系统发育分析的结论外,np样蛋白不是一般贮藏蛋白的发现与其他木本植物属的季节性贮藏蛋白的报道一致。例如,碧桃和云杉sitchensis都是木本多年生植物。在吠叫中碧桃,主要的储存蛋白是一个60 kDa的脱氢蛋白,一个19 kDa的过敏原相关蛋白和一个16 kDa的未知同源蛋白[72]。在青云杉-恩格尔曼尼复合体结果表明,32 kDa的BSPs并不是主要的贮藏蛋白云杉[73]。
我们的结果指向了NP-like基因家族是该目的共同祖先Malpighiales其中白杨,马尼乌特,蓖麻和亚麻属植物代表们[44,74]。而来自白杨、马尼乌特和蓖麻通常在两个群体中聚集在一起,亚麻属植物只有三个np样蛋白聚集在第一类中亚麻属植物失去II族np样蛋白。最近的大会亚麻属植物基因组显示亚麻属植物最近经历了WGD,随后失去了一条或多条染色体[75]。在木薯和蓖麻这个星团靠近杨树储存蛋白表明这些属具有储存功能。
结论
我们的研究的功能意义和np样蛋白和进化历史和基因杨树在整个植物界,这说明了微观进化(如自然选择)和宏观进化(如基因组复制和串联复制)在形成蛋白质家族多样性模式中的重要性。特别值得注意的是,我们发现了迄今为止尚未确定的证据NP-like基因可能在体内发挥功能作用杨树基于表达式数据;这些蛋白质的功能意义尚不清楚。此外,对启动子区域的分析显示,与光反应、植物激素反应、应激和组织特异性相关的基序存在,也支持np样蛋白的功能作用。我们还发现了重要的证据,表明情景多样化选择作用于内部的np样蛋白杨树在这个基因家族的进化过程中,基因表达水平的变化发生的时间相对较近。与其他发现一致,我们推断的系统发育暗示历史上的基因组复制事件和最近的分类群特异性独立复制事件是产生现存np样蛋白多样性的机制杨树。总之,我们的结果是第一个在植物中检测np样蛋白的研究,我们提出了它们在植物中可能具有功能意义的可能性。
方法
植物材料
杨树trichocarpa(托。基因型‘Nisqually-1’扦插在6 L盆栽中,在18°C的受控环境室中生长,PAR范围为310-470μ摩尔米2年代1.移栽一周后施用5 g控释肥(18-3-3)(Nutricote, Florikan, Sarasota, FL, USA)。在LD条件下(16 h光照/8 h暗)8周后,在SD条件下(8 h光照/16 h暗)3、6、8和12周后收集组织样本。在SD的最后4周(即SD的8至12周),温度降至白天10°C /晚上4°C。采集茎尖或顶芽、幼叶(叶质体指数(LPI) 3)、成熟叶(LPI 8)和树皮(LPI 8 - 9),在液氮中冷冻2然后保存在-80°C,直到RNA提取。一个重复由来自三株植物的组织池组成。采用2个重复进行基因表达分析。
植物中np样基因的鉴定及系统发育分析杨树基因组
在。中鉴定np样蛋白杨树基因组进行BLASTP搜索杨树trichocarpaGenome v2.2 (http://www.phytozome.net)使用默认参数(期望(E)阈值= -1,BLOSUM62比较矩阵)与毛卡柏(轨迹名:POPTR_0013s10380)作为查询序列。然后从Phytozome数据库中检索符合这些标准的np样蛋白序列,并用于系统发育分析。
QPCR引物设计与验证
采用MacVector v10 (MacVector Inc., Cary, NC, USA)设计np样基因家族引物和内参基因。所有引物均由Invitrogen公司(Carlsbad, CA, USA)合成。最佳退火温度由温度梯度确定,放大效率由10倍连续稀释的五点校准曲线计算。为了确认单个扩增产物,对所有qPCR反应进行熔体曲线分析。
RNA提取、cDNA合成、qPCR检测及统计分析
使用RNeasy植物迷你试剂盒和QIAcube (Qiagen, Valencia, CA, USA)自动纯化总RNA。组织在液氮中研磨,加入含有1%聚乙烯吡咯烷酮和1% β -巯基乙醇的RLT萃取缓冲液中。将样品涡旋后,加入0.4体积的5 M醋酸钾,pH为6.5,混合在冰上孵育15 min,然后在4℃下,以15,000 g离心15 min。将上清转移到QIAcube上,用柱上dna酶I酶切法提取RNA (Qiagen, Valencia, CA, USA)。使用Experion进行微流控分析TM采用自动电泳系统和RNA StdSens芯片测定RNA的质量和数量(Bio-Rad, Hercules, CA, USA)。使用RevertAid First strand cDNA Synthesis Kit (Thermo Scientific Inc., Rockford, IL, USA)和oligo dT引物,以1 μg的总RNA为原料,经三次反应合成第一链cDNA。收集cDNA作为模板进行qPCR检测。扩增反应采用iQ5实时检测系统(Bio-Rad, Hercules, CA, USA)和Maxima SYBR绿色qPCR主混合物(Thermo Scientific Inc., Rockford, IL, USA)进行。扩增反应包括在95°C下10 min,然后在95°C下15秒,在最佳退火温度下1 min的40个循环。通过geNorm确定每种组织类型的稳定内参基因+在qbase+第三版(http://www.qbaseplus.com)。(附加文件4表S1),使用qbase进行相关表达分析+(附加文件5表2)。相对表达数据被缩放到每个组织的最大相对表达单位。
我们还进行了主成分分析,分析了13个基因在治疗过程中每种组织类型中表达模式的差异。使用ADE4包进行分析[76]在R之内[77]。
在网上启动子序列分析NP-like基因家族杨树
启动子序列位于转录起始位点(ATG密码子)上游2kb处杨树NP-like基因从杨树trichocarpaPhytozome基因组v2.2 (http://www.phytozome.net/)。在网上对这些区域的分析使用预测程序MEME (http://meme.nbcr.net)及MotifClick (http://motifclick.uncc.edu/)以确定代表性过高的8 bp图案[41,42]。MEME使用期望最大化策略来检测过度表示的图案,而MotifClick采用基于图形的算法,通过识别完整子图(也称为cliques)来确定图案[41,42]。在以下设置下进行模因分析:单个模因的重复数任意分布在序列中,最小和最大模因宽度为8 bp,最大可找到的模因数为100。MotifClick分析设置为motif长度为8 bp,最大motif数为100,更多退化位点,结合位点与背景序列的平方和分别为0.06和0.1。在SSD 0.06和0.1下运行的输出相似度为93%,选择SSD 0.06的输出进行进一步比较(附加文件)6表S3)。将得到的8 bp基序与PlantPAN (Plant Promoter Analysis Navigator)确定的这些区域的已知CRE序列进行比较[43]。PlantPAN从PLACE、TRANSFAC、AGRIS和Jaspar中收集了已知的TF结合基序[43(参见附加文件7(表S4为PlantPAN的输出)。选择8 bp的基序长度有三个原因:TFs通常与长度在8 - 10到16-20 bp之间的DNA区域结合,该长度适合于预测程序并与PlantPAN的输出交叉参考[52]。较小的区域可能导致假阳性的数量增加[52]。
我们进行了主成分分析,以阐明基于两个程序预测的基序丰度的蛋白质之间的关系。使用ADE-4软件包进行的分析输入[76]在R之内[77],是每个基因的基序计数矩阵。为了识别PCA产生的非平凡成分,我们执行了一种分析方法[78的10000个随机化矩阵[77]。
系统发育np样区蛋白序列鉴定及基序预测
为了构建整个植物界的np样蛋白之间的进化关系,我们从Phytozome数据库(http://www.phytozome.net),使用带有默认参数的BLASTP搜索毛卡柏(基因座名称:POPTR_0013s10380)蛋白序列进行查询。对于Phytozome中缺失的物种,在NCBI使用“非冗余蛋白序列”数据库(http://www.ncbi.nlm.nih.gov/)。序列马吕斯从蔷薇科数据库(http://www.rosaceae.org/)和序列挪威云杉检索自ConGenIE (http://congenie.org/)。对于所有搜索,只搜索e值低于10的结果4被使用。手动检查序列是否有注释错误,并删除重复序列。共有142个序列用于分析,3个细菌序列作为外群(附加文件)2表5)。为了确认所有植物序列都可以被分类为NPs,我们使用默认设置和e值设置为0.01,对所有142个植物序列进行了保守域数据库(CDD)的批量cd搜索,所有序列都被识别为PNP_UDP_1超家族的成员(Pfam:01048) [79]。使用gapping Local Alignment of Motifs (GLAM2)程序4.8.1对所有具有默认参数和8000次迭代的序列进行保守区鉴定(http://meme.nbcr.net/meme/cgi-bin/glam2.cgi) [80]。预测的基序使用NCBI非冗余蛋白数据库与GLAM2SCAN交叉引用,该数据库在PNP_UDP_1超家族中具有最接近匹配的基序(Pfam:01048)。为了验证GLAM2预测的基序,从CDD (http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml),并通过GLAM2预测了这组序列的基序。然后,我们从每个序列集(植物和非植物)中构建预测基序的比对,并将MUSCLE设置为默认设置,以验证植物中的np样区域[81[附加文件8:图S2)。在植物中验证的NP-like motif被用来去除NP-like motif序列外的区域,得到的序列被用于系统发育树的构建。
的同工区域的坐标杨树trichocarpa基因组v2.2如图所示2可在http://ftp.jgi-psf.org/pub/compgen/phytozome/v5.0/Ptrichocarpa / miscelaneous /同线性/ allPoplarSegments.seg。
系统发育分析和蛋白质家族进化
使用贝叶斯(MrBayes v3.0;[82])和基于最大似然的方法(快速似然推断的遗传算法;GARLI;[83])。我们使用WAG + I + G氨基酸取代模型,由ProtTest [84],用于所有分析(包括在杨树具有全长氨基酸序列)。我们提出了基于观测数据集的1000个重复GARLI分析的最佳拓扑,并评估了1000个bootstrap重复拓扑关系的统计支持度。所有GARLI分析均使用与LATTICE项目相关的计算资源进行[85]。对于贝叶斯分析,我们使用默认设置(两个同时运行四个链的独立分析,其中三个是加热的),但将氨基酸模型设置为混合。我们使用0.01的阈值作为分割频率的标准偏差,作为充分收敛和混合的度量。较小的13蛋白的分析Poplulus数据集和更大的142蛋白数据集运行10次6和107分别代。在这两项分析中,25%的世代都是倦怠的。多序列比对和系统发育树可从TreeBASE (http://purl.org/phylo/treebase/phylows/study/TB2:S14494;[86])。包含细菌群的系统发育分析可以在附加文件中找到3.:图S1
自然选择和基因表达进化的测试
我们测试了13个基因的重组杨树使用遗传重组检测算法(GARD)方法对可能对自然选择分析产生不利影响的数据集进行分析[87在HyPhy包中实现[88]。GARD的结果没有发现任何有统计学意义的重组断点的证据。因此,我们采用Branch-site REL (random effects likelihood,随机效应似然)方法[87]在HyPhy包中实现,以确定13基因内是否有任何基因复制杨树数据集显示了多样化选择的证据。我们选择这种方法检测选择,因为我们的主要兴趣是确定哪些基因重复,而不是位点,在选择。这并没有假设哪些分支可能在选择中,这可能会增加假阳性的发生率[87]。所有分析均使用密码子序列。
我们进行了两种不同的测试来评估四种组织类型中基因表达模式的进化史。首先,我们估计了参数λ,其中0表示完全缺乏系统发育信号,1表示系统发育信号强(即由于共同祖先而具有相似性)。其次,我们通过估计参数δ来测试基因表达水平的进化是否随着时间的推移而增加、减少或保持不变;δ < 1表明基因表达模式差异的进化发生在基因分化的早期,δ > 1表明进化差异发生的时间相对较晚。为了评估λ和δ模型的统计拟合性,我们对特征进化布朗运动模型下的结果进行了似然比检验(LRT)。所有分析均使用GEIGER包进行[89]在R之内[77]。
参考文献
lelewkowicz E, Iribarren A:核苷磷酸化酶。有机化学,2006,(10):1197-1215。10.2174 / 138527206777697995。
王晓明,王晓明,王晓明,王晓明,拟南芥代谢代谢途径的研究进展。植物学报,2009,33(3):1177 - 1177。10.1111 / j.1469-8137.2009.02843.x。
Pugmire MJ, Ealick SE:结构分析揭示了核苷磷酸化酶的两个不同家族。中国生物医学工程学报,2009,32(1):1- 4。10.1042 / 0264 - 6021:3610001。
拉文迪F,甘地V:新型嘌呤核苷类似物治疗t细胞系急性淋巴细胞白血病和淋巴瘤。专家意见调查药物,2006,15:1601-1613。10.1517 / 13543784.15.12.1601。
Batista EL, Deves C, Ayub L, da Silva RG, Filho LCC, Basso LA, Santos DS:嘌呤核苷磷酸化酶在牙周病影响部位的活性和表达上调。[J] .地理学报,2010,35(5):664-671。10.1111 / j.1600-0765.2010.01282.x。
Kojima S, Chiyomaru T, Kawakami K, Yoshino H, Enokida H, Nohata N, Fuse M, Ichikawa T, Naya Y, Nakagawa M, Seki N:肿瘤抑制因子miR-1和miR-133a靶向前列腺癌嘌呤核苷磷酸化酶(PNP)的致癌功能。中国生物医学工程学报,2011,31(2):444 - 444。
Smith PMC, Atkins CA:嘌呤生物合成。在细胞分裂中更大,在氮同化中更大。植物生理学报,2002,28(2):793- 792。10.1104 / pp.010912。
王志强,王志强,王志强,等。氮动员的生物化学:嘌呤环分解代谢。植物学报,2011,16:381-387。10.1016 / j.tplants.2011.03.012。
植物嘌呤分解代谢:黄苹(lupinus luteus L.)种子肌苷核苷酶的纯化及一些特性。植物生理学报,2002,31(2):344-349。10.1104 / pp.70.2.344。
王晓明,王晓明,王晓明,等。马铃薯块茎中嘌呤的合成、回收和降解。植物学报,2006,25(4):379 - 379。10.1007 / s00425 - 006 - 0334 - 9。
Jung B, Hoffmann C, Möhlmann T:拟南芥核苷水解酶参与细胞内和细胞外嘌呤降解。植物科学学报,2011,35(5):779 - 779。10.1111 / j.1365 - 313 x.2010.04455.x。
王晓明,王晓明,王晓明,等。拟南芥黄嘌呤脱氢酶基因的序列定位研究。生物化学学报,2004,29(3):13547-13554。10.1074 / jbc.M312929200。
张春华,张春华。马铃薯块茎损伤诱导的腺苷酸生物合成的研究进展。植物生理学报,2006,44(4):551-555。10.1016 / j.plaphy.2006.09.016。
杨春华,刘春华,李春华,等:拟南芥Atxdh1基因突变体中嘌呤转运的研究进展。植物学报,2008,35(4):496- 496。10.1111 / j.1365 - 313 x.2008.03440.x。
[3]王志强,王志强,王志强,等。杨树氮素储存与季节氮素循环的分子生理学和生态生理学研究。植物学报,2005,32(2):369 - 369。10.1111 / j.1469-8137.2005.01451.x。
魏泽尔,邓志强,陈志强,等。季节性波动的树皮蛋白是3种温带阔叶树氮储存的潜在形式。植物学报,2009,32(2):559 - 561。10.1007 / BF00391854。
杨树营养贮藏蛋白的克隆及其在植物育种中的应用。植物生理学报,2001,32(2):557 - 557。10.1104 / pp.97.3.1017。
杨树林(Populus X Canadensis Robusta)木材蛋白质储存的超微结构、免疫化学研究。植物学报,1991,(3):92-100。
郭广民,陈涛,陈恩斯特,李富昌:杨树树皮贮藏蛋白积累的光周期调控。植物生理学报,2001,26(6):686-692。10.1104 / pp.96.3.686。
杨树树皮贮藏蛋白基因启动子(bspA)的活性与光敏色素介导的光周期感知、芽部生长、谷氨酰胺、钙和蛋白磷酸化有关。植物生理学报,2001,26(4):342-351。10.1104 / pp.126.1.342。
Coleman GD, Banados MP, Chen T .:氮素对杨树树皮贮藏蛋白及相关损伤诱导基因的诱导差异。植物生理学报,1994,26(4):591 - 591。
Lawrence SD, Cooke JEK, Greenwood JS, Korhnak TE, Davis JM:杨树顶芽形成过程中营养贮藏蛋白的表达。[J]中国生物医学工程学报,2001,31(1):1098-1103。10.1139 / x01 - 028。
M M ion C, Lalanne C, Claverol S, Meddour H, Kohler A, Bogeat-Triboulot M- b, Barre A, Le Provost G, Dumazet H, Jacob D, Bastien C, Dreyer E, de Daruvar A, Guehl j M, Schmitter j M, Martin F, Bonneu M:干旱胁迫下杨树蛋白质组的定位及其在干旱胁迫下响应蛋白的发现。生物工程学报,2006,6(6):659 - 659。10.1002 / pmic.200600362。
王晓明,王晓明,王晓明,等。植物氮素储存与再动员的研究进展。植物物理学报,2010,30:1083-1095。10.1093 / treephys / tpq042。
[3]李建平,李建平。植物对营养限制的适应。植物物理学报,2010,30:1047-1049。10.1093 / treephys / tpq076。
Lynch M, Conery JS:重复基因的进化命运和后果。科学通报,2000,29:1151-1155。10.1126 / science.290.5494.1151。
ssammon M, Wolfe KH:基因组复制的后果。信息技术与应用,2007,17(5):555 - 562。10.1016 / j.gde.2007.09.007。
林奇·M:非适应性过程的遗传网络进化。中国生物医学工程学报,2007,28(8):893 - 893。
Chapman BA, Bowers JE, Feltus FA, Paterson AH:同源重复基因对关键功能的缓冲可能有助于被子植物基因组复制的周期性。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.0507782103。
Sugino RP, Innan H:选择更多相同的产物作为加强复制基因协同进化的力量。科学通报,2006,22(2):642-644。10.1016 / j.tig.2006.09.014。
孙S,孙德松DI, Berg OG:新基因功能的进化:扩增模型的模拟与分析。遗传学报,2008,35(5):369 - 369。
大野S:基因和基因组复制的进化。纽约:斯普林格出版社:1970。
王志强,王志强。基因转化压力下的基因功能化。遗传学报,2008,32(1):559 - 559。10.1534 / genetics.107.082933。
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。遗传学报,2009,31(2):531- 545。
力A:通过亚功能化保存重复基因的可能性。遗传学报,2000,34(4):459-473。
王志强,王志强。基因复制模式的分类与区分。地理学报,2010,11(1):97-108。
Veitia RA:基因剂量平衡:缺失,重复和显性。趋势研究,2005,21:33-35。10.1016 / j.tig.2004.11.002。
Marais Des DL, Rausher MD:花青素途径基因复制后逃避适应性冲突。自然科学,2008,45(4):762-765。
Birchler JA, Veitia RA:基因平衡假说:从经典遗传学到现代基因组学。植物学报,2007,19:395-402。10.1105 / tpc.106.049338。
李建平,李建平。基因和基因组复制:剂量敏感性对核基因命运的影响。染色体学报,2009,17:699-717。10.1007 / s10577 - 009 - 9055 - 9。
李建军,李建军。基于期望最大化的混合模型在生物聚合物中发现基序。中国生物医学工程学报,1994,22(2):359 - 361。
张松,李松,牛敏,范培平,苏忠:MotifClick:基于合并团的顺式调控结合位点预测。生物信息学杂志,2011,12:238-10.1186/1471-2105-12-238。
张文超,李天宇,黄红东,黄海燕,潘瑞林:PlantPAN:植物启动子分析导航器——植物基因群中具有距离约束的组合顺式调控元件的识别。生物医学工程学报,2008,9(9):561- 5186 /1471-2164- 561。
王红,Moore MJ, Soltis PS, Bell CD, Brockington SF, Alexandre R, Davis CC, Latvis M, Manchester SR, Soltis DE: Rosid辐射与被子植物森林的快速崛起。自然科学进展,2009,26(6):1053 - 1058。10.1073 / pnas.0813376106。
Lee EK, Cibrian-Jaramillo A, Kolokotronis S-O, Katari MS, Stamatakis A, Ott M, Chiu JC, Little DP, Stevenson DW, McCombie WR, Martienssen RA, Coruzzi G, DeSalle R:种子植物功能系统发育研究。科学通报,2011,27 (7):1002411-10.1371/journal.pgen.1002411。
Tuskan GA, DiFazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q,等:黑棉、毛杨(Torr.)的基因组。&灰色)。科学通报,2006,31(3):1596-1604。10.1126 / science.1128691。
Sterck L, Rombauts S, Jansson S, Sterky F, rouzael P, Van de Peer Y: EST资料表明杨树是一种古老的多倍体。植物学报,2005,32(2):557 - 557。10.1111 / j.1469-8137.2005.01378.x。
杨志强,王志强,王志强,Öst T, Rönnberg-Wästljung A:杨树和杨树类群的高密度连锁定位与进化。中国生物医学工程学报,2010,31(1):391 - 391。
Rodgers-Melnick E, Mane SP, Dharmawardhana P, Slavov GT, Crasta OR, Strauss SH, Brunner AM, DiFazio SP:杨树全基因组与串联复制事件后的进化模式对比。中国生物医学工程学报,2012,22(2):95-105。10.1101 / gr.125146.111。
邹春华,李文华,李文华,王晓华:植物受体样激酶/小粒基因的进化历史和胁迫调控。植物生理学报,2009,32(1):1 - 6。10.1104 / pp.108.134353。
自由M:基因平衡的复制,就像四倍体一样,为增加形态复杂性提供了可预测的动力。中国生物医学工程学报,2006,32(6):559 - 564。10.1101 / gr.3681406。
zzambelli F, Pesole G, Pavesi G:新一代测序时代前后Motif发现和转录因子结合位点。生物信息学报,2012,14:225-237。
刘霞,金玉娟,Muller R, Yumul RE,刘超,潘莹,曹旭,Goodrich J,陈旭:AGAMOUS通过募集多梳蛋白直接抑制WUSCHEL,终止拟南芥花干细胞维持。植物学报,2011,23(3):354 -367。10.1105 / tpc.111.091538。
黄慧,Tudor M, Weiss CA,胡颖,马宏:拟南芥MADS-box基因AGL3广泛表达并编码序列特异性dna结合蛋白。植物学报,1995,28:549-567。10.1007 / BF00020401。
麦康奈尔,Emery J, Eshed Y, Bao N, Bowman J, Barton MK: PHABULOSA和PHAVOLUTA在芽径向模式中的作用。自然科学,2001,11:709-713。10.1038 / 35079635。
张宁,乔志,梁志,梅波,徐志,宋锐:玉米Mays taxilin蛋白通过改变其亚细胞分布负向调控不透明蛋白2的转录活性。PLoS ONE。[j] .科学通报,2012,(7):43822-10.1371/journal.pone.0043822。
禹海鹏,金建辉,金俊,金俊,李宇,宋爱军,金建辉,李海燕,南海鹏,林宝宝:RAV1转录因子正调控拟南芥叶片衰老。[J] .实验学报,2010,61:3947-3957。10.1093 / jxb / erq206。
李春华,李春华。植物全基因组复制与小尺度基因组复制的进化动力学与功能专门化。中国生物医学工程学报,2012,29(3):551 - 551。10.1093 / molbev / mss162。
Paterson AH, Bowers JE, Chapman BA:谷物分化前的古代多倍体化及其对比较基因组学的影响。美国国家科学促进会。[j] .中国科学:地球科学。10.1073 / pnas.0307901101。
Jiao Y, Leebens-Mack J, Ayyampalayam S, Bowers JE, McKain MR, McNeal J, Rolf M, Ruzicka DR, Wafula E, Wickett NJ, Wu X, Zhang Y, Wang J, Zhang Y, Carpenter EJ, Deyholos MK, Kutchan TM, Chanderbali AS, Soltis PS, Stevenson DW, McCombie R, Pires JC, Wong GK-S, Soltis DE, DE Pamphilis CW:与核心基因早期多样性相关的基因组三复制。中国生物医学工程学报,2012,32(3):559 - 559。
李春华,李春华,李春华,李春华,杨杨NBS抗性基因的全基因组分析。植物生理学报,2009,35(6):619-636。10.1007 / s11103 - 008 - 9293 - 9。
DeLuna A, Vetsigian K, Shoresh N, Hegreness M, Colón-González M, Chao S, Kishony R:重复基因对适合度的贡献。生物医学工程学报,2008,40(4):676-681。10.1038 / ng.123。
钱伟,廖保勇,张军,张军。基因冗余与重复基因的维持。遗传学报,2010,26(4):425-430。10.1016 / j.tig.2010.07.002。
Konrad A, Teufel AI, Grahnen JA, Liberles DA:基因复制进化动力学的一般模型。生物学报,2011,31(3):1197-1209。对10.1093 / gbe / evr093。
Moran和Wright-Fisher种群的多步进化速率。中国生物医学工程学报,2011,31(2):379 - 379。10.1016 / j.tpb.2011.07.003。
Proulx SR:亚功能化和基因复制专门化的多种途径。遗传工程学报,2012,29(1):737-751。10.1534 / genetics.111.135590。
何欣,张杰:重复基因进化中伴随长时间和大量新功能化的快速亚功能化。遗传学报,2005,32(1):557 - 564。10.1534 / genetics.104.037051。
Nakagawa A, Sakamoto S, Takahashi M, Morikawa H, Sakamoto A: rnai介导的黄嘌呤脱氢酶沉默影响转基因拟南芥的生长和生育,加速叶片衰老。植物生理学报,2007,32(4):444 - 444。10.1093 /卡式肺囊虫肺炎/ pcm119。
Schuch R, Garibian A, Saxild HH, Piggot PJ, Nygaard P:枯草芽孢杆菌drm-pupG操纵子的碳源研究。微生物学(英国)[j] .中国科学:地球科学。
王晓明,王晓明,王晓明,等。磷酸戊糖在核苷代谢中的应用。中国生物医学工程学报,2006,27(3):1089- 1091。10.1111 / j.1742-4658.2006.05155.x。
Konrad A, Piškur J, Liberles DA:细菌核苷磷酸化酶超家族催化残基的进化和酶机制。中国生物医学工程学报,2012,31(5):554 - 561。10.1016 / j.gene.2012.08.046。
王晓明,王晓明,王晓明,等。不同龄期桃茎和根中季节表达蛋白的分布及部分特征。植物物理学报,2004,24:339-345。10.1093 / treephys / 24.3.339。
林林总总,李建平,李建平,等。云杉苗木秋季适应模式及其与营养贮藏蛋白变化的关系。植物物理学报,1994,14(4):1107-1120。10.1093 / treephys / 14.10.1107。
张志强,张志强,张志强,等。白垩纪被子植物辐射类的系统基因组学分析。美国国家科学促进会。[j] .中国科学:自然科学,2012。10.1073 / pnas.1205818109。
Wang Z, Hobson N, Galindo L, Zhu S, Shi D, McDill J, Yang L, Hawkins S, Neutelings G, Datla R, Lambert G, Galbraith DW, Grassa CJ, Geraldes A, Cronk QC, Cullis C, Dash PK, Kumar PA, Cloutier S, Sharpe A, Wong GK-S, Wang J, Deyholos MK:亚麻(Linum usitatissimum)基因组的短散列序列测序。植物科学学报,2012,32(2):461-473。10.1111 / j.1365 - 313 x.2012.05093.x。
刘建军,刘建军,李建军,等。基于多变量分析的计算机辅助设计软件。统计,1997,7:75-83。10.1023 /: 1018513530268。
R:用于统计计算的语言和环境。维也纳,奥地利:R基金会:2008。
Peres-Neto PR, Jackson DA, Somers KM:给予坐标轴有意义的解释:评估主成分分析中的载荷显著性。生态学报,2003,34(9):2347-2363。10.1890 / 00 - 0634。
Marchler- bauer A, Anderson JB, dewese - scott C, Fedorova ND, Geer LY, He S, Hurwitz DI, Jackson JD, Jacobs AR, Lanczycki CJ, Liebert CA, Liu C, Madej T, Marchler GH, Mazumder R, Nikolskaya AN, Panchenko AR, Rao BS, Shoemaker BA, Simonyan V, Song JS, Thiessen PA, Vasudevan S, Wang Y, Yamashita RA, Yin JJ, Bryant SH: CDD:一个基于Entrez的保守域序列数据库。中国生物医学工程学报,2003,31(3):383-387。10.1093 / nar / gkg087。
Frith MC, Saunders NFW, Kobe B, Bailey TL:发现任意插入和缺失的序列基序。计算机工程学报,2008,25 (4):11008 - 11008 /journal. pcbii . 11008。
Edgar RC: MUSCLE:具有高精度和高通量的多序列比对。中国生物医学工程学报,2004,32(2):992 - 997。10.1093 / nar / gkh340。
Ronquist F, Huelsenbeck JP: MrBayes 3:混合模型下的贝叶斯系统发育推断。生物信息学,2003,19(1):1 - 4。10.1093 /生物信息学/ btg180。
最大似然准则下大型生物序列数据集系统发育分析的遗传算法方法。博士论文。德克萨斯大学奥斯汀分校:2006年。
李建军,李建军,李建军,等。蛋白质进化的最佳拟合模型选择。生物信息学。2005,21:2104-2105。10.1093 /生物信息学/ bti263。
Bazinet AL, Cummings MP: The Lattice Project:一个结合多种网格计算模型的网格研究和生产环境。分布式和网格计算——使人人透明的科学原理、应用和支持社区。编辑:Weber MHW。2008,马尔堡:Tectum出版社,2-13。
Sanderson MJ, Donoghue MJ, Piel WH, Eriksson T: TreeBASE:系统发育分析的原型数据库和浏览生命系统发育的交互式工具。[J] .植物学报,1994,18 (3):369 - 369
李建军,李建军,李建军,李建军,李建军。基于随机效应的生物多样性选择模型研究。中华生物医学工程学报,2011,28(2):393 - 393。10.1093 / molbev / msr125。
Pond SLK, Frost SDW, Muse SV: HyPhy:基于系统发育的假设检验。生物信息学。2005,21:676-679。10.1093 /生物信息学/ bti079。
Harmon LJ, Weir JT, Brock CD, Glor RE, Challenger W: GEIGER:研究进化辐射。生物信息学,2007,24:129-131。
致谢
我们非常感谢csamciile Parmentier-Line (University of Maryland, College Park MD)协助RNA提取并评估RNA的质量和数量。本项目资金来自美国国家科学基金会拨款IOS 0922650 (GC)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者声明,没有竞争的经济利益。
作者的贡献
EP设计了实验装置,完成了所有表达评估和启动子分析的工作,并撰写了稿件。EP和JP进行了分析。GC提供了定向支持。JP和GC参与了稿件编辑。所有作者都认可了这份手稿。
电子补充资料
12870 _2013_1326_moesm1_esm.xls
附加文件1:表S6:已知顺式与匹配的预测基序相对应的调控元件。包括顺式调控元件(CRE)名称、序列、CRE数据库和其他功能信息。(xl48kb)
12870 _2013_1326_moesm2_esm.xls
附加文件2:表S5:系统发育树标识符。图中的标识符7对应于Phytozome位点或NCBI序列标识符。(xl43kb)
12870 _2013_1326_moesm3_esm.pdf
附加文件3:图S1:植物界细菌外群中np样蛋白的系统发育分析。采用贝叶斯和最大似然方法构建系统发育关系。分支上的数字分别表示基于1000次重复的后验概率和自举百分比。括号中的数字对应于Phytozome或NCBI序列标识符,可以在附加文件中找到7表S4。五个主要的分类科由高亮的颜色表示。(pdf 302kb)
12870 _2013_1326_moesm4_esm.xlsx
附加文件4:表S1: QPCR参考基因信息。包括qPCR内参基因符号、基因名称、Phytozome位点名称、引物序列、扩增产物大小、退火温度、PCR效率和内参基因用于归一化计算的组织类型。(xlsx141kb)
12870 _2013_1326_moesm6_esm.xlsx
表S3:由MEME和MotifClick预测的启动子motif。由MEME(左)和MotifClick(右)预测的8个bp启动子基序。(xlsx85kb)
12870 _2013_1326_moesm7_esm.xlsx
附加文件7:表S4:来自PlantPAN的输出。包括独联体-调控元件序列;NP-like基因后的启动子区域,调控元件名称,在启动子区域内的位置,从数据库中检索元件和功能类别。(xlsx51 kb)
12870 _2013_1326_moesm8_esm.pdf
附加文件8:图S2:细菌和植物核苷磷酸化酶(NP)蛋白的模因预测序列比对。细菌氨基酸序列从NP家族(COG0775)的保守结构域数据库(CDD, http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)中检索,植物氨基酸序列从Phytozome (http://www.phytozome.net)。(pdf 2mb)
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
Pettengill, E.A, Pettengill, J.B. & Coleman, G.D.阐明植物核苷磷酸化酶类似物(营养储存蛋白)的进化历史和表达模式杨树还有植物王国。BMC Plant Biol13日,118(2013)。https://doi.org/10.1186/1471-2229-13-118
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-118
关键字
- 核苷磷酸化酶
- 营养贮藏蛋白
- 树皮贮藏蛋白
- 氮循环
- 杨树trichocarpa