摘要
背景
尽管芝麻是一种重要的油料作物,但对芝麻的遗传学和分子生物学的研究直到最近才开始。由于缺乏足够的分子标记,芝麻的高密度遗传图谱尚未发表。特定长度扩增片段测序(slf -seq)是最近发展起来的一种大规模高分辨率测序策略新创SNP发现和基因分型。本研究采用SLAF-seq获取足够的标记,构建芝麻高密度遗传图谱。
结果
测序后共获得28.21 Gb的数据,包含201,488,285对端reads。每个SLAF标记在父本中平均覆盖23.48倍,在母本中平均覆盖23.38倍,在每个F中平均覆盖14.46倍2个人。共检测到71793个高质量slaf,其中多态性标记3673个,多态性标记1272个符合构建遗传图谱的要求。最终图谱包括15个连锁群(LGs)上的1,233个标记,全长1,474.87 cM,相邻标记之间的平均距离为1.20 cM。据我们所知,这张图谱是迄今为止芝麻最密集的遗传连锁图谱。“SNP_only”标记占图谱标记的87.51%。地图上共有205个标记显示出显著的(P< 0.05)偏析变形。
结论
我们在这里报告了芝麻的第一个高密度遗传图谱。地图是用F来构造的2利用slf -seq方法,可以在短时间内高效地开发出大量多态性标记。本研究结果不仅可为芝麻基因/QTL精细定位、基因图谱分离、分子育种等提供平台,还可为在物理图谱上定位序列支架,辅助芝麻基因组序列组装提供参考。
背景
胡麻属indicum芝麻是一种二倍体(2n = 26),是最古老的油料作物之一[1]。全球每年生产约400万吨芝麻(http://faostat.fao.org/)。芝麻是优质油的重要来源,含有天然抗氧化剂,如芝麻素和芝麻醇。尽管芝麻具有重要的经济价值和营养价值,但其遗传和分子生物学研究直到最近才开始。芝麻的基因图谱只发表了一份[2]和QTL定位尚未见报道。
遗传图谱,特别是高密度遗传图谱是QTL定位的重要基础[3.- - - - - -5]和用于锚固顺序脚手架[6,7]。大规模的微卫星标记开发是获得足够数量的标记以构建高密度遗传图谱的重要途径。这种方法被用于烟草(烟草l .) [8]和日本比目鱼(Paralichthys olivaceus) [5],构建标记间平均间隔小于1.5 cM的高密度遗传图谱,所开发的SSR标记也将成为进一步推进基因组分析的重要工具。另一种构建高密度遗传图谱的方法是在一项研究中利用几种类型的可用标记。Troggio等。[9]发表了一份密集的葡萄遗传连锁图谱,该图谱使用483个基于单核苷酸多态性(SNP)的遗传标记、132个简单序列重复(SSRs)和379个扩增片段长度多态性(aflp)构建。基于snp的标记是从bac端序列资源中开发出来的,它们的加入显著增加了图谱的密度。Wang等。[10]构建了白菜高密度遗传连锁图谱,包含602个ssr位点和625个snp位点,总面积为1197.9 cM,相邻位点之间平均为0.98 cM。在这种情况下,SNP标记是通过对一个作图群体的另一个亲本进行重测序而开发的。
snp作为遗传标记比许多传统标记更有用,因为它们是大多数基因组中最丰富和最稳定的遗传变异形式[11]。由单核苷酸多态性引入的序列多态性很容易在数据库中编译,并且对进化研究很有用[9]。利用这些优势,高通量的SNP鉴定和基因分型方法迅速发展起来。Miller等。[12开发了一种使用限制性内切位点相关DNA (RAD)标记的快速且具有成本效益的多态性鉴定和基因分型方法。这种方法被用于许多生物的快速SNP发现和遗传连锁定位,包括棘鱼,粗糙脉孢菌,小菜蛾大麦和葡萄[13- - - - - -16]。Peterson等。[17报道了一种改进的低成本RAD测序(RADseq)方法,称为双消化RADseq,作为一种完整的实验室方案,不需要事先的基因组知识。波兰等。[18]还开发了一种新的双酶测序基因分型(GBS)方法,并用它构建了大麦和小麦的高密度遗传图谱。两种酶的使用不同于最初的GBS方案,因为两种酶文库中的扩增片段都由条形码的正向适配器和共同的反向适配器组成。这种文库构建方法大大简化了测序前文库的定量。最近,Sun等人[19]报道了特定长度扩增片段测序(SLAF)的发展-Seq)作为大规模的高分辨率策略新创SNP发现和基因分型。这种方法的有效性在水稻和大豆的数据上进行了测试,并用于创建鲤鱼的遗传图谱(鲤属carpio这是迄今为止没有参考基因组序列的任何生物体的密度最高的遗传图谱。
自1999年以来,几种标记被用于评估芝麻遗传多样性,包括随机扩增多态性DNA (RAPD) [20.,21],简单序列重复(ISSR) [22,23], aflp [24,25]、序列相关扩增多态性(SRAP) [26,27],微卫星多态性位点(RSAMPL)的随机选择扩增[2],但直到2005年才有了芝麻的SSR标记。Dixit等。[28]开发了第一批芝麻SSR标记,迄今已开发和发表了500多个SSR标记[2,28- - - - - -31]。尽管SSR标记在许多作物中对构建遗传图谱具有重要意义,但由于芝麻SSR标记的多态性率较低,可用的数量不足以满足构建遗传图谱的要求。Wei等。[2]利用120个EST-SSRs、256个aflp和8576个RSAMPLs构建了芝麻的首个遗传图谱,但这些标记中只有220个(8个EST-SSRs、25个aflp和187个RSAMPLs)被定位在30个连锁组中,覆盖面积为936.72 cM,平均标记间距为4.93 cM。Wei等。[2指出需要额外的标记来获得芝麻基因组的精确长度和高密度的图谱覆盖率。随后,Zhang等。[30.]将14个基因ssr整合到该图谱上的9个主要连锁群中。但是由于缺乏足够的分子标记,芝麻的高密度遗传图谱尚未发表。
在这项研究中,我们采用最近开发的slf -seq方法实现了芝麻SNP和插入删除(InDel)标记的首次快速批量发现。利用这些新标记构建了一个高密度遗传图谱,并对其特征进行了分析,讨论了本研究开发标记的方法和该遗传图谱的应用前景。
结果
SLAF-seq数据及SLAF标记物分析
经过SLAF文库构建和高通量测序,共获得28.21 Gb的数据,包含201,488,285对端reads,每个reads长度约为70 bp。Q20(质量分数为20分,有1%的误差,有99%的置信度)比为74.32%,鸟嘌呤-胞嘧啶(GC)含量为40.60%。在这些高质量的数据中,~158 Mb来自父本(2,259,333个reads)和~157 Mb来自母本(2,246,084个reads)。读F中107个人的数字2人口从824263人到3122925人不等,平均人口为1416287人。
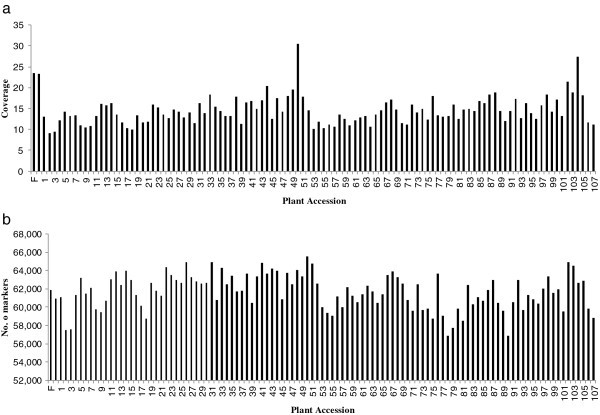
男性和女性父母的slaf数量分别为61909个和60949个。在男性和女性父母中,slaf的读数分别为1,453,579和1,425,202。各标记在父本的平均覆盖率为23.48倍,在母本的平均覆盖率为23.38倍。在F区2个体SLAF标记数在56,852 ~ 65,545个之间,平均为61,705个。slf的阅读数从527,734到2,000,299不等,平均为898,509,覆盖范围从9.18到30.52不等,平均为14.46(图2)1)。
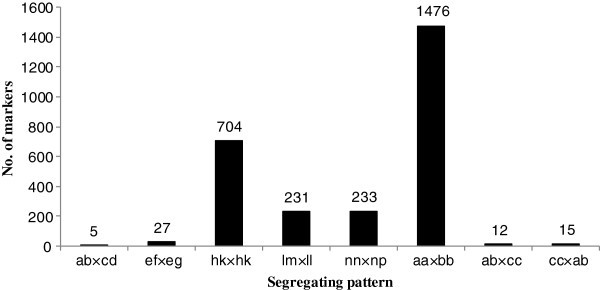
在检测到的71793个高质量slaf中,多态性3673个,多态性率仅为5.12%(表1)1)。在3673个多态slaf中,2703个被划分为8种分离模式(图2)2)。F2总体是通过自旋F1两个基因型为aa或bb的完全纯合亲本杂交的结果。因此,只有aa × bb偏析模式在F2利用群体构建遗传图谱,有1476个标记属于这一类。其中1272个标记亲本序列深度超过20倍,单个序列深度超过14倍,SLAF标签完整性超过80%,用于构建遗传图谱。
遗传图谱的基本特征
经过联动分析,1,233(附加文件)1)(附加文件2)标记被映射到遗传图谱上,而其他39个标记未能与任何群体联系起来。这些标记在父本中覆盖率为23.83倍,在母本中覆盖率为23.32倍,在每个F中覆盖率为14.96倍2个人平均。107个F中每个标记的完整性2个体也是控制地图质量的关键参数。地图上所有标记的平均完整性为98%。
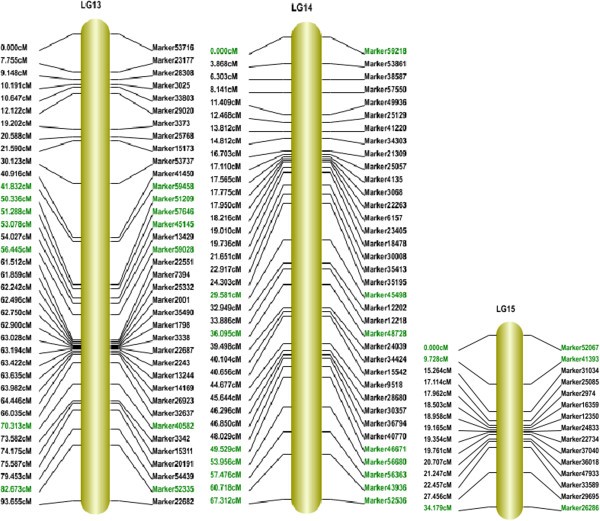
最终的图谱包括15个连锁组的1233个标记(图2)3.,4和5),长1474.87 cM,相邻标记间平均距离1.20 cM。据我们所知,这张图谱是迄今为止芝麻最密集的遗传连锁图谱。如表所示2最大的连锁群(LG)是LG7,共有247个标记,长度为189.68 cM,相邻标记间的平均距离仅为0.77 cM。最小的LG是LG15,仅有15个标记,标记长34.18 cM,标记间平均距离为2.44 cM。标记间的连锁程度体现为‘Gap < = 5’,范围为78.57% ~ 98.85%,平均值为91.76%。这张地图上最大的缺口是29.10 cM,位于LG9。
遗传图谱上标记类型的分布
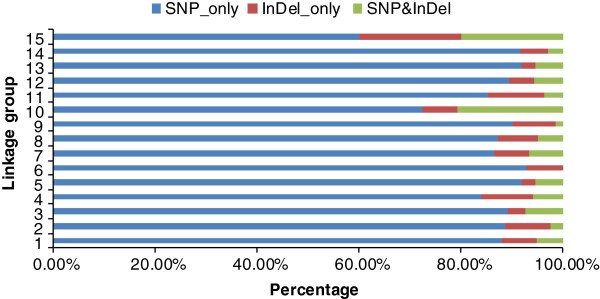
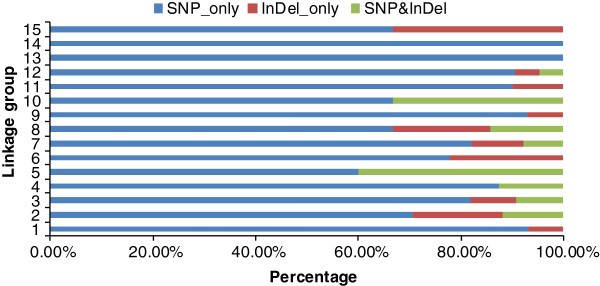
遗传图谱有3种标记类型,包括1079个“SNP_only”标记、86个“InDel_only”标记和68个“SNP&InDel”标记。“SNP_only”是主要的标记类型,占87.51%。研究了15个LGs中每个LGs的标记类型(表2)2,图6)。最小的LG15具有最低的“SNP_only”标记百分比,但具有最高的“InDel_only”标记百分比,分别为60%和20%。LG10中SNP&InDel标记的比例最高,为20.69%。3种标记在最大的LG7上的占比分别为86.64%、6.88%和6.48%,与15个LG7标记类型的平均分布非常接近。
在1079个“SNP_only”型标记中,302个标记有两个或两个以上的SNP位点,其余777个标记有一个SNP位点,占总数的72%。在最终图谱上的1233个标记中,共检测到1669个SNP位点,并调查了不同SNP类型的百分比(表2)3.)。大多数SNP为过渡型SNP,其中Y(T/C)型和R(G/A)型分别占所有SNP标记的31.76%和30.68%。其他4种SNP类型分别为S(G/C)、M(A/C)、K(G/T)和W(A/T),所占比例为8.69% ~ 10.96%,占全部SNP的37.56%。
地图上的隔离扭曲标记
总共有205个标记显示出显著(P< 0.05)的分离畸变被映射到最终的图上(图2)3.,4和5),多分布在有联动的LGs末端。a χ的结果2实验表明,每一个LG上都分布着分离畸变标记,其分布与所有标记的分布非常相似(表1)4), LG6及LG11除外。这意味着一个LG同时拥有更多的标记和分离失真标记。例如,最大的LG (LG7)具有最高的偏析扭曲标记百分比(19.02%),最小的LG (LG15)具有最低的偏析扭曲标记百分比(1.46%)。而LG6和LG11的分离畸变标记出现频率分别为32.14%和37.04%,明显高于其他LG6和LG11。在11个LGs上检测到18个偏析畸变区,其中LG1和LG7的偏析畸变区最多。
205个分离畸变标记为3种类型,其中‘SNP_only’型标记171个,‘Indel_only’型标记20个,‘SNP&Indel’型标记14个,所占比例分别为83.41%、9.76%和6.83%。这种分布与三种类型的所有标记所观察到的百分比相似,但三种类型的隔离扭曲标记在每个LG的分布不同(图2)7)。LG13和LG14上的偏聚扭曲标记均为“SNP_only”型,其余8个LG13上仅观察到两种偏聚扭曲标记。'SNP_only '和'Indel_only '偏析扭曲标记位于LG1、LG6、LG9、LG11和LG15上,'SNP_only '和'SNP&Indel '偏析扭曲标记位于LG4、LG5和LG10上。
讨论
需要通过测序来开发芝麻的标记
根据已发表的芝麻研究结果,种质资源之间的遗传多样性较低[20.- - - - - -22,24,26,27]品种间的遗传差异有限[23,25,30.,32]。遗传亲缘关系密切,可以解释分子标记多态性低的原因。据报道,芝麻中多种标记的多态性率为20%,其中EST-SSR [28EST-SSR为7.50%,AFLP为9.00%,RSAMPL为2.30% [2], 11.59%为遗传ssr [30.], 3.81%为cDNA-SSR [31], SLAF标记(本研究中的SNP和InDel)为5.12%。由于可用标记数量有限,多态性率低,用传统方法几乎不可能构建高密度标记的芝麻遗传连锁图谱。测序基因分型是一种高通量技术,为在短时间内高效开发大量标记,生成足够多的多态性标记,构建高密度遗传图谱打开了大门。
SLAF测序用于开发标记物的优势
与低效、昂贵和耗时的传统标记开发方法相比,生物信息学分析在SLAF测序之前进行;考虑基因组GC含量、重复条件和遗传特征等信息,对芝麻参考基因组(未发表数据)进行分析。我们设计了一种方法来确保标记开发的密度、均匀性和效率。slf -seq方法具有显著的优势,如开发大量的标记,具有较少的测序量,具有较高的准确性,特别适合分析芝麻等多态性较低的物种。本研究首次对芝麻进行了标记的大规模开发,基于高通量测序共开发了71793个SLAF标记,并鉴定了1272个多态性标记,用于构建遗传连锁图谱。标记完整性和准确性高,标记质量和数量满足构建遗传图谱的要求。结果也准确地反映了芝麻的遗传和多态性特征,增加了我们对该物种遗传学的认识。
分离扭曲标记的使用增加了图谱的基因组覆盖率
由于芝麻可用的分子标记数量有限,多态性率低,因此需要利用分离畸变标记等附加标记来构建高密度的遗传图谱。偏析畸变是一种常见的现象,在许多研究中都观察到。分离扭曲标记的基因型频率偏离了典型的孟德尔比率。这种偏差可能是由配子选择引起的[33],合子选择[34或两者兼而有之。本研究首先利用孟德尔标记构建遗传图谱。然后在现有图谱中插入分离失真标记,并重新估计标记之间的重组分数,类似于Wang等人使用的方法。[qh]35]。结果,205个标记(16.63%)被映射到最终的图谱中,每个LG上的分布与所有标记的分布相似。除了增加地图上标记的数量外,地图的基因组覆盖率也增加了。分离扭曲标记的存在不会影响该图谱在QTL图谱等应用中的使用。徐(36表明扭曲标记可以用于QTL定位,对结果没有不利影响,如果使用得当,可能是有益的。Zhang等。[37研究表明,分离扭曲可能导致比非扭曲更高的遗传方差,并有助于提高连锁QTL的检测,因为扭曲标记对QTL分析的位置或效应估计没有太大的影响。Xu和Hu开发了一种使用分离扭曲标记的QTL定位新方法[38]。
基因图谱的未来应用
据我们所知,本文提出的遗传图谱是迄今为止最密集的芝麻遗传图谱,尽管它仍然不饱和,因为它有15个LGs,而不是预期的13个LGs。该地图的宽度为1474.87 cM,平均每LG有82.20个标记,相邻标记之间的平均距离为1.20 cM。这幅图的平均距离远小于Wei等人之前报道的4.25 cM。[2]。更重要的是,该遗传图谱上93.03%的标记是snp,这是个体之间最丰富的遗传变异类型。snp是序列标记标记,具有共显性遗传,因此它们适合比较基因组研究[39]和关联映射[40,41]。本研究结果不仅为芝麻提供了质量标记,而且为芝麻的基因/QTL精细定位、基于图谱的基因分离和分子育种提供了数据。由于这些高密度连锁群是基于全基因组水平的分子标记构建的,因此它们将作为在物理图谱上定位序列支架的参考,以辅助芝麻基因组序列的组装过程。
结论
我们报告了芝麻的第一个高密度遗传图谱。地图是用F来构造的2基于slf -seq方法开发的多态标记,可以在短时间内高效地开发大量标记。本研究结果不仅可以为芝麻基因/QTL精细定位、基因图谱分离和分子育种提供平台,还可以为在物理图谱上定位序列支架和辅助芝麻基因组序列组装过程提供参考。
方法
植物材料和DNA提取
F2作图群体由中国农业科学院武汉油料作物研究所“中植13号”(母本)与“山东嘉祥芝麻”(父本)杂交的107个个体组成。年轻健康的叶片来自双亲和F2收集个体,冷冻在液氮中,用于提取DNA。根据十六烷基三甲基溴化铵(CTAB)法从每株植物中提取总基因组DNA [42],并对CTAB缓冲液的成分进行了一些修改(8.18 g氯化钠和2g CTAB,总体积为100 ml 20 mM EDTA, 100 mM Tris, pH 8.0)。DNA浓度和质量用ND-1000分光光度计(NanoDrop, Wilmington, DE, USA)和用lambda DNA标准的0.8%琼脂糖凝胶电泳进行评估。
SLAF文库构建及高通量测序
手术按照Sun等人的描述进行。[19稍加修改。简要地说,通过SLAF中试实验,建立优化SLAF产率的条件,避免重复SLAF,并获得SLAF均匀分布以获得最大的SLAF-seq效率。根据中试结果,构建SLAF库如下:基因组DNA首先在37°C孵育均方误差I[新英格兰生物实验室(NEB),伊普斯维奇,马萨诸塞州,美国],T4 DNA连接酶(NEB), ATP (NEB),和均方误差我的适配器。限制/连接反应在65℃下热灭活,用EcoR我和论坛I限制性内切酶在37°C。然后,在含有稀释后的限制性内切/结扎样品、dNTP、Taq DNA聚合酶(NEB)和Taq DNA聚合酶的反应溶液中进行聚合酶链反应(PCR)均方误差1.引物包含条形码PCR产物使用E.Z.N.A.®Cycle Pure Kit (Omega Bio-Tek, Norcross, GA, USA)纯化并汇总。混合后的样品在37°C孵育均方误差I, T4 DNA连接酶,ATP和Solexa适配器,使用Quick Spin柱(Qiagen, Hilden, Germany)纯化,并在2%琼脂糖凝胶上运行。使用凝胶提取试剂盒(Qiagen)分离400-450 bp的片段(含索引和接头),用Phusion Master Mix (NEB)和Solexa amplification引物混合物(Illumina, Inc., San Diego, CA, USA)进行PCR扩增,根据Illumina样品制备指南添加条形码2。样品经凝胶纯化后,切除400-450 bp的DNA片段(SLAFs),稀释后在Illumina High-seq 2000测序平台(Illumina, Inc;圣地亚哥,加州,美国)在北京生物标志物技术公司(http://www.biomarker.com.cn/english/)。测序过程中每个周期进行实时监测,计算原始reads中质量分数大于Q20(质量分数为20,表示有1%的错误机会,因此有99%的置信度)的高质量reads的比例,并计算鸟嘌呤-胞嘧啶(GC)含量进行质量控制。
slf -seq数据分组和基因型定义
所有具有明确索引信息的SLAF对端reads均基于BLAT检测到的序列相似性聚类[43[-tileSize = 10 -stepSize = 5]如Sun等人所述,同源性超过90%的序列被归为一个SLAF位点。[19]。使用次要等位基因频率(MAF)评估在每个SLAF中定义等位基因。由于芝麻是二倍体物种,一个位点最多包含4个SLAF标签,因此含有4个以上标签的组被过滤为重复的SLAF。在本研究中,序列深度小于107的SLAFs被定义为低深度SLAFs,并被过滤掉。具有2、3或4个标记的SLAFs被认为是多态SLAFs,并被认为是潜在的标记。多态性标记可分为ab × cd、ef × eg、hk × hk、lm × ll、nn × np、aa × bb、ab × cc和cc × ab 8种分离模式。F2群体是由两个基因型为aa或bb的完全纯合亲本杂交的F1自交获得的。因此,本研究仅使用分离模式为aa × bb的SLAF标记构建遗传图谱。SLAF标记的平均序列深度在亲本大于20倍,在子代大于14倍。任何一个子代含有超过80%的父母SLAF标记,即80%的个体SLAF标记完整性。
偏析畸变分析及基因图谱构建
标记分离比采用卡方检验计算。显示显著(P< 0.05)的隔离畸变最初被排除在地图构建之外,后来作为辅助标记加入。地图上有三个以上相邻位置的区域,显示出显著的(P< 0.05)偏析畸变被定义为偏析畸变区(SDR) [44]。使用JoinMap 4.0软件计算标记物之间的重组率[45],采用对数概率阈值≥4.0,最大重组分数为0.4构建遗传图谱。使用Kosambi制图函数计算厘-摩根的地图距离[46]。
参考文献
- 1.
贝迪安D,哈伦J:古代世界芝麻种植的证据。经济学报,1986,40(4):369 - 369。10.1007 / BF02859136。
- 2.
魏宝玲,张海燕,郑义忠,苗海明,张振华,郭文忠:芝麻遗传连锁图谱的构建(英文)胡麻属indicuml .)。基因工程学报,2009,31(2):199-208。10.1007 / BF03191152。
- 3.
Petroli CD, sanaloni CP, Carling J, Steane DA, Vaillancourt RE, Myburg AA, DA Silva OB, Pappas GJ, Kilian A, Grattapaglia D:桉树DArT标记的高密度连锁分析和物理定位。科学通报,2012,7 (9):e44684-10.1371/journal.pone.0044684。
- 4.
张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,张建军,等。中国生物医学工程学报,2012,31(3):888 - 888。
- 5.
宋伟,庞锐,牛燕,高峰,赵燕,张健,孙健,邵超,廖晓,王磊,等:牙鲆高密度遗传连锁图谱构建及生长相关定量追踪位点定位[J]。Paralichthys olivaceus)。科学通报,2012,7 (11):e50404-10.1371/journal.pone.0050404。
- 6.
王勇,孙生,刘斌,王辉,邓杰,廖勇,王强,程峰,王旭,吴健:基于序列的遗传连锁图谱研究芸苔属植物拉伯pseudochromosome组装。中国生物医学工程学报,2011,32(2):559 - 564。
- 7.
任勇,赵辉,寇强,姜军,郭松,张宏,侯伟,邹旭,孙华,龚刚,等:西瓜基因组高分辨率基因图谱锚定支架。科学通报,2012,7 (1):e29453-10.1371/journal.pone.0029453。
- 8.
Bindler G, Plieske J, Bakaher N, Gunduz I, Ivanov N, Van der Hoeven R, Ganal M, Donini P:烟草高密度遗传图谱(烟草L.)通过大规模微卫星标记开发获得。理论与应用,2011,23(2):219-230。10.1007 / s00122 - 011 - 1578 - 8。
- 9.
Troggio M, Malacarne G, Coppola G, Segala C, Cartwright DA, Pindo M, Stefanini M, Mank R, Moroldo M, Morgante M,等:基于密集单核苷酸多态性的葡萄遗传连锁图谱[j]。葡萄l)锚定黑皮诺细菌人工染色体群。遗传工程学报,2009,35(4):557 - 557。10.1534 / genetics.106.067462。
- 10.
王伟,黄松,刘勇,方志,杨丽,华伟,袁生,刘生,孙军,庄明,等:白菜高密度遗传连锁图谱的构建与分析(英文)芸苔属植物oleraceaL.变种)。中国生物医学工程学报,2012,31(3):523-10.1186/1471-2164-13-523。
- 11.
刘健,黄松,孙敏,刘生,刘勇,王伟,张鑫,王辉,华伟:一种改进的SNP标记分析等位基因特异性PCR引物设计方法及其应用。植物学报,2012,8(1):34-10.1186/1746-4811-8-34。
- 12.
Miller MR, Dunham JP, Amores A, Cresko WA, Johnson EA:限制性内切位点相关DNA (RAD)标记的快速高效多态性鉴定和基因分型。中国生物医学工程学报,2007,17(2):240-248。10.1101 / gr.5681207。
- 13.
Baird NA, Etter PD, Atwood TS, Currey MC, Shiver AL, Lewis ZA, Selker EU, Cresko WA, Johnson EA:利用测序RAD标记快速发现SNP和基因定位。科学通报,2008,3 (10):e3376-10.1371/journal.pone.0003376。
- 14.
Baxter SW, Davey JW, Johnston JS, Shelton AM, Heckel DG, Jiggins CD, Blaxter ML:基于下一代非模式生物RAD测序的连锁定位和比较基因组学。科学通报,2011,6 (4):e19315-10.1371/journal.pone.0019315。
- 15.
Chutimanitsakun Y, Nipper RW, custa - marcos A, Cistue L, Corey A, Filichkina T, Johnson EA, Hayes PM:大麦限制性内切位点相关DNA (RAD)连锁图谱的构建与QTL分析。中国生物医学工程学报,2011,32(4):564 - 564。
- 16.
王宁,方丽,辛慧,王丽,李生:利用下一代限制性位点相关DNA测序构建葡萄高密度遗传图谱。植物生态学报,2012,32(2):559 - 564。
- 17.
张建军,张建军,张建军,张建军,张建军。基于双消化RADseq的生物多样性基因分型研究。科学通报,2012,7 (5):e37135-10.1371/journal.pone.0037135。
- 18.
李建军,李建军,李建军,李建军,等。基于双酶基因分型的小麦和大麦基因图谱的构建。科学通报,2012,7 (2):e32253-10.1371/journal.pone.0032253。
- 19.
孙鑫,刘东,张鑫,李伟,刘宏,洪伟,蒋超,关宁,马超,曾宏,等:SLAF-seq:一种高效的大规模从头SNP发现和高通量测序基因分型方法。科学通报,2013,8 (3):e58700-10.1371/journal.pone.0058700。
- 20.
Ercan AG, Taskin M, Turgut K:土耳其芝麻遗传多样性分析(胡麻属indicumL.)群体使用RAPD标记。遗传资源作物学报,2004,51(6):599-607。
- 21.
Pham TD, Bui TM, Werlemark G, Bui TC, Merker A, Carlsson AS:芝麻遗传多样性研究[j]。胡麻属indicumL.)在越南和柬埔寨的RAPD标记估计。遗传资源作物学报,2009,56(5):679-690。10.1007 / s10722 - 008 - 9393 - z。
- 22.
Kim DH, Zur G, Danin-Poleg Y, Lee SW, Shim KB, Kang CW, Kashi Y:基于简单序列重复序列的芝麻种质资源遗传关系分析。植物育种,2002,21(3):259-262。10.1046 / j.1439-0523.2002.00700.x。
- 23.
Kumar V, Sharma SN:芝麻表型、ISSR和SSR标记的比较潜力(胡麻属indicumL.)来自印度的品种。作物科学与技术,2011,14:163-171。10.1007 / s12892 - 010 - 0102 - z。
- 24.
Laurentin HE, Karlovsky P:利用扩增片段长度多态性(AFLP)分析芝麻(Sesamum indicum L.)种质资源亲缘关系和多样性。生物医学工程学报,2006,7:10-
- 25.
Laurentin H, Karlovsky P:芝麻的AFLP指纹图谱(胡麻属indicumL.)品种:鉴定、亲缘关系及AFLP信息参数比较。遗传资源作物学报,2007,54(7):1437-1446。10.1007 / s10722 - 006 - 9128 - y。
- 26.
张云云,张小荣,华伟,王丽莲,车志:地方芝麻品种遗传多样性分析(胡麻属indicumL.基于SRAP和SSR标记的中国核心种质。基因工程学报,2010,32(3):207-215。10.1007 / s13258 - 009 - 0888 - 6。
- 27.
张勇,张鑫,车志,王磊,魏伟,李东:中国芝麻核心种质的表型和分子标记遗传多样性评价及微核心种质的提取。生物医学工程学报,2012,13:102-
- 28.
Dixit A, Jin MH, Chung JW, Yu JW, Chung HK, Ma KH, Park yjj, Cho EG:芝麻多态性微卫星标记的开发(胡麻属indicuml .)。生物化学学报,2005,35(4):736-738。10.1111 / j.1471-8286.2005.01048.x。
- 29.
魏伟,齐欣,王磊,张勇,华伟,李东,吕红,张欣:芝麻的表征(胡麻属indicumL.)全球转录组利用Illumina配对端测序和EST-SSR标记的开发。中国生物医学工程学报,2011,32(2):551 - 556。
- 30.
张慧,魏玲,苗辉,张涛,王超:基于RNA-seq的芝麻遗传ssr标记的开发与验证。中国生物医学工程学报,2012,13(3):316- 316。
- 31.
王丽,张燕,齐霞,高燕,张霞:油料作物59个多态cDNA-SSR标记的开发与鉴定胡麻属indicum(胡麻科)。植物学报,2012,99(10):394-398。10.3732 / ajb.1200081。
- 32.
张云云,孙佳,张晓荣,王丽丽,车志:中国主要芝麻品种遗传多样性及遗传基础分析。中国农业科学,2011,10(4):509-518。10.1016 / s1671 - 2927 (11) 60031 - x。
- 33.
Faris JD, Laddomada B, Gill BS:黄家鱼分离畸变位点的分子定位。遗传学报,1998,39(1):319-327。
- 34.
王晓明,王晓明,王晓明,等。苏地松近交系退化的地理变异。进化,1996,50(1):111-119。10.2307 / 2410785。
- 35.
王超,朱超,翟辉,万杰:水稻小穗不育性分离扭曲位点和数量性状位点的定位(英文)栽培稻l .)。中国生物医学工程学报,2005,26(2):394 - 394。10.1017 / S0016672305007779。
- 36.
许胜:数量性状位点定位有利于分离失真。遗传工程学报,2008,35(4):591 - 598。10.1534 / genetics.108.090688。
- 37.
张丽,王松,李辉,邓强,郑安,李松,李鹏,李忠,王杰:标记缺失和分离畸变对F2群体QTL定位的影响。理论应用,2010,21(6):1071-1082。10.1007 / s00122 - 010 - 1372 - z。
- 38.
徐松,胡忠:利用扭曲标记定位数量性状位点。植物基因组学学报,2009,29 (1):481 - 481
- 39.
Luo MC, Deal KR, Akhunov ED, Akhunova AR, Anderson OD, Anderson JA, Blake N, Clegg MT, Coleman-Derr D, Conley EJ,等:禾本科植物染色体数目减少和基因组加速进化的优势机制。美国国家科学促进会。中国生物医学工程学报,2009,26(3):559 - 561。10.1073 / pnas.0908195106。
- 40.
Cogan NO, Ponting RC, Vecchies AC, Drayton MC, George J, Dracatos PM, Dobrowolski MP, Sawbridge TI, Smith KF, Spangenberg GC,等:多年生黑麦草基因相关单核苷酸多态性的发现[J]。多年生黑麦草l .)。中国生物医学工程学报,2006,31(2):391 - 391。10.1007 / s00438 - 006 - 0126 - 8。
- 41.
Chan EK, Rowe HC, Kliebenstein DJ:了解防御代谢物的进化拟南芥使用全基因组关联图谱。遗传学报,2010,35(3):991- 997。10.1534 / genetics.109.108522。
- 42.
Doyle JJ, Doyle JL:一种用于少量新鲜叶片组织的快速DNA分离方法。植物化学,1987,19:11-15。
- 43.
Kent WJ: blat -类似blast的对齐工具。中国生物医学工程学报,2002,31(4):656-664。
- 44.
Paillard S, Schnurbusch T, Winzeler M, Messmer M, Sourdille P, Abderhalden O, Keller B, Schachermayr G:冬小麦综合遗传连锁图谱(英文)小麦l .)。理论应用学报,2003,107(7):1235-1242。10.1007 / s00122 - 003 - 1361 - 6。
- 45.
论文P:利用一种新的计算机程序包——连接图谱构建综合遗传连锁图谱。植物学报,1993,3:739-744。10.1111 / j.1365 - 313 x.1993.00739.x。
- 46.
Kosambi DD:从重组值估计地图距离。王志强,刘志强。2008,26(1):1 - 3。
致谢
项目资助:国家自然科学基金(no. 5);国家基础研究计划(973计划)资助项目(no. 31271766);2011CB109304);中央公益性科研机构基础研究基金;CARS-15)。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
XZ, YZ和LW设计并组织了整个项目。LW, DL和XD进行了实验。YZ, HX, CM和WH分析数据。YZ, HX和XZ起草了手稿。所有作者都阅读并批准了最终的手稿。
权利和权限
本文由BioMed Central Ltd.授权发表。这是一篇基于知识共享署名许可(http://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
张勇,王磊,辛浩。et al。基于特定长度扩增片段(SLAF)测序大规模标记开发的高密度芝麻遗传图谱构建。BMC Plant Biol13,141(2013)。https://doi.org/10.1186/1471-2229-13-141
收到了:
接受:
发表:
关键字
- 胡麻属indicuml
- 高密度
- 遗传图谱
- 连锁分析
- 测序