抽象的
背景
豌豆(Pisum sativum.L.)是一种自我授粉,二倍体,凉爽的季节食物豆类。作物产量受到多种生物和非生物应激因子的限制,包括盐度,导致增长和产量降低。基因组学的最新进展允许开发低成本的高通量基因分型系统,允许饱和遗传联系地图的构建,用于鉴定与感兴趣的特征相关的定量性状基因座(QTL)。然后可以在品种改善计划中实现与相关基因组区域密切联系的遗传标记。
结果
本研究开发了与表达序列标签(ESTs)相关的单核苷酸多态性(SNP)标记,并用于构建豌豆综合连锁图谱。从一组36188个变异核苷酸位置检测在网上对768个重组自交系(RIL)群体进行基因分型。共705个snp(91.7%)成功检测到分离多态性。除SNPs外,还将基因组和est衍生的简单序列重复序列(SSRs)分配到遗传图谱中,以获得均匀分布的全基因组覆盖。与标记相关的序列用于与其他豆科植物的基因组比较分析。在基因组中观察到较高水平的保守共染色体Medicago truncatula盖恩。和鹰嘴豆(Cicer Arietinum.L.)比与大豆(甘氨酸最大(L。]Merr.),莲花japonicus.L.和鸽子豌豆(Cajanus毛竹(L。]Millsp.).在幼苗生长阶段筛选父母和Ril后代,以应对盐度应力,通过在浇水溶液中以18ds m的浓度加入NaCl施加-1。盐度诱导的症状显示正常分布,症状的严重程度随着时间的推移而增加。QTL用于盐度耐受性,鉴定在Lingage基团Ps III和VII上,具有适合选择抗性品种的侧翼SNP标记。基于这些SNP标记的序列的比较M. Truncatula.基因组定义了含有与盐胁迫耐受性相关的候选基因的基因组区域.
结论
本研究中开发的SNP测定和相关的遗传联系地图允许鉴定盐度耐受QTL和候选基因。这构成了用于标记辅助选择(MAS)计划的重要工具,旨在实现豌豆品种的性能增强。
背景
豌豆(Pisum sativum.L.)在全球范围内广泛培养,作为人类膳食蛋白摄入和牲畜饲料营养的重要植物作物[1].大田豌豆在与谷物轮作时特别有益,以防止病害和提供土壤氮[2].
在生物和非生物胁迫下持续存在的可持续高产品种的发展是满足世界人口不断增长的食物要求的先决条件。几种作物中的作物改善计划采用了分子育种策略,包括大豆和常见豆类等豆类[3.],并且适用于豌豆的应用。通过优化作物架构(即,减少的节间长度),收获指数和苯版特质,具有生长季节长度和降雨[4,5].育种实践也主要集中在对重要真菌病害(如囊叶枯病、白粉病和霜霉病)以及病毒(如豌豆种子传播花叶病毒(PSbMV)和豆叶卷病毒(BLRV))的抗性基因的堆积。然而,在改善生理上的复杂和推定的多基因性状,如耐盐胁迫等方面的努力相对较少[6].
通过对首选基因的鉴定和选择,新的基因组工具将促进复杂性状的遗传改良。对于豆科作物,直到最近才有有限的基因组资源可用,因此MAS的采用一直很慢[6].然而,DNA测序和基因分型技术的进步最近为Field Pea提供了大规模转录组序列数据集[7,8].这些数据可用于设计基于dna的遗传标记,如SSRs和SNPs,支持连锁定位、遗传多样性分析和性状分析[9,10,以及MAS的基因标记[11].
对于豌豆,先前已经开发了大量的遗传联系地图[10,12- - - - - -18].SSR标记在自然界普遍具有共显性和高多态性,在作物育种和遗传研究中被广泛应用于系谱分析[6].SNP普遍普遍,通常是双层和共同主导的性质,序列标记,并且可用于开发低成本多重标记测定的开发,其可以为复杂性状的解剖提供足够致密的基因组覆盖[19,20].许多方法已经被开发用于SNP检测。中等到高通量基于阵列的SNP基因分型系统现在已经可用,这取决于待分析的样本和标记的数量,如GoldenGate®来自illumina Inc.的infinium,来自Beckman Coulter的Snpstream,以及来自Affymetrix的Genechip [18].
为了了解植物中的复杂生物学过程,具有广泛使用模型物种的对比遗传分析。在音乐会中,具有广泛的基因组资源,可用于许多物种的豆科植物底皮番荔枝(例如)M. Truncatula.[http://www.medicago.org.],L. japonicus.[21],鹰嘴豆[22],大豆[23]和鸽子豌豆[24]),这种分析为翻译基因组学提供了协助培育其他,较少学习的作物豆类的机会,例如豌豆。
由于植物生长受限和作物产量下降,土壤盐碱化是一个严重的全球性问题[25].豌豆的盐度耐受性在澳大利亚越来越重要,由于作物生产的地理转变,以较短的季节,更高的水限制和具有较高瞬态土壤盐度的边缘土壤[26].盐度和钠度的大量效果主要是由于NA的水平+阳离子和澳大利亚通常与高碱性(pH> 8.5)土壤有关[27,28].组合,这些因素可以引起营养素(Fe,K)缺陷和土壤毒性(例如硼的升高水平)限制生长和籽粒产量潜力。对于Fea缺乏的豌豆,相对较高和遗传的遗传耐受性[29硼毒性[30.- - - - - -32.]已被确认。在耐盐性方面,基于生物量减少的初步研究表明,大田豌豆对盐的敏感性明显高于其他常见的澳大利亚宽亩作物,如大麦[33.,34.], 小麦 [35.]和油菜籽[36.],由于低盐度阈值水平[37.]在豌豆。与其他豆类相比,相比之下,豌豆[38.- - - - - -41.],以及蚕豆[42.]看起来比鹰嘴豆更耐受[43.]和扁豆[44.].
其他主要旱地作物,如小麦的研究[45.已经证明,由于与高pH和硼等其他应力因子相互作用的复杂性,难以将基于产量的响应测量作为盐度耐受性的衡量标准。+土壤剖面的变异性,以及根据生长阶段和基因型的不同反应。然而,低成本和可靠的盆栽温室筛选方法已经开发了一系列作物,包括豌豆[41.],可用于在苗期识别有用的变异,以便育种。根据筛选实验的结果,似乎有相当大的遗传改良潜力[41.,46.].因此,对含有耐盐胁迫qtl的基因组区域进行识别和标记,将极大地促进该性状向非适应性种质的定向渗透。
本研究的目的是:新型SNP标记的开发和表征和现有SSR标记的表征;构建基于SSR和SNP的连杆地图,适用于盐度耐受性的豌豆种群;豌豆与亚家庭皮吡喹酮植物和其他豆类的比较遗传分析;与野生豌豆盐度耐受相关的基因组区域和分子遗传标志物的鉴定。
方法
植物材料和DNA提取
单基因型杂交的品种Kaspa(盐度敏感)和Parafield(中度耐受)。这些十字架于2007年在德比-霍舍姆表演,F2产生生成后代。单种子血液从f开始2从2008年到2011年,在温室中进行了4代子代遗传基因型的研究。结果F6绘图人口由134个rils组成。
每个子代基因型的冷冻叶片组织使用Mixer Mill 300 (Retsch®,Haan,德国)研磨,使用DNeasy®96植物试剂盒(QIAGEN, Hilden,德国)提取基因组DNA。DNA重悬于1 × TE缓冲液中,浓度为50 ng/μl, -20℃保存。
SNP发现和验证
从转录组序列数据中鉴定出推测的SNPs [8)使用下基因软件v1.96(Softgenetics,州立大学,帕,美国)。基于四种基因型的高质量序列的对准(包括Kaspa和Parafield [8])与共识参考(由此获得)德诺维组装),鉴定了所有基础变体。所有插入和删除(Indel)变体被排除在进一步的分析之外。随后,使用以下标准过滤高置信SNP:(1)每个基因型内的纯合子状况的碱性变体;(2)等于或大于4的读数;(3)在每个SNP中侧翼的20bp段内没有任何其他基础变体。
选取48个SNPs进行Sanger测序验证。引物对采用Sequencher 4.7 (Gene Codes Corporation, USA)和OligoCalc: oligonucotide Properties Calculator (http://www.basic.northwestern.edu/biotools/oligocalc.html.).PCR反应为12 μl,每对引物5 μM, DNA含量为10 ng。放大条件如下:热从94°C 15分钟,其次是35周期95°C的30年代,50°C 30年代和72°C 1分钟,最后延伸步骤在72°C 7分钟。PCR产物纯化在15μl反应包含0.5 U核酸外切酶(新英格兰生物学实验室),0.5 U虾碱性磷酸酶(USB-VWR国际、宾夕法尼亚州、PCR产物5 μl。测序反应总体积为7.5 μl,每个反应包含3.2 μM引物、BigDye®Terminator v3.1 (Life Technologies Australia Pty Ltd, Victoria, Australia)、BigDye®测序缓冲液(Life Technologies Australia Pty Ltd, Victoria, Australia)。澳大利亚),并遵守BigDye®v.3.1协议中所述的骑行条件。延伸产品通过乙醇/EDTA/乙酸钠沉淀法纯化,在12 μl Hi-Di™甲酰胺中重悬(Life Technologies Australia Pty Ltd, Victoria, Australia),并在ABI3730xl自动毛细管电泳平台上分离。DNA序列分析和比对采用Sequencher 4.7,拼接和SNP验证采用目视法。
SSR基因分型
基因组DNA和EST衍生的SSRS [8,17]筛选在映射父母上进行多态性检测。如前所述进行引物合成和PCR扩增[8,47.].PCR产物与ABI GeneScan LIZ500尺寸标准结合,并根据制造商说明使用ABI3730xl (Life Technologies Australia Pty Ltd, Victoria, Australia)毛细管电泳平台进行分析。使用GeneMapper®3.7软件包(Life Technologies Australia Pty Ltd)对等位基因大小进行评分。
框架遗传地图结构和选择最大重组个体
使用JoinMap®3.0构建框架遗传图[48.],阈值log-of-odds (LOD)评分为3,为使用MapPop 1.0版本在作图群体中选择最大重组个体提供基础[49.].
SNP基因分型
选择了SNP的初步清单,为Goldengate选择了®底漆设计(Illumina Inc.,San Diego,CA,USA)。为Illumina每次SNP计算指定性等级分数(0到1)。最后,选择具有0.7和1.0之间的指定性评分的SNP用于开发Illumina Goldengate®用于基因分型的寡核苷酸池测定(OPA)。根据制造商的使用250 ng模板基因组DNA,个体是SNP基因分型。基因分型测定由Illumina Iscan Reader加工。使用Illumina Genome Studio Software V2011.1实现自动等位基因呼叫,其中GeneCall阈值为0.20并视觉检查输出,以及确认群集特异性。
遗传连锁的映射
遗传连锁图谱使用map Manager软件版本QTXb19生成[50.].标记与A.χ2>10分未纳入进一步分析。地图距离是使用Kosambi地图函数计算的[51.,临界值LOD评分为3。LGs是根据标记位点进行分配的[17与公开可用的豌豆联系地图共同,并与染色体相比M. Truncatula.[52.,53.].使用MapChart软件v 2.2绘制LGS [54.].
比较基因组分析
地图分配的SSR和SNP标记的DNA序列用于与鹰嘴豆(NCBI,PREMPNA175619)的基因组组装进行比较分析,M. Truncatula,v3.5 (http://www.medicago.org.),G.Max.v189 (http://www.plantgdb.org.),L. japonicus,v2.5 (ftp://ftp.kazusa.or.jp/pub/lotus/lotus_r2.5/pseudomolecule/),C. Cajan.v5.0(http://www.icrisat.org/gt-bt/iipg/Genome_Manuscript.html).利用BLASTN对每个基因组序列进行相似性搜索,阈值e为10-10.
表型筛选
Kaspa X Parafield Ril群体被筛选用于反应在幼苗阶段施加的NaCl诱导的应力。在Depi-horsham的半控股(Polyhouse)环境中在2012年秋季进行了实验。通过在13厘米直径的罐中播种六个每个RIL的植物来进行筛选,进入沙子和砾石介质(在两个罐复制中的2厘米的深度)。这为每个Ril提供了12个植物作为复制。培养基由粗河砂和5毫米蓝芯片的1:1比例组成。每壶每天用雨水治疗,播种直到出现。外部后6天,除了用钙源(即硝酸钙)补充外,幼苗用完整的营养溶液(即硝基,NPK比12.2:8.5)浇水。使用电导(EC)仪表测试所需的NaCl浓度,并以3ds m的初始速率施用-1从出苗后第9天开始。施用NaCl的浓度增加3ds m-1以避免突发性的渗透休克,最终速率可达18ds m-1,并在评估之前保持这种浓度。在每罐直接施加到生长培养基表面的速率为200ml的3天间隔,以3天间隔进行3天间隔的所有浇水。用于控制管线(父母基因型)和在实验中随机化,包括在实验中随机化,以消除由于其他应力因子引起的效果的含氟盐施用处理(未添加的NaCl)。如前所述,评估每个罐中的个体植物进行症状发展(症状评分)[41.]从出苗后28天,此后每7个TH.一天,直到植物死亡。还获得了最终植物生物质切割,并每种基因型罐记录种子组。从个体植物评估计算植物症状评分的平均值,并使用Reml空间行列分析来估计症状分数的基因型特异性平均值。指数用于量化基因型盐度耐受值,并根据基于加权症状分数和最终生物质描述耐受性的耐受性水平。
用于植物症状评分的平均值(从个体植物评估计算)和盐指数用于产生频率分布直方图。狭义的感觉秘度(h2),利用最佳线性无偏预测(BLUP)分析方法,考虑试验中性状的空间变化趋势,对性状进行计算。
QTL分析和候选基因选择
使用MapManager QTX软件版本QTXB19进行QTL检测。最初进行标记回归分析,以识别显着与特性变化相关的标记(LOD阈值= 3)。简单的间隔映射(SIM)和复合间隔映射(CIM)方法用于识别和确认与耐盐性相关的QTL。侧翼侧翼含有QTL的间隔的序列的序列被爆炸分析M. Truncatula.基因组识别包含假定候选基因的基因组区域。
结果
SNP发现和验证
从映射父母从MET序列数据库中获得的转录组读数的比较,共鉴定了总共36,188个推定的SNP。观察到两种单倍型之间每kB的平均频率为1.85个SNP。在消除Indels后,选择了初步的21,000个SNP。在基于纯合状态的标准和附近没有其他已知的SNP的标准进一步过滤之后,获得了956个高质量SNP的亚组。其中,共满足了953个满足所需的引物设计标准和最终的768个SNP基因座的最终子集,其中为Goldengate选择了1的可指定级别1®化验。
核苷酸变异的分析显示,比横向更优异(2:1)。两个最常见的SNP变体分别为A / G和C / T,分别表示所有变化的36%和32%。其他SNP变体(T / G,C / G,A / C和A / T)占总数的不到10%(附加文件1).通过在768-Plex SNP OPA合成之前的Sanger测序验证了48个SNP基因座的子集(附加文件2),其中45与预测协调一致(附加文件3.).
框架遗传地图结构和选择最大重组个体
总共96个基因组DNA衍生的SSR和EST-SSRS(40%)揭示了父母基因型之间的多态性,其中选择了78,以基于扩增的一致性筛选在绘图群体上。47个SSR标记的子集产生足够的质量数据以产生框架遗传映射,并且将40个基因座(85%)分配给9 LGS。然后使用这些数据来选择101个最大重组个体以用于箱映射。
SNP基因分型
共使用768个SNP对101个选定的RIL进行基因分型。所有SNP都是目视合格的,大多数在Genome Studio中产生两个主要簇,代表纯合子(AA和BB)基因型类别,但偶尔也观察到第三个小簇杂合子(AB)基因型(附加文件4).映射人口被解除为F.6水平,因此剩余杂合度预计较低(c。5 - 10%). 共有705个SNPs(91.7%)产生了连贯的数据,而那些产生模糊聚类结构的SNPs则从进一步分析中删除。462个SNPs(65%)的子集在Kaspa x旁场作图群体中产生多态性簇,并用于遗传连锁图的构建。
键映射
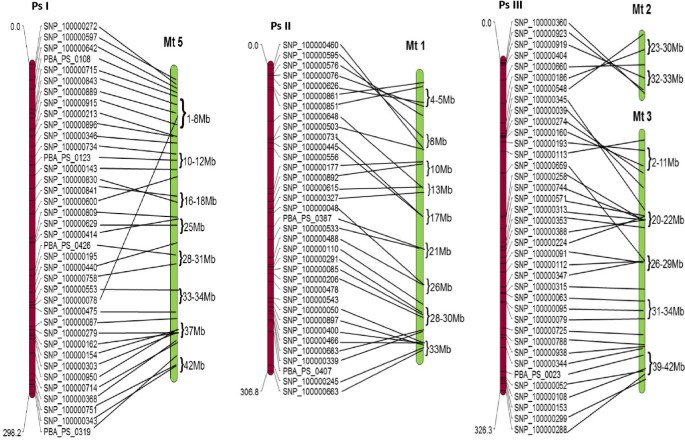
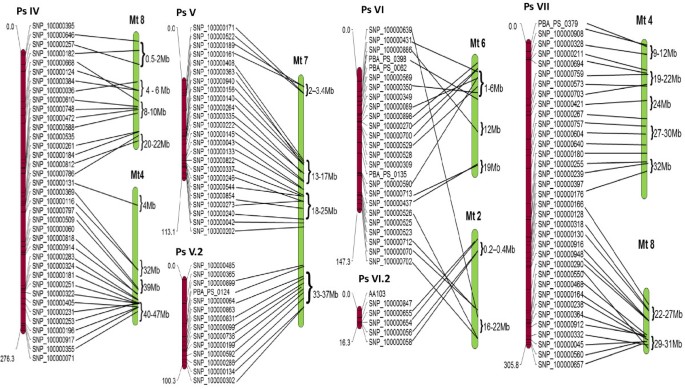
共有73个标记(13.5%)因杂合度过高、数据缺失、偏析或歧义而被排除在连锁分析之外。最后一组467个标记(53个SSR和414个SNP)用于构建连锁图谱。一小部分标记未分组,因此458(98%)个标记包括48个SSR和410个SNP(表1)1)被分配到9 LGS(附加文件5).估计的累积总图长度为1916厘米,平均轨迹间隔为4.2 cm(图1;表格2).LG的身份和定位是通过与M. Truncatula.基因组,以及使用先前将先前映射分配的SSRS作为锚定标记。
比较基因组分析
相应的DNA序列可用于458的映射基因座(15 EST-SSRS和295个SNP)中,其中307检测到的显着序列相似性与至少一种参考基因组序列和130个序列与所有序列显示相似性五个基因组。
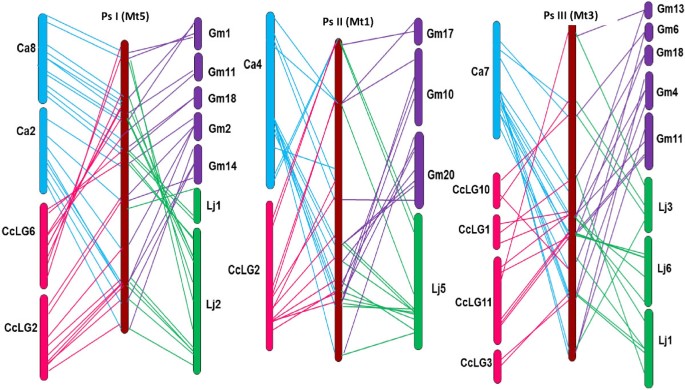
Field PEA映射与鹰嘴豆基因组的比较显示出最多的匹配数量(301:97%)(附加文件6).将豌豆染色体PS II,PS IV,PS V,V.2和PS VII中的每一个相关的同步关系分别对鹰嘴豆PseudoMolueColeCa4,Ca7,Ca 3和Ca6。还观察到一些含有嵌段对多于一个Ca组的块。豌豆 -M. Truncatula.观察到Macorosynteny 292(94%)序列。之中M. Truncatula.染色体,MT5,1,3和7分别显示出豌豆连锁基团PS I,PS II,PS III和PS v的同时性和Colinearity(图2和3.).相反,MT2和6含有最低数量的现场豌豆正端,揭示了与PSLG的更复杂的关系。
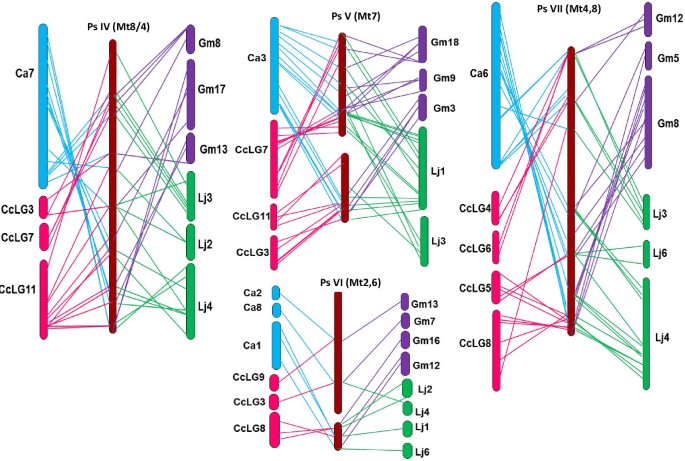
豌豆(LGs PsIV - VII)和豌豆的共线关系示意图M. Truncatula.基因组。详情见图2.
尽管在豌豆和大豆序列之间有大量的匹配(294),在两个基因组之间观察到显着的染色体重排,使得每个PSLG与多于一个大豆染色体表现出大量的同步。比较L. japonicus.鉴定出226个(73%)与片段共线区而非整个染色体关系相匹配。野豌豆-鸽豌豆的共线性分析显示了最低数量的匹配(183),短保守区域分布在不同的染色体上。在大多数情况下,除了CcLG 2和11之外,CcLGs与PsLGs相比顺序颠倒。
利用130个常见的同源序列进一步分析和确认基因组保守程度(图)4和5). 对于大多数PsLGs,鹰嘴豆和豌豆的染色体只鉴定出一条或两条对应的染色体M. Truncatula.,但观察到复杂的关系L. japonicus,鸽豌豆和大豆,与配对比较一致。这些常规模式的例外是PS VI,在所有情况下都显示了复杂的关系。
豌豆(LGS PSIV - VII)与其他豆类基因组的同步关系。详情见图4.
表型分析、QTL检测和候选基因筛选
植物症状响应来自播种阶段的Ril群体的盐度筛选表明,通常分布耐受性的变化(附加文件7和8),因此可能由多个基因控制。估计的狭义感到遗传性(h2)的盐指数为0.55。两种不同的表型测量方法,包括盐指数和平均症状评分(35天的平均症状评分)被用来检测耐盐qtl(图)1),LOD评分为3.2(盐指数)和2.5(症状评分)作为最小意义水平。在PS III和PS VII上鉴定出两种QTL,解释了12%和19%的表型方差(Vp),分别为12%和17%的症状评分(表3.).利用不同时间点(第7、14、21、35天)获得的症状评分进行QTL分析,发现相同的QTL位点,占V的比例相似p(数据没有显示)。在第42、49、56天获得的症状评分的表型数据偏离了正常,因此不能用于基于平均症状评分的QTL分析。
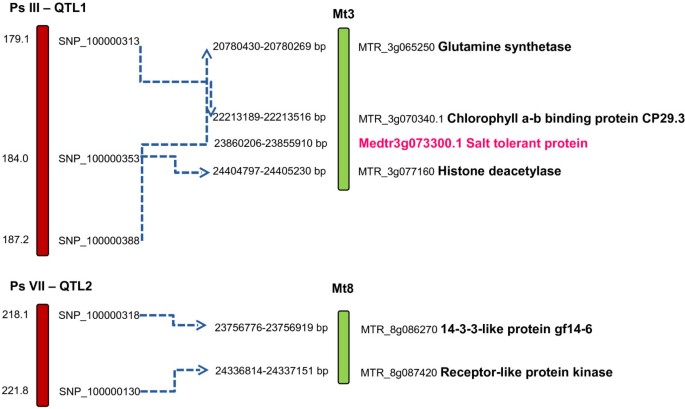
将链接标记相关序列的比较M. Truncatula.基因组直接鉴定具有功能性注释的候选基因,作为受体样蛋白激酶,14-3-3样蛋白质,组蛋白脱甲酰酶和谷氨酰胺合成酶,其据报道参与植物的复合耐盐机制(图6).此外,地区M. Truncatula.检查基因组在链接的SNP相关序列的邻晶片之间的间隔内的间隔内进行候选基因存在。MEDTR3G073300.1基因位于PEA豌豆SNP标记SNP_100000313和SNP_100000353的PS III-QTL1附近的间隔中,并作为耐盐蛋白注释。
讨论
豌豆的SNP变异
植物基因组中的SNP频率显着变化,这取决于生殖习惯(自然或共同的),分析区域评估和地位(编码或非编码)的种群多样性。本研究中豌豆中检测到的SNP频率远低于谷物作物报告的值(小麦/小麦/小麦的16.5个SNP,每kB中的4.2个SNP [55.]),但类似于其他豆类(每kB中0.9个SNPS [豇豆)[56.],每kB中1.96个SNPM. Truncatula.[57.]和2.06个单核苷酸多态性/ kb的大豆[58.])。
核苷酸替换模式显示A/G和C/T是最常见的碱基变化,这与以往对白三叶草等豆科植物的研究一致[59.]和鹰嘴豆[60.].C/T转变的高比例可能部分是由于5-甲基胞嘧啶反应的脱氨,这在进化过程中经常发生,特别是在CpG二核苷酸[61.].
金门的有效性和适用性®以前证明了对基因分型绘制群体和豌豆遗传资源收集的SNP测定[18].本研究提供了额外的SNP标记,可用于分子育种程序。SNP基因分型的成功率(约91%)与之前在豌豆中所做的观察(92.7%)相当[18鹰嘴豆(90.75%)[60.].SNP基因分型的成功取决于许多因素,包括基本变体选择,相邻的SNP频率,重复序列的存在,以及最终,可指分。由于现场豌豆SNP发现基于来自多种基因型的转录组测序[8]但是,少数少数标记(C.35%)并不令人惊讶的是在绘图家庭中未能检测多态性。然而,在所选基因型中包含Kaspa和Parafield确保了高频率的成功。
遗传连锁的映射
此前,随着新的分子标记技术的相继采用,已经开发了若干种豌豆田间连锁图谱[10,12- - - - - -17].本研究中构建的连杆地图表现出常规的标记分布,但是在减数分裂原型的典型的Chiasma频率(1-2个每双偶)基于典型的Chiasma频率(1-2倍)的累积遗传型映射(1916cm)明显更长。豌豆遗传联系地图的这种扩展也被报道(1700cm [62.];2202.7厘米(63.])。不同作图群体的遗传构成、作图策略、作图位点的数量和类型、作图软件的选择、标记数量与群体大小的比例[64.- - - - - -67.].
比较基因组分析
在豌豆和鹰嘴豆之间基因组结构的广泛保存M. Truncatula.与本研究中使用的其他豆科植物相比,这些种之间的系统发育关系更密切。与之前鹰嘴豆和其他豆科植物的比较基因研究结果相比[68.,69.,在本研究中观察到大量的宏观同步。
在8染色体之间观察到染色体结构的广泛保护M. Truncatula.豌豆的7个LGs,以及进化易位的证据[52.,70].先前的一些研究[52.,53.]的研究表明,与Mt1和5相比,保护水平较高;与Mt3、4、7和8相比,保护水平中等;与Mt2和6相比,保护水平较低。与其他Mt染色体不同,Mt6染色体长度短,重复次数多,基因含量低(但有相当数量的NBS-LRR抗病基因),异染色质含量高[71.].PS VI与MT2和6匹配,包含最少数量的正交序列查询,与这些先前的研究一致。通过从PS VI的较大的标记队列的发展可能会弥补这种情况。尽管是一个c。在基因组大小之间的10倍差异M. Truncatula.豌豆[72.[两种基因组之间的广泛同时性表明,随后从C的进化分歧之后,豌豆谱系中没有发生全基因组重复。40 mya [53.,73.].豌豆的较大基因组大小可能是多个转位事件的结果[74.].本研究的结果在很大程度上扩展了对豌豆和豌豆的比较知识M. Truncatula.这些信息可用于候选基因的选择,以进一步应用于育种计划。
相反,跨越整个PSLG的大型同步块与与...的比较不存在L. japonicus,大豆和鸽子豌豆基因组.前者是Papilionoideae亚家族的Galegoid Clade的成员,但与豌豆更省略M. Truncatula.鹰嘴豆和鹰嘴豆,而后两种是Phaseoloid分支的成员,因此观察到的关系符合广泛的系统发育亲缘关系[75.].对于大豆,尽管大量的正交序列,所以越有限的关系也可能反映了该物种的复杂古聚倍性基因组结构[76.].豌豆 -L. japonicus.比较揭示了相似之处,但经常被染色体重排所打断。在介于相似的节段性同步关系L. japonicus.和Galegoid草料豆类白三叶草[77.[还从比较中推断出来M. Truncatula.[78.].豌豆和鸽豌豆之间的广泛染色体重排,同样表明分类分类差异的影响。
表型分析、QTL检测和候选基因筛选
植物对耐盐性的反应受到各种生理机制的影响,这些生理机制很可能是由跨不同环境的多个基因控制的[79.].本研究表明,培养源自调整和高产父母植物豌豆基因型的幼苗诱导盐度耐受的定量基础,以及中等水平的遗传性C.45%的变异是由于非遗传因素。在PS III和PS VII上识别出两个QTL基因座,每个算法适用于v的中等比例p.与耐盐性相关的不同生理特性的研究M. Truncatula.鉴定了总共19个推定的基因组区域,最大数量的QTL位于MT8上,其次是MT5,1,3,4,7,6和2 [80].但由于无法接近,无法进行直接比较QTL分析M. Truncatula.QTL区间两侧标记的相关序列。然而,比较基因组分析显示,Ps III与Mt2/3、Ps VII与Mt4/8之间存在宏观共线关系。因此,本研究鉴定的qtl可能是在Galegoid豆科植物之间保守的。
本研究确定了豌豆盐耐盐机制相关的候选基因。组蛋白脱乙酰酶和谷氨酰胺合成酶在植物中具有耐盐胁迫的关键作用[81.,82.]而14-3-3蛋白质调节各种靶点的活动,并在对盐水应激的反应中发挥重要作用[83.].受体样蛋白激酶参与各种过程,包括生物/非生物应激反应[84.].此外,耐盐蛋白(STO)被鉴定为参与内源性钠调控的基因产物之一+/K+比例,耐盐性的基本过程[85.].因此,在QTL的区域内鉴定的基因是合理的候选者,但验证需要额外的研究。
本研究中鉴定的QTL与幼苗生长阶段盐度耐受性有关。类似地,在许多谷物作物中鉴定了幼苗生长耐受性的QTL,包括稻米[86.],大麦[87.],大豆[88.]和小麦[89.].其他qtl与番茄萌发期生长反应的相关机制[90.,91.)、大米(92.],大麦[93.]和小麦[89.)或在生殖发育期间(水稻[94.],大麦[95.]和番茄[96.])可能对现场pea具有重要意义,值得进一步调查。盐分诱导生长反应的程度和时间在作物物种内部和物种之间的巨大差异突出了这一特性的复杂性。
在MAS中,分子标记的实现很少用于生理复杂性状,如耐盐性[97.].在这种情况下,育种者将需要选择不同种质,不同的筛选环境和不同的组织阶段中发现的不同和多种基因组区域或响应机制。因此,可能需要量化自适应性质[98.根据不同的盐度应力,不同的QTL,并分配基因组值,类似于基于指标的选择。基因组测序和基因分型能力的进展,尤其是基因分型逐个测序(GBS),提供了基因组范围的标记分析的可能性[99.以及不考虑效应大小而识别所有与某种性状(如耐盐性)有关的基因座的能力。这些数据可用于基于所有性状连锁标记的育种价值估算,以确定目标渐渗程序的关键亲本。
结论
本文介绍了利用est衍生的多位点SNPs构建豌豆遗传连锁图谱的方法。在温室条件下的耐盐性评价允许鉴定两个重要的基因组区域。利用序列相关标记确定了大田豌豆与其他5种豆科植物的宏观共线关系,并预测了候选耐盐基因。这一信息可用于耐盐品种标记辅助选择(MAS)的连锁和诊断多态性的开发,基于qtl基因组区域从供体到受体种质的渗透。由于耐盐性是一种复杂的生理性状,未来的研究将需要在不同的筛选环境和不同的个体发生阶段进行评估,以确定额外的相关基因组区域。最后,本研究获得的遗传资源将有助于其他性状分析研究,并有助于相关豆科作物的信息传递,为未来田间豌豆育种提供参考。
参考
- 1.
Kocer A,Albayrak S:豌豆饲料产量和质量的测定(Pisum sativum.与燕麦和大麦的混合物。特克J大田作物。2012, 17: 96-99.
- 2.
低投入种植制度和氮肥对作物生产的影响:土壤氮素动态和玉米作物氮素利用效率。农业环境科学学报,2011,11:282-295。
- 3。
Chamarthi SK, Kumar A, Voung TD, Blair MW, Gaur PM, Nguyen HT, Varshney RK:性状图谱和分子育种。食用豆类的生物学与育种。牛津郡:CAB国际;2011:296 - 313。
- 4。
McPhee柯:豌豆。植物的基因组定位和分子育种:豆类、糖和块茎作物。卷3。编辑:Kole C. Berlin:施普林格;2007:33-47。
- 5。
Redden B,Leonforte T,Ford R,Croser J,Slattery J:Pea(Pisum sativum.l .)。遗传资源,染色体工程和作物改进。编辑:辛格rj。佛罗里达州,美国:CRC媒体;2005年:49-83。
- 6。
Kumar J,Choudhary Ak,Solanki Rk,PRATAP AP:朝着脉冲中的标记辅助选择:审查。植物品种。2011,130:297-313。10.1111 / J.1439-0523.2011.01851.X。
- 7。
Franssen SU, Shrestha RP, Bräutigam A, Bornberg-Bauer E, Weber APM:高度复杂的综合转录组分析Pisum sativum.基因组使用下一代测序。基因组学杂志。2011,12:227-10.1186/1471-2164-12-227。
- 8。
Kaur S,Pembleton LW,Cogan No,Savin Kw,Leonforte T,Paull J,Materne M,Forster JW:Field Pea和Faba Bean的转录ome测序用于发现和验证SSR遗传标记。BMC基因组学。2012,12:265-276。
- 9。
Jing Z, Qu Y, Yu C, Pan D, Fan Z, Chen J, Li C:利用普通野生稻的高级回交群体进行产量相关性状的QTL分析选用高产稻l)。莫尔植物品种。2007,1:1-10。
- 10。
Fondevilla S,KüsterH,Krajinski F,Cubero Ji,Rubiales D:鉴定在抗性反应中差异表达的基因球腔菌属pinodes使用微阵列技术的豌豆。BMC基因组学。2011,13:12-28。
- 11
凯伦BCY,JAHUFER MZZ,BROROWER JB,PANK ECK:标记介绍,定量特质基因座(QTL)映射和作物改进的标记辅助选择:基本概念。Euphytica。2005,142:169-196。10.1007 / s10681-005-1681-5。
- 12
Timmerman-Vaughan GM, McCallum JA, Frew TJ, Weeden NF, Russell AC:控制豌豆种子重量的数量性状连锁作图(Pisum sativum.L.)。应用基因理论。1996,93:431-439.10.1007/BF0022187。
- 13
McCallum J,Timmerman-Vaughan G,Frew TJ,Russell AC:豌豆绿色种子颜色的生化和遗传联系分析(Pisum sativum.l .)。J AM SoC Hort SCI。1997,122:218-225。
- 14。
Weeden NF, Ellis THN, Timmerman-Vaughan GM, Swiecicki WK, Rozov SM, Berdnikov VA:一个一致的连锁图谱Pisum sativum..皮瑟姆·吉奈,1998,30:1-4。
- 15.
[Pilet-Nayel, Muehlbauer FJ, McGee RJ, Kraft JM, Baranger A, Coyne CJ]:豌豆对隐球菌根腐病部分抗性的数量性状位点。《中国科学:地球科学》2002年第1期。
- 16。
Prioul S,Frankewitz A,Deriot G,Morin G,Baranger A:Quantativie特质抵抗力的映射mycosopharella pinodes.在豌豆(Pisum sativum.),在苗期和成株期。中国科学(d辑),2004,28(6):591 - 598。10.1007 / s00122 - 003 - 1543 - 2。
- 17。
Loridon K, McPhee K, Morin J, Dubreuil P, Pilet-Nayel ML, Aubert G, Rameau C, Baranger A, Coyne C, Lejeune-Hènaut I, Burstin J:豌豆微卫星标记多态性与定位(Pisum sativum.l .)。Al Appl Genet。2005,111:1022-1031。10.1007 / s00122-005-0014-3。
- 18。
Deulvot C,Charrel H,Marty A,Jacquin F,Donnadieu C,Lejeune-Hénauti,Burstin J,Aubert G:豌豆遗传测绘和种质多样性研究的高度复用SNP基因分型。BMC基因组学。2010,11:468-10.1186 / 1471-2164-11-468。
- 19。
Krawczak M:双胞胎单核苷酸多态性的信息性评估。电泳。1999年,20:1676-1681。10.1002 /(SICI)1522-2683(199990101)20:8 <1676 :: AID-ELPS1676> 3.0.co; 2-D。
- 20。
王涛,邢国强,邢国强:微卫星、单核苷酸多态性和SNPs复合标记在连锁分析中的比较。BMC Genet. 2005, 6: S29-10.1186/1471-2156-6-S1-S29。
- 21。
佐藤年代,中村Y, T,金子Asamizu E,加藤T,地震区M, Sasamoto年代,渡边,小野,川岛K, Fujishiro T, Katoh M, Kohara M, Kishida Y,南城C, Nakayama年代,Nakazaki N,清水Y, Shinpo年代,高桥C,和田T,山田M, Ohmido N, Hayashi M,福井K,巴巴T, Nakamichi T, Mori H, Tabata年代:豆科植物的基因组结构。植物遗传资源学报,2008,27(5):523 - 527。10.1093 / dnares / dsn008。
- 22。
Varshney RK,Song C,Saxena RK,Azam S,Yu S,Sharpe AG,Cannon S,Baek J,Rosen BD,Tar'an B,Millan T,Zhang X,Ramsay LD,IWATA A,Wang Y,Farmer广告,Gaur PM, Soderlund C, Penmetsa RV, Xu C, Bharti AK, He W, Winter P, Zhao S, Hane JK, Carrasquilla-Garcia N, Condie JA, Upadhyaya HD, Luo MC, N W, et al: Draft genome sequence of chickpea (Cicer Arietinum.)为性状改良提供了资源。生物技术学报。2013,31:240-246。10.1038 / nbt.2491。
- 23.
Schmutz J,Cannon SB,Schlueter J,Ma J,MITROS T,Nelson W,Hyten DL,Song Q,Thelen JJ,Cheng J,Xu D,Hellsten U,May Gd,Yu Y,Sakurai T,Umezawa T,Bhattacharyya MK,Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J, Tian Z, Zhu L, et al: Genome sequence of the palaeopolyploid soybean. Nature. 2012, 463: 178-183.
- 24.
瓦尔沙尼rk,陈某,李伊,bharti ak,萨克拿rk,schlueter ja,donoghue mt,亚萨姆s,风扇g,whaley am,farmer广告,谢里丹j,岩石a,tuteja r,penmetsa rv,wu w,upadhyaya hd,杨sp,shah t,saxena kb,michael t,mccombie wr,yang b,张g,杨h,王j,spillane c,厨师博士,五月gd,xu x等:pigeonpea的草案基因组序列草案(Cajanus毛竹),是资源匮乏的农民的一种孤儿豆类作物。生物技术。2011,30:83-89。10.1038 / nbt.2022。
- 25.
Ondrasek G,Rengel Z,Veres S:作物生产中的土壤盐渍化和盐胁迫。植物中的非生物胁迫-机制和适应。编辑:Shanker A。ISBN:978-953-307-394-1,内政部英特赫:10.5772/22248[http://www.intechopen.com/books/abiotic-stress-in-plants-mechanisms-adaptations/soil-salinisation-and-salt-stress-in-cropuction.],
- 26.
利用遗传耐受性来克服碱性作物土壤中底土的限制。土壤学报,2009,48(4):489 - 494。
- 27。
Rengasamy P:澳大利亚肥皂土壤中旱地农业的瞬态盐度和替补限制:概述。Aust J Exp agr。2002,42:351-361。10.1071 / EA01111。
- 28。
Rengasamy P:世界盐渍化重点在澳大利亚。J Exp Bot。2006,57:1017-1023。10.1093 / JXB / ERJ108。
- 29。
Kabir啊,Paltridge NG,能够AJ,Paull JG,Stangoulis JC:Fe-效率的自然变化与策略I机制的上调和增强的柠檬酸盐和乙烯合成有关Pisum sativum.L. planta。2012,235:1409-1419。10.1007 / s00425-011-1583-9。
- 30。
Bagheri A, paul JG, Rathjen AJ:回应Pisum sativum.土壤硼浓度对羊草种质的影响。草业学报1994,75:9-17。10.1007 / BF00024526。
- 31。
Bagheri A,Paull JG,Rthjen AJ:豌豆中高浓度土壤硼的遗传学(Pisum sativum.l .)。Euphytica。1996,87:69-75。10.1007 / bf00022967。
- 32。
Leonforte A,Noy D,Redden R,Inneking D:改善植物豌豆的硼和盐度耐受性(Pisum sativum.l .)。第14届澳大利亚植物育种(APB)会议和第11届亚洲和大洋洲(Sabrao)大会推进的第11次社会2009年。澳大利亚昆士兰:凯恩斯;2009年。
- 33。
Maas EV:植物的耐盐性。应用的农业res。1986,1:12-26。
- 34。
Saxena NP,Saxena MC,Ruckenbauer P,Rana Rs,El-Fouly MM,Shabana R:凉爽季节食物豆类盐度和矿物质不平衡的筛选技术和耐受性。Euphytica。1994年,73:85-93。10.1007 / BF00027185。
- 35
Francois LE,Maas EV:受盐影响土壤上的作物响应和管理。植物和作物胁迫手册。编辑:Pessarakli M。纽约:马塞尔·德克尔;1994:149-181.
- 36
Steppuhn H,Volkmar Km,Miller Pr:比较油菜,豌豆,干豆和杜兰姆小麦作物在盐水培养基中种植。农作物科学。2001,41:1827-1833。10.2135 / cropsci2001.1827。
- 37
B:蚕豆的反应(维亚比亚法瓦)还有豌豆(Pisum sativum.)品种盐度。印度j agr sci。1989,59:729-731。
- 38。
F:豌豆的耐受性(Pisum sativum.长期盐胁迫与诱导抗氧化防御有关。acta botanica sinica(云南植物学报). 2000,23(4):553 - 562。10.1046 / j.1365-3040.2000.00602.x。
- 39。
El-Hamdaoui A, Redondo-Nieto M, Rivilla R, Bonilla I, Bolaños L:硼钙营养对番茄幼苗生长发育的影响Remuminosarum.- 豌豆(Pisum sativum.)盐胁迫下共生和结瘤的发育。植物细胞环境。2003年,26:1003-1011.10.1046/j.1365-3040.2003.00995.x。
- 40.
长期NaCl胁迫对豌豆超氧化物歧化酶和抗坏血酸过氧化物酶同工酶的影响(Pisum sativum.简历。Puget)叶绿体。J Exp Bot。2004年,55:119-130。
- 41.
Leonforte A,Forster JW,Redden RJ,Nicolas Me,Salisbury Pa:豌豆盐度高耐受性的来源(Pisum sativum.l .)。中国科学:地球科学。10.1007 / s10681 - 012 - 0771 - 4。
- 42.
Cordovilla MP,Ligero F,Lluch C:寄主基因型对蚕豆生长、共生性能和氮同化的影响(维亚比亚法瓦L.)盐胁迫下。植物土壤。1995,172:289-297。10.1007 / bf00011331。
- 43。
Sadiki M,Rabih K:精选鹰嘴豆(Cicer Arietinum.的产量和盐胁迫下共生固氮能力。农业科学。2001,21:659-666。10.1051 /农业:2001158。
- 44。
Maher L,Armstrong R,Connor D:耐盐扁豆 - 未来的可能性?第11届澳大利亚农学会会议:2003年2月。吉朗,维多利亚:澳大利亚农学会;2003年:2-6。
- 45。
Munns R,James R,Lauchli A:提高小麦和其他谷物耐盐性的方法。J Exp Bot。2006,57:1025-1043。10.1093 / JXB / ERJ100。
- 46。
Leonforte A,Noy D,Forster JW,Salisbury P:评估较高耐受Nacl的耐受性Pisum sativum.L.第五届国际研究会议的诉讼程序。土耳其:anatalya;2010年。
- 47.
一种用于PCR片段荧光标记的经济方法。生物工程学报。2000,18:233-234。10.1038/72708。
- 48.
范Ooijen J,Voorrips R:JoinMap 3.0用于计算遗传联系地图的软件。Wageningen,荷兰:植物研究国际;2001年。
- 49.
棕色DG,Vision TJ:Mappop 1.0版:用于选择性映射和BIN映射的软件。2000年,http://www.bio.unc.edu/faculty/vision/lab/mappop/]
- 50。
Manly KF, Cudmore RH, Meer JM: Map Manager QTX,用于基因图谱绘制的跨平台软件。《哺乳动物基因组》,2001,12:930-932。10.1007 / s00335 - 001 - 1016 - 3。
- 51。
Kosambi DD:根据重组值估算地图距离。安·尤金,1944,12:172-175。
- 52。
Choi HK, Mun JH, Kim DJ, Zhu H, Baek JM, Mudge J, Roe B, Ellis N, Doyle J, Kiss GB, Young ND, Cook DR:估算作物和模式豆类物种的基因组保护。中国科学(d辑:地球科学),2004,32(5):591 - 598。10.1073 / pnas.0402251101。
- 53。
Kalo P,Seres A,Taylor SA,Jakab J,Kevei Z,Kereszt A,Endre G,Ellis Thn,Kiss GB:与之间的比较映射Medicago Sativa.和Pisum sativum..Mol Gen Genomics。2004,272:235-246。10.1007 / s00438-004-1055-z。
- 54。
Voorrips RE: MapChart:图形化表达连锁图和qtl的软件。何立德,2002,33:77-78。10.1093 / jhered / 93.1.77。
- 55。
Barker GL,Edwards KJ:全球核苷酸多态性多态性的全基因组分析,在世界各大谷物作物中的单核苷酸多态性多样性。植物Biotechnol J. 2009,7:318-325。10.1111 / J.1467-7652.2009.00412.x。
- 56。
Bhat Pr,Fenton Rd,Wanamaker S,Pottorff M,Hearne S,Cisse N,Fatokun C,Ehlers JD,Roberts Pa,Close TJ:Cowpea的共识遗传地图[豇豆属unguiculata(l)沃尔普。]和基于EST衍生的SNP的同步。Proc Natl Acad Sci U S A. 2009,106:18159-18164。10.1073 / pnas.0905886106。
- 57。
Choi HK, Kim DJ, Uhm T, Limpens E, Lim H, Mun JH, Kalo P, Penmetsa RV, Seres A, Kulikova O, Roe BA, Bisseling T, Kiss GB, Cook DR:基于序列的基因图谱Medicago Truncatul.A和比较标记离罗素性的M. Sativa..遗传学。2004,166:1463-1502。10.1534 / genetics.166.3.1463。
- 58。
Choi IY、Hyden DL、Matukumalli LK、Song Q、Chaky JM、Quigley CV、Chase K、Lark KG、Reiter RS、Yoon MS、Hwang EY、Yi SI、Young ND、Shoemaker RC、van Tassell CP、Specht JE、Cregan PB:大豆转录本图谱:基因分布、单倍型和单核苷酸多态性分析。遗传学。2007,176:685-696.10.1534/genetics.107.070821。
- 59
Hand ML, Ponting RC, Drayton MC, Lawless KA, Cogan NO, Charles Brummer E, Sawbridge TI, Spangenberg GC, Smith KF, Forster JW:基于与祖类群比较的远系异源多倍体物种同源、同源和副同源序列变异的鉴定。中国生物医学工程学报。2008,29(4):429 - 434。10.1007 / s00438 - 008 - 0365 - y。
- 60.
Gaur R,Azam S,Jeena G,Khan AW,Choudhary S,Jain M,Yadav G,Tyagi Ak,Chattopadhyay D,Bhatia S:高通量SNP发现和基因分型,用于构建鹰嘴豆的饱和联系地图(Cicer Arietinum.l .)。DNA Res。2012,19:357-373。10.1093 / DNARES / DSS018。
- 61.
Holliday R,Grigg GW:DNA甲基化和突变。mutatres。1993年,285:61-67。10.1016 / 0027-5107(93)90052-h。
- 62.
埃利斯THN,特纳L,Hellens RP,Lee D,Harker Cl,Enard C,Domoney C,Davies Dr:Linkages Maps在Pea。遗传学。1992,130:649-663。
- 63.
Moreno RR:豌豆产量构成数量性状位点的定位与鉴定,Pisum sativum.SSP。博士论文。伊利诺伊州北部;2009年。
- 64.
Liu G, Bernhardt JL, Jia MH, Wamishe YA, Jia Y:重组粳稻-籼稻杂交自交系群体的分子特征。acta physica sinica, 2008, 59(4): 457 - 461。
- 65.
诺克斯先生,埃利斯THN:豌豆地图扩张。遗传学报。2002,32(4):591 - 598。
- 66.
Sim SC, Durstewitz G, Plieske J, Wieseke R, Ganal MW, Van Deynze A, Hamilton JP, Buell CR, Causse M, Wijeratne S, Francis DM:番茄大型SNP基因分型序列的建立和高密度遗传图谱的生成。PLoS ONE。2012年,7:e40563 - 10.1371 / journal.pone.0040563。
- 67。
白泽明、贝蒂奥利DJ、瓦什尼RK、莫特松MC、莱尔·贝蒂奥利SC、图迪M、潘迪MK、拉米JF、方卡D、高达MV、秦H、郭B、洪Y、梁X、平川H、塔巴塔S、伊索贝S:栽培花生和野生亲缘植物的综合一致性图谱揭示了花生A和B基因组的结构落花生豆科植物基因组的分化。中国生物医学工程学报,2013,30(4):457 - 461。10.1093 / dnares / dss042。
- 68。
Hiremath PJ,Kumar A,Penmetsa RV,Farmer A,Schlueter Ja,Chamarthi Sk,Whaley Am,Carrasquilla-Garcia N,Gaur Pm,Upadhyaya HD,Kavi Kishor PB,Shah TM,Cook Dr,Varshney RK:大规模发展具有成本效益的SNP标记分析,用于鸡肝的多样性评估和遗传映射和豆类中的比较映射。植物Biotechnol J. 2012,10:716-732。10.1111 / J.1467-7652.2012.00710.x。
- 69。
Nayak SN,朱镕基H, Varghese N,达塔,崔港元,Horres R, R丛林,辛格J, Kavi基肖尔PB, Sivaramakrihnan年代,Hoisington哒,卡尔G,冬天P,库克博士,Varshney RK:集成新的SSR和基于基因的SNP标记位点的鹰嘴豆遗传图谱,建立新的锚点Medicago truncatula基因组。Al Appl Genet。2010,120:1415-1441。10.1007 / s00122-010-1265-1。
- 70。
Aubert G, Morin J, Jacque F, Loridon K, Quillet MC, Petit A, Rameau C, Lejeune-Hénaut I, Huguet T, Burstin J:豌豆的功能定位,作为候选基因选择的辅助,并研究与模式豆科植物的同源性Medicago truncatula.Al Appl Genet。2006,112:1024-4。10.1007 / s00122-005-0205-y。
- 71。
Cannon SB、Sterck L、Rombauts S、Sato S、Cheung F、Gouzy J、Wang X、Mudge J、Vasdwani J、Schiex T、Spannagl M、Monaghan E、Nicholson C、Humphray SJ、Schoof H、Mayer KF、Rogers J、Quétier F、Oldr yd GE、DebelléF、Cook DR、Retzel EF、Roe BA、Town CD、Tabata S、Van de Peer Y Y、Young ND:豆科植物基因组进化Medicago truncatula和莲花japonicus.基因组。PROC NATL ACAD SCI。2006,103:14959-14964。10.1073 / pnas.0603228103。
- 72。
Arumuganathan K,Earle Ed:一些重要植物物种的核DNA含量。植物MOL BIOL REP。1991,9:208-218。10.1007 / BF02672069。
- 73。
Wojciechowski MF:重建豆类的系统发育(豆科植物):21世纪初的观点。豆类系统的进展。第10部分编辑:Klitgaard BB,Bruneau A. Royal Botanic Gardens,Kew:更高水平的系统学;2003年:5-35。
- 74。
Vershinin AV, Allnutt TR, Knox MR, Ambrose MJ, Ellis THN:转座因子揭示了基因渗入的影响,而不是置换Pisum.多样性,进化和驯化。mol Biol Evol。2003,20:2067-2075。10.1093 / molbev / msg220。
- 75。
Doyle JJ, Luckow MA:冰山的其余部分,豆科植物的多样性和进化的系统发育背景。植物生理学报。2003,31(4):497 - 503。10.1104 / pp.102.018150。
- 76。
Saxena RK,Penmetsa RV,Upadhyaya HD,Kumar A,Carrasquilla-Garcia N,Schluetes Ja,Farmer A,Whaley Am,Sarma BK,May Gd,Cook Dr,Varshney RK:大规模开发成本效益的单核苷酸多态性贪污遗传映射的标记测定和豆类中的比较映射。DNA Res。2012,19:449-461。10.1093 / DNARES / DSS025。
- 77。
Isobe Sn,Hisano H,Sato S,Hirakawa H,Okumura K,Shirasawa K,Sasamoto S,Watanabe A,Wada T,Kishida Y,Tsuruoka H,Fujishiro T,Yamada M,Kohara M,Tabata S:比较遗传映射和发现白三叶草连锁群中的联动不平衡(Trifolium Repens.l .)。中国生物医学工程学报,2012,29(2):429 - 434。
- 78。
George J, Sawbridge TI, Cogan NOI, Gendall AR, Smith KF, Spangenberg GC, Forster JW:白三叶和白三叶基因组结构的比较Medicago truncatula支持基于保守同义的同源群命名法。基因组学杂志。2008,51:905-911。10.1139 / g08 - 076。
- 79。
马铃薯耐盐基因研究的最新进展。acta botanica sinica(植物研究与开发),2004,34(6):531 - 534。
- 80.
Arraouadi S,Chardon F,Huguet T,Aouani Me,Badri M:与耐盐耐受相关的形态特征的QTL映射Medicago truncatula. 生理植物学报。2011,33:917-929.10.1007/s11738-010-0621-8。
- 81.
王志强,王志强,王志强,等:表观遗传过程在胁迫下植物开花时间调控中的作用。中国生物医学工程学报。2011,32(4):527 - 534。10.1093 / jxb / err177。
- 82.
Aydi S,Sassi S,Debouba M,Hessini K,Larrainzar E,Gouia H,Abdelly C:抵抗力Medicago truncatula盐胁迫与谷氨酰胺合成酶活性和钠螯合有关。J植物Nutr土壤Sc。2010,173:892-899。10.1002 / JPLN.200900235。
- 83.
徐健,SHI WM:14-3-3基因家族的表达谱响应盐胁迫和钾盐和煤矿和铁缺乏症(Solanum lycopersicum)根:通过实时RT-PCR分析。Ann Bot(Lond)。2006,98:965-974。10.1093 / AOB / MCL189。
- 84.
欧阳树清,刘艳芳,刘鹏,雷刚,何淑娟,马波,张文坤,张建生,陈淑英:受体样激酶OsSIK1改善水稻耐旱盐胁迫能力(栽培稻) 植物。工厂J.2010,62:316-329。10.1111 / J.1365-313x.2010.04146.x。
- 85.
Nagaoka S,Takano T:耐盐相关的蛋白质STO与MyB转录因子同源物结合,并赋予耐盐性拟南芥.中国海洋大学学报(自然科学版)。10.1093 / jxb / erg241。
- 86.
Alam R,Rahman Ms,Seraj Zi,Thomson MJ,Ismail Am,Tumimbang-raiz E,Gregorio GB:使用源泉线的幼苗级盐度耐受QTL的研究栽培稻L. Pokkali。植物品种。2011,130:430-437。10.1111 / j.1439-0523.2010.01837.x。
- 87.
周G,约翰逊P,Ryan PR,德尔海泽E,周M:大麦耐盐性的数量性状位点(大麦芽l .)。《昆虫学报》2012,29:427-439。10.1007 / s11032 - 011 - 9559 - 9。
- 88.
Hamwieh广告,Tuyen D,Cong H,Benitez Er,Takahashi R,Xu DH:大豆耐盐耐盐性QTL的识别和验证。Euphytica。2011,179:451-459。10.1007 / s10681-011-0347-8。
- 89.
MA LQ,周EF,Huo NZ,周RH,王GY,佳JZ:小麦重组血统群体耐盐性遗传分析(Triticum Aestivum.l .)。Euphytica。2007,153:109-117。
- 90.
林国英:番茄萌发期耐盐性与营养生长的关系。植物育种。1997,116:363-367。10.1111 / j.1439-0523.1997.tb01013.x。
- 91。
番茄耐盐性及种间杂交相关的RAPD标记Lycospersicon esculentumxl . pennelli).植物细胞播放。1998,17:306-312。10.1007 / S002990050398。
- 92。
程海涛,蒋辉,薛德伟,郭立波,曾东丽,张光华,钱强:水稻萌发期和苗期耐碱QTL的定位。作物学报,2008,34:1719-1727。10.1016 / s1875 - 2780(09) 60006 - 7。
- 93。
Mano Y,Takeda K:在萌发中绘制定量特质,用于散热和大麦的幼苗阶段(大麦芽l .)。Euphytica。1997,94:263-272。10.1023 /答:1002968207362。
- 94。
Manneh B,STAM P,Struik PC,Bruce-Oliver S,Van Eeuwijk Fa:基于QTL的基因型 - 基因型分析,对压力和非应力环境中水稻籽粒产量的基因型逐环相互作用分析。Euphytica。2007,156:213-226。10.1007 / s10681-007-9368-8。
- 95。
薛DW,周MZ,张XQ,陈S,魏克,曾富,毛y,吴FB,张GP:不同生长条件下大麦产量和产量成分的QTLS鉴定。J Zheijang Univ Sci B. 2012,11:169-176。
- 96。
Villalta I,Bernet GP,Carbonell EA,Asins MJ:使用两个水果产量方面的盐度耐受的对比QTL分析Solanum.F-7线的群体。Al Appl Genet。2007,114:1001-1017。10.1007 / s00122-006-0494-9。
- 97。
Ashraf M,Foolad先生:分子标志物时代的耐盐性和标记辅助选择的作物育种。植物品种。2013,132:10-20。10.1111 / PBR.12000。
- 98。
非生物胁迫下的数量性状位点与作物性能:我们的立场是什么?植物营养与肥料学报。2008,27(4):439 - 446。10.1104 / pp.108.118117。
- 99。
Elshire RJ,Glaubitz JC,Sun Q,Poland Ja,Kawamoto K,Buckler Es,Mitchell Se:一种高分多样性物种的鲁棒,简单的基因分序列(GBS)方法。Plos一个。2011,4:E19379-
致谢
这项工作得到了维多利亚环境和初级工业部门以及澳大利亚谷物研究和发展理事会的资助。
作者信息
隶属关系
通讯作者
额外的信息
利益争夺
提交人声明他们没有竞争利益。
作者的贡献
AL表演人口发展,表型评估,数据解释,并为起草稿件进行了贡献。SS进行了标记发现,地图结构,QTL分析,比较基因组学,并有助于起草稿件。NC共同概念化了该项目,促成了数据解释并协助起草手稿。PS和MN共同概念化项目并协助起草稿件。JF,MM和SK共同概念化并协调项目并协助起草手稿。所有作者阅读并认可的终稿。
Antonio Leonforte、Shimna Sudheesh对这项工作做出了同样的贡献。
电子补充材料
12870_2013_1380_moesm1_esm.pptx.
附加文件1:SNP基本变体的百分比。此文件包含一个饼图,描绘了每个SNP基本变量类的百分比。(PPTX 47 KB)
12870_2013_1380_moesm2_esm.xlsx.
附加文件2:768plex SNP-OPA设计的细节。此文件包含用于链接映射的所有SNP标记的名称和序列信息。(XLSX 74 KB)
12870 _2013_1380_moesm3_esm.png
附加文件3:使用Sanger Sequencing的SNP验证。该文件包含由Sanger测序生成的电泳图的示例,以演示SNP验证,并显示不同映射家庭父母之间的两个引起的SNP。(PNG 91 KB)
12870 _2013_1380_moesm4_esm.pptx
附加文件4:IlluminaGoldengate®SNP基因分型测定产生的代表性聚类模式。该文件包含在两个映射群体上从SNP基因分型测定获得的聚类模式的示例。数据点颜色代码表示:红色,AA(纯合);蓝色,BB(纯合);紫色,ab(杂合);黑色,没有呼叫(缺少数据)。a)高品质的多晶型SNP;b)单数态SNP;c)具有大量杂合子的SNP;d)失败的SNP。 (PPTX 106 KB)
12870_2013_1380_moesm5_esm.xlsx.
附加文件5:链接映射统计信息。这个文件包含了不同标记(SSRs和SNPs)的详细信息以及它们在不同lg上对应的位置。(XLSX 19 KB)
同时性分析统计。
附加文件6:此文件详细信息Field Pea LGS,标记序列数量和鹰嘴豆的同时性,M. Truncatula,大豆、L. japonicus.和鸽子染色体。(XLSX 12 KB)
频率分布直方图。
附加文件7:18 dS m盐度处理后Kaspa x Parafield RIL子代盐度指数值和定性评级(T(耐受)、MT-T(中度耐受至耐受)、MS-S(中度敏感至敏感)、S(敏感)、HS(高度敏感)的频率分布-1.(PPTX 77 KB)
频率分布直方图。
附加文件8:在18 dS m的水溶液中施用NaCl后,Kaspa x Parafield RIL后代在7、14、21、35、42、49和56天症状评分的频率分布-1. (PPTX(72 KB)
作者的原始提交的图像文件
权利和权限
开放存取本文在“生物资源”中央有限公司的许可下公布了这是一个开放式访问条款,分配根据创意公约归因许可证的条款(https://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。
关于这篇文章
引用这篇文章
Leonforte,A.,Sudheesh,S.,Cogan,N.O.et al。豌豆耐盐性增强的SNP标记发现、连锁图谱构建及qtl鉴定(Pisum sativum.l .)。BMC植物BIOL.13,161(2013)。https://doi.org/10.1186/1471-2229-13-161
已收到:
接受:
发表:
关键字
- 谷物豆类
- 遗传标记
- 特质解剖
- 比较基因组学
- 非生物胁迫
- 配种