摘要
背景
甘蔗花叶病毒(SCMV)病使易感玉米品种的粮食产量和饲料生物量大幅下降。玉米对SCMV的抗性与两个显性基因有关,Scmv1而且Scmv2,分别位于6号染色体的短臂和3号染色体的着丝粒区附近。我们结合连锁和关联作图来鉴定位置候选基因Scmv1.
结果
Scmv1在来自近等基因系的分离群体中进行精细定位,并使用两个重组自交系群体进一步验证和精细定位。综合结果分配Scmv1根据公开的B73序列预测该区域内的候选基因。三个可能参与抗病反应的预测基因中没有一个与受体样抗性基因相似。利用94个抗SCMV变异自交系进行了候选基因的关联定位。的存在/缺失变异(PAV)Scmv1区域周围有两个多态位点Zmtrx-h基因与SCMV抗性显著相关。
结论
结合链接和关联映射精确点Zmtrx-h作为最有可能的位置候选基因Scmv1.这些结果为克隆Scmv1促进玉米抗马铃薯病毒的标记辅助选择。
背景
甘蔗花叶病毒是最常见的病毒病原体之一,在全球范围内造成玉米和饲草产量的重大损失[1],尤其是在中国和德国[1,2].最近在波兰和法国中部的一份关于SCMV的报告显示,SCMV的发病率持续增加[3.,4].感染SCMV的诊断症状包括发育迟缓、褪绿、植株重量和产量降低[1,5].SCMV原称为玉米矮花叶病毒(MDMV) B株,属于玉米矮花叶病毒科Potyviridae[5].家庭的其他成员Potyviridae同样侵染玉米的病毒包括MDMV、小麦条纹花叶病毒(WSMV)、约翰逊草花叶病毒(JGMV)、高粱花叶病毒(SrMV)和玉米花叶病毒(ZeMV) [6,7].由于病毒通过蚜虫媒介的非持续性传播,农艺和化学手段对SCMV的控制是无效的。因此,部署抗性品种是防止产量损失的最重要途径,这是基于抗性种质的筛选和鉴定[8].
美国自交系Pa405在田间和温室条件下均表现出对MDMV和SCMV的完全抗性[9].在欧洲,对122个早熟玉米自交系进行了抗SCMV的筛选,结果鉴定出三个系D21、D32和FAP1360A对SCMV表现出完全抗性[6].在中国,许多优良自交系在接受SCMV接种后表现出完全抗性[2,10].对抗性品系D32与易感品系D145杂交的数量性状位点(QTL)分析显示,在第3和6号染色体上有两个主要的显性基因,在第1、5和10号染色体上有三个次要的QTL [11,12].使用FAP1360A(完全抗SCMV)和F7(对SCMV高度敏感)杂交后代的高分辨率映射证实了这一点Scmv1(在6号染色体的短臂上)和Scmv2(靠近3号染色体的着丝粒)对玉米抗SCMV至关重要[13].在几个独立的实验中也报告了类似的测绘结果[8,14,15].的Scmv1在中国抗性品系黄早4中也检测到基因座,并在染色体1、3、5和10上检测到额外的QTL [14].在Siyi×Mo17的子代中,两个主基因分别定位在第3染色体(bin 3.04/05)和第6染色体(bin 6.00/01)上,[8].通过使用F9在Huangzao4×Mo17杂交获得的-衍生的永生RIL群体中,在染色体仓6.00和6.01之间检测到一个主要的QTL,占总表型变异的50.0%,使疾病指数下降27.9% [16].在巴西的热带玉米种质中,第3和第6染色体上的位点也得到了证实[15].在B73全基因组序列的帮助下Scmv2区域精细映射到0.28 cM的区域,覆盖物理距离为1342.6 kb。四种可能参与病毒运动的预测基因可能是候选基因Scmv2[17].存在两个紧密相连的抗性基因Scmv1区域已被假定[18,19].有趣的是,一些赋予对各种病毒抗性的QTL都映射到6号染色体上的同一区域[20.].目前尚不确定,是否是单一的多效性Scmv1基因赋予不同的病毒抗性,或不同的抗性基因聚集在这个区域。
尽管使用了不同的测绘人群,但所有的研究都一致地确定了Scmv1而且Scmv2基因组区域被认为是赋予SCMV抗性的关键。在本研究中,我们首先对Scmv1使用大的等基因作图群体,以前显示仅为隔离Scmv1[13,18,19,21,22].其次,我们从对SCMV具有不同抗性的精英亲本系中选取了两个RIL群体,以确认并进一步精确定位Scmv1轨迹。第三,我们确定了有限数量的位置Scmv1基于组合精细定位结果的候选基因。最后,我们利用94个抗SCMV变异玉米自交系进行QTL区域和候选基因的关联作图。
结果
精细定位的Scmv1在一个近等基因的F2人口
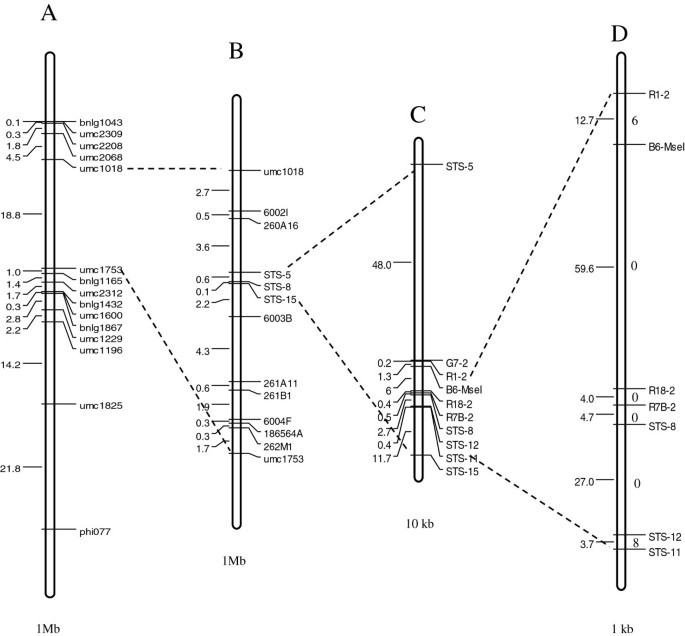
在177个未选中的F250株初次接种后2 ~ 3周内表现出典型的SCMV感染症状。抗性和易感株数以3:1的比例(χ2= 0.83 < χ20.01, 1),与单一显性基因遗传相一致。利用玉米遗传和基因组学数据库(http://www.maizegdb.org/)(附加文件1),Scmv1根据B73物理图,在umc1018和umc1753之间绘制了一个位点,物理距离为18.80 Mb1A).由于缺乏高密度标记,这两个标记之间的28个重组无法解析。据此,我们开发了11个新的标记(表1)基于美国国家生物技术信息中心的GenBank中公开的玉米细菌人工染色体(BAC)序列(http://www.ncbi.nlm.nih.gov).我们用这11个新的标记来解析28个重组体。随后的标记-表型分析使我们能够逐步完善Scmv1在STS-5和STS-15之间形成710.97 kb的间隔(图5 -15)1B),在这两个标记之间仍有两个重组子。然后,利用标记STS-5和STS-15对510f进行基因分型2易感植物,产生13个新的重组。在STS-5/STS-15区间内,单拷贝/低拷贝序列被开发出7个额外的标记(表2 -1)1).这些标记被用于基因型15重组,这使我们能够划分Scmv1由两个最近的标记R1-2和STS-11组成的112.39 kb的区间1C和1D)左边还有6个重组体,右边还有8个重组体Scmv1轨迹(图1D).与B73物理图谱的比较表明,距离最近的两个标记R1-2和STS-11分别位于GenBank中相邻的BACs上:b0129G15和b0598N23。
顺序精细映射过程的示意图。(一)17个多态SSR标记的物理图谱来自MaizeGDB (http://www.maizegdb.org/)在Scmv1基因组区,17个定位不明确的标记中有2个被删除。通过分析177个未选择的F2植物,Scmv1由SSRs umc1018和umc1573缩小到18.8 Mb的间隔。(B)的物理映射Scmv1精细定位后得到28个重组体和13个新开发的标记。(C),(D)最终的物理地图Scmv1利用8个新开发的标记对15个STS-15和STS-5之间的重组进行基因分型。染色体段左侧的数目对应AGPv2的物理距离,A、B、C、D的单位分别为1mb、1mb、10kb、1kb。染色体片段右侧的数字表示两个侧翼标记之间的重组数。
的验证Scmv1在瑞来斯
考虑到Scmv1近等基因的F不能进一步缩小区域2群体中,我们使用了两个RIL群体,其亲本系对SCMV的抗性进行了对比,以确认和精细定位Scmv1地区。因为两者Scmv1而且Scmv2在玉米对SCMV的抗性中,每个RIL的基因型都被研究了Scmv2利用Ingvardsen等人开发的共分离标记DJF004基因座[17].考虑到其中一个Scmv1侧翼标记STS-11未能揭示两个RIL群体亲本之间的多态性,我们在STS-11周围寻找了一个替代标记,最终开发了一个新的标记B4,位于STS11下游约103.75 kb。B4标记和R1-2标记一起用于所有RILs的基因分型Scmv1轨迹。对于每个RIL群体,有4组30个RILScmv1或Scmv2或分别选取两个位点进行表型评价。
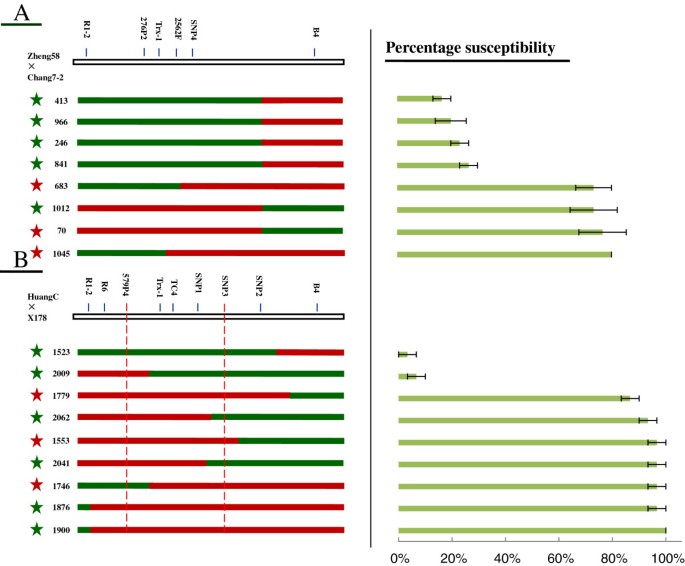
在Zheng58×Chang7-2的RIL群体中,30个RIL中有28个与长7-2具有相同的单倍型Scmv1而且Scmv2基因座(RR/RR)对SCMV高度耐药。30个长7-2单倍型的RILsScmv1,但郑58单倍型在Scmv2(SS/RR),只有1个RIL对SCMV耐药。另外有两组30个RILs与郑58具有相同的单倍型Scmv1基因座(SS/SS和RR/SS)均易受SCMV影响。在X178×HuangC的RIL群体中观察到类似的结果,所有30个RIL都与X178具有相同的单倍型Scmv1而且Scmv2基因座(RR/RR)对SCMV表现出较高的抗性。30个X178单倍型的RILsScmv1黄c单倍型在Scmv2(SS/RR),只有4个RILs对SCMV耐药。另外两组30个RILs与黄c具有相同的单倍型Scmv1基因座(SS/SS或RR/SS)均易发生SCMV。这些发现表明两者都存在Scmv1而且Scmv2基因座是SCMV耐药的先决条件,如以前的报告所述[13].显著差异(P抗性百分比< 0.01)Scmv1而且Scmv2基因座表明在两个RIL群体中存在相同的耐药基因座(表2)2).
进一步的精细映射Scmv1在瑞来斯
根据验证结果,使用侧翼标记B4和R1-2筛选215.47-kb范围内的重组体Scmv1时间间隔。在RIL群体Zheng58×Chang7-2和X178×HuangC中分别检测到8个和9个重组子。为Scmv2基因座,共分离标记DFJ004用于确定每个RIL的基因型。在温室和田间条件下,对所有重组株的抗SCMV性进行了反复研究。另一方面,通过对1145、FAP1360A和黄早4 3个抗病自交系和B4/R1-2区间内的1个易感B73序列的多重比较,开发了10个标记,可解析17个重组体。
从Zheng58×Chang7-2 RIL群体中筛选出的8个重组株中,4个RIL株对SCMV的抗性与抗性亲本长7-2相当,另外4个RIL株对SCMV的抗性与敏感亲本郑58相当。的Scmv270号、683号和1045号三个易感重组体的基因座与易感亲本郑58号的基因座相同,因此不能用于确定易感重组体Scmv1轨迹。4个耐药RIL(413、966、246和841号)和1个易感RIL(1012号)的抗性等位基因为Chang7-2Scmv2轨迹,因此可用于精细映射Scmv1轨迹。4个耐药RIL位于标记B4上游的Chang7-2供体区,1个易感RIL 1012位于标记SNP4下游的Chang7-2供体区。这些数据支持Scmv1基因座位于标记B4的上游(图2A).地图Scmv1Zheng58×Chang7-2 RIL种群的区域跨度为112.39 kbScmv1近等基因的F2人口,因此没有价值进一步缩小Scmv1轨迹。
进一步的精细映射Scmv1在两个RIL群体中。每种重组型的遗传结构用绿色和红色矩形表示(绿色表示来自抗性亲本chang7-2或X178的片段;红色象征来自易感父母的片段,正58或黄c)。星号代表等位基因Scmv23号染色体上的基因座(绿色表示来自长7-2或X178的抗性等位基因;红色代表来自Zheng58或HuangC的易感等位基因)。右边的浅绿色柱状图表示根据温室中三个独立实验得出的具有SCMV症状的植物的平均百分比。一)的Scmv1在RIL群体中,基因座可划分为215.47 kb的区域Zheng58×Chang7-2。B)的Scmv1在RIL群体HuangC×X178中,该基因座可被标记579P4和SNP3(红色虚线)划分为59.21 kb的区域。
在从RIL群体X178×HuangC筛选的9个重组体中,有2个表现出与抗性亲本X178相同的高抗性,而另外7个与亲本HuangC一样对SCMV敏感。1779号、1553号和1746号是3个高易感的RILs,携带黄c易感等位基因Scmv2轨迹。因此,它们被排除在精细测绘Scmv1轨迹。1900号、1876号、2041号和2062号4个RILs在X178位点有抗性等位基因Scmv2轨迹。但均对SCMV易感。这一发现表明X178位点上抗性等位基因的缺失Scmv1这四个RILs的轨迹。其中,1876号和1900号ril在R1-2标记下游存在供体黄c区,2041号和2062号ril在SNP3标记上游存在供体黄c区Scmv1基因座受R1-2和SNP3标记的限制。2009号和1523号两个高抗性株系携带X178抗性等位基因Scmv2轨迹和他们的Scmv1两侧分别标记579P4和B4。因此,Scmv1该基因座可以缩小为59.21 kb的区域,两侧是标记579P4和SNP3(图2B),位于112.39 kb的近等基因F2人口。
预测地图内和周围的假定基因Scmv1地区
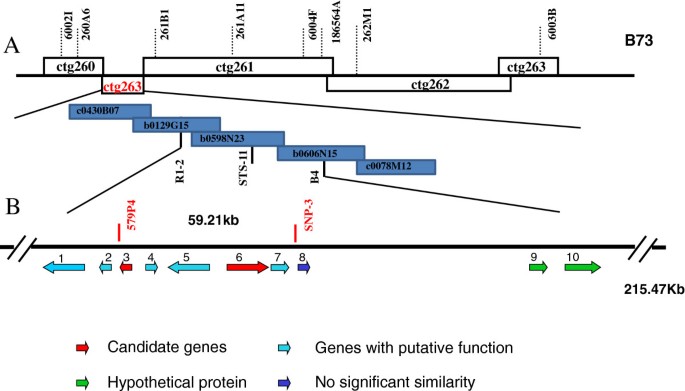
从中提取到B4和R1-2标记的215.47 kb序列http://www.maizesequence.org确定候选人Scmv1基因。该区域仍有7个缺口,表明存在一些缺失序列。转座子占该区域的55.27%,其中82.28%为逆转录转座子,99.87%的逆转录转座子为长端重复元件(数据未显示)。我们使用Fgenesh软件(http://linux1.softberry.com/berry.phtml(2.6版本),从屏蔽序列中预测10个假定基因,包括7个假定功能基因,2个假定基因,1个与已知基因无显著相似性的基因(图3.C).无典型阻力(R)基因在Scmv1地区。基因本体论(GO)和京都基因与基因组百科全书(KEGG)注释的7个预测基因的可能功能分析表明,其中3个基因可能对SCMV具有抗性(表5)3.).
整合三个种群的测绘结果。(一)的组合映射Scmv1.上的contigs的修改顺序Scmv1区域是#260-#263-#261-#262-#264(红色的ctg263是其中Scmv1位于)。Scmv1最终被分配到一个59.21 kb的区域,标记为579P4和SNP3。(B)覆盖目标区域的215.47-kb B73序列的基因预测:每个箭头代表一个预测基因,头部代表基因方向,箭头长度对应基因长度(详细信息见表3.).
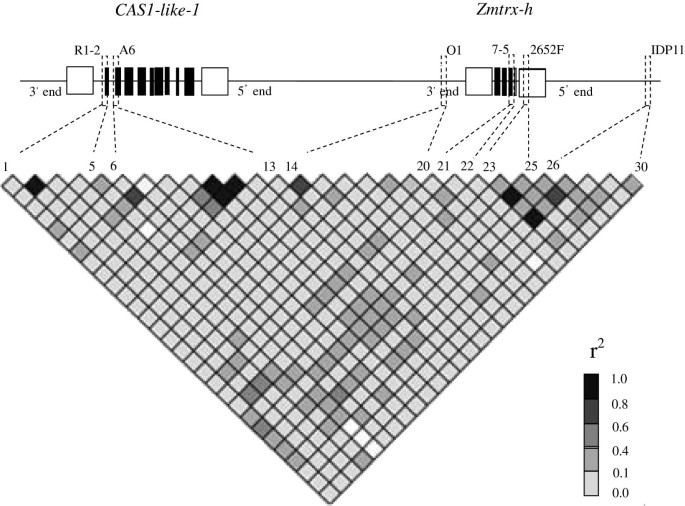
其中两个预测基因是环artenol synthase -like (CAS1-like)与同源基因序列相似度高的基因拟南芥。第一个覆盖率为76%,e值为1.00E-129,而第二个覆盖率为60%,e值为1.00E-74。这两个CAS1-like虽然氨基酸序列长度不同,但相似度为74.18% (204/275),E值为2.00E-117。我们指定较大的基因(403残基)为CAS1-like-1,较小的(360个残差)为CAS1-like-2.另一个候选基因,Zmtrx-h,编码一种假定的119残基蛋白,与硫氧还蛋白高度相似(100%覆盖率;E = 4.00E-64),一种氧化还原酶,作用于硫基团的供体,使用二硫作为受体。
综合作图结果表明Scmv1位于579P4和SNP3两侧的59.21 kb区域3.B)在这个区域的五个预测基因中,CAS1-like-2而且Zmtrx-h比其他三个基因更有可能对SCMV产生抗性。
候选基因基于关联映射Scmv1轨迹
为了验证候选区域,并通过识别数量性状核苷酸多态性来改进分析,我们使用94个自交系进行了关联映射研究(附加文件)2),基于70个SSR标记将其分为6个亚组(附加文件3.).多序列比对分析在6个信息性扩增子中鉴定出30个多态性。连锁不平衡(LD)分析显示r较低2在这些多态性之间(图4),占r的91.03%2< 0.20。两个(单核苷酸多态性)SNP位点S454和S126分别位于基因的3 '端和内含子上Zmtrx-h与SCMV抗性显著相关(P <0.05;表格4和附加文件4).尽管如此,低r20.01表示有两个独立的功能变异。
考虑到94个玉米自交系中有19个(包括Mo17)无法通过聚合酶链式反应(PCR)扩增,其中17个引物对分散在玉米自交系中Scmv1区域,我们确定了一大片PAV覆盖Scmv1地区。该PAV由施普林格等人验证。[23],在6号染色体上有2.6 mb的区域,包括Scmv1在B73基因组中存在,而在Mo17基因组中不存在。引人注目的是,所有19个自交系都对SCMV易感,从而与SCMV抗性有很强的关联(P= 9.96)的军医。
讨论
精确的表型评价对标记-性状关联分析至关重要[24- - - - - -27],尤其对于外显性和表型可塑性不完全的性状。玉米对SCMV的抗性除受两大基因的影响外,还受遗传背景和各种环境因素的影响[13,17,18].为了获得可靠的表型数据,我们开发了F2从近等基因对中定位群体,其中抗性等位基因固定在Scmv2轨迹,而且只有Scmv1正在隔离[28].此外,从F2人口的精细测绘Scmv1基因座降低了错误分类的可能性,因为杂合子对SCMV接种的反应更具可塑性。
多种基因组平台和分析方法的进步使重要农艺性状QTL的精细定位和克隆成为可能[29].在目前的研究中,在112.39 kb中开发了多态标记Scmv1区域是一个挑战,因为高度重复的序列。因此,我们对1145、FAP1360A和Huangzao4 BAC文库的阳性克隆进行测序,以掩盖重复序列Scmv1位点,以揭示单拷贝/低拷贝序列用于标记发育。一般来说,对更多玉米自交系进行重测序[30.]以及其他可公开获取的序列信息的可用性(例如Mo17:http://www.phytozome.net/maize.php;和Palomero:http://www.palomerotoluqueno.org/index.php)将促进目标基因组区域标记的发展。在精细制图过程中,我们发现BAC contigs的发表顺序(http://www.maizesequence.org)Scmv1区域配置错误。基于BAC的B73全基因组物理图谱表明,相邻的BAC contigs #260和#261应该在物理上彼此接近。然而,由于遗传距离太大,在umc1018/umc1753区间内的重组都不能被基于261号contig的新标记进一步解析。只有从263染色体发育而来的标记才能解析重组。这表明contig #263位于contig #260和#261之间,并且正确的contig顺序必须是#260-#263-#261-#262-#264(图2)3.A).此正确的contig顺序已释放(http://www.maizesequence.org).
寻找具有明确表型的重组子对于精确的图谱绘制至关重要。因此,我们只使用F2对人口进行精细映射,这允许我们精细映射Scmv1位点转化为112.39 kb的区域,两侧为R1-2和STS-11。从事实来看Scmv1位点已多次检测到[8,12,14,21,我们推断Scmv1可能存在于各种抗性自交系中。基于这一假设,我们使用了两个RIL群体来精细定位Scmv1基因座进入一个59.21 kb的区域,两侧有标记579P4和SNP3。
公开的B73序列显示了两个预测基因,即CAS1-like homoolog andZmtrx-h吉恩,是最有可能Scmv1由于它们在疾病防御反应中的潜在作用[31,32],尽管假设蛋白质的潜在影响Scmv1区域对SCMV的抗性不能排除。与完整的比较CAS1基因拟南芥,编码759个残基,表明假定的CAS1-like该基因可能是含有萜烯合成酶基序的部分基因。CAS1参与类固醇和非甾体次级代谢产物的生物合成,如菜油甾醇和豆甾醇[31].菜油甾醇是油菜素内酯的前体,油菜素内酯是一类重要的植物激素,在细胞信号转导中起作用,而豆甾醇的积累刺激了细菌感染叶片后发生的重要植物代谢过程[32].考虑到表型变异与Scmv1区域受植物发育阶段的影响[19],目前还不清楚情况是否与Scmv1非常类似于款Hm2.的删减副本Hm1.相比Hm1,款Hm2主要作用于成体植物,对叶斑病和穗霉病具有抗性石竹蜗1[33,34].
硫氧还蛋白是细胞氧化还原状态的主要调节因子[35],据报道h型硫氧还蛋白在病毒防御反应中起作用[36]和真菌[37].考虑到Zmtrx-h蛋白质Scmv1区域缺乏保守的WC(G/P)PC基序,这对氧化还原蛋白的活性至关重要,但似乎不太可能Zmtrx-h影响细胞氧化还原状态。因此,需要进一步研究这三个候选基因的作用,以揭示SCMV耐药的机制。
连锁分析和关联分析是绘制基因或QTL的两种常用方法。两者可以互补地用于分析感兴趣性状的遗传基础[38,39],以及精确定位目标QTL的致病变异[40].用6对引物检测基因序列中特定片段的相关性Scmv1具有抗SCMV的区域。一般线性模型(GLM)分析强调了三个与SCMV抗性相关的多态性。PAV和SCMV耐药性之间的显著相关性证实了抗肿瘤药物的作用Scmv1而其他两个SNPs与SCMV耐药性之间的关联提供了统计学支持Zmtrx-h作为竞选的主要候选人Scmv1.而且,这两个SNP位于内含子和3 '端Zmtrx-h,似乎对基因表达有影响。的CAS1-like-2目前还不能排除基因,因为在其附近没有引物对可用。因此,仍然需要通过转化进行补充功能测试,以确定哪些候选基因定义Scmv1.这些转基因实验正在我们的实验室进行。
广谱耐药性(BSR),即对两种或两种以上类型的病原体或同一病原体的大多数种的耐药性[41],对提高作物对各种疾病的抵抗力至关重要。数量抗性基因Lr34最近克隆的小麦具有抗叶锈病、条锈病、白粉病和其他多种小麦病害的能力[42].玉米群体中存在多种抗病基因的证据[43揭示了定量BSR可以由单个基因授予。的Scmv1据报道,该地区有抵抗其他成员的同伙Potyviridae家族,包括西米氏病毒及中米氏病毒[44,45].近等基因系F7RR / RR在本研究中被用作抗性亲本系,也被发现对MDMV和ZeMV具有抗性[28].根据寇提出的模型[46],赋予BSR的单个基因可能在基础抗性途径、赋予种族特异性抗性的重叠途径或不同防御途径之间的交叉对话位点上发挥作用。考虑到这两个候选基因在基础抗性中都有假定的作用,Scmv1可能会产生BSR,这在植物受到多种病原体威胁的环境中是有利的。然而,这一推测还有待于人工接种具有适当致病菌范围的转基因植物和重组体来验证。
结论
我们绘制了精细地图Scmv1通过整合不同种群的测绘数据,得到59.21 kb的区域。这为启动分子育种以提高玉米抗SCMV能力提供了不可或缺的数据,玉米是一种对世界粮食安全至关重要的作物。我们确定了两个候选人,其中Zmtrx-h吉恩似乎最有希望。基于地图的克隆Scmv1转基因分析将揭示这种重要的植物-病毒相互作用的分子机制。
方法
用于精细绘图的种群
F7近等基因系F7RR / RR、F7RR /党卫军,和F7SS / RR由两个早熟欧洲玉米自交系FAP1360A(完全抗SCMV)和F7(对SCMV高度易感)培育而成,如Ingvardsen [17](附加文件5).总共2200华氏度2源自F7杂交的植物RR / RR×F7RR /党卫军被收集作为映射群体进行精细映射Scmv1.最初,一组177株植物从F2映射人口,被用作未选定的映射人口,以划定Scmv1地区。每株机械接种SCMV两次,每隔一周进行一次评分,以评估对SCMV的抗性。的附加标记Scmv1对未选择的F进行基因分型2人口。与此同时,剩下的2023个F2研究了植物对SCMV的抗性,结果鉴定出510株具有典型SCMV症状的植物与其易感亲本F7难以区分。这510种易感F2植物,以及28个来自未选择的F2映射人口,形成F2子种群进行精细映射Scmv1.
验证Scmv1两个RIL群体的基因座
两颗单籽下降F6RIL群体被用于验证和进一步精细映射Scmv1轨迹。一种是郑58(抗SCMV)和长7-2(抗SCMV)的杂交,后者是一种目前在中国广泛种植的商业杂交品种(ZD958)。另一个RIL群体来自X178(完全抗SCMV)和黄c(对SCMV高度敏感)的杂交,黄c是另一个在中国广泛种植的商业杂交(ND108)。总共2,206 F62010年夏,在北京上庄试验站种植了1285株(Zheng58×Chang7-2)和921株(X178×HangC)。所有RILs均用ril侧翼的R1-2和B4标记进行基因型Scmv1基因座,以及同分离标记DJF004Scmv2轨迹(17].对于每个RIL群体,有4组30个RILScmv1或Scmv2分别选择基因座或两个基因座进行表型评价,而所有R1-2和B4之间的重组子都对SCMV抗性进行了研究。
人工接种
在温室条件下,将种子播种在0.80 l的土壤/蛭石混合物中。从感染scmv的易感F7植株中制备病毒接种体(中国分离株)。将具有典型花叶症状的幼叶组织均质于5体积0.01 M磷酸钠缓冲液中,pH为7.0。在液液中加入碳化硅以促进摩擦接种。每株叶片均在3 ~ 4叶期进行摩擦接种。接种1周后,所有无症状株均重新接种,防止脱毒。在制备液液和机械接种过程中,将接种体置于冰上以保持低温条件。
为评价自交系和选育ril的表型,每系12粒在4个0.80 l的播种盆中播种。10株接种病毒,2株未接种病毒。这些RILs连同它们的亲本系作为所有测试的阳性和阴性对照进行评估。具有相同症状的RILs (P> 0.05, t检验)作为易感亲本或耐药亲本作为易感(S)或耐药(R)。为确保表型准确,于2010年9月至2011年7月进行了3次独立实验。
DNA提取和分子分析
从每个个体身上采集未成熟的叶子样本,并立即在液氮中冷冻。DNA提取采用Murray和Thompson描述的SDS方法[47].
除了从公共BAC序列中开发的新标记外,所有SSR标记均从玉米遗传与基因组学数据库(http://www.maizegdb.org)(附加文件1),由Invitrogen公司(北京,中国)合成。为了设计特定的标记进行精细定位,我们首先将bin 6.01中的QTL锚定在基于B73 bac的全基因组物理图上(http://www.maizesequence.org/)使用侧翼SSR标记。接下来我们提取BACs,表达序列标签和Indel多态序列(http://www.ncbi.nlm.nih.gov/;http://magi.plantgenomics.iastate.edu/browseMarkers.do),以开发新的多态性标记(SSR、STS、SNP和CAPS)。用RepeatMasker (http://www.repeatmasker.org/cgi-bin/WEBRepeatMasker),然后对玉米高通量基因组序列进行BLAST分析和基因组序列调查,去除冗余序列[48].所有单拷贝/低拷贝序列的引物均采用PRIMER 5.0软件设计,引物长度为20个核苷酸,GC含量为40-60%,无二级结构,不存在相同核苷酸的连续片段。微卫星搜索工具SSRHunter1.3 [49]用于挖掘>2碱基对的4-6个串联拷贝的SSR序列。
PCR检测SSR和STS标记时,反应体积为10 μL,含100 ng基因组DNA, 10× PCR缓冲液1 μL, dNTP各0.2 mM,正向和反向引物各0.2 μM, Taq聚合酶1u。在50 μL的反应中,模板DNA 300 ng, 10× PCR缓冲液5 μL, dNTP各1 mM,正向和反向引物各1 μM, 5 U Taq聚合酶(北京Transgen Biotech Co.LTD .)进行SNP和CAPS标记检测。所有PCR反应均使用PTC-200 Peltier热循环仪(MJ Research Inc.,加拿大)进行,每个反应都有特定的退火温度和延伸时间。PCR产物用1%琼脂糖凝胶(使用美国Bio-Rad Laboratories Inc.的凝胶成像系统显示)或6%聚丙烯酰胺凝胶(用银染色显示扩增产物)进行电泳。SNP标记在北京基因组研究所测序。
候选基因分析
为了预测候选基因,我们检索了基因的DNA序列Scmv1来自B73全基因组序列的区域(http://www.maizesequence.org)用于候选基因预测。的重复序列Scmv1使用RepeatMasker 3.3.0版本(http://www.repeatmasker.org).我们使用Fgenesh程序(http://linux1.softberry.com/berry.phtml(版本2.6)来预测单拷贝/低拷贝中的基因Scmv1地区。为了进行功能分析,我们将每个推测基因的氨基酸序列与国家生物技术信息中心网站(http://blast.ncbi.nlm.nih.gov/).详细的GO和KEGG注释应用于BLAST搜索预测的基因序列(http://www.geneontology.org/而且http://www.genome.jp/kegg/).
的关联映射Scmv1地区
在温室条件下(2007年10月和2010年5月),采用与RIL群体相同的人工接种程序,对94个玉米自交系进行了表型分析。同样的94个自交系使用70个SSR标记进行基因分型,这些标记均匀分布在玉米基因组中(附加文件)6).基于B73序列,使用Primer 3 (http://frodo.wi.mit.edu/)或Premier 5.00 (Premier Biosoft International, USA),我们设计了17对PCR引物(附加文件7)在三个候选基因的侧翼。我们使用这些引物首先筛选了F7、FAP1360A、Zheng58、Chang7-2、HuangC和X178之间的多态性。大多数引物对由于扩增不可靠而被丢弃。利用6对具有多态产物的引物对对这94个自交系进行扩增,以鉴定潜在的多态位点。在这6个多态扩增子中,2个和3个位于CAS1-like-1而且Zmtrx-h,而周围没有引物对CAS1-like-2是可用的。核苷酸序列比对使用多序列比对软件MUSCLE [50],然后手工提炼。Q由STRUCTURE 2.2推断[51,52]有70个SSR标记。进行了五次独立的试验,设置种群数量(k)从1到10,燃烧时间和MCMC(马尔科夫链蒙特卡罗)复制数均为50万,并建立了混合和相关等位基因频率的模型。k值由STRUCTURE输出中的LnP(D)和一个基于LnP(D)在连续k之间的变化率的临时统计量Δk确定。隶属概率≥0.75的线被分配到相应的簇;隶属概率< 0.75的系被分配到混合组。使用类似的方法将组进一步细分为子组。用最符合育种家关于系谱知识的运行来将品系分配到集群中。使用TASSEL2.0计算两个位点之间的LD水平[53].在TASSEL2.0中,利用GLM分析了小等位基因频率(MAF)≥0.05的多态位点与SCMV抗性的相关性。
缩写
- BAC:
-
细菌人工染色体
- BSR:
-
广谱抗
- 中科院:
-
Cycloartenol synthase1-like
- 走:
-
基因本体论
- 凯西:
-
亲属关系
- KEGG:
-
京都基因和基因组百科全书
- LD:
-
连锁不平衡
- 加:
-
小等位基因频率
- JGMV:
-
约翰逊草花叶病毒
- 全球语言监测机构:
-
一般线性模型
- 聚合酶链反应:
-
聚合酶链反应
- 问:
-
人口结构
- 奶油水果蛋白饼:
-
存在/没有变化
- QTL:
-
数量性状位点
- 瑞来斯:
-
重组自交系
- SCMV:
-
甘蔗花叶病毒
- SNP:
-
单核苷酸多态性
- SrMV:
-
高粱花叶病毒
- 苏维埃社会主义共和国:
-
简单序列重复
- WSMV:
-
小麦条纹花叶病毒
- ZeMV:
-
玉米花叶病毒。
参考文献
- 1.
Fuchs E, gruntzzig M:甘蔗花叶病毒(SCMV)和玉米矮花叶病毒(MDMV)对2个玉米品种生长和产量的影响。中国植物病害学报,2004,22(1):1 -5。
- 2.
王志辉,李晓红,袁丽霞,韩晓青,张世生:玉米抗SCMV优良种质的遗传多样性。作物学报,2003,29(3):391-396。
- 3.
波兰首次报告甘蔗花叶病毒感染玉米。植物保护学报,2009,29(3):344 - 344。
- 4.
Marie-Jeanne V, Hariri D, Doucet R, Signoret PA:法国中部地区玉米上甘蔗花叶病毒的首次报告。植物学报,2011,95(1):70-70。
- 5.
Shukla DD, Ward CW:基于衣蛋白序列数据和血清学的poty病毒的鉴定和分类。中国寄生虫病杂志,1998,26(4):344 - 344。
- 6.
陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明,等:早熟欧洲玉米种质资源对甘蔗花叶病毒和玉米矮花叶病毒的抗性分析。植物遗传资源学报,1997,26(5):379 - 379。10.1111 / j.1439-0523.1997.tb01038.x。
- 7.
Seifers DL, Salomon R, Marie-Jeanne V, Alliot B, Signoret P, Haber S, Loboda A, Ens W, She YM, Standing KG:从以色列玉米中分离出的一种新型potyvirus的特征。植物病理学杂志,2000,20(5):357 - 357。10.1094 / PHYTO.2000.90.5.505。
- 8.
吴建勇,丁建强,杜友友,徐亚波,张晓春:玉米抗甘蔗花叶病毒两个显性互补基因的遗传分析和分子定位。自然科学进展,2007,26(3):355-364。10.1007 / s10681 - 007 - 9384 - 8。
- 9.
刘志刚,刘志刚,王志刚:玉米品种对玉米矮花叶病毒和玉米绿斑矮病毒的反应。作物科学,1990,30(6):1210-1215。10.2135 / cropsci1990.0011183X003000060010x。
- 10.
刘赵江H,白Y, Y, W,江H,白YJ,赵YM,刘WZ:研究评价抗性的玉米(玉米)种质菌株B的玉米矮花叶病毒在中国东北。玉米科学,2002,10(1):84-87。
- 11.
梅青格,李志刚,李志刚,李志刚,李志刚。欧洲玉米种质抗甘蔗花叶病毒的遗传基础。应用理论,1998,96(8):1151-1161。10.1007 / s001220050851。
- 12.
夏晓春,杨晓明,杨晓明,杨晓明,杨晓明。玉米抗甘蔗花叶病毒的数量性状定位。植物病理学杂志,1999,29(3):366 - 366。10.1094 / PHYTO.1999.89.8.660。
- 13.
徐明明,夏晓春,刘志军,刘志军,等:利用RFLP、SSR和AFLP标记对玉米抗甘蔗花叶病毒基因座进行高分辨率定位。中国生物医学工程学报,1999,26(3):574-581。10.1007 / s004380051003。
- 14.
张双生,李晓红,王志辉,George ML, Jeffers D,王福飞,刘晓东,李明松,袁丽霞:中国玉米种质抗SCMV的QTL定位。学报学报,2003,48(4):307-312。
- 15.
Prazeres De Souza IR, Schuelter AR, Guimaraes CT, Schuster I, De Oliveira E, Redinbaugh M:热带玉米抗SCMV QTL定位。遗传学报,2008,145(4):167-173。10.1111 / j.0018-0661.2008.02006.x。
- 16.
刘小红,谭志斌,荣志涛:玉米抗SCMV主要QTL的分子定位。中国生态学报,2009,37(2):379 - 379。10.1007 / s10681 - 008 - 9874 - 3。
- 17.
邢永忠,王志刚,王志刚,王志刚,王志刚:玉米抗病毒基因Scmv2的遗传和物理精细定位。应用理论,2010,20(8):1621-1634。10.1007 / s00122 - 010 - 1281 - 1。
- 18.
Dussle CM, Quint M, Melchinger AE, Xu ML, Lubberstedt T:利用SSR和AFLP标记对牛血清白蛋白对SCMV抗性的两个染色体区域的饱和。应用理论与实践,2003,26(3):485-493。
- 19.
袁lx, Dussle CM, Melchinger AE, Utz HF, Lubberstedt T:玉米抗SCMV QTL的聚类分析。学报学报,2003,48(1):55-62。
- 20.
王志刚,王志刚,王志刚:玉米抗病性遗传结构的研究进展。植物病理学杂志,2006,36(2):344 - 344。10.1094 /发朵- 96 - 0120。
- 21.
杜斯乐,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明,杨晓明Scm1而且Scm2,两个主要QTL有助于玉米抗SCMV。植物学报,2000,29(4):389 - 397。10.1046 / j.1439-0523.2000.00509.x。
- 22.
袁lx, Dussle CM, Muminovic J, Melchinger AE, Lubberstedt T: BSA的靶向定位Scmv1而且Scmv2赋予抗SCMV使用PstI / MseI相比之下,EcoRI / MseI妊娠标记。植物遗传资源学报,2004,29(5):344 - 344。10.1111 / j.1439-0523.2004.00966.x。
- 23.
吴伟,叶婷婷,吴玮,Richmond T, Kitzman J, Rosenbaum H,等:玉米自交系基因组含量存在大量拷贝数变异(CNV)和存在/缺失变异(PAV)。公共科学学报,2009,5 (11):e1000734-10.1371/journal.pgen.1000734。
- 24.
孟建平,李志刚,李志刚:植物遗传研究中的新型通量表型平台。植物科学进展,2007,12(10):433-436。10.1016 / j.tplants.2007.08.006。
- 25.
Myles S, Peiffer J, Brown PJ, Ersoz ES, Zhang ZW, Costich DE, Buckler ES:关联映射:从基因分型到实验设计的关键因素转移。植物细胞学报,2009,21(8):2194-2202。10.1105 / tpc.109.068437。
- 26.
Rafalski JA:作物改良中的关联遗传学。植物学报,2010,13(2):174-180。10.1016 / j.pbi.2009.12.004。
- 27.
闫建平,王志强,王志强,等。关联作图技术在玉米遗传改良中的应用。作物科学,2011,51(2):433-449。10.2135 / cropsci2010.04.0233。
- 28.
邢永忠,王晓明,杨晓明,王晓明,等:玉米花叶病毒抗性分析及其对玉米花叶病毒抗性的影响。中国生物工程学报,2006,49(10):1274-1282。10.1139 / g06 - 070。
- 29.
植物qtl克隆与不克隆:当前与未来的挑战。植物科学进展,2005,10(6):297-304。10.1016 / j.tplants.2005.04.008。
- 30.
赖建军,李仁强,徐旭,金文文,徐明明,赵海恩,项志科,宋文文,应坤,张明,焦亚平,倪鹏平,张建刚,李东,郭新新,叶克新,简明,王波,郑海辉,梁海强,张晓强,王素生,陈世杰,李建军,付勇:玉米优良自交系遗传变异的全基因组模式。植物学报,2010,42 (11):1027-U1158。10.1038 / ng.684。
- 31.
Babiychuk E, bouier - nave P, Compagnon V, Suzuki M, Muranaka T, Van Montagu M, Kushnir S, Schaller H:拟南芥环artenol合成酶1在细胞活力和质体生物发生中的独特功能美国国家科学院。2008, 35(8): 366 - 366。10.1073 / pnas.0712190105。
- 32.
李志刚,张志刚,张志刚,等。谷甾醇转化为豆甾醇在植物-病原菌相互作用中的作用。植物学报,2010,32(2):344 - 344。10.1111 / j.1365 - 313 x.2010.04235.x。
- 33.
Sindhu A, Chintamanani S, Brandt AS, Zanis M, Scofield SR, Johal GS:草的守护者:草系中一种独特抗病基因的特定起源和保护。美国国家科学院。2008, 35(5): 457 - 457。10.1073 / pnas.0711406105。
- 34.
Chintamanani S, Multani DS, Ruess H, Johal GS:玉米的剂量依赖性和发育调控抗性具有独特的机制款Hm2基因。植物微生物学报,2008,21(1):79-86。10.1094 / mpmi - 21 - 1 - 0079。
- 35.
Tada Y, Spoel SH, Pajerowska-Mukhtar K,牟志林,宋建强,王超,左小雷,董晓宁:植物免疫需要构象电荷NPR1通过s -亚硝基化和硫氧还蛋白。科学通报,2008,29(4):344 - 344。10.1126 / science.1156970。
- 36.
孙丽娟,任海燕,刘瑞霞,李蓓,吴tq,孙芳,刘红梅,王晓明,董海生:h型硫氧还蛋白在烟草对两种病毒和非生物氧化应激防御反应中的作用。Mol植物微生物,2010,23(11):1470-1485。10.1094 / mpmi - 01 - 10 - 0029。
- 37.
Sweat TA, Wolpert TJ:硫氧还蛋白h5是拟南芥CC-NBS-LRR基因介导的维克托林敏感性所必需的。植物细胞学报,2007,19(2):673-687。10.1105 / tpc.106.047563。
- 38.
陆玉玲,张士生,沙涛,谢彩霞,郝泽峰,李晓辉,Farkhari M, Ribaut JM,曹敏,荣志涛,徐亚波:联合连锁-连锁不平衡作图是检测玉米耐旱性数量性状位点的有效方法。美国国家科学院。2010, 107(45): 195 -195。10.1073 / pnas.1006105107。
- 39.
田飞,Bradbury PJ, Brown PJ, Hung H, Sun Q, Flint-Garcia S, Rocheford TR, McMullen MD, Holland JB, Buckler ES:玉米巢状关联定位群体叶片结构的全基因组关联研究。植物学报,2011,43 (2):159-U113。10.1038 / ng.746。
- 40.
李林,李华,李强,杨晓辉,郑德宝,Warburton M,柴玉春,张鹏,郭永强,严建彪,李建军:11-bp插入玉米脂肪降低了玉米籽粒脂肪酸中棕榈酸的含量。公共科学学报,2011,6 (9):e24699-10.1371/journal.pone.0024699。
- 41.
孙倩,孙强,李志刚,李志刚,李志刚:水稻基因组中与广谱定量抗病性相关区域的鉴定与鉴定。中国生物医学工程学报,2005,26(4):379 - 379。10.1534 / genetics.104.036327。
- 42.
Krattinger SG, Lagudah ES, Spielmeyer W, Singh RP, Huerta-Espino J, McFadden H, Bossolini E, Selter LL, Keller B:一种假定的ABC转运蛋白赋予小麦对多种真菌病原体的持久抗性。科学通报,2009,29(5):528 - 528。10.1126 / science.1166453。
- 43.
Kump KL, Bradbury PJ, Wisser RJ, Buckler ES, Belcher AR, Oropeza-Rosas MA, Zwonitzer JC, Kresovich S, McMullen MD, Ware D,等:玉米巢状关联定位群体对南叶枯病定量抗性的全基因组关联研究。植物学报,2011,43 (2):163-U120。10.1038 / ng.747。
- 44.
王晓明,王晓明,王晓明,王晓明,等:3玉米近交系PA405抗小麦条纹花叶病毒的遗传位点分析。植物微生物学报,1994,7(6):708-712。10.1094 / mpmi - 7 - 0708。
- 45.
琼斯MW,雷丁堡MG,路易RMdm1玉米矮花叶病毒抗性基因座与玉米。中国植物学报,2007,29(2):379 - 379。10.1094 / pdi - 91 - 2 - 0185。
- 46.
寇玉军,王素萍:广谱与持久性:对定量抗病的理解。植物学报,2010,13(2):181-185。10.1016 / j.pbi.2009.12.010。
- 47.
Murray MG, Thompson WF:高分子量植物DNA的快速分离。中国生物医学工程学报,2000,8(19):4321-4325。10.1093 / nar / 8.19.4321。
- 48.
杨强,殷格明,郭玉玲,张东峰,陈世杰,徐明兰:玉米抗赤霉病的一个主要QTL。应用物理学报,2010,41(4):673-687。10.1007 / s00122 - 010 - 1339 - 0。
- 49.
李强,万建民:SSRHunter: SSR位点本地搜索软件的开发。hereditas。2005,27(5): 808-810。
- 50.
Edgar RC: MUSCLE:多序列比对,高精度,高通量。中国生物医学工程学报,2004,32(5):1792-1797。10.1093 / nar / gkh340。
- 51.
李志强,李志强,李志强,等。基于多位点基因型数据的种群结构推断。遗传学报,2000,15(2):945-959。
- 52.
王晓明,王晓明,王晓明,等:基于多位点基因型数据的群体结构分析。中国生物医学工程学报,2003,34(4):357 - 357。
- 53.
张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚,张志刚。生物信息学,2007,23(19):2633-2635。10.1093 /生物信息学/ btm308。
确认
国家自然科学基金(31371625)、农业部(2011ZX08009-003-001)、国家高新技术发展计划(2012AA10A306、2012AA101104)资助。我们感谢中国农业大学杨晓红博士对本研究中关联分析的重要指导。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
LJ、QL、CRI和UKF在分离种群时进行了映射实验。BW和JL提供了2个RIL群体。YT, QL,在这两个RIL群体中进行了精细定位。YZ、RZ和YT完成了关联映射工作。MX和TL监督了研究,设计了实验并参与了数据分析。YT撰写稿件初稿,MX和TL编辑修改稿件。所有作者都阅读并批准了最终的手稿。
陶永福、蒋璐、刘青青对这项工作也有同样的贡献。
电子辅助材料
用于测绘的公共SSR标记列表
附加文件1:Scmv1。(docx15kb)
12870 _2013_1372_moesm2_esm.docx
附加文件2:94个玉米自交系及其抗SCMVR性能表示抗SCMV, S表示易感SCMV。(docx18kb)
12870 _2013_1372_moesm3_esm.docx
附加资料3:利用70个ssr估计的94个玉米自交系的群体结构。采用structure方法评估人口结构。每个个体用竖条表示,分割成彩色片段,每个片段的长度代表个体基因组在k = 6组中的比例。对于所有类,一个给定的组表示为:red, REID;蓝色、P;绿色,局域网;黄色,TSPT;粉色,Z330;青绿色,其他。(docx86 kb)
SCMV抗性与多形性位点之间的关系
附加文件4:Scmv1基因组区域。a:使用GLM计算的p值。#:与SCMV抗性相关的多态位点。(docx16kb)
开发的程序
附加文件5:Scmv1人口的映射。(docx29 kb)
12870 _2013_1372_moesm7_esm.docx
附加文件7:为关联映射设计的引物。*:这些引物扩增的PCR产物含有多态位点,可用于关联作图。(docx15kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
陶勇,蒋,李,刘,Q。et al。结合链接和关联映射显示了候选的Scmv1这是玉米抗甘蔗花叶病毒(SCMV)的主要基因座。BMC植物生物学13,162(2013)。https://doi.org/10.1186/1471-2229-13-162
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-162
关键字
- 玉米
- SCMV
- QTL
- 精细定位
- 关联映射