摘要
背景
成熟日期是新鲜水果销售的重要因素,特别是那些保质期有限的水果,例如桃子(碧桃L. Batsch):选择几个MD不同的品种将有利于覆盖和延长销售季节。这项工作的目的是对先前在桃连锁群4上发现的主要成熟日期位点的候选基因进行精细定位和鉴定。为了提高目标基因座2f的遗传分辨率2分别用Sequenom和9K Illumina Peach Chip SNP平台对来自竞争者x Ambra (CxA, 306个个体)和PI91459 (NJ Weeping) x Bounty (WxBy, 103个个体)的群体进行基因分型。
结果
重组个体来自WxBy F2群体将成熟日期位点定位到桃基因组的220kb区域。在这个区间内的25个被注释的基因中,功能分类鉴定出ppa00757m和ppa008301m是最有可能的候选基因,它们都编码NAC的转录因子(NAM /一个TAF1 2 /CUC2)的家庭。对四个亲本进行重测序并与参考基因组序列进行比较,发现ppa007577m上游区域缺失232 bp,该区域在NJ中为纯合子,在Ambra、Bounty和WxBy F中为杂合子1的父母。然而,这种变异并没有在cxaf中分离2人口是美国联邦调查局1参考等位基因为亲本纯合。因此,第二个基因被检查为成熟日期的候选。对ppa008301m进行重测序,发现最后一个外显子在CxA和WxBy F中与成熟日位点共分离的帧内插入9bp2人群。
结论
利用两个不同的分离群体,将成熟日期位点的图谱位置从3.56 Mb细化到220 kb。NAC基因ppa008301m的序列变异与成熟日期位点共分离,表明该基因是控制桃子成熟时间的候选基因。如果在其他遗传材料上得到证实,该变异体可用于不同成熟期品种的标记辅助育种。
背景
果实成熟是一个复杂的过程,涉及许多代谢途径的协调调节,影响许多性状,如色、香、味。在成熟过程中,果实组织的软化与糖、酸和挥发性化合物的积累同时发生。总之,这些特征有助于增加可口性。其他特征,如颜色、大小和到期日,都被选择来提供提高消费者鉴赏力的产品。由于实际原因,水果在生理成熟之前就被收获,因此经常被消费者认为质量差。因此,现代桃子育种所追求的理想特征是提高果实质量,使其易于收获(果实质地更紧实或软化速度很慢),以便在消费时达到最佳果实质量。果实成熟度是一项重要的性状,提前成熟可以通过延长生产季节的长度来促进市场增长。对多种植物物种的研究已经提供了对介导果实成熟相关过程的遗传机制的见解,如色素合成、细胞壁和糖代谢[qh]1- - - - - -4]。
更年期水果如桃子的特点是呼吸增加和乙烯生产的自催化爆发在果实发育后期,这是正常果实成熟所必需的。阻止乙烯合成或感知阻止成熟[5- - - - - -7]。一些番茄突变体(如:rin,也不,中国北车)即使在外源乙烯的作用下也不能成熟,尽管在激素信号转导途径中没有受损[8];以及其中的参考文献]。这些突变体的分子分析和相应基因的鉴定表明,它们是成熟过程中乙烯依赖基因和非依赖基因表达所必需的,作用于乙烯的上游,也可能与乙烯平行[8]。在香蕉(另一种更年期水果)中,NAC转录因子已被证明与乙烯信号的下游组分乙烯不敏感3 (EIN3)样蛋白相互作用,该蛋白在成熟过程中下调[9]。因此,NAC TFs对成熟的控制与激素调控一样重要。
在桃中,控制MD的qtl被定位在不同的染色体上,主要的qtl位于连锁群(LG) 4和6上[10- - - - - -13]。通过对竞争者x Ambra (CxA) F2我们已经证明,在LG4上检测到的QTL表现为孟德尔性状,具有多效性效应,掩盖了不同果实性状的QTL效应[10]。在目前的工作中,我们把这个轨迹称为qMD4.1。最近,在杏的共线区也发现了一个主要的QTL。p . armeniacaL.)和甜樱桃(p .鸟结核L.)表明可能有一种共同的机制控制着果实的成熟李属物种(13]。
在本研究中,qMD4.1为进一步了解桃MD的控制机制,并更好地了解该位点与其他果实性状的遗传相互作用,对该位点进行了遗传解剖。为此,绘制了精细的地图qMD4.1构建基因座,增加CxA群体的标记和个体数量,并分析另一个F2在这个群体中,MD作为一种孟德尔特征正在分离。分析MD QTL候选基因所在区域,并扫描公开获得的CxA F重测序数据1亲本在窝藏区域发现多态性qMD4.1[14]。在这两个群体中,NAC候选基因的一个序列变异被证明与MD性状共分离,可用于早/晚成熟基因型的选择。
结果与讨论
精细映射qMD4.1轨迹
本研究的目的是绘制精细地图qMD4.1即Eduardo描述的MD位点等。[10]在CxA F的LG4上2人口。利用169个个体和31个ssr位点和2个表型标记构建了该位点的遗传图谱,其中侧翼标记M12a和EPPISF032定义了6.6 cM的间隔,对应于桃树参考基因组的3.56 Mb。为了精细地图qMD4.1,该群体的个体数增加到306个,在原图谱区间周围和区间内增加了14个snp。这些snp是人工从公开的CxA F基因组重测序数据中选择的1自花授粉亲本(NCBI Sequence Read Archive,生物样本SRS335631)。同时,我们还分析了另一个F2PI91459 (N.J. Weeping)杂交得到的103个群体;W)和Bounty (By),也对MD进行了分离,并使用9K SNP桃阵列v1进行了基因分型[15(达席尔瓦)等,未发表)。
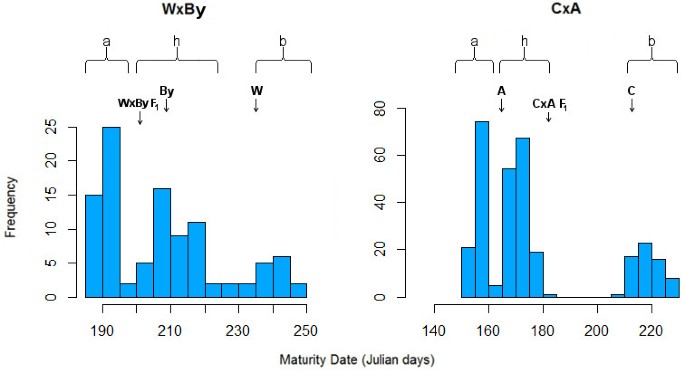
在这两个人群中,MD呈三峰分布(图2)1),年份之间的相关性非常高(0.92)1).这种分布表明了一种孟德尔行为。因此,对MD进行了全基因组QTL分析,以独立使用两个群体进行确认,目的是排除其他MD QTL的可能分离(数据未显示)。尽管在CxA和WxBy F中分离是一个孟德尔特征2在其他种群中,MD表现为数量性状,如F1由Bolero和OroA杂交而来的种群[10]。
数字2显示了qMD4.1在CxA和WxBy这两张地图上,与Eduardo等。[10]。在两幅图中,在目标区域观察到高度的隔离畸变(表1)2).等位基因分析表明,低代表等位基因分别来自CxA的C基因组和WxBy的W基因组,均与晚熟等位基因相对应。
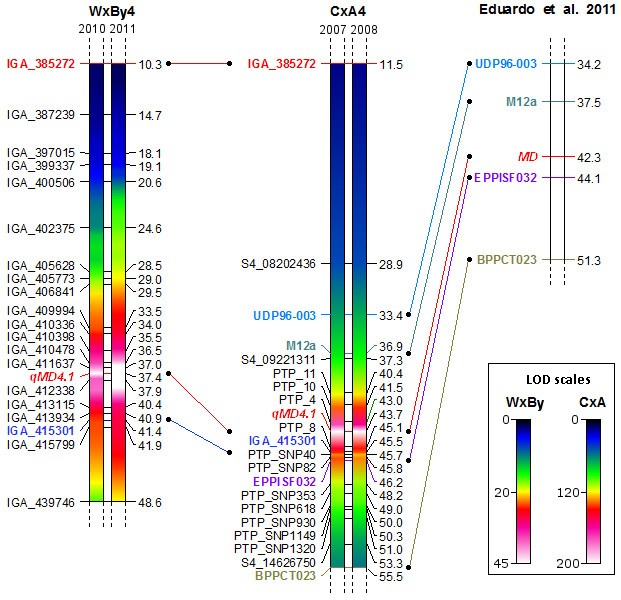
WxBy与CxA的连锁群4的图谱比较,以及Eduardo已发表的CxA的SSR图谱等。[[10]]。彩色线表示WxBy和CxA之间的共同标记(snp)和本研究的CxA与Eduardo的CxA图谱等。的位置qMD4.1在两个研究群体(WxBy为2010年和2011年;(a)按区间映射确定为2007年和2008年。标记名称列在左侧,遗传距离(cM)列在右侧。qtl以对应各自LOD尺度的颜色梯度表示(见右下角),并由Harry Plotter软件(http://genomics.research.iasma.it/download.html).
为了确定候选基因分析的目标区域并固定两个图谱,我们使用Sequenom平台对CxA F进行基因分型2同时包含在9K SNP桃阵列v1中的2个SNP(即SNP_IGA_385272和SNP_IGA_415301)的后代;数字2).
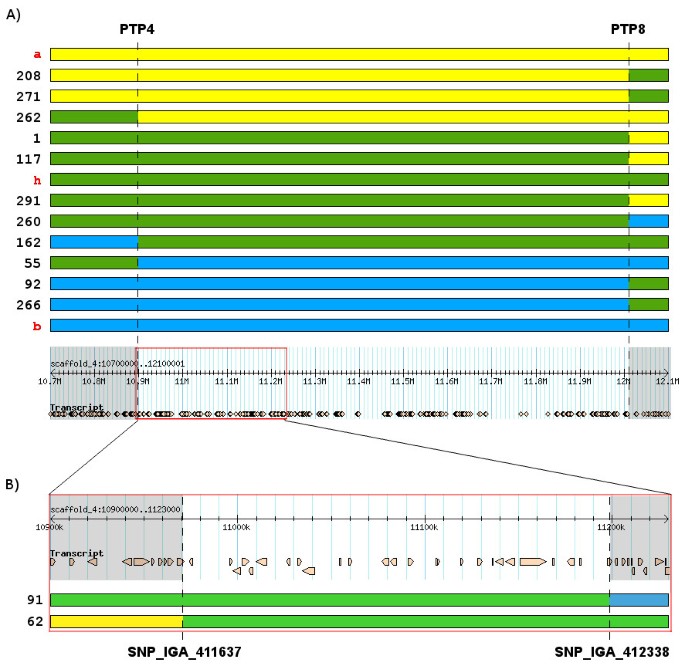
考虑到两个群体中锚点标记SNP_IGA_385272和SNP_IGA_415301之间的区域,在CxA图谱中,该区域跨度为33.99 cM,包含2个ssr和6个SNPs,而在WxBy精细图谱中,该区域包含16个SNPs,跨度为31.12 cM。来自WxBy的完整图谱将在其他地方描述,并在这里用于对MD性状进行QTL分析,显示该群体中没有其他与MD分离的QTL(数据未显示)。精细图谱分析表明,在CxA中,qMD4.1位于标记PTP4和PTP8之间的2.13 cM窗口,横跨10.87-12.09 Mb的染色体区域,而在WxBy图谱中qMD4.1位于标记SNP_IGA_411637和SNP_IGA_412338之间0.98 cM的窗口,跨越10.97-11.19 Mb的染色体区域(表2).此外,在CxA后代中,LOD评分为qMD4.1区域达到220以上(占解释方差的97%),而在WxBy中LOD得分约为50,占解释方差的90%以上(表1)2).因此,这是我们重点寻找候选基因的区域。
尽管CxA F2种群有更多的个体,WxBy F2群体具有较多的分离基因型标记,这为定位提供了较好的分辨率qMD4.1(图3.).这些结果显示了新的基因组工具的实用性,例如9K SNP桃阵列v1 [15],以提高桃制图研究的效率和分辨率。此外,使用WxBy F2种群使我们能够确定一个相对较小的区域,与CxA - F相比,候选基因数量有限2人口(图3.).
区域中MD的qtl与qMD4.1已经被其他组织确认了[11,16]。比如,德勒万格等。[13在LG4上用F标记了一个MD的主要QTL2由Ferjalou Jalousia和Fantasia (JxF)杂交得来的种群:基于现有数据,qMD4.1SSRs UDP97-402和AMPA 103在JxF中定义了该区域,分别位于支架4的10,486,180和13,509,355位置(相距约3.02 Mb),似乎与不同年份的置信区间重叠。与孟德尔的行为相反qMD4.1在我们的人群中,在这些先前的研究中,MD作为一个数量性状分离出来,qtl定位在不同的染色体上。有趣的是,这些研究表明,区域跨越qMD4.1承载了一组与其他性状相关的qtl,如果糖和蔗糖含量、可滴定酸度、pH、山梨醇、麦芽糖、柠檬酸和奎宁酸含量[11]。最近,一项关联作图研究表明,在4号染色体起始位置与SSR标记CPPCT028相关的另一个位点(2.1 Mb)与MD性状的变异密切相关[17]。这表明其他控制MD的基因座可以通过不同的分离群体或关联图谱来识别。此外,本研究还发现MD、SSC、黄酮类化合物、山梨醇和总糖水平之间存在很强的相关性[17]。
候选基因的鉴定与分析
基于重叠的地图位置qMD4.1在CxA和WxBy F的位点2在种群中,我们的假设是同一基因的等位基因变异可以解释两个种群中MD性状的分离。因此,我们在两个WxBy重组体鉴定的10.97-11.19 Mb区域内寻找基因组变异(图2)3.).在桃基因组的这一区域有25个基因被注释(表2)3.),我们将注意力集中在ppa007577m和ppa008301m上,这两个基因都被预测编码NAC转录因子(tf)。NACs是最大的植物TF家族之一,是发育程序和胁迫反应的关键调节因子[18- - - - - -20.],以及其中的参考文献]。首先,我们Sanger对四个亲本C、A、W和ppa007577m进行了重新测序,试图确定该基因内部的多态性,以及1.5 kb的上下游侧翼序列。序列分析未发现编码区内存在多态性,但与参考基因组(Lovell)相比,在起始密码子上游579 bp处发现了232 bp的缺失。该区域的PCR扩增结果显示,C为参考Lovell等位基因纯合,W为缺失等位基因纯合,而A和By均为杂合(数据未显示)。此外,对CxA F1结果表明,该个体与参考Lovell等位基因(C单倍型)纯合。因此,不期望在F2CxA人口。为了进一步验证这一假设,我们筛选了CxA F的三个表型类别(早、中、晚成熟)的一些个体,对这一缺失进行了检验2人口。正如父母分析所预期的那样,在所分析的个体中不存在232 bp的缺失。相比之下,在WxBy F的选定个体中,232 bp的缺失与表型共分离2人口是F1亲本杂合在此位点。总之,这些结果促使我们寻找其他的MD性状候选基因,因为ppa007577m在两个种群中都不与MD性状分离。
因此,我们将注意力集中在第二个NAC候选基因ppa008301m上。桑格重排序的四个父母和两个F1个体发现了一个帧内9 bp的插入(与参考基因组相比),导致c端结构域的三个氨基酸串联重复(图2)4).A、C和By是杂合的,而W是纯合的,没有9bp的插入。CxA和WxBy F1该多态性为杂合子,进一步分析了F2通过个体分析验证其与MD性状的正确分离。为此,使用用于微卫星分析的基因分型方案对9bp插入/缺失(INDEL)进行评分(数据未显示)。等位基因变异与两个群体中所有个体的MD共分离2早熟个体显示9bp插入,晚熟个体携带Lovell参考等位基因(附加文件)1).为了进一步验证该图谱的位置,我们将这些数据添加到之前的基因分型数据集中,用于图谱构建和QTL分析。我们发现这个INDEL与MD表型标记共定位(附加文件)2).最后,在两个群体中重复QTL分析,发现该INDEL与qMD4.1(附加文件2).
进一步证实在NAC基因ppa008301m的最后一个外显子中发现的9bp INDEL是最可能的多态性qMD4.1区域,我们检索了公开可获得的CxA F的成对端全基因组下一代测序数据1原始目标区域内序列变异的父序列。表格4报告此分析的结果。大多数已鉴定的多态性位于基因间区。在编码区内或附近仅发现两个多态性:第一个多态性位于β-葡萄糖苷酶18基因(ppa025660m)的5 ' -UTR,参与碳水化合物和苯丙素代谢,第二个多态性位于NAC候选基因ppa008301m的9 bp插入。即使基因间区域的多态性可以顺式调节靶基因的表达,但该NAC候选基因编码序列中9bp插入的存在支持了NAC TF ppa008301m是CxA和WxBy群体中控制MD的强候选基因的假设。
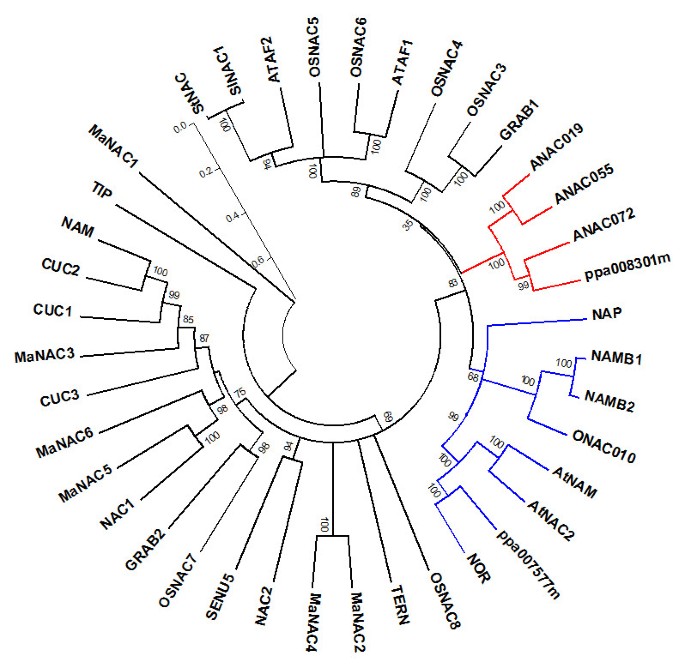
中NACs的系统发育分析拟南芥在水稻、小麦、马铃薯、香蕉和番茄中,NAC基因ppa00757m和ppa008301m属于两个不同的进化枝(图2)5).NAC ppa007577m与已知参与果实成熟的番茄NOR(非成熟)聚类(GeneBank登录号为AY573802)。首先由Giovannoni在10号染色体上绘制等。[21),也不基因似乎对果实成熟有全局影响,可能作用于乙烯合成的上游[22]。相关的拟南芥基因AtNAC2和AtNAM(即NARS1和NARS2,分别)(图5)通过调节胚珠被毛的发育和退化来控制胚胎发生[23]。ppa007577m的进化支还包括水稻ONAC10和小麦NAC TFs NAM_B1和NAM_B2(图2)5).NAM-B1和NAM-B2已被证明参与延迟种子成熟,并对籽粒蛋白质、锌和铁含量产生多效性影响[j]。24]。的功能分析ONAC10最初被作者选为与小麦NAM_B1最相似的水稻NAC TF,排除了在种子成熟中的作用,但表明它对正确的花药发育至关重要[20.]。总的来说,来自ppa00757.7枝的基因似乎经历了功能多样化,在不同的生物过程中发挥作用。NAC TF支系ppa008301m中主要参与应激反应的基因为拟南芥ANAC072、ANAC055及ANAC019 [25]。最近,研究表明,密切相关的ANAC019和ANAC055在发育性衰老过程中发挥着不同的作用[26]。事实上,ANAC019可能通过控制和激活类黄酮和花青素的生物合成参与触发衰老,而ANAC055似乎参与了对几丁质的反应[26]。此外,这些基因在茉莉酸和水杨酸途径的调控中显示出相反的作用[26]。不幸的是,目前关于NAC TFs参与成熟的文献非常少。在香蕉中,四种不同成熟特征果实的基因表达谱显示MaNAC果皮和果肉成熟过程中基因表达差异[9]。此外,MaNAC1和MaNAC2与mail5相互作用(参与乙烯信号传导),表明这些TFs可能通过与乙烯信号传导成分相互作用参与香蕉果实成熟。
桃树NAC TF ppa007577m和ppa008301m与其他品种NAC TF的系统发育分析。基因名称和UniProtKB/Swiss-Prot加入号如下。栽培稻: OsNAC3, OsNAC4, OsNAC5, OsNAC6, OsNAC7, OsNAC8, ONAC010;答:芥: ATAF1、ATAF2、NAP、CUC1、CUC2、CUC3、AtNAC2、NAC1、NAC2、AtNAM、TIP;Anac019, anac055, anac072;佩妮矮牵牛:南;茄属植物lycopersicum: NOR, SlNAC1;茄属植物tuberosum: StNAC1;Lycopersicon esculentum: SENU5;烟草:燕鸥;小麦属植物sp.: grab1, grab2, nam-b1, nam-b2;穆萨acuminata: MaNAC1, MaNAC2, MaNAC3, MaNAC4, MaNAC5, MaNAC6。红色和蓝色分支分别表示ppa008301m和ppa007577m两个聚类组。分支上的数字表示引导值。细条表示刻度。所选蛋白序列的加入编号见方法。
我们的研究结果也支持ppa008301m参与桃子果实成熟的控制,这表明该支系的基因可能已经进化到实现ppa00757m支系的不同生物学功能。虽然同源物通常被认为在不同的生物体中保持相同的功能,但偏离这种情况并不罕见[9,27]。nac是最大的TF家族之一,每个成员的精确功能指示需要实验验证。候选基因ppa008301m的9 bp INDEL导致苏氨酸-天冬氨酸-脯氨酸延伸的重复,可能对蛋白质功能产生影响。简单的氨基酸重复序列和富含丝氨酸和苏氨酸、脯氨酸和谷氨酰胺的区域,或酸性残基在NAC蛋白的c端区域是常见的[28- - - - - -33]。虽然很少有研究关注NAC翻译后修饰,但番茄SlNAC1 c端区域赖氨酸268和269对其降解有辅助作用,而用两种天冬氨酸取代赖氨酸残基可以显著稳定SlNAC1 c端蛋白序列[34]。此外,几种NAC蛋白的c端区域作为转录激活域起作用[19];以及其中的参考文献]。对域进行删除分析拟南芥NAC基因ATAF1的研究表明,该反激活活性是由c端结构域赋予的,而不需要n端结构域[35]。因此,ppa008301m中的三个氨基酸INDEL可能通过修饰c端结构域结构,影响蛋白的稳定性或其转录激活能力,从而影响靶基因的转录激活。
有趣的是,我们发现在两个种群中,早熟个体在蛋白质序列中具有三个氨基酸插入,而延迟成熟则与参考等位基因相关。
结论
在本研究中,我们报告了在两个不同的群体中,桃子成熟日期性状的一个主要控制位点的精细定位。该策略使我们能够将先前描述的位点从3.56 Mb限制到0.22 Mb。NAC基因ppa008301m被确定为该区域的强候选基因,并且在其最后外显子中插入一个一致的9 bp的插入被认为是可能导致早熟的变异。该INDEL的功能意义还需要进一步的实验来探索。在任何情况下,基于该序列多态性开发的标记为区分CxA、WxBy和可能的其他育种群体中的早熟和晚熟个体提供了方便的分子工具。考虑到提出的多效性效应qMD4.1在其他重要的果实性状上,我们的研究结果为更好地确定果实品质改良的育种方案提供了依据。
方法
植物材料和表型
两个F2本研究使用的群体是:306个个体来自于竞争者(C)和Ambra (a)的杂交群体,另一个103个个体来自于N.J. Weeping (W)和Bounty (By)的杂交群体。树木位于意大利艾米利亚罗马涅城堡和特巴诺的ASTRA果园。CxA和WxBy子代树木在各自的根上种植,行内间距为1 m,行间间距为4 m,训练成细长纺锤(一茎,短侧支架)。每年进行修剪,并采用标准的栽培方法。果实在果核硬化前被削薄,根据活力每棵树只有30-40个果实,以便在不受竞争限制的情况下充分表现果实大小。
根据视觉颜色变化和手工硬度评估,每棵树在商业成熟时收获20个果实,并记录日期。在2007-2008年和2010-2011年分别获得了CxA和WxBy的成熟日(MD)表型。MD是用收获时的儒略日来定义的。
DNA提取和基因分型
采用dnasy 96 Plant Kit (QIAGEN)提取幼叶DNA。微卫星(SSR)扩增是按照多重准备PCR协议进行的[10]。
CxA人群的SNP选择、设计和基因分型
对于CxA F2人口,根据Eduardo确定的置信区间等。[10],对306个个体进行了精细定位,其中16个新开发的snp分布在LG4的9-12 Mb区间。假设的SNPs是人工从F基因测序中选择的1CxA和相关的周围序列(300 bp)从IGA网站(http://www.appliedgenomics.org/).使用Mass ARRAY Assay Design 3.1软件设计包含选定snp的多重反应。使用iPLEX Gold技术进行基因分型[36和Mass ARRAY高通量DNA分析质谱法(Sequenom, Inc)。所有选定的14个snp都提供了高质量的结果,并用于进一步分析。关于用于基因分型的引物的信息在附加文件中列出3.。
WxBy群体的SNP选择、设计和基因分型
WxBy F的103个人2利用新近开发的Illumina 9000 SNP阵列v1对桃进行基因分型。15]。采用dnasy 96 Plant kit (Qiagen)提取DNA,稀释至50 ng/μl,送至IASMA研究与创新中心(San Michele all’adige, Italy)进行基因分型。按照Verde中描述的制造商建议进行基因分型等。[15]。使用GenomeStudio数据分析软件(Illumina Inc.)对SNP数据进行评分,GenCall阈值为0.15。GenTrain评分< 0.6和分离失真严重的snp (χ2检验,p < 10)6),并排除了超过1%的缺失数据。
遗传连锁图谱
利用joinmap4进行遗传连锁分析和图谱构建[37]。在joinmap4软件中生成CxA和WxBy图作为“F2”种群进行分析。将重组阈值设置为0.40,利用Kosambi映射函数将重组频率转换为地图距离。显示分离扭曲的标记被纳入连锁分析。CxA和WxBy图谱的所有连锁群均以最小LOD评分为5计算。连结图使用MapChart 2.1软件绘制[38]。
法分析
采用MAPQTL 6.0版软件进行QTL分析[39]。经1万个排列检验,xa图谱2007年和2008年的LOD阈值分别为3.9和3.8,WxBy图谱2010年和2011年的LOD阈值分别为3.6和3.5,达到QTL显著性。为了获得p = 0.05左右的总体显著性水平,我们采用严格的显著性水平p = 0.005作为单个检验的QTL检测阈值,Van Ooijen [39]。使用MapChart 2.1软件绘制qtl [38]。对于两种植物F2基因模型ppa008301m中9bp插入/缺失的群体、成熟期表型数据和基因型报告于Additional file1。为了使遗传图谱和QTL特征可视化,QTL- lod图谱被绘制成热图,热图是由Edmund Mach基金会开发的Harry Plotter软件(http://genomics.research.iasma.it/download.html).
NAC ppa007577m和ppa008301 9 bp INDELs的发现及基因分型
候选基因ppa00757m和ppa008301m分别跨越111,126,755 - 111,131,088和111,105,608 -11,107,907区域进行Sanger测序,引物组合如Additional file所述4。分别用正向引物5′-CTACTCATACCCGCCAAGGA-3′和反向引物AACGTCGTCATGAGGTACCC扩增ppa007577m基因232 bp缺失。PCR反应含有1-20 ng基因组DNA, 1倍PCR反应缓冲液(16 mM, (NH)4)2所以4, 67 mM Tris-Cl pH 8.8和0.1% Tween-20), 3 mM MgCl2,每个dNTP 0.2 mM,正向和反向特异性引物40 nM (Sigma Life Sciences), EuroTaq DNA聚合酶0.1 U (EuroClone),无菌蒸馏水至20 μl终体积。扩增设置为在95°C下初始变性步骤2 min;30个扩增周期,94°C 30s, 60°C 30s, 72°C 30s, 72°C最终延长5min。PCR产物装在0.8%琼脂糖凝胶上(Sigma Life Sciences)。SSR扩增按照Hayden描述的多重准备PCR协议进行等。经过一些修改后[40]。合成前向NAC ppa008301- indel特异性引物,在5′端添加序列5′-ACGACGTTGTAAAA-3′。该方案还包括使用短的通用引物tagF,用VIC荧光染料荧光标记(5 ' -ACGACGTTGTAAAA-3 ')。PCR反应含有1-20 ng基因组DNA, 1倍PCR反应缓冲液(16 mM, (NH)4)2所以4, 67 mM Tris-HCl pH 8.8和0.1% Tween-20), 3 mM MgCl2,每个dNTP 0.2 mM,每个标签引物100 nM, nac - indel特异性引物40 nM(正向:5 ' -AGAACTCAGCGGGTTGATAACT-3 ';相反:5“-TGCACCCCTACTCGATTTCT-3”;Sigma Life Sciences), 0.01 U EuroTaq DNA聚合酶(EuroClone),无菌蒸馏水至8 μl终体积。扩增程序包括在95°C下初始变性步骤2 min;预扩增周期20个,92℃30s, 60℃30s, 72℃30s;40个扩增/标记周期,在92°C下15 s, 54°C下30 s, 72°C下30 s;最后在72℃下延长5min,然后在25℃下延长25min。
CxA基因多态性检测1来自全基因组重测序数据的亲本
公开的对端全基因组重测序数据碧桃文献(研究SRP013437)下载自NCBI Sequence Read Archive (SRA,http://www.ncbi.nlm.nih.gov/Traces/sra, (41]。在本研究中,我们考虑了生物样品SRS335634 (Lovell Clone PLov2-2 - N,运行SRR502985)和SRS335631 (F1竞争者x Ambra,运行SRR502997)。将每次运行的SRA数据转储为fastq格式fastq-dumpNCBI sratoolkit v.2.1.16软件(http://www.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software);正向和反向配对读取将每个样本分成两个单独的文件(选项2)将文件拆分).使用Trimmomatic v.0.22对读数进行质量修剪[42],修剪前尾碱基低于20的质量阈值,同时去除平均质量低于20的读取(以8 bp长的滑动窗口计算)和修剪的读取短于24 bp。对于每个样本,只有通过质量过滤的读数作为匹配对被保留并与整体对齐碧桃参考基因组v1.0 (http://www.rosaceae.org, (14])使用Burrows-Wheeler对齐工具(BWA) v.0.6.2 [43]。的aln(IS线性时间算法)和sampe(所有默认选项除外- n25 -N25)命令分别用于构建每个reads的良好命中的后缀数组(SA)坐标,并将其转换为染色体坐标并配对。生成的SAM文件被Picard Tools v.1.77 (http://picard.sourceforge.net/)按顺序使用工具CleanSam,SamFormatConverter和AddOrReplaceReadGroups。符合GATK的BAM文件提交到GATK v.2.3-3 [44]用于预处理程序,包括indel重新调整,重复删除和基本质量分数重新校准(BQSR)。BQSR中重新校准步骤所需的数据表是根据来自Peach 9k芯片阵列的验证SNP数据手动生成的[15]。然后使用GATK同时在CxA和Lovell样品中使用全基因组重新校准比对应用变异发现程序HaplotypeCaller应用标准硬过滤参数的工具[45]。
系统发育分析
已知的NAC家族蛋白收集自文献和UniProtKB/Swiss-Prot (http://www.uniprot.org) [46]。基因名称和UniProtKB7Swiss-Prot加入号如下:栽培稻: OsNAC3 [Q7EZT1]、OsNAC4 [Q52QH4]、OsNAC5 [Q53NF7]、OsNAC6 [Q7F2L3]、OsNAC7 [Q5Z6B6]、OsNAC8 [Q7GCL7]、ONAC010 [Q8H4S4] [31];答:芥: ATAF1 [Q39013]及ATAF2 [Q9C598] [32], nap [o49255] [47]、CUC1 [Q9FRV4]、CUC2[04017]、AtNAC2 [Q9LD44]、[48], [q9s851] [49], nac1 [q84te6] [50], nac2 [q84k00] [46], [Q9ZNU2] [29[qh], [qh] [qh]51]、ANAC019 [Q9C932]、ANAC055 [Q9LDY8]、ANAC072 [Q93VY3] [52];佩妮矮牵牛: nam [q40880] [32];茄属植物lycopersicum[q56up7] [46], SlNAC1 [Q6RH27] [34];茄属植物tuberosum: StNAC1 [Q948Z2] [46];Lycopersicon esculentum: senu5 [q43521] [53];烟草: term [q9sxq0] [46];小麦属植物sp.: GRAB1[Q9ZRZ3]和GRAB2 [Q9ZRZ2] [33]、NAM-B1 [A0SPJ4]及NAM-B2 [A0SPJ6] [24];穆萨acuminata: MaNAC1 [M0TAJ5], MaNAC2 [M0TBV3], MaNAC3 [M0U410], MaNAC4 [M0TDM6], MaNAC5 [M0U3M1], MaNAC6 [M0RNB9] [9]。采用Neighbor-Joining法进行系统发育分析[54]。从1000个重复中推断出的bootstrap共识树[55]代表所分析的分类群的进化史[55]。在少于50%的引导复制中复制的分区对应的分支将崩溃。进化距离采用泊松校正法计算[56],并以每个位点的氨基酸取代数表示。分析共涉及39个蛋白序列,包括ppa007577m和ppa008301m。每个序列对的所有模糊位置都被移除。最终数据集中共有708个位置。对MEGA5进行了进化分析[57]。
参考文献
- 1.
王晓明,王晓明,王晓明,等。番茄果实发育过程中代谢调控的研究进展。[J] .中国生物医学工程学报,2006,27(5):883- 897。
- 2.
李建军,李建军,李建军,李建军,李建军。草莓果实发育过程中瘦果和花托代谢网络的重构。植物生理学报,2009,32(4):559 - 563。
- 3.
Moing A, Aharoni A, Biais B, Rogachev I, Meir S, Brodsky L, Allwood JW, Erban A, Dunn WB, Kay L,等:空间和发育组合代谢组学研究揭示甜瓜果实广泛代谢串扰。中国生物医学工程学报,2011,31(2):683-696。
- 4.
张静,王鑫,于奥,唐健,顾鑫,万鑫,方翀:草莓果实发育和成熟过程中的代谢特征分析。[J] .中国生物医学工程学报,2011,32(2):1103-1118。
- 5.
Ziliotto F, Begheldo M, Rasori A, Bonghi C, Tonutti P: 1-MCP处理油桃果实的转录组分析。实验学报,2008,(5):2781-2791。
- 6.
Schaffer RJ, Friel EN, Souleyre EJ, Bolitho K, Thodey K, Ledger S, Bowen JH, Ma JH, Nain B, Cohen D,等:基因组学方法揭示了苹果香气产生主要受乙烯控制的生物合成途径的最后一步。植物生理学报,2007,44(4):559 - 561。
- 7.
张建军,张建军,张建军,等。乙烯在番茄果实发育和成熟过程中的分子生物学研究。植物科学,2008,35(5):1093 - 1093。
- 8.
李洪军,乔凡诺尼京,等。番茄果实成熟和品质性状的遗传与控制。[j] .科学通报,2011,45(5):441 - 559。
- 9.
单伟,邝建峰,陈玲,谢慧,彭海红,肖玉云,李晓平,陈文霞,何清国,陈建勇,卢文杰:香蕉果实成熟过程中NAC转录因子及其与乙烯信号成分EIL的相互作用。实验学报,2012,32(3):5171-5187。
- 10.
Eduardo I, Pacheco I, Chietera G, Bassi D, Pozzi C, Vecchietti A, Rossini L:两个桃种内群体果实品质性状的QTL分析及成熟期多效效应的重要性。基因工程学报,2011,27(7):323-335。
- 11.
Quilot B, Wu BH, Kervella J, Genard M, Foulongne M, Moreau K:桃李与野生近缘种大花李高级回交品质性状的QTL分析。理论与应用,2004,(10):884-897。
- 12.
Verde I, Quarta R, Cedrola C, Dettori MT: BC1桃群体农艺性状的QTL分析。第五届国际桃子学术研讨会论文集。植物学报,2002,29(2):291-297。
- 13.
Dirlewanger E, Quero-Garcia J, Le Dantec L, Lambert P, Ruiz D, Dondini L, Illa E, Quilot-Turion B, Audergon JM, Tartarini S,等:桃树、杏和甜樱桃3种李属植物花期和成熟期两个关键物候性状的遗传确定性比较。遗传(Edinb)。中国机械工程,2012,31(2):391 - 391。
- 14.
Verde I, Abbott AG, Scalabrin S, Jung S, Shu S, Marroni F, Zhebentyayeva T, Dettori MT, Grimwood J, Cattonaro F,等:桃树(Prunus persica)高质量基因组草图的遗传多样性、驯化和基因组进化模式。生物医学工程学报,2013,45(5):487-494。
- 15.
Verde I, Bassil N, Scalabrin S, Gilmore B, Lawley CT, Gasic K, Micheletti D, Rosyara UR, Cattonaro F, Vendramin E,等:桃子9K SNP阵列的构建及其在育种种质中的应用。科学通报,2012,27 (7):356 - 356
- 16.
Dirlewanger E, Moing A, Rothan C, Svanella L, Pronier V, Guye A, Plomion C, Monet R:桃果实品质控制qtl的定位蟠桃。理论与应用,1999,98:18-31。
- 17.
Font I, Forcada C, Oraguzie N, Igartua E, Moreno MÁ, Gogorcena Y:桃和油桃品种果实性状的群体结构和标记性状关联。基因工程学报,2013,29(9):331-349。
- 18.
李建军,李建军,李建军,李建军。拟南芥NAC转录因子家族在逆境胁迫下的表达及其调控作用。中国生物医学工程学报,2010,26(4):559 - 564。
- 19.
李建军,陈建军,李建军,等。NAC转录因子的研究进展。植物科学进展,2005,10:79-87。
- 20.
李春华,李春华,李春华,李春华,李春华。小麦和水稻同源NAC转录因子的克隆与分析。植物生理学报,2012,31(2):515-524。
- 21.
Giovannoni JJ, Noensie EN, Ruezinsky DM, Lu X, Tracy SL, Ganal MW, Martin GB, Pillen K, Alpert K, Tanksley SD:番茄成熟抑制基因和非成熟基因位点的分子遗传分析:果实成熟基因遗传图谱克隆的第一步。中国生物医学工程学报,1997,26(4):559 - 561。
- 22.
Osorio S, Alba R, Damasceno CM, Lopez-Casado G, Lohse M, Zanor MI, Tohge T, Usadel B, Rose JK, Fei Z,等。番茄果实发育的系统生物学:番茄转录因子(nor, rin)和乙烯受体(Nr)突变体的联合转录、蛋白质和代谢产物分析。植物生理学报,2011,32(4):557 - 557。
- 23.
Kunieda T, Mitsuda N, ohme takagi M, Takeda S, Aida M, Tasaka M, Kondo M, Nishimura M, Hara-Nishimura I:拟南芥外被NAC家族蛋白NARS1/NAC2和NARS2/NAM调控胚胎发生。植物学报,2008,26(2):631- 642。
- 24.
王晓明,王晓明,王晓明,等。NAC基因对小麦籽粒蛋白质、锌和铁含量的影响。科学通报,2006,31(4):1298-1301。
- 25.
陈国良,Nakashima K, Sakuma Y, Simpson SD, Fujita Y, Maruyama K, Fujita M, Seki M, Shinozaki K, Yamaguchi-Shinozaki K:拟南芥干旱胁迫诱导NAC转录因子的分离和功能分析。植物学报,2004,16(6):481- 498。
- 26.
Hickman R, Hill C, Penfold CA, Breeze E, Bowden L, Moore JD, Zhang P, Jackson A, Cooke E, Bewicke-Copley F,等:拟南芥叶片NAC转录因子在胁迫应答和衰老中的局部调控网络。植物学报,2013,31(1):26-39。
- 27.
王晓明,王晓明,王晓明,等。基因同源性的功能和进化意义。水土保持学报,2013,14(4):369 - 369。
- 28.
Aida M, Ishida T, Fukaki H, Fujisawa H, Tasaka M:拟南芥杯形子叶突变体的器官分离相关基因分析。植物学报,1997,9:841-857。
- 29.
杜华,谢福峰,金世荣,Thomas TL:拟南芥NAC结构域超家族成员AtNAM的分子特征。植物化学学报,2002,25(5):387 - 398。
- 30.
李建军,李建军,李建军,李建军,李建军。甘蓝型油菜NAC结构域转录激活因子的研究进展。中国生物医学工程学报,2003,35(3):383-397。
- 31.
Kikuchi K, guguchi - tanaka M, Yoshida KT, Nagato Y, Matsusoka M, Hirano HY:水稻NAC基因家族的分子分析。生物医学工程学报,2002,26(2):447 - 451。
- 32.
soer E, van Houwelingen A, Kloos D, Mol J, Koes R:牵牛胚和花形态形成所需的无顶分生组织基因在分生组织和原基边界表达。细胞。1996,85:159-170。
- 33.
谢强,Sanz-Burgos AP,郭宏,Garcia JA, Gutierrez C: NAC结构域家族的新成员- GRAB蛋白与双病毒蛋白的相互作用。植物化学学报,1999,19(3):647-656。
- 34.
黄伟,苗明,库杰,牛鑫,欧阳波,张军,叶芝,Kuhl JC,刘勇,肖峰:SlNAC1是应激相关转录因子,在转录和翻译后水平上均有调控。中国生物医学工程学报,2013,32(1):444 - 444。
- 35.
陈陆PL,新西兰,R,苏Z, BS,任F,陈J,王XC:小说drought-inducible基因,ATAF1编码NAC家族蛋白,负调节拟南芥的逆境应答基因的表达。植物化学学报,2007,32(3):559 - 559。
- 36.
Jurinke C, van den Boom D, Cantor CR, Koster H: DNA MassArray技术的自动基因分型。方法:中华生物医学杂志,2002,18(3):179-192。
- 37.
Van Ooijen J: joinmap4,用于计算实验群体遗传连锁图谱的软件。荷兰瓦赫宁根:Kyazma BV;2006.
- 38.
Voorrips RE: MapChart:用于链接图和qtl图形化表示的软件。中国生物医学工程学报,2002,32(3):559 - 561。
- 39.
Van Ooijen J: MapQTL 6,二倍体植物实验群体数量性状位点定位软件。2009年,荷兰瓦赫宁根:Kyazma BV
- 40.
Hayden MJ, Nguyen TM, Waterman A, Chalmers KJ:一种新的SSR和SNP基因分型方法。生物医学工程学报,2008,(9):80- 80
- 41.
Wheeler DL, Barrett T, Benson DA, Bryant SH, Canese K, Chetvernin V, Church DM, Dicuccio M, Edgar R, Federhen S,等:国家生物技术信息中心数据库资源。核酸学报,2008,36(6):913 - 921。
- 42.
Lohse M, Bolger AM, Nagel A, Fernie AR, Lunn JE, Stitt M, Usadel B: RobiNA:基于rna - seq的转录组学的用户友好集成软件解决方案。中国生物医学工程学报,2012,35(4):622- 627。
- 43.
李宏,Durbin R:基于burrows-wheeler变换的快速精确短读对齐。生物信息学报,2009,25(5):1754-1760。
- 44.
麦凯纳A,汉娜M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo MA:基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。中国生物医学工程学报,2010,32(2):444 - 444。
- 45.
DePristo MA, Banks E, Poplin R, Garimella KV, Maguire JR, Hartl C, Philippakis AA, del Angel G, Rivas MA, Hanna M,等:基于下一代DNA测序数据的变异发现和基因分型框架。生物医学工程学报,2011,33(4):491-498。
- 46.
在通用蛋白质资源(UniProt)上重组蛋白质空间。中国生物医学工程学报,2012,35(4):771 - 775。
- 47.
Sablowski RW, Meyerowitz EM: NO APICAL分生系统的同源物是花同源基因APETALA3/雌蕊的直接目标。细胞,1998,32(2):93-103。
- 48.
Takada S, Hibara K, Ishida T, Tasaka M:拟南芥茎尖分生组织形成的杯状COTYLEDON1基因。发展,2001,28:1127-1135。
- 49.
Vroemen CW, Mordhorst AP, Albrecht C, Kwaaitaal MA, de Vries SC:拟南芥分生组织边界和茎部分生组织形成所需的杯形子叶don3基因。植物学报,2003,15(5):1563-1577。
- 50.
谢强,Frugis G, Colgan D, chunh:拟南芥NAC1在TIR1下游转导生长素信号促进侧根发育。基因工程学报,2000,14(4):334 - 336。
- 51.
任涛,曲峰,Morris TJ: HRT基因功能需要NAC蛋白与病毒衣壳蛋白相互作用才能获得对萝卜皱病毒的抗性。植物细胞,2000,12:1917-1926。
- 52.
Ooka H, Satoh K, Doi K, Nagata T, Otomo Y, Murakami K, Matsubara K, Osato N, Kawai J, Carninci P,等:水稻和拟南芥NAC家族基因的综合分析。中国生物医学工程学报,2003,25(3):379 - 379。
- 53.
李建军,李建军,李建军,李建军,等。番茄叶片衰老相关cdna的克隆与鉴定。植物学报,1997,33(3):641-651。
- 54.
齐涛,李晓明,李晓明,等。一种新的系统发育树重建方法。中华生物医学工程学报,1997,22(4):444 - 444。
- 55.
Felsenstein J:系统发育的置信限:一种使用自举法的方法。生物学进展,1985,39:783-791。
- 56.
朱建平,李建平,李建平,等。蛋白质的进化分化与趋同。进化的基因和蛋白质。编辑:Bryson V, Vogel HJ。纽约:学术出版社;1965.
- 57.
李建军,李建军,李建军,等。基于最大似然、进化距离和最大简约方法的分子进化遗传分析。中华生物医学工程学报,2011,28(2):731- 739。
致谢
我们要感谢S. Foschi和M. Lama在田间林木管理和采伐方面的宝贵贡献,M. Valdameri和C. Colombi在SSR基因分型方面的贡献,M. Troggio在SNP基因分型方面的帮助。这项工作得到了意大利向DB提供的赠款的支持,该赠款由私人和公共机构“MAS”资助。PES:杏和桃的分子辅助选择育种”,由意大利农业、食品和林业部(MiPAAF)通过“DRUPOMICS”项目(批准号DM14999/7303/08)资助。知识产权局和知识产权局的工作得到了意大利里卡多大学(MIUR)教育部博士奖学金的支持。
作者感谢博洛尼亚应用生物医学研究中心(CRBA)对Sequenom分析的宝贵贡献。作者还感谢博洛尼亚Cassa di Risparmio基金会对CRBA的支持。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者证实不存在相互竞争的利益。
作者的贡献
RP(设计实验;进行实验;分析数据;写稿);IE(分析数据;写稿);IP(收集和分析表型和基因型数据;修改原稿);分析数据;修改原稿); MM (analysed data); IV (provided re-sequencing data prior to publication); ST (genotyping, analysed data; revised the manuscript); LD (genotyping; analysed data; revised the manuscript); GP (analysed data); DB (selected the genetic materials and developed the populations; revised the manuscript); LR (conceived and designed the study; analysed data; helped writing the manuscript). All authors read and approved the final manuscript.
电子补充资料
表报告了CxA和WxBy F的成熟日期和相应的基因型
附加文件1:2本研究分析的个体。(xl50kb)
12870 _2013_1370_moesm2_esm.xls
附加文件2:重复QTL分析,包括在ppa008301m候选基因(INDEL)中发现的9 bp INDEL的基因分型数据。CxA_2007和CxA_2008表分别报告了2007年和2008年CxA种群的QTL分析。WxBy_2010和WxBy_2011表分别报告了2010年和2011年WxBy种群的QTL分析。位置:地图在cM中的位置;基因座:标记id;LOD: LOD评分;# iter。:the number of iteration needed to reach the tolerance criterium; mu_A, mu_H, and mu_B: estimated means of the distribution of the quantitative trait associated with the a-, h-, and b-genotypes; variance: the residual variance after fitting the QTL;%Expl.: percentage of the variance explained by the QTL; Additive: the estimated additive effect; Dominance: the estimated dominance effect; GIC: genotypic information coefficient. (XLS 84 KB)
使用Sequenom平台进行CxA F SNP基因分型的引物细节
附加文件3:2人口。(xls22kb)
12870 _2013_1370_moesm4_esm.xls
附加文件4:两个NAC候选基因及其上下游区域的Sanger筛选引物序列。(xl24kb)
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
R.皮罗纳,I.爱德华多,I.帕切科。et al。桃成熟日期主要控制位点候选基因的精细定位与鉴定。BMC Plant Biol13日,166(2013)。https://doi.org/10.1186/1471-2229-13-166
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-166
关键字
- 到期日
- 碧桃
- QTL
- 候选基因