摘要
背景
Argonaute蛋白是RNA干扰(RNAi)的关键成分,在RNA定向基因沉默中发挥重要作用。各种类型的Argonaute基因已经从植物中鉴定出来,并可能参与发育调控。然而,人们对小麦中的这些基因知之甚少。小麦).
结果
本研究从小麦中克隆了两个Argonaute的全长cdna,命名为TaAGO1b和TaAGO4.的cDNATaAGO1b全长3273 bp,编码868个氨基酸,预测分子量为~97.78 kDa, pI为9.29。3157年英国石油公司TaAGO4编码916个氨基酸,分子量为102.10 kDa, pI为9.12。基因组学分析显示,TaAGO1b和TaAGO4分别含有20和18个内含子。蛋白结构分析表明,TaAGO1b和TaAGO4均存在典型的PAZ和PIWI结构域。从高度保守的PIWI结构域,我们检测到保守的Asp-Asp-His (DDH)基序,它作为催化三联体起作用,在RNAi机制中序列特异性切割过程中起关键作用。结构模型表明,这两个TaAGOs都可以折叠成特定的α/β结构。此外,三个排列的DDH残基在PIWI结构域的“切片器”位置空间上彼此接近。表达分析表明,这两个基因在根、茎、叶、花药、胚珠和种子等营养和生殖器官中普遍表达。然而,它们在萌发胚乳组织中有差异表达。我们很感兴趣地了解到这两个TaAGOs在发育中的小麦植株中也有差异表达,并且它们的表达模式受到春化处理的不同影响。进一步的研究表明,它们可以由春化过程中的冷积累引起。
结论
两个假定的小麦Argonaute基因TaAGO1b和TaAGO4,都是克隆的。系统发育分析、保守结构域和催化基序预测以及它们的蛋白质结构建模表明,它们编码功能性Argonaute蛋白。时空表达分析表明,这些基因可能参与小麦植株的发育调控。
背景
众所周知,RNA干扰途径在转录后基因沉默和触发染色质修饰中起着关键作用[1]。遗传、生化和结构研究表明,Argonaute (AGO)蛋白是RNAi效应复合物RISC的催化核心;第一个AGO基因在拟南芥[2,3.]。植物AGO蛋白是高度保守的碱性蛋白(约100 kDa),具有典型的结构域或氨基酸基序。这些结构域包括具有rnase - h样折叠的c端PIWI (p -元件诱导的弱睾丸)结构域,通过靶RNA的3'端结合小RNA的中心PAZ (PIWI Argonaute和Zwille)结构域,以及位于PAZ和PIWI结构域之间的MID结构域,该结构域将小RNA的5'磷酸端固定在Argonaute蛋白上。4,5]。基于它们的系统发育关系和它们与小rna结合的能力,人们可以将这些蛋白质分为三组:I,这是ago样亚家族(类似于ago2的AGO1)拟南芥结合微小rna (miRNAs)和小干扰rna (sirna)的;II, PIWI样亚家族(与PIWI蛋白密切相关)黑腹果蝇),其成员与piwi相互作用rna (pirna)结合;第三,与次级sirna结合的WAGO亚家族(蠕虫特异性Argonautes) [5- - - - - -7]。
AGO-small-RNA复合物可以抑制基因的转录,靶向mRNA进行位点特异性切割或一般降解,或阻止mRNA翻译成蛋白质。一些证据表明,Argonaute蛋白在rna导向的基因沉默中起着至关重要的作用,并参与植物的发育调节[8- - - - - -10]。通过基因组注释,鉴定出10个和18个AGO基因拟南芥米饭(栽培稻),分别[4,11]。虽然这些AGO成员中的一些已经被表征,但大多数在植物中尚未被探索。
小麦(小麦)是一种全球重要作物,占人类消耗热量的20% [12]。从分子水平研究小麦发育调控机制对加快小麦改良进程具有重要意义。AGO蛋白作为rna导向基因沉默机制的关键组分,在发育调控中发挥重要作用[j]。9,10]。然而,人们对小麦中的这些基因知之甚少。在此,我们克隆并鉴定了两个小麦AGO基因,并分析了它们的时空表达模式。我们还研究了它们在发育特异性基因调控中的潜在参与。
结果
克隆TaAGOs带全长cDNA
我们在NCBI EST(表达序列标签)数据库中进行TBLASTX分析(http://www.ncbi.nlm.nih.gov/dbEST/)用两个拟南芥Argonaute基因,AGO1[GenBank: NM_179453]和AGO4[基因库:NM_128262]。结果表明,两组小麦ESTs具有高度同源性拟南芥AGO1和AGO4。根据其EST序列的保守区域,我们设计了特定的引物(附加文件)1)克隆小麦Argonaute基因。
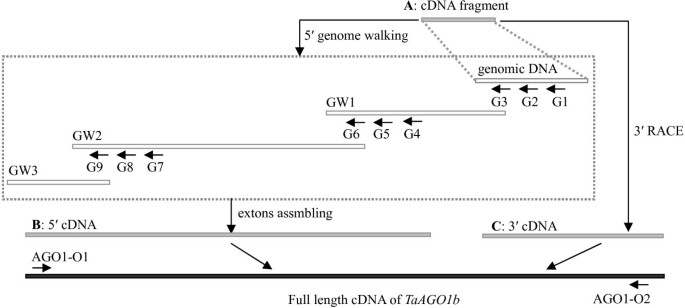
采用TaAGO1-1F和-1R的引物组合进行RT-PCR(逆转录聚合酶链反应)扩增。一个412bp的cDNA片段(图1-A)是克隆的。为了延长小麦Argonaute基因的序列,根据克隆的序列设计了新的引物,并应用于5'-和3'- race (cDNA末端快速扩增)中。虽然进行了多次5′-RACE分析,但没有得到满意的结果。因此,我们选择了基因组行走策略来克隆该5'区域。首先,cDNA区域对应的基因组片段(图2)1a)被克隆和基因组行走引物(附加文件1)是根据序列设计的。三轮基因组行走(图1(GW1-3),得到3958 bp的基因组DNA。最后,5'-cDNA区域(图21-B)由外显子(http://genes.mit.edu/GENSCAN.html).3’-cDNA片段(图21-C)用3′-RACE分析。将上述三个片段组装在一起,得到全长cDNA(图1)1a - c),通过RT-PCR克隆和测序证实了AGO1-O1和-O2之间的cDNA序列。该小麦AGO基因全长3273 bp,编码一个含有868个氨基酸残基的推测蛋白,与水稻OsAGO1b高度同源[Swiss-Prot: Q7XSA2.3],被命名为TaAGO1b[基因库:JQ805149]。
利用引物TaAGO4-1F和-1R,通过RT-PCR扩增,克隆了一个498 bp的小麦cDNA片段。测序结果显示该cDNA与拟南芥AGO4。根据克隆的序列,通过5′-和3′- race获得其全长cDNA。序列分析表明,该基因来自小麦AGO4全长3157 bp,编码一种由916个氨基酸残基组成的蛋白质。在NCBI中的BLASTX分析显示其高度同源拟南芥ag4 [GenBank: AEC07929.1]。因此,我们将其命名为TaAGO4[基因库:JQ805150]。
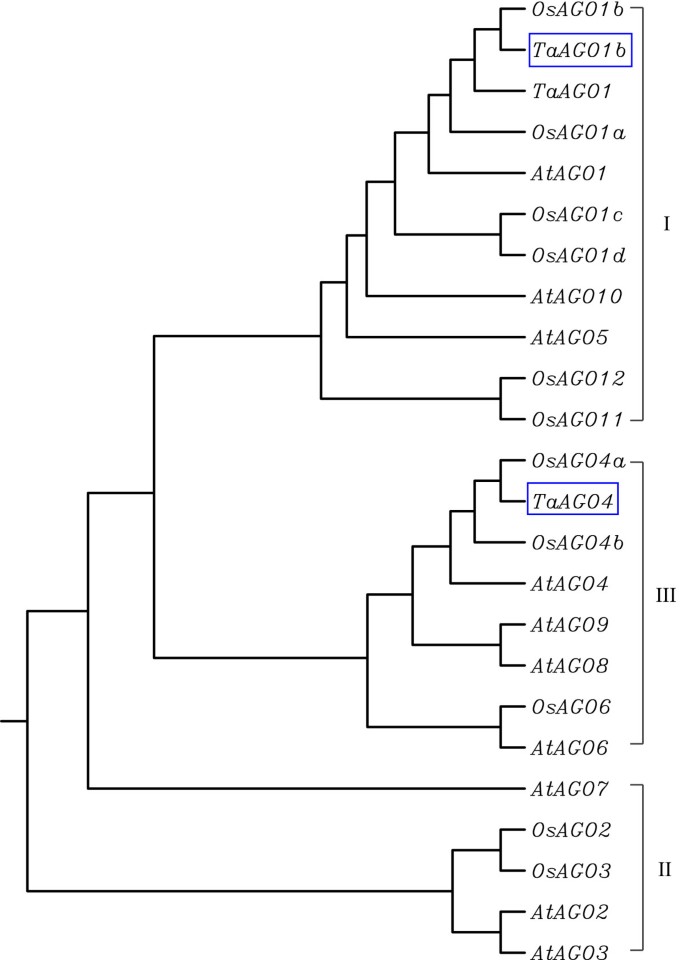
AGO植物蛋白(包括来自水稻的AGO)的系统发育分析拟南芥结果表明,TaAGO1b和TaAGO4可分为上述三组中的两组(图2)2).TaAGO1b与OsAGO1b、TaAGO1、OsAGO1a、AtAGO1具有高度同源性,归属于第I族。TaAGO4与水稻OsAGO4a、OsAGO4b、OsAGO6属于同一个单系亚类,属于第III群;和拟南芥AtAGO4, AtAGO6, AtAGO8,和AtAGO9。三个拟南芥将AGOs (AtAGO2、3、7)和2个水稻AGOs (OsAGO2、3)分为第二组(图2)2).
塔古斯的特点
ExPaSy (http://www.expasy.org) [13,14结果表明,TaAGO1b包含868个氨基酸,预测分子量为~97.78 kDa,理论pI为9.29。TaAGO4全长916个氨基酸,理论pI为9.12,分子量约为102.10 kDa。
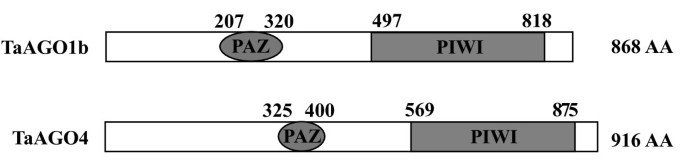
两个TaAGOs都包含典型的PAZ和PIWI保守区域(图2)3.).TaAGO1b的PAZ结构域由114个氨基酸组成(来自残基207 - 320),与其他植物AGO1蛋白具有82 - 95%的同源性。TaAGO4的PAZ结构域包含76个氨基酸(残基325 - 400),与其他植物AGO4成员仅有有限的同源性。TaAGO1b的c端PIWI结构域包含322个氨基酸(残基497 - 818),与其他植物AGO1成员如OsAGO1b(97%)、OsAGO1a(96%)和AtAGO1(92%)具有高度相似性。TaAGO4(残基569 - 875)的PIWI结构域与OsAGO4a、OsAGO4b和AtAGO4的PIWI结构域同源性分别为90%、85%和77%。一般来说,植物Argonaute成员的PIWI结构域具有较高的相似性,这支持了先前关于PIWI结构域保守性较高而PAZ结构域保守性较差的报道[15]。
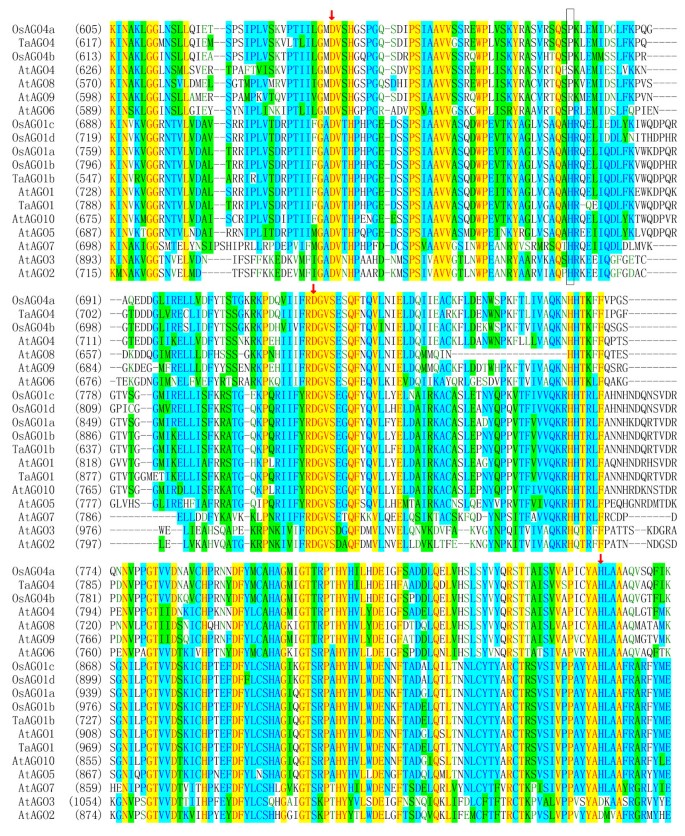
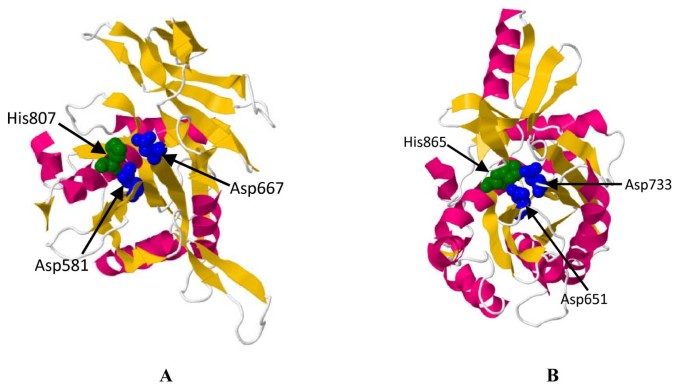
当我们对齐植物AGOs的PIWI结构域时,包括水稻和水稻的TaAGOs和类似物拟南芥我们在TaAGO1b和TaAGO4中检测到三个保守的金属螯合氨基酸——天冬氨酸、天冬氨酸和组氨酸(DDH)4).这些特定的DDH残基在rna定向切割靶rna的过程中发挥关键作用,起到催化三联体的作用[16]。因此,在两种小麦AGOs中都包含一个DDH三联体,这表明它们在功能上与之前表征的rna定向基因沉默的AGOs相似。先前的研究表明,AtAGO1的798 (H798)位置的保守组氨酸对于在体外内切酶活性[17];在TaAGO1b中也检测到该残基。相比之下,在TaAGO4中,以及在OsAGO4a、OsAGO4b、AtAGO6和AtAGO8中,798位的组氨酸被脯氨酸(P)取代,而在AtAGO4和AtAGO9中,该位点分别被丝氨酸(S)和精氨酸(R)取代(图2)4).
PIWI结构域的结构建模,Argonaute蛋白[PDB: 2004]来自海床furiosus作为模板,表明TaAGOs可以折叠成特定的α/β结构,该结构以中心混合β-片为主导,两侧有两个长α-螺旋。在这里,三个排列的DDH残基(Asp, Asp和His)在空间上彼此靠近,位于PIWI结构域的“切片器”位置(图2)5).
基因组序列分析TaAGO1b和TaAGO4
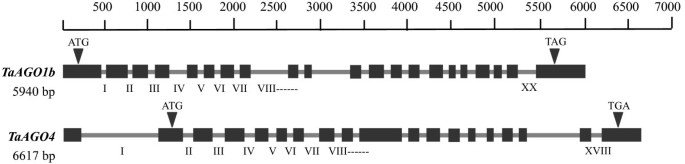
基因组DNA序列分析采用DNA扩增或基因组行走,使用特定的引物。TaAGO1b内含子20个,长度从72 ~ 449 bp不等。相比之下,18个内含子,长78 ~ 858 bpTaAGO4(图6).最大的内含子(858 bp)TaAGO4位于mRNA 5' UTR对应的区域。
的空间表现模式TaAGOs
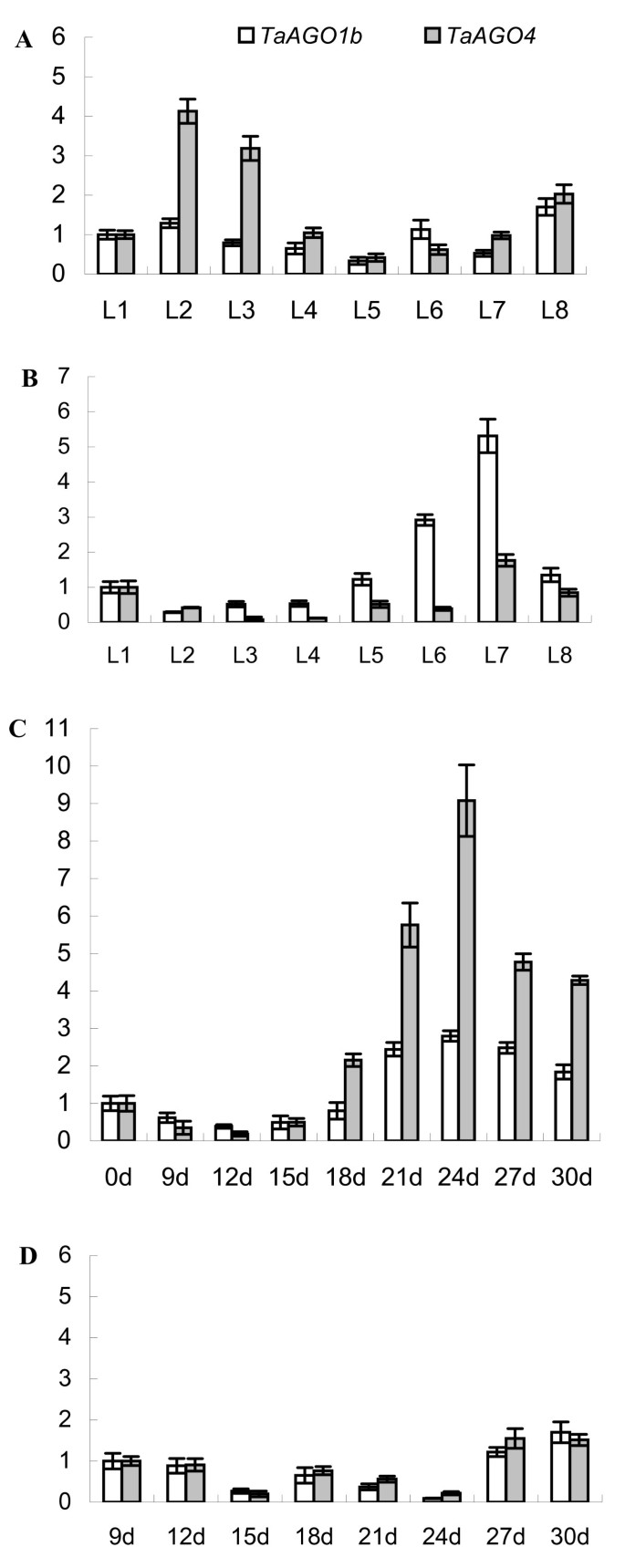
半RT-PCR表达分析证实两者均有TaAGO1b和TaAGO4在所有被检测的组织中都高度表达,包括根、茎、叶、花药、胚珠和成熟种子(图7-A),以及小麦籽粒的发育(图7- b)。在发芽,TaAGO1b和TaAGO4在胚胎组织中普遍表达(图7-C),但仅在胚乳中差异表达(图2)7- d)。成绩单水平TaAGO4小麦种子萌发过程中胚乳组织中磷含量明显减少。
的变化TaAGO春化反应的表达
的表达模式TaAGOs随着时间的推移,我们收获了小麦1至8叶发育阶段的叶片,并进行了实时定量RT-PCR。的表达TaAGO4在2叶和3叶期显著上调,而TaAGO1b在我们的观察期间没有明显的变化(图8一)。春化处理(在播种前将发芽的种子在4°C的黑暗环境下暴露30 d)显著影响了它们的表达模式TaAGO4在2叶和3叶阶段不再上调,但是TaAGO1b在6叶和7叶期显著上调(图2)8- b)。
为了进一步研究春化治疗的效果,我们监测了TaAGOs在冷积累期间。均在茎部组织中诱导,表达TaAGO4在开始冷处理后的18 ~ 30 d显著上调,但诱导TaAGO1b相对较低(图1)8- c)。然而,没有明显的诱导TaAGOs在冷积累过程中,在根组织中检测到(图8- d)。这些结果表明,这两个基因在春化反应中都有重要作用,但它们的功能可能涉及不同的调节机制。
在网上塔古斯地图
从一组小麦非整倍体和缺失群体中,在小麦染色体/染色体箱中绘制了超过16000个ESTs [18]。这些结果是非常有用的在网上映射分析。通过BLASTN搜索,我们确定了与小麦AGO基因同源的已定位ESTs(附加文件)2).根据序列的相似性和位置,我们可以绘制出TAGO1小矮人的长臂和TaAGO4到小麦染色体上3A、3B和3D的短臂。
讨论
转录后水平的基因表达调控在植物发育中起着至关重要的作用。RNAi途径参与转录后基因沉默和染色质修饰。Argonaute蛋白包括一个高度保守的家族,其成员与植物中的RNAi和相关途径有关[15]。我们分离了两个小麦AGO基因,并得到了全长cDNA;TaAGO1b和TaAGO4.这两个TaAGOs都是碱性蛋白(pI ~ 9.0),预测分子量为~100 kDa,与真核生物中其他AGO成员相似[j]。6,19]。Argonautes具有两个高度保守的结构域PAZ和PIWI,它们在rnai介导的基因调控过程中发挥重要作用[4]。PAZ结构域是与小rna 3'端相互作用的关键区域,决定了Argonautes的rna结合特异性。PIWI结构域与5'端结合,并与靶RNA相互作用[5]。在水稻中,PAZ结构域的同源性相对较低(~21%),而PIWI结构域高度保守,同源性>90% [16]。我们还发现,AGOs的PIWI结构域高度保守拟南芥大米和小麦。该结构域与RNase H具有广泛的同源性;一些Argonaute蛋白切割具有与小rna互补序列的靶rna [17]。在这一序列特异性裂解过程中,PIWI结构域中三个保守的金属螯合残基作为催化三联体发挥了关键作用[16,20.]。此外,具有退化活性催化基序为“D-D- h /D/E/K”的PIWI蛋白显示出推断的切片机或类似切片机的活性[6]。我们在TaAGO1b和TaAGO4中都检测到这种保守的DDH三联体。结构建模表明,DDH残基在空间上彼此接近,位于PIWI结构域的“切片器”位置。这些特征证明了TaAGOs在RNAi机制中的调控功能。
在植物中,rnai依赖通路广泛参与基因表达的控制和参与各种发育事件[21,22]。RNAi机制的组成部分参与了茎尖分生组织(SAM)中未分化细胞的维持、SAM和花分生组织中侧枝器官原基的起始以及雄性和雌性生殖细胞的形成[11,23,24]。在拟南芥,AGO1和AGO10通过miR172和miR165/166两个microrna调控花干细胞的终止[10]。在水稻中,基于微阵列的表达谱分析表明拟南芥AGO1和AGO4在花发育初期显著上调[16]。在我们的研究中,TaAGO1b和TaAGO4在营养器官和生殖器官中普遍表达,但在萌发胚乳组织中表达差异。的表达TaAGO4在种子吸胀后,这些胚乳组织中的TaAGO4立即大幅减少,这表明TaAGO4参与了胚乳中某些靶基因(如编码淀粉酶的基因)的rna定向沉默。此外,TaAGO4的下调与萌发过程中酶的上调是一致的,这些酶是将胚乳淀粉分解成糖以滋养生长的幼苗所必需的。
我们也很感兴趣TaAGO4在2叶期和3叶期叶片中显著上调,且春化处理对该表达模式也有显著影响。然而,无论是TaAGO1b也不TaAGO4在4°C条件下处理15 d诱导。然而,在21 d冷积累后,两者均显著上调。这表明TaAGO1b和TaAGO4参与了小麦春化过程中芽部某些开花抑制因子的rna定向沉默。在根组织中,没有明显的诱导作用TaAGOs在冷应激下被检测到,这一发现与之前的报告一致TaAGO1的同系物TaAGO1b)对低温胁迫没有反应,但受到脱水的显著调节[25]。因此,我们的结果表明,潜在的参与TaAGO1b和TaAGO4在发育特异性基因调控机制中。这为TaAGOs的可能功能提供了新的见解,并为进一步研究小麦种子萌发和春化过程中rnai依赖的基因调控提供了新的起点。
结论
Argonaute蛋白是rna定向基因沉默的关键组分,在植物发育调控中发挥重要作用。我们从小麦中克隆并鉴定了两个Argonaute基因,TaAGO1b和TaAGO4.我们的研究结果表明它们编码功能性AGO蛋白。有趣的是,这两个基因在种子萌发和植物生长过程中表现出不同的表达模式,以及对春化处理的响应。这证明了TaAGO1b和TaAGO4可能参与了小麦植株的发育调控。
方法
植物样品制备
对冬小麦品种京841的根、茎、叶、花药、胚珠和成熟种子进行了取样。分别于5、10、15、20和30 DAP采集大田植株的发育种子。在萌发处理中,成熟种子在5%次氯酸钠中表面灭菌15分钟,并用灭菌水洗涤4次(每次2分钟)。然后,他们在温度控制的培养室(25°C下16小时的光周期)中从培养皿中的潮湿滤纸中吸收水分。分别于吸胀后6、12、24、48和72 h分离胚和胚乳组织。
为了监测小麦AGOs随时间的表达,我们将种子置于盆栽中,并在25°C的温控室中培养16小时。叶片在1至8叶阶段收获。
为了研究春化处理对AGO表达的影响,种子吸水24 h,然后转移到冷室(黑暗中4°C) 30 d。春化后,移栽于盆栽中,在25℃的温控室中培养,光周期为16 h。叶片在1至8叶阶段收获。为了研究AGOs在春化处理期间的表达模式,我们在第0、9、12、15、18、21、24、27和30天对茎和根组织进行了取样。所有材料均由每个独立处理的3个重复制备。
总RNA和基因组DNA的制备
按照前面的描述提取总RNA [26],并通过琼脂糖凝胶(0.8%)电泳评估这些样品的完整性。RNA的浓度和纯度由A260/A280比值测定,使用UC800核酸蛋白分析仪(Beckman Co., USA)。采用CTAB (hexadecyltrimethylammonium bromide)法从小麦叶片中提取基因组DNA [27]。
cdna的克隆
两个拟南芥Argonaute基因,AGO1[GenBank: NM_179453]和AGO4[GenBank: NM_128262],在我们针对EST数据库的TBLASTX搜索中使用。由此,我们鉴定出两组小麦EST序列——GenBank CF133307、CA741561、CK155094、CJ900837、CA726349和BE586111;与GenBank的BT009411、BQ841772、HX190168、CB307539、BJ244578、CJ679989高度同源AGO1和AGO4,分别。根据每个小麦类群的保守区域,我们设计了引物对TaAGO1-1F/1R和TaAGO4-1F/1R(附加文件)1)用于小麦基因的cDNA克隆。RT-PCR以2 μl cDNA为模板,每种引物125 pmol,反应体系为20 μl, dNTP各0.2 mM, MgCl各1.5 mM2和1u (Taq聚合酶。PCR程序如下:94°C, 5 min;然后在95°C下1分钟,在53°C下1分钟,在72°C下1分钟进行40次循环;在72°C下延长10分钟。产物经1%琼脂糖凝胶电泳分离后,恢复其条带,并将其亚克隆到pBS-T载体(TakaRa, China)中进行测序。RACE技术采用TaKaRa试剂盒(5”-完整的RACE套件和3”-完整的RACE核心集版本2.0),根据制造商的说明。具体的引物在附加文件中列出1.基因特异性引物GP1与3的外引物结合”-RACE, GP3与5相结合”运动中。特异性引物GP2和GP4与3结合”-和5”-分别RACE内引物。
全长cdna的确认
为了验证组装的序列,我们设计了基因特异性引物并对RT-PCR产物进行了测序。引物TaAGO1-O1和TaAGO1-O2克隆TaAGO1b,引物TaAGO4- o1和TaAGO4- o2扩增TaAGO4(附加文件)1).
基因组特征TaAGOs
我们使用基因组DNA扩增的特异性引物来分析TaAGO1b.引物TaAGO1-1F/R和TaAGO1-2F/R分别克隆3′-区域,5′-区域用日本Takara公司的基因组行走试剂盒扩增。这些基因组行走引物(TaAGO1-G1至TaAGO1-G9)在Additional文件中列出1其位置如图所示1.利用6对基因特异性引物(AGO4-1F/R ~ AGO4-6F/R)扩增TaAGO4的基因组DNA。
基因序列分析在网上映射
在NCBI进行了BLAST搜索。在ExPaSy上推导了氨基酸序列并预测了其结构。13,14]。ClustalW (http://www.ebi.ac.uk/clustalw/)进行比对和系统发育分析。使用HHpred服务器,使用modelmodel8.0构建PIWI域的结构模型[28]。选择的模板结构(即HHpred的最佳命中)是P.furiosusArgonaute蛋白;UniProt代码:Q8U3D2, PDB id 1U04) [19]。PIWI结构域的模型包括残基825 ~ 1070。在网上在Graingenes (http://wheat.pw.usda.gov/GG2/blast.shtml).
基因表达分析
采用基因特异性引物对AGO1-F/R和AGO4-1F/R进行RT-PCR分析,以肌动蛋白基因[GenBank: AB181991]为内源对照。引物包括TaAc-F: gttccaatctaggagggatacacgc和TaAc-R: GAACCTCCACTGAGAACAACATTACC。RT-PCR以2 μl cDNA为模板,每种引物125 pmol,反应体系为20 μl, dNTP各0.2 mM, MgCl各1.5 mM2和1u (Taq聚合酶。PCR程序如下:94°C, 5 min;然后在95°C下1分钟,在53°C下1分钟,在72°C下1分钟,循环28次;在72°C下延长10分钟。产物经1%琼脂糖凝胶电泳分离。如前所述进行实时定量RT-PCR [26],以泛素基因(GenBank登录号:AY297059)作为内源对照。引物包括UF: ATCCAGGACAAGGAGGGCA和UR: CGGAGACGGAGCACCAAG。所有其他引物在附加文件中列出1.
缩写
- 前:
-
Argonaute
- CTAB:
-
Hexadecyltrimethylammonium溴化
- 衣冠楚楚的:
-
授粉后几天
- 美国东部时间:
-
表达序列标签
- 巴斯:
-
Piwi Argonaute和Zwille
- 匹威:
-
p元素诱发的睾丸萎缩
- 种族:
-
cDNA末端的快速扩增
- RNAi:
-
RNA干扰
- rt - pcr:
-
逆转录聚合酶链反应
参考文献
Rivas FV, Tolia NH, Song JJ, Aragon JP, Liu J, Hannon GJ, Joshua-Tor L:纯化Argonaute2和siRNA形成重组人RISC。中华生物医学工程学报,2005,31(4):349 -349。10.1038 / nsmb918。
RNA沉默在植物中的应用。自然科学,2004,31(7):356-363。10.1038 / nature02874。
Bohmert K, Camus I, Bellini C, Bouchez D, Caboche M, Benning C: AGO1定义了一种新的基因位点拟南芥控制叶片发育。中华微生物学杂志,1998,17(1):170-180。10.1093 / emboj / 17.1.170。
Vaucheret H:植物ARGONAUTES。植物科学进展,2008,13(7):350-358。10.1016 / j.tplants.2008.04.007。
王晓东,王晓东,王晓东,等。Argonaute蛋白在RNA沉默中的作用。中华生物医学工程学报,2008,29(1):22-32。10.1038 / nrm2321。
托利亚·NH,约书亚·托尔·L:《切片机》和《角斗士》。生物化学学报,2007,31(1):36-43。10.1038 / nchembio848。
Yigit E, Batista PJ, Bei Y, Pang KM, Chen CC, Tolia NH, Joshua-Tor L, Mitani S, Simard MJ, Mello CC:气候变化分析秀丽隐杆线虫Argonaute家族揭示了不同的Argonaute在RNAi期间的顺序行为。Cell. 2006, 127(4): 747-757。10.1016 / j.cell.2006.09.033。
Cenik ES, Zamore PD: Argonaute蛋白。中国生物医学工程学报,2011,21(12):446-449。10.1016 / j.cub.2011.05.020。
Carrington JC, carcarbonell A, Fahlgren N, Garcia-Ruiz H, Gilbert KB, Montgomery TA, Nguyen T, Cuperus JT:三种功能分析拟南芥阿尔戈英雄使用有切片缺陷的突变体。植物细胞,2012,24(9):3613-3629。10.1105 / tpc.112.099945。
纪丽,刘霞,闫军,王伟,于木然,金玉杰,丁婷婷,刘健,崔旭,郑波,等:ARGONAUTE10和ARGONAUTE1通过两个microrna调控花干细胞的终止拟南芥.科学通报,2011,7 (3):e1001358-10.1371/journal.pgen.1001358。
noonomura K, Morohoshi A, Nakano M, Eiguchi M, Miyao A, Hirochika H, Kurata N: ARGONAUTE家族生殖细胞特异性基因对水稻减数分裂过程中有丝分裂和减数分裂进程至关重要。植物细胞,2007,19(8):2583-2594。10.1105 / tpc.107.053199。
Brenchley R, Spannagl M, Pfeifer M, Barker GLA, D'Amore R, Allen AM, McKenzie N, Kramer M, Kerhornou A, Bolser D,等:面包小麦全基因组霰弹枪测序分析。自然科学学报,2012,41(4):705-710。10.1038 / nature11650。
李建军,李建军,李建军,等。基于gis的蛋白质结构同源性建模研究。生物信息学,2006,22(2):195-201。10.1093 /生物信息学/ bti770。
李建军,张建军,张建军,张建军,等。基于gis的蛋白质结构同源性研究。自然科学学报,2009,41(1):1-13。
张明强,张明强,张建军:Argonaute家族:触角伸向RNAi,发育控制,干细胞维持和肿瘤发生。基因工程学报,2002,16(21):2733-2742。10.1101 / gad.1026102。
Kapoor M, Arora R, Lama T, Nijhawan A, Khurana JP, Tyagi AK, Kapoor S:水稻Dicer-like, Argonaute和RNA依赖性RNA聚合酶基因家族的全基因组鉴定、组织和系统发育分析及其在生殖发育和胁迫中的表达分析。中国生物医学工程学报,2008,29(9):559 - 561。
Baumberger N, Baulcombe DC:拟南芥ARGONAUTE1是一种选择性招募microrna和短干扰RNA的RNA切片器。美国国家科学促进会。中国生物医学工程学报,2009,32(3):559 - 563。10.1073 / pnas.0505461102。
Lazo GR, Chao S, Hummel DD, Edwards H, Crossman CC, Lui N, Matthews DE, Carollo VL, Hane DL, You FM,等:小麦表达序列标签(EST)资源的开发(英文)小麦L.): EST生成、单基因分析、探针选择和生物信息学对一个16000个位点的双圈定图谱。遗传学报,2004,18(2):585—593。
宋俊杰,Smith SK, Hannon GJ, Joshua-Tor L: Argonaute晶体结构及其对RISC切片机活性的影响。科学通报,2004,35(6):334 - 337。10.1126 / science.1102514。
李建军,李建军,李建军,等。基于sirna - sirna复合物的rna识别研究。自然科学学报,2005,34(7):663-666。10.1038 / nature03462。
一种microRNA作为aptala2的翻译抑制因子拟南芥花的发育。科学通报,2004,33(6):1222 - 1225。10.1126 / science.1088060。
李建军,李建军,李建军,等。植物转录后基因沉默的一种小反义RNA。科学,1999,28(4):950-952。10.1126 / science.286.5441.950。
长崎H, Itoh J, Hayashi K, Hibara K, satoto - nagasawa N, Nosaka M, Mukouhata M, Ashikari M,北野H,松冈M,水稻茎部分生组织的形成需要小干扰RNA的产生途径。美国国家科学促进会。中国生物医学工程学报,2007,32(3):444 - 444。10.1073 / pnas.0704339104。
刘波,李鹏,李霞,刘超,曹生,褚超,曹旭:OsDCL1功能缺失影响水稻microRNA积累,导致发育缺陷。植物生理学报,2009,39(1):394 - 394。10.1104 / pp.105.063420。
刘涛,李军,顾军,陆伟,郭超,李锐,肖凯:小麦argonaute基因家族成员TaAGO1的克隆及分子特征分析(英文)小麦l .)。中国生物医学工程学报,2011,35(6):444 - 444。
李勇,孟峰,尹军,刘辉,司志,倪忠,孙青,任军,牛辉:小麦6个MBD基因的分离及比较表达分析。生物化学,2008,1779(2):90-98。10.1016 / j.bbagrm.2007.09.004。
马建军,李建军,李建军,等。大麦核糖体DNA间隔长度多态性:孟德尔遗传、染色体定位和群体动态。美国国家科学促进会。中国生物工程学报,1994,18(4):444 - 444。10.1073 / pnas.81.24.8014。
Eswar N, Webb B, marti - renm MA, Madhusudhan MS, Eramian D, Shen MY, Pieper U, Sali A, Eswar N, Webb B, marti - renm MA, Madhusudhan MS, Eramian D, Shen MY, Pieper U, Sali A:基于MODELLER的蛋白质结构比较建模。中国生物医学工程学报,2007,32(2):559 - 559。
致谢
国家自然科学基金项目(31071410和30971771)资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
FM进行了生物信息学、基因克隆和系统发育分析。HJ、NL、YX和HL参与了基因的克隆和表达分析。KW参与了生物信息学分析。FM, JY, YL设计并准备了稿件。所有作者都阅读并批准了最终的手稿。
贾海英、凌娜、薛银蕾对这项工作也有同样的贡献。
权利和权限
开放获取本文由BioMed Central Ltd.授权发表。这是一篇开放获取的文章,在知识共享署名许可(https://creativecommons.org/licenses/by/2.0),允许在任何媒体上不受限制地使用、分发和复制,前提是正确引用原创作品。
关于本文
引用本文
孟芳,贾洪,凌,宁。et al。小麦2个Argonaute基因的克隆与特性分析小麦l .)。BMC Plant Biol13,18(2013)。https://doi.org/10.1186/1471-2229-13-18
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-18
关键字
- Argonaute
- 克隆
- 发展
- 基因表达
- 小麦(小麦l .)