摘要
背景
玉米是世界上最重要的作物之一。随着人口呈指数级增长,对粮食和饲料生产的需求不断增加,增加玉米(以及大米、小麦和其他谷物)的产量将至关重要。玉米籽粒发育主要从形态、激素反应和贮藏积累的角度来理解。这包括对胚胎发育和成熟过程中基因表达的各种研究,但直到最近,对胚胎基因表达的全球研究才成为可能。转录组分析是了解胚胎成熟的遗传基础的一个强有力的新工具。
结果
我们对一个用于转基因植株杂交的优良玉米种质系授粉后15、21和27天的正常成熟胚胎进行了转录组分析。通过这种方法分析了超过19,000个基因,挑战是选择对胚胎发育和成熟至关重要的基因子集进行初步分析。通过转录组分析,我们描述了与初级代谢途径、DNA合成、胚胎发生晚期蛋白和胚胎储存蛋白相关的基因的表达变化,并使用qRT-PCR证实了转录组中某些基因的转录水平。
结论
许多与胚胎成熟相关的基因已经被鉴定出来,其中许多基因在15 - 27 DAP的过程中表达水平发生变化。一组预期的参与初级代谢的基因被鉴定出来。此外,超过30%的转录本代表未注释的基因,还有许多功能有待发现。特别令人感兴趣的是储存蛋白基因,球蛋白-1,球蛋白-2和一个未知的cupin家族基因。在玉米中表达外源蛋白时,最常用的是球蛋白-1启动子,但这种cupin家族基因的表达量要高得多,可能是玉米胚胎中外源基因表达的更好候选基因。诸如此类的结果允许识别可能无法使用的候选基因和启动子。NCBI SRA中mRNA seq数据存档;登录号:ACC=SRA060791 subid=108584。
背景
我们实验室正在利用玉米胚胎表达外源蛋白用于工业应用[1- - - - - -3.].例如,一种内纤维素酶E1和一种外纤维素酶CBH I的基因已经转化为玉米,植物被回收,种子被收集。原始玉米组织培养种质Hi-II具有转化能力,但不具有农艺生产力[4].因此,必须将高表达转化子选育成优良种质,以提高田间性能,优化生产系统,实现商业化。在每种情况下,当原始转化子选育成优良种质时,通过选择可以获得更高的目标蛋白积累[3.,5,6].虽然这种现象在玉米中已被经验地观察到多次,但这种现象的机制尚不清楚。为了了解这一机制的遗传基础,了解参与正常胚胎发育的基因是至关重要的。胚胎成熟阶段对我们的研究至关重要,因为我们感兴趣的外源基因,即纤维素酶,是由球蛋白-1启动子表达的,球蛋白-1启动子是一种胚胎种子储存蛋白启动子,在胚胎发育的中期成熟阶段很活跃[7].
从形态、贮藏、蛋白质积累和激素反应等方面了解玉米种子发育过程[8- - - - - -12].例如,Kiesselbach [10他发表了最早的关于玉米发育的论文之一,包括配子、胚胎和种子。他主要是使用光学显微镜进行视觉研究,虽然在发育时间表上有优雅的细节,但亚细胞细节有限。此外,Kriz [11]通过显示球蛋白(1和2)是胚胎中最丰富的储存蛋白质,增加了储备的细节。这些蛋白质在成熟过程中形成,在萌发过程中降解,为生长的幼苗提供碳和氮源。在胚乳中,醇溶蛋白是主要的储存储备,其积累具有复杂的阶段[9].这些储备在萌发过程中也被降解,以供生长的胚胎使用。麦卡蒂(8]研究了玉米胎生突变体在胚胎发育成熟过程中对ABA和GAs的表型反应。ABA以其控制植物对胁迫的反应而闻名,包括干旱[13].同样,种子的干燥也受ABA的控制[8].总的来说,这些研究描述了一个定义种子发育和成熟的框架。然而,为了充分理解这些过程,我们必须利用现有的新技术,如转录组测序。
特别是胚胎,Kiesselbach [10]的研究结果表明,授粉后13-15天,胚仅具有胚轴(子叶)和分生组织的基本结构。到21dap,胚结构明显,茎尖和根尖可见。到25dap,胚结构完全形成,胚芽尖和胚根分别被胚囊鞘和胚囊鞘覆盖[10].根据Vernoud等人。[8]时,胚胎在15dap开始成熟,在21dap时储存量开始积累。我们利用这些早期研究设计了成熟胚胎的转录组测序实验。
以往的基因表达研究描述了种子发育和成熟的调控,包括细胞分裂、DNA复制、形态变化的诱导、储存蛋白质的积累和休眠的干燥[14- - - - - -17].Vernoud等人。[15]提供了玉米胚胎发生过程中基因表达的概述,包括突变体和克隆基因的描述。Lee等人对胚胎发育过程中的基因表达变化进行了微阵列分析。[14].他们的定制阵列包括来自EST文库的900个基因,这些基因被预测参与代谢和胚胎发生。他们发现了在种子发育的不同阶段表达的基因组,从早期的细胞分裂活动到储存储备合成和干燥。全基因组的microRNA基因也正在研究中,以最终确定它们在种子生长发育调节中的功能作用[17].
我们对一个优秀种质系的正常成熟胚胎进行了转录组分析,该种质系用于与Hi II种质资源中最初产生的转基因事件的杂交。在这项研究中,我们分析了15、21和27 DAP的胚胎转录组,以确定在这些时间点表达的基因。通过这种方法分析了超过19,000个基因,挑战是选择对胚胎发育和成熟至关重要的基因子集进行初步分析。我们描述了与初级代谢途径、DNA合成、胚胎发生晚期蛋白和胚胎储存蛋白相关的基因表达的变化。我们描述了通过转录组分析显示的变化,并通过qRT-PCR确认了转录组中一个基因子集的表达水平。
结果
胚胎收集
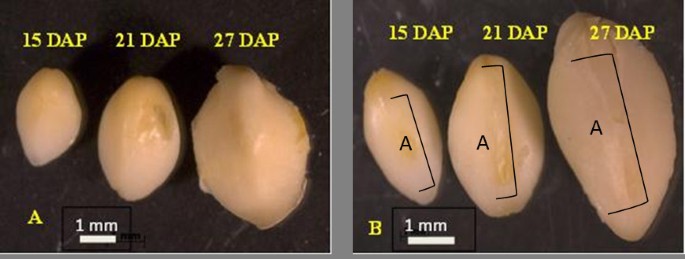
分析胚胎的精英自交系SP114是Stiff Stalk品种种质资源(usp# 6,252,148)。一个平行的兰开斯特品种被用作杂种的补充,但在这里没有描述。胚胎是在无菌条件下分离的,使用的是在16小时光照和8小时黑暗的温室中生长的植物的未成熟耳朵。结合基因型(SP114)和收获时间,我们选择在授粉后15、21和27天(DAP)三个时间点(S15、S21和S27)研究玉米胚胎基因的表达。我们对活跃成熟的最大范围内的基因表达变化感兴趣,这三个时间对应于胚胎成熟的早期、中期和晚期(图1).第一个观察到的是在这段时间内的巨大变化。显然,在15 - 27dap之间发生了很大的生长,以及胚胎轴的显著发育。我们想要分析胚胎中储存蛋白质积累的阶段,因为驱动转基因表达的启动子是球蛋白-1启动子,一种胚胎储存蛋白质。选择的三个时间点与球蛋白-1基因的表达模式相对应。已证明球蛋白-1基因的转录本在大约18dap开始积累,在24dap达到峰值,并在27dap开始下降[7].
玉米胚胎转录组概述
在每个采样时间,从单个耳朵的池化成熟胚胎中分离总RNA。RNA测序使用Illumina GA II/Solexa仪器(Tufts Core Facility, Tufts University School School, Boston, MA)进行。对72个核苷酸进行单端读取。这三个样本总共生成了5620万个原始reads,其中5290万个修剪后的reads可用于定位玉米参考基因组ZmB73AGPv1(表2)1).实际映射到参考基因组的修剪reads的数量为4290万,但这些reads中只有1040万(20%)唯一地映射到单个位点。几乎70%的唯一映射reads都是在已知的外显子内映射的。表中显示了每个样品的修剪和校准摘要1.
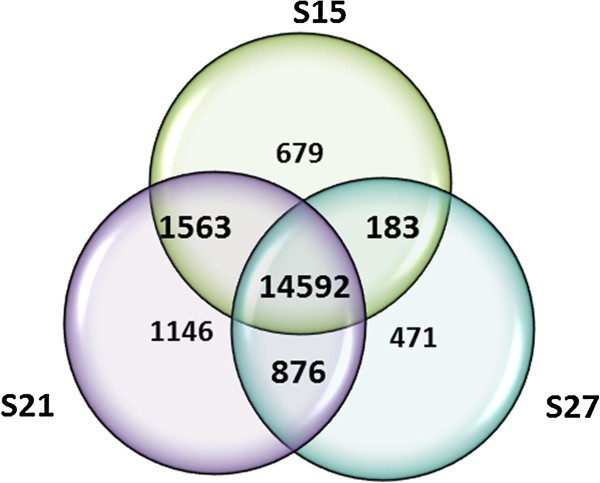
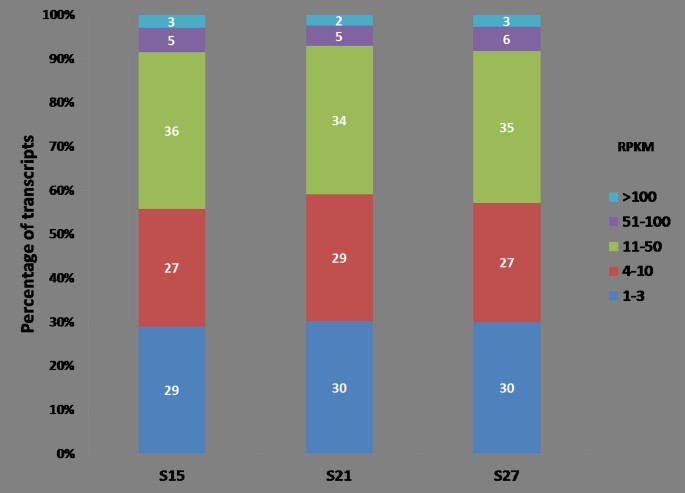
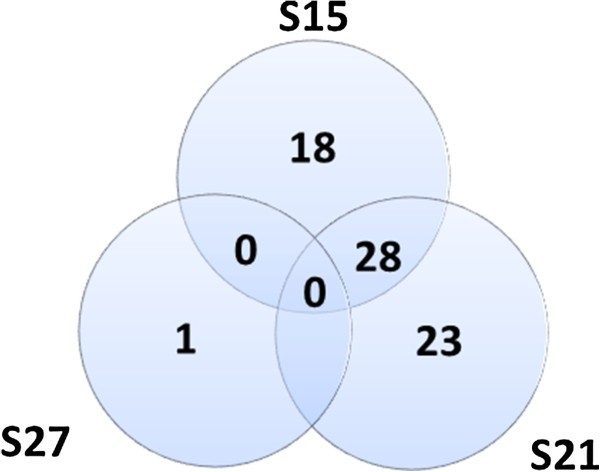
标准化reads被称为每千碱基外显子每百万映射reads (RPKM),用于估计整个胚胎成熟过程中表达的基因总数。RPKM方法纠正了总基因外显子大小的偏差,并对从每个样本中获得的每个文库的读取序列总数进行了归一化[18].RPKM值≥1用于估计表达的基因数量。在成熟胚胎中统计的基因总数为19510个,几乎占玉米转录组注释的60%。其中,17,017个(87%)表达于S15, 18,177个(93%)表达于S21, 16,122个(83%)表达于S272).数字2显示在每个阶段中唯一表达的基因数量,或与一个或两个其他阶段共享的基因数量。在这项研究中,14,592(75%)的表达基因在我们分析的胚胎成熟的所有三个阶段都有表达,12%的表达在单个阶段。共享基因表达的差异对于发现它们在胚胎成熟过程中如何变化很有意义。此外,单个基因之所以引起人们的兴趣,是因为它们在这一阶段具有潜在的重要性。胚胎成熟每个阶段的基因表达水平根据其RPKM值分为五类:极低(1-5 RPKM)、低(>5-10 RPKM)、中等(>10-50 RPKM)、高(>50-100)和极高(>100 RPKM)(图3.).只有一小部分基因属于高丰度(5-6%)和非常高丰度(2-3%)类别。
玉米胚胎基因的差异表达
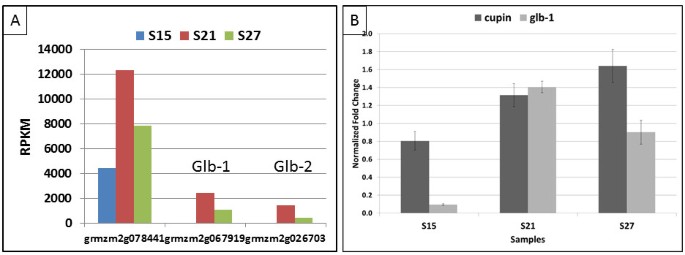
在成熟胚胎中表达的19510个基因中,差异表达的基因数量(p < 0.001和q < 0.001)为7124个,约占胚胎转录组的36.5%。在所有三个成对比较中,超过一半的差异表达基因在表达水平上至少显示了两倍的变化(表3.).对于表现出至少两倍变化的基因,几乎有同样多的基因上调和下调,除了S27和S15之间,下调的基因比例更高,56%比44%上调(表3.).差异表达基因的比例在S27和S15比较中最高(46%),在S21和S27比较中最低(36%)(补充文件)1:图S1)。我们用qRT-PCR方法验证了11个转录本的表达水平,发现两者高度相关(R2= 0.941)的mRNA-seq数据和qRT-PCR(附加文件2:表S1)。qRT-PCR最显著的结果是在储存蛋白质积累过程中cupin基因的表达水平高于球蛋白-1基因。(见图4详情)。
胚胎成熟遵循胚胎图案的形成和分化,从15dap开始,持续约30天[15].胚胎成熟的标志是生长,储备物质的积极积累和一些发育事件。生长的特征是前体生物合成、DNA合成和细胞分裂活跃的一段时间[19,20.],储备积累的特征是储存蛋白质、脂肪酸和淀粉的生物合成和沉积[21- - - - - -23].
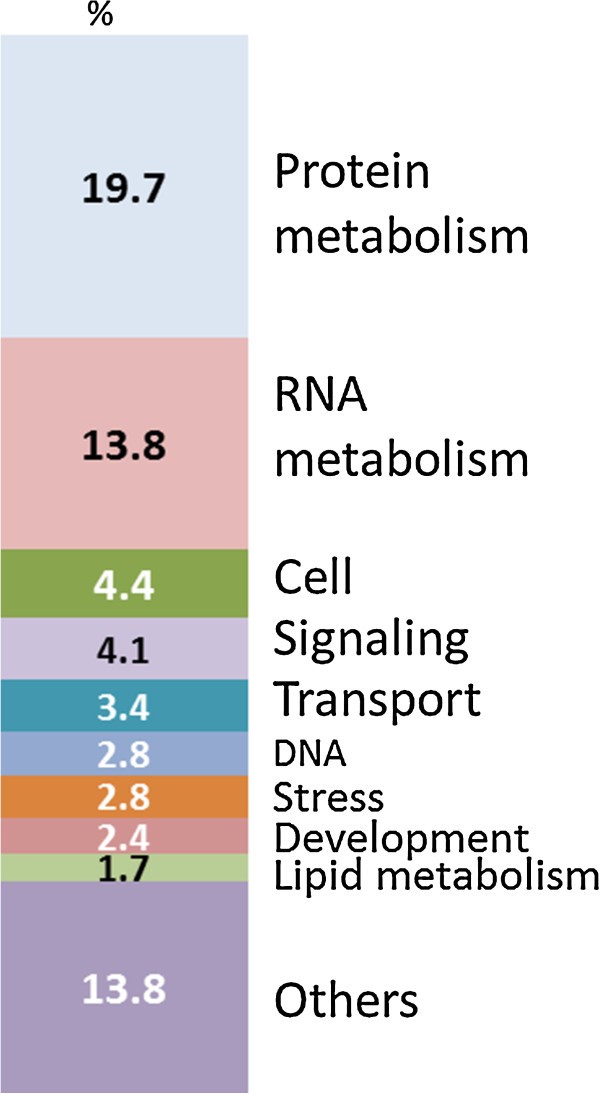
我们使用映射文件Zm_Genome_Release_09对SP114胚胎转录组进行Mapman注释[24,25将基因划分为35个功能类别(附加文件3.:表S2)。蛋白质代谢和RNA代谢是显示转录基因数量最多的两个功能类别。归一化读在前九个功能类别中的分布如图所示5不包括属于“未分配或未知”类别的30%(第35类)。
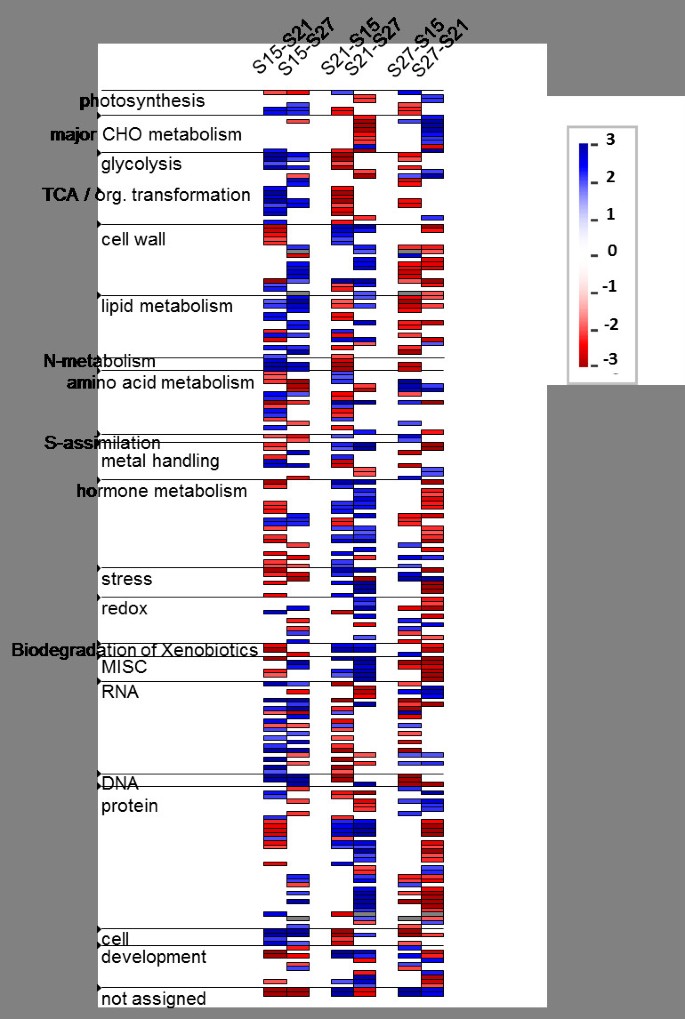
我们使用PageMan [26]以获得三种成对比较(S15 vs S21, S21 vs S27和S27 vs S15)中丰富功能类别的基于统计的概述。转录组数据加载到PageMan和Wilcoxon测试[27]被应用到每个类别。Wilcoxon检验比较了一个功能类别的基因与所有不在该类别的基因的log以2倍为基数的变化值。这揭示了某个特定类别的基因是否与所有其他基因表现不同。该分析通过删除没有显示出显著不同变化的类别,并使用假彩色热图式显示显示出显著变化的类别来显示上调或下调的类别,从而浓缩和压缩了基因。Wilcoxon检验结果的可视化显示显示,在三个成对比较中,每一个都丰富了特定的功能类别(图6).编码不依赖光的光合反应、糖酵解、TCA循环、脂质代谢、RNA和DNA合成以及细胞功能的酶的基因在胚胎成熟早期强烈上调(S15)。在21dap时,基因富集转移到功能类别,包括细胞壁,金属处理,激素代谢,应激,异种生物的生物降解和存储蛋白的合成。在胚胎成熟后期(S27),除了主要碳水化合物代谢、硫同化和氨基酸代谢亚类、RNA和蛋白质外,几乎所有功能类的基因都下调。
糖酵解
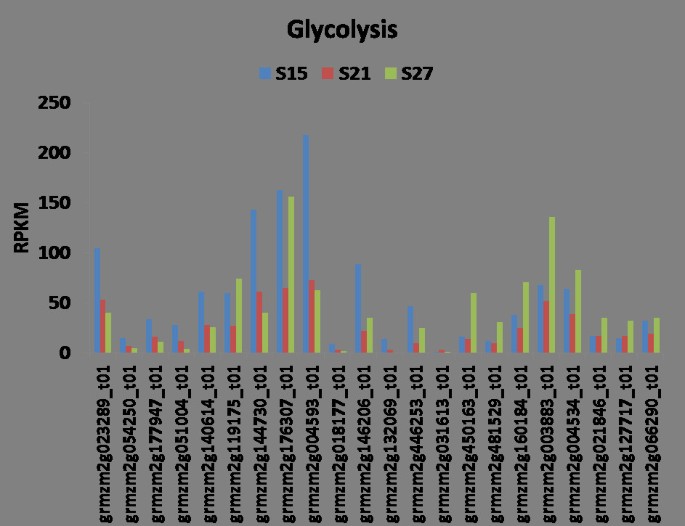
糖酵解和线粒体TCA循环是胚胎成熟的重要途径。它们提供积极生长所需的能量。分配到这两种途径的转录本显示了两组不同的基因(图7&8).第一组在胚胎成熟早期具有高表达,其特征是整体水平的转录物积累。第二组在27dap上调,其特征是转录物积累的相对水平要低得多。几乎所有糖酵解和TCA循环中的基因在21dap时的表达量都很低(图7和图8).
脂质代谢
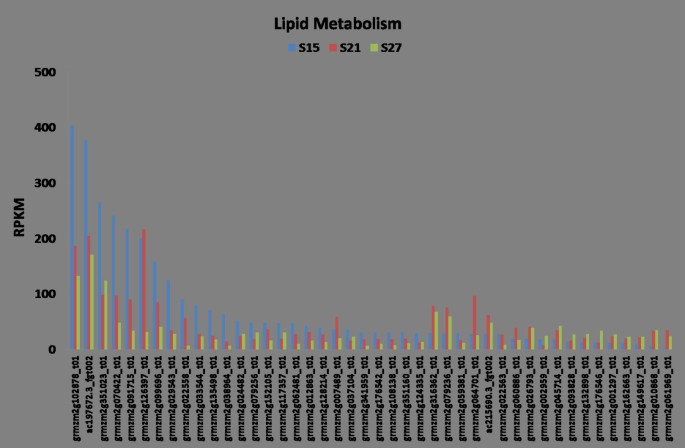
胚是果仁中产生活跃脂肪酸的部位。成熟时,脂质的积累可达玉米胚干重的50% [28].活性脂肪酸的生物合成发生在胚胎成熟的早期,并持续到约21dap。三组基因被分配到脂质代谢(图9).第一个由大约25个基因组成,在成熟早期被诱导。它们主要与脂肪酸合成和脂肪酸延伸有关。该组的特点是整体转录本积累高于其他两组。在21 DAP诱导的第二个较小的基因组包括编码与脂质修饰相关的各种酶的基因,如ACP去饱和酶和三酰甘油(TAG)的生物合成。第三组基因包括一些与鞘脂的生物合成有关的基因,鞘脂是由类异戊二烯如固醇和角鲨烯衍生的脂类,以及与脂质降解有关。
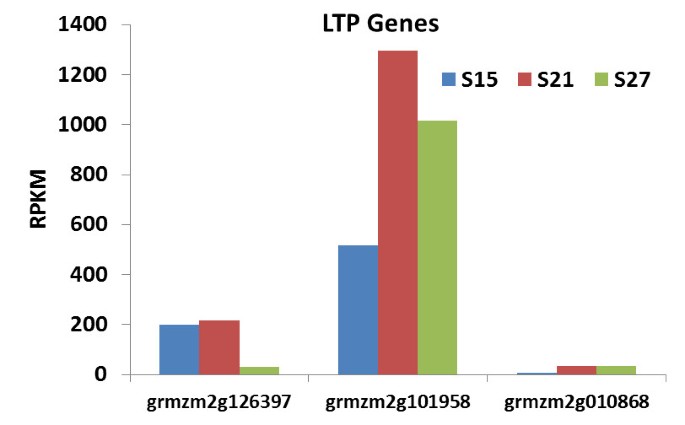
三个基因被分配到脂质转移蛋白(LTPs)。脂质转移蛋白已被证明可以促进在体外脂质在膜间的转移,被认为在膜生物发生中发挥作用[29].其中两个基因LTP1和LTP2的表达量非常高,但它们的表达模式彼此不同。LTP1转录本grmzm2g126397_t01的积累在15dap时很高,并持续到21dap,但在27dap时急剧下降(图10).另一方面,LTP2转录本的积累,grmzm2g101958_t01,在15 DAP开始处于高位,在21 DAP急剧上升,并维持到27 DAP。LTP3在低到中等水平表达,表现出与LTP2相似的表达模式。
DNA合成
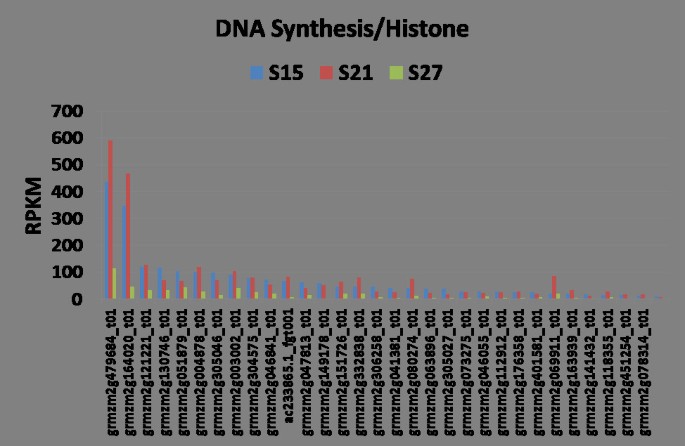
与DNA合成相关的基因,如那些控制染色质结构和组蛋白修饰的基因,在胚胎成熟的早期阶段是高的(图11),反映在S15和S21胚胎中大量的基因和相对较高的转录本水平,要么急剧下降27dap,要么在某些情况下根本不检测到。许多与染色质结构相关的基因在27dap时表现出高表达(附加文件)4:图S2),但都不是组蛋白基因。两个组蛋白基因,grmzm2g164020和grmzm2g479684分别编码组蛋白H1和组蛋白H4,在整个胚胎成熟期表达异常高,特别是在15和21 DAP(图11).
发展
属于库品家族存储蛋白发育类别编码的基因,包括球蛋白蛋白、油酸蛋白和胚胎发育后期丰富蛋白(LEA)(图4,数据12- - - - - -13).与参与DNA合成和细胞功能的基因相反,属于发育类的基因在15dap时表达量低,在21dap时达到峰值,然后随着胚胎进入后期成熟阶段略有下降。这类基因的转录本在21dap时积累到异常高的水平。
在胚胎中编码储存蛋白质的三个主要基因,grmzm2g067919 (glb1), grmzm2g026703 (glb2)和功能未知的cupin家族基因grmzm2g078441高表达(图4).这三个基因的转录本加在一起约占所有存储蛋白基因转录本的99%。球蛋白蛋白被认为是成熟胚胎中的主要储存蛋白[11].然而,转录组数据显示,它们的表达水平没有grmzm2g078441那么高。grmzm2g078441的转录水平即使在最低水平(15dap)也高于峰值水平glb1而且glb2(图4A).由于我们对外源基因的胚胎表达感兴趣,我们进行了定量PCR,从转录组中确认了这些关键基因的高水平。如图所示4B,未知cupin基因在15dap时表达量比glb-1高8倍,在21dap时约相等,在27dap时高65%。尽管qPCR结果与转录组结果不完全一致,但这个未知的cupin基因值得进一步分析。
脂质贮存蛋白是种子脂质体周围单层膜结构单元的组成部分。7个基因被分配到油苷中,其中3个在成熟胚胎转录组中高度代表(图12).最具代表性的油蛋白基因是grmzm2g337229 (oleosin 1),在21 DAP时,它在转录组中占所有油蛋白转录本的75%。
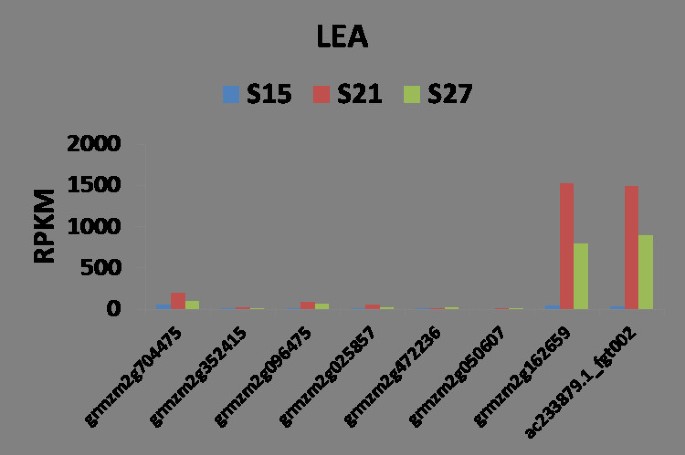
在我们转录组数据中的8个LEA基因中,有两个非常突出。这两个LEA转录本grmzm2g162659_t01和ac233879.1 .1_fgt002分别是拟南芥ATEM1和ATEM6的同源物。这两个LEA转录本从15dap到21dap显著增加(30- 50倍)。LEA转录本已被证明可被脱落酸(ABA)诱导,但不需要vp1[30.,31].其他LEA基因的转录本没有积累到一个显著的水平(图13).
基因本体论
对玉米胚胎转录本中表达的基因本体论(GO)术语的分析显示,在每个胚胎成熟阶段都有独特的GO术语以及在S15和S21之间共享的GO术语(图)14).S15特有的最显著丰富的GO术语是微管相关复合物、微管运动活性、微管运动、运动活性、微管基础过程、细胞骨架部分、脂肪酸代谢过程、脂肪酸生物合成过程和脂类生物合成过程。对于S21来说,最引人注目的独特GO术语是细胞成分和细胞成分组织,与蛋白质相关的生物过程和分子功能,如蛋白质寡聚化和蛋白质- dna复合物组装以及与应激反应相关的GO术语。只有一个GO项在S27中显著富集,即养分库活性。GO术语的描述列表在附加文件中给出5:表S3。
单核苷酸多态性的发现
玉米通常被认为是高度多态的,具有相对较高的SNPs频率。snp的高频率,加上测序技术的改进和高通量基因分型方法,如DNA芯片、等位基因特异性PCR和引物扩展方法,使snp成为特别有吸引力的遗传标记[32用于发现基因和鉴定种质。表格4显示了每条染色体发现的SNPs的数量。已发现的snp及其上下文序列见表4.snp数量最多的是染色体1和3,分别为4076个和3382个,而最少的是染色体8(717个)。
讨论
转录组学是分析任何生命系统中基因表达的有力工具。在一个实验中产生的数据将回答有关该系统的许多问题。这些数据还将产生一系列新的问题,可以使用这些相同的技术对相关样本进行更详细的探索。这一代大数据集的缺点是,对数据的分析需要强大的计算能力、多个软件包和基于价值判断的决策,即如何将数据解析成有意义且可以从生物学角度解释的包大小。
Mapman通路和Pageman程序使我们能够观察到与玉米胚胎成熟期相关的基因表达模式的变化。在15dap时,胚胎处于发育晚期和早期成熟之间的过渡阶段,因此许多与胚胎发育早期事件相关的基因的表达水平开始下降。例如,编码前体生物合成(脂质、氨基酸)、DNA和RNA合成以及细胞分裂和组织功能的基因在15dap比后期更有代表性(图)5).在21岁和27岁的胚胎成熟阶段,生长和储备积累等活动的基因表达增加,而早期阶段的基因表达下降。这些结果与Lee等人观察到的结果相似。[14]使用微阵列技术。与基于有限数量基因序列的微阵列相比,转录组学相对于微阵列的优势在于RNA测序的发现潜力的广度。
在我们的转录组中,几乎30%的基因没有指定的功能。这些基因中有许多是胚胎特定阶段所特有的,而另一些则在成对比较中表现出巨大的表达水平差异。其中一些未分类基因如grmzm2g409101和grmzm2g075042表达水平非常高,并与球蛋白-1基因协调表达(数据未显示)。进一步的功能分析将对这些基因在胚胎成熟或储存蛋白质积累中的作用提供更深入的了解,这些作用将通过突变和网络预测和操作来确定。我们的转录组数据还显示,总表达基因数在21 DAP时最高,随着胚胎成熟的增加而减少。这与Lee等人发表的数据一致。[14]和罗等人。[33],在玉米籽粒发育过程中个体mrna的积累在25dap后显著降低。21 DAP胚胎也代表了从15 DAP到27 DAP的过渡阶段,21 DAP与其他两个阶段有大量的基因共享。戴维森等人[34]发现在25个DAP B73胚胎中表达了22493个基因,非常接近我们在SP114成熟胚胎中发现的数量。
我们集中研究了几组基因,以展示我们的数据在理解胚胎成熟特定时间点的基因表达变化方面的效用。例如,编码组蛋白H1和H4的2个组蛋白基因在整个成熟过程中表现出非常高的表达,这表明这两个基因在这一关键时期发挥了非常重要的作用。相比之下,LEA基因被认为在耐旱性中发挥作用[35,36这可能解释了它们在胚胎成熟后期表达水平的增加。我们还对玉米胚胎中储存蛋白的积累感兴趣,因为了解这些基因的表达模式可能有助于我们了解这些基因是如何被调节的。我们最有趣的观察结果之一与cupin家族中编码胚胎内存储蛋白质的基因有关。在我们的生物工厂实验中,我们使用球蛋白-1启动子[7]驱动外源基因表达,用于工业酶生产[3.,5].生物工厂生产系统的目标是尽可能地增加外源蛋白的积累,以降低生产成本。因此,在图中观察到cupin家族中未知基因grmzm2g078441的转录本4,在15和27 DAP的表达量明显高于球蛋白-1基因(grmzm2g067919),提示其启动子可能是更有效的外源基因表达启动子。贝朗格和克里兹[7发现glb-1和2是胚胎中含量最多的蛋白质。问题是,如果是这样的话,为什么cupin转录本的表达水平似乎高于球蛋白。一种解释可能是,转录本不产生蛋白质,而是一种伪基因。另一种解释是,这些研究是在不同类型的玉米上进行的,1991年的研究是W64A和Va26,这次的研究是SP114。此外,球蛋白是由多基因家族产生的,可能不是由排列完全解释的,而cupin基因的转录本可能更高,尽管蛋白质没有那么丰富。这些可能性都可以在进一步的研究中加以解决。
“GO项目开发了三个结构化的受控词汇表(本体论),以一种与物种无关的方式描述基因产物的相关生物过程、细胞成分和分子功能”(http://www.geneontology.org/GO.doc.shtml).由于基因注释语言的一致性,这个有用的工具允许跨物种的基因功能比较。它还将基因表达类别的复杂性降低到3个功能类别,以便在特定的转录组中找到独特的功能。高度过度表示的GO术语以其活动而闻名,并描述了在特定时间和地点正在进行的基本功能。当GO分析应用于胚胎成熟转录组时,发现在每个阶段有一些术语是唯一的或共享的(图14).这些术语将在未来引发一些有趣的分析。
Alexandrovet al。[16],通过对玉米cdna的大规模测序,显示了与其启动子、转录起始位点预测因子和GC含量相关的mRNA特征的分布,尤其是在密码子的第三位。根据这些作者的分析,了解在特定时间特定组织中表达的基因是否在特定结构特征上存在差异将是很有趣的。例如,在一个或另一个组织或发育事件中,一个GC含量模式组中的基因组可能是首选的。
这里报道的结果为进一步研究单个基因或基因组提供了一个基线,这将阐明玉米胚胎如何成熟并开始停止休眠。这些类型的研究结果可用于增强基因,以提高粮食或饲料产量。在这个人口不断增长的世界,这样的结果将是至关重要的。
结论
许多与胚胎成熟相关的基因已经被鉴定出来,其中许多基因在15 - 27 DAP的过程中表达水平发生了显著变化。一组预期的参与初级代谢的基因被鉴定出来。特别令人感兴趣的是储存蛋白基因,球蛋白-1,球蛋白-2和一个未知的cupin家族基因。在玉米中表达外源蛋白时,最常用的是球蛋白-1启动子,但这种cupin家族基因的表达量要高得多,可能是玉米胚胎中外源基因表达的更好候选基因。诸如此类的结果允许识别可能无法使用的候选基因和启动子。转录组数据显示了参与胚胎发育和储存蛋白质积累的不同基因的表达模式。转录组数据也将成为玉米基因功能表征的宝贵资源,因为超过30%的转录本代表未注释的基因,还有许多功能有待发现。随着拟南芥和其他模式系统中基因的标注,其中一些新基因的鉴定将会完成。
方法
植物材料和生长条件
玉米(玉米L)自交系SP114 (usp# 6,252,148)在位于Metro Mix 200 (SunGro Horticulture, Bellevue, WA)无土培养基的阿肯色州生物科学研究所的温室中生长,并用Osmocote施肥。温度和光照周期设置为27°C至31°C,光照16小时,20°C至24°C,黑暗8小时。在授粉后15、21和27天(DAP)在无菌条件下分离胚胎,在液氮中冷冻,并在−80°C保存,直到用于RNA提取。
RNA提取
冷冻胚胎在液氮中研磨成细粉,并在TRI试剂溶液中均质(Ambion, Austin, TX)。按照Invitrogen公司(Carlsbad, CA)的RNA分离方案分离15,21和27dap的总RNA,并使用Qiagen RNAeasy Mini Spin柱(Qiagen, Valencia, CA)进行纯化。使用ND-1000分光光度计纳米滴系统(Thermo Scientific, Wilmington, DE)和RNA凝胶电泳(甲醛缓冲系统)测定总RNA的浓度和纯度。
cDNA文库构建及转录组测序
cDNA文库的构建和转录组的测序被承包给位于马萨诸塞州波士顿的塔夫茨大学医学院的塔夫茨核心设施。cDNA文库按照制造商手册(Illumina, Inc, San Diego, CA)中概述的程序构建。转录组测序使用Illumina基因组分析仪II /Solexa (Illumina, Inc, San Diego, CA)完成。21 DAP (S21)的cDNA文库在一个Illumina流细胞通道中运行,15 DAP (S15)和27 DAP (S27)的cDNA文库分别与一个相同年龄的转基因样品配对。S15的修正读数为1160万,S21为1880万,S27为1540万。单端读取的长度范围从64 (S15和S27)到66 (S21)碱基。
基因组测序和数据分析
RNA-seq的映射读取
通过定制的Data2Bio (Ames, Iowa)修剪脚本对原始读取进行修剪以去除低质量的核苷酸。GSNAP (Genomic Short-read Nucleotide Alignment Program, version 2010-07-37) [37],它允许有间隙的比对,包括内含子跨越比对,被用于将修剪过的reads映射到参考基因组。后续分析只使用参考基因组中唯一最佳匹配且每36 bp不匹配≤2个,尾数≤3 bp的reads。每个基因的读取深度是基于所绘制的reads的坐标和基因在参考基因组中的注释位置来计算的。
所有reads均与参考基因组对齐玉米ZmB73AGPv1;线粒体(AY506529.1)和叶绿体(X8563.2)。转录组数据的比对和初步分析由Data2Bio (Ames, Iowa)完成。进一步的分析在内部使用CLC Genomics Workbench (Cambridge, MA)进行。地图和路径的可视化是使用公开的软件进行的,包括Mapman (http://mapman.gabipd.org/) [38]及综合基因组观察器(http://www.broadinstitute.org/software/igv/) [39].
通过Fisher精确测试鉴别差异表达基因
归一化是使用一种方法进行的,该方法纠正了由RNA组成引入的偏差和两个样本中映射reads总数的差异[40].归一化读计数用于计算折叠变化(FC)和统计显著性。Fisher精确检验用于检验原假设,即给定基因的表达在两个样本之间没有差异。只有两个样本中至少有50个基因被检测。通过校正多重测试进一步筛选被鉴定为差异表达候选基因[41],错误发现率为0.1% (q值)。统计学上显著的变异可能是两个样本之间基因表达的生物学或技术变异的结果。
基因本体(GO)分析
软件gotoools (https://github.com/tanghaibao/goatools)进行GO分析。根据费雪的精确测试,确定了某些GO术语的过度和不足代表性。两个多重校正控制(Bonferroni和置换控制错误发现率)[42]被执行。
单核苷酸多态性的发现
通过与参考基因组ZmB73AGPv1的比较,snp被称为。通过基因组短读核苷酸比对程序(GSNAP)识别的序列变体被进一步筛选,以使用唯一映射的reads识别snp。如果SNP位点有≥3个reads支持,最小SNP基础质量值≥15,并且所有reads中的罕见等位基因覆盖率必须超过0.8,则称为SNP位点,这严格控制了可能由序列错误和谬误引起的虚假SNP发现。
实时聚合酶链反应
为了验证RNA-seq结果,采用SYBR green (Bio-Rad)和CFX384 real-time PCR检测系统(Bio-Rad, Hercules, CA)进行实时定量PCR。qPCR的SYBR®绿色引物使用AlleleID®7软件设计(Premier Biosoft, Palo Alto CA)。为了确保目标特异性,基因序列在非冗余数据库(GenBank, NCBI)中进行blast,以确定与其他序列的交叉同源性。为了避免在这些区域设计引物,对基因序列进行二级结构分析。引物设计用于获得75 - 200 bp之间的产物。引物由加州圣地亚哥的Integrated DNA Technologies, Inc.合成。采用SYBR Green检测化学方法进行两步RT-qPCR。cDNA由1 μg总RNA和寡核苷酸(dT)引物合成,使用iScript™Select cDNA Synthesis kit (Bio-Rad),遵循制造商的程序。定量实时PCR的总体积为5 uL,其中模板0.5 uL,主混合4.5 uL。采用以下扩增程序:95°C变性30 s, 40次扩增(95°C 10 s, 60°C 30 s)和熔解曲线程序(从65°C到95°C,增加0.5°C 5 s)。3个内参基因(附加文件2:表S1)用于归一化表达,然后将这些值与每千碱基外显子每百万映射reads (RPKM)估计值进行比较。所有PCR反应都在384孔全裙PCR板上重复进行(USA Scientific, Ocala, FL)。
作者的信息
EEH研究玉米胚胎外源基因表达已有15年,并对胚胎发育感兴趣,因为它与蛋白质积累有关。
缩写
- 衣冠楚楚的:
-
授粉后几天
- 存在:
-
定量实时聚合酶链式反应
- 阿坝:
-
脱落酸
- 遗传算法:
-
Gibberllic酸
- GSNAP:
-
基因组短读核苷酸比对程序
- 舰队指挥官:
-
Fold-changes
- 走:
-
基因本体论
- RPKM:
-
每千碱基的外显子每百万的映射读取数
- 柠檬酸:
-
三羧酸循环
- 标签:
-
三酰甘油
- LTP:
-
脂质转移蛋白
- 李:
-
晚期胚胎发生丰富
- glb2:
-
球蛋白2基因
- SNP:
-
单核苷酸多态性
参考文献
- 1.
Clough RC, Pappu K, Thompson K, Beifuss K, Lane J, Delaney DE, Harkey R, Drees C, Howard JA, Hood EE:来自白腐真菌黄孢Phanerochaete chrysosporium的锰过氧化物酶具有酶活性,并在转基因玉米种子中积累到高水平。植物学报,2006,4(1):53-62。10.1111 / j.1467-7652.2005.00157.x。
- 2.
Hood EE, Bailey MR, Beifuss K, magallane - lundback M, Horn ME, Callaway E, Drees C, Delaney DE, Clough R, Howard JA:一种真菌漆酶基因在转基因玉米中高水平表达的标准。植物生态学报,2004,19(2):379 - 379。10.1046 / j.1467-7652.2003.00014.x。
- 3.
Hood E, Love R, Lane J, Bray J, Clough R, Pappu K, Drees C, Hood K, Yoon S, Ahmad A:亚细胞靶向是转基因玉米种子中纤维素酶蛋白高水平积累的关键条件。植物生态学报,2007,26(6):389 - 397。10.1111 / j.1467-7652.2007.00275.x。
- 4.
Armstrong C, Green C, Phillips R: II型培养形成反应高的种质资源的开发与利用。农学通报,1998,25(3):397 - 397。
- 5.
胡德华,胡德华,胡德华,王晓明,等:玉米种质资源重组蛋白积累的研究进展。植物科学进展,2012,30(4):344 - 344。10.1111 / j.1467-7652.2011.00627.x。
- 6.
Streatfield S, Mayor J, Barker D, Brooks C, Lamphear B, Woodard S, Beifuss K, Vicuna D, Massey L, Horn M,等:玉米抗肠毒素大肠杆菌亚单位疫苗的研制。中国生物医学工程学报,2002,38(1):11-17。10.1079 / IVP2001247。
- 7.
杨晓明,王晓明,王晓明,等。玉米球蛋白-1基因等位基因多态性的分子分析。遗传学报,1998,29(3):863-872。
- 8.
种子发育中成熟和萌发途径的遗传控制和整合。植物化学学报,2004,19(4):531 - 531。10.1146 / annurev.pp.46.060195.000443。
- 9.
高崎K,张志刚,张志刚,张志刚:转基因烟草胚乳蛋白体中玉米[γ]-Zein对[α]-Zein的隔离和稳定。植物细胞学报,2012,8(12):2335-2345。
- 10.
基塞尔巴赫TA:玉米的结构和繁殖。中国农业勘定,1949年,96-185103272版
- 11.
Kriz AR:由玉米编码的胚胎球蛋白的特征Glb基因。生物化学学报,1998,27(3-4):239-251。
- 12.
吴艳梅,胡德威,胡德威,荣荣荣:玉米胚乳中玉米蛋白基因表达的基因组学分析。植物细胞学报,2001,13(10):2297-2317。
- 13.
Nakashima K, Fujita Y, Katsura K, Maruyama K, Narusaka Y, Seki M, Shinozaki K, Yamaguchi-Shinozaki K: ABI3-和aba反应基因的转录调控包括RD29B而且RD29A在种子,发芽胚胎和幼苗中拟南芥.植物分子生物学,2006,60(1):51-68。10.1007 / s11103 - 005 - 2418 - 5。
- 14.
李建民,李建民,李建民,李建民。玉米胚发育过程中基因表达变化的DNA序列分析。中国生物医学工程学报,2002,2(1):13-27。10.1007 / s10142 - 002 - 0046 - 6。
- 15.
杨晓明,杨晓明,杨晓明,Depège玉米胚胎发育研究进展。Maydica, 2005, 5万
- 16.
张华,张海华,张永平,陆玉萍,鲍克J, Flavell R,等:基于cDNA序列的玉米基因序列分析。植物分子生物学,2009,69(1):179-194。10.1007 / s11103 - 008 - 9415 - 4。
- 17.
张玲,贾建明,Kumari S, Stein JC,刘智,Narechania A, Maher CA, Guill K, McMullen MD, Ware D:玉米MicroRNA基因的全基因组特征。公共科学学报,2009,5 (11):e1000716-10.1371/journal.pgen.1000716。
- 18.
Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B:利用RNA-Seq绘制和量化哺乳动物转录组。光子学报,2008,5(7):621-628。10.1038 / nmeth.1226。
- 19.
Grafi G, Larkins BA:玉米胚乳的核内复制:M相促进因子抑制和S相相关激酶的诱导。科学通报,1995,29(4):344 - 344。10.1126 / science.269.5228.1262。
- 20.
koules RV, Phillips RL:玉米胚乳核在籽粒发育过程中的DNA扩增模式。自然科学进展,2004,26(3):379 - 379。10.1073 / pnas.82.20.7010。
- 21.
王晓明,刘志刚,刘志刚,刘志刚,王志刚,王志刚。拟南芥种子发育过程的基因芯片分析。中国农业科学,2004,27(4):457 - 457。10.1104 / pp.124.4.1570。
- 22.
赫尔曼EM,拉金斯BA:蛋白质储存体和液泡。植物学报,1999,11(4):601-614。
- 23.
李志刚,李志刚。脂质生物合成。中国生物医学工程学报,2004,27(3):357 - 357。
- 24.
李萍,彭娜娜,王玲,斯杨,陶斯塔,Kebrom TH, Provart N, Patel R, Myers CR,等:玉米叶片转录组的发育动态。植物学报,2010,42(12):1060-1067。10.1038 / ng.703。
- 25.
Hayano-Kanashiro C, Calderón-Vázquez C, Ibarra-Laclette E, Herrera-Estrella L, Simpson J:干旱胁迫和恢复灌溉对3个墨西哥地方玉米基因表达和生理响应的影响分析。公共科学学报,2009,4 (10):e7531-10.1371/journal. peer .0007531。
- 26.
Usadel B, Poree F, Nagel A, Lohse M, Czedik-Eysenberg A, Stitt M:使用MapMan可视化和比较植物组学数据的指南:玉米作物物种的案例研究。植物细胞环境,2009,32(9):1211-1229。10.1111 / j.1365-3040.2009.01978.x。
- 27.
Wilcoxon F:排名方法的个体比较。植物学报。1945,1(6):80-83。10.2307 / 3001968。
- 28.
鲍曼VB,黄V,黄AH:玉米种子发育过程中脂质体蛋白基因的表达。空间、时间和荷尔蒙的调节。中国生物医学工程学报,1998,26(3):344 - 344。
- 29.
Kader JC:植物细胞中磷脂、半乳糖脂和脂肪酸的细胞内转移。中国生物医学工程学报,2000,26(4):344 - 344。
- 30.
Pla M, Gómez J, Goday A, Pagès M:脱落酸应答基因rab28在玉米胎生突变体中的调控。Mol Gen Genet MGG。1991, 23(3): 394-400。10.1007 / BF00280296。
- 31.
胡晓明,李志刚,李志刚,李志刚:玉米胚胎发育后期蛋白质的积累:脱落酸和胎生1基因产物的作用。植物营养学报,2002,23(2):357 - 357。10.1104 / pp.99.2.607。
- 32.
battamakki D, Rafalsk A:植物单核苷酸多态性标记的发现和应用。植物基因分型:植物的DNA指纹。编著:RJ H. Wallingford: CABI Publishing, 2001, 179-192。
- 33.
罗敏,刘娟,李仁仁,郭伯忠:玉米自交系Tex6发育过程中籽粒基因表达谱的研究。中国植物学报,2008,27(6):569-578。10.1111 / j.1439-0523.2008.01538.x。
- 34.
Davidson RM, Hansey CN, Gowda M, Childs KL, Lin H, Vaillancourt B, Sekhon RS, de Leon N, Kaeppler SM, Jiang N,等:RNA测序在玉米生殖转录组分析中的应用。植物学报,2011,4(3):191-203。10.3835 / plantgenome2011.05.0015。
- 35.
王志强,王志强,王志强,等:棉花中6种Lea蛋白及其基因的序列分析。植物分子生物学,1988,11(3):277-291。10.1007 / BF00027385。
- 36.
Dure L, Crouch M, Harada J, Ho T- hd, Mundy J, Quatrano R, Thomas T, Sung ZR:高等植物LEA蛋白常见氨基酸序列域。植物分子生物学,1989,12(5):475-486。10.1007 / BF00036962。
- 37.
Wu TD, Nacu S:短读中复杂变异和剪接的快速和耐snp检测。生物信息学,2010,26(7):873-881。10.1093 /生物信息学/ btq057。
- 38.
Thimm O, Bläsing O, Gibon Y, Nagel A, Meyer S, Krüger P, Selbig J, Müller LA, Rhee SY, Stitt M: mapman:一种用户驱动的工具,可以将基因组学数据集显示在代谢途径和其他生物过程的图表上。植物学报,2004,37(6):914-939。10.1111 / j.1365 - 313 x.2004.02016.x。
- 39.
Thorvaldsdóttir H, Robinson JT, Mesirov JP:整合基因组Viewer (IGV):高性能基因组数据可视化和探索。生物工程学报,2012,1:15-doi:10.1093/bib/bbs017。
- 40.
Robinson MD, Oshlack A: RNA-seq数据差异表达分析的标度归一化方法。中国生物工程学报,2010,11:R25-10.1186/gb-2010-11-3 r25。
- 41.
Benjamini Y, Hochberg Y:控制错误发现率:一种实用而强大的多重测试方法。中国统计杂志,1995,第5期:379 - 379。
- 42.
Zeeberg B, Qin H, Narasimhan S, Sunshine M, Cao H, Kane D, Reimers M, Stephens R, Bryant D, Burt S,等:高通量GoMiner,一种用于多微阵列实验解释的“工业强度”集成基因本体工具,应用于常见可变免疫缺陷(CVID)研究。中国生物医学工程学报,2005,30(6):366 - 366。
确认
Data2Bio, Pat Schnable,吴伟;工厂动力生产(P3)中心由国家科学基金会和阿肯色州科学技术管理局通过RII:阿肯色州资产计划(AR EPSCoR) I (EPS-0701890)和II (EPS-1003970)资助。这项研究得到了国家研究资源中心(5P20RR016460-11)和国家普通医学科学研究所(8P20GM103429-11)的国家卫生研究所的部分资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们之间没有利益冲突。
作者的贡献
KT:实验设计、执行及数据分析、稿件准备;DVR:部分PCR和稿件准备;SD:部分PCR和手稿准备;DJ:协助数据分析;XH:协助数据分析;JH:协助项目开发;EH:项目开发、设计和融资。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2012_1253_moesm4_esm.png
附加文件4:图S2。:Gene distribution in each functional category and number of transcripts detected. (PNG 98 KB)
作者提交的图片原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
赵国强,李志强,d.v.,德瓦雅,S.P.et al。玉米胚成熟的转录组分析。BMC植物生物学13,19(2013)。https://doi.org/10.1186/1471-2229-13-19
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/1471-2229-13-19
关键字
- 转录组
- 玉米
- 胚胎
- 成熟
- 中存在