摘要
背景
草莓属vesca,一种二倍体草莓,通常被称为高山草莓或林地草莓,是一个多功能的实验植物系统和蔷薇科的一个新兴模型。一个祖先f . vesca八倍体甜点草莓(F。×ananassa),现有的基因组与蔷薇科其他重要的商业成员,如苹果和桃子,表现出同步性。为了在特定的组织和细胞类型上提供花器官和果实发育的分子描述,从自交系的花和发育早期的果实组织中提取rnaf . vesca利用Illumina HiSeq2000对YW5AF7株进行cDNA文库测序。为了便于访问和挖掘这个二维(阶段和组织)转录组数据集,我们开发了一个基于web的数据库——草莓基因组资源(SGR)。
描述
SGR是一个包含样本描述、样本统计、基因注释和基因表达分析的web可访问数据库。该信息可以从一个基于网络的界面公开访问http://bioinformatics.towson.edu/strawberry/Default.aspx.SGR网站为数据库中存储的所有数据提供用户友好的搜索和浏览功能。用户可以使用基因ID或描述搜索基因,或者通过输入不同的比较参数获得差异表达的基因。搜索结果可以下载表格格式兼容微软excel应用程序。通过基因组浏览器可以显示单个基因和外显子/内含子结构的对齐阅读,方便个人用户重新注释基因。
结论
SGR数据库的建立是为了促进林地草莓花和果实转录组数据的传播和数据挖掘。它使用户能够以不同的方式挖掘数据,以研究生殖发育的不同途径或生物过程。
背景
草莓是世界公认的美味和健康促进食品。草莓是美国重要的水果作物,年市场价值超过10亿美元(加州草莓委员会,http://www.calstrawberry.com).商业上种植的甜点草莓草莓属×ananassa是起源于西半球的两个八倍体物种的异源八倍体杂种。的二倍体f . vesca,被认为对人类基因组有贡献F。×ananassa,通常被称为林地草莓或高山草莓。f . vesca有一个小的测序基因组(240 Mb),一个小的身材和短的种子到种子周期,以及有性繁殖和植物性繁殖的能力,所有这些都有助于它作为一个属的参考植物的有用性[1].此外,f . vesca是可变形的,根癌土壤杆菌[2,3.因此,这种小型多年生植物也是蔷薇科功能基因组分析的理想候选,蔷薇科包括许多重要的果树作物,如苹果、桃子和樱桃。虽然蔷薇科植物和果实类型有广泛的多样性,但草莓、桃子和苹果基因组之间仍有足够的同源性[4),这样f . vesca可以被认为是一个理想的系统,用它来研究花的发育,并开始了解不同的水果在家族内的发展基础。
由于草莓果实的经济价值,早期对果实的分子研究主要集中在经济上重要的过程,如风味和香气的形成、营养属性、硬度和成熟等[5].相比之下,对草莓花器官和早期果实发育的分子调控知之甚少。从农业的观点来看,受精后的果实发育需要适当的花器官和配子的形成。从生物学和进化的基本观点来看,孢子体和配子体细胞之间以及瘦果和花托之间的信号传递对种子的成熟、果实的成熟和种子的传播至关重要。下一代测序(Illumina RNA Seq)用于分析早期果实发育的转录组,包括5种果实组织类型和从开花到扩大果实的5个发育阶段[6].最终目标是让科学家研究水果发育的分子机制。目前SGR中有来自50个文库(每个组织类型有两个副本)的RNA-seq数据,这些数据将随着花朵发育转录组等进一步数据的获得而更新。草莓生殖发育的广泛的二维(组织和阶段)数字数据集可以被任何研究者挖掘,是一种有价值的资源。
结构和内容
SGR数据库的设计、实现和托管使用Microsoft SQL Server 2008 R2企业版。采用Microsoft Visual Studio 2008进行网页的设计与实现。NET framework 2.35与c#编程语言。SGR数据库和网站都托管在位于美国马里兰州巴尔的摩陶森大学的同一个网络服务器上。此服务器运行Microsoft Windows server 2003和Internet Information Services (IIS V6.0)。
SGR数据库存储描述每个复制的研究样本,每个样本的数量的读取,读取的过滤率,质量的一致性读的基因组,读取的对齐的基因,基因功能信息,基因本体论(去)作业,植物本体(PO)作业,和基因表达分析使用两种不同的工具,DEGseq [7]及DESeq [8].
Gbrowse 2.0 [9]以图形方式显示了基因组序列,并带有显示每个样本的预测基因模型和所有研究样本的短读的轨迹。七个f . vesca伪分子组装文件和非锚定支架文件从Rosaceae Genome Database, GDR,http://www.rosaceae.org/species/fragaria/fragaria_vesca/genome_v1.1),并合并在一起展示,代表基因组的七个连锁组。GeneMark杂交基因模型的GFF3文件(ftp://ftp.bioinfo.wsu.edu/species/Fragaria_vesca/Fvesca-genome.v1.1/genes/fvesca_v1.1_genemark_hybrid.gff3.gz)下载并导入到MySQL服务器5.1.67。使用samtools将所有对齐输出文件转换为GBrowse可接受的格式[10,从而使它们被视为单独的轨道。GBrowse和MySQL托管在运行Red Hat Enterprise Linux server 6.4版的Linux服务器上。
效用和讨论
SGR是首个用于林地草莓果实发育早期基因组规模转录组分析的网络可访问数据库。该网站向公众提供转录组数据,用于分析所有已测序组织发育过程中伴随形态变化的潜在分子变化,如[6].
RNA-sequencing分析
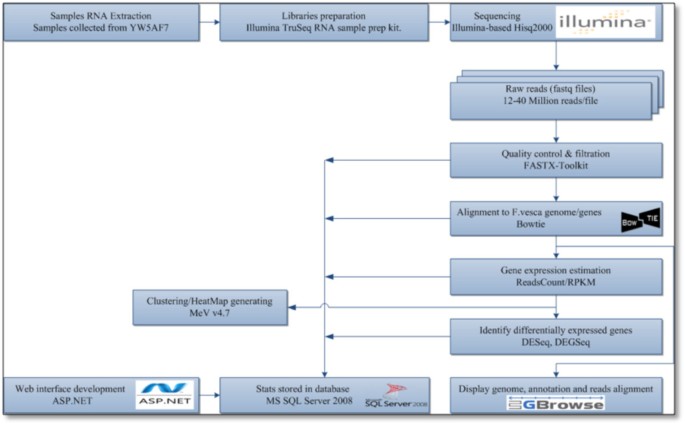
RNA-Seq在深入的转录组研究中迅速胜过微阵列。利用Illumina Hiseq 2000平台从25个复制库中每个库获得12 ~ 4000万51 bp的单端读,共获得约70 Gb测序数据。短读被映射到f . vesca栽培夏威夷4 × 4基因组,已测序f . vesca品种(1].绘制图谱后,对基因组中已知基因的读取次数进行计算。然后将这些计数归一化,以考虑到单个基因的大小和总样本量。为此,我们使用RPKM(每千碱基每百万映射读的读数)方法。数字1说明了用于RNA-Seq分析的生物信息学管道。差异表达基因和折叠变化是基于DESeq和DEGSeq的阶段或组织间比较得出的[7,8].
苹果真正的数据库
一个关系数据库草莓基因组资源(SGR)被创建来存放RNA-seq数据并允许用户查询。SGR存储样本信息,包括详细的样本描述、读取统计、比对结果、基因表达原始计数和标准化数据(RPKM)。不同表达的基因和折叠变化可以由用户检索,并指定特定的成对比较。
Web界面
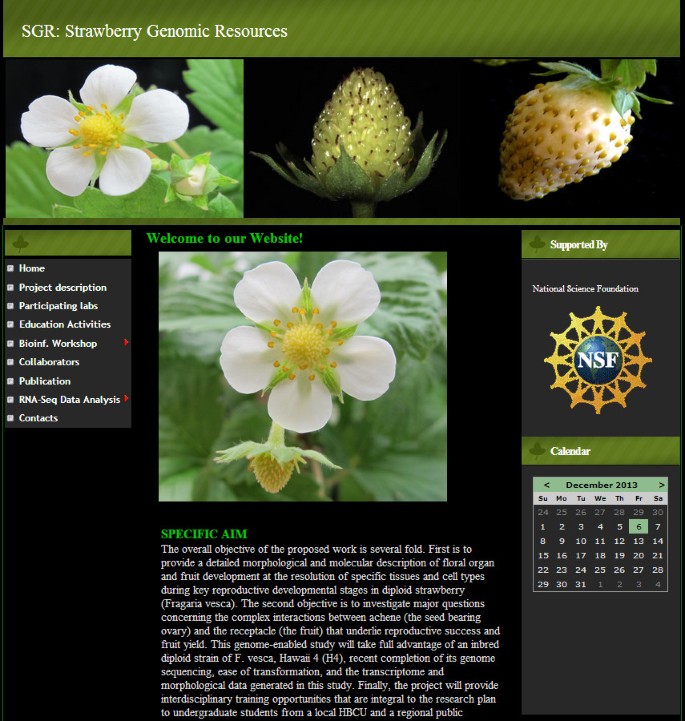
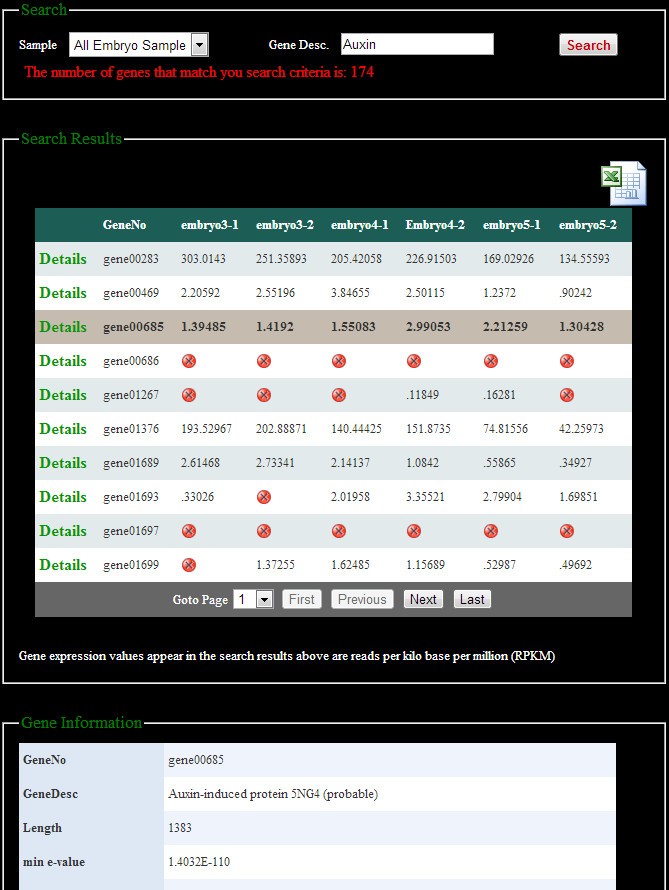
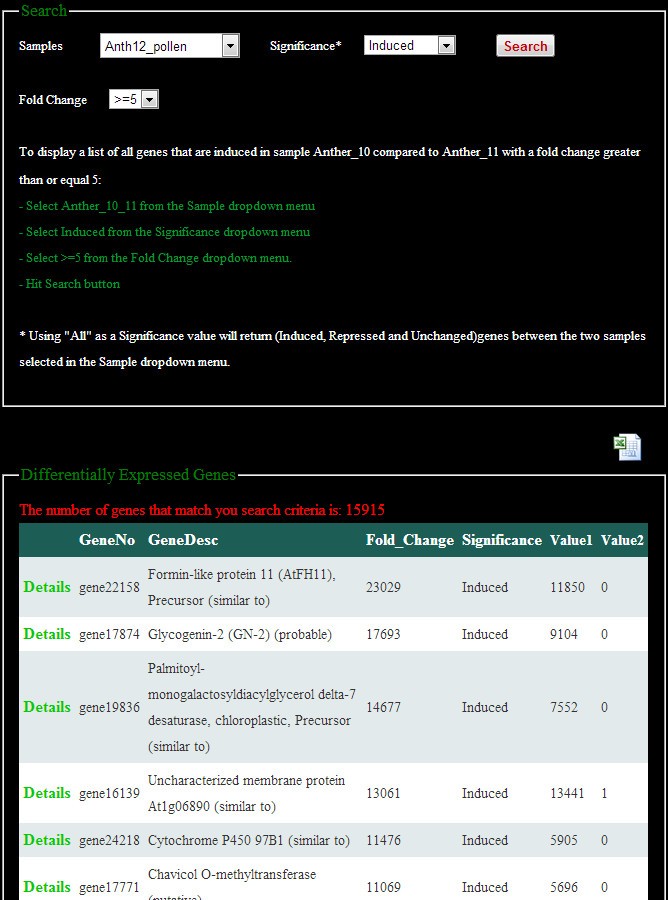
我们开发了一个用户友好的网站,允许快速远程访问存储在SGR的所有数据。网站主页提供了项目描述,并有链接到网站的所有其他部分(图)2).该网站提供搜索、浏览、下载分析结果等功能。用户可以通过示例名称浏览基因表达式数据或搜索特定的基因(图3.).搜索结果可在用户的电脑上以表格形式下载。该网站还具有通过精确或部分基因描述搜索基因的功能。在差异表达式页面(图4)用户可以选择任意两个感兴趣的样本进行比较,并通过DEGSeq和DESeq两种不同的工具获得差异表达的基因名称和折叠变化。用户可以通过选择差异的显著性(通过p值)以及基因表达趋势(诱导或抑制)来过滤他们的搜索。Fold change也可以作为一种减少返回基因数量的切断方法。
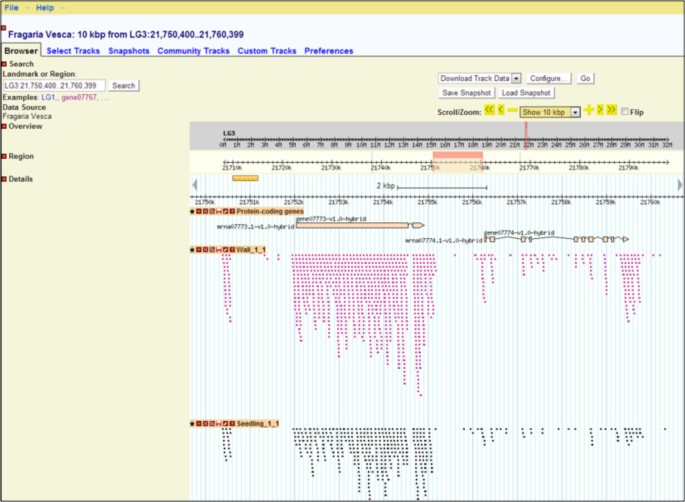
SGR GBrowse支持大多数基因组浏览器功能,包括定性和定量跟踪。GBrowse版本2.0支持下一代测序。我们部署了GBrowse来可视化短读比对输出文件与参考基因组和杂交genmark预测基因。GBrowse允许通过读比对和读计数直接洞察基因表达水平和基因结构(图)5).
未来的视角
来自发育中的草莓花组织和器官的转录组数据,包括雄性和雌性器官和配子体,将被添加到数据库中。这些数据将帮助科学家们描述草莓花发育的分子过程,以及蔷薇科密切相关成员(如树莓和玫瑰)的类似过程。
此外,转录组数据目前被用于改进转录基因的注释f . vesca基因组。目前的基因模型是基于GeneMark的混合预测[1]或Gnomon (NCBI Refseq组件GCF_000184155.1)。通过数据库中大量的转录组数据,我们可以得到更准确的基因模型。最后,我们计划鉴定和显示不同f . vesca其中,夏威夷4 × 4 (Hawaii 4 × 4)和YW5AF7 (YW5AF7)是研究花和果实发育的亲缘材料。
结论
SGR为研究草莓和其他植物的科学家提供了重要的基因组资源蔷薇科其中包括许多重要的水果作物。此外,该数据库将有助于调查植物生殖发育的基本问题。这些数据、分析和工具将扩大我们识别调控植物繁殖和作物产量的关键基因和途径的能力。
可用性和需求
该资料库可于以下网址免费索取:http://bioinformatics.towson.edu/strawberry/.
SGR GBrowse可通过以下网址自由访问http://mb3.towson.edu/gb2/gbrowse/F.Vesca/.
SGR和SGR GBrowse都可以通过任何标准的互联网浏览器使用。
参考文献
- 1.
Shulaev V, Sargent DJ, Crowhurst RN, Mockler TC, Folkerts O, Delcher AL, Salama DY:林地草莓(Fragaria vesca)的基因组。acta photonica sinica, 2010, 43(2): 109-116。
- 2.
关键词:草莓(Fragaria vesca),功能基因组学,高效转化植物学报。2006,223(6):1219-1230。10.1007 / s00425 - 005 - 0170 - 3。
- 3.
草莓二倍体Fragaria vesca f. semperflorens的近交系,用于蔷薇科基因组和分子遗传学研究。植物学报。2009,5(1):15。
- 4.
Jung S, Cestaro A, Troggio M, Main D, Zheng P, Cho I, Sargent DJ: Fragaria、Prunus和Malus的全基因组比较揭示了不同的蔷薇亚科进化模式。中国生物医学工程学报。2012,13(1):129。10.1186 / 1471-2164-13-129。
- 5.
草莓第3部分:结构和功能基因组学。浆果的遗传学、基因组学和育种。编辑:Folta KM, Cole C. Enfield, NH:科学出版社;2011年,240 - 308。
- 6.
刘志伟,刘志伟,刘志伟:草莓果实发育早期的基因组尺度转录组学研究。植物学报。2013,25(6):1960-1978。10.1105 / tpc.113.111732。
- 7.
王磊,冯志强,王旭东,张晓霞:DEGseq:一种用于RNA-seq数据差异表达基因鉴定的R包。生物信息学。2010,26(1):136-138。10.1093 /生物信息学/ btp612。
- 8.
Anders S, Huber W:序列计数数据的差异表达分析。中国生物医学工程学报。2010,11 (10):R106。10.1186 / gb - 2010 - 11 - 10 - r106。
- 9.
Stein LD:使用GBrowse 2.0可视化和共享下一代序列数据。生物通报。2013,14(2):162-171。10.1093 /龙头/ bbt001。
- 10.
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Durbin R:序列比对/映射格式和SAMtools。生物信息学。2009,25(16):2078-2079。10.1093 /生物信息学/ btp352。
确认
这项工作得到了美国国家科学基金MCB0923913的资助。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
作者的贡献
OD设计开发了数据库和用户界面。SH协助建立了Genome Browser。CK、CH和AG生成测序的RNA样本。JS, ZL和NA设计研究并监督工作。OD, JS, NA, ZL撰写手稿。所有作者阅读并批准了最终的手稿。
作者为图像提交的原始文件
权利和权限
开放获取本文由BioMed Central Ltd授权发表。这是一篇开放获取的文章,是根据知识共享署名许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、分发和复制,但须注明原作的出处。创作共用及公共领域专用豁免书(https://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
达维什,斯洛文,J.P,康,C。et al。林地草莓的在线基因组资源。BMC植物杂志13,223(2013)。https://doi.org/10.1186/1471-2229-13-223
收到了:
接受:
发表:
关键字
- 草莓
- 转录组
- RNA-seq
- 数据库
- gBrowse
- 水果
- 花
- 蔷薇科