摘要
背景
在植物中,复杂的细胞壁保护细胞并限定它们的形状。纤维素原纤维在细胞壁基质内形成多层网络,在控制细胞膨胀中起着直接作用。解决该网络的结构将使我们能够理解纤维素原纤维定向和生长的关系。
荧光染料Pontamine Fast Scarlet 4BS (PFS)对纤维素具有很高的特异性,可以在共聚焦显微镜下观察到拟南芥根表皮细胞细胞壁中的纤维素束。共聚焦显微镜的分辨率极限约为200纳米英寸xy550纳米英寸Z在绿光下,纤维素只能直接显现为相对较大的束状结构,而直径小于10 nm的纤维素微纤丝的结构仍然无法分辨。在过去的十年里,人们开发了几种所谓的超分辨率显微镜方法;在本文中,我们探讨了这些方法的潜力,以直接可视化的纤维素。
结果
为了确保最佳成像,我们确定了pfs染色组织的光谱特性.PFS对洋葱鳞片表皮细胞活力无明显影响。通过直接随机光学重建显微镜(dSTORM)和全内反射荧光显微镜(TIRF)的结合,我们首次展示了植物细胞壁中纤维素束的超分辨率图像。由于TIRF限制了对细胞表面的观察,我们测试了替代3d结构照明显微镜(3D-SIM)和共聚焦显微镜,结合图像反卷积。这两种方法的分辨率都低于STORM,但支持3D成像。3D-SIM产生了较强的伪影,反褶积得到了较好的结果。与传统的共聚焦显微镜相比,分辨率得到了提高,该方法可以用来显示细胞壁不同层的原纤维方向的差异,以及胞间连丝周围特殊的纤维素强化。
结论
pfs染色的纤维素纤维的超分辨率光学显微镜是可能的,而且比传统方法更高的分辨率使其成为研究细胞壁结构的有价值的工具。这是方法发展的一个步骤,将填补更多侵入性技术的差距,如原子力和电子显微镜。
背景
纤维素微纤维构成植物细胞复杂细胞壁的主干。纤维素纤维网络的精确结构对我们在细胞水平上理解植物生长具有重要的意义,因为它在控制细胞伸长方面有直接作用[1-3.].此外,对细胞壁结构的精确了解有助于开发在生物燃料生产中有效分解植物细胞壁的方法[4.那5.].
Pontamine Fast Scarlet 4BS (PFS)是一种高特异性结合纤维素的荧光染料,已被用于观察拟南芥根表皮细胞细胞壁中的纤维素束[6.].在共聚焦显微镜下,随着时间的推移,可以跟踪纤维素束的方向,这第一次直接证明了在细胞伸长过程中纤维素束的被动重定向。然而,由于传统光学显微镜在xy方向上的分辨率限制在250 nm左右,只能看到最大的纤维素束。纤维素微纤维的直径小于10nm [7.那8.],但可以捆绑成大纤维。提供纳米级分辨率的破坏性方法,如扫描电子显微镜和原子力显微镜,表明大纤维不一定显示出与大多数微纤维相同的方向[9.那10.].
为了更完整和动态地了解植物细胞壁中纤维素网络的结构,需要提高光学显微镜的分辨率,以显示原生状态的细胞壁及其在活细胞中的分化。扫描电子显微镜(SEM)和原子力显微镜(AFM)等侵入性高分辨率技术的广泛制备与活体显微镜不兼容,并讨论了它们对纤维素网络结构的深刻影响[9.那11.].
光学显微镜领域的最新发展显著提高了荧光显微镜的分辨能力,采用不同的方法(Schermelleh等人综述[12.])。基本上,这些方法通过绕过ernst abbe定义的显微镜衍射限制来改善分辨率[13.].我们使用了两种所谓的超分辨率显微镜技术,3d结构照明显微镜(3D-SIM)和直接随机光学重建显微镜(dSTORM),来测试pfs染色的纤维素纤维的可视化。第三种已建立的超分辨率技术,受激发射耗尽显微镜(STED)与绿色植物组织不兼容,因为耗尽激光波长被样品中的叶绿素高度吸收。与宽视野荧光显微镜相比,3D-SIM和STORM分别将荧光信号的分辨率提高了2倍xyz和10倍xy [12.].此外,我们还测试了解卷积对共聚焦图像堆栈的影响,通过去除点扩散函数的扭曲效应来导致微量图像。该功能是任何光学系统的固有因素,可以通过适当的算法来补偿。
本研究中使用的洋葱鳞片表皮已被确定为研究细胞如何控制生长的理想模型系统[14.]因此,了解细胞壁中纤维素网络的确切结构具有直接意义。
结果和讨论
桥胺原位快速猩红的光谱特性和细胞毒性
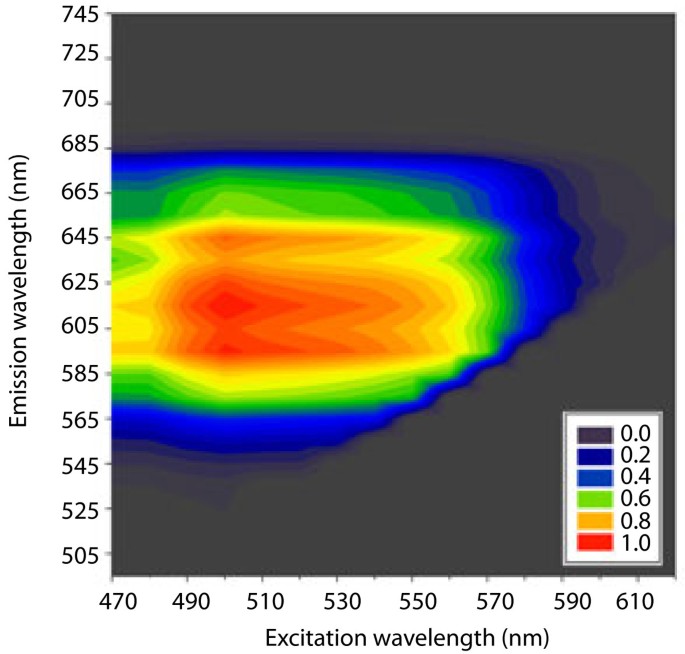
PFS染色洋葱鳞片表皮细胞壁上的同时激发和发射扫描显示了485至565 nm的宽激发范围(图1)。因此,它非常适合与流行的488nm和561nm激光线一起使用。结果镜像早期测量在体外[6.那15.].在红色光谱中检测到发射最大值,在615 nm附近有一个宽峰(图)1)因此,它很容易与在488 nm激发的GFP、YFP、FITC和其他常用荧光团区分。在大多数情况下,叶绿素自发荧光和PFS荧光发射光谱的部分重叠不是问题,因为叶绿体和细胞壁很容易区分。
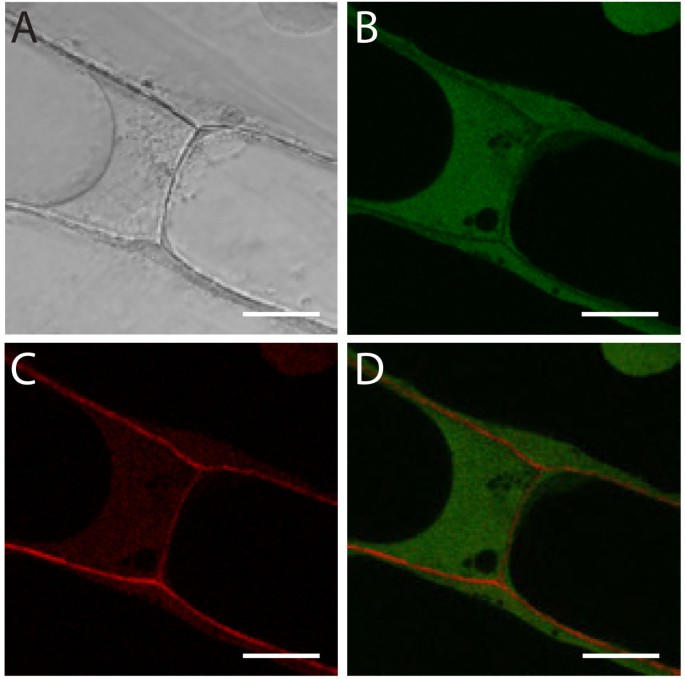
用羧基荧光素-二乙酸酯(CFDA)检测PFS染色后的植物细胞活力[16.].非极性CFDA分子能够通过质膜。在细胞质中,它们通过内源性酯酶的裂解获得荧光特性。因此,完整的细胞在细胞质中显示绿色信号,CFDA在细胞质中聚集。死细胞中酯酶活性的缺乏阻止了荧光的激活。在共聚焦显微镜下(图2), PFS染色3h后仍可在洋葱鳞片表皮细胞胞质中观察到CFDA信号。这并不随着培养时间的延长而改变(数据未显示),这表明在长期活细胞实验中,PFS作为一般细胞壁染色剂的潜力。碘化丙啶是最常见的一般细胞壁染料,在红色光谱中发射,相反,它具有诱变性,对样本和科学家的健康构成威胁[16.].
直接随机光学重建显微镜显示细胞表面有PFS染色的纤维素纤维
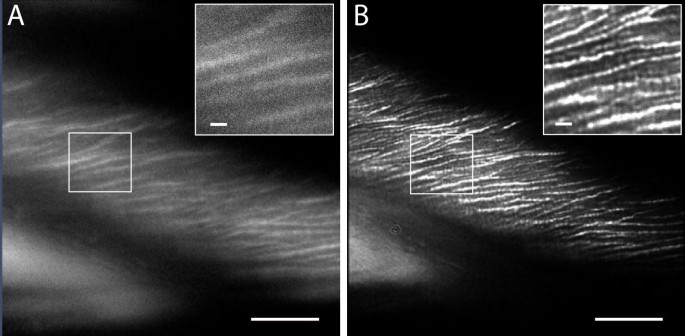
在直接风暴(dSTORM)中,当使用适当的激发波长和强度时,荧光色素可以在荧光和暗状态之间切换。由于荧光状态的随机特性,它被记录为闪烁,并允许相邻荧光团分子在时间上分离。如果在单次闪烁事件中记录足够数量的光子,分辨率可以提高到30纳米[17.].这种方法可以最有效地用于可光开关的荧光团,但已经证明,基于其固有的开关能力,dSTORM也可以用于传统染料[18.].dSTORM的一个先决条件是,激励被限制在一个狭窄的光学部分,以排除在大z体积中存在的局部不确定性。这通常是通过全内部反射(TIRF)显微镜实现的。pfs染色后,使用TIRF可在洋葱鳞片表皮细胞的细胞壁中看到纤维素原纤维(图)3.A).强激发激光漂白后,进一步降低PFS荧光信号,到目前为止可以记录到单次闪烁事件。数字3.B示出了基于本地化结果的超分辨率图像的渲染产生了显着的增益。
从根本上说,有两个因素对一个好的dSTORM染料很重要:1)每个染料分子检测到的光子数量。这将决定染料分子定位的精度。2)开关占空比。Dempsey等人将开关占空比定义为:“荧光团在开启状态下所花费的时间百分比”。即使在染料分子完全定位的情况下,占空比也会影响结构的分辨率[19.]就这些因素而言,PFS不是dSTORM的理想探针。观察到的闪烁事件表明,当PFS与纤维素结合时,通常在不可逆光破坏之前只有几个开关周期,并且具有长的暗态,这意味着占空比非常低。此外,在5–10 ms的开启时间内,只有相对的实际上,这意味着为了获得一幅完整的超分辨率图像,必须采集大量的图像。
通过使用含有氧清除系统和伯硫醇作为还原化合物的成像缓冲液可以改善该方法。减少条件稳定荧光团的偏离状态,从而防止其不可逆的光散脱离。同时,缓冲条件诱导荧光团分子的自发恢复到它们的状态。硫醇浓度和激发激光强度的优化可能导致每帧和占空比增加的局部数量增加。这应该导致更快的成像和更高分辨率[20.].
图中的纤维素原纤维3.B的直径约为100nm,间距清晰,在不同位置出现分支。细胞表面的这种结构与内层明显不同,内层通常显示更窄、更均匀的原纤维[9.].然而,观察到的形式和模式反映了细胞扩张过程中实现的结构变化,因此,可以给出这一过程如何发生的指示。这里获得的dSTORM图像可以指出在重组过程中原纤维聚集增加的可能性,尽管用类似方法获得的其他细胞壁层的数据需要进行比较。
PFS不适用于三维结构照明下的纤维素可视化
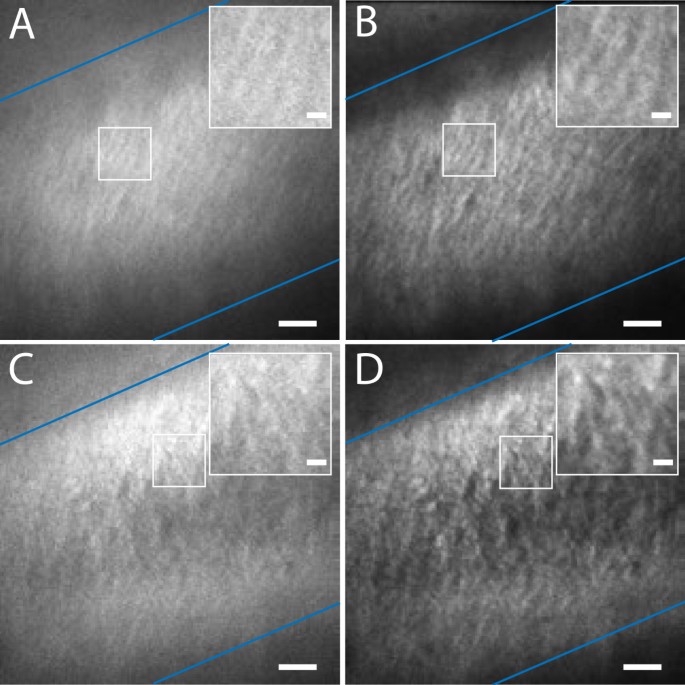
在3D-SIM中,模式激发光用于拍摄不同相位、旋转和聚焦平面的图像。通过傅里叶空间的数学重构,可以得到比宽场图像更高分辨率的图像。通常,这种技术非常适合在植物组织中成像,因为它与传统探针和中等厚度的标本一起工作[21那22].然而,洋葱鳞片表皮细胞外细胞壁的纤维素纤维不可能被清晰地分解。额外的文件1:图S1显示了具有两个不同噪声滤波器设置的宽场和3D-SIM模式中PFS染色的外壁表皮的比较。虽然标准噪声滤波导致任何结构信息的丢失,但最小的噪声滤波导致典型的旋转对称3D-SIM人工制品,如附加文件中心所示1:图S1。图像中只有一小部分显示出纤维状结构。不能排除来自其他类型细胞(如厚壁组织纤维)的排列强烈的细胞壁大纤维会被3D-SIM分解。然而,从真实结构中区分人工制品似乎很困难——无论如何,这里使用的例子都是如此。
不同滤波器设置的影响表明PSF染色洋葱电池中的背景信号电平太高。在这方面,3D-SIM模式的表现类似于宽场显微镜而不是共聚焦显微镜,其中在记录下已经强烈降低了焦点荧光。
共聚焦成像结合图像碎片卷积可以显示3D中的纤维素纤维网络
图像反褶积是一种从模糊和噪声中恢复信号的数学运算,虽然通常不被认为是一种超分辨率技术,但它也可以用来提高传统显微镜的分辨率,使其超越衍射极限。与STORM和3D-SIM相比,分辨率增益不大,但图像反卷积具有适用于共焦图像数据的优势。这意味着不需要特殊的成像硬件,而由于共聚焦显微镜的光学切片能力,可以对标本内部深处的结构进行成像。
数据可以通过点扫描以及旋转圆盘共焦系统获得。我们使用旋转圆盘共焦,它提供了更高的捕获率和光漂白较低的点共焦。在获取图像堆栈后,将其导入到适当的图像处理软件中,其中开源ImageJ和商业惠更斯SVI被认为能给出最好的结果[23].对图像进行反卷积的关键因素是点扩散函数,它可以通过实验或理论来确定。我们选择理论计算来确定实验点扩散函数对于厚的样本,如全组织样本是不可靠的[24].然而,如果在成像过程中包括实际焦平面上的参考物体,例如附着在细胞外部的荧光珠,然后用于计算样品固有的点扩散函数,反褶积效率可能会得到提高。
将反卷积应用于洋葱表皮细胞细胞壁中pfs染色的纤维素图像堆栈,提高了图像质量,提供了更清晰的纤维素原纤维方向(图)4.)和胞间连丝周围纤维素沉积(图5.)。原始数据显示,类似于拟南芥根表皮细胞的情况[6.,纤维素原纤维的方向可以从相对较大的束中推断出来,都在外面(图4.A)以及在内部(图4.C)细胞壁。虽然反卷积图像显示的主要是相同的原纤维,但增强的分辨率可以清晰地区分单个原纤维(图)4.B,D)。这对于精确定量至关重要,例如原纤维和电池的纵向轴线之间的角度。测量20原纤维,在电池表面下方的焦平面100nm处确定46.2°(标准误差3.1°)的平均角度,而电池壁的20个原纤维300nm较深的300nm布置在92°的n平均角度(标准错误1.8°)。具有不同纤维素取向的两个不同的层反射了不同的细胞膨胀相,其中在被动重新定向假设中提出的XYZ沉积后,外层中的纤维素在XYZ沉积中被动地重新定位[25]该信息可以补充洋葱表皮生长研究,该研究仅分析整个细胞壁中的平均纤维素方向[3.那14.].
在PFS的最大发射波长(600 nm)下,采用物镜(N.A = 1.4),光学分辨率约为200 × 550 nm (x × z),在200 nm焦距下观察到的原纤取向变化似乎是不可能的。然而,反卷积的强度恰恰在于它能够通过消除齐柏林型点扩散函数的模糊效应来提高三维分辨率。这种技术不允许在光轴上的两个点(=Z在该距离内,但能够为在此处使用的样品中的宏观原纤维来这样做。这意味着两个微纤维在彼此顶部平行运行,距离为200nmZ不能分开,但那些是纵横交错的。在这里,反卷积算法能够充分改善聚焦光纤和200 nm以下的间隙(无荧光)之间的对比度,以便可视化。
除了纤维素取向外,pfs染色洋葱表皮细胞壁图像的反卷积也揭示了细胞连接结构的特征(图)5.)。虽然使用愈合特异性染料anilin蓝共染色的原始数据表明,在细胞壁中核糖中的纤维素的不一致域(图5.A,B),这在反卷积图像上变得更清楚(图5.C, D),甚至显示了残留在坑内的薄细胞壁板。有趣的是,在3D视图或反卷积图像的最大强度投影中,很明显,与壁的其余部分相比,纤维素在胼胝质周围2 μm区域沉积的密度要高得多(图)5.F)。该纤维素套环可能潜在地形成对胼ins沉积物的限制,以便堵塞Plasmodesmata [26].
结论
宽视野荧光显微镜可以研究细胞壁的自然状态,但进一步的细节,其亚结构可以提高显微镜分辨率。在这里,我们证明了纤维素特异性染料PFS适用于TIRF显微镜和STORM,实现了纤维素纤维低于100 nm厚度的分辨率。然而,由于TIRF显微镜只能观察细胞表面,不能从更深的细胞壁层获得信息。虽然3D-SIM主要实现了生物标本内100 μm以上的超分辨率成像,但细胞壁中PFS的高信号密度阻碍了对洋葱表皮纤维亚结构的正确数学重建。作为STORM和3D-SIM的替代方案,我们使用了传统的共焦显微镜和采集后的图像反卷积。与其他方法相比,这使得分辨率有了更适度的提高,但利用了共聚焦显微镜的z形切片和深度成像能力。这种方法获得的数据使我们能够评估纤维素原纤维的方向,这在细胞壁的两层中是不同的。它还被用于与另一种细胞壁成分的染料共染实验,揭示了相邻细胞间胞间连丝周围细胞壁结构的新特征。
未来的发展,如3D-STORM的改编[27毫无疑问,对于植物组织将有助于进一步关闭非侵入性光学显微镜和侵入性技术(如AFM和SEM)之间的分辨率差距。此处提出的结果显示了如何利用目前可用的超分辨率技术实现这一目标的第一步,证明他们的潜力是在高于的分辨率下调查生活植物细胞的细胞壁结构的宝贵工具以前在技术上是什么。
方法
染料和染色
Pontamine Fast Scarlet 4 BS购自Aldrich稀有化学品图书馆(目录号S479896; S479896; Sigma-Aldrich,美国圣路易斯,美国)。它现在可以使用同义词直接红色。在0.1M磷酸盐缓冲盐水(PBS)pH 7.2中,5mg / ml的储备溶液可以在-4℃或-20℃下储存几周。取决于组织,将工作溶液稀释至0.1-0.01mg / mL,并始终新鲜制备。孵育时间通常是20分钟,但复杂组织中的更长。染色之后是短暂洗涤PBS。
羧基荧光素二乙酸酯(Molecular Probes, Life Technologies Corporation, Carlsbad, USA)在0.5 μg/ml的PBS中使用。在10 ~ 15分钟内对组织进行染色,然后在磷酸盐缓冲液中洗涤5分钟。
苯胺蓝(Sigma-Aldrich, St. Louis, USA)工作液在PBS中稀释至50 μg/ml,染色时间5 ~ 10分钟。
样品制备
从中制备洋葱灯泡量表表皮Allium Cepa.在超市购买的灯泡。用剃须刀从新鲜的鳞片上剪下0.5 × 0.5 cm的鳞片,用镊子将鳞片内侧的表皮剥落。
光谱分析
同时励磁和发射扫描(Lambda2-scan)进行了原位使用徕卡SP5-X共聚焦激光扫描显微镜对洋葱鳞片表皮细胞壁进行PFS染色(德国曼海姆徕卡微系统公司)。使用经过校准以在可用光谱上保持恒定输出功率的白光激光器作为激发源。在470 nm和620 nm之间每隔10 nm进行激发扫描。AOBS定义的10 nm宽检测窗口从激发波长+15 nm(中心位置)移动在ImageJ中进行图像分析。
图像采集与处理
在细胞活力测试中使用徕卡TCS SP2/MP (Leica Microsystems, Mannheim, Germany)进行共聚焦成像。PFS和cFDA在488 nm氩气激光下激发。定义了以下检测窗口:PFS: 560-605 nm, cFDA: 485-505 nm。
超分辨率成像是在蔡司Elyra PS.1显微镜(蔡司,德国耶拿)和安道尔iXion 860 EMCCD相机(安道尔技术,爱尔兰贝尔法斯特)上进行的,使用561 nm固体激光器进行激发,带通滤波器为DM 561 nm,EM 570–650 nm。
对于3D-SIM,采用63 × 1.2 Zeiss Apochromat水浸和63 × 1.4 Zeiss Apochromat油浸物镜,外加23 μm光栅。z堆栈记录5个相位变化和5个光栅旋转的每个部分。使用蔡司Zen 2011软件中的3D-SIM向导进行图像处理,设置如下:SR频率称重1,基线切割,理论PSF和图像图例中指定的噪声滤波。
对于STORM,使用相同的显微镜,使用100 × 1.46蔡司复色油浸物镜。通过epi-荧光模式聚焦洋葱表皮细胞表面,然后切换到TIRF模式,并增加角度,直到纤维素纤维可见(约60°)。对于一个图像系列,采集了20000张图像。这些都是在蔡司Zen 2011软件中使用“掌心向导”进行处理的。与使用相对较低卡方和背景阈值的光开关蛋白时通常使用的设置相比,过滤器设置被选择为允许的。通过设置最小光子数为100来过滤假阳性。根据定位精度矢量图,以75 nm的定位精度绘制背景校正点扩散函数。在单独的图像序列中确定PFS荧光团的启动时间和暗状态间隔。仅读取128 × 128像素区域的相机传感器,以使图像采集时间约为3ms每幅图像。
使用旋转盘共焦(Andor Revolution XD, 100 × 1.4 Olympus UPlanSApo物镜,Andor iXion 897 EMCCD相机,561 nm固态激光器,EX 564 nm, DM 561 nm, EM 570-650 nm)记录光学切片后,对共焦图像进行反褶积。反褶积在ImageJ中使用插件PSF Generator和DeconvolutionLab进行,这两个插件都是由École Polytechnique de Lausanne生物医学成像组开发的[28].利用PSF Generator,利用Born and Wolf算法生成理论点扩展函数[29,其中考虑了数值孔径、折射率、波长、像素大小和z步距。Richardson和Lucy开发的反褶积算法使用得到的点扩散函数[30]这种基于贝叶斯的方法通常被认为是最适合共焦数据反褶积的方法之一[24].输出数据与未经进一步图像调整的原始数据进行比较。
参考文献
- 1。
植物细胞壁的各向异性膨胀。《生物医学工程学报》,2005,21(1):93 - 97。10.1146 / annurev.cellbio.20.082503.103053。
- 2。
Geitmann A:初级植物细胞壁的力学建模和结构分析。植物营养与肥料学报。2010,13:693-699。10.1016 / j.pbi.2010.09.017。
- 3.
纤维素取向决定了洋葱表皮细胞壁的力学各向异性。中国生物医学工程学报。2006,33(6):593 - 598。10.1093 / jxb / erj177。
- 4.
Himmel Me,Ding Sy,Johnson DK,Adney Ws,Nimlos Mr,Brady JW,Foust Td:生物量批量:工程植物和生物燃料生产的酶。科学。2007,315:804-807。10.1126 / Science.1137016。
- 5.
Keegstra K:植物细胞壁。植物物理学报。2010,44(4):489 - 494。10.1104 / pp.110.161240。
- 6.
拟南芥细胞壁扩张过程中纤维素定向的实时成像。植物物理学报,2010,46(6):757 - 762。10.1104 / pp.109.150128。
- 7.
Ha MA, Apperley DC, Evans BW, Huxham IM, Jardine WG, Vietor RJ, Reis D, Vian B, Jarvis MC:纤维素微纤维的精细结构:来自洋葱和温柏的核磁共振证据。植物学报1998,16:183-190。10.1046 / j.1365 - 313 x.1998.00291.x。
- 8.
Kennedy CJ, Cameron GJ, Sturcova A, Apperley DC, Altaner C, Wess TJ, Jarvis MC:芹菜角组织纤维素的微原纤维直径:x射线散射和核磁共振证据。纤维素。2007,14:235-246。10.1007 / s10570 - 007 - 9116 - 1。
- 9.
丁淑玲,王玉玲,王玉玲:玉米原代细胞壁微原丝的研究进展。食品科学。2006,54:597-606。10.1021 / jf051851z。
- 10.
McCann MC, Bush M, Milioni D, Sado P, Stacey NJ, Catchpole G, Defernez M, Carpita NC, Hofte H, Ulvskov P, et al .:植物细胞壁功能结构的理解方法。植物化学学报。2001,57:811-821。10.1016 / s0031 - 9422(01) 00144 - 3。
- 11.
Sarkar P,Bosneaga E,奥尔M:整个进化中的植物细胞壁:朝着他们对设计原则的分子理解。J Exp Bot。2009,60:3615-3635。10.1093 / JXB / ERP245。
- 12.
Schermelleh L, Heintzmann R, Leonhardt H:超分辨率荧光显微镜指南。中国生物医学工程杂志。10.1083 / jcb.201002018。
- 13
阿贝E:对显微镜理论和微观视觉本质的贡献。Proc Bristol Nat Soc. 1874, 1: 200-261。
- 14
Suslov D,Verbelen JP,Vissenberg K:洋葱表皮作为一种研究高等植物生长各向异性控制的新模型。J Exp Bot.2009,60:4175-4187.10.1093/jxb/erp251。
- 15.
Hoch HC,Galvani CD,Szarowski DH,Turner JN:两种适用于真菌细胞壁可视化的新型荧光染料。Myologia.2005,97:580-588.10.3852/Myologia.97.3.580。
- 16.
Oparka KJ, Read N:使用荧光探针研究活的植物细胞。植物细胞生物学:实用方法。编辑:Harris N, Oparka KJ。牛津:牛津大学出版社;1994年,27-50。
- 17.
Heilemann M,Van de Linde S,Schuttpelz M,Kasper R,Seefeldt B,Mukherjee A,Tinnefeld P,Sauer M:具有常规荧光探针的Subtiffraction分辨率荧光成像。Angew Chem Int编辑。2008,47:6172-6176。10.1002 / ANIE.200802376。
- 18.
Shim Sh,夏克,钟GS,Babcock惠普,沃恩JC,黄B,王X,XU C,BI GQ,壮XW:带有光学膜探针的活细胞器的超级分辨率荧光成像。Proc Natl Acad Sci U S A. 2012,109:13978-13983。10.1073 / pnas.1201882109
- 19.
邓普西GT, Vaughan JC,陈浩华,Bates M,庄旭:基于局部超分辨率成像的荧光团优化性能评估。中国生物医学工程学报。2011,8:1027-1036。10.1038 / nmeth.1768。
- 20。
Vogelsang J,Cordes T,Thephtmann C,Steinhauer C,Tinnefeld P:控制单分子切换和超级化学显微镜的常见月嗪染料的荧光。Proc Natl Acad Sci USA。2009,106:8107-8112。10.1073 / PNAS.0811875106。
- 21.
Fitzgibbon J, Bell K, King E, parka K:利用三维结构照明显微镜对胞间连丝进行超分辨率成像。植物物理学报。2010,33(6):753 - 756。10.1104 / pp.110.157941。
- 22.
Linnik O, Liesche J, Tilsner J, Oparka KJ:超分辨率解开病毒复制复合物的结构。植物科学学报,2013,4:6。
- 23。
Griffa A, Garin N, Sage D:反褶积软件在3D显微镜中的比较。用户的观点。吉林大学学报(医学版). 2010,1:41-43。
- 24。
demonvel JB, Le Calvez S, Ulfendahl M:共聚焦显微镜图像恢复:提高反卷积的极限,并应用于哺乳动物听觉器官的可视化。acta botanicologica sinica(云南植物学报),2001,30(4):457 - 461。10.1016 / s0006 - 3495(01) 76214 - 5。
- 25
Chan J:植物细胞和器官生长期间的微管和纤维素微纤维取向。J MicroSC。2012,247:23-32。10.1111 / J.1365-2818.2011.03585.x。
- 26
王志强,王志强:胞间连丝的结构与发育。胞间连丝。编辑:Oparka K. Oxford: Blackwell Publishing, 1-31。年度工厂审查;2005.
- 27.
Huang B,Jones SA,Brandenburg B,庄XW:全池3D风暴揭示了纳米尺度分辨率蜂窝结构之间的相互作用。NAT方法。2008,5:1047-1052。10.1038 / nmeth.1274。
- 28.
生物医学成像组算法。http://bigwww.epfl.ch/algorithms.html..
- 29.
《光学原理》第7版。剑桥:剑桥大学出版社;2002年。
- 30.
基于贝叶斯的图像恢复迭代方法。中国海洋大学学报(自然科学版),2012,32(4):457。10.1364 / JOSA.62.000055。
确认
所有显微镜检查均在丹麦先进生物成像中心进行。我们感谢哥本哈根大学的Michael Hansen在超分辨率图像处理方面的建议。
作者信息
隶属关系
通讯作者
额外的信息
利益争夺
两位作者宣称他们没有相互竞争的利益。
作者的贡献
JL构思和设计了该研究,进行了实验并写了稿件。根据实验设计和讨论图像数据和稿件的分析和解释。IZ辅助解释超分辨率图像数据和图像处理。所有作者阅读并认可的终稿。
电子辅料
A.
附加文件1:图S1:洋葱鳞片表皮细胞pfs染色纤维素纤维的3d结构照明;在大视场图像()上,背景荧光强度相对于聚焦区域是明显的。对同一幅图像进行标准噪声滤波处理(B)所有结构信息将丢失。使用最小噪音滤波代替(C),文物涵盖大部分焦点区域。一些高分辨率结构信息可能会被保留(框,相同的区域在插入中放大)。秤条10μm;1μm插入物。(PDF 613 KB)
作者为图像提交的原始文件
权限
本文在BioMed Central Ltd.的许可下发布了这是一个开放的访问文章,根据Creative Commons归因许可(http://creativecommons.org/licenses/by/2.0)提供任何介质中的不受限制使用,分发和再现,所以提供了正确的工作。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
A. A.&Schulz,A的Liesche,J.,Ziomkiewicz,A.具有Pontamine Fast Scarlet 4bs的超分辨率成像,可以直接可视化洋葱表皮细胞中的纤维素取向和细胞连接架构。BMC植物杂志13,226(2013)。https://doi.org/10.1186/1471-2229-13-226
已收到:
公认:
出版:
关键字
- 荧光染料
- 细胞壁
- 纤维素
- 风暴
- 结构化的照明
- 超分辨率显微镜
- TIRF
- 反褶积