摘要
背景
籽粒蛋白质含量(GPC)是大麦作为麦芽、饲料和食品的重要品质决定因素。它是由一个复杂的遗传系统控制的。GPC在不同的大麦基因型之间差异很大,在不同的环境中也有差异。了解大麦GPC的遗传控制,识别不同环境下变异较少的基因型是当务之急。
结果
本研究利用59个栽培大麦和99个西藏野生大麦基因型进行全基因组关联研究(GWAS)和多平台候选基因关联分析,以鉴定与GPC相关的分子标记。西藏野生大麦的GPC高于栽培大麦。GPC与凝析功率(DP)和麦芽提取物之间的显著相关,证实了GPC在判断麦芽品质方面的重要性。采用GWAS技术检测大麦GPC相关的DArT标记。此外,GWAS还揭示了两个HvNAM基因作为控制GPC的候选基因。两者之间没有发现关联HvNAM1多态性和GPC,而单核苷酸多态性(SNP) (798, P < 0.01),位于的第二内含子HvNAM2,与GPC相关。单倍型间相关性显著HvNAM1,HvNAM2和GPC。
结论
基于GWAS和候选基因的关联研究可以有效地确定大麦GPC的遗传变异。DArT标记及其多态性HvNAM本研究鉴定的基因对今后培育优质大麦品种具有重要意义。HvNAM基因可能在控制大麦GPC中发挥作用。
背景
籽粒蛋白质含量是决定谷类作物品质的重要因素。在大麦中,GPC与饲料和麦芽品质密切相关。蛋白质含量较高有利于饲料品质,而麦芽大麦蛋白质含量较低或中等。GPC在许多方面影响麦芽质量,包括酵母营养、啤酒中烟雾的形成和酶活性[1,2].
大麦GPC处于多基因控制下,在7条染色体上都定位了许多数量性状位点,主要分布在2H、4H、5H和6H上[3.,4].所有这些位点都已通过QTL作图确定。近年来,全基因组关联研究(genome-wide association study, GWAS)已发展起来,用于分析植物的多种复杂性状[5,6].GWAS与传统的QTL定位相比具有优势,即GWAS可以对许多基因型进行定位。虽然用于传统QTL定位的群体是由双亲杂交开发的,但由于相关遗传位点之间的重组不足,仅允许检测植物内的一个位点/等位基因子集,并提供有限的分辨率。因此,GWAS在群体水平上可能比传统QTL定位具有更广泛的遗传变异和更高的表型和性状定位分辨率[6].在大麦中,利用GWAS对7个麦芽品质性状和一些重要的农艺性状进行了有效分析[7- - - - - -9].
青藏高原有丰富的大麦种质资源,被认为是世界上最早的栽培大麦中心之一[10].SSR标记分析表明,西藏野生大麦的多态性信息含量(PIC)值高于中国地方品种,且野生大麦比栽培大麦具有更多的独特等位基因[11- - - - - -13].因此,推测西藏野生大麦在控制GPC的基因上具有更大的变异性[11- - - - - -13].因此,利用世界各地的西藏野生大麦和栽培大麦衍生群体,可以为大麦GPC的GWAS提供较高的分辨率。
一个控制小麦GPC的QTL,命名为Gpc-B1基因克隆,发现一种转录因子(NAM-B1)通过调控衰老和蛋白再动员与GPC相关[14].的两个同源基因(Genbank登录号DQ869678和DQ869679)TtNAM-B1分别在染色体6H和2H上发现[14].单核苷酸多态性(SNP)分析表明,NAM-1基因的等位变异可能与GPC变异相关大麦属。表达的差异HvNAM-1或大麦品种间的其他基因均可归因于GPC变异[15].然而,关于大麦的研究很少HvNAM2最新的,除了序列的HvNAM2已出版[16].
本研究的目的是(1)探讨GPC与麦芽品质的相关性;(2)利用GWAS技术在大麦定位群体中鉴定与GPC相关的分子标记,并确定控制GPC的候选基因;与(3)之间的关联分析HvNAM基因和GPC。
方法
植物材料
利用158份大麦种质资源进行关联作图和GPC分析。这些种质包括59个大麦品种(h . vulgareL.)和99种西藏野生大麦(h . spontaneuml .)。所有大麦品种和种质均在浙江大学花家池校区种植(浙江杭州,120.0°E)。30.5°N)在2008年和2009年初冬。每次增加播种于2行地,长2 m,行与行之间间隔0.24 m,每行播种40粒。各小区施氮量均为150 kg/ha,其中播前施氮肥40 kg/ha,双叶期和孕穗期施氮肥110 kg/ha,均等量。此外,播种前施用180公斤/公顷氯化钾。实验被安排在一个组块设计中,有两个重复。在每个区块中,158个大麦品种被随机排列。所有其他农艺管理,包括杂草和疾病控制,都与当地应用的相同。苗期采集各基因型叶片进行DNA提取。 The harvested seeds were stored at 4°C prior to malting. GPC and malt quality of all samples were measured, three measurements were done for each sample.
GPC测定
成熟的颗粒在Cyclotec 1093样品磨机(Tecator AB, Hoganas,瑞典)中研磨,并通过0.5 mm的筛网。GPC采用凯氏定氮法测定[17].蛋白质含量是用N含量乘以6.25来计算的。
麦芽和质量分析
谷物样品(约200克)在微麦芽设备(Phoenix系统,阿德莱德,澳大利亚)中进行微麦芽,使用以下制度:6小时浸泡,14小时静置,8小时浸泡,14小时静置和4小时浸泡,然后96小时萌发——所有都在15°C下进行。然后将麦芽在65°C下烧制24小时,去根并使用配有0.5毫米筛的Tecator旋风磨机进行研磨。根据Analytica EBC官方方法(1975年欧洲啤酒厂公约)测定麦芽中可溶性蛋白和总蛋白含量(SPC和TPC)以及麦芽质量参数(麦芽提取物、Kolbach指数、粘度和DP)。
DNA提取及基因型分析
根据Uzunova等人的描述,从大麦幼苗的幼叶中分离出基因组DNA样本。18].简而言之,将叶片组织研磨,并用CTAB(十六烷基三甲基溴化铵)缓冲液(pH 5.0)重新悬浮得到的粉末。为了纯化DNA,通过离心去除不溶性颗粒。dna从水相中析出,并彻底清洗以去除污染盐。
使用Barley PstI (BstNI) version 1.7阵列分析所有DNA样本中的DArT全基因组谱[19]在澳大利亚的Diversity Arrays Technology Pty Ltd。大约有1500个DArT标记,在广泛的大麦品种中有多态性,在野生大麦品种中检测到1000个标记(http://www.triticarte.com.au/content/barley_diversity_analysis.html).在报道的1576个标记和1319个多态DArT标记中,P值< 0.05的标记被用于本研究。
引物对采用Primer3设计[20.]基于HvNAM1而且HvNAM2序列(Genbank登录号DQ869678和DQ869679, NCBI)。5”-atgggcagcccggactcatcctcc-3”和5”-tacagggattccagttcacgccggat-3”, 5”-atgggcagctcggactcatcttcc-3”和5”-tcagggattccagttcacgccgga-3”被用来放大HvNAM1而且HvNAM2,分别。聚合反应混合物中含有20 mM Tris-HCl, 50 mM KCl, 2 mM MgCl2,各dNTP 1 M,引物各5 pmol,基因组DNA 50-100 ng, 1单位TaqDNA聚合酶(迈杰生物,上海,中国)。反应最初在95°C变性5分钟,然后进行35个循环,分别为95°C 45秒、60°C 45秒和72°C 1.5分钟。在72°C终止PCR 10分钟。使用BigDye Terminator v3.1循环测序试剂盒(Applied Biosystems, Foster City, CA, USA)进行测序。使用Bioedit软件分析完整的基因序列(http://www.mbio.ncsu.edu/bioedit/bioedit.html).
数据分析
采用SPSS 13.0和SigmaPlot 10.0软件对GPC、SPC、TPC与麦芽品质参数进行Pearson相关分析。所有序列的比对由ClustalW [21].在澳大利亚diversity Arrays Technology Pty Ltd公司,用1319个随机分布的大麦DArT标记检测了基因组的遗传多样性。利用来自1319个DArT标记的遗传多态性数据,利用结构软件2.3.3版本,使用混合模型和5个独立重复的100,000次马尔可夫链迭代来检测群体结构[22,23].根据软件的说明,通过10万次迭代的老化和10万次马尔科夫链蒙特卡罗(MCMC)迭代,测试了从1到10的K值。使用SAS GLM (SAS Institute, Cary, North Carolina, USA)测试了种群结构对GPC的影响。该模型包含了STRUCTURE 2.2.3得到的Q矩阵的分量,用来说明种群结构。R2(由模型解释的方差)被认为是由群体结构解释的表型变异比例的估计。对1319个DArT标记的基因型数据进行主成分分析(PCA),这些标记首先使用un扰码器9.7 (CAMO PROCESS AS, Oslo, Norway)进行标准化。流苏采用2.01计算基于参数r的连杆不平衡(LD)2,这是对一对变量之间相关性的测量[23].使用1319个DArT标记计算成对关系矩阵(k -矩阵),进一步用于关联模型的总体校正流苏2.01[23].对两年的GPC数据进行平均,以便日后进行关联分析。DArT标记之间基于k矩阵的结构关联分析,HvNAM基因和GPC的计算流苏2.01[23].采用混合线性模型(MLM)测试DArT标记与总性状变异之间的相关性流苏2.01.P值用置换试验进行调整,使用在临床试验中实施的降压MinP程序流苏2.01.调整后的P值< 0.05或<0.01被认为是关联的标准。用R软件2.14.2版本绘制DArT标记和P值的Manhattan plot (http://www.r-project.org/).关联图使用MapDraw 2.1版本构建[24].
序列HvNAM1而且HvNAM2使用VectorNTI 10.0 (Invitrogen Corporation, Carlsbad, USA)或CLC主工作台5 (CLC bio, Aarhus, Denmark)进行对齐,并使用BioEdit软件手动编辑对齐。单倍型是用软件推断出来的流苏2.01[23].一个大麦附加被推断为罕见的单倍型,被排除在进一步的分析之外。根据单倍型分组HvNAM利用SAS 9.0软件(SAS Institute, Cary, North Carolina, USA)对59份栽培大麦和99份西藏野生大麦的GPC变异进行了分析。为了进一步分析共158份资料的单倍型与GPC之间的相关性,采用SAS 9.0软件(SAS Institute, Cary, North Carolina, USA)进行方差分析(ANOVA)和差异不显著的多重比较分析(LSD),平均差异在0.05水平上显著。
结果
蛋白质含量和Kolbach指数的变化
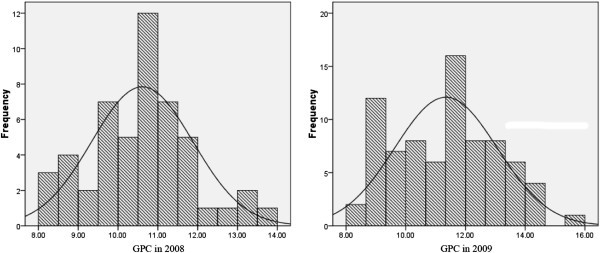
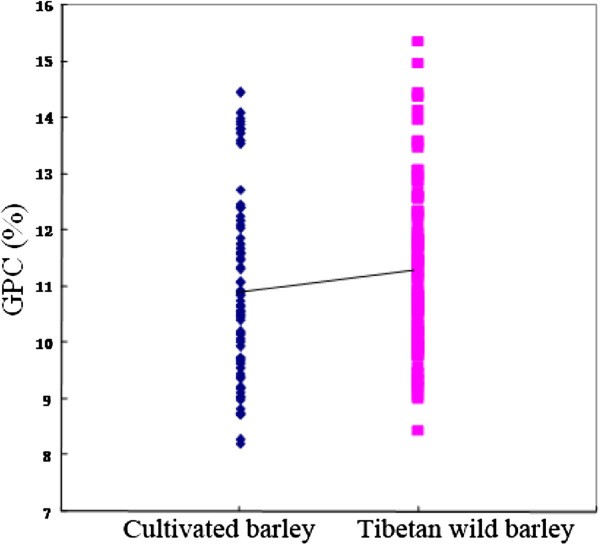
59个栽培大麦和99个野生大麦品种2008年的GPC变化范围为8.02% ~ 13.50%,平均为10.56%;2009年的GPC变化范围为8.28% ~ 14.45%,平均为10.87%1).总体而言,西藏野生大麦的GPC高于栽培大麦(图2).GPC的正态分布模式如图所示1表明大麦GPC由多个基因/ qtl控制。158份资料的SPC、TPC和Kolbach指数也存在较大差异(图3.).
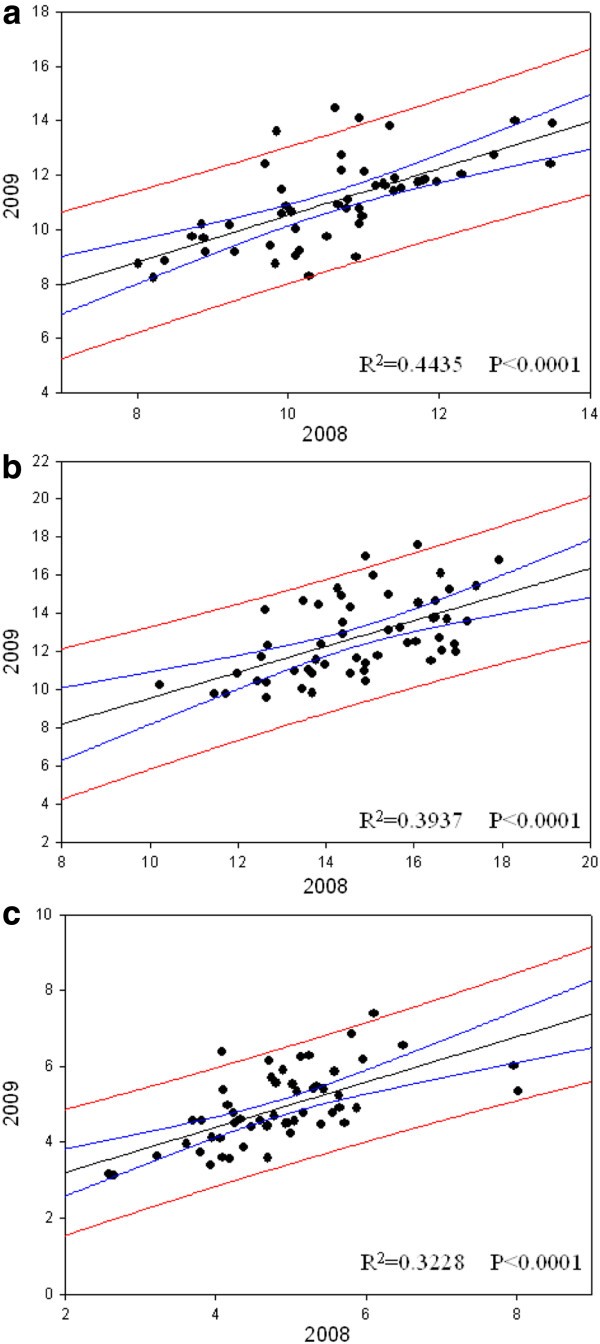
2008 ~ 2009年GPC、SPC、TPC值呈显著正相关(R2= 0.4435**GPC;R2= 0.3937**程控;R2= 0.3937**适用于TPC)(图3.),而2008年Kolbach指数的数据可占2009年变化的55.11%。由此可见,GPC、SPC、TPC和Kolbach指数主要受遗传因素控制,同时也受环境变异的影响。
GPC、SPC、TPC与四种麦芽品质参数的关系
GPC在这两年给出了相似的结果(图3.),故相关分析采用两年的平均值。结果表明,GPC与SPC (0.628, P < 0.01)、TPC (0.847, P < 0.01)、DP (0.340, P < 0.05)呈极显著正相关,与麦芽浸膏呈负相关(−0.347,P < 0.01)(表1)1).GPC与粘度、Kolbach指数无显著相关性。SPC与TPC (0.759, P < 0.01)、Kolbach指数(0.626,P < 0.01)、DP (0.456, P < 0.01)呈正相关,与粘度(−0.356,P < 0.01)呈负相关,说明SPC对判断麦芽品质具有重要意义。TPC与DP呈显著正相关(0.465,P < 0.01),与麦芽提取物呈显著负相关(−0.326,P < 0.01)1).
种群结构及其对GPC变化的影响
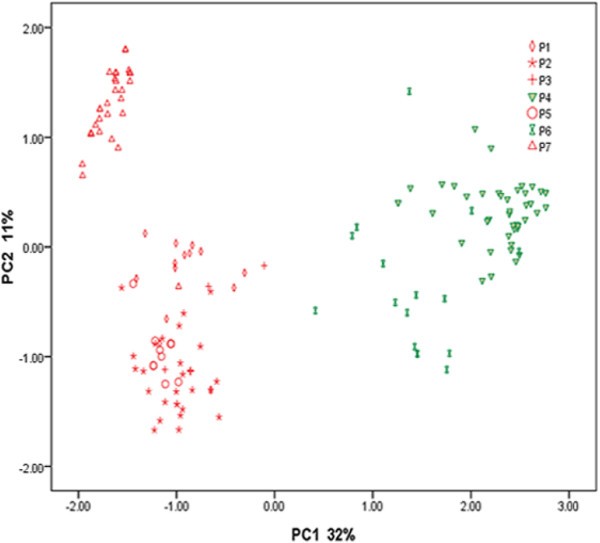
本研究的主要目的之一是确定GWAS是否可用于大麦GPC与遗传标记的关联分析。因此,我们得到LD (r2)在这个实验中使用的人口。获得LD的范围扩展到0.40 cM以上(附加文件1:图S1), 1319个DArT随机分布在整个大麦基因组上,保证了DArT标记在大麦基因组上的良好覆盖。这些群体中存在的群体分层和等位基因的不平等分布可能导致非功能性和虚假的关联[25,26].因此,本研究考虑了种群结构。利用1319个DArT标记对59个栽培大麦和99个西藏野生大麦基因型亚群进行了评价。采用结构分析和主成分分析对大麦群体进行分层检测。用STRUCTURE软件计算出的亚群体(K值)的最高似然值为K= 7(附加文件2:表S1,附加文件3.:图S2),表明有7个亚种群的方差最稳定。此外,对种群结构进行了主成分分析。有趣的是,PCA将栽培大麦和西藏野生大麦明显分为两组(图4),栽培大麦品种属于亚群体4和亚群体6,而野生大麦品种属于亚群体1、2、3、5和7(图2)4和附加文件4:表S2)。总的来说,这7个组成部分占遗传变异的65.18%。第一个组成部分解释了32%的变异,而第二个组成部分解释了11%的遗传变异(图4).然后,采用7个亚种群的Q矩阵进行进一步分析。对2008年和2009年GPC数据的方差分析表明,种群结构解释了总变异的10.6%,说明种群结构对GPC存在影响。
DArT标记与GPC的关联及候选基因的确定
一般来说,一个严格的模型可能会导致较少的假背景关联。在目前的研究中,基于结构的关联分析与k矩阵计算使用流苏2.01[23].
2008年和2009年的两次试验显示了类似的结果;因此,我们将这两年的数据结合起来进行关联分析。采用降压MinP法调整后的P值进行排列试验[27].作为P值调整后的标记。被认为是显著的,则整个假设集拒绝单个真零假设的概率保持在<0.05。该检验考虑了假设之间的依赖性,不像其他多重检验校正程序那样假设假设是独立的[23].在这里,DArT和GPC的关联显示在曼哈顿的地块(附加文件)5:图S3)。当调整后的P值<0.01时,有3、8、1、1和7个DArT标记,分别与1H、2H、3H、5H和7H的GPC相关。有趣的是,本研究中有5个分子标记与之前报道的GPC遗传标记接近(表1 - 2)2).其中,bPb-1628和bPb-1072与标记物HVBKASI接近,鉴定为HVBKASIHvNAM2[14].此外,bPb-8986和bPb-3412与HVM36和Bmag0751标记位点较为接近。
据报道,关联分析结果受环境因素影响[28,29].因此,严格的显著性标准可能会使研究偏离检测显示显著基因型-环境相互作用的因果关系[30.].2008 - 2009年GPC的相关分析表明,GPC主要受遗传因素控制,但也受环境条件的影响。因此,我们将关联分析的阈值设置为0.05,以便检测出可能与GPC相关的标记。当调整后的P值<0.05时,我们发现大麦GPC处于多基因控制状态,相关基因/ qtl几乎位于除4H外的所有染色体上,主要位于2H和7H染色体上(图5).在1H和5H染色体上有10个与GPC相关的DArT标记,2H染色体上有20个,3H染色体上有13个,6H染色体上有11个,7H染色体上有20个。相关标记占GPC方差的比例为2.2 ~ 18.0%。一些与GPC相关的DArT标记紧密定位于基因组内。因此,我们认为10 cM以内的相关DArT标记是相同的位点。结果在1H、2H、3H、5H、6H、7H染色体上分别有5、7、6、5、6、8个位点。其中,一个占总变异量40%的主要QTL与标记abg458、hvm74和mwg2029非常接近,可能与位于小麦6BS染色体上的GPC - b1基因同源。的Gpc-B1与小麦籽粒蛋白质的增加有关[1],该QTL被鉴定为HvNAM1在大麦中[14)(表2).同样,在我们的研究中,与大麦GPC相关的标记bPb-7179、bPb-5822和bPb-9522与标记abg458、hvm74和mwg2029接近。此外,最佳邻居连接树表明HvNAM基因与小麦NAM基因距离最近[14],同时揭示了大麦和小麦NAM位点的共线性[16].然后,我们推断HvNAM基因可能与大麦GPC有关。因此,HvNAM1而且HvNAM2作为GPC进一步关联分析的候选基因。
大麦籽粒蛋白质含量(GPC)关联图。地图使用MapDraw 2.1版本构建[20.].星号表示与GPC相关的分集阵列技术(DArT)标记。括号表示10 cM内与GPC相关的DArT标记。
协会HvNAMGPC基因
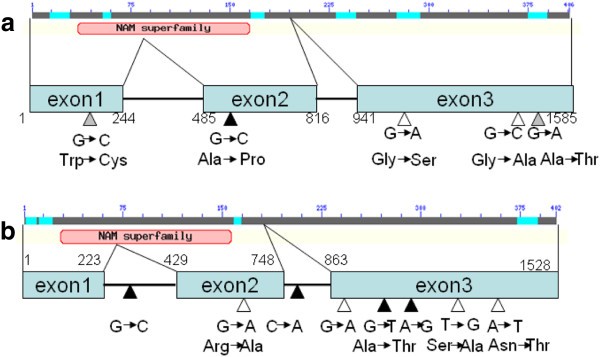
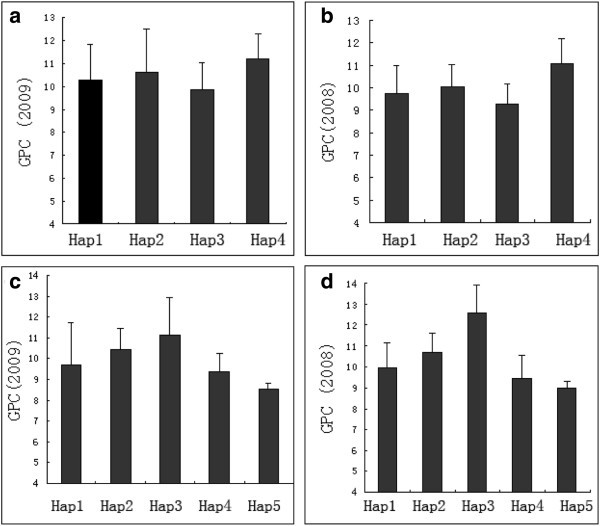
的序列HvNAM1而且HvNAM2对照NCBI文献(登录号为DQ869678和DQ869679)进行基因分析。的结构和snpHvNAM1而且HvNAM2如图所示6.的放大长度HvNAM1基因长度为1585 bp,包含3个外显子,2个内含子和一个NAM超家族结构域,位于氨基酸35 ~ 165。通过与参考序列(DQ869678)的比较,得到了该序列HvNAM1本研究共发现5个SNPs,分别位于栽培大麦的234、544和1433碱基上,西藏野生大麦的544、1190和1427碱基上6和附加文件6:图S4)。所有snp均位于编码区内,并导致5个氨基酸取代,其中Trp、Ala、Gly、Gly和Ala分别被Cys、Pro、Ser、Ala和Thr取代。两者之间没有联系HvNAM1多态性和GPC。因为没有SNPHvNAM1发现基因与GPC相关,进行单倍型相关分析。使用软件流苏2.01推断单倍型HvNAM1在该基因中,我们发现了5个单倍型。在59个栽培大麦基因型和99个野生大麦基因型中分别发现3个和4个单倍型。栽培大麦和藏大麦分别有1个和2个独特的单倍型(表2)3.).为了分析单倍型对GPC可能存在的差异影响,考虑了种群结构。的单倍型HvNAM1在被测人群中解释了20.6%的GPC方差。在整个品系中观察到,该品系携带4号单倍型HvNAM1GPC最高,而单倍型为3的HvNAM1两年来平均平均收入最低(图7).
的放大HvNAM2基因包含3个外显子和2个内含子,在氨基酸28 ~ 157之间有一个NAM超家族结构域,长度为1528bp。的多肽序列HvNAM2有80%的相似度HvNAM1.西藏野生大麦的307、732、798、962、979、991、1034和1289碱基上有8个snp位点,栽培大麦的307、798、979和991碱基上有4个多态性位点。其中,SNP307和SNP797位于内含子内,其余位于编码序列内。SNP732、SNP979、SNP1034和SNP1289导致氨基酸替换,特别是Arg、Ala、Ser和Asn分别被Lys、Thr、Ala和Tyr取代。根据栽培大麦和藏野大麦的多态性可分为6个单倍型。此外,栽培大麦的1个单倍型和西藏野生大麦的2个单倍型是独特的(表2)3.).西藏野生大麦新多态性的存在,为大麦的遗传改良提供了新的遗传资源。然而,只有1个SNP (798, P < 0.05)位于基因的第二内含子内HvNAM2(图6和附加文件7:图S5)与59个栽培大麦基因型和99个西藏野生大麦基因型连续2年的GPC相关。此外,为了分析的效果HvNAM2大麦GPC上的单倍型,从6个单倍型中排除了1个增加的单倍型HvNAM2.的单倍型HvNAM2解释了人群中7.2%的GPC方差。我们观察到的单倍型3HvNAM2GPC较高,而单倍型5的HvNAM2在这两年的平均国民生产总值最低(图7).
讨论
用于酿造麦芽的大麦GPC应低于11.5%。GPC在很大程度上受基因型和环境的影响[31,32].在目前的研究中,多样性面板的表型为了解大麦GPC的范围和分布提供了一些有价值的信息。比较连续两年的GPC数据,基因型和环境的相互作用确实很明显。结果表明,部分GPC较高的西藏野生品种可用于饲料大麦和食用大麦品种的选育。虽然连续两年各基因型间GPC、SPC和TPC均存在显著差异,但性状的一致性较高,主要受遗传因素控制。
GPC与麦芽提取物呈负相关,与DP呈正相关[33].同样,在本研究中,我们发现TPC与麦芽提取物呈负相关,与DP呈正相关。有趣的是,SPC与除麦芽提取物外的所有麦芽品质参数均相关。显然,谷物和麦芽中的蛋白质含量与麦芽品质密切相关。因此,在麦芽大麦育种中,开发GPC稳定的大麦品种势在必行。
基于双亲杂交群体的GWAS相比传统QTL定位的优势已被证实[34].与QTL作图相比,GWAS增加了在单个实验中可以调查的自然变异的范围,以及可能被识别的重要区域的数量[12].因此,GWAS可以提供比QTL定位更高的分辨率,促进精细定位和基因发现。GWAS研究使用的材料包括59个世界栽培大麦品种和99个西藏野生大麦品种,这些品种涵盖了世界上大多数大麦产区的代表性品种。
相关分析表明,GPC主要受遗传因素控制,同时也受环境变异的影响。然而,严格的显著性标准可能会使研究偏离检测显示显著基因型-环境相互作用的因果关系[30.].因此,我们选择0.01和0.05作为关联分析的阈值,以检测可能与GPC相关的标记。结果表明,GWAS在1H、2H、3H、5H、6H和7H染色体上分别鉴定出5、7、6、5、6和8个与大麦GPC相关的位点。这些结果表明,与传统的QTL定位相比,GWAS可以检测到更多与GPC相关的分子标记。
除了发现GPC的DArT标记外,GPC关联图谱的完成是朝着克隆GPC相关基因迈出的重要一步。所鉴定的GPC标记对于评价和筛选GPC合理的大麦种质具有重要意义。与以往的研究相比[1,4,15,31],我们在本研究中发现了更多的标记,分别在6H染色体、2H染色体和5H染色体上发现了3个、3个和1个标记(表2).利用低粒蛋白六棱品种“Karl”与高粒蛋白二棱品种“Lewis”杂交的大麦定位群体,在染色体6H和2H上鉴定出三个主要qtl。3个qtl可解释GPC总遗传方差的56% [1].其中两人被确认为HvNAM1而且HvNAM2一种NAC转录因子(NAM-B1)的同源物,通过调节小麦的衰老来增加GPC [14].因此,我们认为HvNAM1而且HvNAM2作为控制GPC的候选基因。由于基因-靶标关联在大麦中识别SNP标记的作用[35],即两个候选基因之间的联系,HvNAM1而且HvNAM2,并对GPC进行分析,以研究GPC的遗传结构,鉴定GPC在大麦中的位点。
Jamar等人发现功能性NAM-1基因的等位变异可能与属内GPC变异相关大麦[15],他们研究中使用的13个基因型可以分为3个单倍型:11个欧洲品种h . vulgare收集为单倍型1,1h . spontaneum(Hs)和1大麦bulbosum(Hb)分别被分类为单倍型2 (Genbank登录号EU908210)和单倍型3 (Genbank登录号EU908211)。通过与参考序列(DQ869678)比较,在其中的355、483和554碱基上鉴定出3个snpHvNAM1.然而,我们在目前的研究中并没有发现这些snp。相反,我们在栽培大麦中发现了3个位于碱基234、544和1433上的snp,在西藏野生大麦中发现了3个位于碱基544、1190和1427上的snp。的多态性之间没有发现关联HvNAM1与GPC呈显著正相关HvNAM1单倍型和GPC。此外,内部有8个snpHvNAM2在西藏野生大麦中分别位于307、732、798、962、979、991、1034和1289碱基上,而在栽培大麦中仅在307、798、979和991碱基上存在4个snp。有趣的是,单SNP (798, P < 0.05)在HvNAM2基因位于第二内含子上,与GPC相关。为了获得更深入的了解,之间的相关性HvNAM2在大麦中进行单倍型和GPC分析,其中DArT标记接近HvNAM1和HvNAM2的单倍型解释了18%和6.4%的GPC方差HvNAM1和HvNAM2占GPC方差的20.6%和7.2%。综合分析,包括NAM基因在拟南芥和其他作物中的初代GWAS、大麦和小麦NAM位点共线性、NAM基因的最佳邻居连接树以及NAM基因的关联分析HvNAM基因表明HvNAM基因可以驱动大麦GPC的变异。此外,结果还表明,调整后的P值< 0.05对于寻找受环境因素影响较大性状相关的分子标记是合理的。事实上,我们在GPC的初次GWAS中使用P <0.05的阈值确保了DArT标记物的识别,而在P <0.01的阈值分析中没有检测到DArT标记物。其中一个候选基因,HvNAM1经校正P值<0.05进行关联分析,发现与GPC相关。目前的结果表明,调整后的P值<0.05适用于识别与GPC相关的分子标记。同样,在其他研究中,以调整后的P值<0.05作为关联分析的标准[36].
最终,鉴定的snp和单倍型HvNAM基因可以为GPC开发有用的分子标记。在此,关联分析可能提供一些分子标记HvNAM对麦芽大麦育种早期选择具有潜在重要性的基因。
更重要的是,这将有助于揭示栽培大麦和野生大麦GPC基因型差异的分子机制。此外,这些标记的确切染色体区域对于研究人员理解GPC的遗传学将是有趣的,因为这些区域中的大多数还没有根据它们的功能进行注释。然而,关联作图仅为所识别基因的功能提供了统计和间接证据,因此我们将在未来的研究中针对GPC和麦芽品质的潜在分子机制提供一些直接证据。
结论
本研究结果表明,蛋白质含量与麦芽品质参数之间存在密切的相关性,表明开发具有稳定GPC的大麦品种势在必行。本研究鉴定的大麦GPC标记将为GPC合理的大麦种质资源的评价和筛选提供参考。的单倍型HvNAM1而且HvNAM2与大麦GPC相关的SNP和DArT标记可为小麦品质性状的选择提供关键分子标记。此外,GWAS对寻找候选基因非常有用,可能为鉴定影响大麦GPC的不同位点提供有力的工具。
参考文献
- 1.
参见DK, Kephart V, Blake K:大麦籽粒蛋白质浓度变异的基因图谱。作物科学,2002,42:680-685。10.2135 / cropsci2002.0680。
- 2.
杨晓明,李文杰,李志强,等:大麦品种淀粉酶qtl的比较分析。北美大麦基因组计划。作物科学,2003,43:1043-1052。10.2135 / cropsci2003.1043。
- 3.
Ullrich SE:大麦饲料品质属性的遗传育种。大麦科学:从分子生物学到产量和品质农学的最新进展。编辑:Slafer GA, Molina-Cano JL, Savin R, Araus JL, Romagosa I.食品出版社:食品出版社;2002: 115 - 142。
- 4.
Emebiria LC, Moodya DB, Horsleyb R, Panozzoa J, Read BJ:澳洲和北美双排大麦杂交双单倍体群体籽粒蛋白质含量变异的遗传控制。粮食科学,2005,41:107-114。10.1016 / j.jcs.2004.08.012。
- 5.
杨晓明,李志强,李志强,等。玉米抗北叶枯病抗性的研究进展。美国国家科学研究院。2010, 38(4): 457 - 457。
- 6.
Hu VW, Addington A, Hyman A:通过对已发表的GWAS数据进行数量性状和亚表型关联分析,揭示了新的自闭症亚型依赖遗传变异。公共科学学报,2011,6 (4):e19067。10.1371 / journal.pone.0019067。
- 7.
Beattie AD, Edney MJ, Scoles GJ, Rossnagel BG:加拿大西部两行大麦合作试验麦芽质量数据的关联映射。作物科学,2010,50:1649-1663。10.2135 / cropsci2009.06.0334。
- 8.
Pasam RK, Sharma R, Malosetti M, Van Eeuwijk FA, Haseneyer G, Kilian B, Graner A:全球春大麦农艺性状的全基因组关联研究。中国生物医学工程学报,2012,29(3):329。10.1186 / 1471-2229-12-16。
- 9.
Stracke S, Haseneyer G, Veyrieras JB, Geiger HH, Sauer S, Graner A, Piepho HP:关联作图揭示了大麦开花时间决定中的基因作用和相互作用。理论与应用,2009,18(1):59- 61。10.1007 / s00122 - 008 - 0896 - y。
- 10.
Glémin S, Bataillon TA:驯化下草类进化的比较观点。环境科学学报,2009,29(3):344 - 344。10.1111 / j.1469-8137.2009.02884.x。
- 11.
Nevo E:野生大麦的起源、进化、群体遗传学和育种资源,大麦spontaneum在新月沃土。大麦:遗传学。编辑:Shewry PR.分子生物学和生物技术,CAB国际,沃灵福德:生物化学;1992: 19-43。
- 12.
金晓亮,蔡光胜,韩艳,王军,魏凯,张桂平:水稻遗传变异HvGlb1西藏一年生野生大麦和栽培大麦的差异及其与麦芽品质的相关性。粮食科学,2011,53:59-64。10.1016 / j.jcs.2010.09.006。
- 13.
邱林,吴德泽,阿里生,蔡国胜,戴峰,金晓亮,吴方波,张广平:盐渍化油菜耐盐性评价及等位基因功能分析HvHKT1而且HvHKT2在西藏野生大麦中应用物理学报,2011,32(3):366 - 366。10.1007 / s00122 - 010 - 1479 - 2。
- 14.
Uauy C, Distelfeld A, Fahima T, Blechl A, Ubcovsky J:一个调控衰老的NAC基因可以提高小麦籽粒蛋白质、锌和铁含量。科学通报,2006,29(4):344 - 344。10.1126 / science.1133649。
- 15.
Jamar C, Loffet F, Frettinger P, Ramsay L, Fauconnier M-L, Jardin P:康南1号Hordeum基因多态性与籽粒蛋白质含量的关系。植物营养学报,2010,27(3):357 - 357。10.1016 / j.jplph.2009.10.014。
- 16.
Distelfeld A, Korol A, Dubcovsky J, Uauy C, Blake T, Fahima T:大麦染色体臂6HS粒蛋白含量QTL与小麦GPC - b1区共线性关系分子育种,2008,22:25-38。10.1007 / s11032 - 007 - 9153 - 3。
- 17.
凯氏定氮法:测定有机质中氮的一种新方法。化学学报,2003,22:366-382。
- 18.
吴晓燕,王晓燕,王晓燕,Röbbelen G:油菜基因组图谱分析(芸苔属植物显著l .)。一、种子硫代葡萄糖苷含量RFLP连锁图谱构建及qtl定位。应用理论,1995,90:194-204。
- 19.
温泽尔P, Carling K, Kudrna D, Jaccoud D, Huttner E, Kleinhofs A, Kilian A:大麦全基因组测序的多样性阵列技术(DArT)。美国国家科学研究院。2004, 30(4): 457 - 457。10.1073 / pnas.0401076101。
- 20.
万维网上的Primer3为普通用户和生物学家程序员。生物信息学方法与协议:分子生物学方法。编辑:Rozen S, Skaletsky HJ, Krawetz S, Misener S. Totowa: Humana Press;2000: 365 - 386。
- 21.
Thompson JD, Higgins DG, Gibson TJ: CLUSTAL W:通过序列加权、位置特定间隙惩罚和权重矩阵选择提高渐进式多序列对齐的灵敏度。核化学学报,1994,22:439 - 439。10.1093 / nar / 22.22.4673。
- 22.
Hubisz MJ, Falush D, Stephens M, Pritchard JK:利用样本群信息推断弱种群结构。中国生物医学工程学报,2009,29(3):344 - 344。10.1111 / j.1755-0998.2009.02591.x。
- 23.
李志强,李志强,李志强,等。基于多位点基因型数据的种群结构推断。遗传学报,2000,37(3):344 - 344。
- 24.
刘rh,孟JL: MapDraw:一个微软Excel宏,用于根据给定的遗传连锁数据绘制遗传连锁地图。Heraditas(北京)。2003, 25: 317-321。
- 25.
Knowler WC, Williams RC, Pettitt DJ, Steinberg AG: Gm3-5, 13,14和2型糖尿病:美国印第安人与遗传混合物的关系。王文杰,王文杰,1998,(3):1 - 4。
- 26.
沙贝尔TF, Haubold B, Mitchell-Olds T:拟南芥的遗传隔离:生物地理学与欧洲冰川后殖民。分子生物学学报,2000,9:2109-2118。10.1046 / j.1365 - 294 x.2000.01122.x。
- 27.
葛永昌,杜doit S,速度TP:基于重采样的微阵列多重测试。数据分析。社会统计调查操作检验。2003,12(1):1-77。
- 28.
陈ek, Rowe HC, Hansen BG, Kliebenstein DJ:代谢组的复杂遗传结构。公共科学学报,2010,6:e1001198。10.1371 / journal.pgen.1001198。
- 29.
Manolio TA, Collins FS, Cox NJ, Goldstein DB, Hindorff LA:寻找复杂疾病的缺失遗传性。自然科学进展,2009,29(4):344 - 344。10.1038 / nature08494。
- 30.
刘玉娟,Papasian CJ,刘金峰,Hamilton J,邓慧文:复制是验证全基因组关联发现的金标准吗?公共科学学报,2008,3:e4037。10.1371 / journal.pone.0004037。
- 31.
Jukanti AK, Fischer AM:大麦高粒蛋白含量基因座(大麦芽) 6号染色体与旗叶蛋白水解和氮再动员的增加有关。中国生物医学工程学报,2008,29(4):344 - 344。10.1111 / j.1399-3054.2007.01044.x。
- 32.
大麦种子蛋白及其对麦芽和酿造质量的影响。植物学报,1990,3:63-80。
- 33.
王建民,张广平,陈建新,丁绍荣,周泰天:大麦籽粒和麦芽品质受品种和环境影响的变异。中国农业科学,2003,2:699-705。
- 34.
金晓亮,魏坤,张广平:西藏野生大麦蛋白质分数含量数量性状位点的全基因组关联分析。生物技术通报,2012,34:159-165。10.1007 / s10529 - 011 - 0736 - z。
- 35.
Yan JB, Kandianis CB, Harjes CE, Bai L, Kim EH, Yang XH, Skinner DJ, Fu ZY, Mitchell S, Li Q, Fernandez MGS, Zaharieva M, Babu R, Fu Y, Palacios N, Li JS, Penna D, Brutnell T, Buckler ES, Warburton ML, Rocheford T:罕见遗传变异玉米crtRB1增加玉米籽粒β-胡萝卜素。王文杰,2010,34(3):332 -327。10.1038 / ng.551。
- 36.
王楠,钱伟,Suppanz D,魏丽娟,毛伯忠,龙勇,孟JL, Müller AE,荣昌:油菜花期变异(芸苔属植物显著L.)与FRIGIDA同源体的等位变异相关BnaA。FRI.a.实验学报,2011,62(15):5641-5658。10.1093 / jxb / err249。
确认
本研究得到国家自然科学基金项目(No.30800681, 31129005)、浙江省自然科学基金项目(Y3100044)、中央高校基本科研业务费项目(2011FZA6005和2012FZA6011)、浙江省钱江人才计划项目(No. 2011R10079)的资助。我们也感谢陈忠华博士(西悉尼大学)对稿件的意见和修改。
作者信息
从属关系
相应的作者
额外的信息
相互竞争的利益
作者宣称他们没有竞争利益。
作者的贡献
实验构思与设计:XJ。进行实验:XJ、SC、YH、XC、XJ。数据分析:XJ。贡献试剂/材料/分析工具:XJ, GZ。论文作者:XJ,广州。所有作者都阅读并批准了最终的手稿。
电子辅助材料
12870 _2012_1237_moesm1_esm.doc
附加文件1:图S1:基于1319个DArT标记的158个群体的连锁不平衡衰减。LD衰减方程为y =−0.01ln(x) + 0.091,遗传距离衰减为0.40 cM (r2= 0.1)。x轴表示遗传距离,y轴表示r2,等位基因频率相关性的平方,这是一对变量之间相关性的测量。(doc 80kb)
12870 _2012_1237_moesm2_esm.doc
附加文件2:表S1:数据似然概率的对数摘要(现况(D))用于大麦基因型全基因组关联研究(GWAS)的群体结构。注:Ln p(D),数据概率的自然对数。概率计算是在10次独立的10万次迭代中进行的,然后使用不允许混合和相关等位基因频率的模型进行10万次迭代。K值设置为1 ~ 10,共使用1319个DArT标记进行分析。(doc 32kb)
12870 _2012_1237_moesm3_esm.doc
附加资料3:图S2:基于1319个DArT标记检测的59个栽培大麦和99个西藏野生大麦种质资源遗传多样性的群体结构P1到P7代表7个亚种群。(doc 83kb)
12870 _2012_1237_moesm4_esm.doc
附加文件4:表S2: 158个大麦品种的种群子结构。注:C、W分别代表栽培大麦和西藏野生大麦。(doc 112kb)
12870 _2012_1237_moesm5_esm.doc
附加文件5:图S3。:的Manhattan plot of DArT markers used in association analysis. The DArT markers with unknown genetic location were excluded from the Manhattan plot. The P values were adjusted with permutation test using a step-down MinP procedure. 1H to 7H on the X-axis denoted the barley chromosomes from 1H to 7H, respectively. The Y-axis showed that the –Log10(P),两条虚线表示P值= 0.05和0.01。(doc 41 kb)
12870 _2012_1237_moesm6_esm.doc
附加文件6:图S4:多个序列对齐HvNAM-1不同单倍型的基因。序列对齐下的符号表示相同的残差(*),以及CLUSTALW (http://align.genome.jp/)的强保守(:)和弱保守(.)替换。属于外显子的核苷酸用灰色阴影表示。SNPs用红色标记。(doc 38kb)
12870 _2012_1237_moesm7_esm.doc
附加文件7:图S5:多个序列对齐HvNAM-2不同单倍型的基因。序列对齐下的符号表示相同的残差(*),以及CLUSTALW (http://align.genome.jp/)的强保守(:)和弱保守(.)替换。属于外显子的核苷酸用灰色阴影表示。SNPs用红色标记。(doc 40kb)
权利和权限
开放获取本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用归属许可协议(https://creativecommons.org/licenses/by/2.0),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。
关于本文
引用本文
蔡邵,余刚,陈X。et al。大麦籽粒蛋白质含量变化及其相关性分析。BMC植物生物学13,35(2013)。https://doi.org/10.1186/1471-2229-13-35
收到了:
接受:
发表:
关键字
- 籽粒蛋白质含量
- 麦芽质量
- 分子多态性
- 西藏野大麦(大麦spontaneuml .)
- 关联映射